Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Quantitative Fluoreszenz In Situ Hybridisierung (FISH) und Immunfluoreszenz (IF) spezifischer Genprodukte in KSHV-infizierten Zellen
In diesem Artikel
Zusammenfassung
Wir beschreiben ein Protokoll, das fluoreszenz-in-situ-Hybridisierung (FISH) verwendet, um mehrere herpesvirale RNAs in lytisch infizierten menschlichen Zellen zu visualisieren, entweder in Suspension oder anhaftend. Dieses Protokoll enthält die Quantifizierung der Fluoreszenz, die ein nukleozytoplasmatisches Verhältnis erzeugt, und kann für die gleichzeitige Visualisierung von Wirts- und viralen Proteinen mit Immunfluoreszenz (IF) erweitert werden.
Zusammenfassung
Mechanistische Erkenntnisse stammen aus einer sorgfältigen Untersuchung und Quantifizierung bestimmter RNAs und Proteine. Die relativen Positionen dieser Biomoleküle in der Zelle zu bestimmten Zeiten können mit Fluoreszenz-In-situ-Hybridisierung (FISH) und Immunfluoreszenz (IF) erfasst werden. Während einer lytischen Herpesvirus-Infektion entführt das Virus die Wirtszelle, um bevorzugt virale Gene auszudrücken, was zu Veränderungen in der Zellmorphologie und dem Verhalten von Biomolekülen führt. Lytische Aktivitäten konzentrieren sich auf Atomfabriken, die als Viralreplikationsfächer bezeichnet werden und nur mit FISH und IF erkennbar sind. Hier beschreiben wir ein anpassungsfähiges Protokoll von RNA FISH- und IF-Techniken für Kaposi-Sarkom-assoziierte Herpesvirus-infizierte Zellen, sowohl anhaftend als auch in Suspension. Die Methode umfasst Schritte zur Entwicklung spezifischer Anti-Sense-Oligonukleotide, Doppel-RNA-FISH, RNA-FISH mit IF und quantitative Berechnungen von Fluoreszenzintensitäten. Dieses Protokoll wurde erfolgreich auf mehrere Zelltypen, nicht infizierte Zellen, latente Zellen, lytische Zellen, Zeitläufe und Zellen angewendet, die mit Inhibitoren behandelt wurden, um die räumlich-zeitlichen Aktivitäten bestimmter RNAs und Proteine sowohl vom menschlichen Wirt als auch von Proteinen aus dem menschlichen Wirt und KSHV.
Einleitung
In ihrer lytischen (aktiven) Phase kapern Herpesviren die Wirtszelle, was zu Veränderungen in der Zellmorphologie und Lokalisierung biologischer Moleküle führt, um Virionen zu erzeugen. Die Operationsbasis ist der Kern, in dem das doppelsträngige DNA-Virusgenom repliziert undin eine Proteinhülle, Capsid 1, verpackt wird. Zu Beginn drückt das Virus seine eigenen Proteine aus, entführt Wirtsmaschinen und verhindert die Expression von nicht-essentiellen Wirtsgenen, ein Prozess, der als Wirtsabschalteffekt bezeichnet wird. Der Großteil dieser Aktivität ist lokalisiert auf spezifische 4-,6-Diamidino-2-Phenylindole (DAPI)-freie Kernregionen, die als Viralreplikationskompartimenten bezeichnet werden und aus Wirts- und Viralproteinen, RNAs und viraler DNA2bestehen. Die Zelle wird überholt, um Platz und Ressourcen für die Replikationsräume und damit die Montage von viralen Kapsiden bereitzustellen. Sobald das Capsid den Kern verlässt, ist unklar, wie das Capsid in das Zytoplasma eingehüllt wird, um ein membrangebundenes virales Teilchen zu produzieren, das auch als Virion bekannt ist. Das Verständnis der Lokalisierung und räumlichen Verschiebungen von Wirts- und viralen Biomolekülen während der lytischen Phase bietet tiefere mechanistische Einblicke in die Anordnung des Replikationsfachs, den Wirtsabschalteffekt, den Virion-Egress-Pfad und andere Prozesse im Zusammenhang mit herpesviralen Infektionen und Replikationen.
Derzeit ist die beste Methode, um diese Veränderungen zu erkennen und zu untersuchen, die Visualisierung von Proteinen und RNAs in infizierten Zellen mit Immunfluoreszenz (IF) bzw. fluoreszierender In-situ-Hybridisierung (FISH). Die Verwendung eines Zeitverlaufs mit diesen Techniken zeigt die Lokalisierung von Biomolekülen an zentralen Punkten der lytischen Phase oder einfach, raumzeitliche Daten. FISH und IF ergänzen andere biochemische Techniken, wie z. B. Hemmung eines zellulären Prozesses (z. B. Hemmung der viralen DNA-Replikation), RT-qPCR (Echtzeit-Polymerase-Kettenreaktion), RNA-Sequenzierung, Nordflecken, Massenspektrometrie, Western Blotting und Analyse der viralen DNA-Produktion, die ein globaleres Bild der zellulären Aktivitäten liefern kann.
Wir entwickelten RNA-FISH-Strategien, um die RNA-Produkte aus bestimmten Genen zu untersuchen, und eine Rechneranalyse, die das nukleozytoplasmatische Verhältnis eines bestimmten Genprodukts quantitativ berechnet. Die Probenvorbereitung, modifiziert aus früheren Veröffentlichungen von Steitz und Kollegen3,4, ist relativ einfach und kann sowohl für Haftzellen als auch für suspendierte Zellen verwendet werden. Das Protokoll ist auch für den gleichzeitigen Einsatz mehrerer RNA FISH-Strategien (Double RNA FISH) oder RNA FISH mit IF-Strategien anpassungsfähig. Die Entwicklung einer spezifischen FISH-Strategie ist eine Herausforderung, aber es werden Vorschläge zur Verbesserung des Erfolgs skizziert. Die hier beschriebene Datenanalyse ist quantitativ, wenn fluoreszierende Perlen und starke Marker von Fachgrenzen verwendet werden und bietet zusätzliche Einblicke in die Mikrographen, Erkenntnisse, die Beobachtungsverzerrungen entfernen. Das detaillierte Protokoll ist sowohl für latente als auch für lytische Zellen konzipiert, die mit dem Mit dem Kaposi-Sarkat-assoziierten Herpesvirus (KSHV) infiziert sind, und kann mit nicht infizierten Zellen oder Zellen verwendet werden, die mit anderen Herpesviren infiziert sind5. Die Methoden der Quantifizierung sind auf Studien über nukleozytoplasmatische Verschiebungen oder Relokalisierung zwischen subzellulären Kompartimenten in den meisten Zellen anwendbar.
Protokoll
1. Entwicklung von Fluoreszenz in situ (FISH) Anti-Sense-Oligonukleotide zum Nachweis einer spezifischen herpesviralen Transkription
- Wählen Sie 25 bis 40 nt Segmente aus der Sequenz der RNA von Interesse und konvertieren Sie, um Anti-Sinn zu sein. Eine erfolgreiche FISH-Strategie kann von einem bis zu zehn oder mehr verschiedenen Anti-Sense-Oligonukleotiden enthalten. Berücksichtigen Sie bei der Auswahl von Sequenzen Folgendes:
- Wenn die RNA von Interesse einen einzigartigen Wiederholungsbereich enthält, dann nutzen Sie dieses Feature und entwerfen Sie ein Anti-Sense-Oligonukleotid, um die Wiederholungssequenz zu zielen.
HINWEIS: Tycowski und Kollegen5 liefern ein Beispiel für diese Strategie mit Rhesus rhadinovirus (RRV) polyadenylierte Kernkraft (PAN) RNA. - Wenn die RNA von Interesse eine bekannte Protein-Bindungsstelle oder Stammschleifenstruktur enthält, entwerfen Sie Oligonukleotide, die diese Regionen vermeiden.
- Je nach den Zielen der Experimente sollten Sie die intronische Reihenfolge berücksichtigen und prüfen, ob ein Anti-Sense-Oligonukleotid dafür entwirft wird.
- Wenn die RNA von Interesse einen einzigartigen Wiederholungsbereich enthält, dann nutzen Sie dieses Feature und entwerfen Sie ein Anti-Sense-Oligonukleotid, um die Wiederholungssequenz zu zielen.
- Führen Sie einfache Rechenanalysen an den ausgewählten Anti-Sense-Sequenzen durch, um die Bindungsspezifität zu gewährleisten und die Aggregation des Anti-Sense-Oligonukleotids zu reduzieren.
- Die Sequenzen müssen ca. 50% GC-reich (hoher Guanin- und Cytosingehalt) sein und eine Schmelztemperatur im Bereich von 60 bis 70 °C aufweisen.
- Verwenden Sie ein Sequenzanalysewerkzeug, um Sequenzen auszuwählen, die sich nicht selbst dimerisieren oder Haarnadeln mit Schmelztemperaturen über 37 °C bilden, die Hybridisierungstemperatur.
- Führen Sie eine NCBI BLASTn (National Center for Biotechnology Information Basic Local Alignment Search Tool for Nukleotide Alignments) durch, um die ausgewählten Sequenzen sowohl mit dem Host als auch mit viralen Transkriptomen unter Verwendung der Einstellung "etwas ähnlich" zu durchsuchen. Diese Suche identifiziert einzigartige Anti-Sense-Oligonukleotide, die wahrscheinlich nicht an andere Host- oder virale Transkripte binden.
HINWEIS: Wenn Transkriptome nicht verfügbar sind, führen Sie die BLAST-Suche mit den genomischen Sequenzen durch. Es ist ideal, wenn die Suchvorgänge auf die Sequenzen aus dem Virus durchgeführt werden, das von den infizierten Zellen isoliert wird, die im Experiment verwendet werden, da wilde Stämme dazu neigen, zu diversifizieren und eine Kombination von Sequenzen aus verschiedenen Laborstämmen enthalten.
- Order gereinigte DNA-Oligonukleotide entsprechend der Anti-Sinn-Sequenz, die rechnerisch verifiziert wurde, um eindeutig zu sein und wahrscheinlich an die Ziel-RNA zu binden. Es müssen keine besonderen Modifikationen in die Oligonukleotide eingeführt werden.
- Testen Sie die entworfenen Anti-Sense-Oligonukleotide auf Bindungsspezifität durch FISH und Northern Blot.
- Unter Verwendung einer nicht infizierten Zelllinie von der gleichen Wirtsart (z.B. 293T) und idealerweise desselben Zelltyps führen Sie eine Transfektion mit einem Plasmid durch, das die RNA von Interesse von einem robusten Promotor (CMV, Cytomegalievirus) und einer mit dem leeren Vektor (z. B. pcDNA3) ausdrückt. Verwenden Sie eine Positivkontrolle für die Transfektion, wie z. B. Die Kotransfektion mit einem GFP-Plasmid (grünefluoreszierende Proteine) (z. B. pmaxGFP) oder dem GfP-Vektor.
HINWEIS: Es ist wichtig, Zelllinien zu vermeiden, die mit dem Herpesvirus Epstein-Barr Virus (EBV) verewigt wurden, da es Sequenzähnlichkeiten zwischen Herpesviren gibt. - Führen Sie FISH wie in Abschnitt 3 beschrieben auf beiden Gruppen von Zellen mit den Anti-Sense-Oligonukleotiden, wie in diesem Protokoll beschrieben. Schließen Sie die erfolgreichen Kandidaten ab, indem Sie FISH-Experimente mit einzelnen, Paaren oder Sätzen der Anti-Sense-Oligonukleotide vergleichen. Verwenden Sie eine positive Kontrolle für das FISH-Protokoll wie U2 snRNA (kleine kernnukleare RNA) FISH, vorhanden bei 500.000 Kopien pro menschlichem Zellkern6 (Tabelle 1).
- Das fluoreszierende Signal sollte spezifisch und stark in der Zelle sein, die die RNA von Interesse enthält. Entwerfen Sie zusätzliche Anti-Sense-Oligonukleotide, um das Signal zu stärken und Anti-Sense-Oligonukleotide zu entfernen, die nicht spezifisch von der Betrachtung binden. Die Signalstärke muss über dem Hintergrund und der Autofluoreszenz liegen.
- Testen Sie die Bindungsspezifität durch Northern Blot.
- Unter Verwendung einer nicht infizierten Zelllinie von der gleichen Wirtsart (z.B. 293T) und idealerweise desselben Zelltyps führen Sie eine Transfektion mit einem Plasmid durch, das die RNA von Interesse von einem robusten Promotor (CMV, Cytomegalievirus) und einer mit dem leeren Vektor (z. B. pcDNA3) ausdrückt. Verwenden Sie eine Positivkontrolle für die Transfektion, wie z. B. Die Kotransfektion mit einem GFP-Plasmid (grünefluoreszierende Proteine) (z. B. pmaxGFP) oder dem GfP-Vektor.
2. Oligonukleotid und Zellzubereitung
- Verwenden Sie nach den Anweisungen des Herstellers die Anti-Sense-Oligonukleotide mit Dioxigenin(DIG)-dUTP oder, wenn sie stark bindend sind, direkt mit einem fluoreszierenden Nukleotid wie Alexa Fluor 594-5-dUTP. Nach der Etikettierung ist keine zusätzliche Reinigung erforderlich. Beschriftete Oligonukleotide bei -20 °C bis zu mehreren Jahren und in Zinnfolie lagern, wenn sie direkt beschriftet sind, um Photobleichmittel zu verhindern.
VORSICHT: Die Etikettierlösung enthält ein giftiges Material, Kaliumkokodylat. Behandeln Sie Etikettierreaktionen mit Handschuhen.
HINWEIS: Um Ressourcen zu schonen, können mehrere verschiedene Anti-Sense-Oligonukleotide in einer Reaktion gekennzeichnet werden. Dieses Protokoll verwendet 3'-End-Kennzeichnung. Die interne Kennzeichnung ist eine Herausforderung, da die chemische Gruppe (z. B. DIG oder das Fluorophor) gefangen wird oder nicht in die aktive Stelle einer DNA-Polymerase gelangen kann. Ein Experiment mit zwei verschiedenen RNAs kann mit direkt gekennzeichneten Anti-Sense-Oligonukleotiden und Anti-DIG-Immunfluoreszenz mit einem anderen Fluorophor (z. B. FITC (Fluorescein) oder Alexa Fluor 488 mit Alexa Fluor 594 durchgeführt werden. - Befestigen Sie die Zellen an den Achtkammer-Dias.
HINWEIS: Achtkammer-Dias ermöglichen mehrere gleichzeitige Experimente bei gleichzeitiger Minimierung wertvoller Ressourcen wie Antikörper. Eine Alternative zu Achtkammer-Dias ist eine sechswellige Gewebekulturplatte mit Standard-Abdeckungen (22 mm x 22 mm), die beide steril sind. Eine ähnliche Anordnung ist mit kreisförmigen Abdeckungen und einer 24-Well-Gewebekulturplatte möglich. Erhöhen Sie für beide die in diesem Protokoll genannten Volumina um das 10-15-fache (z. B. 1,75 ml Hybridisierungslösung) bzw. die 4x (z. B. 600 L Hybridisierungslösung).- Für anhaftende lytische Zellen 1x Trypsin/PBS bei 37 °C und 5% CO2 für 10 min verwenden, um Zellen auszusetzen und auf 60% Konfluenz zu verdünnen.
HINWEIS: In FISH-Experimenten verwendete Haftzellen umfassten 293T, iSLK.2197und iSLK-BAC36-Zellen8. - Tragen Sie 200 l Zellsuspension auf jede Kammer der sterilen Achtkammerschlitten auf und lassen Sie das Saatgutwachstumfür 12-24 h bei 37 °C und 5% CO2 zu. Passen Sie bei Bedarf für langsam e0er oder schnell wachsende Zellen und für Zellen, die leicht durch Trypsin beschädigt werden.
HINWEIS: Das Ziel besteht darin, gleichmäßig verteilte Zellen fest an der Folie befestigt zu haben. Erwägen Sie, eine lytische Phase nach der Adhäsion zu induzieren, wenn die lytischen Zellen zerbrechlich sind. Die Schlussfolgerungen aus Experimenten mit iSLK-Zellen sind begrenzt9. - Für lytische Suspensionszellen achtkammerige Dias mit 1:10 Poly-L-Lysin für 5 min unter der Gewebekulturhaube vorbehandeln. Dann lassen Sie die Rutschen über Nacht bei Raumtemperatur oder 1 h bei 65 °C trocknen. Inkubieren Sie 800 l lytische Zellen in einer Konzentration von 1 x 106 Zellen/ml mit den kammergefilterten Dias für 30 min bis 1 h bei 37 °C und 5% CO2 .
HINWEIS: Suspensionszellen setzen sich in einer Monoschicht ab, kleben an dem Poly-L-Lysin und daher sind überschüssige Zellen im Vergleich zu anhaftenden Zellen kein Problem. Lytische Zellen, die Traubenhaufen bilden, sollten, wenn möglich, durch sanfteWirbel oder chemische Mittel getrennt werden. Zwar hatten die Autoren bei lytischen BJAB-RRV-GFP-Zellen wenig Erfolg mit solchen Empfehlungen. Wenn Suspensionszellen nicht gut haften, sollten Sie entweder die Zeit oder Konzentration der Poly-L-Lysin-Inkubation erhöhen.
- Für anhaftende lytische Zellen 1x Trypsin/PBS bei 37 °C und 5% CO2 für 10 min verwenden, um Zellen auszusetzen und auf 60% Konfluenz zu verdünnen.
3. Fixierung, Immunfluoreszenz (optional), Hybridisierung und Visualisierung viraler RNAs
- Entfernen Sie Medien und überschüssige Zellen. Verwenden Sie während dieses Protokolls Vakuumabsaugung, um Lösungen und sanftes Mikropipetting zu entfernen, um Lösungen hinzuzufügen.
HINWEIS: Die Festigkeit eines Vakuums kann reduziert werden, indem eine 200-L-Mikropipette-Spitze über die glasgläserne Pasteurpipette platziert wird. Ersetzen Sie die Mikropipettespitze zwischen den Waschstufen, um eine Kontamination zu verhindern. Jeder Waschschritt muss schnell durchgeführt werden, da es zwingend notwendig ist, dass die Zellen nie austrocknen. - Fixieren Sie die Zellen mit vorgekühlten 4% Formaldehyd/PBS (Phosphat-gepufferte Saline) 30 min auf Eis. Waschen Sie die Zellen dreimal mit 200 l 1x PBS auf 4 °C abgekühlt und 5 min bei Raumtemperatur oder auf Eis inkubieren.
- Permeabilisieren Sie die festen Zellen mit 200 l vorgekühlten 0,5% Triton-X/PBS (Phosphat gepufferte Saline) für 10 min auf Eis oder 750 l vorgekühltem 70% Ethanol bei 4 °C für 1 h (min) bis 7 d (max).
HINWEIS: Sammeln Sie Protein-, RNA-Gesamt- und genomische DNA-Proben am Fixierungspunkt, um die Konsistenz zwischen Bildern und biochemischen Assays zu gewährleisten. Alle Wässerungen in diesem Protokoll werden auf die gleiche Weise ausgeführt, sofern nicht anders angegeben. 70% Ethanol lockert den Kleber zwischen den Kammern und dem Schlitten, was die spätere Trennung erleichtert und auch eine signifikante Pause im Protokoll ermöglicht. Verwenden Sie jedoch Paraffinfolie um die Kammerrutsche, um die Verdunstung zu reduzieren und den Gehalt des Ethanols in jeder Kammer etwa alle 8 h zu überprüfen. 70% Ethanol glättet auch die Zellen, wodurch ein knackigeres Bild entsteht, während Triton-X die Zellen nicht dehydriert und die Abmessungen der Zelle.
- Permeabilisieren Sie die festen Zellen mit 200 l vorgekühlten 0,5% Triton-X/PBS (Phosphat gepufferte Saline) für 10 min auf Eis oder 750 l vorgekühltem 70% Ethanol bei 4 °C für 1 h (min) bis 7 d (max).
- Entfernen Sie die Kammern vorsichtig, um risse am Schlitten zu verhindern. Wenn das Experiment die Immunfluoreszenz (IF) eines viralen oder Wirtsproteins mit einem polyklonalen Primärantikörper enthält, führen Sie das IF wie unten beschrieben durch, bevor Sie mit RNA FISH fortfahren. Wenn die Immunfluoreszenz einen monoklonalen Primärantikörper verwendet, führen Sie die Immunfluoreszenz gemäß Schritt 3.3.1 nach Schritt 3.11 durch.
HINWEIS: Verwenden Sie eine frische Entnahmevorrichtung oder eine mit sehr wenig Restkleber vom Hersteller zur Verfügung gestellt und sanft die Kammern zu erleichtern, um zu verhindern, dass der Schlitten risse. Die Verwendung von 70% Ethanol als permeabilisierendes Reagenz für 4 h reduziert die Wahrscheinlichkeit von Rissen erheblich. Im Falle eines Risses, setzen Sie das Protokoll über Kammern, die nicht durch den Riss betroffen sind, fort und achten Sie auf die höhere Oxidationsrate von unvollkommen versiegelten Dias (d. h. verminderte Lagerdauer).- Zellen mit vorgekühlten 1x PBS spülen und mit vorgekühlten 4% BSA (Rinderserumalbumin)/1x PBS für 30 min bei 4 °C blockieren.
HINWEIS: Die Verwendung von BSA in diesem Protokoll schränkt die unspezifische Kennzeichnung ein. - Entfernen Sie die Blockierlösung und inkubieren Sie die Zellen mit 1:200 oder einem anderen polyklonalen Primärantikörper in 0,1% BSA/1x PBS für 1 h bei 4 °C. Dann dreimal mit 1x PBS waschen.
HINWEIS: Bei 1:200 Verdünnung wurde ein Antikörper10 zum Nachweis von SSB/ORF6 (virales einsträngiges DNA-Bindungsprotein) verwendet. - Inkubieren Sie die Zellen mit einem sekundären Antikörper mit Fluorophor, der mit dem FISH-detektierenden Antikörper für 1 h bei 4 °C kompatibel ist. Dreimal mit 1x PBS waschen. Dann fixieren Sie mit 4% Formaldehyd/1x PBS für 10-15 min und permeabilisieren sie entweder mit Triton-X oder 70% Ethanol, wie zuvor beschrieben, bevor Sie zu FISH gehen. Abdeckung Sauge mit Zinnfolie, um fluoreszierendes Signal zu erhalten und Photobleichmittel zu verhindern.
- Zellen mit vorgekühlten 1x PBS spülen und mit vorgekühlten 4% BSA (Rinderserumalbumin)/1x PBS für 30 min bei 4 °C blockieren.
- Waschen Sie die Zellen einmal mit 2x SSC (Salin-Natriumcitrat) und wenden Sie dann 45 l Hybridisierungslösung an, die aus 50% Formamid, 10% Dextransulfat, 2x SSC, 0,1% BSA, 500 g/ml Lachssperma-DNA, 125 g/ml E. coli tRNA und 1 mM Vanadylribonucleosid Komplexe. Inkubieren Sie für 1 h bei 37 °C in einer Feuchtigkeitskammer, die eine 150 mm Petrischale mit befeuchteten sterilen Tüchern sein kann.
HINWEIS: Bereiten Sie eine neue Hybridisierungslösung mindestens eine Stunde vor Gebrauch vor. Lösen Sie das Dextransulfat zuerst in Wasser, wirbeln häufig und in einem 37 °C-Wasserbad. - Berechnen Sie eine empfohlene Konzentration von 25 -M Oligonukleotiden in 35-uL-Hybridisierungslösung pro Kammer. Stellen Sie die Konzentration von Anti-Sense-Oligonukleotid nach Bedarf ein. Fügen Sie destilliertes Wasser zu den Oligonukleotiden hinzu, um das Denaturierungsvolumen auf 10 l zu erhöhen.
HINWEIS: Nach der Etikettierungsreaktion werden die Oligonukleotide in der abgeschreckten Lösung mit 0,18 M Kaliumcacodylat, 23 mM Tris-HCl, 0,23 mg/mL BSA, 4,5 mM CoCl2, 18 mM EDTA, 2,7 mM K-Phosphat und 6,8 mM KCl, 45 mM 2-Mercaptoethanol, 0,02% Triton X-100 und 2% Glycerin. Die Konzentrationen sind hoch genug, dass die Verdünnung mit Wasser die Denaturierungslösung in Konzentrationen nahe 1x TE (10 mM Tris-HCl und 1 mM EDTA), einem Standard-Oligonukleotid-Denaturierungspuffer, bringt. - Denaturieren Sie die DIG- und/oder Alexa Fluor 594-markierten Oligonukleotide bei 95 °C für 5 min. Dann fügen Sie die denaturierte Oligonukleotide pro vorgesehener Kammer mit einer frischen Hybridisierungslösung von 35 l ein. Bei doppelter FISH-Ausführung können beide Sätze von Anti-Sense-Oligonukleotiden denaturiert und hybridisiert werden.
- Entfernen Sie die Vorhybridisierungslösung, und fügen Sie dann den Zellen eine Hybridisierungslösung hinzu, die die markierten Oligonukleotide enthält. Über Nacht in der Feuchtigkeitskammer bei 37 °C mit Zinnfolie inkubieren, um die fluorophor-markierten Oligonukleotide zu schützen.
HINWEIS: Die Inkubation sollte mindestens 10 h und nicht mehr als 24 h betragen. - Am nächsten Tag die Zellen zweimal mit 2x SSC für 10 min bei 37 °C und dann zweimal mit 1x SSC für 10 min bei 25 °C waschen.
- Fixieren Sie die Zellen mit vorgekühlten 4% Formaldehyd/1x PBS für 10-15 min auf Eis. Dann waschen Sie die Zellen mit PBS dreimal und permeabilisieren für 1 h mit vorgekühltem 70% Ethanol oder für 10 min mit vorgekühlten 0,5% Triton-X/1x PBS bei 4 °C.
- Inkubieren Sie die Zellen mit 1:200 Anti-DIG FITC in vorgekühlten 0,1% BSA/1x PBS für 1 h bei 4 °C. Entfernen Sie die Antikörperlösung und waschen Sie sie dreimal mit 1x PBS.
- Mit vorgekühlten 4% Formaldehyd/1x PBS für 10-15 min bei 4 °C fixieren und dann dreimal mit 1x PBS waschen. Wenn Sie die Immunfluoreszenz für ein Wirt oder virales Protein mit einem monoklonalen Primärantikörper durchführen, permeabilisieren Sie die Zellen und führen Sie dann das in Schritt 3.3.1 beschriebene IF-Protokoll durch. Fahren Sie andernfalls mit der DAPI-Färbung fort.
- Inkubieren Sie die Zellen mit 0,4 g/ml DAPI in vorgekühlten 0,5% Triton-X/1x PBS für 15 min auf Eis und waschen Sie dann dreimal mit 1x PBS.
- Montieren Sie Rutschen mit fluoreszierenden Perlen (optional) und einem Montagemedium. Dann versiegeln Sie den Deckelrutsch mit klarem Nagellack an die Rutsche.
- Sammeln Sie mit einem konfokalen Mikroskop innerhalb einer Stunde bis zu einer Woche Bilder der Proben, wenn Sie das Protokoll bei 630-facher Vergrößerung durchführen. Tragen Sie mehrere Schichten Nagellacke auf, um den Deckelschlupf zu versiegeln und die Lebensdauer des Fluorophors zu verlängern, indem Sie die Oxidationsrate reduzieren.
HINWEIS: Verwenden Sie kein DAPI-haltiges Montagemedium. Wenn Sie die Bilder sammeln, fügen Sie die Maßstabsleiste für die spätere Quantifizierung auf jedem Bild ein. Fluoreszierende Perlen dienen als Steuerung der Fluoreszenzintensität zwischen Dias und Probenpräparaten11. Erfassen Sie Bilder am Mittelteil der Zelle für die zweidimensionale (2D) Quantifizierung in Schritt 4.
4. Quantifizierung von FISH- und IF-Bildern zur Hervorhebung der subzellulären Lokalisation und zur Bestimmung des nukleozytoplasmatischen Fluoreszenzverhältnisses
- Führen Sie eine Bildanalyse auf einem zusammengebauten Stapel der verschiedenen fluoreszierend gefärbten und zusammengeführten Bilder durch, um konsistenzfähig zu sein. Legen Sie den Maßstab der Bildanalysesoftware mithilfe der Maßstabsleiste fest, die bei der Erfassung der Bilder enthalten war.
- Um die Fluoreszenzintensität über mehrere Kanäle und in Bezug auf den nuklearen DAPI-Fleck zu quantifizieren, verwenden Sie ein Linienwerkzeug und eine Plotprofilfunktion. Geben Sie dann die Zeile dauerhaft auf einer Kopie des Bildes mit Markern an, die das Urteil des Betrachters nicht behindern oder beeinflussen.
- Legen Sie Kriterien fest, um zu steuern, wo die Linie gezeichnet wird, z. B. eine Spur, die eine Vielzahl von topographischen Merkmalen, Gipfeln und Tälern entlang einer zentralen Achse oder einer Linie erfasst, die keine übersättigten Bereiche durchquert.
HINWEIS: Diese Linienspuren zeigen rohe Fluoreszenz in einer Zelle und beschränken sich daher auf Vergleiche der Positionen eines Flecks, nicht der Intensität. Um Die Intensitäten desselben Flecks zwischen Dias, Behandlungen oder Zubereitungen zu vergleichen, fügen Sie dem Dia in Schritt 3.13 eine fluoreszierende Perle als interne Kontrolle hinzu. Die leuchtstoffzierende Perle muss während des Montageprozesses zugegeben und mit den gleichen Einstellungen am Anregungslaser und Photomultiplierrohr (konfokad) erfasst werden.
- Legen Sie Kriterien fest, um zu steuern, wo die Linie gezeichnet wird, z. B. eine Spur, die eine Vielzahl von topographischen Merkmalen, Gipfeln und Tälern entlang einer zentralen Achse oder einer Linie erfasst, die keine übersättigten Bereiche durchquert.
- Um eine Verschiebung der subzellulären Lokalisation zu quantifizieren, berechnen Sie die nukleozytoplasmatischen Verhältnisse von Zellen, die verschiedenen Behandlungen unterzogen werden.
- Messen Sie die Fläche und die Rohefluoreszenzintensität sowohl des Kerns als auch des Zytoplasmas mithilfe des nuklearen DAPI-Flecks, um die innere Grenze festzulegen. Umfassen Sie kerntechnische und zytoplasmatische Kontrollen wie eine kernnukleare RNA (z. B. KSHV PAN RNA) und zytoplasmatische RNA (z. B. Host GAPDH mRNA). Berechnen Sie außerdem die Hintergrundintensität für drei zellähnliche Bereiche und durchschnittlich die Werte pro Pixel oder2.
HINWEIS: Intensitätswerte neigen dazu, Einheiten zu fehlen, und daher wird der Begriff "Einheiten" verwendet. - Normalisieren Sie sowohl nukleare als auch zelluläre Rohintensitätswerte, indem Sie zunächst den durchschnittlichen Hintergrund für denselben Bereich bestimmen und dann diesen individualisierten Wert von der Rohintensität des Bereichs subtrahieren.
- Zum Beispiel hat ein Kern einer lytischen B-Zelle eine Fläche von 133,4 m2 und eine Rohe intensität von 75976 Einheiten, während die Hintergrundintensität für dasselbe fluoreszierende Signal auf 0,67 Einheiten pro m2bestimmt wurde. Die normalisierte nukleare Intensität würde
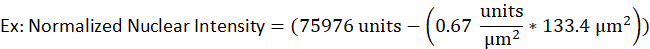
- Zum Beispiel hat ein Kern einer lytischen B-Zelle eine Fläche von 133,4 m2 und eine Rohe intensität von 75976 Einheiten, während die Hintergrundintensität für dasselbe fluoreszierende Signal auf 0,67 Einheiten pro m2bestimmt wurde. Die normalisierte nukleare Intensität würde
- Geben Sie die Werte in der folgenden Gleichung ein.

HINWEIS: Diese Berechnung steuert Änderungen im subzellulären Bereich. Lytische Induktion und medikamentöse Behandlungen können den Zellkern vergrößern bzw. die Größe der Zelle verändern. - Um die Ergebnisse zu interpretieren, erstellen Sie ein Box-Whisker-Plot. Eine gleichmäßige Verteilung des fluoreszierenden Signals wäre nahe Null, während eine kerntechnische Verteilung einen positiven Verhältniswert begünstigen würde und eine zytoplasmatische Verteilung zu einem negativen Verhältniswert tendieren würde.
- Messen Sie die Fläche und die Rohefluoreszenzintensität sowohl des Kerns als auch des Zytoplasmas mithilfe des nuklearen DAPI-Flecks, um die innere Grenze festzulegen. Umfassen Sie kerntechnische und zytoplasmatische Kontrollen wie eine kernnukleare RNA (z. B. KSHV PAN RNA) und zytoplasmatische RNA (z. B. Host GAPDH mRNA). Berechnen Sie außerdem die Hintergrundintensität für drei zellähnliche Bereiche und durchschnittlich die Werte pro Pixel oder2.
Ergebnisse
Die in diesem Manuskript beschriebenen FISH- und IF-Methoden sind in Abbildung 1 zusammen mit der Quantifizierung der Ergebnisse durch Linienspuren der fluoreszierenden Intensität dargestellt. Die hier vorgestellten Ergebnisse sind semiquantitativ und bieten Einblicke in die Lokalisation und nicht in Vergleiche zwischen Intensitäten verschiedener fluoreszierender Flecken, da Experimente keine fluoreszierende Perle in die Diavorbereitung einbeziehen.
Diskussion
Das in diesem Bericht beschriebene Protokoll kann an verschiedene Zelltypen angepasst werden und enthält Schritte für doppel-RNA-FISH und RNA-FISH mit IF, wobei sowohl monoklonale als auch polyklonale Primärantikörper verwendet werden. Obwohl vorbereitete Dias in der Regel mit einem konfokalen Mikroskop abgebildet werden, kann die Bildgebung mit einem STED-Mikroskop (stimulierte Emissionserschöpfung) nach Modifikationen der erhöhten Antikörperkonzentration und einem anderen Montagemedium durchgeführt werden. Zur ...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken Jonathan Rodenfels, Kazimierz Tycowski und Johanna B. Withers für die Beratung zur Datenanalyse. Wir danken auch G. Hayward für den Anti-SSB-Antikörper. Diese Arbeit wurde durch die Stipendien T32GM007223 und T32AI055403 von den National Institutes of Health (an TKV) und NIH Grant (CA16038) (an JAS) unterstützt. JAS ist ein Forscher des Howard Hughes Medical Institute. Die Abbildungen 1-3 und Tabelle 1 wurden mit Genehmigung der American Society for Microbiology unter einer Creative Commons Attribution Lizenz der folgenden Publikation reproduziert: Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi es Sarcoma-Associated Herpesvirus mRNA Akkumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), doi:10.1128/JVI.00220-18, (2018).
Materialien
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
Referenzen
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81 (2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300 (2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten