Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Obtención de esferas de células madre de cáncer de cáncer de tumores ginecológicos y de cáncer de mama
* Estos autores han contribuido por igual
En este artículo
Resumen
El objetivo de esta metodología es identificar células madre cancerosas (CSC) en líneas celulares cancerosas y muestras de tumores humanos primarios con el protocolo de formación de esferas, de manera robusta, utilizando ensayos funcionales y caracterización fenotípica con citometría de flujo y occidental Blot.
Resumen
Las células madre cancerosas (CSC) son una pequeña población con auto-renovación y plasticidad que son responsables de la tumorigenesis, la resistencia al tratamiento y la enfermedad recurrente. Esta población se puede identificar por marcadores de superficie, actividad enzimática y un perfil funcional. Estos enfoques per se son limitados, debido a la heterogeneidad fenotípica y la plasticidad CSC. Aquí, actualizamos el protocolo de formación de esferas para obtener esferas CSC a partir de cánceres mamarios y ginecológicos, evaluando las propiedades funcionales, los marcadores CSC y la expresión de proteínas. Las esferas se obtienen con sembrado de una sola célula a baja densidad en cultivo de suspensión, utilizando un medio semisólido de metilcelulosa para evitar la migración y los agregados. Este protocolo rentable se puede utilizar en líneas celulares de cáncer, pero también en tumores primarios. El cultivo tridimensional de suspensión no adherente que se cree que imita el microambiente tumoral, particularmente el nicho CSC, se complementa con factor de crecimiento epidérmico y factor de crecimiento básico de fibroblastos para garantizar la señalización de CSC. Con el objetivo de una identificación sólida de la CSC, proponemos un enfoque complementario, que combine la evaluación funcional y fenotípica. La capacidad de formación de esferas, la autorenovación y el área de proyección de esferas establecen propiedades funcionales de CSC. Además, la caracterización comprende la evaluación de la citometría de flujo de los marcadores, representada por CD44+/CD24- y CD133, y Western blot, considerando ALDH. El protocolo presentado también fue optimizado para muestras de tumores primarios, después de un procedimiento de digestión de muestra, útil para la investigación traslacional.
Introducción
Las poblaciones de cáncer son heterogéneas, con células que presentan diferentes morfologías, proliferación y capacidad de invasión, debido a la expresión génica diferencial. Entre estas células, existe una población minoritaria denominada células madre cancerosas (CSC)1, que tienen la capacidad de auto-renovación, recapitulando la heterogeneidad del nicho tumoral primario y produciendo progenitores aberrantes diferenciadores que no responden adecuadamente a los controles homeostáticos2. Las propiedades de CSC se pueden traducir directamente en la práctica clínica, dada la asociación con eventos, como la tumorogenicidad o la resistencia a la quimioterapia3. La identificación de la CSC puede conducir al desarrollo de terapias dirigidas que pueden incluir bloqueo de marcadores superficiales, promoción de la diferenciación de CSC, bloqueo de los componentes de la vía de señalización de CSC, destrucción de nichos y mecanismos epigenéticos4.
El aislamiento de CSC se ha realizado en líneas de células y en muestras de tumores primarios5,6,7,8. El perfil funcional descrito para CSC incluye la capacidad clonogénica, la población lateral y la formación de tumorosfera9. El fenotipobajo CD44alto/CD24 se ha asociado consistentemente con la CSC mamaria, que ha demostrado ser tumorigénico in vivo y ya se ha asociado con la transición epitelial a mesenquimal5,10. La alta actividad de ALDH también se ha asociado con la talloy y la transición epitelial a mesenquimal (EMT) en varios tipos de tumores sólidos11. La expresión ALDH se ha asociado con la resistencia a la quimioterapia y al fenotipo CSC in vitro12,13,14,15,16. Varios otros marcadores se han vinculado a propiedades CSC en diferentes tipos de tumores, como CD133, CD49f, ITGA6, CD1663,4 y otros, como se describe en la Tabla 1.
Las tumoresferas consisten en un modelo tridimensional para el estudio y la expansión de la CSC. En este modelo, las suspensiones celulares de las líneas celulares y de muestras de sangre o tumores se cultivan en un medio complementado con factores de crecimiento, a saber, factor de crecimiento epidérmico (EGF) y factor de crecimiento básico de fibroblastos (bFGF), sin suero bovino fetal y en condiciones no adherentes17. La inhibición de la adhesión celular resulta en la muerte por anoikis de células diferenciadas18. Las esferas se derivan del crecimiento clonal de una célula aislada. Para ello, las celdas se distribuyen a baja densidad para evitar la fusión de celdas y la agregación19. Otra estrategia incluye el uso de metilcelulosa semisólida20.
El protocolo de formación de esferas ganó popularidad en el aislamiento y expansión de CSC, debido al tiempo y costo y razones técnicas, rentables y reproducibles21,22. A pesar de algunas reservas sobre la extensión de la cual la formación de esferas refleja CSC, existe una propensión a que las células madre crezcan en condiciones no adherentes con el fenotipo característico, que se asemeja al microambiente nativo21. Ninguno de los métodos disponibles para el aislamiento de la CSC de los tumores sólidos tiene una eficiencia completa, lo que pone de relieve la importancia de desarrollar marcadores o combinaciones más específicas de metodologías y marcadores.
En este protocolo, detallamos el aislamiento de CSC con el protocolo de formación de esferas, con el principio de crecimiento de una sola célula en condiciones no adherentes y la capacidad de producir un fenotipo diferenciado. En la Figura 1se representa una representación esquemática de este procedimiento. También describimos la caracterización con marcadores de superficie y expresión ALDH para CSC, tanto para líneas de células tumorales mamarias como ginecológicas y muestras de tumores primarios.
Protocolo
Este protocolo se realizó cumpliendo con las directrices éticas del Banco de Tumores del Hospital y Centro Universitario de Coimbra (CHUC), y fue aprobado por el Comité de ética de salud de la CHUC y por la Comisión Nacional de Protección de Datos de Portugal.
1. Protocolo de formación de esferas y poblaciones adherentes derivadas de cultivos celulares continuos
NOTA: Realice todos los procedimientos bajo estrictas condiciones estériles.
- Preparación de matraces o placas de cultivo de suspensión no adherentes cubriendo la superficie de crecimiento con poli(2-hidroxietilo-metacrilato (poli-HEMA)
- Preparar una solución de 15 mg/ml agitando poly-HEMA en etanol absoluto a 65 oC. Recubrir matraces de cultivo celular con 50 l/cm2.
- Dejar secar a 37oC en un horno de secado. Si es necesario, envuelva los platos y guárdelos a temperatura ambiente.
- Preparación de los medios de cultivo de la esfera (SCM)
- Preparar una solución del 2% de metilcelulosa en agua ultrapura y esterilizar en el autoclave. La metilcelulosa tiende a ser más fácil de solubilizar por enfriamiento; por lo tanto, disperse el polvo en agua a 80 oC y revuelva hasta que se enfríe23.
- Preparar una solución dos veces concentrada de SCM (solución de stock). SCM solución de trabajo contiene DMEM-F12, complementado con 100 mM putrescine, 1% insulina, transferrina, selenio y 1% solución antibiótica-antimicótica (10.000 U/ml de penicilina, 10 mg/ml de estreptomicina y 25 g/ml de anfotericina B).
- Para preparar el SCM, mezcle volúmenes iguales de la solución de stock de SCM con la solución del 2% de metilcelulosa.
- Complete el medio inmediatamente antes de su uso añadiendo 10 ng/ml de factor de crecimiento epidérmico (EFG) y factor de crecimiento de fibroblastobásico básico de 10 ng/ml (bFGF).
- Si se utilizan líneas celulares más fastidiosas, complementar el medio con 0.4% albúmina sérica bovina, que podría ser una ventaja.
- Comience con un matraz de cáncer de mama MCF7 o HCC1806 o células cancerosas de endometrio ECC-1 o RL95-2 (u otra línea celular cancerosa de elección) con una confluencia del 80% al 90%.
- Deseche los medios de cultivo celular, lave con solución salina tamponada de fosfato (PBS) y separe las células con trippsina-EDTA (1 a 2 ml para un matraz de cultivo de75 cm 2 células).
- Añadir medios de cultivo celular (2 a 4 ml para un matraz de cultivo celular de 75 cm2) y centrífuga a 200 x g durante 5 min para desechar enzimas.
- Suspenda el pellet en un volumen conocido de medios de cultivo celular y pipetas hacia arriba y hacia abajo para asegurar una suspensión de una sola célula. Para ello, se puede utilizar un colador de células de 40 m.
- Cuente las células en el hemocitómetro y calcule la concentración celular de la suspensión celular. Aproveche este paso para asegurar la observación de una suspensión de una sola célula. El recuento cuidadoso de células es esencial para cuantificar con precisión los efectos de los tratamientos.
- Suspenda la cantidad determinada de suspensión celular en medio completo de SCM y transfiera a platos recubiertos con poli-HEMA. Como valor de referencia para la densidad de sembrado, considere entre 500 y 2000 celdas/cm2.
NOTA: Se recomienda encarecidamente la optimización de la densidad de sembrado y el tiempo de cultivo de cada línea celular24. - Incubar a 37oC y 5%CO2 durante 2 días sin perturbar las placas.
- Restablezca la concentración de factores de crecimiento añadiendo EFG de 10 ng/ml y bFGF de 10 ng/ml al medio de cultivo celular. Repita este paso cada dos días.
- Incubar a 37oC y 5%co2 hasta 5 días después del enchapado (esto puede variar de 3 a 12 días según la línea celular) para obtener esferas, que presentan la morfología de las colonias celulares en forma de bola de suspensión.
- Utilice o recoja las esferas, mediante pipeteo, para los experimentos.
- Para obtener poblaciones adherentes derivadas, coloque las esferas en condiciones de cultivo estándar, respectivamente de la línea celular utilizada. 1 a 2 días después, es posible observar una monocapa de células que crecen alrededor de las esferas adherentes, que presenta una morfología similar a la línea de origen celular.
2. Protocolo de formación de esferas a partir de muestras de tumores humanos
NOTA: El uso de muestras humanas con fines de investigación debe cumplir con la legislación de cada país y ser aprobado por el Comité de ética de las Instituciones involucradas.
- Preparar los medios de transporte que contengan DMEM/F12, complementados con suero bovino fetal al 10% (FBS) y solución antibiótica antibiótica antibiótica (10.000 U/ml de penicilina, 10 mg/ml de estreptomicina y 25 g/ml de anfotericina B).
- Preparar los medios de digestión que contengan DMEM/F12, complementados con 10% FBS, 1% solución antibiótica antimicótica, 1 mg/ml de colagenasa tipo IV y 100 g/ml de DNAse I.
- Preparar los medios de inactivación enzimáticos que contengan DMEM/F12, complementados con 10% de FBS y 1% de solución antibiótica-antimicótica (10.000 U/ml de penicilina, 10 mg/ml de estreptomicina y 25 g/ml de anfotericina B).
- Prepare el SCM como se describe en la sección 1.2.
- Obtener la muestra durante el estudio macroscópico de la pieza operativa tan pronto como sea posible después de la extracción quirúrgica.
- Coloque las muestras en medios de transporte y transfieralas al laboratorio para realizar su procesamiento. El procesamiento de muestras debe comenzar dentro de 1 h después de la recopilación para mejorar la tasa de éxito del procedimiento. Tenga cuidado en la recolección de muestras. Manipule las muestras con cuidado. Evite el uso de zonas necróticas o cauterizadas.
- Debajo de la cámara de flujo estéril, transfiera la muestra a un plato y corte en trozos más pequeños (alrededor de 1 mm3) con un bisturí.
- Incubar el tejido humano en un tubo con medios de digestión en una coctelera giratoria de hasta 180 min, a 37oC. Identifique este tubo como Tubo A.
- Reemplace la solución enzimática cada 15 minutos.
- Recoger los medios de digestión (sin extraer ningún fragmento de tejido) y transferirlo a través de un colador celular de 40 m a un nuevo tubo medio lleno de medios de inactivación enzimáticos. Mantenga este tubo a temperatura ambiente e identifíquelo como Tubo B.
- Añadir nuevos medios de digestión al tubo A y devolverlo a la coctelera giratoria a 37 oC.
- En cada colección, compruebe la viabilidad de la celda utilizando el método de exclusión azul trypan.
- Repita este procedimiento durante 180 min o hasta que el recuento de células sea significativamente menor.
- Incubar los fragmentos de tejido en el tubo A en una segunda solución de digestión que contenga partes iguales de accutase y trippsina-EDTA, agitando durante 10 min a 37 oC.
- Agregue el medio de inactivación de enzimas al tubo A y filtre el contenido a través de un colador de células de 40 m en el tubo B.
- Centrifugar la suspensión celular en el tubo B a 200 x g durante 10 min.
- Suspenda el pellet en SCM y compruebe la concentración celular utilizando un hemocitómetro.
- Suspenda la cantidad determinada de suspensión celular en SCM y transfiera a platos recubiertos con poli-HEMA (ver paso 1.1) con una densidad de semilla de 4000 celdas/cm2.
- Incubar a 37oC y 5%CO2 durante 2 días sin perturbar las placas.
- Restablezca la concentración de factores de crecimiento añadiendo EFG de 10 ng/ml y bFGF de 10 ng/ml al medio de cultivo celular.
NOTA: Debe hacerlo cada dos días. - Incubar a 37oC y 5%co2 hasta 5 días después del enchapado (esto puede variar hasta 12 días) para obtener esferas, que presentan la morfología de las colonias celulares en forma de bola de suspensión.
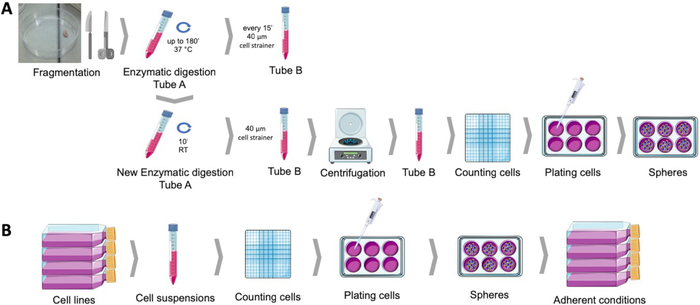
Figura 1: Obtención de células madre cancerosas de muestras de tumores endometriales humanos (A) y líneas celulares de cáncer de mama y ginecológico (B). Las muestras de tumores humanos se fragmentan, digieren enzimáticamente y se enmarcan en esfera que cultuculen medio en platos recubiertos de poli-HEMA. Las líneas celulares cancerosas se separan, se cuentan las suspensiones celulares y las células individuales se distribuyen a baja densidad en placas recubiertas de poli-HEMA en condiciones adecuadas. Las esferas obtenidas, cuando se colocan bajo condiciones de cultivo adherentes, producen poblaciones adherentes derivadas. Haga clic aquí para ver una versión más grande de esta figura.
3. Capacidad de formación de esferas, auto-renovación y área de proyección de esferas
NOTA: La capacidad de formación de esferas es la capacidad de una población de células tumorales para producir esferas. La autorenovación es la capacidad de las células esferas para producir nuevas colonias de células esféricas en suspensión. El área de proyección de la esfera es representativa del área ocupada por la esfera y por lo tanto expresiva de su tamaño y el número de divisiones celulares experimentadas en un cierto período de tiempo.
- Determinación de la capacidad de formación de esferas
- Después de completar el protocolo de formación de esferas, recoger las esferas en un tubo de centrífuga y centrífuga a 125 x g durante 5 min.
- Deseche el SCM y suspenda suavemente el pellet en un volumen conocido de medios frescos. Con el objetivo de concentrar las esferas para facilitar el conteo, suspenda las esferas en un pequeño volumen de medios. Tenga cuidado de no molestar a las esferas.
- Utilice un hemocitómetro para contar las esferas con más de 40 m de diámetro. Alternativamente, las esferas se pueden contar directamente en la placa mediante el uso de un microscopio equipado con una retícula25 o utilizando un sistema automatizado26,27.
- Calcular la proporción porcentual de esferas obtenidas frente al número de celdas inicialmente chapadas.
- Determinación de la autorenovación
- Después de completar el protocolo de formación de esferas, recoger las esferas en un tubo de centrífuga y centrífuga a 125 x g durante 5 min.
- Deseche los medios de cultivo de la esfera y suspenda suavemente el pellet en trippsin-EDTA.
- Incubar hasta 5 min a 37oC.
- Agregue medios de inactivación enzimática y pipetas hacia arriba y hacia abajo para asegurar una suspensión de una sola célula.
- Usando un hemocitómetro y el método de exclusión azul trypan, cuente las células viables en la suspensión.
- Inicie el protocolo de formación de esferas como se describe en la sección 1.
- Después de 8 días, utilice un hemocitómetro para contar las esferas con más de 40 m de diámetro.
- Calcular la proporción porcentual de esferas obtenidas frente al número de celdas inicialmente chapadas.
- Determinación del área de proyección de la esfera
- Para evaluar el área ocupada por las esferas, obtener imágenes de al menos 10 campos aleatorios por condición, en un microscopio invertido equipado con un módulo de adquisición de imágenes. Se recomienda un aumento de 100X a 400X.
- Analizar imágenes utilizando software de imágenes, como el software ImageJ28,dibujando áreas de interés correspondientes a las esferas y midiendo su área en píxeles.
- Calcule el área de proyección de esfera como el área media de píxeles medida.
4. Evaluación del marcador de células madre del cáncer con citometría de flujo
NOTA: CD44+/CD24-/fenotipo bajo se asoció constantemente con células madre de cáncer de mama y ginecológico. El procedimiento descrito se puede utilizar para evaluar este y otros marcadores de superficie celular.
- Después de completar el protocolo de formación de esferas, recoger las esferas en un tubo de centrífuga y centrífuga a 125 x g durante 5 min.
- Deseche el SCM y suspenda suavemente el pellet en trippsin-EDTA.
- Incubar hasta 5 min a 37oC.
- Agregue medios de inactivación enzimática y pipetas hacia arriba y hacia abajo para asegurar una suspensión de una sola célula.
- Centrifugar a 125 x g durante 5 min, desechar el sobrenadante y suspender suavemente las células en PBS.
- Deje que las células descansen en suspensión durante 30 minutos para asegurar la recuperación de la conformación de la membrana.
- Usando un hemocitómetro y el método de exclusión azul trypan, cuente las células en la suspensión.
- Ajuste el volumen de la suspensión celular a 106 celdas/500 l.
- Incubar con los anticuerpos monoclonales de acuerdo con las instrucciones de los proveedores (concentración, tiempo, temperatura y luz/oscuridad) y teniendo en cuenta el conjunto de experimentos representado en el Cuadro 2 o los marcadores indicados en el Cuadro 1.
- Inmediatamente después de la tinción, realice el análisis citométrico de flujo utilizando un catómetro de flujo con módulos de detección adecuados.
- Estandarizar la configuración del citómetro, siguiendo los protocolos establecidos por el EuroFlow Consortium29.
- Configure las puertas primarias en función de la dispersión hacia adelante y lateral, excluyendo los desechos y las células muertas. Esto puede mejorarse mediante el etiquetado concomitante con células anexionantes V y células negativas de gating.
- Establezca puertas de fluorescencia basadas en las muestras no retenidas y compensación para una superposición espectral utilizando controles de tinta individuales.
5. Evaluación del marcador de células madre del cáncer con Western Blot
NOTA: Además de la actividad DE ALDH1, la alta expresión de este marcador se asoció constantemente con las células madre de cáncer de mama y ginecológico13,14. El procedimiento descrito se puede utilizar para evaluar este y otros marcadores de celda.
- Después de completar el protocolo de formación de esferas, recoger las esferas en un tubo de centrífuga y centrífuga a 125 x g durante 5 min.
- Preparación de los líticos de células enteras
- Coloque los tubos de centrífuga sobre hielo y deseche el sobrenadante sin interrumpir el pellet.
- Lavar el pellet con 1 ml de PBS frío y desechar por centrifugación.
- Suspenda el pellet en un pequeño volumen (200-500 l) de tampón de lisis RIPA30 (NaCl 150 mM, Tris-HCl 1,50 mM pH 7,4, Triton-X100 1% vol./vol., ácido desoxicólico de sodio 0,5% wt./vol., dodecyl sulfato sódico 0,5% wt./vol.) complementado con cOmplete Mini y ditiothreitol 1 mM.
- Manteniendo las muestras frías (sobre hielo), enviarlas al vórtice y sonicación con una amplitud del 30%.
- Centrifugar las muestras durante 15 min a 14.000 x g en una centrífuga refrigerada establecida en 4oC.
- Transfiera los sobrenatantes a microtubos nuevos y debidamente identificados.
- Determinar las concentraciones de proteínas utilizando los ensayos BCA o Bradford31.
- Si es necesario, almacene las muestras a -80 oC hasta que se pueda analizar la mancha occidental.
- Realizar la desnaturalización de la muestra, electroforesis, transferencia de electrones y detección de proteínas de acuerdo con los protocolos de hinchamiento occidentales estándar, como se describe32,33,34.
Resultados
El protocolo de formación de esferas permite obtener colonias esféricas en suspensión de varias líneas celulares de cáncer de mama y endometriales (Figura 2A) o después de una suave digestión enzimática del tejido a partir de muestras de tumores humanos(Figura 2E). En ambos casos, pocos días después del enchapado, se obtienen colonias esféricas monoclonales en suspensión. Tanto las esferas del endometrio como las de ...
Discusión
Este protocolo detalla un enfoque para obtener tumoresferas a partir de líneas celulares cancerosas y muestras humanas primarias. Las tumorferas se enriquecen en una subpoblación con propiedades similares a células madre36. Este enriquecimiento en CSC depende de la viabilidad en un entorno libre de anclaje, mientras que las células diferenciadas dependen de la adhesión a un sustrato37. Como el revestimiento primario de células tumorales en un entorno de baja adherenci...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue financiado por la Sociedad Portuguesa de Ginecología a través del Premio de Investigación 2016 y por CIMAGO. Cnc. IBILI cuenta con el apoyo de la Fundación para la Ciencia y la Tecnología, Portugal (UID/NEU/04539/2013), y cofinanciado por FEDER-COMPETE (POCI-01-0145-FEDER-007440). El Banco de Tumores del Hospital y Centro Universitario de Coimbra (CHUC), aprobado por el Comité de ética de la Salud de la CHUC y por la Comisión Nacional de Protección de Datos de Portugal, fue la fuente de muestras endometriales de pacientes seguidas en el Servicio de Ginecología de la institución. La Figura 1 fue producida usando Servier Medical Art, disponible en www.servier.com.
Materiales
| Name | Company | Catalog Number | Comments |
| Absolute ethanol | Merck Millipore | 100983 | |
| Accutase | Gibco | A1110501 | StemPro Accutas Cell Dissociation Reagent |
| ALDH antibody | Santa Cruz Biotechnology | SC166362 | |
| Annexin V FITC | BD Biosciences | 556547 | |
| Antibiotic antimycotic solution | Sigma | A5955 | |
| BCA assay | Thermo Scientific | 23225 | Pierce BCA Protein Assay Kit |
| Bovine serum albumin | Sigma | A9418 | |
| CD133 antibody | Miteny Biotec | 293C3-APC | Allophycocyanin (APC) |
| CD24 antibody | BD Biosciences | 658331 | Allophycocyanin-H7 (APC-H7) |
| CD44 antibody | Biolegend | 103020 | Pacific Blue (PB) |
| Cell strainer | BD Falcon | 352340 | 40 µM |
| Collagenase, type IV | Gibco | 17104-019 | |
| cOmplete Mini | Roche | 118 361 700 0 | |
| Dithiothreitol | Sigma | 43815 | |
| DMEM-F12 | Sigma | D8900 | |
| DNAse I | Roche | 11284932001 | |
| ECC-1 | ATCC | CRL-2923 | Human endometrium adenocarcinoma cell line |
| Epidermal growth factor | Sigma | E9644 | |
| Fibroblast growth factor basic | Sigma | F0291 | |
| Haemocytometer | VWR | HERE1080339 | |
| HCC1806 | ATCC | CRL-2335 | Human mammary squamous cell carcinoma cell line |
| Insulin, transferrin, selenium Solution | Gibco | 41400045 | |
| MCF7 | ATCC | HTB-22 | Human mammary adenocarcinoma cell line |
| Methylcellulose | AlfaAesar | 45490 | |
| NaCl | JMGS | 37040005002212 | |
| Poly(2-hydroxyethyl-methacrylate | Sigma | P3932 | |
| Putrescine | Sigma | P7505 | |
| RL95-2 | ATCC | CRL-1671 | Human endometrium carcinoma cell line |
| Sodium deoxycholic acid | JMS | EINECS 206-132-7 | |
| Sodium dodecyl sulfate | Sigma | 436143 | |
| Tris | JMGS | 20360000BP152112 | |
| Triton-X 100 | Merck | 108603 | |
| Trypan blue | Sigma | T8154 | |
| Trypsin-EDTA | Sigma | T4049 | |
| ß-actin antibody | Sigma | A5316 |
Referencias
- Hardin, H., Zhang, R., Helein, H., Buehler, D., Guo, Z., Lloyd, R. V. The evolving concept of cancer stem-like cells in thyroid cancer and other solid tumors. Laboratory Investigation. 97 (10), 1142 (2017).
- Plaks, V., Kong, N., Werb, Z. The Cancer Stem Cell Niche: How Essential Is the Niche in Regulating Stemness of Tumor Cells?. Cell Stem Cell. 16 (3), 225-238 (2015).
- Visvader, J. E., Lindeman, G. J. Cancer stem cells in solid tumours accumulating evidence and unresolved questions. Nature reviews. Cancer. 8, 755-768 (2008).
- Allegra, A., et al. The Cancer Stem Cell Hypothesis: A Guide to Potential Molecular Targets. Cancer Investigation. 32 (9), 470-495 (2014).
- Al-Hajj, M., Wicha, M. S., Benito-Hernandez, A., Morrison, S. J., Clarke, M. F. Prospective identification of tumorigenic breast cancer cells. Proceedings of the National Academy of Sciences. 100 (7), 3983-3988 (2003).
- Friel, A. M., et al. Functional analyses of the cancer stem cell-like properties of human endometrial tumor initiating cells. Cell Cycle. 7 (2), 242-249 (2008).
- Zhang, S., et al. Identification and Characterization of Ovarian Cancer-Initiating Cells from Primary Human Tumors. Cancer Research. 68 (11), 4311-4320 (2008).
- Bapat, S. A., Mali, A. M., Koppikar, C. B., Kurrey, N. K. Stem and progenitor-like cells contribute to the aggressive behavior of human epithelial ovarian cancer. Cancer research. 65 (8), 3025-3029 (2005).
- Carvalho, M. J., Laranjo, M., Abrantes, A. M., Torgal, I., Botelho, M. F., Oliveira, C. F. Clinical translation for endometrial cancer stem cells hypothesis. Cancer and Metastasis Reviews. 34 (3), 401-416 (2015).
- Morel, A. P., Lièvre, M., Thomas, C., Hinkal, G., Ansieau, S., Puisieux, A. Generation of Breast Cancer Stem Cells through Epithelial-Mesenchymal Transition. PLoS ONE. 3 (8), e2888 (2008).
- Tirino, V., et al. Cancer stem cells in solid tumors: an overview and new approaches for their isolation and characterization. The FASEB Journal. 27 (1), 13 (2013).
- Ajani, J. A., et al. ALDH-1 expression levels predict response or resistance to preoperative chemoradiation in resectable esophageal cancer patients. Molecular Oncology. 8 (1), 142-149 (2014).
- Carvalho, M. J., et al. Endometrial Cancer Spheres Show Cancer Stem Cells Phenotype and Preference for Oxidative Metabolism. Pathology and Oncology Research. , (2018).
- Laranjo, M., et al. Mammospheres of hormonal receptor positive breast cancer diverge to triple-negative phenotype. The Breast. 38, 22-29 (2018).
- Cui, M., et al. Non-Coding RNA Pvt1 Promotes Cancer Stem Cell–Like Traits in Nasopharyngeal Cancer via Inhibiting miR-1207. Pathology & Oncology Research. , (2018).
- Deng, S., et al. Distinct expression levels and patterns of stem cell marker, aldehyde dehydrogenase isoform 1 ALDH1), in human epithelial cancers. PloS one. 5 (4), e10277 (2010).
- Weiswald, L. B., Guinebretière, J. M., Richon, S., Bellet, D., Saubaméa, B., Dangles-Marie, V. In situ protein expression in tumour spheres: development of an immunostaining protocol for confocal microscopy. BMC Cancer. 10 (1), 106 (2010).
- Weiswald, L. B., Bellet, D., Dangles-Marie, V. Spherical Cancer Models in Tumor Biology. Neoplasia. 17 (1), 1-15 (2015).
- Picon-Ruiz, M., et al. Low adherent cancer cell subpopulations are enriched in tumorigenic and metastatic epithelial-to-mesenchymal transition-induced cancer stem-like cells. Scientific Reports. 6 (1), 1-13 (2016).
- Dontu, G., et al. In vitro propagation and transcriptional profiling of human mammary stem/progenitor cells. Genes & development. 17 (10), 1253-1270 (2003).
- Ballout, F., et al. Sphere-Formation Assay: Three-Dimensional in vitro Culturing of Prostate Cancer Stem/Progenitor Sphere-Forming Cells. Frontiers in Oncology. 8 (August), 1-14 (2018).
- Ishiguro, T., Ohata, H., Sato, A., Yamawaki, K., Enomoto, T., Okamoto, K. Tumor-derived spheroids: Relevance to cancer stem cells and clinical applications. Cancer Science. 108 (3), 283-289 (2017).
- Noseda, M., Nasatto, P., Silveira, J., Pignon, F., Rinaudo, M., Duarte, M. Methylcellulose, a Cellulose Derivative with Original Physical Properties and Extended Applications. Polymers. 7 (5), 777-803 (2015).
- Shaw, F. L., et al. A detailed mammosphere assay protocol for the quantification of breast stem cell activity. Journal of Mammary Gland Biology and Neoplasia. 17 (2), 111-117 (2012).
- Zhou, M., et al. LncRNA-Hh Strengthen Cancer Stem Cells Generation in Twist-Positive Breast Cancer via Activation of Hedgehog Signaling Pathway. Stem cells (Dayton, Ohio). 34 (1), 55-66 (2016).
- Ha, J. R., et al. Integration of Distinct ShcA Signaling Complexes Promotes Breast Tumor Growth and Tyrosine Kinase Inhibitor Resistance. Molecular cancer research MCR. 16 (5), 894-908 (2018).
- Jurmeister, S., et al. Identification of potential therapeutic targets in prostate cancer through a cross-species approach. EMBO molecular medicine. 10 (3), (2018).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature methods. 9 (7), 671-675 (2012).
- Kalina, T., et al. EuroFlow standardization of flow cytometer instrument settings and immunophenotyping protocols. Leukemia. 26 (9), 1986-2010 (2012).
- Peach, M., Marsh, N., Miskiewicz, E. I., MacPhee, D. J. . Solubilization of Proteins: The Importance of Lysis Buffer Choice. , 49-60 (2015).
- Olson, B. J. S. C. Assays for Determination of Protein Concentration. Current Protocols in Pharmacology. , A.3A.1-A.3A.32 (2016).
- Eslami, A., Lujan, J. Western Blotting: Sample Preparation to Detection. Journal of Visualized Experiments. (44), 1-2 (2010).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. Journal of Visualized Experiments. 84 (84), 1-8 (2014).
- Oldknow, K. J., et al. A Guide to Modern Quantitative Fluorescent Western Blotting with Troubleshooting Strategies. Journal of Visualized Experiments. 8 (93), 1-10 (2014).
- Serambeque, B. . Células estaminais do cancro do endométrio - a chave para o tratamento personalizado? [Stem Cells of Endometrial Cancer: The Key to Personalized Treatment?]. , (2018).
- Lee, C. H., Yu, C. C., Wang, B. Y., Chang, W. W. Tumorsphere as an effective in vitro platform for screening anti-cancer stem cell drugs. Oncotarget. 7 (2), (2015).
- De Luca, A., et al. Mitochondrial biogenesis is required for the anchorage-independent survival and propagation of stem-like cancer cells. Oncotarget. 6 (17), (2015).
- Masuda, A., et al. An improved method for isolation of epithelial and stromal cells from the human endometrium. Journal of Reproduction and Development. 62 (2), 213-218 (2016).
- Del Rio-Tsonis, K., et al. Facile isolation and the characterization of human retinal stem cells. Proceedings of the National Academy of Sciences. 101 (44), 15772-15777 (2004).
- Wang, L., Guo, H., Lin, C., Yang, L., Wang, X. I. Enrichment and characterization of cancer stem-like cells from a cervical cancer cell line. Molecular Medicine Reports. 9 (6), 2117-2123 (2014).
- Chen, Y. C., et al. High-throughput single-cell derived sphere formation for cancer stem-like cell identification and analysis. Scientific Reports. 6 (April), 1-12 (2016).
- Kim, J., Jung, J., Lee, S. J., Lee, J. S., Park, M. J. Cancer stem-like cells persist in established cell lines through autocrine activation of EGFR signaling. Oncology Letters. 3 (3), 607-612 (2012).
- Hwang-Verslues, W. W., et al. Multiple Lineages of Human Breast Cancer Stem/Progenitor Cells Identified by Profiling with Stem Cell Markers. PloS one. 4 (12), e8377 (2009).
- Feng, Y., et al. Metformin reverses stem cell-like HepG2 sphere formation and resistance to sorafenib by attenuating epithelial-mesenchymal transformation. Molecular Medicine Reports. 18 (4), 3866-3872 (2018).
- Wang, H., Paczulla, A., Lengerke, C. Evaluation of Stem Cell Properties in Human Ovarian Carcinoma Cells Using Multi and Single Cell-based Spheres Assays. Journal of Visualized Experiments. (95), 1-11 (2015).
- Stebbing, J., Lombardo, Y., Coombes, C. R., de Giorgio, A., Castellano, L. Mammosphere Formation Assay from Human Breast Cancer Tissues and Cell Lines. Journal of Visualized Experiments. (97), 1-5 (2015).
- Zhao, H., et al. Sphere-forming assay vs. organoid culture: Determining long-term stemness and the chemoresistant capacity of primary colorectal cancer cells. International Journal of Oncology. 54 (3), 893-904 (2019).
- Bagheri, V., et al. Isolation and identification of chemotherapy-enriched sphere-forming cells from a patient with gastric cancer. Journal of Cellular Physiology. 233 (10), 7036-7046 (2018).
- Kaowinn, S., Kaewpiboon, C., Koh, S., Kramer, O., Chung, Y. STAT1-HDAC4 signaling induces epithelial-mesenchymal transition and sphere formation of cancer cells overexpressing the oncogene, CUG2. Oncology Reports. , 2619-2627 (2018).
- Lonardo, E., Cioffi, M., Sancho, P., Crusz, S., Heeschen, C. Studying Pancreatic Cancer Stem Cell Characteristics for Developing New Treatment Strategies. Journal of Visualized Experiments. (100), 1-9 (2015).
- Lu, H., et al. Targeting cancer stem cell signature gene SMOC-2 Overcomes chemoresistance and inhibits cell proliferation of endometrial carcinoma. EBioMedicine. 40, 276-289 (2019).
- Bu, P., Chen, K. Y., Lipkin, S. M., Shen, X. Asymmetric division: a marker for cancer stem cells. Oncotarget. 4 (7), (2013).
- Islam, F., Qiao, B., Smith, R. A., Gopalan, V., Lam, A. K. Y. Cancer stem cell: fundamental experimental pathological concepts and updates. Experimental and molecular pathology. 98 (2), 184-191 (2015).
- Liu, W., et al. Comparative characterization of stem cell marker expression, metabolic activity and resistance to doxorubicin in adherent and spheroid cells derived from the canine prostate adenocarcinoma cell line CT1258. Anticancer research. 35 (4), 1917-1927 (2015).
- Broadley, K. W. R., et al. Side Population is Not Necessary or Sufficient for a Cancer Stem Cell Phenotype in Glioblastoma Multiforme. STEM CELLS. 29 (3), 452-461 (2011).
- Cojoc, M., Mäbert, K., Muders, M. H., Dubrovska, A. A role for cancer stem cells in therapy resistance: Cellular and molecular mechanisms. Seminars in Cancer Biology. 31, 16-27 (2015).
- Batlle, E., Clevers, H. Cancer stem cells revisited. Nature Medicine. 23 (10), 1124-1134 (2017).
- Zhang, X. L., Jia, Q., Lv, L., Deng, T., Gao, J. Tumorspheres Derived from HCC Cells are Enriched with Cancer Stem Cell-like Cells and Present High Chemoresistance Dependent on the Akt Pathway. Anti-cancer agents in medicinal chemistry. 15 (6), 755-763 (2015).
- Fukamachi, H., et al. CD49fhigh Cells Retain Sphere-Forming and Tumor-Initiating Activities in Human Gastric Tumors. PLoS ONE. 8 (8), e72438 (2013).
- Gao, M. Q., Choi, Y. P., Kang, S., Youn, J. H., Cho, N. H. CD24+ cells from hierarchically organized ovarian cancer are enriched in cancer stem cells. Oncogene. 29 (18), 2672-2680 (2010).
- Cariati, M., et al. Alpha-6 integrin is necessary for the tumourigenicity of a stem cell-like subpopulation within the MCF7 breast cancer cell line. International Journal of Cancer. 122 (2), 298-304 (2008).
- López, J., Valdez-Morales, F. J., Benítez-Bribiesca, L., Cerbón, M., Carrancá, A. Normal and cancer stem cells of the human female reproductive system. Reproductive Biology and Endocrinology. 11 (1), 53 (2013).
- Alvero, A. B., et al. Molecular phenotyping of human ovarian cancer stem cells unravels the mechanisms for repair and chemoresistance. Cell Cycle. 8 (1), 158-166 (2009).
- Charafe-Jauffret, E., Ginestier, C., Birnbaum, D. Breast cancer stem cells: tools and models to rely on. BMC Cancer. 9 (1), 202 (2009).
- Leccia, F., et al. ABCG2, a novel antigen to sort luminal progenitors of BRCA1- breast cancer cells. Molecular Cancer. 13 (1), 213 (2014).
- Croker, A. K., Allan, A. L. Inhibition of aldehyde dehydrogenase (ALDH) activity reduces chemotherapy and radiation resistance of stem-like ALDHhiCD44+ human breast cancer cells. Breast Cancer Research and Treatment. 133 (1), 75-87 (2012).
- Sun, M., et al. Enhanced efficacy of chemotherapy for breast cancer stem cells by simultaneous suppression of multidrug resistance and antiapoptotic cellular defense. Acta Biomaterialia. 28, 171-182 (2015).
- Shao, J., Fan, W., Ma, B., Wu, Y. Breast cancer stem cells expressing different stem cell markers exhibit distinct biological characteristics. Molecular Medicine Reports. 14 (6), 4991-4998 (2016).
- Croker, A. K., et al. High aldehyde dehydrogenase and expression of cancer stem cell markers selects for breast cancer cells with enhanced malignant and metastatic ability. Journal of Cellular and Molecular Medicine. 13 (8b), 2236-2252 (2009).
- Cheung, S. K. C., et al. Stage-specific embryonic antigen-3 (SSEA-3) and β3GalT5 are cancer specific and significant markers for breast cancer stem cells. Proceedings of the National Academy of Sciences. 113 (4), 960-965 (2016).
- Meyer, M. J., Fleming, J. M., Lin, A. F., Hussnain, S. A., Ginsburg, E., Vonderhaar, B. K. CD44 pos CD49f hi CD133/2 hi Defines Xenograft-Initiating Cells in Estrogen Receptor–Negative Breast Cancer. Cancer Research. 70 (11), 4624-4633 (2010).
- Ahn, S. M., Goode, R. J. A., Simpson, R. J. Stem cell markers: Insights from membrane proteomics?. PROTEOMICS. 8 (23-24), 4946-4957 (2008).
- Chefetz, I., et al. TLR2 enhances ovarian cancer stem cell self-renewal and promotes tumor repair and recurrence. Cell Cycle. 12 (3), 511-521 (2013).
- Alvero, A. B., et al. Stem-Like Ovarian Cancer Cells Can Serve as Tumor Vascular Progenitors. Stem Cells. 27 (10), 2405-2413 (2009).
- Yin, G., et al. Constitutive proteasomal degradation of TWIST-1 in epithelial–ovarian cancer stem cells impacts differentiation and metastatic potential. Oncogene. 32 (1), 39-49 (2013).
- Wei, X., et al. Mullerian inhibiting substance preferentially inhibits stem/progenitors in human ovarian cancer cell lines compared with chemotherapeutics. Proceedings of the National Academy of Sciences. 107 (44), 18874-18879 (2010).
- Meirelles, K., et al. Human ovarian cancer stem/progenitor cells are stimulated by doxorubicin but inhibited by Mullerian inhibiting substance. Proceedings of the National Academy of Sciences. 109 (7), 2358-2363 (2012).
- Shi, M. F., et al. Identification of cancer stem cell-like cells from human epithelial ovarian carcinoma cell line. Cellular and Molecular Life Sciences. 67 (22), 3915-3925 (2010).
- Meng, E., et al. CD44+/CD24− ovarian cancer cells demonstrate cancer stem cell properties and correlate to survival. Clinical & Experimental Metastasis. 29 (8), 939-948 (2012).
- Witt, A. E., et al. Identification of a cancer stem cell-specific function for the histone deacetylases, HDAC1 and HDAC7, in breast and ovarian. Oncogene. 36 (12), 1707-1720 (2017).
- Wu, H., Zhang, J., Shi, H. Expression of cancer stem markers could be influenced by silencing of p16 gene in HeLa cervical carcinoma cells. European journal of gynaecological oncology. 37 (2), 221-225 (2016).
- Huang, R., Rofstad, E. K. Cancer stem cells (CSCs), cervical CSCs and targeted therapies. Oncotarget. 8 (21), 35351-35367 (2017).
- Zhang, X., et al. Imatinib sensitizes endometrial cancer cells to cisplatin by targeting CD117-positive growth-competent cells. Cancer Letters. 345 (1), 106-114 (2014).
- Luo, L., et al. Ovarian cancer cells with the CD117 phenotype are highly tumorigenic and are related to chemotherapy outcome. Experimental and Molecular Pathology. 91 (2), 596-602 (2011).
- Zhao, P., Lu, Y., Jiang, X., Li, X. Clinicopathological significance and prognostic value of CD133 expression in triple-negative breast carcinoma. Cancer Science. 102 (5), 1107-1111 (2011).
- Ferrandina, G., et al. Expression of CD133-1 and CD133-2 in ovarian cancer. International Journal of Gynecologic Cancer. 18 (3), 506-514 (2008).
- Rutella, S., et al. Cells with characteristics of cancer stem/progenitor cells express the CD133 antigen in human endometrial tumors. Clinical cancer research an official journal of the American Association for Cancer Research. 15 (13), 4299-4311 (2009).
- Friel, A. M., et al. Epigenetic regulation of CD133 and tumorigenicity of CD133 positive and negative endometrial cancer cells. Reproductive Biology and Endocrinology. 8 (1), 147 (2010).
- Nakamura, M., et al. Prognostic impact of CD133 expression as a tumor-initiating cell marker in endometrial cancer. Human Pathology. 41 (11), 1516-1529 (2010).
- Saha, S. K., et al. KRT19 directly interacts with β-catenin/RAC1 complex to regulate NUMB-dependent NOTCH signaling pathway and breast cancer properties. Oncogene. 36 (3), 332-349 (2017).
- LV, X., Wang, Y., Song, Y., Pang, X., Li, H. Association between ALDH1+/CD133+ stem-like cells and tumor angiogenesis in invasive ductal breast carcinoma. Oncology Letters. 11 (3), 1750-1756 (2016).
- Ruscito, I., et al. Exploring the clonal evolution of CD133/aldehyde-dehydrogenase-1 (ALDH1)-positive cancer stem-like cells from primary to recurrent high-grade serous ovarian cancer (HGSOC). A study of the Ovarian Cancer Therapy–Innovative Models Prolong Survival (OCTIPS). European Journal of Cancer. 79, 214-225 (2017).
- Sun, Y., et al. Isolation of Stem-Like Cancer Cells in Primary Endometrial Cancer Using Cell Surface Markers CD133 and CXCR4. Translational Oncology. 10 (6), 976-987 (2017).
- Rahadiani, N., et al. Expression of aldehyde dehydrogenase 1 (ALDH1) in endometrioid adenocarcinoma and its clinical implications. Cancer Science. 102 (4), 903-908 (2011).
- Mamat, S., et al. Transcriptional Regulation of Aldehyde Dehydrogenase 1A1 Gene by Alternative Spliced Forms of Nuclear Factor Y in Tumorigenic Population of Endometrial Adenocarcinoma. Genes & Cancer. 2 (10), 979-984 (2011).
- Mukherjee, S. A., et al. Non-migratory tumorigenic intrinsic cancer stem cells ensure breast cancer metastasis by generation of CXCR4+ migrating cancer stem cells. Oncogene. 35 (37), 4937-4948 (2016).
- Lim, E., et al. Aberrant luminal progenitors as the candidate target population for basal tumor development in BRCA1 mutation carriers. Nature Medicine. 15 (8), 907-913 (2009).
- Liang, Y. J., et al. Interaction of glycosphingolipids GD3 and GD2 with growth factor receptors maintains breast cancer stem cell phenotype. Oncotarget. 8 (29), 47454-47473 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados