É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Obtenção de esferas de células-tronco cancerígenas de tumores ginecológicos e de câncer de mama
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
O objetivo dessa metodologia é identificar células-tronco cancerígenas (CSC) em linhas de células cancerosas e amostras primárias de tumor humano com o protocolo de formação da esfera, de forma robusta, utilizando ensaios funcionais e caracterização phenotípica com citometria de fluxo e ocidental Borrão.
Resumo
As células-tronco cancerígenas (CSC) são uma pequena população com autorenovação e plasticidade que são responsáveis pela tumorigênese, resistência ao tratamento e doenças recorrentes. Essa população pode ser identificada por marcadores de superfície, atividade enzimática e um perfil funcional. Essas abordagens em si são limitadas, devido à heterogeneidade penópeba e plasticidade csc. Aqui, atualizamos o protocolo de formação da esfera para obter esferas csc a partir de cânceres de mama e ginecológico, avaliando propriedades funcionais, marcadores CSC e expressão proteica. As esferas são obtidas com semeade de células únicas em baixa densidade na cultura da suspensão, utilizando um meio semissólido de metilcelulose para evitar migração e agregados. Esse protocolo rentável pode ser usado em linhas de células cancerosas, mas também em tumores primários. A cultura tridimensional de suspensão não aderente pensada para imitar o microambiente tumoral, particularmente o nicho csc, é complementada com fator de crescimento epidérmico e fator básico de crescimento do fibroblasto para garantir a sinalização do CSC. Visando uma identificação robusta do CSC, propomos uma abordagem complementar, combinando avaliação funcional e phenotipica. Capacidade de formação da esfera, auto-renovação e área de projeção da esfera estabelecem propriedades funcionais csc. Além disso, a caracterização compreende a avaliação da citometria de fluxo dos marcadores, representados por CD44+/CD24- e CD133, e mancha ocidental, considerando aldH. O protocolo apresentado também foi otimizado para amostras de tumor primário, seguindo um procedimento de digestão amostral, útil para pesquisa translacional.
Introdução
As populações cancerígenas são heterogêneas, com células apresentando diferentes morfologias, capacidade de proliferação e invasão, devido à expressão genética diferencial. Entre essas células, existe uma população minoritária chamada células-tronco cancerígenas (CSC)1, que têm a capacidade de autorenovação, recapitulando a heterogeneidade do nicho tumoral primário e produzindo progenitores aberrantemente diferenciadores que não respondem adequadamente aos controles homeostáticos2. As propriedades do CSC podem ser traduzidas diretamente na prática clínica, dada a associação com eventos, como tumorigenicidade ou resistência à quimioterapia3. A identificação do CSC pode levar ao desenvolvimento de terapias direcionadas que possam incluir bloqueio de marcadores de superfície, promoção da diferenciação do CSC, bloqueio de componentes da via de sinalização CSC, destruição de nicho e mecanismos epigenéticos4.
O isolamento do CSC tem sido realizado nas linhas de células e em amostras de tumores primários5,6,7,8. O perfil funcional descrito para CSC inclui capacidade clonogênica, população lateral e formação de tumorosfera9. O fenótipobaixo CD44high/CD24 tem sido consistentemente associado ao CSC mamário, que tem se mostrado tumorigênico in vivo e já foi associado com transição epitelial ao mesenquimal5,10. A alta atividade de ALDH também tem sido associada à caule e transição epitelial à mesenquimal (EMT) em vários tipos de tumores sólidos11. A expressão ALDH tem sido associada à resistência à quimioterapia e ao fenótipo CSC in vitro12,13,14,15,16. Vários outros marcadores foram ligados a propriedades csc em diferentes tipos de tumores, como CD133, CD49f, ITGA6, CD1663,4 e outros, conforme descrito na Tabela 1.
As tumoresferas consistem em um modelo tridimensional para o estudo e expansão do CSC. Nesse modelo, as suspensões celulares das linhas celulares e de amostras de sangue ou tumor são cultivadas em um meio complementado com fatores de crescimento, ou seja, fator de crescimento epidérmico (EGF) e fator básico de crescimento do fibroblasto (bFGF), sem soro bovino fetal e em condições não aderentes17. A inibição da adesão celular resulta na morte por anoikis de células diferenciadas18. As esferas são derivadas do crescimento clonal de uma célula isolada. Para isso, as células são distribuídas em baixa densidade para evitar a fusão celular e agregação19. Outra estratégia inclui o uso de metilcelulose semissólida20.
O protocolo de formação da esfera ganhou popularidade no isolamento e expansão do CSC, devido ao tempo e custo e ao custo e razões técnicas, rentáveis e reprodutíveis21,22. Apesar de algumas reservas sobre a extensão de qual a formação da esfera reflete csc, há uma propensão de células-tronco para crescer em condições não aderentes com o fenótipo característico, que se assemelha ao microambiente nativo21. Nenhum dos métodos disponíveis para isolamento do CSC de tumores sólidos tem total eficiência, destacando a importância de desenvolver marcadores mais específicos ou combinações de metodologias e marcadores.
Neste protocolo, detalhamos o isolamento do CSC com o protocolo de formação da esfera, com o princípio do crescimento unicelular em condições não aderentes e a capacidade de produzir um fenótipo diferenciado. Uma representação esquemática deste procedimento está representada na Figura 1. Descrevemos também a caracterização com marcadores de superfície e expressão ALDH para CSC, tanto para linhas de células tumorais mama quanto ginecológicas e amostras de tumores primários.
Protocolo
Esse protocolo foi realizado em conformidade com as diretrizes éticas do Hospital Coimbra e do Núcleo De Tumores do Centro Universitário (CHUC), e foi aprovado pelo Comitê de Ética para a Saúde da CHUC e pela Comissão Nacional de Proteção de Dados.
1. Protocolo de formação da esfera e populações adeptos derivadas de culturas celulares contínuas
NOTA: Realize todos os procedimentos em condições estéreis rigorosas.
- Preparação de frascos de cultura de suspensão não aderentes ou placas, revestindo a superfície de crescimento com poli (2-hidroxiyethyl-methacrito (poli-HEMA)
- Prepare uma solução de 15 mg/mL mexendo poli-HEMA em etanol absoluto a 65 °C. Frascos de cultura celular de revestimento ou placas com 50 μL/cm2.
- Deixe secar a 37 °C em um forno de secagem. Se necessário, enrole as placas e armazene à temperatura ambiente.
- Preparação da mídia de culturinga da esfera (SCM)
- Prepare uma solução de 2% de metilcelulose em água ultrapura e esterilizar no autoclave. A metilcelulose tende a ser mais fácil de solarubilizar pelo resfriamento; portanto, disperse o pó em água a 80 °C e mexa até esfriar23.
- Prepare uma solução concentrada duas vezes do SCM (solução de estoque). A solução de trabalho Do SCM contém DMEM-F12, complementada com 100 mM putrescine, 1% insulina, transferrina, selênio e 1% solução antiminética antibiótico-antimicótica (10.000 penicilina U/mL, 10 mg/mL estreptomicina e 25 μg/mL amphotericina B).
- Para preparar o SCM, misture volumes iguais da solução de estoque SCM com a solução de 2% de metilcelulose.
- Complete o meio imediatamente antes do uso adicionando 10 ng/mL fator de crescimento epidérmico (EFG) e 10 ng/mL fator de crescimento básico do fibroblasto (bFGF).
- Se forem utilizados linhas celulares mais exigentes, complemente o meio com albumina de soro bovino de 0,4%, o que pode ser uma vantagem.
- Comece com um frasco de câncer de mama MCF7 ou HCC1806 ou células cancerígenas endometrial Endometrial (ou outra linha de escolha de células cancerígenas) com 80% a 90% de confluência.
- Descarte a mídia de cultura celular, lave com solução salina tampão de fosfato (PBS) e desapego as células com trypsin-EDTA (1 a 2 mL para um frasco de cultura celular de 75 cm2).
- Adicione mídia de cultura celular (2 a 4 mL para um frasco de cultura celular de 75 cm2) e centrífuga em 200 x g para 5 min para descartar enzimas.
- Suspenda a pelota em um volume conhecido de mídia de cultura celular e pipeta para cima e para baixo para garantir uma única suspensão celular. Para isso, pode ser utilizado um filtro de 40 μm de células.
- Conte as células no hemocímetro e calcule a concentração celular da suspensão celular. Aproveite esta etapa para garantir a observação de uma suspensão de uma única célula. A contagem cuidadosa das células é essencial para quantificar com precisão os efeitos dos tratamentos.
- Suspender a quantidade determinada de suspensão celular no meio completo do SCM e transferir para pratos revestidos poli-HEMA. Como valor de referência para a densidade de semeade, considere de 500 a 2000 células/cm2.
NOTA: A otimização da densidade de semeação e do tempo de cultura para cada linha celular é altamente recomendada24. - Incubar a 37 °C e 5% de CO2 por 2 dias sem perturbar as placas.
- Restabelecer a concentração de fatores de crescimento adicionando 10 ng/mL EFG e 10 ng/mL bFGF à mídia de cultura celular. Repita esse passo a cada dois dias.
- Incuba-se a 37 °C e 5% DE CO2 até 5 dias após o revestimento (isso pode variar de 3 a 12 dias de acordo com a linha celular) para obter esferas, que apresentam a morfologia das colônias de células em forma de bola suspensa.
- Use ou colete as esferas, por tubulação, para os experimentos.
- Para obter populações adeptos derivadas, coloque as esferas em condições culturais padrão, respectivamente da linha celular utilizada. 1 a 2 dias depois, é possível observar uma monocamada de células crescendo em torno de esferas aderentes, que apresenta uma morfologia semelhante à linha de origem celular.
2. Protocolo de formação da esfera a partir de amostras de tumor humano
NOTA: O uso de amostras humanas para fins de pesquisa deve cumprir a legislação de cada país e ser aprovado pelo Comitê de Ética das Instituições envolvidas.
- Prepare os meios de transporte contendo DMEM/F12, complementados com soro bovino 10% fetal (FBS) e 2% solução antiminóltica antibiótico-antiminética (10.000 penicilina U/mL, 10 mg/mL streptomicin e 25 μg/mL amphotericina B).
- Prepare a mídia de digestão contendo DMEM/F12, complementado com 10% de FBS, 1% de solução antiminística antibiótico, 1 mg/mL tipo IV colagenase e 100 μg/mL DNAse I.
- Prepare a enzima inativação de mídia contendo DMEM/F12, complementada com 10% de FBS e 1% de solução antimiocrática antibiótico(10.000 penicilina U/mL, 10 mg/mL estreptomicina e 25 μg/mL anhoticina B).
- Prepare o SCM conforme descrito na seção 1.2.
- Obtenha a amostra durante o estudo macroscópico da peça operativa o mais rápido possível após a remoção cirúrgica.
- Coloque as amostras nos meios de transporte e transfira-as para o laboratório para onde o processamento. O processamento amostral deve começar dentro de 1h após a coleta para melhorar a taxa de sucesso do procedimento. Aplique cautela na coleta de amostras. Manuseie as amostras cuidadosamente. Evite o uso de zonas necroticóticas ou cauterizadas.
- a câmara de fluxo estéril, transfira a amostra para um prato e corte em pedaços menores (cerca de 1 mm3) com um bisturi.
- Incubar o tecido humano em um tubo com mídia de digestão em um shaker rotativo de até 180 min, a 37 °C. Identifique este tubo como tubo A.
- Substitua a solução enzimática a cada 15 min.
- Colete a mídia de digestão (sem remover fragmentos de tecido) e transfira-o através de um filtro de 40 μm para um novo tubo semi-cheio de mídia de inativação enzimática. Mantenha este tubo à temperatura ambiente e identifique-o como tubo B.
- Adicione novos meios de digestão ao Tubo A e devolva-o ao agitador rotativo a 37 °C.
- A cada coleção, verifique a viabilidade celular usando o método de exclusão azul trypan.
- Repita este procedimento por 180 min ou até que a contagem de células seja significativamente menor.
- Incubar os fragmentos de tecido no Tubo A em uma segunda solução de digestão contendo partes iguais de accutase e trypsin-EDTA, mexendo por 10 min a 37 °C.
- Adicione a enzima inativação à tela A e filtre o conteúdo através de um filtro de 40 μm de célulano Tubo B.
- Centrífuga a suspensão celular no Tubo B a 200 x g por 10 min.
- Suspenda a pelota no SCM e verifique a concentração celular usando um hemocímetro.
- Suspender a quantidade determinada de suspensão celular no SCM e transferir para pratos revestidos poli-HEMA (ver passo 1.1) com densidade de semeadedes de 4000 células/cm2.
- Incubar a 37 °C e 5% de CO2 por 2 dias sem perturbar as placas.
- Restabelecer a concentração de fatores de crescimento adicionando 10 ng/mL EFG e 10 ng/mL bFGF à mídia de cultura celular.
NOTA: Você deve fazer isso a cada dois dias. - Incuba-se a 37 °C e 5% CO2 até 5 dias após o revestimento (isso pode variar até 12 dias) para obter esferas, que apresentam a morfologia das colônias de células em forma de bola suspensa.
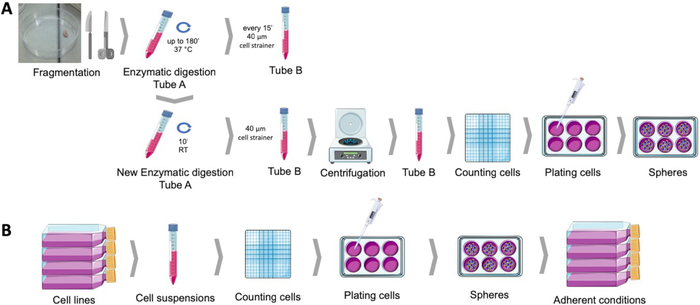
Figura 1: Obtenção de células-tronco cancerígenas de amostras de tumores endometrial humanos (A) e linhas de células cancerígenas de mama e ginecológica (B). As amostras de tumor humano são fragmentadas, enzimáticamente digeridas e emplacadas em meio de culturinga em pratos revestidos poli-HEMA. As linhas de células cancerígenas são separadas, são contadas suspensões celulares, e as células únicas são distribuídas em baixa densidade em placas revestidas de poli-HEMA em condições apropriadas. As esferas obtidas, quando colocadas condições culturais adeptos, produzem populações adeptos derivadas. Clique aqui para ver uma versão maior deste valor.
3. Capacidade formadora de esferas, autorenovação e Área de Projeção da Esfera
NOTA: A capacidade de formação da esfera é a capacidade de uma população celular tumoral produzir esferas. A autorenovação é a capacidade das células esferas de produzir novas colônias de células esféricas em suspensão. A área de projeção da esfera é representativa da área ocupada pela esfera e, portanto, expressiva de seu tamanho e do número de divisões celulares submetidas em um determinado período de tempo.
- Determinando a capacidade de formação da esfera
- Após a conclusão do protocolo de formação da esfera, colete as esferas em um tubo de centrífuga e centrífuga a 125 x g por 5 min.
- Descarte o SCM e suspenda suavemente a pelota em um volume conhecido de mídia fresca. Com o objetivo de concentrar as esferas para facilitar a contagem, suspenda as esferas em um pequeno volume de mídia. Tenha cuidado para não perturbar as esferas.
- Use um hemocímetro para contar as esferas com mais de 40 μm de diâmetro. Alternativamente, as esferas podem ser contadas diretamente na placa usando um microscópio equipado com um graticule25 ou usando um sistema automatizado26,27.
- Calcule a razão percentual das esferas obtidas versus o número de células inicialmente emplacadas.
- Determinando a auto-renovação
- Após a conclusão do protocolo de formação da esfera, colete as esferas em um tubo de centrífuga e centrífuga a 125 x g por 5 min.
- Descarte a esfera culturing media e suspenda suavemente a pelota em trypsin-EDTA.
- Incubar até 5 min a 37 °C.
- Adicione a enzima inativação de mídia e pipeta para cima e para baixo para garantir uma suspensão celular única.
- Usando um hemocímetro e o método de exclusão azul trypan, conte as células viáveis na suspensão.
- Inicie o protocolo de formação da esfera conforme descrito na seção 1.
- Após 8 dias, use um hemocímetro para contar as esferas com mais de 40 μm de diâmetro.
- Calcule a razão percentual das esferas obtidas versus o número de células inicialmente emplacadas.
- Determinando a área de projeção da esfera
- Para avaliar a área ocupada pelas esferas, obtenha imagens de pelo menos 10 campos aleatórios por condição, em um microscópio invertido equipado com um módulo de aquisição de imagens. Recomenda-se uma ampliação de 100X a 400X.
- Analisar imagens usando software de imagem, como o software ImageJ28,desenhando áreas de interesse correspondentes às esferas e medindo sua área em pixels.
- Calcule a área de projeção da esfera como a área média dos pixels medidos.
4. Avaliação do marcador de células-tronco do câncer com citometria de fluxo
NOTA: CD44+/CD24-/fenótipo baixo foi consistentemente associado às células-tronco do câncer de mama e ginecológico. O procedimento descrito pode ser usado para avaliar este e outros marcadores de superfície celular.
- Após a conclusão do protocolo de formação da esfera, colete as esferas em um tubo de centrífuga e centrífuga a 125 x g por 5 min.
- Descarte o SCM e suspenda delicadamente a pelota em trypsin-EDTA.
- Incubar até 5 min a 37 °C.
- Adicione a enzima inativação de mídia e pipeta para cima e para baixo para garantir uma suspensão celular única.
- Centrífuga a 125 x g por 5 min, descarte o supernatante e suspenda suavemente as células na PBS.
- Permita que as células descansem em suspensão por 30 min para garantir a recuperação da conformação da membrana.
- Usando um hemocímetro e o método de exclusão azul trypan, conte as células na suspensão.
- Ajuste o volume de suspensão celular para 106 células/500 μL.
- Incubar com os anticorpos monoclonais de acordo com as instruções dos fornecedores (concentração, tempo, temperatura e luz/escuro) e considerando o conjunto de experimentos representado sem tabela 2 ou os marcadores dados na Tabela 1.
- Imediatamente após a coloração, realize a análise citométrica de fluxo usando um citómetro de fluxo com módulos de detecção adequados.
- Padronizar a configuração do címetro, seguindo protocolos estabelecidos pelo Consórcio EuroFlow29.
- Coloque os portões primários baseados na dispersão dianteira e lateral, excluindo detritos e células mortas. Isso pode ser melhorado por rotulagem concomitante com anexarV e gating células negativas.
- Coloque os portões de fluorescência baseados nas amostras não manchadas e compensação por uma sobreposição espectral usando controles manchados únicos.
5. Avaliação do marcador de células-tronco do câncer com mancha ocidental
NOTA: Além da atividade aldh1, a alta expressão desse marcador foi consistentemente associada às células-tronco de câncer de mama e ginecológico13,14. O procedimento descrito pode ser usado para avaliar este e outros marcadores celulares.
- Após a conclusão do protocolo de formação da esfera, colete as esferas em um tubo de centrífuga e centrífuga a 125 x g por 5 min.
- Preparação de toda a célula lisates
- Coloque os tubos de centrífuga no gelo e descarte o supernatante sem interromper a pelota.
- Lave a pelota com 1 mL de PBS frio e descarte por centrífuga.
- Suspender a pelota em um pequeno volume (200-500 μL) de tampão de lise RIPA30 (NaCl 150 mM, Tris-HCl 1,50 mM pH 7.4, Triton-X100 1% vol./vol., ácido desoxicolic de sódio 0,5% wt./vol., sulfato dodecyl de sódio 0,5% wt./vol.) suplementado com cOmplete Mini e dithiothreitol 1 mM.
- Mantendo as amostras frias (no gelo), submeta-as ao vórtice e à sonsonação com uma amplitude de 30%.
- Centrífuga as amostras por 15 min a 14.000 x g em uma centrífuga refrigerada fixada em 4 °C.
- Transfira os supernatantes para microtubos novos e devidamente identificados.
- Determine as concentrações proteicas usando os ensaios BCA ou Bradford31.
- Se necessário, armazene as amostras a -80 °C até uma análise mais aprofundada da mancha ocidental.
- Realizar desnaturação amostral, eletroforese, transferência de elétrons e detecção de proteínas de acordo com protocolos padrão de manchas ocidentais, conforme descrito32,33,34.
Resultados
O protocolo de formação da esfera permite que colônias esféricas sejam obtidas em suspensão de várias linhas de células endómetrial e câncer de mama (Figura 2A) ou após a digestão enzimática suave de tecido de amostras de tumor humano (Figura 2E). Em ambos os casos, poucos dias após o revestimento, são obtidas colônias esféricas monoclonais em suspensão. Tanto as esferas endometrial quanto o câncer de mama dão...
Discussão
Este protocolo detalha uma abordagem para obter esferas tumoralais de linhas de células cancerosas e amostras humanas primárias. As esferas tumoralizadas são enriquecidas em uma subpopulação com propriedades semelhantes a células-tronco36. Esse enriquecimento no CSC depende da viabilidade em um ambiente livre de ancoragem, enquanto as células diferenciadas dependem da adesão a um substrato37. Como o revestimento primário das células tumorais em um ambiente de baix...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este estudo foi financiado pela Sociedade Portuguesa de Ginecologia através do Prêmio de Pesquisa de 2016 e da CIMAGO. Cnc. A IBILI é apoiada através da Fundação para ciência e tecnologia, Portugal (UID/NEU/04539/2013), e co-financiada pela FEDER-COMPETE (POCI-01-0145-FEDER-007440). O Banco tumordo do Hospital de Coimbra e do Centro Universitário (CHUC), aprovado pelo Comitê de Ética da CHUC para a Saúde e pela Comissão Nacional de Proteção de Dados de Coimbra, foi fonte de amostras endometrial de pacientes acompanhados no Serviço de Ginecologia da instituição. A Figura 1 foi produzida utilizando a Servier Medical Art, disponível a partir de www.servier.com.
Materiais
| Name | Company | Catalog Number | Comments |
| Absolute ethanol | Merck Millipore | 100983 | |
| Accutase | Gibco | A1110501 | StemPro Accutas Cell Dissociation Reagent |
| ALDH antibody | Santa Cruz Biotechnology | SC166362 | |
| Annexin V FITC | BD Biosciences | 556547 | |
| Antibiotic antimycotic solution | Sigma | A5955 | |
| BCA assay | Thermo Scientific | 23225 | Pierce BCA Protein Assay Kit |
| Bovine serum albumin | Sigma | A9418 | |
| CD133 antibody | Miteny Biotec | 293C3-APC | Allophycocyanin (APC) |
| CD24 antibody | BD Biosciences | 658331 | Allophycocyanin-H7 (APC-H7) |
| CD44 antibody | Biolegend | 103020 | Pacific Blue (PB) |
| Cell strainer | BD Falcon | 352340 | 40 µM |
| Collagenase, type IV | Gibco | 17104-019 | |
| cOmplete Mini | Roche | 118 361 700 0 | |
| Dithiothreitol | Sigma | 43815 | |
| DMEM-F12 | Sigma | D8900 | |
| DNAse I | Roche | 11284932001 | |
| ECC-1 | ATCC | CRL-2923 | Human endometrium adenocarcinoma cell line |
| Epidermal growth factor | Sigma | E9644 | |
| Fibroblast growth factor basic | Sigma | F0291 | |
| Haemocytometer | VWR | HERE1080339 | |
| HCC1806 | ATCC | CRL-2335 | Human mammary squamous cell carcinoma cell line |
| Insulin, transferrin, selenium Solution | Gibco | 41400045 | |
| MCF7 | ATCC | HTB-22 | Human mammary adenocarcinoma cell line |
| Methylcellulose | AlfaAesar | 45490 | |
| NaCl | JMGS | 37040005002212 | |
| Poly(2-hydroxyethyl-methacrylate | Sigma | P3932 | |
| Putrescine | Sigma | P7505 | |
| RL95-2 | ATCC | CRL-1671 | Human endometrium carcinoma cell line |
| Sodium deoxycholic acid | JMS | EINECS 206-132-7 | |
| Sodium dodecyl sulfate | Sigma | 436143 | |
| Tris | JMGS | 20360000BP152112 | |
| Triton-X 100 | Merck | 108603 | |
| Trypan blue | Sigma | T8154 | |
| Trypsin-EDTA | Sigma | T4049 | |
| ß-actin antibody | Sigma | A5316 |
Referências
- Hardin, H., Zhang, R., Helein, H., Buehler, D., Guo, Z., Lloyd, R. V. The evolving concept of cancer stem-like cells in thyroid cancer and other solid tumors. Laboratory Investigation. 97 (10), 1142 (2017).
- Plaks, V., Kong, N., Werb, Z. The Cancer Stem Cell Niche: How Essential Is the Niche in Regulating Stemness of Tumor Cells?. Cell Stem Cell. 16 (3), 225-238 (2015).
- Visvader, J. E., Lindeman, G. J. Cancer stem cells in solid tumours accumulating evidence and unresolved questions. Nature reviews. Cancer. 8, 755-768 (2008).
- Allegra, A., et al. The Cancer Stem Cell Hypothesis: A Guide to Potential Molecular Targets. Cancer Investigation. 32 (9), 470-495 (2014).
- Al-Hajj, M., Wicha, M. S., Benito-Hernandez, A., Morrison, S. J., Clarke, M. F. Prospective identification of tumorigenic breast cancer cells. Proceedings of the National Academy of Sciences. 100 (7), 3983-3988 (2003).
- Friel, A. M., et al. Functional analyses of the cancer stem cell-like properties of human endometrial tumor initiating cells. Cell Cycle. 7 (2), 242-249 (2008).
- Zhang, S., et al. Identification and Characterization of Ovarian Cancer-Initiating Cells from Primary Human Tumors. Cancer Research. 68 (11), 4311-4320 (2008).
- Bapat, S. A., Mali, A. M., Koppikar, C. B., Kurrey, N. K. Stem and progenitor-like cells contribute to the aggressive behavior of human epithelial ovarian cancer. Cancer research. 65 (8), 3025-3029 (2005).
- Carvalho, M. J., Laranjo, M., Abrantes, A. M., Torgal, I., Botelho, M. F., Oliveira, C. F. Clinical translation for endometrial cancer stem cells hypothesis. Cancer and Metastasis Reviews. 34 (3), 401-416 (2015).
- Morel, A. P., Lièvre, M., Thomas, C., Hinkal, G., Ansieau, S., Puisieux, A. Generation of Breast Cancer Stem Cells through Epithelial-Mesenchymal Transition. PLoS ONE. 3 (8), e2888 (2008).
- Tirino, V., et al. Cancer stem cells in solid tumors: an overview and new approaches for their isolation and characterization. The FASEB Journal. 27 (1), 13 (2013).
- Ajani, J. A., et al. ALDH-1 expression levels predict response or resistance to preoperative chemoradiation in resectable esophageal cancer patients. Molecular Oncology. 8 (1), 142-149 (2014).
- Carvalho, M. J., et al. Endometrial Cancer Spheres Show Cancer Stem Cells Phenotype and Preference for Oxidative Metabolism. Pathology and Oncology Research. , (2018).
- Laranjo, M., et al. Mammospheres of hormonal receptor positive breast cancer diverge to triple-negative phenotype. The Breast. 38, 22-29 (2018).
- Cui, M., et al. Non-Coding RNA Pvt1 Promotes Cancer Stem Cell–Like Traits in Nasopharyngeal Cancer via Inhibiting miR-1207. Pathology & Oncology Research. , (2018).
- Deng, S., et al. Distinct expression levels and patterns of stem cell marker, aldehyde dehydrogenase isoform 1 ALDH1), in human epithelial cancers. PloS one. 5 (4), e10277 (2010).
- Weiswald, L. B., Guinebretière, J. M., Richon, S., Bellet, D., Saubaméa, B., Dangles-Marie, V. In situ protein expression in tumour spheres: development of an immunostaining protocol for confocal microscopy. BMC Cancer. 10 (1), 106 (2010).
- Weiswald, L. B., Bellet, D., Dangles-Marie, V. Spherical Cancer Models in Tumor Biology. Neoplasia. 17 (1), 1-15 (2015).
- Picon-Ruiz, M., et al. Low adherent cancer cell subpopulations are enriched in tumorigenic and metastatic epithelial-to-mesenchymal transition-induced cancer stem-like cells. Scientific Reports. 6 (1), 1-13 (2016).
- Dontu, G., et al. In vitro propagation and transcriptional profiling of human mammary stem/progenitor cells. Genes & development. 17 (10), 1253-1270 (2003).
- Ballout, F., et al. Sphere-Formation Assay: Three-Dimensional in vitro Culturing of Prostate Cancer Stem/Progenitor Sphere-Forming Cells. Frontiers in Oncology. 8 (August), 1-14 (2018).
- Ishiguro, T., Ohata, H., Sato, A., Yamawaki, K., Enomoto, T., Okamoto, K. Tumor-derived spheroids: Relevance to cancer stem cells and clinical applications. Cancer Science. 108 (3), 283-289 (2017).
- Noseda, M., Nasatto, P., Silveira, J., Pignon, F., Rinaudo, M., Duarte, M. Methylcellulose, a Cellulose Derivative with Original Physical Properties and Extended Applications. Polymers. 7 (5), 777-803 (2015).
- Shaw, F. L., et al. A detailed mammosphere assay protocol for the quantification of breast stem cell activity. Journal of Mammary Gland Biology and Neoplasia. 17 (2), 111-117 (2012).
- Zhou, M., et al. LncRNA-Hh Strengthen Cancer Stem Cells Generation in Twist-Positive Breast Cancer via Activation of Hedgehog Signaling Pathway. Stem cells (Dayton, Ohio). 34 (1), 55-66 (2016).
- Ha, J. R., et al. Integration of Distinct ShcA Signaling Complexes Promotes Breast Tumor Growth and Tyrosine Kinase Inhibitor Resistance. Molecular cancer research MCR. 16 (5), 894-908 (2018).
- Jurmeister, S., et al. Identification of potential therapeutic targets in prostate cancer through a cross-species approach. EMBO molecular medicine. 10 (3), (2018).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature methods. 9 (7), 671-675 (2012).
- Kalina, T., et al. EuroFlow standardization of flow cytometer instrument settings and immunophenotyping protocols. Leukemia. 26 (9), 1986-2010 (2012).
- Peach, M., Marsh, N., Miskiewicz, E. I., MacPhee, D. J. . Solubilization of Proteins: The Importance of Lysis Buffer Choice. , 49-60 (2015).
- Olson, B. J. S. C. Assays for Determination of Protein Concentration. Current Protocols in Pharmacology. , A.3A.1-A.3A.32 (2016).
- Eslami, A., Lujan, J. Western Blotting: Sample Preparation to Detection. Journal of Visualized Experiments. (44), 1-2 (2010).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. Journal of Visualized Experiments. 84 (84), 1-8 (2014).
- Oldknow, K. J., et al. A Guide to Modern Quantitative Fluorescent Western Blotting with Troubleshooting Strategies. Journal of Visualized Experiments. 8 (93), 1-10 (2014).
- Serambeque, B. . Células estaminais do cancro do endométrio - a chave para o tratamento personalizado? [Stem Cells of Endometrial Cancer: The Key to Personalized Treatment?]. , (2018).
- Lee, C. H., Yu, C. C., Wang, B. Y., Chang, W. W. Tumorsphere as an effective in vitro platform for screening anti-cancer stem cell drugs. Oncotarget. 7 (2), (2015).
- De Luca, A., et al. Mitochondrial biogenesis is required for the anchorage-independent survival and propagation of stem-like cancer cells. Oncotarget. 6 (17), (2015).
- Masuda, A., et al. An improved method for isolation of epithelial and stromal cells from the human endometrium. Journal of Reproduction and Development. 62 (2), 213-218 (2016).
- Del Rio-Tsonis, K., et al. Facile isolation and the characterization of human retinal stem cells. Proceedings of the National Academy of Sciences. 101 (44), 15772-15777 (2004).
- Wang, L., Guo, H., Lin, C., Yang, L., Wang, X. I. Enrichment and characterization of cancer stem-like cells from a cervical cancer cell line. Molecular Medicine Reports. 9 (6), 2117-2123 (2014).
- Chen, Y. C., et al. High-throughput single-cell derived sphere formation for cancer stem-like cell identification and analysis. Scientific Reports. 6 (April), 1-12 (2016).
- Kim, J., Jung, J., Lee, S. J., Lee, J. S., Park, M. J. Cancer stem-like cells persist in established cell lines through autocrine activation of EGFR signaling. Oncology Letters. 3 (3), 607-612 (2012).
- Hwang-Verslues, W. W., et al. Multiple Lineages of Human Breast Cancer Stem/Progenitor Cells Identified by Profiling with Stem Cell Markers. PloS one. 4 (12), e8377 (2009).
- Feng, Y., et al. Metformin reverses stem cell-like HepG2 sphere formation and resistance to sorafenib by attenuating epithelial-mesenchymal transformation. Molecular Medicine Reports. 18 (4), 3866-3872 (2018).
- Wang, H., Paczulla, A., Lengerke, C. Evaluation of Stem Cell Properties in Human Ovarian Carcinoma Cells Using Multi and Single Cell-based Spheres Assays. Journal of Visualized Experiments. (95), 1-11 (2015).
- Stebbing, J., Lombardo, Y., Coombes, C. R., de Giorgio, A., Castellano, L. Mammosphere Formation Assay from Human Breast Cancer Tissues and Cell Lines. Journal of Visualized Experiments. (97), 1-5 (2015).
- Zhao, H., et al. Sphere-forming assay vs. organoid culture: Determining long-term stemness and the chemoresistant capacity of primary colorectal cancer cells. International Journal of Oncology. 54 (3), 893-904 (2019).
- Bagheri, V., et al. Isolation and identification of chemotherapy-enriched sphere-forming cells from a patient with gastric cancer. Journal of Cellular Physiology. 233 (10), 7036-7046 (2018).
- Kaowinn, S., Kaewpiboon, C., Koh, S., Kramer, O., Chung, Y. STAT1-HDAC4 signaling induces epithelial-mesenchymal transition and sphere formation of cancer cells overexpressing the oncogene, CUG2. Oncology Reports. , 2619-2627 (2018).
- Lonardo, E., Cioffi, M., Sancho, P., Crusz, S., Heeschen, C. Studying Pancreatic Cancer Stem Cell Characteristics for Developing New Treatment Strategies. Journal of Visualized Experiments. (100), 1-9 (2015).
- Lu, H., et al. Targeting cancer stem cell signature gene SMOC-2 Overcomes chemoresistance and inhibits cell proliferation of endometrial carcinoma. EBioMedicine. 40, 276-289 (2019).
- Bu, P., Chen, K. Y., Lipkin, S. M., Shen, X. Asymmetric division: a marker for cancer stem cells. Oncotarget. 4 (7), (2013).
- Islam, F., Qiao, B., Smith, R. A., Gopalan, V., Lam, A. K. Y. Cancer stem cell: fundamental experimental pathological concepts and updates. Experimental and molecular pathology. 98 (2), 184-191 (2015).
- Liu, W., et al. Comparative characterization of stem cell marker expression, metabolic activity and resistance to doxorubicin in adherent and spheroid cells derived from the canine prostate adenocarcinoma cell line CT1258. Anticancer research. 35 (4), 1917-1927 (2015).
- Broadley, K. W. R., et al. Side Population is Not Necessary or Sufficient for a Cancer Stem Cell Phenotype in Glioblastoma Multiforme. STEM CELLS. 29 (3), 452-461 (2011).
- Cojoc, M., Mäbert, K., Muders, M. H., Dubrovska, A. A role for cancer stem cells in therapy resistance: Cellular and molecular mechanisms. Seminars in Cancer Biology. 31, 16-27 (2015).
- Batlle, E., Clevers, H. Cancer stem cells revisited. Nature Medicine. 23 (10), 1124-1134 (2017).
- Zhang, X. L., Jia, Q., Lv, L., Deng, T., Gao, J. Tumorspheres Derived from HCC Cells are Enriched with Cancer Stem Cell-like Cells and Present High Chemoresistance Dependent on the Akt Pathway. Anti-cancer agents in medicinal chemistry. 15 (6), 755-763 (2015).
- Fukamachi, H., et al. CD49fhigh Cells Retain Sphere-Forming and Tumor-Initiating Activities in Human Gastric Tumors. PLoS ONE. 8 (8), e72438 (2013).
- Gao, M. Q., Choi, Y. P., Kang, S., Youn, J. H., Cho, N. H. CD24+ cells from hierarchically organized ovarian cancer are enriched in cancer stem cells. Oncogene. 29 (18), 2672-2680 (2010).
- Cariati, M., et al. Alpha-6 integrin is necessary for the tumourigenicity of a stem cell-like subpopulation within the MCF7 breast cancer cell line. International Journal of Cancer. 122 (2), 298-304 (2008).
- López, J., Valdez-Morales, F. J., Benítez-Bribiesca, L., Cerbón, M., Carrancá, A. Normal and cancer stem cells of the human female reproductive system. Reproductive Biology and Endocrinology. 11 (1), 53 (2013).
- Alvero, A. B., et al. Molecular phenotyping of human ovarian cancer stem cells unravels the mechanisms for repair and chemoresistance. Cell Cycle. 8 (1), 158-166 (2009).
- Charafe-Jauffret, E., Ginestier, C., Birnbaum, D. Breast cancer stem cells: tools and models to rely on. BMC Cancer. 9 (1), 202 (2009).
- Leccia, F., et al. ABCG2, a novel antigen to sort luminal progenitors of BRCA1- breast cancer cells. Molecular Cancer. 13 (1), 213 (2014).
- Croker, A. K., Allan, A. L. Inhibition of aldehyde dehydrogenase (ALDH) activity reduces chemotherapy and radiation resistance of stem-like ALDHhiCD44+ human breast cancer cells. Breast Cancer Research and Treatment. 133 (1), 75-87 (2012).
- Sun, M., et al. Enhanced efficacy of chemotherapy for breast cancer stem cells by simultaneous suppression of multidrug resistance and antiapoptotic cellular defense. Acta Biomaterialia. 28, 171-182 (2015).
- Shao, J., Fan, W., Ma, B., Wu, Y. Breast cancer stem cells expressing different stem cell markers exhibit distinct biological characteristics. Molecular Medicine Reports. 14 (6), 4991-4998 (2016).
- Croker, A. K., et al. High aldehyde dehydrogenase and expression of cancer stem cell markers selects for breast cancer cells with enhanced malignant and metastatic ability. Journal of Cellular and Molecular Medicine. 13 (8b), 2236-2252 (2009).
- Cheung, S. K. C., et al. Stage-specific embryonic antigen-3 (SSEA-3) and β3GalT5 are cancer specific and significant markers for breast cancer stem cells. Proceedings of the National Academy of Sciences. 113 (4), 960-965 (2016).
- Meyer, M. J., Fleming, J. M., Lin, A. F., Hussnain, S. A., Ginsburg, E., Vonderhaar, B. K. CD44 pos CD49f hi CD133/2 hi Defines Xenograft-Initiating Cells in Estrogen Receptor–Negative Breast Cancer. Cancer Research. 70 (11), 4624-4633 (2010).
- Ahn, S. M., Goode, R. J. A., Simpson, R. J. Stem cell markers: Insights from membrane proteomics?. PROTEOMICS. 8 (23-24), 4946-4957 (2008).
- Chefetz, I., et al. TLR2 enhances ovarian cancer stem cell self-renewal and promotes tumor repair and recurrence. Cell Cycle. 12 (3), 511-521 (2013).
- Alvero, A. B., et al. Stem-Like Ovarian Cancer Cells Can Serve as Tumor Vascular Progenitors. Stem Cells. 27 (10), 2405-2413 (2009).
- Yin, G., et al. Constitutive proteasomal degradation of TWIST-1 in epithelial–ovarian cancer stem cells impacts differentiation and metastatic potential. Oncogene. 32 (1), 39-49 (2013).
- Wei, X., et al. Mullerian inhibiting substance preferentially inhibits stem/progenitors in human ovarian cancer cell lines compared with chemotherapeutics. Proceedings of the National Academy of Sciences. 107 (44), 18874-18879 (2010).
- Meirelles, K., et al. Human ovarian cancer stem/progenitor cells are stimulated by doxorubicin but inhibited by Mullerian inhibiting substance. Proceedings of the National Academy of Sciences. 109 (7), 2358-2363 (2012).
- Shi, M. F., et al. Identification of cancer stem cell-like cells from human epithelial ovarian carcinoma cell line. Cellular and Molecular Life Sciences. 67 (22), 3915-3925 (2010).
- Meng, E., et al. CD44+/CD24− ovarian cancer cells demonstrate cancer stem cell properties and correlate to survival. Clinical & Experimental Metastasis. 29 (8), 939-948 (2012).
- Witt, A. E., et al. Identification of a cancer stem cell-specific function for the histone deacetylases, HDAC1 and HDAC7, in breast and ovarian. Oncogene. 36 (12), 1707-1720 (2017).
- Wu, H., Zhang, J., Shi, H. Expression of cancer stem markers could be influenced by silencing of p16 gene in HeLa cervical carcinoma cells. European journal of gynaecological oncology. 37 (2), 221-225 (2016).
- Huang, R., Rofstad, E. K. Cancer stem cells (CSCs), cervical CSCs and targeted therapies. Oncotarget. 8 (21), 35351-35367 (2017).
- Zhang, X., et al. Imatinib sensitizes endometrial cancer cells to cisplatin by targeting CD117-positive growth-competent cells. Cancer Letters. 345 (1), 106-114 (2014).
- Luo, L., et al. Ovarian cancer cells with the CD117 phenotype are highly tumorigenic and are related to chemotherapy outcome. Experimental and Molecular Pathology. 91 (2), 596-602 (2011).
- Zhao, P., Lu, Y., Jiang, X., Li, X. Clinicopathological significance and prognostic value of CD133 expression in triple-negative breast carcinoma. Cancer Science. 102 (5), 1107-1111 (2011).
- Ferrandina, G., et al. Expression of CD133-1 and CD133-2 in ovarian cancer. International Journal of Gynecologic Cancer. 18 (3), 506-514 (2008).
- Rutella, S., et al. Cells with characteristics of cancer stem/progenitor cells express the CD133 antigen in human endometrial tumors. Clinical cancer research an official journal of the American Association for Cancer Research. 15 (13), 4299-4311 (2009).
- Friel, A. M., et al. Epigenetic regulation of CD133 and tumorigenicity of CD133 positive and negative endometrial cancer cells. Reproductive Biology and Endocrinology. 8 (1), 147 (2010).
- Nakamura, M., et al. Prognostic impact of CD133 expression as a tumor-initiating cell marker in endometrial cancer. Human Pathology. 41 (11), 1516-1529 (2010).
- Saha, S. K., et al. KRT19 directly interacts with β-catenin/RAC1 complex to regulate NUMB-dependent NOTCH signaling pathway and breast cancer properties. Oncogene. 36 (3), 332-349 (2017).
- LV, X., Wang, Y., Song, Y., Pang, X., Li, H. Association between ALDH1+/CD133+ stem-like cells and tumor angiogenesis in invasive ductal breast carcinoma. Oncology Letters. 11 (3), 1750-1756 (2016).
- Ruscito, I., et al. Exploring the clonal evolution of CD133/aldehyde-dehydrogenase-1 (ALDH1)-positive cancer stem-like cells from primary to recurrent high-grade serous ovarian cancer (HGSOC). A study of the Ovarian Cancer Therapy–Innovative Models Prolong Survival (OCTIPS). European Journal of Cancer. 79, 214-225 (2017).
- Sun, Y., et al. Isolation of Stem-Like Cancer Cells in Primary Endometrial Cancer Using Cell Surface Markers CD133 and CXCR4. Translational Oncology. 10 (6), 976-987 (2017).
- Rahadiani, N., et al. Expression of aldehyde dehydrogenase 1 (ALDH1) in endometrioid adenocarcinoma and its clinical implications. Cancer Science. 102 (4), 903-908 (2011).
- Mamat, S., et al. Transcriptional Regulation of Aldehyde Dehydrogenase 1A1 Gene by Alternative Spliced Forms of Nuclear Factor Y in Tumorigenic Population of Endometrial Adenocarcinoma. Genes & Cancer. 2 (10), 979-984 (2011).
- Mukherjee, S. A., et al. Non-migratory tumorigenic intrinsic cancer stem cells ensure breast cancer metastasis by generation of CXCR4+ migrating cancer stem cells. Oncogene. 35 (37), 4937-4948 (2016).
- Lim, E., et al. Aberrant luminal progenitors as the candidate target population for basal tumor development in BRCA1 mutation carriers. Nature Medicine. 15 (8), 907-913 (2009).
- Liang, Y. J., et al. Interaction of glycosphingolipids GD3 and GD2 with growth factor receptors maintains breast cancer stem cell phenotype. Oncotarget. 8 (29), 47454-47473 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados