Method Article
Aplicación de células madre embrionarias haploides partegenéticas del ratón como sustituto de los espermatozoides
En este artículo
Resumen
Este artículo tiene como objetivo demostrar el uso de células madre embrionarias heploideas partefenogenéticas como sustituto de los espermatozoides para la construcción de embriones semi-clonados.
Resumen
En organismos con reproducción sexual, las células germinales son la fuente de células totipotentes que se convierten en nuevos individuos. En ratones, la fertilización de un ócito por un espermatozoide crea un cigoto totipotente. Recientemente, varias publicaciones han informado de que las células madre embrionarias haploides (haESC) pueden ser un sustituto de los genomas áticos y contribuir a los embriones, que se convierten en ratones. Aquí, presentamos un protocolo para aplicar haESC partenogenéticos como sustituto de los espermatozoides para construir embriones mediante inyección intracitoplasmática en ocitos. Este protocolo consiste en pasos para preparar los haESC como reemplazo de espermatozoides, para la inyección de cromosomas haESC en ocitos y para el cultivo de embriones semi-clonados. Los embriones pueden producir ratones semi-clonados fértiles después de la transferencia de embriones. El uso de haESCs como reemplazo de espermatozoides facilita la edición del genoma en la línea germinaria, los estudios de desarrollo embrionario y la investigación de la impresión genómica.
Introducción
En los mamíferos, los gametos son las únicas células que transmiten información genética a la próxima generación. La fusión de un ócito y un espermatozoide forma un cigoto diploide que se convierte en un animal adulto. Las mutaciones en genomas áticos son heredadas por la descendencia e impulsan la variación genética en la especie1. Se ha aplicado la introducción de mutaciones en la línea germinal para producir animales genéticamente modificados para diversos estudios biológicos, incluida la caracterización de la función génica y el modelado de enfermedades. Tanto los ocitos como los espermatozoides son células diferenciadas terminalmente y altamente especializadas que han dejado de proliferar. Por lo tanto, la modificación directa de gametos es técnicamente difícil, y se han desarrollado enfoques especializados. Las modificaciones genéticas pueden introducirse en la línea germinal del ratón mediante la inyección de ESC genéticamente modificados en blastocquistas, donde se integran en el embrión en desarrollo y colonizan la línea germinal. Además, la modificación genética de los cigotos utilizando enfoques de edición del genoma, incluido el sistema CRISPR/Cas9, se ha adoptado ampliamente2.
Recientemente, se ha notificado un enfoque excepcional, que aplica haESCs como sustituto de un genomagametic 3,4,5,6,7,8. Los haESC son líneas de células madre derivadas de la masa celular interna de blastocistos haploides partefenogenéticos o androgenéticos y poseen un solo conjunto de cromosomas4,7,9,10. Se ha demostrado que tanto los haESC partenogenéticos como los haESC androgenéticos pueden contribuir al genoma de ratones semi-clonados después de la inyección intracitoplasmática en ocitos. A diferencia de otros enfoques, los genomas de los haESC pueden modificarse directamente en el cultivo debido a su capacidad de autorretración. La introducción de modificaciones genéticas en la línea germinales mediante la sustitución de espermatozoides por haESC es un método importante para los estudios biológicos. Prevé la posibilidad de cultivo separado y manipular el genoma materno o paterno, que se derivan de haESCs partefenogenéticos o androgenéticos, respectivamente. A continuación, los HaESC se pueden utilizar como reemplazo del genoma ático, lo que es especialmente ventajoso para los estudios de la impresión genómica, la expresión específica del alelo y los procesos específicos de los padres.
En ratones, se requiere información genómica materna y paterna para el desarrollo normal del embrión11. Por lo tanto, los cachorros a término no pudieron obtenerse cuando se inyectaron haESC partenogenéticos de tipo salvaje (fasfascos) para reemplazar el genoma del esperma5,8. Para superar el bloque de desarrollo, es necesario corregir la impresión genómica del genoma materno de los haESC partefenogenéticos a una configuración paterna. Esto se puede lograr mediante la manipulación de las regiones metiladas diferencialmente (DMR). Hasta la fecha, se han estudiado eliminaciones específicas del H19-DMR, Gtl2-Dlk1 IG-DMR y Rasgrf1-DMR para reprimir los genes expresados maternamente en fases3,5,8,12. Estos estudios demostraron que las eliminaciones tanto del H19-DMR como del IG-DMR son suficientes para convertir a un materno en una configuración de impresión paterna que puede sustituir a los cromosomas de espermatozoides. La inyección intracitoplasmática de falanges que transportan las dos eliminaciones dmr en ocitos produjo cachorros semi-clonados con una frecuencia entre el 5,1% y el 15,5% de los embriones transferidos de 2 células.
Este protocolo se basa en la aplicación de fases con eliminaciones de H19-DMR e IG-DMR, a las que denominamos fases eliminatorias dobles (DKO-phaESCs). Proporcionamos instrucciones detalladas para la modificación de la impresión genómica en fases para establecer líneas DKO-phaESC, para la inyección de DKO-fasfas en ocitos como sustituto de un genoma de espermatozoides, para el cultivo de embriones semi-clonados a blastocistas y para la obtención de ratones semi-clonados. Este protocolo es una referencia para los investigadores que requieren una manipulación precisa y directa del genoma paterno y la generación de embriones y ratones semi-clonados.
Protocolo
Todos los experimentos con animales se realizaron bajo la licencia ZH152/17 de acuerdo con las normas y reglamentos de la Comisión Cantonal de Ética de Zúrich y la instalación animal EPIC en el Instituto de Ciencias de la Salud Molecular, ETH Zurich.
NOTA: Este protocolo comienza con la eliminación de los H19- y IG-DMR en los phaESC. Para obtener más información sobre cómo establecer líneas phaESC, consulte los informes publicados10,13. En la Figura 1A se proporciona una visión general y un marco temporal de este protocolo (pasos 1–14); medios, soluciones y búferes se enumeran en la Tabla 1. El procedimiento para establecer líneas DKO-phaESC (pasos 1–6) se muestra en la Figura 1B,y la estrategia para la construcción de embriones semi-clonados (pasos 7–14) se representa en la Figura 1C.
1. Transfección de plásmidos para la eliminación de H19-DMR e IG-DMR en fases
- Prepare plásmidos CRISPR/Cas9 para la coexpresión de las nucleases Cas9 y guíe a los ANR para dirigirse a las eliminaciones de H19-DMR e IG-DMR. Ligar cuatro pares de oligos de ARN guía (H19-DMR-gRNA1-F, R; H19-DMR-gRNA2-F, R; IG-DMR-gRNA1-F, R; IG-DMR-gRNA2-F, R enumerada en la Tabla 2) en plásmidos pX330.
NOTA: Consulte el protocolo publicado sobre el procedimiento detallado para la preparación de estos 4 plásmidos CRISPR/Cas914. Alternativamente, los plásmidos disponibles para cepas generales de ratón también son accesibles a través de un repositorio (Tabla de Materiales). - Cubra la superficie de un pozo de una placa de 6 pozos con 1 ml de solución de gelatina al 0,2% mediante incubación a 37 °C durante 10 minutos.
- Placa 2 × 105 fasfas de tipo salvaje en el pozo recubierto de gelatina en medio haESC sin antibióticos, e incubar la placa a 37 °C en una atmósfera de CO2 del 5% durante 1 día.
NOTA: Los antibióticos se omiten del medio para aumentar la eficiencia de la lipofección posterior. Usamos fasfas de tipo salvaje en el paso 10. Recomendamos utilizar fases de paso temprano, pero se ha utilizado con éxito una variedad de números de pasaje8. Actualmente se desconoce la correlación entre el número de pasaje y la eficiencia de la obtención de embriones y ratones semi-clonados. - Fases de transfectos en el pozo de una placa de 6 pozos (desde el paso 1.3) con 6 plásmidos simultáneamente utilizando reactivo de lipofección: plásmido piggyBac de 50 ng que transporta un transgénero CAG-EGFP, Plásmido transposasa 50 ng piggyBac y 600 ng de cada uno de los 4 plásmidos CRISPR/Cas9 (del paso 1.1). Consulte el protocolo del fabricante sobre el procedimiento detallado de la transfección.
NOTA: Dos plásmidos piggyBac se utilizan para integrar un transposon para la expresión omnipresente de proteína fluorescente verde mejorada (EGFP) en el genoma de los phaESC. Si no se requiere el marcado GFP de las células, estos dos plásmidos pueden ser sustituidos por un plásmido CRISPR/Cas9 para la expresión transitoria de proteínas de fluorescencia (por ejemplo, plásmido pX458) en lugar de uno de los plásmidos pX330. La expresión EGFP transitoria se puede utilizar para ordenar celdas transfectadas. - Dos días después de la transfección, aspirar el medio, y añadir 800 μL de trippsina.
- Incubar la placa a 37 °C en una atmósfera de CO2 del 5% durante 5 minutos. A continuación, agregue 2 ml de búfer de lavado para apagar la trypsin y pipetear varias veces para obtener una sola suspensión celular.
- Transfiera la suspensión celular a un tubo de 15 ml.
- Centrífuga el tubo a 160 x g durante 5 minutos, y retire el sobrenadante.
- Resuspend el pellet celular en 400 μL de búfer de mantenimiento haESC complementado con 15 μg/mL Hoechst 33342.
NOTA: Para reducir la toxicidad potencial de Hoechst 33342, se han utilizado 1 μg/mL Hoechst 33342 y 50 μM verapamil en lugar de 15 μg/mL Hoechst 3334215. - Incubar la suspensión celular a 37 °C en una atmósfera de CO2 del 5% durante 15 min. Después de la incubación, transfiera la suspensión celular a un tubo de 5 ml a través de una tapa del colador celular y mantenga el tubo a 4 °C hasta que esté listo para usarse en el siguiente paso (sección 2).
2. Chapado de una sola célula de fases transfectadas utilizando un citómetro de flujo
- Un día antes de ordenar los falosescos transfectados, placa irradiado fibroblastos embrionarios de ratón (MEF) en placas de 96 pozos recubiertas de gelatina a una densidad de 4 × 104 células/cm2 en medio MEF. Normalmente, 6 placas están preparadas para establecer una línea phaESC con eliminaciones específicas. Incubar las placas a 37 °C en una atmósfera de CO2 del 5%.
NOTA: Los MEF irradiados están disponibles comercialmente. Utilizamos MEF derivados de embriones E12.5 de ratones DR4. Aunque los haESC pueden crecer en placas recubiertas de gelatina sin MEF, recomendamos mefs para aumentar la viabilidad de los haESC individuales ordenados. - El día de la clasificación, aspirar el medio MEF de las placas de 96 pozos, y añadir 120 μL de haESC fresco medio por pozo. Mantenga las placas a 37 °C en una atmósfera de CO2 del 5%.
- Configure una clasificador de celdas con una boquilla de 100 μm de acuerdo con las instrucciones del fabricante. Un láser UV de 355 nm y un láser azul de 488 nm se utilizan para la excitación de hoechst 33342 y fluorescencia EGFP, respectivamente.
NOTA: Alternativamente, Hoechst 33342 se puede detectar por excitación con 405 nm. - Ordene las fases transfectadas en el tubo de 5 ml (desde el paso 1.10) utilizando una puerta para recoger células haploides en la fase G1/S que muestran la expresión EGFP. Deposite una sola celda en cada pozo de las placas de 96 pozos desde el paso 2.2.
NOTA: La detección de la tinción de Hoechst 33342 generalmente distingue 3 picos de células con un contenido de ADN de 1n, 2n y 4n, que corresponden a células haploides en fase G1, una mezcla de células haploides en células de fase G2/M y diploides en fase G1, y células diploides en fase G2/M, respectivamente. Las células haploides en fase G1/S se identifican como el pico con menor intensidad de fluorescencia Hoechst 33342. Un resultado representativo y una puerta de ordenación se muestran en la figura 2A. - Después del chapado, incuba las placas de 96 pozos a 37 °C en una atmósfera de CO2 al 5%.
3. Subagulación de fases transfectadas
- Tres días después del chapado de una sola célula, las colonias se pueden observar en varios pozos de las placas de 96 pozos bajo un microscopio. Marca los pozos en los que sólo crecen las colonias individuales.
NOTA: En nuestra experiencia, se observaron colonias individuales en el 20%-40% de los pozos de las placas de 96 pozos. - El día 4 después del chapado unicelular, reemplace la mitad del medio por un nuevo medio haESC en los pozos por colonias individuales.
- Un día antes de pasar (en el día 4 o 5 después del chapado de una sola célula), placa irradiada MEF en placas de 96 pozos recubiertas de gelatina a una densidad de 4 × 104 celdas/cm2 en medio MEF. Mantenga las placas a 37 °C en una atmósfera de CO2 del 5%.
- Después de 5 o 6 días de chapado de una sola célula, seleccione los pozos de las placas de 96 pozos que contienen colonias individuales con un diámetro superior a 150 μm.
NOTA: Se seleccionan aproximadamente 100 pozos preferiblemente para establecer una línea phaESC con las eliminaciones dmr específicas. - Aspirar el medio en los pozos seleccionados y añadir 30 μL de trippsina. Incubar las placas de 96 pozos a 37 °C en una atmósfera de CO2 del 5% durante 5 minutos. A continuación, agregue 30 μL de tampón de lavado a cada pozo para apagar la trypsina.
- Agregue 140 μL de medio haESC en cada pozo, y pipeta varias veces para obtener células individuales.
- Aspirar el medio MEF de los pozos de las placas de 96 pozos preparadas en el paso 3.3.
- Transfiera las fases de cada pozo desde el paso 3.6 a un pozo fresco de la nueva placa de 96 pozos desde el paso 3.7.
- Incubar las placas de 96 pozos que contienen subclones phaESC a 37 °C en una atmósfera de CO2 del 5%.
- Al día siguiente, aspirar todo el medio de cada pozo, y añadir 120 μL de nuevo medio haESC. Incubar las placas a 37 °C en una atmósfera de CO2 del 5%.
- Un día antes de que las células se vuelvan confluentes para pasar, platee los MEF irradiados en placas de 24 pozos recubiertas de gelatina a una densidad de 4 × 104 células/cm2 en medio MEF. Mantenga las placas a 37 °C en una atmósfera de CO2 del 5%.
- Cuando los phaESC se hayan vuelto confluentes para el passaging, aspirar el medio y añadir 30 μL de trippsina. Incubar las placas de 96 pozos a 37 °C durante 5 minutos.
- Agregue 90 μL de búfer de lavado en cada pozo para apagar la trypsina. Pipeta varias veces para obtener células individuales.
- Aspirar el medio MEF de los pozos de las placas de 24 pozos desde el paso 3.11, y añadir 600 μL de haESC fresco medio.
- Transfiera 60 μL de la suspensión de subclones phaESC de cada pozo en el paso 3.13 a un nuevo pozo de las placas de 24 pozos desde el paso 3.14. Mantenga la suspensión restante de cada subclone phaESC para la extracción de ADN y genotipado en el paso 4.
- Incubar las placas de 24 pozos con las subclones phaESC a 37 °C en una atmósfera de CO2 del 5%.
- Un día antes de que los cultivos celulares alcancen la densidad para pasar, platee los MEF irradiados en placas de 6 pozos recubiertas de gelatina a una densidad de 4 × 104 células/cm2 en medio MEF. Mantenga las placas a 37 °C en una atmósfera de CO2 del 5%.
- Cuando los phaESC se vuelvan lo suficientemente confluentes para el passaging, aspirar el medio y añadir 250 μL de trippsina. Incubar las placas de 24 pozos a 37 °C durante 5 minutos.
NOTA: Después del genotipado en el paso 4.9, solamente las líneas phaESC con las eliminaciones del H19-DMR y el IG-DMR necesitan ser pasadas. - Agregue 750 μL de tampón de lavado en cada pozo para saciar la trypsina. Pipeta varias veces para obtener una sola suspensión celular. Transfiera la suspensión celular a un tubo de 15 ml.
- Centrífuga el tubo a 160 x g durante 5 minutos, retire el sobrenadante y resuspend el pellet celular en 2 ml de medio haESC.
- Aspirar el medio MEF desde los pozos de las placas de 6 pozos desde el paso 3.17. Transfiera la suspensión phaESC de cada tubo del paso 3.20 a un nuevo pozo. Mantenga las placas a 37 °C en una atmósfera de CO2 del 5%.
- Expanda los clones celulares repitiendo los pasos 3.17 a 3.21 y aumentando el tamaño de la placa y los volúmenes de trypsin, buffer de lavado y medio haESC. Prepare un matraz T25 para cada línea phaESC sub-clonada de MEF para el paso 5.
NOTA: Recomendamos congelar una coartada de cada línea phaESC sub-clonada en 300 μL de medio de congelación y mantener un criostock en el almacenamiento de nitrógeno líquido antes de proceder al paso 5.
4. Primer genotipado de líneas phaESC sub-clonadas con MEF
- Para extraer ADN genómico de la suspensión celular restante del paso 3.15, agregue 200 μL de tampón de lisis a cada pozo de las placas de 96 pozos. Transfiera la suspensión celular a un tubo de 1,5 ml. Enjuague cada pozo con 200 μL adicionales de tampón de lisis para recuperar todas las células restantes y recoger en el mismo tubo de 1,5 ml.
- Incubar el tubo de 1,5 ml a 55 °C durante 3 h con mezcla.
- Después de la incubación, añadir 460 μL de isopropanol a cada tubo de 1,5 ml, y mezclar suavemente hasta que un precipitado de ADN se haga visible.
- Centrífuga los tubos a ≥ 10.000 x g durante 5 minutos y retire el sobrenadante. Lave los pellets de ADN con 200 μL de etanol al 70%.
- Centrífuga los tubos a ≥ 10.000 x g durante 5 minutos y retire el sobrenadante.
- Seque los tubos en aire durante 10 minutos y luego resuspend el ADN en 20 μL de agua.
- Realice la reacción en cadena de la polimerasa (PCR) utilizando la polimerasa de ADN termoestable siguiendo el protocolo del fabricante.
NOTA: Los pares de imprimación para PCR se enumeran en la Tabla 2 y se utilizan de la siguiente manera: H19-DMR-P1 y P3 (407 bp para H19-DMR eliminado); H19-DMR-P2 y P3 (623 bp para wildtype H19-DMR); IG-DMR-P1 y P3 (319 bp para IG-DMR eliminado); IG-DMR-P2 y P3 (492 bp para wildtype IG-DMR). El perfil de temperatura de PCR para todos los pares de imprimación es el siguiente: 30 s 98 °C, 35 x (10 s 98 °C, 20 s 56 °C, 30 s 72 °C), 5 min 72 °C. La longitud de los fragmentos de ADN amplificados para las eliminaciones H19-DMR e IG-DMR muestra cierta variación debido a la unión final no homóloga asociada con la edición mediada por CRISPR/Cas9. Los FADC fueron cultivados con MEF, que contienen el wildtype H19-DMR y IG-DMR DNA. Por lo tanto, los pares de imprimación de H19-DMR-P2/P3 e IG-DMR-P2/P3, que amplifican fragmentos de ADN de tipo salvaje, no son informativos. Sin embargo, estos pares de imprimación se incluyen como controles y deben dar una banda en todas las reacciones. - Analice los fragmentos de PCR mediante electroforesis de gel de agarose. Consulte el protocolo publicado sobre el procedimiento detallado de la electroforesis16.
- Identifique las líneas celulares con las eliminaciones de H19-DMR y IG-DMR. Una imagen representativa de la electroforesis se muestra en la Figura 2B.
NOTA: En nuestro caso, se identificaron ocho líneas celulares con eliminaciones de H19-DMR e IG-DMR entre 135 líneas phaESC sub-clonadas.
5. Purificación celular haploide de líneas phaESC sub-clonadas
- Cuando los cultivos phaESC sub-clonados en los matraces T25 del paso 3.22 se vuelven lo suficientemente densos como para pasar, aspirar el medio y añadir 1,5 ml de trypsin. Incubar el matraz a 37 °C durante 5 min. A continuación, agregue 4,5 ml de tampón de lavado y pipeta varias veces para obtener una suspensión de una sola celda.
- Transfiera cada suspensión celular a un tubo de 15 ml y centrífuga el tubo a 160 x g durante 5 minutos. Retire el sobrenadante y resuspend los pellets celulares en 400 μL de búfer de mantenimiento haESC complementado con 15 μg/mL Hoechst 33342.
- Incubar las suspensiones celulares a 37 °C durante 15 min. Después de la incubación, transfiera las suspensiones celulares a un tubo de 5 ml a través de una tapa de colador celular. Enjuague la tapa del colador celular con 400 μL adicionales de búfer de mantenimiento haESC y recoja las células restantes en el mismo tubo de 5 ml. Mantenga el tubo a 4 °C hasta que esté listo para ordenarse.
- Configure un cytómetro de flujo con una boquilla de 100 μm de acuerdo con las instrucciones del fabricante.
NOTA: Hoechst 33342 se puede detectar por excitación a 405 nm. Aquí, se utiliza un láser UV de 355 nm para la detección de Hoechst 33342. - Configure la suspensión celular (desde el paso 5.3) y un nuevo tubo de 15 ml que contenga 2 ml de búfer de mantenimiento haESC para recoger células ordenadas en el cytómetro de flujo.
- Inicie el análisis y configure la puerta de clasificación para recoger celdas haploides en la fase G1/S. Refiera al histograma en la Figura 2A para identificar la población de fase G1/S phaESC.
NOTA: Algunas líneas phaESC sub-clonadas pueden no contener ninguna celda haploide debido a la diploidización completa o el a cuadro erróneo de células diploides en el paso 2. Si no se observan células haploides en la fase G1/S, proceda a otra muestra sin ordenar. En nuestro caso, 5 líneas celulares contenían células haploides y 3 líneas celulares contenían solo células diploides de 8 líneas phaESC sub-clonadas. - Después de la clasificación celular, agregue 5 ml de búfer de lavado a lo largo de la pared del tubo de recolección de 15 ml. Centrífuga el tubo a 160 x g durante 5 minutos. Retire el sobrenadante.
- Seleccione una placa de tamaño adecuado para la culturing dependiendo del número de celdas ordenadas. Utilice un solo pozo de una placa de 96 pozos, una placa de 24 pozos y una placa de 12 pozos para cultivo de 1.000-40.000 células, 40.000-200.000 células y 200.000-400.000 células, respectivamente. Resuspend el pellet celular en 120 μL, 600 μL y 1,2 ml de haESC medio, respectivamente.
NOTA: Platee las células a alta densidad porque la baja confluencia puede causar la muerte celular de las células después de la clasificación. A partir de este momento, los phaESC se cultivan en pozos recubiertos de gelatina sin MEF para facilitar el genotipado en el paso 6 y la posterior aplicación de inyección intracitoplasmática a partir del paso 9. - Después de transferir la suspensión celular a un pozo recubierto de gelatina del tamaño adecuado, incubar la placa a 37 °C en una atmósfera de CO2 del 5%.
- Continúe expandiendo los cultivos phaESC repitiendo los pasos 3.18 a 3.21 con el aumento de los tamaños de placa y el aumento de los volúmenes de trypsina, tampón de lavado y medio haESC. Las células se cultivan en pozos recubiertos de gelatina sin MEF.
- Para cada línea phaESC sub-clonada, prepare una cultura en un pozo de una placa de 24 pozos y un pozo de una placa de 6 pozos para los pasos 6 y 9, respectivamente.
NOTA: Algunas células de cada línea phaESC sub-clonada deben congelarse en 300 μL de medio de congelación como criostock en un tanque de almacenamiento de nitrógeno líquido antes de proceder al paso 9.
6. Segundo genotipado de líneas phaESC sub-clonadas sin MEF
NOTA: Se realiza una segunda ronda de genotipado para confirmar que las líneas phaESC sub-clonadas poseen eliminaciones de los DMR H19- e IG- , y que los alelos de wildtype están ausentes después de la eliminación de los MEF.
- Confirme bajo el microscopio que los cultivos en pozos de las placas de 24 pozos del paso 5.11 están libres de MEF.
NOTA: Si se observan MEF, es necesario seguir pasando las culturas hasta que los MEF hayan desaparecido para evitar contaminar el PCR con ADN de tipo salvaje de los MEF. - Aspirar el medio de cultivos confluentes y añadir 400 μL de tampón de lisis por pozo de la placa de 24 pozos. Después de pipetear varias veces, transfiera la suspensión celular a un tubo de 1,5 ml.
- Incubar el tubo de 1,5 ml a 55 °C durante 3 h con mezcla.
- Después de la incubación, añadir 400 μL de isopropanol al tubo de 1,5 ml, y mezclar suavemente hasta que un precipitado de ADN se haga visible.
- Centrífuga el tubo a ≥ 10.000 x g durante 5 minutos y retire el sobrenadante. Lave el pellet de ADN con 200 μL de etanol al 70%.
- Centrífuga el tubo a ≥ 10.000 x g durante 5 minutos y retire el sobrenadante.
- Seque el tubo en aire durante 10 minutos y luego resuspend el ADN en 50 μL de agua.
- Realice el PCR genotipado siguiendo el paso 4.7 y gel electroforesis en el paso 4.8 para identificar las líneas celulares, que poseen eliminaciones de los H19- y IG- DMR y están libres de alelos de wildtype.
NOTA: Una imagen de un segundo análisis genotipado típico se muestra en la Figura 2B como referencia. En nuestro caso, las 5 líneas celulares seleccionadas después de la purificación celular haploide (paso 5) estaban libres de alelos de tipo salvaje y poseían sólo los alelos de eliminación de los H19- y IG-DMR. - Utilice las líneas phaESC sub-clonadas seleccionadas después de este segundo genotipado como líneas DKO-phaESC.
7. Superovulación de ratones
- Para la producción de ocitos MII, inicie la superovulación mediante inyección intraperitoneal de 5 UI de solución de gonadotropina sérica de yegua embarazada (PMSG) en cada ratón hembra B6D2F1 (4-5 semanas de edad) 63-65 h antes de la recolección de oocitos.
NOTA: La cepa del ratón B6D2F1 se recomienda para este protocolo porque los ocitos B6D2F1 toleran bien la inyección intracitoplasmática y muestran un alto potencial de desarrollo después del procedimiento17. - Cuarenta y ocho horas después de la inyección de PMSG, inyectar por vía intraperitoneally 5 UI de solución de gonadotropina coriónica humana en cada ratón.
8. Colección de ocitos
- Prepare una placa de 4 pozos que contenga 700 μL de medio hialuronidasa en un pozo y 700 μL de M2 en los 3 pozos restantes. Además, prepara un plato de 6 cm con 7 ml de M2 medio y un plato de centro-pozo con 900 μL de M16 medio. Precalente el plato y los platos a 37 °C en un ambiente de 5% CO2.
- El día de la inyección intracitoplasmática, eutanasia a las hembras superovuladas (desde el paso 7.2) por dislocación cervical o inhalación de CO2 alrededor de las 8 de la mañana. Disecciona los oviductos usando pinzas y tijeras. Coloque los oviductos en el plato de 6 cm con el medio M2.
- Suelte los complejos de cúmulos-ócitos (COC) desgarrando la ampolla de los oviductos con una aguja de 30 G. Transfiera los COC al medio hialuronidase precalentado y manténgalo a 37 °C en una atmósfera de CO2 del 5%.
- Después de 2-3 min, recoger ócitos libres de cúmulos con una pipeta bucal y lavar los ócitos 3 veces transfiriéndolos al medio fresco M2 en los otros 3 pozos de la placa de 4 pozos.
- Recoge ocitos metafásicos II (MII), que poseen los primeros cuerpos polares, en un plato de centro-pozo con medio M16 y mantén el plato a 37 °C en una atmósfera de CO2 del 5% hasta su uso para inyección intracitoplasmática en el paso 12.
9. Tratamiento y recopilación de DKO-phaESCs
- Prepare un cultivo DKO-phaESC en un pozo de una placa de 6 pozos sin MEF al 60-80% de confluencia un día antes de la inyección intracitoplasmática (desde el paso 5.11).
- Para inducir la detención del ciclo celular en fase M, aspirar el medio por completo y añadir 2 ml de medio haESC que contiene 0,05 mg/ml demecolcina.
- Después de 8 h de tratamiento con demecolcina, aspirar el medio y añadir 800 μL de trippsina.
- Incubar la placa a 37 °C en una atmósfera de CO2 del 5% durante 5 minutos, luego añadir 2 ml de tampón de lavado para apagar la triptina, y pipeta varias veces para obtener una suspensión de una sola célula.
- Transfiera la suspensión celular a un tubo de 15 ml. Centrífuga el tubo a 160 x g durante 5 minutos y retire el sobrenadante.
- Resuspend el pellet celular en 400 μL de búfer de mantenimiento haESC que contiene 15 μg/mL Hoechst 33342.
- Incubar la suspensión celular a 37 °C en una atmósfera de CO2 del 5% durante 15 min. Después de la incubación, transfiera la suspensión celular a un tubo de 5 ml a través de una tapa del colador celular y mantenga el tubo a 4 °C hasta que la clasificación de la célula en el paso 10.
10. Purificación de DKO-phaESCs detenidos en fase M
- Configure un cytómetro de flujo con una boquilla de 100 μm de acuerdo con las instrucciones del fabricante.
NOTA: Hoechst 33342 se puede detectar por excitación a 405 nm. Aquí, se utiliza un láser UV de 355 nm para la detección de Hoechst 33342. - Establecer el DKO-phaESCs detenido en fase M a partir del paso 9.7 e iniciar el análisis. Seleccione una puerta de clasificación adecuada para recoger las células de fase M haploides (2n) de la muestra tratada con demecolcina.
NOTA: Después del tratamiento de la demecolcina, se esperan 2 poblaciones celulares, correspondientes a células haploides y diploides detenidas en fase M como se muestra en la Figura 3B. La detención del ciclo celular después del tratamiento con demecolcina está completa, por lo tanto, no se observa ningún pico de ADN haploide 1n. Esto es importante ya que las células haploides de fase M y las células diploides G1 poseen el mismo contenido de ADN (2n) y producirían picos superpuestos. - Configure un tubo de 15 ml que contenga 2 ml de búfer de mantenimiento haESC para recoger las celdas ordenadas en el cytómetro de flujo. Inicie la clasificación de celdas.
- Después de la clasificación celular, agregue 5 ml de búfer de lavado a lo largo de la pared del tubo de recolección. Centrífuga el tubo a 160 x g durante 5 minutos y retire el sobrenadante.
- Resuspend las células en un volumen adecuado de búfer de mantenimiento haESC para obtener una concentración final de 5 x 105 celdas/ml.
- Transfiera la suspensión celular a un tubo de 1,5 ml. Mantenga el tubo sobre hielo hasta que esté listo para la inyección intracitoplasmática en el paso 12.
11. Preparación de pipetas de sujeción y microinjección
NOTA: Para realizar la inyección intracitoplasmática (paso 12), se requieren varias pipetas de sujeción y microinyección(Figura 4A). Estas pipetas se pueden comprar a petición a medida de un proveedor comercial o hechas de capilares de vidrio adecuados utilizando un extractor de micropipette y un microforge.
- Tire de capilares de vidrio borosilicato en un extractor de micropipette. Para extraer capilares de vidrio borosilicato sin filamento (0,78 x 1,00 x 80 mm) se indican los siguientes parámetros para un extractor horizontal en llamas(Tabla de Materiales)como referencia, pero diferirán para otros instrumentos y tipos capilares de vidrio: Calor 510 (Prueba de rampa 480), Extracción 0, Velocidad 150, Tiempo 175 y Presión 200 para la sujeción de pipetas; Heat 510 (Prueba de rampa 480), Extracción 90, Velocidad 140, Tiempo 125 y Presión 200 para pipetas de microinjeción.
NOTA: Los parámetros óptimos deben definirse individualmente porque varios factores, incluyendo la humedad, el modelo de un tirador de micropipette y la gran cantidad de capilares de vidrio pueden afectar la forma de las pipetas de inyección. Se debe apuntar a una forma alargada con una inclinación gradual. - Preparación de la celebración de pipetas
- Ajuste un capilar tirado preparado en el paso 11.1 en un microforge. Coloque el capilar sobre el cordón de vidrio en el filamento y baje el capilar para hacer un contacto con la cuenta de vidrio mientras calienta el filamento.
- Romper el capilar apagando el calentamiento y desprendiéndose de la cuenta de vidrio de tal forma que su diámetro exterior es de 60-100 μm.
- Coloque la punta capilar horizontalmente para enfrentarse al cordón de vidrio en el filamento.
- Caliente el filamento para permitir que el diámetro interno de la punta capilar se derrita a un diámetro de 10-20 μm.
- Mueva el capilar para que las posiciones de cordón de vidrio en un punto ~ 1 mm de la punta capilar sin contacto. Caliente el filamento para permitir que el capilar se doble en un ángulo de 20°. Desmontar el capilar, denominado pipeta de sujeción, del microforge.
NOTA: Para medir el tamaño del capilar, preferiblemente se instala un retícula ocular en el microforge.
- Preparación de pipetas de microinjección
- Ajuste un capilar tirado preparado en el paso 11.1 en un microforge. Coloque el capilar sobre el cordón de vidrio en el filamento y baje el capilar para hacer un contacto con la cuenta de vidrio mientras calienta el filamento.
- Rompa el capilar apagando el calentamiento y desprendiéndose de la cuenta de vidrio en una posición en la que su diámetro exterior es de 6 μm.
- Mueva el capilar para que las posiciones de cordón de vidrio en un punto ~ 1 mm de la punta capilar sin contacto. Caliente el filamento para permitir que el capilar se doble hacia arriba en un ángulo de 20°. Desmontar la pipeta de microinjección del microforge y almacenarla en una caja segura para su uso posterior.
NOTA: Las pipetas de microinjección se preparan con las siguientes especificaciones: diámetro exterior, 6 μm; diámetro interior, 4,5–5 μm; ángulo de flexión, 20°. Definir el diseño óptimo de la pipeta de microinjección es importante para el éxito de la inyección intracitoplasmática. Un diámetro interno demasiado grande puede evitar la rotura de la membrana plasmática del donante DKO-phESCs (ver sección de discusión). Si el diámetro interno es demasiado estrecho, puede impedir la pipeteo suave del donante DKO-phESCs. Un ángulo de pliegue < 30° es preferible ya que un ángulo de pliegue alto impide el efecto de los pulsos piezo.
12. Inyección intracitoplasmática de DKO-phaESCs
- Antes de la inyección intracitoplasmática (a partir del paso 12.2), prepare una solución de polivinilpirrolidona (PVP) añadiendo 5 ml de medio M2 en un tubo de 50 ml que contenga 0,6 g de PVP y agitando el tubo en un balancín a 4 °C durante 2 días. Después de que el PVP se haya disuelto por completo, la solución se filtra estérilmente y se almacena a 4 °C.
- El día de la inyección intracitoplasmática, prepara un plato de pozo central con 900 μL de KSOM medio y pre-calienta el plato a 37 °C en una atmósfera de CO2 al 5%.
- Prepare un plato de micromanipulación alineando gotas de 5 μL de solución PVP y 20 μL de medio M2 en una tapa de un plato de 10 cm que se coloca al revés. Cubra las gotas con aceite mineral y coloque el plato en el escenario del microscopio de inyección.
NOTA: La disposición recomendada de la antena de micromanipulación se muestra en la Figura 4B. - Instale una pipeta de sujeción en el micromanipulador. Llene la pipeta de microinjeción con el aceite de fluorocarbono mediante una punta de microcargador y colócela en el actuador piezo.
- Sumerja la pipeta de microinjeción en una gota con solución PVP y pipeta arriba y abajo varias veces para recubrir el vidrio con PVP y hacerlo menos pegajoso. Cargue un pequeño volumen de solución PVP en la pipeta de microinyección y mueva la pipeta a una gota con el medio M2.
- Sumerja la pipeta de sujeción en el medio M2 y concéntrese en la pipeta en la parte inferior de la gota.
- Transfiera aproximadamente 2 μL de suspensión DKO-phaESC del paso 10.6 a la caída media M2.
- Transfiera 10 ócitos MII del paso 8.5 a la misma caída mediana M2 mediante una pipeta bucal.
- Para inyección, gire un ócito en la caída media M2 para que el espacio perivitellina se enfrente a la pipeta de microinjección, y la placa MII no se encuentra en el camino de la pipeta de microinjección (Figura 4A). Sujete el ócito aplicando presión negativa a través de la pipeta de sujeción.
NOTA: Una placa MII se identifica visualmente como una protuberancia de ooplasma que se conoce como una "joroba" y a menudo se encuentra junto al primer cuerpo polar. La placa MII contiene el husillo meiotico con cromosomas unidos. El contacto de la pipeta de microinjección y la placa MII debe evitarse, ya que el daño mecánico del husillo y los cromosomas pueden interrumpir el desarrollo del embrión. - Cargue un DKO-phaESC en la punta de la pipeta de microinjección aplicando una presión negativa suave. Romper la membrana plasmática de un DKO-phaESC mediante pipeteo para evitar la inyección de un DKO-phaESC intacto(Figura 3C;ver discusión).
NOTA: En caso de que la membrana plasmática de un DKO-phaESC no se rompa por pipeteo, deseche el DKO-phaESC y cargue otro DKO-phaESC. - Coloque la pipeta de microinjeción en contacto con la zona pellucida del ócito y aplique una pequeña cantidad de presión negativa dentro de la pipeta de microinjeción.
- Aplicar impulsos piezo (intensidad, 20; frecuencia, 4) para romper la zona mientras empuja la punta de la pipeta de microinjeción hacia el espacio perivitellina. Confirme que la placa MII, que contiene un husillo y cromosomas, no se encuentra en el camino de la pipeta de microinjeción.
NOTA: Ajuste empíricamente el ajuste a los pulsos piezozo más bajos para perforar a través de la zona para minimizar la posibilidad de daños en el oolemma. - Deseche el fragmento de la zona pellucida de la pipeta de microinjección y coloque el DKO-phaESC en el borde de la pipeta.
- Penetrar en el ócito con la pipeta de microinjección para que el oolema llegue al lado opuesto.
NOTA: No toque la placa MII para evitar daños en el husillo y los cromosomas. - Aplicar un pulso piezo (intensidad, 6; frecuencia, 1) para perforar el oolema. Asegúrese de que el oolemma se relaje a lo largo del eje de la pipeta de microinjección.
NOTA: Defina empíricamente el ajuste más bajo del pulso piezozo para romper el oolema para minimizar el daño al ócito. - Inyecte la DKO-phaESC con un volumen mínimo de medio en el ooplasma y retire la pipeta de microinjección suavemente del ócito.
- Suelte el ócito inyectado de la pipeta de sujeción y colóquelo en el lado de la microdrop para su posterior recolección.
- Repita el procedimiento de inyección de los pasos 12.9 a 12.17 para los otros ócitos MII en la caída media M2.
NOTA: Evite mantener los ocitos fuera de la incubadora durante más de 20 minutos. En nuestra experiencia, un lote de 10 ócitos se puede manipular cómodamente en un radio de 15 minutos después del entrenamiento adecuado. - Transfiera el lote de ocitos inyectados de la caída media M2 a un plato de pozo central pre-calentado con el medio KSOM.
- Mantenga el plato durante 1 h a 37 °C en un ambiente de CO2 del 5%.
- Repita la manipulación de ocitos de los pasos 12.5 y 12.20 con grupos adicionales de ócitos MII para obtener suficientes ócitos inyectados.
13. Activación de embriones semi-clonados construidos
- Prepare dos platos de centro-pozo con 900 μL cada uno de KSOM medio y medio de activación. Prepare una placa de 4 pozos con 700 μL de medio KSOM en cada pozo. Precalente los platos y el plato a 37 °C en un ambiente de CO2 del 5%.
- Después de 1 h en el medio KSOM, transfiera los ocitos inyectados del paso 12.21 al plato de pozo central pre-calentado con medio de activación.
- Mantenga el plato durante 6 h a 37 °C en una atmósfera de CO2 del 5% para la activación.
- Después de la activación, observar algunos embriones semi-clonados forman tres cuerpos polares, que son el primero y el segundo cuerpo polar del oocito, y el cuerpo pseudo polar de la DKO-phaESC (Figura 3E).
- Lave los embriones 3 veces transfiriéndolos a nuevos pozos con el medio KSOM en una placa de 4 pozos.
- Transfiera los embriones al plato central con el medio KSOM y mantenga el plato a 37 °C en una atmósfera de CO2 del 5% para un mayor desarrollo.
14. Desarrollo de embriones semi-clonados construidos
- Después de 1 día de cultivo en el medio KSOM desde el paso 13.6, varios embriones semi-clonados llegan a la etapa de 2 células (Figura 5A).
- Para un mayor desarrollo de embriones preimplantacionales in vitro, continúe culturando los embriones semi-clonados en el medio KSOM a 37 °C en una atmósfera de CO2 del 5%. Transfiera los embriones semi-clonados al medio fresco KSOM en el día 2. En el día 4, varios embriones llegarán a la etapa de blastocista(Figura 5A).
- Para la derivación de ratones semi-clonados, transfiera embriones de 2 células del paso 14.1 a los oviductos de las hembras receptoras pseudo-embarazadas. Identificar a las hembras pseudo-embarazadas apareándose a machos vasectomizados un día antes de la transferencia del embrión y seleccionarlas sobre la base de la presencia de un tapón claramente visible en la mañana del día para la transferencia del embrión (0,5 días después del coitum (dpc)). Alrededor de 19.5 dpc, cachorros a término se entregan naturalmente de las hembras receptoras (Figura 5B).
Resultados
El propósito de este protocolo es aplicar haESCs como sustituto de los espermatozoides para obtener embriones y ratones semi-clonados. Para ello, se generó una línea DKO-phaESC que transportaba el transgénero CAG-EGFP y se utilizó para la inyección intracitoplasmática en ócitos MII. Para obtener una línea phaESC adecuada con una configuración de impresión paterna, realizamos ingeniería genética utilizando núcleos Cas9. Una línea ESC haploide contiene células haploides y diploides que surgen debido a una tendencia inherente de los ESC haploides para la diploidización10. Un conjunto de cromosomas haploides es un requisito previo para la sustitución exitosa del genoma del esperma. El análisis de contenido de ADN por citometría de flujo muestra la distribución de células haploides y diploides en fases G0/G1,S y G2/M(Figura 2A).
Para establecer las líneas DKO-phaESC, las líneas phaESC de tipo salvaje se transfectaron con construcciones de transposon piggyBac para una expresión egfp transgénica estable y con plásmidos CRISPR/Cas9 para obtener eliminaciones de los H19- y IG-DMR. Para excluir los ESC diploides y aislar solo los ESC haploid que expresan EGFP, se definió una puerta de ordenación específica (Figura 2A). Las células positivas de egfp haploides individuales fueron entonces chapadas en pozos individuales de placas de 96 pozos para obtener sub-clones. Los alimentadores MEF se utilizaron para aumentar la eficiencia de chapado y la supervivencia de las fases transfectadas. Después de la expansión de las culturas, PCR llevó a cabo una ronda inicial de genotipado para identificar sub-clones que llevan eliminaciones de ambos DMR. Después de que los alimentadores MEF habían sido eliminados de las culturas, se realizó un segundo genotipado para confirmar la ausencia de alelos de wildtype en el H19- y IG-DMR (Figura 2B). De un total de 135 sub-clones, obtuvimos 5 líneas haploid DKO-phESC que llevaban los alelos eliminados y estaban libres de alelos de tipo salvaje tanto del H19-DMR como del IG-DMR.
Se introdujo un transgénero CAG-EGFP en DKO-fasfas para estudiar su contribución a embriones semi-clonados mediante la visualización de fluorescencia verde bajo un microscopio (Figura 3A). Para la inyección intracitoplasmática, los DKO-phaESC fueron tratados con demecolcina para arrestarlos en fase M. Por lo tanto, el ciclo celular de DKO-phaESCs se sincronizó con el de los ocitos MII. El análisis de citometría de flujo mostró 2 poblaciones correspondientes a la fase G2/M detenidas haploides (2n) y células diploides (4n) después del tratamiento con demecolcina (Figura 3B). La ausencia del pico haploide 1n indicó que la detención del ciclo celular estaba en gran parte completa. A continuación, se ordenaron e inyectaron ESC haploides en fase M en ócitos. Para ello, se cargó una sola DKO-phaESC en la pipeta de microinjección e se inyectó en el citoplasma de un ócito MII (Figura 3C). La membrana plasmática del DKO-phaESC se rompió al pipetear en la punta de la pipeta de microinjeción.
Después de la inyección, rara vez se detectó la expresión EGFP en los embriones semi-clonados construidos, ya que el citoplasma de DKO-fasfas se había dispersado en el gran citoplasma del citocitocito (Figura 3D). En raras ocasiones, se podría observar un punto redondo de expresión intensa de EGFP dentro del ooplasma. Esta observación probablemente fue causada por la inyección involuntaria de DKO-phaESCs intactos. Es probable que no se rompa la membrana celular DKO-phaESC, es probable que no sea compatible con un mayor desarrollo embrionario y se debe evitar. Una hora después de la inyección, los embriones se activaron mediante tratamiento con cloruro de estroncio18. Seis horas después del inicio de la activación, se observaron hasta 3 cuerpos polares bajo el microscopio(Figura 3E). Estos cuerpos polares corresponden a la primera y segunda masa polar del ócito y un cuerpo pseudo polar del DKO-phaESC7. Además, se observaron dos pronuclei bajo un microscopio de contraste de interferencia diferencial, que se asemejaba a la etapa pronuclear de los cigotos después de la fertilización normal con espermatozoides.
Para demostrar la competencia en el desarrollo, los embriones semi-clonados fueron cultivados a la etapa blastocista (Figura 5A). Además, se obtuvo un ratón a término después de transferir embriones de etapa semi-clonados de 2 células a los oviductos de una hembra receptora(Figura 5B). Como era de esperar, el ratón derivado de un embrión semi-clonado era una hembra, ya que ni los ocitos ni los falosos derivados de ocitos llevan un cromosoma Y. El ratón semi-clonado era excesivamente normal y producía descendencia sana cuando se apareaba con un macho webster suizo de tipo salvaje. Hasta ahora, el ratón semi-clonado ha estado vivo por más de 600 días sin ningún problema de salud aparente.
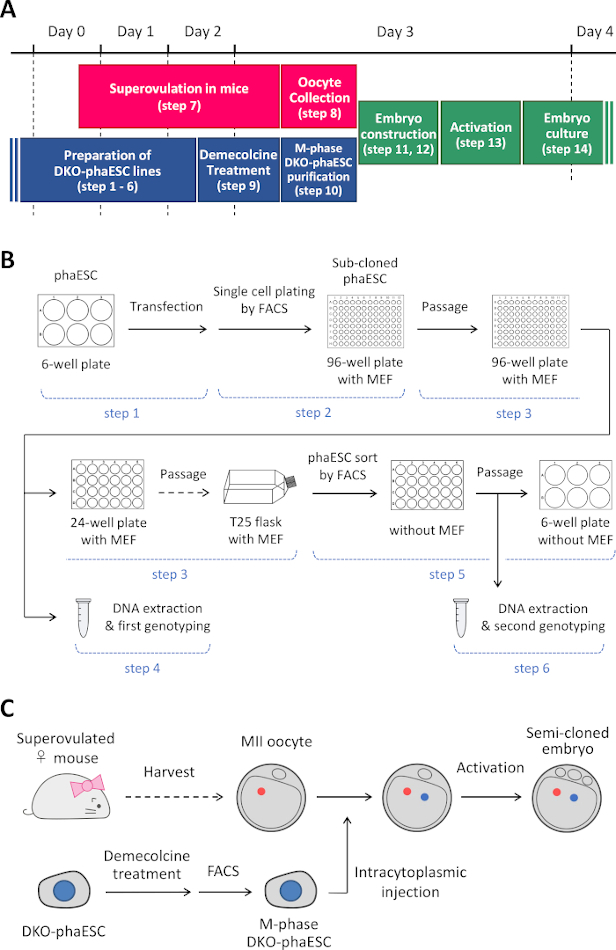
Figura 1: Visión general de la aplicación de DKO-phaESCs como reemplazo de espermatozoides. (A) Un plazo de los procedimientos del protocolo. (B) El esquema muestra los pasos para establecer líneas DKO-phaESC (pasos 1–6). (C) El esquema muestra los pasos para construir embriones semi-clonados mediante inyección intracitoplasmática de un DKO-phaESC en un ócito MII (pasos 7–14). Abreviaturas: DKO = doble nocaut; phaESC = célula madre embrionaria heploidea partegenética; FACS = clasificación celular activada por fluorescencia; MEF = fibroblasto embrionario del ratón. Haga clic aquí para ver una versión más grande de esta figura.
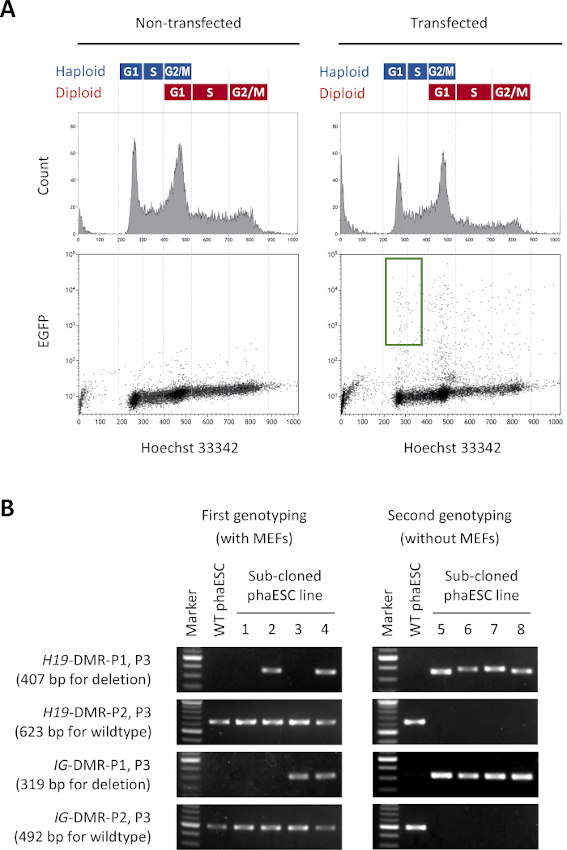
Figura 2: Establecimiento de líneas DKO-phaESC mediante eliminaciones mediadas por CRISPR/Cas9 de H19- e IG-DMR. (A) Análisis de citometría de flujo de fases después de la transfección con plásmidos piggyBac para una expresión egfp estable y con 4 plásmidos CRISPR/Cas9. Los fascos no transfectados se muestran como control. El perfil de ADN (arriba) muestra la distribución del ciclo celular de las células haploides y diploides. Las células haploides G1/S fase que expresan EGFP se indican por la puerta verde (abajo, derecha). (B) Genotipado de sub-clones phaESC que se cultivaron en MEF (primer genotipado) y después de la eliminación de mefs (segundo genotipado). Las líneas phaESC sub-clonadas 1, 2, 3 y 4 representan celdas de tipo salvaje, celdas con una eliminación H19-DMR, con una eliminación IG-DMR, y con eliminaciones combinadas de H19- e IG-DMR, respectivamente. Las líneas DKO-phaESC 5–8 poseen eliminaciones H19-DMR e IG-DMR y están libres de alelos de tipo salvaje. Abreviaturas: DKO = doble nocaut; phaESC = célula madre embrionaria heploidea partegenética; DMR = región metilada diferencial; MEF = fibroblasto embrionario del ratón; EGFP = proteína fluorescente verde mejorada; WT = wildtype. Haga clic aquí para ver una versión más grande de esta figura.
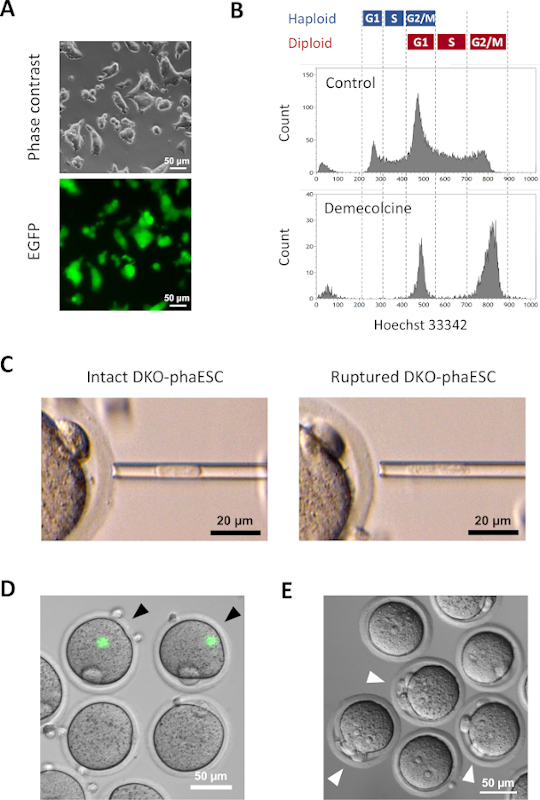
Figura 3: Inyección de DKO-phaESCs mitóticamente detenidos en omacitos MII. (A) Morfología de una referencia cultural DKO-phaESC que lleve un transgénero CAG-EGFP y eliminaciones de los DMR H19 e IG;; barra de escala = 50 μm. (B) Análisis representativo de citometría de flujo de DKO-fasfas después de la detención con demecolcina durante 8 h. DKO-phaESCs sin tratamiento de demecolcina se muestran como control. (C) Morfología de DKO-fasfas en la pipeta de microinjección. Se muestra un solo DKO-phaESC intacto antes (izquierda) y después (derecha) de la rotura de la membrana plasmática mediante pipeteo; barra de escala = 20 μm. (D) Embriones construidos a 1 h después de la inyección de DKO-phaESCs en ocitos MII. Se muestra una imagen combinada de fluorescencia EGFP y campo brillante. Las puntas de flecha negras indican puntos redondos de expresión EGFP intensa después de la inyección de DKO-phaESCs intactos, que deben evitarse; barra de escala = 50 μm. (E) Se muestra una imagen de contraste de interferencia diferencial de embriones semi-clonados 6 h después del inicio de la activación con cloruro de estroncio. Las puntas de flecha blancas indican embriones con 3 cuerpos polares, incluyendo un cuerpo pseudo polar del DKO-phaESC inyectado; barra de escala = 50 μm. Abreviaturas: DKO = doble nocaut; phaESC = célula madre embrionaria heploidea partegenética; EGFP = proteína fluorescente verde mejorada. Haga clic aquí para ver una versión más grande de esta figura.
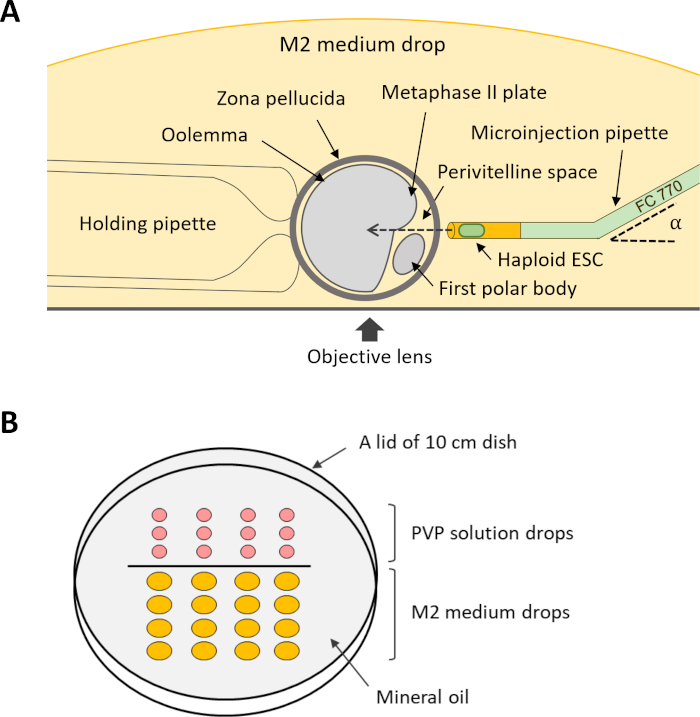
Figura 4: Esquema de la configuración para la inyección intracitoplasmática de DKO-phaESCs en ocitos. (A) Se muestra la disposición de una pipeta de inyección, una pipeta de sujeción y un ócito en la cámara de inyección. α, ángulo de flexión de la pipeta de microinyección. (B) Disposición de las gotas en el plato de micromanipulación para inyección intracitoplasmática. Abreviaturas: DKO = doble nocaut; phaESC = célula madre embrionaria heploidea partegenética. Haga clic aquí para ver una versión más grande de esta figura.
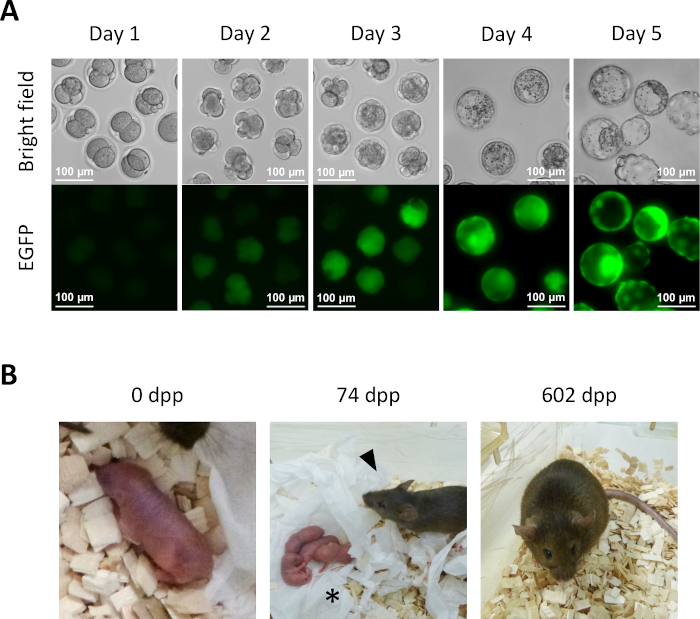
Figura 5: Desarrollo de embriones semi-clonados. (A) Desarrollo preimplantacional de embriones semi-clonados in vitro. La expresión EGFP se observa inicialmente en embriones de cuatro células en el día 2 después de la inyección intracitoplasmática. En el día 4, se puede observar una expresión intensa de egfp en blastocistas; barra de escala = 100 μm. (B) Un ratón semi-clonado obtenido después de la transferencia de embriones de 2 células a una hembra receptora. A los 74 dpp, el ratón semi-clonado (punta de flecha) entregó a sus primeros cachorros (asterisco) por nacimiento natural después de aparearse con un macho webster suizo de tipo salvaje. Los embriones semi-clonados y el ratón semi-clonado que se muestra en esta figura son idénticos a los reportados en Aizawa et al.3. Abreviaturas: EGFP = proteína fluorescente verde mejorada; .dpp, días después del parto. Haga clic aquí para ver una versión más grande de esta figura.
| Solución de gelatina | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| Agua | - | 500 mL | - |
| gelatina | - | 3 % | 0.2% |
| Medio de células madre embrionarias haploides (HaESC) | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| NDiff 227 | - | 10 mL | - |
| CHIR 99021 | 10 mM | 3 μL | 3 μM |
| PD 0325901 | 10 mM | 1 μL | 1 μM |
| Lif | 1 x 106 UI/ml | 10 μL | 1.000 UI/mL |
| Penicilina-Estreptomicina | 100x | 100 μL | 1x |
| Búfer de mantenimiento HaESC | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| Medio HaESC | - | 1 ml | - |
| Solución HEPES | 2 . | 20 μL | 20 mM |
| Tampone de lavado | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| DMEM / F-12 | - | 100 mL | - |
| Fracción BSA | 7.5% | 7,1 ml | 0.5% |
| Medio MEF | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| DMEM | - | 500 mL | - |
| GB2 | - | 56 mL | 10% |
| β-Mercaptoetanol | 14.3 mol/L | 3.9 μL | 100 μM |
| Penicilina-Estreptomicina | 100x | 5,6 ml | 1x |
| Tampón de lisis | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| Agua | - | 8.25 mL | - |
| Tris-HCl (pH 8.5) | 1 M | 1 ml | 100 mM |
| EDTA | 0.4 l | 100 μL | 5 mM |
| Solución SDS | 10% | 200 μL | 0.2% |
| NaCl | 6 . | 400 μL | 200 mM |
| Proteinasa K | 20 mg/ml | 50 μL | 100 μg/ml |
| Medio de activación | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| KSOM | - | 1 ml | - |
| Cloruro de estroncio | 2 . | 5 μL | 5 mM |
| EGTA | 0,5 M, pH 8,0 | 4 μL | 2 mM |
| Solución PVP | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| Medio M2 | - | 5 ml | - |
| Pvp | - | 0,6 g | 12% |
| Solución PMSG | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| Pbs | - | 450 μL | - |
| PMSG | 500 IU/mL | 50 μL | 50 UI/mL |
| solución hCG | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| Pbs | - | 450 μL | - |
| hCG | 500 IU/mL | 50 μL | 50 UI/mL |
| Medio hialuronidase | |||
| componente | Concentración de existencias | Volumen / peso | Concentración final |
| Medio M2 | - | 380 μL | - |
| hialuronidasa | 10 mg/ml | 20 μL | 0,5 mg/ml |
| Abreviaturas: LIF = factor inhibitorio de la leucemia; MEF = fibroblasto embrionario del ratón; | |||
| FBS = suero bovino fetal; DMEM = Dulbecco's Modified Eagle Medium; BSA = albúmina sérica bovina; | |||
| EDTA = ácido etilenodiaminetetraacetic; SDS = dodecylsulfato sódico; EGTA = etileno-bis(oxyetilenonitrilo)ácido tetraacético; | |||
| PBS = solución salina tamponada de fosfato; PVP = polivinylpyrrolidone; hCG = gonadotropina coriónica humana; | |||
| PMSG = gonadotropina sérica de yegua embarazada. | |||
Tabla 1: Receta de medio, búfer y solución.
| nombre | Secuencia (5' a 3') | aplicación | |
| H19-DMR-P1 | GTG GTT AGT TCT ATA TGG GG | Genotipado | |
| H19-DMR-P2 | AGA TGG GGT CAT TCT TTT CC | Genotipado | |
| H19-DMR-P3 | TCT TAC AGT CTG GTC TTG GT | Genotipado | |
| IG-DMR-P1 | TGT GCA GCA GCA AAG CTA AG | Genotipado | |
| IG-DMR-P2 | CCA CAA AAA CCT CCC TTT CA | Genotipado | |
| IG-DMR-P3 | ATA CGA TAC GGC AAC CAA CG | Genotipado | |
| H19-DMR-gRNA1-F | CAC CCA TGA ACT CAG AAG AGA CTG | gRNA | |
| H19-DMR-gRNA1-R | AAA CCA GTC TCT TCT GAG TTC ATG | gRNA | |
| H19-DMR-gRNA2-F | CAC CAG GTG AGA ACC ACT GCT GAG | gRNA | |
| H19-DMR-gRNA2-R | AAA CCT CAG CAG TGG TTC TCA CCT | gRNA | |
| IG-DMR-gRNA1-F | CAC CCG TAC AGA GCT CCA TGG CAC | gRNA | |
| IG-DMR-gRNA1-R | AAA CGT GCC ATG GAG CTC TGT ACG | gRNA | |
| IG-DMR-gRNA2-F | CAC CCT GCT TAG AGG TAC TAC GCT | gRNA | |
| IG-DMR-gRNA2-R | AAA CAG CGT AGT ACC TCT AAG CAG | gRNA | |
Tabla 2: Lista de oligonucleótidos.
Discusión
La clonación de mamíferos por transferencia nuclear de células somáticas (SCNT) ha sido pionera en los años1990 19,20,21. Estos desarrollos siguieron a estudios de clonación realizados 30 años antes en anfibios22. El considerable retraso refleja la dificultad de la embriología y la impresión genómica en mamíferos. El desarrollo del SCNT mamífero es la base para la aplicación de haESC para sustituir espermatozoides, que se detalla en este protocolo.
La sincronización del ciclo de celda es un factor importante para el éxito de SCNT23. Este es también el caso de inyección de haESC en este protocolo. La introducción de un genoma de donante en un receptor requiere que las fases del ciclo celular se igualan para evitar roturas cromosómicas o aneuploidies que abroguen el desarrollo del embrión. La semi-clonación tiene la complejidad adicional de que dos genomas y un citoplasto necesitan ser compatibles. Un informe anterior ha demostrado que la inyección de haESC androgenéticas detenidas en fase M produjo mejores tasas de desarrollo de embriones semi-clonados que la inyección de haESC en fase G1 en ocitos7. Este informe sugiere la fase M como un punto de sincronización adecuado para la semiagulación. En consecuencia, se detuvo mitoticamente a los faásicos con demecolcina e inyectaron en el ooplasma de ocitos MII, que fueron detenidos naturalmente en metafase II de meiosis. Es importante destacar que la detención en fase M se puede lograr en los ESC del ratón con alta eficiencia, proporcionando una excelente sincronización entre los ciclos celulares de donantes y receptores.
Durante la mitosis, la membrana nuclear se descompone y se forma un husillo al que se adhieren los cromosomas replicados. Después de la inyección de DKO-phaESCs de fase M, los cromatátidos hermanos se segregan en un cuerpo pseudo polar y el cigoto7 (Figura 3E). En consecuencia, un solo conjunto de cromosomas de un DKO-phaESC contribuye al embrión semi-clonado. Es fundamental que los cromosomas hermanos de los cromosomas DKO-phaESC se puedan segregar correctamente después de la inyección. La membrana plasmática de un DKO-phaESC intacto evita la segregación en un cuerpo pseudo polar. De hecho, observamos casos raros en los que la membrana plasmática de los DKO-phaESCs no se rompió, y los embriones exhibieron DKO-phaESCs intactos en el ooplasma después de la inyección (Figura 3D). Por lo tanto, se debe tener cuidado de eliminar la membrana plasmática de los DKO-FASC mediante pipeteo. Durante la inyección, es igualmente importante evitar la interrupción del husillo meiotico del ócito, que podría conducir a defectos de segregación cromosómica e inducir la aneuploidía también.
En los mamíferos, la impresión genómica limita la aplicación de fases como reemplazo de espermatozoides. Los haESC partenogenéticos poseen una configuración materna de impresiones genómicas, mientras que los espermatozoides poseen una configuración paterna. Por lo tanto, la generación de cachorros a término no se ha producido después de la inyección de fasfas de wildtype como reemplazo de espermatozoides. Para superar esta limitación, las eliminaciones de los IG- y H19-DMR se diseñan en fases. La modificación de la expresión impresa en el Igf2-H19 materno y gtl2-Dlk1 loci es suficiente para cambiar la configuración de las impresiones genómicas para permitir la generación de ratones semi-clonados con una frecuencia de más del 5,1%, basado en embriones transferidos de 2 células. Estas observaciones sugieren que apuntar a dos genes impresos cambia el genoma de los fasc en una configuración paterna funcional que puede reemplazar a los espermatozoides en ratones. No obstante, para esta estrategia es necesaria una modificación genética permanente de las fases. Como estrategia alternativa, se puede considerar la haESC androgenética. Los haESC androgenéticos se derivan del genoma del esperma y poseen huellas paternas. Se ha informado de que el tipo salvaje y los haESC androgenéticos contribuyeron como reemplazo de espermatozoides para generar cachorros a término a una frecuencia entre el 1,3% y el 1,9% de los embriones transferidos4,7,24. Los cachorros a término también se han obtenido inyectando haESC androgenéticos con eliminaciones del IG- y H19- DMR a una frecuencia del 20,2% de los embriones de 2 células transferidos24. El aumento de la eficiencia de la semi-clonación utilizando haESC androgenéticos modificados es probable porque las impresiones pueden volverse inestables en el cultivo. Los defectos de impresión se corrigen mediante las deleciones genéticas de los DMR críticos.
Teniendo en cuenta la dificultad de introducir modificaciones genéticas directamente en los genomas de los ócitos o espermatozoides, los haESC son una herramienta valiosa para manipular los genomas parentales por separado. El uso de haESCs como sustituto de los espermatozoides proporciona una ventaja notable para la edición del genoma en la línea germino del ratón. Un estudio reciente ha combinado la edición del genoma basada en CRISPR/Cas9 con la aplicación de haESC para la caracterización de regiones de impresión que son críticas para el desarrollo embrionario12. Este estudio analizó el papel del Rasgrf1-DMR en combinación con el H19-y IG-DMR en el desarrollo de ratones bimaternal, y la función de 7 DMR diferentes en el desarrollo de ratones bipaternales. El método para sustituir los haESC por espermatozoides formó la base para enfoques genéticos de detección para identificar aminoácidos clave dentro de la proteína DND1 en el desarrollo primordial de células germinales y para identificar genes en el desarrollo óseo24,25,26. Los estudios sobre la impresión genómica y el cribado genético para identificar factores clave en el desarrollo embrionario son enfoques considerables para la aplicación de los haESC como genomas áticos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos al Dr. Giulio Di Minin la derivación de líneas phaESC y al Dr. Remo Freimann por el funcionamiento de la citometría de flujo. También reconocemos a la Sra. Michèle Schaffner y al Sr. Thomas M. Hennek por apoyo técnico con transferencia de embriones. Este trabajo fue apoyado por la Fundación Nacional suiza de Ciencias (subvención 31003A_152814/1).
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mLTube | Eppendorf | 0030 125.150 | haESC culture |
| 15 ml Tube | Greiner Bio-One | 188271 | haESC culture |
| 12-well plate | Thermo Scientific | 150628 | haESC culture |
| 24-well plate | Thermo Scientific | 142475 | haESC culture |
| 6-well plate | Thermo Scientific | 140675 | haESC culture |
| 96-well plate | Thermo Scientific | 167008 | haESC culture |
| β-Mercaptoethanol | Merck | M6250 | haESC culture |
| BSA fraction | Gibco | 15260-037 | haESC culture |
| CHIR 99021 | Axon | 1386 | haESC culture |
| Demecolcine solution | Merck | D1925 | haESC culture |
| DMEM | Gibco | 41965-039 | haESC culture |
| DMEM / F-12 | Gibco | 11320-033 | haESC culture |
| DR4 mouse | The Jackson Laboratory | 3208 | haESC culture |
| EDTA | Merck | E5134 | haESC culture |
| FBS | biowest | S1810-500 | haESC culture |
| Freezing medium | amsbio | 11897 | haESC culture |
| Gelatin | Merck | G1890 | haESC culture |
| Hepes solution | Gibco | 15630-056 | haESC culture |
| Irradiated MEF | Gibco | A34966 | haESC culture |
| LIF (Leukemia inhibitory factor) | (Homemade) | - | haESC culture |
| Lipofection reagent | Invitrogen | 11668019 | haESC culture |
| NDiff 227 | Takara | Y40002 | haESC culture |
| PD 0325901 | Axon | 1408 | haESC culture |
| Pen Strep | Gibco | 15140-122 | haESC culture |
| piggyBac plasmid carrying a CAG-EGFP transgene | Addgene | #40973 | haESC culture |
| piggyBac transposase plasmid | System Biosciences | PB210PA-1 | haESC culture |
| pX330 plasmid | Addgene | #42230 | haESC culture |
| pX458 plasmid | Addgene | #48138 | haESC culture |
| Trypsin | Gibco | 15090-046 | haESC culture |
| DNA polymerase | Thermo Scientific | F549S | Genotyping |
| dNTP Mix | Thermo Scientific | R0192 | Genotyping |
| Ethanol | Merck | 100983 | Genotyping |
| Hydrochloric acid | Merck | 100317 | Genotyping |
| Isopropanol | Merck | 109634 | Genotyping |
| Proteinase K | Axon | A3830 | Genotyping |
| SDS | Merck | 71729 | Genotyping |
| Sodium chloride | Merck | 106404 | Genotyping |
| ThermoMixer | Epeendorf | 5384000012 | Genotyping |
| Tris | Merck | T1503 | Genotyping |
| 5 mL Tube | Falcon | 352063 | Flow cytometry |
| 5 mL Tube with cell strainer cap | Falcon | 352235 | Flow cytometry |
| Hoechst 33342 | Invitrogen | H3570 | Flow cytometry |
| MoFlo Astrios EQ | Beckman Coulter | B25982 | Flow cytometry |
| 1 mL syringe | Codan | 62.1640 | Superovulation |
| 30-gauge 1/2 inch needle | Merck | Z192362-100EA | Superovulation |
| B6D2F1 mouse | The Jackson Laboratory | 100006 | Superovulation |
| hCG | MSD animal health | 49451 | Superovulation |
| PBS | Gibco | 10010015 | Superovulation |
| PMSG | Avivasysbio | OPPA1037 5000 IU | Superovulation |
| 10-cm dish | Thermo Scientific | 150350 | Embryo manipulation |
| 4-well plate | Thermo Scientific | 179830 | Embryo manipulation |
| 50 mL tube | Greiner Bio-One | 227261 | Embryo manipulation |
| 6 cm dish | Thermo Scientific | 150288 | Embryo manipulation |
| Center-well dish | Corning | 3260 | Embryo manipulation |
| Digital rocker | Thermo Scientific | 88880020 | Embryo manipulation |
| EGTA | AppliChem | A0878 | Embryo manipulation |
| Hyaluronidase | Merck | H4272 | Embryo manipulation |
| KSOM | Merck | MR-020P-5F | Embryo manipulation |
| M16 medium | Merck | M7292 | Embryo manipulation |
| M2 medium | Merck | M7167 | Embryo manipulation |
| Mineral oil | Merck | M8410 | Embryo manipulation |
| Glass capillary | Merck | P1049-1PAK | Embryo manipulation |
| Stereomicroscope | Nikon | SMZ745 | Embryo manipulation |
| Strontium chloride | Merck | 13909 | Embryo manipulation |
| 5ml syringe | Codan | 62.5607 | Microinjection |
| Borosilicate glass cappilary | Science product | GB100T-8P | Microinjection |
| CellTram 4r Oil | Eppendorf | 5196000030 | Microinjection |
| Fluorinert FC-770 | Merck | F3556 | Microinjection |
| Holding pipette | BioMedical Instruments | - | Microinjection |
| Inverted microscope | Nikon | Eclipse Ti-U | Microinjection |
| Microforge | Narishige | MF-900 | Microinjection |
| Microinjection pipette | BioMedical Instruments | - | Microinjection |
| Microlaoder tips | Eppendorf | 5252 956.003 | Microinjection |
| Micromanipulaor arms | Narishige | NT-88-V3 | Microinjection |
| Micropipette puller | Sutter Instrument | P-1000 | Microinjection |
| PiezoXpert | Eppendorf | 5194000016 | Microinjection |
| PVP (Polyvinylpyrrolidine) | Merck | PVP360 | Microinjection |
| Syringe filter | SARSTEDT | 83.1826.001 | Microinjection |
Referencias
- Ellegren, H., Galtier, N. Determinants of genetic diversity. Nature Reviews: Genetics. 17 (7), 422-433 (2016).
- Huijbers, I. J. Generating genetically modified mice: A decision guide. Methods in Molecular Biology. 1642, 1-19 (2017).
- Aizawa, E., Dumeau, C. E., Freimann, R., Di Minin, G., Wutz, A. Polyploidy of semi-cloned embryos generated from parthenogenetic haploid embryonic stem cells. PloS One. 15 (9), 0233072 (2020).
- Li, W., et al. Androgenetic haploid embryonic stem cells produce live transgenic mice. Nature. 490 (7420), 407-411 (2012).
- Li, Z., et al. Birth of fertile bimaternal offspring following intracytoplasmic injection of parthenogenetic haploid embryonic stem cells. Cell Research. 26 (1), 135-138 (2016).
- Wan, H., et al. Parthenogenetic haploid embryonic stem cells produce fertile mice. Cell Research. 23 (11), 1330-1333 (2013).
- Yang, H., et al. Generation of genetically modified mice by oocyte injection of androgenetic haploid embryonic stem cells. Cell. 149 (3), 605-617 (2012).
- Zhong, C., et al. Parthenogenetic haploid embryonic stem cells efficiently support mouse generation by oocyte injection. Cell Research. 26 (1), 131-134 (2016).
- Elling, U., et al. Forward and reverse genetics through derivation of haploid mouse embryonic stem cells. Cell Stem Cell. 9 (6), 563-574 (2011).
- Leeb, M., Wutz, A. Derivation of haploid embryonic stem cells from mouse embryos. Nature. 479 (7371), 131-134 (2011).
- Surani, M. A., Barton, S. C., Norris, M. L. Development of reconstituted mouse eggs suggests imprinting of the genome during gametogenesis. Nature. 308 (5959), 548-550 (1984).
- Li, Z. K., et al. Generation of bimaternal and bipaternal mice from hypomethylated haploid ESCs with imprinting region deletions. Cell Stem Cell. 23 (5), 665-676 (2018).
- Elling, U., et al. Derivation and maintenance of mouse haploid embryonic stem cells. Nature Protocols. 14 (7), 1991-2014 (2019).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shuai, L., et al. Generation of mammalian offspring by haploid embryonic stem cells microinjection. Current Protocols in Stem Cell Biology. 31, 1-15 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923 (2012).
- Behringer, R., Gertsenstein, M., Nagy, K. V., Nagy, A. . Manipulating the Mouse Embryo: A Laboratory Manual. , (2014).
- O'Neill, G. T., Rolfe, L. R., Kaufman, M. H. Developmental potential and chromosome constitution of strontium-induced mouse parthenogenones. Molecular Reproduction and Development. 30 (3), 214-219 (1991).
- Campbell, K. H., McWhir, J., Ritchie, W. A., Wilmut, I. Sheep cloned by nuclear transfer from a cultured cell line. Nature. 380 (6569), 64-66 (1996).
- Wakayama, T., Perry, A. C., Zuccotti, M., Johnson, K. R., Yanagimachi, R. Full-term development of mice from enucleated oocytes injected with cumulus cell nuclei. Nature. 394 (6691), 369-374 (1998).
- Wilmut, I., Schnieke, A. E., McWhir, J., Kind, A. J., Campbell, K. H. Viable offspring derived from fetal and adult mammalian cells. Nature. 385 (6619), 810-813 (1997).
- Gurdon, J. B. Adult frogs derived from the nuclei of single somatic cells. Developmental Biology. 4, 256-273 (1962).
- Campbell, K. H., Alberio, R. Reprogramming the genome: role of the cell cycle. Reproduction Supplement. 61, 477-494 (2003).
- Zhong, C., et al. CRISPR-Cas9-mediated genetic screening in mice with haploid embryonic stem cells carrying a guide RNA library. Cell Stem Cell. 17 (2), 221-232 (2015).
- Bai, M., et al. Targeted genetic screening in mice through haploid embryonic stem cells identifies critical genes in bone development. PLoS Biology. 17 (7), 3000350 (2019).
- Li, Q., et al. CRISPR-Cas9-mediated base-editing screening in mice identifies DND1 amino acids that are critical for primordial germ cell development. Nature Cell Biology. 20 (11), 1315-1325 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados