Method Article
Application des cellules souches embryonnaires haploïdes parthénogénétiques de souris comme substitut du sperme
Dans cet article
Résumé
Cet article vise à démontrer l’utilisation de cellules souches embryonnaires haploïdes parthénogénétiques comme substitut au sperme pour la construction d’embryons semi-clonés.
Résumé
Dans les organismes ayant une reproduction sexuelle, les cellules germinales sont la source de cellules totipotentes qui se développent en nouveaux individus. Chez la souris, la fécondation d’un ovocyte par un spermatozoon crée un zygote totipotent. Récemment, plusieurs publications ont rapporté que les cellules souches embryonnaires haploïdes (HAESCs) peuvent remplacer les génomes gametiques et contribuer aux embryons, qui se développent en souris. Ici, nous présentons un protocole pour appliquer les haESCs parthénogénétiques comme substitut du sperme pour construire des embryons par injection intracytoplasmique dans les ovocytes. Ce protocole consiste en des étapes de préparation des HAESCs comme substitut du sperme, pour l’injection de chromosomes haESC dans les ovocytes, et pour la culture d’embryons semi-clonés. Les embryons peuvent donner des souris semi-clonées fertiles après le transfert d’embryons. L’utilisation des haESCs comme substitut de sperme facilite l’édition du génome dans la lignée germinale, les études sur le développement embryonnaire et l’étude de l’empreinte génomique.
Introduction
Chez les mammifères, les gamètes sont les seules cellules qui transmettent des informations génétiques à la prochaine génération. La fusion d’un ovocyte et d’un spermatozoon forme un zygote diploïde qui se développe en animal adulte. Les mutations dans les génomes gametiques sont ainsi héritées par la progéniture et conduisent à des variations génétiques chez lesespèces 1. L’introduction de mutations dans la lignée germinale a été appliquée pour produire des animaux génétiquement modifiés pour diverses études biologiques, y compris la caractérisation de la fonction génétique et la modélisation des maladies. Les ovocytes et le spermatozoa sont des cellules en phase terminale différenciées et hautement spécialisées qui ont cessé la prolifération. Par conséquent, la modification directe des gamètes est techniquement difficile, et des approches spécialisées ont été développées. Des modifications génétiques peuvent être introduites dans la lignée germinale de la souris par l’injection d’ESCs génétiquement modifiés dans les blastocystes, où ils s’intègrent dans l’embryon en développement et colonisent la lignée germinale. En outre, la modification génétique des zygotes à l’aide d’approches d’édition du génome, y compris le système CRISPR/Cas9, est devenue largementadoptée 2.
Récemment, une approche exceptionnelle a été signalée, qui applique haESCs comme un substitut à un génome gametic3,4,5,6,7,8. Les HAESCs sont des lignées de cellules souches dérivées de la masse cellulaire interne des blastocystes haploïdes parthénogénétiques ou androgènes et possèdent un seul ensemble de chromosomes4,7,9,10. Il a été démontré que les haESCs parthénogénétiques et androgènes peuvent contribuer au génome des souris semi-clonées après injection intracytoplasmique dans les ovocytes. Contrairement à d’autres approches, les génomes des HAESC peuvent être directement modifiés en culture en raison de leur capacité d’auto-renouvellement. L’introduction de modifications génétiques dans la lignée germinale en remplaçant le sperme par des haESCs est une méthode importante pour les études biologiques. Il prévoit la possibilité de culturer et de manipuler séparément le génome maternel ou paternel, qui sont dérivés des haESCs parthénogénétiques ou androgènes, respectivement. Les HAESC peuvent ensuite être utilisés comme remplacement du génome gametique, ce qui est particulièrement avantageux pour les études d’empreinte génomique, d’expression spécifique à l’allèle et de processus spécifiques aux parents.
Chez la souris, des informations génomiques maternelles et paternelles sont nécessaires pour le développement normal del’embryon 11. Par conséquent, les petits à terme n’ont pas pu être obtenus lorsque des haESCs parthénogénétiques de type sauvage (phaESCs) ont été injectés pour remplacer le génome du sperme5,8. Pour surmonter le bloc de développement, l’empreinte génomique du génome maternel des haESCs parthénogénétiques doit être corrigée à une configuration paternelle. Ceci peut être réalisé par la manipulation des régions différemment méthylées (DMRs). À ce jour, des suppressions ciblées du H19-DMR, gtl2-Dlk1 IG-DMR, et Rasgrf1-DMR ont été étudiées pour réprimer les gènes exprimés maternellement dans les phaESCs3,5,8,12. Ces études ont démontré que les suppressions du H19-DMR et de l’IG-DMRsont suffisantes pour convertir une mère en une configuration d’empreinte paternelle qui peut remplacer les chromosomes du sperme. L’injection intracytoplasmique de phaESCs qui transportent les deux suppressions de DMR dans les ovocytes a donné des chiots semi-clonés avec une fréquence entre 5,1% et 15,5% des embryons transférés à 2 cellules.
Ce protocole est basé sur l’application de phaESCs avec suppressions de H19-DMR et IG-DMR, que nous qualions de phaESCs à double KO (DKO-phaESCs). Nous fournissons des instructions détaillées pour la modification de l’empreinte génomique dans les phaESCs pour établir des lignées DKO-phaESC, pour l’injection de DKO-phaESCs dans les ovocytes comme un substitut à un génome de sperme, pour la culture d’embryons semi-clonés aux blastocystes, et pour l’obtention de souris semi-clonées. Ce protocole est une référence pour les chercheurs qui nécessitent une manipulation précise et directe du génome paternel et de la génération d’embryons et de souris semi-clonés.
Protocole
Toutes les expériences animales ont été réalisées sous licence ZH152/17 conformément aux normes et règlements de la Commission cantonale d’éthique zurichoise et de l’installation animale EPIC de l’Institut des sciences moléculaires de la santé (ETH Zurich).
REMARQUE : Ce protocole commence par la suppression des DMR H19- et IG-dans les phaESCs. Pour plus de détails sur la façon d’établir des lignes phaESC, veuillez consulter les rapportspubliés 10,13. Une vue d’ensemble et un calendrier de ce protocole (étapes 1 à 14) sont fournis à la figure 1A; les médias, les solutions et les tampons sont répertoriés dans le tableau 1. La procédure d’établissement des lignées DKO-phaESC (étapes 1-6) est indiquée à la figure 1B,et la stratégie de construction d’embryons semi-clonés (étapes 7 à 14) est représentée à la figure 1C.
1. Transfection des plasmides pour la suppression de H19-DMR et IG-DMR dans les phaESCs
- Préparer les plasmides CRISPR/Cas9 à la co-expression des nucléases Cas9 et guider les ARN pour cibler les suppressions de H19-DMR et IG-DMR. Ligate quatre paires d’oligos d’ARN guide (H19-DMR-gRNA1-F, R; H19-DMR-gRNA2-F, R; IG-DMR-gRNA1-F, R; IG-DMR-gRNA2-F, R répertorié dans le tableau 2) en plasmides pX330.
REMARQUE : Reportez-vous au protocole publié sur la procédure détaillée de préparation de ces plasmides CRISPR/Cas914. Alternativement, les plasmides disponibles pour les souches générales de souris sont également accessibles par l’intermédiaire d’un référentiel(Tableau des matériaux). - Enduire la surface d’un puits d’une plaque de 6 puits de 1 mL de solution gélatineuse de 0,2 % par incubation à 37 °C pendant 10 min.
- Plaque 2 × 105 phaESCs de type sauvage sur le puits recouvert de gélatine dans le milieu haESC sans antibiotiques, et incuber la plaque à 37 °C dans une atmosphère de CO2 de 5 % pendant 1 jour.
REMARQUE : Les antibiotiques sont omis du milieu pour augmenter l’efficacité de la lipofection suivante. Nous avons utilisé des phaESCs de type sauvage au passage 10. Nous vous recommandons d’utiliser des phaESCs de passage précoce, mais une variété de nombres de passage ont été utilisés avecsuccès 8. La corrélation entre le nombre de passages et l’efficacité de l’obtention d’embryons semi-clonés et de souris n’est pas connue à l’heure actuelle. - PhaESCs transfect dans le puits d’une plaque de 6 puits (à partir de l’étape 1.3) avec 6 plasmides utilisant simultanément le reagent de lipofection : 50 ng piggyBac plasmide portant un transgène CAG-EGFP, 50 ng piggyBac transposase plasmide, et 600 ng de chacun des plasmides CRISPR/Cas9 (à partir de l’étape 1.1). Consultez le protocole du fabricant sur la procédure détaillée de la transfection.
REMARQUE : Deux plasmides piggyBac sont utilisés pour intégrer un transposon pour l’expression omniprésente de protéines fluorescentes vertes améliorées (EGFP) dans le génome des phaESCs. Si le marquage GFP des cellules n’est pas nécessaire, ces deux plasmides peuvent être remplacés par un plasmide CRISPR/Cas9 pour l’expression transitoire de protéines de fluorescence (pX458 plasmide par exemple) au lieu de l’un des plasmides pX330. L’expression TRANSITOIRE EGFP peut ensuite être utilisée pour trier les cellules transfectées. - Deux jours après la transfection, aspirer le milieu et ajouter 800 μL de trypsine.
- Incuber la plaque à 37 °C dans une atmosphère de CO2 à 5 % pendant 5 min. Ensuite, ajouter 2 mL de tampon de lavage pour étancher la trypsine, et pipette plusieurs fois pour obtenir une suspension à cellule unique.
- Transférer la suspension cellulaire dans un tube de 15 mL.
- Centrifuger le tube à 160 x g pendant 5 min, et enlever le supernatant.
- Resuspendez la pastille cellulaire dans 400 μL de tampon de maintenance haESC complété par 15 μg/mL Hoechst 33342.
REMARQUE : Pour réduire la toxicité potentielle de Hoechst 33342, 1 μg/mL Hoechst 33342 et 50 μM verapamil ont été utilisés au lieu de 15 μg/mL Hoechst 3334215. - Incuber la suspension cellulaire à 37 °C dans une atmosphère de CO2 de 5 % pendant 15 min. Après l’incubation, transférer la suspension cellulaire dans un tube de 5 mL à travers un bouchon de passoire cellulaire, et garder le tube à 4 °C jusqu’à ce qu’il soit prêt à être utilisé à l’étape suivante (section 2).
2. Placage à cellules simples de phaESCs transfectés à l’aide d’un cytomètre à écoulement
- Un jour avant le tri des phaESCs transfected, plaque irradiée fibroblastes embryonnaires de souris (MEFs) sur les plaques gélatineuses de 96 puits à une densité de 4 × 104 cellules/cm2 dans le milieu mef. En règle générale, 6 plaques sont prêtes à établir une ligne phaESC avec des suppressions ciblées. Incuber les assiettes à 37 °C dans une atmosphère de CO2 à 5 %.
REMARQUE : Les CEM Irradiés sont disponibles dans le commerce. Nous utilisons des MEF dérivés d’embryons E12.5 de souris DR4. Bien que les HAESC puissent pousser sur des plaques recouvertes de gélatine sans CEM, nous recommandons aux CEM d’accroître la viabilité des haESCs simples triés. - Le jour du tri, aspirer le milieu MEF à partir des plaques de 96 puits et ajouter 120 μL de haESC frais moyen par puits. Gardez les assiettes à 37 °C dans une atmosphère de CO2 à 5 %.
- Installez un trieur de cellules avec une buse de 100 μm selon les instructions du fabricant. Un laser UV de 355 nm et un laser bleu de 488 nm sont utilisés pour l’excitation de la fluorescence Hoechst 33342 et EGFP, respectivement.
REMARQUE: Alternativement, Hoechst 33342 peut être détecté par excitation avec 405 nm. - Trier les phaESCs transfectés dans le tube de 5 mL (à partir de l’étape 1.10) à l’aide d’une barrière pour la collecte des cellules haploïdes dans la phase G1/S qui montrent l’expression EGFP. Déposez une seule cellule dans chaque puits des plaques de 96 puits à partir de l’étape 2.2.
REMARQUE : La détection de la coloration hoechst 33342 distingue généralement 3 pics de cellules ayant une teneur en ADN de 1n, 2n et 4n, qui correspondent aux cellules haploïdes en phase G1, un mélange de cellules haploïdes en phase G2/M et de cellules diploïdes en phase G1, et de cellules diploïdes en phase G2/M, respectivement. Les cellules haploïdes à la phase G1/S sont identifiées comme le pic avec une intensité plus faible de fluorescence Hoechst 33342. Un résultat représentatif et une porte de tri sont indiqués dans la figure 2A. - Après placage, incuber les plaques de 96 puits à 37 °C dans une atmosphère de CO2 à 5 %.
3. Sous-clonage des phaESCs transfectés
- Trois jours après le placage unicelliste, des colonies peuvent être observées dans plusieurs puits des plaques de 96 puits au microscope. Marquez les puits dans lesquels ne poussent que des colonies.
REMARQUE : D’après notre expérience, des colonies individuelles ont été observées dans 20 à 40 % des puits des plaques de 96 puits. - Le jour 4 après placage unicelliste, remplacer la moitié du milieu par un nouveau milieu haESC dans les puits par des colonies simples.
- Un jour avant le passage (au jour 4 ou 5 après le placage unicellule), plaque irradiée MEFs sur des plaques gélatineuses de 96 puits à une densité de 4 × 104 cellules/cm2 dans le milieu MEF. Gardez les assiettes à 37 °C dans une atmosphère de CO2 à 5 %.
- Après 5 ou 6 jours de placage unicelliste, sélectionnez les puits des plaques de 96 puits contenant des colonies individuelles d’un diamètre supérieur à 150 μm.
REMARQUE : Environ 100 puits sont de préférence sélectionnés pour établir une ligne phaESC avec les suppressions ciblées du DMR. - Aspirer le milieu dans les puits sélectionnés et ajouter 30 μL de trypsine. Incuber les plaques de 96 puits à 37 °C dans une atmosphère de CO2 à 5 % pendant 5 min. Ajouter ensuite 30 μL de tampon de lavage à chaque puits pour étancher la trypsine.
- Ajouter 140 μL de milieu haESC dans chaque puits, et pipette plusieurs fois pour obtenir des cellules individuelles.
- Aspirez le milieu MEF à partir des puits des plaques de 96 puits préparées à l’étape 3.3.
- Transférez les phaESCs de chaque puits de l’étape 3.6 dans un puits frais de la nouvelle plaque de 96 puits de l’étape 3.7.
- Incuber les plaques de 96 puits contenant des sous-vêtements phaESC à 37 °C dans une atmosphère de CO2 à 5 %.
- Le lendemain, aspirez tout le milieu de chaque puits et ajoutez 120 μL de nouveau milieu haESC. Incuber les assiettes à 37 °C dans une atmosphère de CO2 à 5 %.
- Un jour avant que les cellules deviennent confluentes pour le passage, plaquez les MEF irradiés sur les plaques gélatineuses de 24 puits à une densité de 4 × 104 cellules/cm2 dans le milieu mef. Gardez les assiettes à 37 °C dans une atmosphère de CO2 à 5 %.
- Lorsque les phaESC sont devenus confluents pour le passage, aspirez le milieu et ajoutez 30 μL de trypsine. Incuber les plaques de 96 puits à 37 °C pendant 5 min.
- Ajouter 90 μL de tampon de lavage dans chaque puits pour étancher la trypsine. Pipette plusieurs fois pour obtenir des cellules individuelles.
- Aspirez le milieu MEF des puits des plaques de 24 puits à partir de l’étape 3.11, et ajoutez 600 μL de milieu haESC frais.
- Transférer 60 μL de la suspension des sous-vêtements phaESC de chaque puits à l’étape 3.13 dans un nouveau puits des plaques de 24 puits de l’étape 3.14. Gardez la suspension restante de chaque sous-vêtements phaESC pour l’extraction de l’ADN et le génotypage à l’étape 4.
- Incuber les plaques de 24 puits avec les sous-vêtements phaESC à 37 °C dans une atmosphère de CO2 à 5 %.
- Un jour avant que les cultures cellulaires atteignent la densité de passage, plaquez les MEF irradiés sur des plaques 6 puits recouvertes de gélatine à une densité de 4 × 104 cellules/cm2 dans le milieu MEF. Gardez les assiettes à 37 °C dans une atmosphère de CO2 à 5 %.
- Lorsque les phaESC deviennent suffisamment confluents pour passer, aspirez le milieu et ajoutez 250 μL de trypsine. Incuber les plaques de 24 puits à 37 °C pendant 5 min.
REMARQUE : Après le génotypage à l’étape 4.9, seules les lignes phaESC avec suppressions du H19-DMR et de l’IG-DMR doivent être passages. - Ajouter 750 μL de tampon de lavage dans chaque puits pour étancher la trypsine. Pipette plusieurs fois pour obtenir une suspension à cellule unique. Transférer la suspension cellulaire dans un tube de 15 mL.
- Centrifuger le tube à 160 x g pendant 5 min, enlever le supernatant, et resuspendre la pastille cellulaire dans 2 mL de milieu haESC.
- Aspirez le milieu MEF des puits des plaques de 6 puits à partir de l’étape 3.17. Transférer la suspension phaESC de chaque tube de l’étape 3.20 dans un nouveau puits. Gardez les assiettes à 37 °C dans une atmosphère de CO2 à 5 %.
- Élargissez les clones cellulaires en répétant les étapes 3.17 à 3.21 et en augmentant la taille de la plaque et les volumes de trypsine, tampon de lavage, et milieu haESC. Préparez un flacon T25 pour chaque ligne phaESC sous-clonée de MEFs pour l’étape 5.
REMARQUE : Nous recommandons de congeler un aliquot de chaque lignée phaESC sous-clonée dans 300 μL de milieu de congélation et de conserver un cryostock dans le stockage de l’azote liquide avant de passer à l’étape 5.
4. Premier génotypage de lignes phaESC sous-clonées avec des CEM
- Pour extraire l’ADN génomique de la suspension cellulaire restante de l’étape 3.15, ajoutez 200 μL de tampon de lyse à chaque puits des plaques de 96 puits. Transférer la suspension cellulaire dans un tube de 1,5 mL. Rincez chaque puits avec un tampon supplémentaire de 200 μL de lyse pour récupérer toutes les cellules restantes et recueillir dans le même tube de 1,5 mL.
- Incuber le tube de 1,5 mL à 55 °C pendant 3 h avec le mélange.
- Après l’incubation, ajouter 460 μL d’isopropanol à chaque tube de 1,5 mL et mélanger délicatement jusqu’à ce qu’un précipité d’ADN devienne visible.
- Centrifuger les tubes ≥ 10.000 x g pendant 5 min et enlever le supernatant. Laver les granulés d’ADN avec 200 μL d’éthanol à 70 %.
- Centrifuger les tubes ≥ 10.000 x g pendant 5 min et enlever le supernatant.
- Séchez les tubes dans l’air pendant 10 min, puis réutilisez l’ADN dans 20 μL d’eau.
- Effectuer la réaction en chaîne de polymése (PCR) à l’aide de polymése d’ADN thermostable suivant le protocole du fabricant.
REMARQUE : Les paires d’amorce pour PCR sont répertoriées dans le tableau 2 et sont utilisées comme suit : H19-DMR-P1 et P3 (407 bp pour H19-DMR supprimé); H19-DMR-P2 et P3 (623 bp pour wildtype H19-DMR); IG-DMR-P1 et P3 (319 bp pour supprimé IG-DMR); IG-DMR-P2 et P3 (492 bp pour wildtype IG-DMR). Le profil de température du PCR pour toutes les paires d’amorce est le suivant : 30 s 98 °C, 35 x (10 s 98 °C, 20 s 56 °C, 30 s 72 °C), 5 min 72 °C. La longueur des fragments d’ADN amplifiés pour les suppressions H19-DMR et IG-DMRmontre une certaine variation en raison de l’assemblage final non homologue associé à l’édition crispr/cas9-24. Les PhaESC ont été cultivés avec des MEF, qui contiennent de l’ADNwildtype H19 -DMR et IG-DMR. Par conséquent, les paires d’amorce de H19-DMR-P2/P3 et IG-DMR-P2/P3, qui amplifient les fragments d’ADN de type sauvage, ne sont pas informatives. Toutefois, ces paires d’amorce sont incluses comme contrôles et devraient donner une bande dans toutes les réactions. - Analyser les fragments de PCR par électrophorèse gel agarose. Reportez-vous au protocole publié sur la procédure détaillée de l’électrophoresis16.
- Identifier les lignées cellulaires avec des suppressions de H19-DMR et IG-DMR. Une image représentative de l’électrophoresis est affichée dans la figure 2B.
NOTE : Dans notre cas, huit lignes cellulaires avec des suppressions des lignes H19-DMR et IG-DMRont été identifiées parmi 135 lignes phaESC sous-clonées.
5. Purification des cellules haploïdes des lignées phaESC sous-clonées
- Lorsque les cultures phaESC sous-clonées dans les flacons T25 de l’étape 3.22 deviennent suffisamment denses pour passer, aspirez le milieu et ajoutez 1,5 mL de trypsine. Incuber le flacon à 37 °C pendant 5 min. Ensuite, ajoutez 4,5 mL de tampon de lavage et pipette plusieurs fois pour obtenir une suspension à une seule cellule.
- Transférer chaque suspension cellulaire dans un tube de 15 mL et centrifuger le tube à 160 x g pendant 5 min. Retirer le supernatant et résuspendre les granulés cellulaires dans 400 μL de tampon de maintenance haESC complété par 15 μg/mL Hoechst 33342.
- Incuber les suspensions cellulaires à 37 °C pendant 15 min. Après l’incubation, transférer les suspensions cellulaires dans un tube de 5 mL à travers un bouchon de passoire cellulaire. Rincez le bouchon de la passoire cellulaire avec 400 μL supplémentaires de tampon d’entretien haESC, et recueillez les cellules restantes dans le même tube de 5 mL. Garder le tube à 4 °C jusqu’à ce qu’il soit prêt à trier.
- Installez un cytomètre d’écoulement avec une buse de 100 μm selon les instructions du fabricant.
REMARQUE: Hoechst 33342 peut être détecté par excitation à 405 nm. Ici, un laser UV de 355 nm est utilisé pour la détection de Hoechst 33342. - Configurer la suspension cellulaire (à partir de l’étape 5.3) et un nouveau tube de 15 mL contenant 2 mL de tampon de maintenance haESC pour recueillir les cellules triées dans le cytomètre d’écoulement.
- Commencez l’analyse et installez la barrière de tri pour recueillir les cellules haploïdes dans la phase G1/S. Reportez-vous à l’histogramme de la figure 2A pour identifier la population de phase G1/S du CESE.
REMARQUE : Certaines lignées phaESC sous-clonées peuvent ne pas contenir de cellules haploïdes en raison de la diploïdisation complète ou du placage erroné des cellules diploïdes à l’étape 2. Si les cellules haploïdes ne sont pas observées dans la phase G1/S, procéder à un autre échantillon sans tri. Dans notre cas, 5 lignées cellulaires contenaient des cellules haploïdes et 3 lignées cellulaires ne contenaient que des cellules diploïdes sur 8 lignées phaESC sous-clonées. - Après le tri cellulaire, ajouter 5 mL de tampon de lavage le long du mur du tube de collecte de 15 mL. Centrifuger le tube à 160 x g pendant 5 min. Retirer le surnatant.
- Sélectionnez une plaque de taille appropriée pour la culture en fonction du nombre de cellules triées. Utilisez un seul puits d’une plaque de 96 puits, d’une plaque de 24 puits et d’une plaque de 12 puits pour la culture de 1 000 à 40 000 cellules, 40 000 à 200 000 cellules et 200 000 à 400 000 cellules, respectivement. Resuspendez la pastille cellulaire en 120 μL, 600 μL et 1,2 mL de milieu haESC, respectivement.
REMARQUE : Plaquez les cellules à haute densité parce qu’une faible confluence peut causer la mort cellulaire des cellules après le tri. À partir de ce moment, les phaESC sont cultivés sur des puits recouverts de gélatine sans MEFs pour faciliter le génotypage à l’étape 6 et l’application subséquente pour l’injection intracytoplasmique à partir de l’étape 9. - Après avoir transféré la suspension cellulaire dans un puits recouvert de gélatine de la taille appropriée, incuber la plaque à 37 °C dans une atmosphère de CO2 à 5 %.
- Continuez d’élargir les cultures phaESC en répétant les étapes 3.18 à 3.21 avec l’augmentation de la taille des plaques et l’augmentation des volumes de trypsine, tampon de lavage, et haESC moyen. Les cellules sont cultivés sur des puits recouverts de gélatine sans MEF.
- Pour chaque ligne phaESC sous-clonée, préparez une culture dans un puits d’une plaque de 24 puits et un puits d’une plaque de 6 puits pour les étapes 6 et 9, respectivement.
REMARQUE : Certaines cellules de chaque lignée phaESC sous-clonée devraient être congelées dans 300 μL de milieu de congélation comme cryostock dans un réservoir de stockage d’azote liquide avant de passer à l’étape 9.
6. Deuxième génotypage des lignes phaESC sous-clonées sans MEFs
REMARQUE : Une deuxième série de génotypage est effectuée pour confirmer que les lignées phaESC sous-clonées possèdent des suppressions des DMR H19- et IG-et que les allèles de type sauvage sont absents après l’enlèvement des CEM.
- Confirmez au microscope que les cultures dans les puits des plaques de 24 puits de l’étape 5.11 sont exemptes de CEM.
REMARQUE : Si l’on observe les CEM, il est nécessaire de continuer à passer les cultures jusqu’à ce que les CEM aient disparu pour éviter de contaminer le PCR avec l’ADN sauvage des CEM. - Aspirer le milieu des cultures confluentes et ajouter 400 μL de tampon de lyse par puits de la plaque de 24 puits. Après avoir passé plusieurs fois, transférer la suspension cellulaire dans un tube de 1,5 mL.
- Incuber le tube de 1,5 mL à 55 °C pendant 3 h avec le mélange.
- Après l’incubation, ajouter 400 μL d’isopropanol au tube de 1,5 mL et mélanger délicatement jusqu’à ce qu’un précipité d’ADN devienne visible.
- Centrifugez le tube ≥ 10 000 x g pendant 5 min et retirez le supernatant. Laver la pastille d’ADN avec 200 μL d’éthanol à 70 %.
- Centrifugez le tube ≥ 10 000 x g pendant 5 min et retirez le supernatant.
- Séchez le tube dans l’air pendant 10 min, puis réutilisez l’ADN dans 50 μL d’eau.
- Effectuez le Génotypage PCR suivant l’étape 4.7 et l’électrophorèse gel dans l’étape 4.8 pour identifier les lignées cellulaires, qui possèdent des suppressions des H19- et IG-DMRs et sont exempts d’allèles de type sauvage.
REMARQUE : Une image d’une deuxième analyse génotypage typique est affichée à la figure 2B pour référence. Dans notre cas, les 5 lignées cellulaires sélectionnées après la purification des cellules haploïdes (étape 5) étaient exemptes d’allèles de type sauvage et ne possédaient que les allèles de suppression du H19- et IG-DMRs. - Utilisez les lignes phaESC sous-clonées sélectionnées après ce deuxième génotypage sous forme de lignes DKO-phaESC.
7. Superovulation de souris
- Pour la production d’ovocytes MII, initier la superovulation par injection intraperitoneal de 5 UI de la solution de gonadotropine de sérum de jument enceinte (PMSG) dans chaque souris femelle B6D2F1 (4-5 semaines) 63-65 h avant la collecte d’ovocyte.
REMARQUE : La souche de souris B6D2F1 est recommandée pour ce protocole parce que les ovocytes B6D2F1 tolèrent bien l’injection intracytoplasmique et montrent un potentiel de développement élevé aprèsla procédure 17. - Quarante-huit heures après l’injection de PMSG, injectez intraperitoneally 5 UI de solution chorionic humaine de gonadotropine dans chaque souris.
8. Collection d’ovocytes
- Préparer une assiette de 4 puits contenant 700 μL de milieu hyaluronidase dans un puits et 700 μL de milieu M2 dans les 3 puits restants. En outre, préparer un plat de 6 cm avec 7 mL de M2 moyen et un plat centre-bien avec 900 μL de M16 moyen. Préchauffer l’assiette et les plats à 37 °C dans une atmosphère de CO2 à 5 %.
- Le jour de l’injection intracytoplasmique, euthanasier les femelles superovulées (à partir de l’étape 7.2) par dislocation cervicale ou inhalation de CO2 vers 8 heures du matin. Disséquer les oviductes à l’aide de pinces à épiler et de ciseaux. Placer les oviductes dans le plat de 6 cm avec le M2 moyen.
- Relâchez les complexes cumulus-ovocytes (COC) en déchirant l’ampulla des oviductes à l’aide d’une aiguille de 30 G. Transférer les COC dans un milieu hyaluronidase préchauffé et les maintenir à 37 °C dans une atmosphère de CO2 à 5 %.
- Après 2-3 min, recueillir les ovocytes sans cumulus avec une pipette buccae et laver les ovocytes 3 fois en les transférant au milieu M2 frais dans les 3 autres puits de la plaque de 4 puits.
- Recueillir les ovocytes méthaphase II (MII), qui possèdent les premiers corps polaires, dans un plat central avec milieu M16 et maintenir la plaque à 37 °C dans une atmosphère de CO 2 de 5 %jusqu’à utilisation pour l’injection intracytoplasmique à l’étape 12.
9. Traitement et collecte des DKO-phaESCs
- Préparer une culture DKO-phaESC dans un puits d’une plaque de 6 puits sans MEFs à 60-80% confluency un jour avant l’injection intracytoplasmique (à partir de l’étape 5.11).
- Pour induire l’arrêt du cycle cellulaire en phase M, aspirer complètement le milieu et ajouter 2 mL de milieu haESC contenant 0,05 mg/mL demecolcine.
- Après 8 h de traitement par démécolcine, aspirer le milieu et ajouter 800 μL de trypsine.
- Incuber la plaque à 37 °C dans une atmosphère de CO2 à 5 % pendant 5 min, puis ajouter 2 mL de tampon de lavage pour étancher la trypsine et pipette plusieurs fois pour obtenir une suspension à cellule unique.
- Transférer la suspension cellulaire dans un tube de 15 mL. Centrifuger le tube à 160 x g pendant 5 min et enlever le supernatant.
- Resuspendez la pastille cellulaire dans 400 μL de tampon d’entretien haESC contenant 15 μg/mL Hoechst 33342.
- Incuber la suspension cellulaire à 37 °C dans une atmosphère de CO2 de 5 % pendant 15 min. Après l’incubation, transférer la suspension cellulaire dans un tube de 5 mL à travers un bouchon de passoire cellulaire, et garder le tube à 4 °C jusqu’à ce que le tri cellulaire à l’étape 10.
10. Purification des DKO-phaESCs arrêtés en phase M
- Installez un cytomètre d’écoulement avec une buse de 100 μm selon les instructions du fabricant.
REMARQUE: Hoechst 33342 peut être détecté par excitation à 405 nm. Ici, un laser UV de 355 nm est utilisé pour la détection de Hoechst 33342. - Configurer les DKO-phaESCs arrêtés en phase M à partir de l’étape 9.7 et commencer l’analyse. Choisissez une porte de tri appropriée pour la collecte des cellules haploïdes de phase M (2n) de l’échantillon traité avec de la démécolcine.
REMARQUE : Après traitement par démécolcine, on s’attend à ce que deux populations cellulaires correspondent à des cellules haploïdes et diploïdes arrêtées par phase M, comme le montre la figure 3B. L’arrêt de cycle cellulaire après traitement demecolcine est complet, ainsi, aucun pic haploïde d’ADN 1n n’est observé. Ceci est important car les cellules haploïdes de phase M et les cellules diploïdes G1 possèdent la même teneur en ADN (2n) et produiraient des pics qui se chevauchent. - Installez un tube de 15 mL contenant 2 mL de tampon de maintenance haESC pour recueillir les cellules triées dans le cytomètre d’écoulement. Commencez le tri cellulaire.
- Après le tri cellulaire, ajouter 5 mL de tampon de lavage le long du mur du tube de collecte. Centrifuger le tube à 160 x g pendant 5 min et enlever le supernatant.
- Resuspendez les cellules dans un volume approprié de tampon de maintenance haESC pour obtenir une concentration finale de 5 x 105 cellules/mL.
- Transférer la suspension cellulaire dans un tube de 1,5 mL. Gardez le tube sur la glace jusqu’à ce qu’il soit prêt pour l’injection intracytoplasmique à l’étape 12.
11. Préparation des pipettes de fixation et de microinjection
REMARQUE : Pour effectuer l’injection intracytoplasmique (étape 12), plusieurs pipettes de détention et de microinjection sont nécessaires (figure 4A). Ces pipettes peuvent être achetées sur mesure auprès d’un fournisseur commercial ou fabriquées à partir de capillaires en verre appropriés à l’aide d’un pull de micropipette et d’une microforge.
- Tirez les capillaires en verre borosilicate sur un puller micropipette. Pour tirer les capillaires en verre borosilicate sans filament (0,78 x 1,00 x 80 mm), les paramètres suivants sont donnés pour un pull horizontal enflammé(Table of Materials)comme référence, mais diffèrent pour les autres instruments et types capillaires en verre : Heat 510 (Ramp test 480), Pull 0, Velocity 150, Time 175 et Pressure 200 pour les pipettes de tenue; Heat 510 (Ramp test 480), Pull 90, Velocity 140, Time 125 et Pressure 200 pour les pipettes de microinjection.
REMARQUE : Les paramètres optimaux doivent être définis individuellement parce que plusieurs facteurs, y compris l’humidité, le modèle d’un puller de micropipette et le sort des capillaires en verre peuvent affecter la forme des pipettes d’injection. Une forme allongée avec un cône progressif devrait être visée. - Préparation des pipettes de fixation
- Réglez un capillaire tiré préparé à l’étape 11.1 à une microforge. Placez le capillaire sur la perle de verre sur le filament et abaissez le capillaire pour entrer en contact avec la perle de verre tout en chauffant le filament.
- Briser le capillaire en éteigneant le chauffage et en se détachant de la perle de verre de sorte que son diamètre extérieur est de 60-100 μm.
- Placez la pointe capillaire horizontalement pour faire face à la perle de verre sur le filament.
- Chauffer le filament pour permettre au diamètre intérieur de la pointe capillaire de fondre à un diamètre de 10 à 20 μm.
- Déplacez le capillaire de sorte que la perle de verre se positionne à un point ~ 1 mm de la pointe capillaire sans contact. Chauffer le filament pour permettre au capillaire de se plier à un angle de 20°. Démonter le capillaire, appelé pipette de fixation, de la microforge.
REMARQUE : Pour mesurer la taille du capillaire, un reticle oculaire est de préférence installé dans la microforge.
- Préparation de pipettes de microinjection
- Réglez un capillaire tiré préparé à l’étape 11.1 à une microforge. Placez le capillaire sur la perle de verre sur le filament et abaissez le capillaire pour entrer en contact avec la perle de verre tout en chauffant le filament.
- Briser le capillaire en éteignent le chauffage et en se détachant de la perle de verre à une position que son diamètre extérieur est de 6 μm.
- Déplacez le capillaire de sorte que la perle de verre se positionne à un point ~ 1 mm de la pointe capillaire sans contact. Chauffer le filament pour permettre au capillaire de se plier vers le haut à un angle de 20°. Démonter la pipette de microinjection de la microforge et la conserver dans une boîte sécurisée pour une utilisation ultérieure.
REMARQUE : Les pipettes de microinjection sont préparées selon les spécifications suivantes : diamètre extérieur, 6 μm; diamètre intérieur, 4,5-5 μm; angle de flexion, 20°. La définition de la conception optimale de la pipette de microinjection est importante pour le succès de l’injection intracytoplasmique. Un diamètre intérieur trop grand peut empêcher la rupture de la membrane plasmatique du donneur DKO-phESCs (voir la section discussion). Si le diamètre intérieur est trop étroit, il peut empêcher le pipetage lisse du donneur DKO-phESCs. Un angle de flexion < 30° est préférable car un angle de courbure élevé entrave l’effet des impulsions piezo.
12. Injection intracytoplasmique de DKO-phaESCs
- Avant l’injection intracytoplasmique (à partir de l’étape 12.2), préparez une solution de polyvinylpyrrolidone (PVP) en ajoutant 5 mL de milieu M2 dans un tube de 50 mL contenant 0,6 g de PVP et en agitant le tube sur un rocker à 4 °C pendant 2 jours. Une fois que le PVP s’est complètement dissous, la solution est filtrée stérilement et stockée à 4 °C.
- Le jour de l’injection intracytoplasmique, préparer un plat centré avec 900 μL de KSOM moyen et préchauffer le plat à 37 °C dans une atmosphère de CO2 à 5 %.
- Préparer un plat de micromanipulation en alignant des gouttes de 5 μL de solution PVP et de 20 μL de M2 moyen sur un couvercle d’un plat de 10 cm placé à l’envers. Couvrir les gouttes d’huile minérale et placer le plat sur la scène du microscope à injection.
REMARQUE : L’arrangement recommandé du plat de micromanipulation est indiqué dans la figure 4B. - Installez une pipette de fixation sur le micromanipulateur. Remplissez la pipette de microinjection avec l’huile de fluorocarbone à l’aide d’une pointe de microchargeur, et montez-la sur l’actionneur piezo.
- Immerger la pipette de microinjection dans une goutte avec la solution PVP et pipette de haut en bas à plusieurs reprises pour enrober le verre avec PVP et le rendre moins collant. Chargez un petit volume de solution PVP dans la pipette de microinjection, et déplacez la pipette à une baisse avec le milieu M2.
- Immerger la pipette de fixation dans le milieu M2, et se concentrer sur la pipette dans le fond de la goutte.
- Transférez environ 2 μL de suspension DKO-phaESC de l’étape 10.6 à la chute moyenne M2.
- Transférer 10 ovocytes MII de l’étape 8.5 dans la même goutte moyenne M2 à l’aide d’une pipette buccae.
- Pour l’injection, faites pivoter un ovocyte dans la goutte moyenne M2 de sorte que l’espace périvitelline fait face à la pipette de microinjection, et la plaque MII n’est pas située sur le chemin de la pipette de microinjection (Figure 4A). Maintenez l’ovocyte en appliquant une pression négative à travers la pipette de fixation.
REMARQUE : Une plaque MII est visuellement identifiée comme une saillie de l’ooplasme qui est appelée « bosse » et souvent située à côté du premier corps polaire. La plaque MII contient le fuseau méiotique avec des chromosomes attachés. Le toucher de la pipette de microinjection et de la plaque MII doit être évité car les dommages mécaniques du fuseau et des chromosomes peuvent perturber le développement de l’embryon. - Chargez un DKO-phaESC dans le bout de la pipette de microinjection en appliquant la pression négative douce. Rupture de la membrane plasmatique d’un DKO-phaESC par pipetting pour éviter l’injection d’un DKO-phaESC intact(figure 3C; voir discussion).
REMARQUE : Dans le cas où la membrane plasmatique d’un DKO-phaESC n’est pas rompue par pipetage, jetez le DKO-phaESC et chargez un autre DKO-phaESC. - Placez la pipette de microinjection en contact avec la zona pellucida de l’ovocyte, et appliquez une petite quantité de pression négative dans la pipette de microinjection.
- Appliquer des impulsions piezo (intensité, 20; fréquence, 4) pour percer la zona tout en poussant la pointe de la pipette de microinjection vers l’espace périvitelline. Confirmez que la plaque MII, contenant un fuseau et des chromosomes, n’est pas située sur le chemin de la pipette de microinjection.
REMARQUE : Ajustez empiriquement le réglage aux impulsions piezo les plus basses pour le forage à travers la zona afin de minimiser les risques de dommages à l’oolemma. - Jetez le fragment de la zona pellucida de la pipette de microinjection, et placez le DKO-phaESC au bord de la pipette.
- Pénétrer dans l’ovocyte avec la pipette de microinjection de sorte que l’oolemma atteint le côté opposé.
REMARQUE : Ne touchez pas la plaque MII pour éviter les dommages au fuseau et aux chromosomes. - Appliquer une impulsion piezo (intensité, 6; fréquence, 1) pour percer l’oolemma. Assurez-vous que l’oolemma se détend le long de l’arbre de la pipette de microinjection.
REMARQUE : Définissez empiriquement le réglage le plus bas de l’impulsion piezo pour briser l’oolemma afin de minimiser les dommages à l’ovocyte. - Injecter le DKO-phaESC avec un volume minimal de milieu dans l’ooplasme, et retirer la pipette de microinjection en douceur de l’ovocyte.
- Relâchez l’ovocyte injecté de la pipette de fixation, et placez-le sur le côté de la microdrop pour la collecte ultérieure.
- Répétez la procédure d’injection des étapes 12.9 à 12.17 pour les autres ovocytes MII dans la chute moyenne M2.
REMARQUE : Évitez de garder les ovocytes hors de l’incubateur pendant plus de 20 min. D’après notre expérience, un lot de 10 ovocytes peut être manipulé confortablement dans les 15 minutes suivant une formation appropriée. - Transférer le lot d’ovocytes injectés de la goutte moyenne M2 dans un plat avant réchauffé au centre avec un milieu KSOM.
- Gardez le plat pendant 1 h à 37 °C dans une atmosphère de CO2 à 5 %.
- Répétez la manipulation des ovocytes à partir des étapes 12.5 et 12.20 avec des groupes supplémentaires d’ovocytes MII pour obtenir suffisamment d’ovocytes injectés.
13. Activation d’embryons semi-clonés construits
- Préparer deux plats bien au centre avec 900 μL chacun de milieu KSOM et milieu d’activation. Préparer une assiette de 4 puits avec 700 μL de milieu KSOM dans chaque puits. Préchauffer les plats et l’assiette à 37 °C dans une atmosphère de CO2 à 5 %.
- Après 1 h dans le milieu KSOM, transférer les ovocytes injectés de l’étape 12.21 dans le plat avant réchauffé centre-puits avec milieu d’activation.
- Gardez le plat pendant 6 h à 37 °C dans une atmosphère de CO2 à 5 % pour l’activation.
- Après activation, observez quelques embryons semi-clonés former trois corps polaires, qui sont les premier et deuxième corps polaires de l’ovocyte, et le corps pseudo polaire du DKO-phaESC (Figure 3E).
- Lavez les embryons 3 fois en les transférant dans de nouveaux puits avec le milieu KSOM dans une assiette de 4 puits.
- Transférer les embryons dans le plat du puits central avec le milieu KSOM, et garder le plat à 37 °C dans une atmosphère de CO2 de 5% pour un développement ultérieur.
14. Développement d’embryons semi-clonés construits
- Après 1 jour de culture dans le milieu KSOM à partir de l’étape 13.6, plusieurs embryons semi-clonés atteignent le stade à deux cellules (Figure 5A).
- Pour le développement ultérieur d’embryons préimplantatoires in vitro, continuer à culturer les embryons semi-clonés dans le milieu KSOM à 37 °C dans une atmosphère de CO2 de 5%. Transférer les embryons semi-clonés dans un milieu KSOM frais au jour 2. Au jour 4, plusieurs embryons atteindront le stade blastocyste (Figure 5A).
- Pour la dérivation des souris semi-clonées, transférez des embryons à 2 cellules de l’étape 14.1 dans les oviductes des femelles bénéficiaires pseudo-enceintes. Identifier les femelles pseudo-enceintes en s’accoupler à des mâles vasectomisés un jour avant le transfert de l’embryon et les sélectionner en fonction de la présence d’une prise clairement visible le matin de la journée pour le transfert d’embryons (0,5 jour après le coït (dpc)). Environ 19,5 dpc, chiots à terme sont naturellement livrés à partir de femelles bénéficiaires (Figure 5B).
Résultats
Le but de ce protocole est d’appliquer des haESCs comme substitut du sperme pour obtenir des embryons et des souris semi-clonés. À cette fin, une ligne DKO-phaESC transportant le transgène CAG-EGFP a été générée et utilisée pour l’injection intracytoplasmique dans les ovocytes MII. Pour obtenir une ligne phaESC appropriée avec une configuration d’empreinte paternelle, nous avons effectué le génie génétique en utilisant des nucléases Cas9. Une lignée haploïde d’ESC contient des cellules haploïdes et diploïdes qui surgissent en raison d’une tendance inhérente des ESCs haploïdes à la diploïdisation10. Un ensemble de chromosomes haploïdes est une condition préalable au remplacement réussi du génome du sperme. L’analyse du contenu de l’ADN par cytométrie de débit montre la distribution des cellules haploïdes et diploïdes aux phases G0/G1-, S et G2/M (figure 2A).
Pour établir les lignes DKO-phaESC, les lignes phaESC de type sauvage ont été transfectées avec des constructions transposon piggyBac pour l’expression transgénique stable d’EGFP et avec des plasmides CRISPR/Cas9 pour obtenir des suppressions des H19- et des IG-DMRs. Pour exclure les ESC diploïdes et isoler uniquement les ESC haploïdes exprimant l’EGFP, une porte de tri spécifique a été définie (figure 2A). Les cellules haploïdes individuelles positives à l’EGFP ont ensuite été plaquées dans des puits individuels de plaques de 96 puits pour obtenir des sous-clones. Les mangeoires MEF ont été utilisées pour augmenter l’efficacité du placage et la survie des phaESCs transfectés. Après l’expansion des cultures, un premier cycle de génotypage a été effectué par PCR pour identifier les sous-clones porteurs de suppressions des deux DMR. Après que les mangeoires MEF aient été retirées des cultures, un deuxième génotypage a été effectué pour confirmer l’absence d’allèles de type sauvage au H19- et aux DMR IG(figure 2B). Sur un total de 135 sous-clones, nous avons obtenu 5 lignées haploïdes DKO-phESC qui transportaient les allèles supprimés et étaient exemptes d’allèles de type sauvage du H19-DMR et de l’IG-DMR.
Un transgène CAG-EGFP a été introduit dans les DKO-phaESC pour étudier leur contribution aux embryons semi-clonés par visualisation de la fluorescence verte au microscope (Figure 3A). Pour l’injection intracytoplasmique, DKO-phaESCs ont été traités avec demecolcine pour les arrêter dans la phase M. Ainsi, le cycle cellulaire des DKO-phaESCs a été synchronisé avec celui des ovocytes MII. L’analyse de cytométrie de flux a montré 2 populations correspondant à la phase G2/M arrêté haploïde (2n) et cellules diploïdes (4n) après le traitement par démécolcine (Figure 3B). L’absence du pic haploïde de 1n a indiqué que l’arrestation de cycle cellulaire était en grande partie complète. Les ESC haploïdes de phase M ont ensuite été triés et injectés dans les ovocytes. Pour cela, un seul DKO-phaESC a été chargé dans la pipette de microinjection et injecté dans le cytoplasme d’un ovocyte MII (Figure 3C). La membrane plasmatique du DKO-phaESC a été rompue par pipetting dans la pointe de la pipette de microinjection.
Après l’injection, l’expression egfp a été rarement détectée dans les embryons semi-clonés construits car le cytoplasme des DKO-phaESCs s’était dispersé dans le grand cytoplasme de l’ovocyte (figure 3D). Dans de rares cas, une tache ronde d’expression intense d’EGFP a pu être observée dans l’ooplasme. Cette observation a probablement été causée par l’injection accidentelle de DKO-phaESCs intacts. L’échec de la rupture de la membrane cellulaire DKO-phaESC n’est probablement pas compatible avec le développement ultérieur de l’embryon et doit être évité. Une heure après l’injection, les embryons ont été activés par le traitement au chlorure de strontium18. Six heures après le début de l’activation, jusqu’à 3 corps polaires ont été observés au microscope (figure 3E). Ces corps polaires correspondent aux premier et deuxième corps polaires de l’ovocyte et à un pseudo corps polaire du DKO-phaESC7. En outre, deux pronuclei ont été observés sous un microscope différentiel de contraste d’interférence, qui a ressemblé au stade pronucléaire des zygotes après fertilisation normale avec le sperme.
Pour démontrer leur compétence en matière de développement, des embryons semi-clonés ont été cultivés jusqu’au stade blastocyste (figure 5A). De plus, une souris à terme a été obtenue après avoir transféré des embryons semi-clonés à deux cellules dans les oviductes d’une femelle receveur (figure 5B). Comme prévu, la souris dérivée d’un embryon semi-cloné était une femelle car ni les ovocytes ni les phaESCs dérivés des ovocytes ne portent un chromosome Y. La souris semi-clonée était trop normale et produisait une progéniture en bonne santé lorsqu’elle était attelé à un mâle Webster suisse de type sauvage. Jusqu’à présent, la souris semi-clonée était vivante depuis plus de 600 jours sans aucun problème de santé apparent.
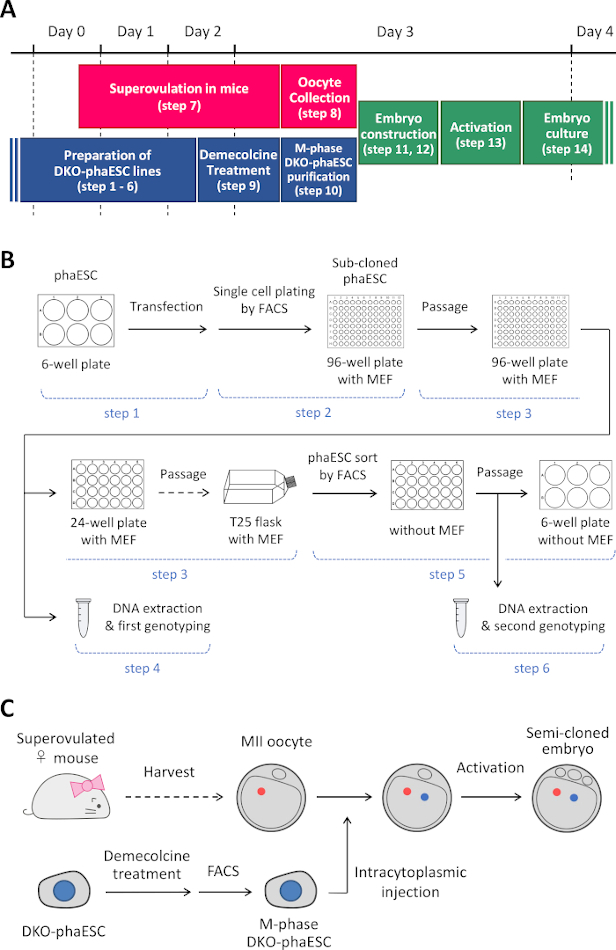
Figure 1 : Aperçu de l’application des DKO-phaESCs comme substitut de sperme. (A) Un calendrier des procédures du protocole. (B) Le schéma montre les étapes pour établir les lignes DKO-phaESC (étapes 1-6). (C) Le schéma montre les étapes pour construire des embryons semi-clonés par injection intracytoplasmique d’un DKO-phaESC dans un ovocyte MII (étapes 7-14). Abréviations: DKO = double-KNOCKOUT; phaESC = cellules souches embryonnaires haploïdes parthogénétiques; FACS = tri cellulaire activé par fluorescence; MEF = fibroblaste embryonnaire de souris. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
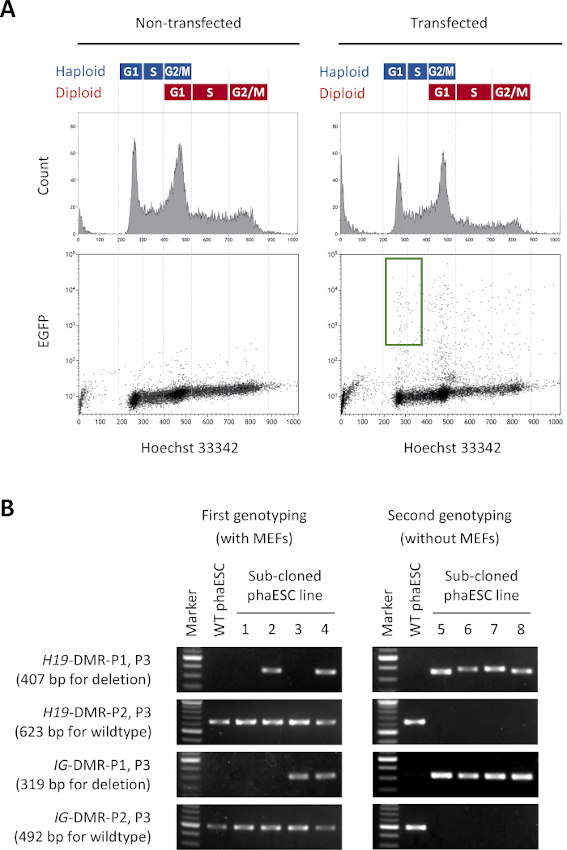
Figure 2 : Création de lignes DKO-phaESC par des suppressions de H19 à 19et d’IG-DMR. (A) Analyse de cytométrie de flux des phaESCs après transfection avec des plasmides de piggyBac pour l’expression stable d’EGFP et avec 4 plasmides CRISPR/Cas9. Les phaESCs non transfected sont montrés comme contrôle. Le profil d’ADN (en haut) montre la distribution du cycle cellulaire des cellules haploïdes et diploïdes. Les cellules haploïdes de phase G1/S exprimant l’EGFP sont indiquées par la porte verte (en bas, à droite). (B) Génotypage des sous-clones phaESC qui ont été cultivés sur les MEFs (premier génotypage) et après l’enlèvement des MEF (deuxième génotypage). Les lignées phaESC sous-clonées 1, 2, 3 et 4 représentent des cellules de type sauvage, des cellules avec une suppression H19-DMR, avec une suppression IG-DMR, et avec des suppressions combinées H19- et IG-DMR, respectivement. Les lignes DKO-phaESC 5-8 possèdent à la fois des suppressions H19-DMR et IG-DMRet sont exemptes d’allèles de type sauvage. Abréviations: DKO = double-KNOCKOUT; phaESC = cellules souches embryonnaires haploïdes parthogénétiques; DMR = région méthylé différemment; MEF = fibroblaste embryonnaire de souris ; EGFP = protéine fluorescente verte améliorée; WT = wildtype. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
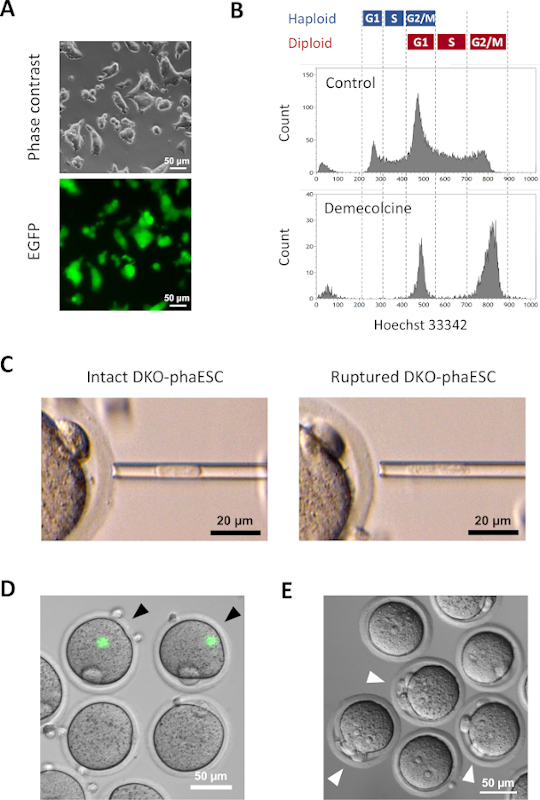
Figure 3 : Injection de DKO-phaESCs arrêtés mitotiquement dans les ovocytes MII. (A) Morphologie d’une culture DKO-phaESC qui porte un transgène CAG-EGFP et des suppressions des DMR H19 et IG; barre d’échelle = 50 μm. (B) Analyse représentative de cytométrie de flux des DKO-phaESCs après arrestation avec demecolcine pendant 8 h. DKO-phaESCs sans traitement de démécolcine sont montrés comme contrôle. (C) Morphologie de DKO-phaESCs dans la pipette de microinjection. Un seul DKO-phaESC intact avant (à gauche) et après (à droite) la rupture de la membrane plasmatique par pipetting est montré; barre d’échelle = 20 μm. (D) Embryons construits à 1 h après injection de DKO-phaESCs dans les ovocytes MII. Une image fusionnée de fluorescence EGFP et de champ lumineux est affichée. Les pointes de flèches noires indiquent des taches rondes d’expression intense de l’EGFP après injection de DKO-phaESCs intacts, qui devraient être évités; barre d’échelle = 50 μm. (E) Une image différentielle de contraste d’interférence des embryons semi-clonés 6 h après l’initiation de l’activation avec le chlorure de strontium est montrée. Les pointes de flèche blanches indiquent des embryons avec 3 corps polaires comprenant un corps pseudo polaire du DKO-phaESC injecté ; barre d’échelle = 50 μm. Abréviations: DKO = double-KNOCKOUT; phaESC = cellules souches embryonnaires haploïdes parthogénétiques; EGFP = protéine fluorescente verte améliorée. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
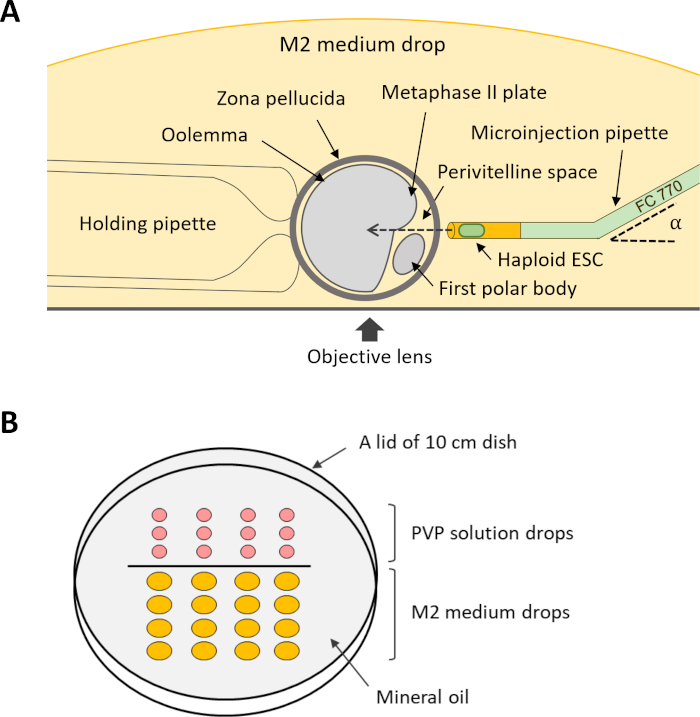
Figure 4 : Schéma de la configuration de l’injection intracytoplasmique de DKO-phaESCs dans les ovocytes. (A) L’arrangement d’une pipette d’injection, d’une pipette de fixation, et d’un ovocyte dans la chambre d’injection est montré. α, angle de courbure de la pipette de microinjection. (B) Disposition des gouttes dans le plat de micromanipulation pour injection intracytoplasmique. Abréviations: DKO = double-KNOCKOUT; phaESC = cellules souches embryonnaires haploïdes parthogénétiques. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
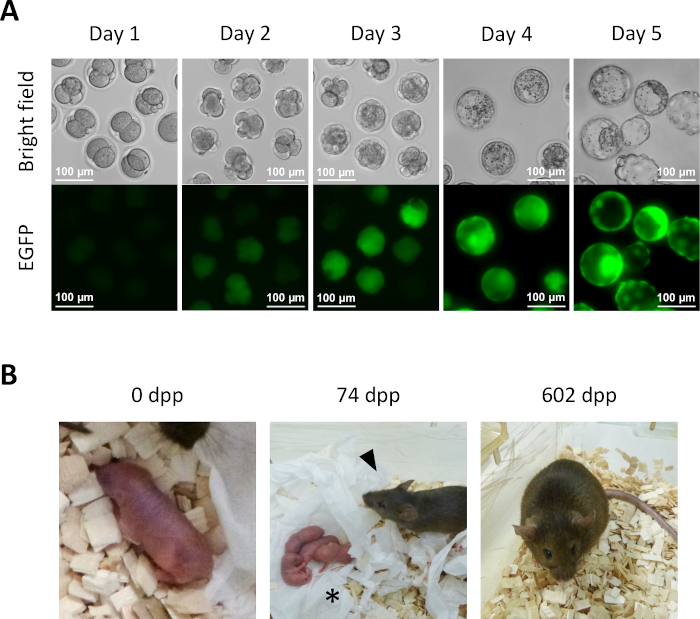
Figure 5 : Développement d’embryons semi-clonés. (A) Développement préimplantatoire d’embryons semi-clonés in vitro. L’expression d’EGFP est au commencement observée dans les embryons à quatre cellules au jour 2 après injection intracytoplasmique. Au jour 4, l’expression intense d’EGFP peut être observée dans les blastocystes ; barre d’échelle = 100 μm. (B) Une souris semi-clonée obtenue après transfert d’embryons à 2 cellules à une femelle receveur. À 74 dpp, la souris semi-clonée (pointe de flèche) a livré ses premiers chiots (astérisque) par naissance naturelle après s’être accouplé avec un mâle Webster suisse de type sauvage. Les embryons semi-clonés et la souris semi-clonée indiquée dans cette figure sont identiques à ceux rapportés dans Aizawa et coll.3. Abréviations: EGFP = protéines fluorescentes vertes améliorées; .dpp, jours post-partum. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
| Solution gélatine | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| Eau | - | 500 mL | - |
| gélatine | - | 1 g | 0.2% |
| Milieu de cellules souches embryonnaires haploïdes (HaESC) | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| NDiff 227 | - | 10 mL | - |
| SCHIR 99021 | 10 mM | 3 μL | 3 μM |
| 0325901 | 10 mM | 1 μL | 1 μM |
| Frv | 1 x 106 UI/mL | 10 μL | 1 000 UI/mL |
| Pénicilline-Streptomycine | 100x (100x) | 100 μL | 1x (1x) |
| Tampon de maintenance HaESC | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| Milieu HaESC | - | 1 mL | - |
| Solution HEPES | 1 M | 20 μL | 20 mM |
| Tampon de lavage | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| DMEM / F-12 | - | 100 mL | - |
| Fraction BSA | 7.5% | 7,1 mL | 0.5% |
| Support MEF | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| DMEM (DMEM) | - | 500 mL | - |
| FBS (FBS) | - | 56 mL | 10% |
| β-Mercaptoéthanol | 14,3 mol/L | 3,9 μL | 100 μM |
| Pénicilline-Streptomycine | 100x (100x) | 5,6 mL | 1x (1x) |
| Tampon de lyse | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| Eau | - | 8,25 mL | - |
| Tris-HCl (pH 8,5) | 2 . | 1 mL | 100 mM |
| Edta | 0,5 M | 100 μL | 5 mM |
| Solution SDS | 10% | 200 μL | 0.2% |
| NaCl (NaCl) | 5 M | 400 μL | 200 mM |
| Proteinase K | 20 mg/mL | 50 μL | 100 μg/mL |
| Moyen d’activation | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| KSOM (KSOM) | - | 1 mL | - |
| Chlorure de strontium | 2 . | 5 μL | 5 mM |
| EGTA (egta) | 0,5 M, pH 8,0 | 4 μL | 2 mM |
| Solution PVP | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| M2 moyen | - | 5 mL | - |
| Pvp | - | 0,6 g | 12% |
| Solution PMSG | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| Pbs | - | 450 μL | - |
| PMSG (pmsg) | 500 UI/mL | 50 μL | 50 UI/mL |
| solution hCG | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| Pbs | - | 450 μL | - |
| hCG (hCG) | 500 UI/mL | 50 μL | 50 UI/mL |
| Hyaluronidase moyen | |||
| composant | Concentration des stocks | Volume / poids | Concentration finale |
| M2 moyen | - | 380 μL | - |
| Hyaluronidase | 10 mg/mL | 20 μL | 0,5 mg/mL |
| Abréviations : LIF = facteur inhibiteur de leucémie ; MEF = fibroblaste embryonnaire de souris ; | |||
| FBS = sérum bovin fœtal; DMEM = Dulbecco’s Modified Eagle Medium; BSA = albumine de sérum bovin; | |||
| EDTA = acide éthylèneediaminetetraacetic; SDS = dodecylsulfate de sodium; EGTA = éthylène-bis (oxyethylenenitrilo)acide tétracétique; | |||
| PBS = solution saline tamponnée de phosphate; PVP = polyvinylpyrrolidone; hCG = gonadotropine chorionique humaine; | |||
| PMSG = gonadotropine de sérum de jument enceinte. | |||
Tableau 1 : Recette de moyen, tampon et solution.
| nom | Séquence (5' à 3') | application | |
| H19-DMR-P1 | GTG GTT AGT TCT ATA TGG GG | Génotypage | |
| H19-DMR-P2 | AGA TGG GGT CAT TCT TTT CC | Génotypage | |
| H19-DMR-P3 | TCT TAC AGT CTG GTC TTG GT | Génotypage | |
| IG-DMR-P1 | TGT GCA GCA GCA AAG CTA AG | Génotypage | |
| IG-DMR-P2 | CCA CAA AAA CCT CCC TTT CA | Génotypage | |
| IG-DMR-P3 | ATA CGA TAC GGC AAC CAA CG | Génotypage | |
| H19-DMR-gRNA1-F | CAC CCA TGA ACT CAG AAG AGA CTG | gRNA (en) | |
| H19-DMR-gRNA1-R | AAA CCA GTC TCT TCT GAG TTC ATG | gRNA (en) | |
| H19-DMR-gRNA2-F | CAC CAG GTG AGA ACC ACT GCT GAG | gRNA (en) | |
| H19-DMR-gRNA2-R | AAA CCT CAG CAG TGG TTC TCA CCT | gRNA (en) | |
| IG-DMR-gRNA1-F | CAC CCG TAC AGA GCT CCA TCGG CAC | gRNA (en) | |
| IG-DMR-gRNA1-R | AAA CGT GCC ATG GAG CTC TGT ACG | gRNA (en) | |
| IG-DMR-gRNA2-F | CAC CCT GCT TAG AGG TAC TAC GCT | gRNA (en) | |
| IG-DMR-gRNA2-R | AAA CAG CGT AGT ACC TCT AAG CAG | gRNA (en) | |
Tableau 2 : Liste des oligonucléotides.
Discussion
Le clonage des mammifères par transfert nucléaire de cellules somatiques (SCNT) a été pionnier dans les années 199019,20,21. Ces développements ont suivi des études de clonage menées 30 ans plus tôt chez lesamphibiens 22. Ce retard considérable reflète la difficulté de l’embryologie et de l’empreinte génomique chez les mammifères. Le développement du SCNT mammifère est à la base de l’application de l’haESC pour remplacer le sperme, qui est détaillée dans ce protocole.
La synchronisation du cycle cellulaire est un facteur important pour le succès de SCNT23. C’est également le cas pour l’injection de haESC dans ce protocole. L’introduction d’un génome donneur dans un receveur nécessite l’appariement des phases du cycle cellulaire afin d’éviter la rupture chromosomique ou les aneuploïdes qui abrogeraient le développement embryonnaire. Le semi-clonage a la complexité supplémentaire que deux génomes et un cytoplaste doivent être compatibles. Un rapport précédent a démontré que l’injection d’haESCs androgènes arrêtés en phase M a donné de meilleurs taux de développement d’embryons semi-clonés que l’injection d’haESCs de phase G1 dans les ovocytes7. Ce rapport suggère la phase M comme point de synchronisation approprié pour le semi-clonage. En conséquence, les phaESCs ont été mitotically arrêtés dans la metaphase avec la mémécolcine et injectés dans l’ooplasme des ovocytes de MII, qui ont été naturellement arrêtés dans la métaphe II de méiose. Fait important, l’arrestation en phase M peut être réalisée dans les ESC de souris avec une efficacité élevée, offrant une excellente synchronisation entre les cycles cellulaires des donneurs et des receveurs.
Pendant la mitose, la membrane nucléaire se décompose et un fuseau se forme auquel les chromosomes répliqués s’attachent. Après l’injection de DKO-phaESCs de phase M, les chromatides sœurs sont séparés en un corps pseudo polaire et le zygote7 (Figure 3E). Par conséquent, un seul ensemble de chromosomes provenant d’un DKO-phaESC contribue à l’embryon semi-cloné. Il est essentiel que les chromatismes sœurs des chromosomes DKO-phaESC puissent être correctement séparés après injection. La membrane plasmatique d’un DKO-phaESC intact empêche la ségrégation dans un corps pseudo polaire. Nous avons en effet observé de rares cas où la membrane plasmatique des DKO-phaESCs n’a pas été rompue, et les embryons ont montré des DKO-phaESCs intacts dans l’ooplasme après injection (figure 3D). Par conséquent, il faut prendre soin d’enlever la membrane plasmatique des DKO-phaESCs par pipetting. Pendant l’injection, il est tout aussi important d’éviter la perturbation du fuseau méiotique de l’ovocyte, qui pourrait conduire à des défauts de ségrégation chromosomique et induire une aneuploïde ainsi.
Chez les mammifères, l’empreinte génomique limite l’application de phaESCs comme substitut du sperme. Les haESCs parthénogénétiques possèdent une configuration maternelle des empreintes génomiques, tandis que le sperme possède une configuration paternelle. Par conséquent, la génération de chiots à terme n’a pas eu lieu après l’injection de phaESCs de type sauvage comme remplacement du sperme. Pour surmonter cette limitation, les suppressions des DMR IG- et H19- sont conçues en phaESCs. La modification de l’expression imprimée aux loci maternelles Igf2-H19 et Gtl2-Dlk1 est suffisante pour modifier la configuration des empreintes génomiques pour permettre la génération de souris semi-clonées avec une fréquence de plus de 5,1 %, basée sur des embryons transférés à 2 cellules. Ces observations suggèrent que le ciblage de deux gènes imprimés transforme le génome des phaESCs en une configuration paternelle fonctionnelle qui peut remplacer le sperme chez la souris. Néanmoins, une modification génétique permanente des phaESCs est nécessaire pour cette stratégie. Comme stratégie alternative, l’haESC androgène peut être considéré. Les haESCs androgènes sont dérivés du génome du sperme et possèdent des empreintes paternelles. Il y a eu des rapports que les haESCs androgènes de wildtype ont contribué en tant que remplacement de sperme pour produire des chiots à terme à une fréquence entre 1.3% et 1.9% des embryonstransférés 4,7,24. Des chiots à terme ont également été obtenus en injectant des haESCs androgènes avec des suppressions de l’IG- et h19-DMRs à une fréquence de 20,2% des embryons transférés à 2 cellules24. L’efficacité accrue du semi-clonage à l’aide d’haESCs androgènes modifiés est probablement parce que les empreintes peuvent devenir instables dans la culture. Les défauts d’impression sont corrigés par les suppressions génétiques des DMR critiques.
Compte tenu de la difficulté d’introduire des modifications génétiques directement dans les génomes des ovocytes ou des spermatozoïdes, les hésics sont un outil précieux pour manipuler les génomes parentaux séparément. L’utilisation d’haESCs comme substitut du sperme offre un avantage remarquable pour l’édition du génome dans la lignée germinale de la souris. Une étude récente a combiné l’édition du génome à base de CRISPR/Cas9 avec l’application d’haESCs pour la caractérisation des régions d’impression qui sont critiques pour le développement embryonnaire12. Cette étude a analysé le rôle des Rasgrf1-DMR en combinaison avec le H19-et IG-DMRs dans le développement de souris bimaternales, et la fonction de 7 DMR différents dans le développement de souris bipaternales. La méthode de substitution des HAESCs au sperme a servi de base à des approches de dépistage génétique pour identifier les acides aminés clés dans la protéine MDN1 dans le développement primordial des cellules germinales et pour identifier les gènesdans le développement osseux 24,25,26. Les études sur l’empreinte génomique et le dépistage génétique afin d’identifier les facteurs clés du développement embryonnaire sont des approches considérables pour l’application des HAESCs comme génomes gametiques.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions le Dr Giulio Di Minin pour la dérivation des lignes phaESC et le Dr Remo Freimann pour l’opération de cytométrie du débit. Nous reconnaissons également Mme Michèle Schaffner et M. Thomas M. Hennek pour leur soutien technique au transfert d’embryons. Ces travaux ont été soutenus par la Fondation nationale suisse pour la science (31003A_152814/1).
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mLTube | Eppendorf | 0030 125.150 | haESC culture |
| 15 ml Tube | Greiner Bio-One | 188271 | haESC culture |
| 12-well plate | Thermo Scientific | 150628 | haESC culture |
| 24-well plate | Thermo Scientific | 142475 | haESC culture |
| 6-well plate | Thermo Scientific | 140675 | haESC culture |
| 96-well plate | Thermo Scientific | 167008 | haESC culture |
| β-Mercaptoethanol | Merck | M6250 | haESC culture |
| BSA fraction | Gibco | 15260-037 | haESC culture |
| CHIR 99021 | Axon | 1386 | haESC culture |
| Demecolcine solution | Merck | D1925 | haESC culture |
| DMEM | Gibco | 41965-039 | haESC culture |
| DMEM / F-12 | Gibco | 11320-033 | haESC culture |
| DR4 mouse | The Jackson Laboratory | 3208 | haESC culture |
| EDTA | Merck | E5134 | haESC culture |
| FBS | biowest | S1810-500 | haESC culture |
| Freezing medium | amsbio | 11897 | haESC culture |
| Gelatin | Merck | G1890 | haESC culture |
| Hepes solution | Gibco | 15630-056 | haESC culture |
| Irradiated MEF | Gibco | A34966 | haESC culture |
| LIF (Leukemia inhibitory factor) | (Homemade) | - | haESC culture |
| Lipofection reagent | Invitrogen | 11668019 | haESC culture |
| NDiff 227 | Takara | Y40002 | haESC culture |
| PD 0325901 | Axon | 1408 | haESC culture |
| Pen Strep | Gibco | 15140-122 | haESC culture |
| piggyBac plasmid carrying a CAG-EGFP transgene | Addgene | #40973 | haESC culture |
| piggyBac transposase plasmid | System Biosciences | PB210PA-1 | haESC culture |
| pX330 plasmid | Addgene | #42230 | haESC culture |
| pX458 plasmid | Addgene | #48138 | haESC culture |
| Trypsin | Gibco | 15090-046 | haESC culture |
| DNA polymerase | Thermo Scientific | F549S | Genotyping |
| dNTP Mix | Thermo Scientific | R0192 | Genotyping |
| Ethanol | Merck | 100983 | Genotyping |
| Hydrochloric acid | Merck | 100317 | Genotyping |
| Isopropanol | Merck | 109634 | Genotyping |
| Proteinase K | Axon | A3830 | Genotyping |
| SDS | Merck | 71729 | Genotyping |
| Sodium chloride | Merck | 106404 | Genotyping |
| ThermoMixer | Epeendorf | 5384000012 | Genotyping |
| Tris | Merck | T1503 | Genotyping |
| 5 mL Tube | Falcon | 352063 | Flow cytometry |
| 5 mL Tube with cell strainer cap | Falcon | 352235 | Flow cytometry |
| Hoechst 33342 | Invitrogen | H3570 | Flow cytometry |
| MoFlo Astrios EQ | Beckman Coulter | B25982 | Flow cytometry |
| 1 mL syringe | Codan | 62.1640 | Superovulation |
| 30-gauge 1/2 inch needle | Merck | Z192362-100EA | Superovulation |
| B6D2F1 mouse | The Jackson Laboratory | 100006 | Superovulation |
| hCG | MSD animal health | 49451 | Superovulation |
| PBS | Gibco | 10010015 | Superovulation |
| PMSG | Avivasysbio | OPPA1037 5000 IU | Superovulation |
| 10-cm dish | Thermo Scientific | 150350 | Embryo manipulation |
| 4-well plate | Thermo Scientific | 179830 | Embryo manipulation |
| 50 mL tube | Greiner Bio-One | 227261 | Embryo manipulation |
| 6 cm dish | Thermo Scientific | 150288 | Embryo manipulation |
| Center-well dish | Corning | 3260 | Embryo manipulation |
| Digital rocker | Thermo Scientific | 88880020 | Embryo manipulation |
| EGTA | AppliChem | A0878 | Embryo manipulation |
| Hyaluronidase | Merck | H4272 | Embryo manipulation |
| KSOM | Merck | MR-020P-5F | Embryo manipulation |
| M16 medium | Merck | M7292 | Embryo manipulation |
| M2 medium | Merck | M7167 | Embryo manipulation |
| Mineral oil | Merck | M8410 | Embryo manipulation |
| Glass capillary | Merck | P1049-1PAK | Embryo manipulation |
| Stereomicroscope | Nikon | SMZ745 | Embryo manipulation |
| Strontium chloride | Merck | 13909 | Embryo manipulation |
| 5ml syringe | Codan | 62.5607 | Microinjection |
| Borosilicate glass cappilary | Science product | GB100T-8P | Microinjection |
| CellTram 4r Oil | Eppendorf | 5196000030 | Microinjection |
| Fluorinert FC-770 | Merck | F3556 | Microinjection |
| Holding pipette | BioMedical Instruments | - | Microinjection |
| Inverted microscope | Nikon | Eclipse Ti-U | Microinjection |
| Microforge | Narishige | MF-900 | Microinjection |
| Microinjection pipette | BioMedical Instruments | - | Microinjection |
| Microlaoder tips | Eppendorf | 5252 956.003 | Microinjection |
| Micromanipulaor arms | Narishige | NT-88-V3 | Microinjection |
| Micropipette puller | Sutter Instrument | P-1000 | Microinjection |
| PiezoXpert | Eppendorf | 5194000016 | Microinjection |
| PVP (Polyvinylpyrrolidine) | Merck | PVP360 | Microinjection |
| Syringe filter | SARSTEDT | 83.1826.001 | Microinjection |
Références
- Ellegren, H., Galtier, N. Determinants of genetic diversity. Nature Reviews: Genetics. 17 (7), 422-433 (2016).
- Huijbers, I. J. Generating genetically modified mice: A decision guide. Methods in Molecular Biology. 1642, 1-19 (2017).
- Aizawa, E., Dumeau, C. E., Freimann, R., Di Minin, G., Wutz, A. Polyploidy of semi-cloned embryos generated from parthenogenetic haploid embryonic stem cells. PloS One. 15 (9), 0233072 (2020).
- Li, W., et al. Androgenetic haploid embryonic stem cells produce live transgenic mice. Nature. 490 (7420), 407-411 (2012).
- Li, Z., et al. Birth of fertile bimaternal offspring following intracytoplasmic injection of parthenogenetic haploid embryonic stem cells. Cell Research. 26 (1), 135-138 (2016).
- Wan, H., et al. Parthenogenetic haploid embryonic stem cells produce fertile mice. Cell Research. 23 (11), 1330-1333 (2013).
- Yang, H., et al. Generation of genetically modified mice by oocyte injection of androgenetic haploid embryonic stem cells. Cell. 149 (3), 605-617 (2012).
- Zhong, C., et al. Parthenogenetic haploid embryonic stem cells efficiently support mouse generation by oocyte injection. Cell Research. 26 (1), 131-134 (2016).
- Elling, U., et al. Forward and reverse genetics through derivation of haploid mouse embryonic stem cells. Cell Stem Cell. 9 (6), 563-574 (2011).
- Leeb, M., Wutz, A. Derivation of haploid embryonic stem cells from mouse embryos. Nature. 479 (7371), 131-134 (2011).
- Surani, M. A., Barton, S. C., Norris, M. L. Development of reconstituted mouse eggs suggests imprinting of the genome during gametogenesis. Nature. 308 (5959), 548-550 (1984).
- Li, Z. K., et al. Generation of bimaternal and bipaternal mice from hypomethylated haploid ESCs with imprinting region deletions. Cell Stem Cell. 23 (5), 665-676 (2018).
- Elling, U., et al. Derivation and maintenance of mouse haploid embryonic stem cells. Nature Protocols. 14 (7), 1991-2014 (2019).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shuai, L., et al. Generation of mammalian offspring by haploid embryonic stem cells microinjection. Current Protocols in Stem Cell Biology. 31, 1-15 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923 (2012).
- Behringer, R., Gertsenstein, M., Nagy, K. V., Nagy, A. . Manipulating the Mouse Embryo: A Laboratory Manual. , (2014).
- O'Neill, G. T., Rolfe, L. R., Kaufman, M. H. Developmental potential and chromosome constitution of strontium-induced mouse parthenogenones. Molecular Reproduction and Development. 30 (3), 214-219 (1991).
- Campbell, K. H., McWhir, J., Ritchie, W. A., Wilmut, I. Sheep cloned by nuclear transfer from a cultured cell line. Nature. 380 (6569), 64-66 (1996).
- Wakayama, T., Perry, A. C., Zuccotti, M., Johnson, K. R., Yanagimachi, R. Full-term development of mice from enucleated oocytes injected with cumulus cell nuclei. Nature. 394 (6691), 369-374 (1998).
- Wilmut, I., Schnieke, A. E., McWhir, J., Kind, A. J., Campbell, K. H. Viable offspring derived from fetal and adult mammalian cells. Nature. 385 (6619), 810-813 (1997).
- Gurdon, J. B. Adult frogs derived from the nuclei of single somatic cells. Developmental Biology. 4, 256-273 (1962).
- Campbell, K. H., Alberio, R. Reprogramming the genome: role of the cell cycle. Reproduction Supplement. 61, 477-494 (2003).
- Zhong, C., et al. CRISPR-Cas9-mediated genetic screening in mice with haploid embryonic stem cells carrying a guide RNA library. Cell Stem Cell. 17 (2), 221-232 (2015).
- Bai, M., et al. Targeted genetic screening in mice through haploid embryonic stem cells identifies critical genes in bone development. PLoS Biology. 17 (7), 3000350 (2019).
- Li, Q., et al. CRISPR-Cas9-mediated base-editing screening in mice identifies DND1 amino acids that are critical for primordial germ cell development. Nature Cell Biology. 20 (11), 1315-1325 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon