Method Article
Applicazione delle cellule staminali embrionali aploidi partinogenetiche del topo come sostituto dello sperma
In questo articolo
Riepilogo
Questo articolo mira a dimostrare l'uso di cellule staminali embrionali aploidi aploidi partenogenetiche come sostituto dello sperma per la costruzione di embrioni semi-clonati.
Abstract
Negli organismi con riproduzione sessuale, le cellule germinali sono la fonte di cellule totipotenti che si sviluppano in nuovi individui. Nei topi, la fecondazione di un ovocita da parte di uno spermatozoo crea uno zigote totipotente. Recentemente, diverse pubblicazioni hanno riferito che le cellule staminali embrionali aploidi (ESES) possono sostituire i genomi gametici e contribuire agli embrioni, che si sviluppano in topi. Qui, presentiamo un protocollo per applicare gli esHC partenogenetici come sostituto dello sperma per costruire embrioni mediante iniezione intracitoplasmatica negli ovociti. Questo protocollo consiste in fasi per preparare gli ESCS come sostituzione dello sperma, per l'iniezione di cromosomi haESC negli ovociti e per la coltura di embrioni semi-clonati. Gli embrioni possono produrre fertili topi semi-clonati dopo il trasferimento dell'embrione. L'uso degli haESC come sostituzione dello sperma facilita l'editing del genoma nella linea germinale, studi sullo sviluppo embrionale e indagini sull'imprinting genomico.
Introduzione
Nei mammiferi, i gameti sono le uniche cellule che trasmettono informazioni genetiche alla prossima generazione. La fusione di un ovocita e di uno spermatozoo forma uno zigote diploide che si sviluppa in un animale adulto. Le mutazioni nei genomi gametici sono quindi ereditate dalla prole e guidano la variazione genetica nellaspecie 1. L'introduzione di mutazioni nella linea germinale è stata applicata per produrre animali geneticamente modificati per diversi studi biologici, tra cui la caratterizzazione della funzione genica e la modellazione delle malattie. Sia gli ovociti che gli spermatozoi sono cellule terminali differenziate e altamente specializzate che hanno cessato la proliferazione. Pertanto, la modifica diretta dei gameti è tecnicamente difficile e sono stati sviluppati approcci specializzati. Le modifiche genetiche possono essere introdotte nella linea germinale del topo mediante iniezione di ESC geneticamente modificati nelle blastocsti, dove si integrano nell'embrione in via di sviluppo e colonizzano la linea germinale. Inoltre, la modificazione genetica degli zigoti utilizzando approcci di editing del genoma, incluso il sistema CRISPR/Cas9, è diventata ampiamenteadottata 2.
Recentemente è stato segnalato un approccio eccezionale, che applica gli haESC come sostituto di un genoma gametico3,4,5,6,7,8. Gli HaESC sono linee cellulari derivate dalla massa cellulare interna delle blastoc aploidi partenogenetiche o androgenetiche e possiedono un singolo insieme di cromosomi4,7,9,10. È stato dimostrato che sia gli haESC partenogenetici che androgenetici possono contribuire al genoma dei topi semi-clonati dopo l'iniezione intracitoplasmatica negli ovociti. A differenza di altri approcci, i genomi degli haESC possono essere modificati direttamente in coltura grazie alla loro capacità di autori ricambio. L'introduzione di modifiche genetiche nella linea germinale sostituendo lo sperma con gli eSCS è un metodo importante per gli studi biologici. Prevede la possibilità di coltura separata e manipolare il genoma materno o paterno, che derivano rispettivamente da haESC partenogenetici o androgenetici. Gli HaESC possono quindi essere usati come sostituzione del genoma gametico, il che è particolarmente vantaggioso per gli studi sull'imprinting genomico, sull'espressione specifica dell'allele e sui processi specifici dei genitori.
Nei topi, sono necessarie informazioni genomiche materne e paterna per il normale sviluppodell'embrione 11. Pertanto, i cuccioli a tempo pieno non hanno potuto essere ottenuti quando gli haESC partenogenetici di tipo selvatico (faESC) sono stati iniettati per sostituire il genoma dello sperma5,8. Per superare il blocco dello sviluppo, l'imprinting genomico del genoma materno degli haESC partenogenetici deve essere corretto in una configurazione paterna. Ciò può essere ottenuto manipolazione delle regioni metilate differenzialmente (DR). Ad oggi, sono state studiate decezioni mirate di H19-DMR, Gtl2-Dlk1 IG-DMR e Rasgrf1-DMR per reprimere i geni espressi maternamente nelle fasi3,5,8,12. Questi studi hanno dimostrato che le decezioni sia dell'H19-DMR che dell'IG-DMR sono sufficienti per convertire una materna in una configurazione di impronta paterna che può sostituire i cromosomi spermatici. L'iniezione intracitoplasmatica di faESC che trasportano le due deprezzazioni DMR in ovociti ha prodotto cuccioli semi-clonati con una frequenza compresa tra il 5,1% e il 15,5% degli embrioni a 2 cellule trasferiti.
Questo protocollo si basa sull'applicazione di faESC con soppressioni sia di H19-DMR che di IG-DMR, che noi terminiamo faESC a doppio knockout (DKO-faSEC). Forniamo istruzioni dettagliate per la modifica dell'imprinting genomico nei faESC per stabilire linee DKO-phaESC, per l'iniezione di DKO-faESC negli ovociti come sostituto di un genoma spermatico, per la coltura di embrioni semi-clonati a blastocsti e per ottenere topi semi-clonati. Questo protocollo è un riferimento per i ricercatori che richiedono una manipolazione precisa e diretta del genoma paterno e la generazione di embrioni e topi semi-clonati.
Protocollo
Tutti gli esperimenti sugli animali sono stati eseguiti con la licenza ZH152/17 in conformità con gli standard e i regolamenti della Commissione etica cantonale di Zurigo e della struttura animale EPIC presso l'Istituto di scienze della salute molecolare, Zurigo del 1° Zurigo.
NOTA: questo protocollo inizia con l'eliminazione dei DLF H19 e IGnei FASI. Per informazioni dettagliate su come stabilire le righe phaESC, fare riferimento alle relazionipubblicate 10,13. Una panoramica e un intervallo di tempo di questo protocollo (passaggi da 1 a 14) sono forniti nella figura 1A; i supporti, le soluzioni e i buffer sono elencati nella tabella 1. La procedura per stabilire le linee DKO-phaESC (passaggi da 1 a 6) è illustrata nella figura 1Be la strategia per la costruzione di embrioni semi-clonati (passaggi da 7 a 14) è illustrata nella figura 1C.
1. Trasfezione di plasmidi per la cancellazione di H19-DMR e IG-DMR nei faSESC
- Preparare i plasmidi CRISPR/Cas9 per la coespressioni delle nucleasi Cas9 e guidare gli RNA per indirizzare le decezioni di H19-DMR e IG-DMR. Ligate quattro coppie di oligo di RNA guida (H19-DMR-gRNA1-F, R; H19-DMR-gRNA2-F, R; IG-DMR-gRNA1-F, R; IG-DMR-gRNA2-F, R elencato nella tabella 2) in pX330 plasmidi.
NOTA: Fare riferimento al protocollo pubblicato sulla procedura dettagliata per la preparazione di questi 4 plasmidi CRISPR/Cas914. In alternativa, i plasmidi disponibili per ceppi generali di topi sono accessibili anche attraverso un repository (Table of Materials). - Rivestire la superficie di un pozzo di una piastra a 6 pozze con 1 mL di soluzione di gelatina allo 0,2% per incubazione a 37 °C per 10 min.
- La piastra 2 × 105 faESC di tipo selvatico sul pozzo rivestito di gelatina in mezzo eSE senza antibiotici e incubare la piastra a 37 °C in un'atmosfera di CO2 del 5% per 1 giorno.
NOTA: Gli antibiotici vengono omessi dal mezzo per aumentare l'efficienza della successiva lipofezione. Abbiamo usato i faESC di tipo selvatico al passaggio 10. Si consiglia di utilizzare i faESC a passaggio precoce, ma una varietà di numeri di passaggio sono stati utilizzati con successo8. La correlazione tra il numero di passaggio e l'efficienza di ottenere embrioni e topi semi-clonati non è attualmente nota. - Transfette phaESC nel pozzo di una piastra a 6 pozzetti (dal passaggio 1.3) con 6 plasmidi contemporaneamente usando il reagente di lipofezione: 50 ng piggyBac plasmide che trasporta un transgene CAG-EGFP, 50 ng piggyBac trasposasi plasmide e 600 ng di ciascuno dei 4 plasmidi CRISPR / Cas9 (dal passo 1.1). Fare riferimento al protocollo del produttore sulla procedura dettagliata della trasfezione.
NOTA: Due plasmidi piggyBac sono utilizzati per integrare un transposon per l'espressione onnipresente della proteina fluorescente verde migliorata (EGFP) nel genoma dei feSC. Se la marcatura GFP delle cellule non è richiesta, questi due plasmidi possono essere sostituiti da un plasmide CRISPR/Cas9 per l'espressione transitoria delle proteine della fluorescenza (ad esempio, pX458 plasmide) invece di uno dei plasmidi pX330. L'espressione EGFP transitoria può quindi essere utilizzata per ordinare le celle trasfette. - Due giorni dopo la trasfezione, aspirare il mezzo e aggiungere 800 μL di tripina.
- Incubare la piastra a 37 °C in un'atmosfera di CO2 del 5% per 5 minuti. Quindi, aggiungere 2 mL di tampone di lavaggio per temprare la tripina e pipettare più volte per ottenere una sospensione a cella singola.
- Trasferire la sospensione cellulare in un tubo da 15 ml.
- Centrifugare il tubo a 160 x g per 5 minuti e rimuovere il supernatante.
- Rimescolare il pellet cellulare in 400 μL di tampone di manutenzione haESC integrato con 15 μg/mL Hoechst 33342.
NOTA: Per ridurre la tossicità potenziale della Hoechst 33342, sono stati utilizzati 1 μg/mL Hoechst 33342 e 50 μM verapamil invece di 15 μg/mL Hoechst 3334215. - Incubare la sospensione cellulare a 37 °C in un'atmosfera di CO2 del 5% per 15 minuti. Dopo l'incubazione, trasferire la sospensione cellulare in un tubo da 5 ml attraverso un tappo del filtro cellulare e mantenere il tubo a 4 °C fino a quando non è pronto per l'uso nella fase successiva (sezione 2).
2. Placcatura a cella singola di fasci trasfettati utilizzando un citometro a flusso
- Un giorno prima di smistare i FESC trasfetturati, i fibroblasti embrionali di topo irradiati a piastre (MEF) irradiati su piastre a 96 pozzetti rivestiti di gelatina ad una densità di 4 ×10 4 cellule/cm2 nel mezzo MEF. In genere, 6 piastre sono preparate per stabilire una linea phaESC con eliminazioni mirate. Incubare le piastre a 37 °C in un'atmosfera di CO2 al 5%.
NOTA: I MEF irradiati sono disponibili in commercio. Utilizziamo MEF derivati da embrioni E12.5 di topi DR4. Sebbene gli eSIC possano crescere su piastre rivestite di gelatina senza MEF, raccomandiamo i MEF per aumentare la vitalità dei singoli ESCS ordinati. - Il giorno dello smistamento, aspirare il mezzo MEF dalle piastre da 96 poggia e aggiungere 120 μL di mezzo fresco haESC per pozzo. Mantenere le piastre a 37 °C in un'atmosfera di CO2 del 5%.
- Impostare uno smistatore di celle con un ugello da 100 μm secondo le istruzioni del produttore. Un laser UV a 355 nm e un laser blu a 488 nm sono utilizzati per l'eccitazione della fluorescenza Hoechst 33342 ed EGFP, rispettivamente.
NOTA: In alternativa, Hoechst 33342 può essere rilevato per eccitazione con 405 nm. - Ordinare i feSC trasfettati nel tubo da 5 ml (dal passaggio 1.10) utilizzando un cancello per la raccolta di cellule aploidi nella fase G1/S che mostrano l'espressione EGFP. Depositare una singola cella in ogni pozzo delle piastre a 96 potte dal passo 2.2.
NOTA: Il rilevamento della colorazione Hoechst 33342 distingue generalmente 3 picchi di cellule con un contenuto di DNA 1n, 2n e 4n, che corrispondono rispettivamente alle cellule aploidi in fase G1, una miscela di cellule aploidi in fase G2/M e cellule diploidi in fase G1 e cellule diploidi in fase G2/M. Le cellule aploidi in fase G1/S sono identificate come il picco con minore intensità di fluorescenza hoechst 33342. Un risultato rappresentativo e un cancello di smistamento sono riportati nella figura 2A. - Dopo la placcatura, incubare le piastre a 96 po 'a 37 °C in un'atmosfera di CO2 del 5%.
3. Sottoclonazione di faESC trasfette
- Tre giorni dopo la placcatura a una cellula, le colonie possono essere osservate in diversi pozzi delle piastre a 96 pozzi al microscopio. Segna i pozzi in cui crescono solo singole colonie.
NOTA: Secondo la nostra esperienza, singole colonie sono state osservate nel 20%-40% dei pozzi delle piastre a 96 pozzi. - Il giorno 4 dopo la placcatura a cella singola, sostituire metà del mezzo con un nuovo mezzo haESC nei pozzi con singole colonie.
- Un giorno prima della passatura (al giorno 4 o 5 dopo la placcatura a singola cellula), i MEF irradiati a piastre di gelatina rivestite con 96 possedimento ad una densità di 4 ×10 4 celle/cm2 in mezzo MEF. Mantenere le piastre a 37 °C in un'atmosfera di CO2 del 5%.
- Dopo 5 o 6 giorni di placcatura a cella singola, selezionare i pozzi delle piastre a 96 potte contenenti singole colonie con un diametro superiore a 150 μm.
NOTA: Circa 100 pozzi sono preferibilmente selezionati per stabilire una linea phaESC con le eliminazioni DMR mirate. - Aspirare il mezzo nei pozzatti selezionati e aggiungere 30 μL di tripina. Incubare le piastre da 96 pozzetti a 37 °C in un'atmosfera di CO2 del 5% per 5 minuti. Quindi, aggiungere 30 μL di tampone di lavaggio a ciascun pozzo per temprare la tripina.
- Aggiungere 140 μL di mezzo haESC in ogni pozzo e pipettare più volte per ottenere singole cellule.
- Aspirare il mezzo MEF dai pozzi delle piastre da 96 pozzi preparate al passaggio 3.3.
- Trasferire i faESC da ogni pozzo dal passaggio 3.6 in un pozzo fresco della nuova piastra da 96 pozze dal passo 3.7.
- Incubare le piastre da 96 potte contenenti subcloni phaESC a 37 °C in un'atmosfera di CO2 al 5%.
- Il giorno successivo, aspirare tutto il mezzo da ogni pozzo e aggiungere 120 μL di nuovo mezzo haESC. Incubare le piastre a 37 °C in un'atmosfera di CO2 al 5%.
- Un giorno prima che le cellule diventino confluenti per la passatura, placcare i MEF irradiati su piastre a 24 possedimento rivestite di gelatina ad una densità di 4 × 104 celle/cm2 in mezzo MEF. Mantenere le piastre a 37 °C in un'atmosfera di CO2 del 5%.
- Quando i faESC sono diventati confluenti per il passaging, aspirare il mezzo e aggiungere 30 μL di tripina. Incubare le piastre da 96 pozzi a 37 °C per 5 minuti.
- Aggiungere 90 μL di tampone di lavaggio in ogni pozzo per temprare la tripina. Pipetta più volte per ottenere singole celle.
- Aspirare il mezzo MEF dai pozzi delle piastre a 24 pozza dal passo 3.11 e aggiungere 600 μL di mezzo fresco haESC.
- Trasferire 60 μL della sospensione dei subcloni phaESC da ogni pozzo nel passaggio 3.13 in un nuovo pozzo delle piastre a 24 pozza dal passo 3.14. Mantenere la sospensione rimanente di ogni subclone phaESC per l'estrazione e la genotipizzazione del DNA nella fase 4.
- Incubare le piastre a 24 po 'con i subcloni phaESC a 37 °C in un'atmosfera di CO2 al 5%.
- Un giorno prima che le colture cellulari raggiungano la densità di passaging, placcare i MEF irradiati su piastre a 6 porci rivestite di gelatina ad una densità di 4 × 104 celle/cm2 in mezzo MEF. Mantenere le piastre a 37 °C in un'atmosfera di CO2 del 5%.
- Quando i faESC diventano abbastanza confluenti per il passaging, aspirare il mezzo e aggiungere 250 μL di tripina. Incubare le piastre a 24 pozzi a 37 °C per 5 minuti.
NOTA: Dopo il genotipizzazione nel passaggio 4.9, è necessario passare solo le righe phaESC con eliminazioni sia dell'H19-DMR che dell'IG-DMR. - Aggiungere 750 μL di tampone di lavaggio in ogni pozzo per temprare la tripina. Pipetta più volte per ottenere una singola sospensione cellulare. Trasferire la sospensione cellulare in un tubo da 15 ml.
- Centrifugare il tubo a 160 x g per 5 minuti, rimuovere il supernatante e rimescolare il pellet cellulare in 2 ml di mezzo eSESC.
- Aspirare il mezzo MEF dai pozzi delle piastre a 6 pozzi dal passo 3.17. Trasferire la sospensione phaESC da ogni tubo dal passo 3.20 in un nuovo pozzo. Mantenere le piastre a 37 °C in un'atmosfera di CO2 del 5%.
- Espandere i cloni cellulari ripetendo i passaggi da 3.17 a 3.21 e aumentando le dimensioni della piastra e i volumi di tripina, buffer di lavaggio e mezzo haESC. Preparare un pallone T25 per ogni riga di MEF phaESC sotto-clonata per il passaggio 5.
NOTA: Si consiglia di congelare un'aliquota di ogni linea phaESC sub-clonata in 300 μL di mezzo di congelamento e di mantenere un criostock in conservazione di azoto liquido prima di procedere al passaggio 5.
4. Primo genotipizzazione di linee phaESC sotto-clonate con MEF
- Per estrarre il DNA genomico dalla sospensione cellulare rimanente dal passaggio 3.15, aggiungere 200 μL di tampone di lysis ad ogni pozzo delle piastre a 96 potte. Trasferire la sospensione cellulare su un tubo da 1,5 ml. Risciacquare ogni bene con un ulteriore tampone di lisi di 200 μL per recuperare tutte le cellule rimanenti e raccogliere nello stesso tubo da 1,5 ml.
- Incubare il tubo da 1,5 ml a 55 °C per 3 ore con miscelazione.
- Dopo l'incubazione, aggiungere 460 μL di isopropanolo a ciascun tubo da 1,5 ml e mescolare delicatamente fino a quando un precipitato di DNA diventa visibile.
- Centrifugare i tubi a ≥ 10.000 x g per 5 minuti e rimuovere il supernatante. Lavare i pellet di DNA con 200 μL di 70% di etanolo.
- Centrifugare i tubi a ≥ 10.000 x g per 5 minuti e rimuovere il supernatante.
- Asciugare i tubi nell'aria per 10 minuti e quindi rimorsi il DNA in 20 μL di acqua.
- Eseguire la reazione a catena della polimerasi (PCR) utilizzando la DNA polimerasi termostabile seguendo il protocollo del produttore.
NOTA: Le coppie di primer per PCR sono elencate nella tabella 2 e sono utilizzate come segue: H19-DMR-P1 e P3 (407 bp per H19-DMR eliminato); H19-DMR-P2 e P3 (623 bp per wildtype H19-DMR); IG-DMR-P1 e P3 (319 bp per IG-DMR cancellato); IG-DMR-P2 e P3 (492 bp per wildtype IG-DMR). Il profilo di temperatura della PCR per tutte le coppie di primer è il seguente: 30 s 98 °C, 35 x (10 s 98 °C, 20 s 56 °C, 30 s 72 °C), 5 min 72 °C. La lunghezza dei frammenti di DNA amplificati per le deezioni H19-DMR e IG-DMR mostra alcune variazioni a causa dell'unione finale non omologa associata all'editing mediato da CRISPR / Cas9. I FESC sono stati coltivati con MEF, che contengono DNA di tipo selvatico H19-DMR e IG-DMR. Pertanto, le coppie primer di H19-DMR-P2/P3 e IG-DMR-P2/P3, che amplificano frammenti di DNA di tipo selvatico, non sono informative. Tuttavia, queste coppie di primer sono incluse come controlli e dovrebbero dare una banda in tutte le reazioni. - Analizzare i frammenti pcr per elettroforesi del gel di agarosio. Fare riferimento al protocollo pubblicato sulla procedura dettagliata dell'elettroforesi16.
- Identificare le linee cellulari con eliminazioni di H19-DMR e IG-DMR. Un'immagine rappresentativa dell'elettroforesi è mostrata nella figura 2B.
NOTA: Nel nostro caso, sono state identificate otto linee cellulari con cancellazioni di H19-DMR e IG-DMR tra 135 linee phaESC sotto-clonate.
5. Purificazione delle cellule aploidi delle linee phaESC sub-clonate
- Quando le colture phaESC sotto-clonate nei contenitori T25 del passaggio 3.22 diventano abbastanza dense da essere passate, aspirate il mezzo e aggiungete 1,5 mL di tripside. Incubare il pallone a 37 °C per 5 minuti. Quindi, aggiungere più volte 4,5 mL di tampone di lavaggio e pipetta per ottenere una sospensione a cella singola.
- Trasferire ogni sospensione cellulare in un tubo da 15 ml e centrifugare il tubo a 160 x g per 5 minuti. Rimuovere il supernatante e rimescolare il pellet cellulare in 400 μL di tampone di manutenzione haESC integrato con 15 μg/mL Hoechst 33342.
- Incubare le sospensioni cellulari a 37 °C per 15 min. Dopo l'incubazione, trasferire le sospensioni cellulari in un tubo da 5 ml attraverso un tappo del filtro cellulare. Risciacquare il tappo del filtro cellulare con altri 400 μL di tampone di manutenzione haESC e raccogliere le celle rimanenti nello stesso tubo da 5 ml. Mantenere il tubo a 4 °C fino a quando non è pronto per l'ordinamento.
- Impostare un citometro di flusso con un ugello da 100 μm secondo le istruzioni del produttore.
NOTA: Hoechst 33342 può essere rilevato per eccitazione a 405 nm. Qui, un laser UV a 355 nm viene utilizzato per il rilevamento di Hoechst 33342. - Impostare la sospensione cellulare (dal passaggio 5.3) e un nuovo tubo da 15 ml contenente 2 ml di tampone di manutenzione haESC per raccogliere le cellule ordinate nel citometro di flusso.
- Avviare l'analisi e impostare il cancello di smistamento per raccogliere le cellule aploidi nella fase G1/S. Fare riferimento all'istogramma della figura 2A per identificare la popolazione della fase G1/S.
NOTA: Alcune linee phaESC sotto-clonate potrebbero non contenere cellule aploidi a causa della diploidizzazione completa o della placcatura errata delle cellule diploidi nel passaggio 2. Se le cellule aploidi non si osservano nella fase G1/S, procedere a un altro campione senza smistamento. Nel nostro caso, 5 linee cellulari contenevano cellule aploidi e 3 linee cellulari contenevano solo cellule diploidi su 8 linee phaESC sotto-clonate. - Dopo lo smistamento delle celle, aggiungere 5 ml di tampone di lavaggio lungo la parete del tubo di raccolta da 15 ml. Centrifugare il tubo a 160 x g per 5 min. Rimuovere il supernatante.
- Selezionare una lastra di dimensioni adeguate per la ristrutturazione a seconda del numero di celle ordinate. Usa un singolo pozzo di una piastra da 96 po' , una piastra da 24 po' e una piastra da 12 po' per coltura rispettivamente 1.000-40.000 celle, 40.000-200.000 cellule e 200.000-400.000 cellule. Rimescolare il pellet cellulare rispettivamente in 120 μL, 600 μL e 1,2 mL di mezzo haESC.
NOTA: Placcare le cellule ad alta densità perché una bassa confluenza può causare la morte cellulare delle cellule dopo lo smistamento. Da questo momento in poi, i feSC vengono coltivati su pozzi rivestiti di gelatina senza MEF per facilitare la genotipizzazione nella fase 6 e la successiva applicazione per l'iniezione intracitoplasmatica dal passaggio 9. - Dopo aver trasferito la sospensione cellulare in un pozzo rivestito di gelatina delle dimensioni appropriate, incubare la piastra a 37 °C in un'atmosfera di CO2 al 5%.
- Continuare ad espandere le colture phaESC ripetendo i passaggi da 3.18 a 3.21 con l'aumento delle dimensioni delle lastre e l'aumento dei volumi di tripina, tampone di lavaggio e mezzo haESC. Le cellule sono coltivate su pozzi rivestiti di gelatina senza MEF.
- Per ogni linea phaESC sotto-clonata, preparare una coltura in un pozzo di una piastra da 24 po 'e un pozzo di una piastra a 6 poggia-poggia per i passaggi 6 e 9, rispettivamente.
NOTA: Alcune cellule di ciascuna linea phaESC sub-clonata devono essere congelate in 300 μL di mezzo di congelamento come criostock in un serbatoio di stoccaggio dell'azoto liquido prima di procedere al passaggio 9.
6. Seconda genotipizzazione di linee phaESC sottodolonate senza MEF
NOTA: Viene eseguito un secondo ciclo di genotipizzazione per confermare che le linee phaESC sotto-clonate possiedono eliminazioni sia dei DLF H19- che IG- e che gli alleli wildtype sono assenti dopo la rimozione dei MEF.
- Confermare al microscopio che le colture nei pozzi delle piastre a 24 pozzi del passaggio 5.11 sono libere da MEF.
NOTA: Se si osservano MEF, è necessario continuare a far passare le colture fino a quando i MEF non sono scomparsi per evitare di contaminare la PCR con DNA di tipo selvatico dai MEF. - Aspirare il mezzo da colture confluenti e aggiungere 400 μL di tampone di lysis per pozzo della piastra da 24 porri. Dopo aver pipettato più volte, trasferire la sospensione cellulare su un tubo da 1,5 ml.
- Incubare il tubo da 1,5 ml a 55 °C per 3 ore con miscelazione.
- Dopo l'incubazione, aggiungere 400 μL di isopropanolo al tubo da 1,5 ml e mescolare delicatamente fino a quando un precipitato di DNA diventa visibile.
- Centrifugare il tubo a ≥ 10.000 x g per 5 minuti e rimuovere il supernatante. Lavare il pellet di DNA con 200 μL di 70% di etanolo.
- Centrifugare il tubo a ≥ 10.000 x g per 5 minuti e rimuovere il supernatante.
- Asciugare il tubo nell'aria per 10 minuti e quindi rimospendre il DNA in 50 μL di acqua.
- Eseguire la PCR genotipizzante dopo il passaggio 4.7 e l'elettroforesi del gel nel passaggio 4.8 per identificare le linee cellulari, che possiedono eliminazioni sia dei DR H19- che IG- e sono libere da alleli wildtype.
NOTA: Un'immagine di una tipica seconda analisi di genotipizzazione è mostrata nella figura 2B come riferimento. Nel nostro caso, tutte e 5 le linee cellulari selezionate dopo la purificazione delle cellule aploidi (fase 5) erano prive di alleli di tipo selvatico e possedevano solo gli alleli di eliminazione dei DR H19- e IG- . - Utilizzare le linee phaESC sottodolonate selezionate dopo questo secondo genotipizzazione come linee DKO-phaESC.
7. Superovulazione dei topi
- Per la produzione di ovociti MII, avviare la superovulazione mediante iniezione intraperitoneale di 5 UI di soluzione di gonadotropina del siero di mare incinta (PMSG) in ogni topo femmina B6D2F1 (4-5 settimane) 63-65 ore prima della raccolta degli ovociti.
NOTA: Lo sforzo del mouse B6D2F1 è raccomandato per questo protocollo perché gli ovociti B6D2F1 tollerano bene l'iniezione intracitoplasmatica e mostrano un elevato potenziale di sviluppo dopo la procedura17. - Quarantotto ore dopo l'iniezione di PMSG, iniettare per via intraperitoneale 5 UI di soluzione di gonadotropina corionica umana in ogni topo.
8. Raccolta degli ovocite
- Preparare una piastra di 4 porvili contenente 700 μL di mezzo ialuronidasi in un pozzo e 700 μL di mezzo M2 nei restanti 3 pozzi. Inoltre, preparare un piatto di 6 cm con 7 mL di mezzo M2 e un piatto centrale-bene con 900 μL di mezzo M16. Preripidi il piatto e i piatti a 37 °C in un'atmosfera del 5% di CO2.
- Il giorno dell'iniezione intracitoplasmatica, eutanasiare le femmine superovulate (dal passo 7.2) per lussazione cervicale oinalazione di CO 2 intorno alle 8 del mattino. Sezionare gli ovidotti usando pinzette e forbici. Posizionare gli ovidotti nel piatto da 6 cm con mezzo M2.
- Rilasciare i complessi cumulo-ovocite (COC) strappando l'ampolla degli ovidotti con un ago da 30 G. Trasferire i COC in un mezzo di ialuronidasi pre-riscaldato e mantenere a 37 °C in un'atmosfera di CO2 al 5%.
- Dopo 2-3 minuti, raccogli gli ovociti senza cumulo con una pipetta per la bocca e lava gli ovociti 3 volte trasferendoli su mezzo M2 fresco negli altri 3 pozzi della piastra a 4 pozzetti.
- Raccogliere gli ovociti metafase II (MII), che possiedono i primi corpi polari, in un piatto centrale con mezzo M16 e mantenere la piastra a 37 °C in un'atmosfera di CO2 del 5% fino all'uso per l'iniezione intracitoplasmatica nella fase 12.
9. Trattamento e raccolta di DKO-fasi
- Preparare una coltura DKO-phaESC in un pozzo di una piastra a 6 porvili senza MEF al 60-80% di confluenza il giorno prima dell'iniezione intracitoplasmatica (dal passaggio 5.11).
- Per indurre l'arresto del ciclo cellulare in fase M, aspirare completamente il mezzo e aggiungere 2 mL di mezzo eSE contenente 0,05 mg/mL di demecolicina.
- Dopo 8 ore di trattamento con demecolicina, aspirare il mezzo e aggiungere 800 μL di tripside.
- Incubare la piastra a 37 °C in un'atmosfera di CO2 del 5% per 5 minuti, quindi aggiungere 2 mL di tampone di lavaggio per temprare la triparina e pipettare più volte per ottenere una sospensione a cella singola.
- Trasferire la sospensione cellulare in un tubo da 15 ml. Centrifugare il tubo a 160 x g per 5 minuti e rimuovere il supernatante.
- Resuspend il pellet cellulare in 400 μL di tampone di manutenzione haESC contenente 15 μg/mL Hoechst 33342.
- Incubare la sospensione cellulare a 37 °C in un'atmosfera di CO2 del 5% per 15 minuti. Dopo l'incubazione, trasferire la sospensione cellulare in un tubo da 5 ml attraverso un tappo del filtro cellulare e mantenere il tubo a 4 °C fino allo smistamento cellulare nel passaggio 10.
10. Purificazione dei DKO-phaESC arrestati in fase di fase M
- Impostare un citometro di flusso con un ugello da 100 μm secondo le istruzioni del produttore.
NOTA: Hoechst 33342 può essere rilevato per eccitazione a 405 nm. Qui, un laser UV a 355 nm viene utilizzato per il rilevamento di Hoechst 33342. - Impostare i DKO-phaESC arrestati in fase di fase M dal passaggio 9.7 e avviare l'analisi. Selezionare un cancello di smistamento adatto per la raccolta delle cellule aploidi della fase M (2n) dal campione trattato con demecolicina.
NOTA: Dopo il trattamento con demecolicina, sono previste 2 popolazioni cellulari, corrispondenti a cellule aploidi e diploidi arrestate in fase M, come mostrato nella figura 3B. L'arresto del ciclo cellulare dopo il trattamento con demecolicina è completo, quindi non si osserva alcun picco di DNA aploide 1n. Questo è importante in quanto le cellule aploidi della fase M e le cellule G1 diploidi possiedono lo stesso contenuto di DNA (2n) e produrrebbero picchi sovrapposti. - Impostare un tubo da 15 ml contenente 2 ml di tampone di manutenzione haESC per raccogliere le celle ordinate nel citometro di flusso. Avviare l'ordinamento delle celle.
- Dopo lo smistamento delle celle, aggiungere 5 ml di tampone di lavaggio lungo la parete del tubo di raccolta. Centrifugare il tubo a 160 x g per 5 minuti e rimuovere il supernatante.
- Rimescolare le cellule in un volume appropriato di tampone di manutenzione haESC per ottenere una concentrazione finale di 5 x 105 celle/mL.
- Trasferire la sospensione cellulare in un tubo da 1,5 ml. Tenere il tubo sul ghiaccio fino a quando non è pronto per l'iniezione intracitoplasmatica nella fase 12.
11. Preparazione di pipette di tenuta e microiniezione
NOTA: Per eseguire l'iniezione intracitoplasmatica (fase 12), sono necessarie diverse pipette di tenuta e microiniezione(figura 4A). Queste pipette possono essere acquistate su richiesta su misura da un fornitore commerciale o realizzate con adeguati capillari in vetro utilizzando un estrattore di micropipette e una microforgia.
- Tirare i capillari di vetro borosilicato su un estrattore di micropipette. Per estrarre i capillari di vetro borosilicato senza filamento (0,78 x 1,00 x 80 mm) vengono forniti come riferimento i seguenti parametri per un estrattore orizzontale fiammeggiante(Tabelladei materiali), ma differiranno per altri strumenti e tipi capillari di vetro: Calore 510 (Ramp test 480), Pull 0, Velocità 150, Tempo 175 e Pressione 200 per il possesso di pipette; Calore 510 (rampa test 480), Pull 90, Velocità 140, Tempo 125 e Pressione 200 per pipette di microiniezione.
NOTA: I parametri ottimali devono essere definiti individualmente perché diversi fattori tra cui l'umidità, il modello di un estrattore di micropipette e il lotto dei capillari di vetro possono influenzare la forma delle pipette di iniezione. Una forma allungata con una conica graduale dovrebbe essere mirata. - Preparazione delle pipette di tenuta
- Impostare un capillare tirato preparato nel passaggio 11.1 su una microforgia. Posizionare il capillare sopra la perla di vetro sul filamento e abbassare il capillare per effettuare un contatto con la perla di vetro riscaldando il filamento.
- Rompere il capillare spegnendo il riscaldamento e staccandosi dalla perla di vetro in modo che il suo diametro esterno sia di 60-100 μm.
- Posizionare la punta capillare orizzontalmente per affrontare il tallone di vetro sul filamento.
- Riscaldare il filamento per consentire al diametro interno della punta capillare di sciogliersi ad un diametro di 10-20 μm.
- Spostare il capillare in modo che il tallone di vetro si posizioni in un punto ~ 1 mm dalla punta capillare senza contatto. Riscaldare il filamento per consentire al capillare di piegarsi con un angolo di 20°. Smontare il capillare, direttamente una pipetta di tenuta, dalla microforgia.
NOTA: Per misurare le dimensioni del capillare, nel microforgia è preferibilmente installato un reticolo oculare.
- Preparazione di pipette di microiniezione
- Impostare un capillare tirato preparato nel passaggio 11.1 su una microforgia. Posizionare il capillare sopra la perla di vetro sul filamento e abbassare il capillare per effettuare un contatto con la perla di vetro riscaldando il filamento.
- Rompere il capillare spegnendo il riscaldamento e staccandosi dal tallone di vetro in una posizione in cui il suo diametro esterno è di 6 μm.
- Spostare il capillare in modo che il tallone di vetro si posizioni in un punto ~ 1 mm dalla punta capillare senza contatto. Riscaldare il filamento per consentire al capillare di piegarsi verso l'alto con un angolo di 20°. Smontare la pipetta di microiniezione dalla microforgia e conservarsi in una scatola sicura per un uso successivo.
NOTA: Le pipette di microiniezione sono preparate con le seguenti specifiche: diametro esterno, 6 μm; diametro interno, 4,5-5 μm; angolo di piegatura, 20°. Definire il design ottimale della pipetta di microiniezione è importante per il successo dell'iniezione intracitoplasmatica. Un diametro interno troppo grande può impedire la rupturing della membrana plasmatica dei DKO-PHESC donatori (vedi sezione discussione). Se il diametro interno è troppo stretto, può impedire una pipettazione liscia dei DKO-PHESC donatori. Un angolo di piegatura < 30° è preferibile in quanto un angolo di piegatura elevato impedisce l'effetto degli impulsi piezo.
12. Iniezione intracitoplasmatica di DKO-fasi
- Prima dell'iniezione intracitoplasmatica (dal passaggio 12.2), preparare una soluzione di polivinilpirrolidone (PVP) aggiungendo 5 ml di mezzo M2 in un tubo da 50 ml contenente 0,6 g di PVP e agitando il tubo su un rocker a 4 °C per 2 giorni. Dopo che il PVP si è completamente sciolto, la soluzione viene filtrata sterilemente e conservata a 4 °C.
- Il giorno dell'iniezione intracitoplasmatica, preparare un piatto centrale con 900 μL di mezzo KSOM e pre-riscaldare il piatto a 37 °C in un'atmosfera di CO2 al 5%.
- Preparare una piastra di micromanipolazione allineando gocce di 5 μL di soluzione PVP e 20 μL di mezzo M2 su un coperchio di un piatto di 10 cm che viene posizionato capovolto. Coprire le gocce con olio minerale e posizionare il piatto sul palco del microscopio a iniezione.
NOTA: La disposizione raccomandata della parabola di micromanipolazione è illustrata nella figura 4B. - Installare una pipetta di tenuta sul micromanipolatore. Riempire la pipetta di microiniezione con l'olio di fluorocarbonio utilizzando una punta del microloader e montarla sull'attuatore piezo.
- Immergere la pipetta di microiniezione in una goccia con soluzione PVP e pipetta su e giù più volte per rivestire il vetro con PVP e renderlo meno appiccicoso. Caricare un piccolo volume di soluzione PVP nella pipetta di microiniezione e spostare la pipetta in una goccia con mezzo M2.
- Immergere la pipetta di tenuta nel mezzo M2 e concentrarsi sulla pipetta nella parte inferiore della goccia.
- Trasferire circa 2 μL di sospensione DKO-phaESC dal passo 10.6 nella caduta media M2.
- Trasferire 10 ovociti MII dal passo 8.5 nella stessa goccia media M2 utilizzando una pipetta per la bocca.
- Per l'iniezione, ruotare un ovocita nella caduta media M2 in modo che lo spazio perivitellino sia fronte alla pipetta di microiniezione e la piastra MII non si trovi nel percorso della pipetta di microiniezione (Figura 4A). Tenere l'ovocita applicando una pressione negativa attraverso la pipetta di tenuta.
NOTA: Una piastra MII è visivamente identificata come una sporgenza di ooplasma che viene definita una "gobba" e spesso situata vicino al primo corpo polare. La lastra MII contiene il mandrino meiotico con cromosomi attaccati. Il contatto della pipetta di microiniezione e della piastra MII deve essere evitato in quanto i danni meccanici del mandrino e dei cromosomi possono interrompere lo sviluppo dell'embrione. - Caricare un DKO-phaESC nella punta della pipetta di microiniezione applicando una leggera pressione negativa. Rottura della membrana plasmatica di un DKO-phaESC mediante pipettazione per evitare l'iniezione di un DKO-phaESC intatto (Figura 3C; vedi discussione).
NOTA: Nel caso in cui la membrana plasmatica di un DKO-phaESC non si rompono con la pipettazione, scartare il DKO-phaESC e caricare un altro DKO-phaESC. - Mettere la pipetta di microiniezione a contatto con la zona pellucida dell'ovocita e applicare una piccola quantità di pressione negativa all'interno della pipetta di microiniezione.
- Applicare impulsi piezoici (intensità, 20; frequenza, 4) per sfondare la zona spingendo la punta della pipetta di microiniezione verso lo spazio perivitellino. Verificare che la piastra MII, contenente un mandrino e cromosomi, non si trovi nel percorso della pipetta di microiniezione.
NOTA: Regolare empiricamente l'impostazione agli impulsi piezoici più bassi per la perforazione attraverso la zona per ridurre al minimo la possibilità di danni all'oolemma. - Scartare il frammento della zona pellucida dalla pipetta di microiniezione e posizionare il DKO-phaESC sul bordo della pipetta.
- Penetrare l'ovocita con la pipetta di microiniezione in modo che l'oolemma raggiunga il lato opposto.
NOTA: Non toccare la piastra MII per evitare danni al mandrino e ai cromosomi. - Applicare un impulso piezo (intensità, 6; frequenza, 1) per perforare l'oolemma. Assicurarsi che l'oolemma si rilassi lungo l'albero della pipetta di microiniezione.
NOTA: Definire empiricamente l'impostazione più bassa dell'impulso piezo per rompere l'oolemma per ridurre al minimo il danno all'ovocita. - Iniettare il DKO-phaESC con un volume minimo di mezzo nell'ooplasma e ritirare la pipetta di microiniezione senza intoppi dall'ovocita.
- Rilasciare l'ovocito iniettato dalla pipetta di tenuta e posizionarlo sul lato della microdrop per una successiva raccolta.
- Ripetere la procedura di iniezione dai passaggi da 12.9 a 12.17 per gli altri ovociti MII nella caduta media M2.
NOTA: Evitare di tenere gli ovociti fuori dall'incubatrice per più di 20 minuti. Nella nostra esperienza, un lotto di 10 ovociti può essere manipolato comodamente entro 15 minuti dopo un allenamento appropriato. - Trasferire il lotto di ovociti iniettati dalla goccia media M2 a un piatto centrale-pozzo preri riscaldato con mezzo KSOM.
- Conservare il piatto per 1 h a 37 °C in un'atmosfera di CO2 al 5%.
- Ripetere la manipolazione degli ovociti dai passaggi 12.5 e 12.20 con gruppi aggiuntivi di ovociti MII per ottenere abbastanza ovociti iniettati.
13. Attivazione di embrioni semi-clonati costruiti
- Preparare due piatti centro-bene con 900 μL ciascuno di mezzo KSOM e mezzo di attivazione. Preparare una piastra a 4 porvili con 700 μL di mezzo KSOM in ogni pozzo. Preripidi i piatti e il piatto a 37 °C in un'atmosfera di CO2 al 5%.
- Dopo 1 h nel mezzo KSOM, trasferire gli ovociti iniettati dal passo 12.21 nel piatto centro-pozzo prerifoto con mezzo di attivazione.
- Conservare il piatto per 6 ore a 37 °C in un'atmosfera di CO2 al 5% per l'attivazione.
- Dopo l'attivazione, osservare alcuni embrioni semi-clonati formare tre corpi polari, che sono il primo e il secondo corpo polare dell'ovocita, e lo pseudo corpo polare del DKO-phaESC (Figura 3E).
- Lavare gli embrioni 3 volte trasferendoli in nuovi pozzi con mezzo KSOM in una piastra a 4 poggi.
- Trasferire gli embrioni nel piatto centrale con mezzo KSOM e mantenere il piatto a 37 °C in un'atmosfera di CO2 al 5% per un ulteriore sviluppo.
14. Sviluppo di embrioni semi-clonati costruiti
- Dopo 1 giorno di coltura nel mezzo KSOM dal passo 13.6, diversi embrioni semi-clonati raggiungono lo stadio a 2 cellule (Figura 5A).
- Per un ulteriore sviluppo di embrioni preimpianto in vitro, continuare a coltivare gli embrioni semi-clonati nel mezzo KSOM a 37 °C in un'atmosfera di CO2 del 5%. Trasferire gli embrioni semi-clonati su un mezzo KSOM fresco al giorno 2. Al giorno 4, diversi embrioni raggiungeranno lo stadio di blastocista (Figura 5A).
- Per la derivazione di topi semi-clonati, trasferire embrioni a 2 cellule dal passaggio 14.1 negli ovidotti delle femmine riceventi pseudo-gravide. Identificare le femmine pseudo-gravide accoppiandole ai maschi vasectomizzati un giorno prima del trasferimento dell'embrione e selezionarle sulla base della presenza di una spina chiaramente visibile al mattino del giorno per il trasferimento dell'embrione (0,5 giorni dopo il coitum (dpc)). Circa 19,5 dpc, i cuccioli a tempo pieno vengono consegnati naturalmente dalle femmine riceventi (Figura 5B).
Risultati
Lo scopo di questo protocollo è quello di applicare i SEBC come sostituti dello sperma per ottenere embrioni e topi semi-clonati. A tal fine, è stata generata e utilizzata una linea DKO-phaESC che trasportava il transgene CAG-EGFP per l'iniezione intracitoplasmatica negli ovociti MII. Per ottenere una linea phaESC adatta con una configurazione di impronta paterna, abbiamo eseguito ingegneria genetica utilizzando nucleasi Cas9. Una linea ESC aploide contiene cellule aploidi e diploidi che sorgono a causa di una tendenza intrinseca degli ESC aploidi per la diploidizzazione10. Un set cromosomico aploide è un prerequisito per una sostituzione riuscita del genoma dello sperma. L'analisi del contenuto di DNA mediante citometria del flusso mostra la distribuzione di cellule aploidi e diploidi nelle fasi G0/G1, S-e G2/M (Figura 2A).
Per stabilire le linee DKO-phaESC, le linee phaESC di tipo selvatico sono state trasfette con costrutti di transposon piggyBac per l'espressione transgenica stabile di EGFP e con plasmidi CRISPR / Cas9 per ottenere eliminazioni degli H19- e IG-DMR. Per escludere gli ESC diploidi e isolare solo gli ESC aploidi che esprimono EGFP, è stato definito un ordinamento specifico (Figura 2A). Le singole cellule aploidi EGFP-positive sono state quindi placcate in singoli pozzi di piastre da 96 pozzi per ottenere sub-cloni. Gli alimentatori MEF sono stati utilizzati per aumentare l'efficienza di placcatura e la sopravvivenza dei FESC trasfetati. Dopo l'espansione delle culture, un primo ciclo di genotipizzazione è stato effettuato da PCR per identificare i sotto-cloni che trasportano eliminazioni di entrambi i D DMR. Dopo che gli alimentatori MEF sono stati rimossi dalle colture, è stato eseguito un secondo genotipizzazione per confermare l'assenza di alleli di tipo selvatico all'H19- e agli IG-DMR (Figura 2B). Su un totale di 135 sotto-cloni, abbiamo ottenuto 5 linee Aploidi DKO-phESC che trasportavano gli alleli cancellati ed erano privi di alleli wildtype sia dell'H19-DMR che dell'IG-DMR.
Un transgene CAG-EGFP è stato introdotto nei DKO-feSC per studiare il loro contributo agli embrioni semi-clonati mediante visualizzazione della fluorescenza verde al microscopio (Figura 3A). Per l'iniezione intracitoplasmatica, i DKO-fasi sono stati trattati con demecolicina per arrestarli in fase M. Pertanto, il ciclo cellulare dei DKO-faESC è stato sincronizzato con quello degli ovociti MII. L'analisi della citometria del flusso ha mostrato 2 popolazioni corrispondenti all'aploide arrestato in fase G2/M (2n) e alle cellule diploidi (4n) dopo il trattamento con demecolcina(figura 3B). L'assenza del picco aploide di 1n indicava che l'arresto del ciclo cellulare era in gran parte completo. Gli ESC aploidi di fase M sono stati quindi ordinati e iniettati in ovociti. Per questo, un singolo DKO-phaESC è stato caricato nella pipetta di microiniezione e iniettato nel citoplasma di un ovocita MII (Figura 3C). La membrana plasmatica del DKO-phaESC è stata interrotta tubazione nella punta della pipetta di microiniezione.
Dopo l'iniezione, l'espressione EGFP è stata raramente rilevata negli embrioni semi-clonati costruiti poiché il citoplasma dei DKO-feSC si era disperso nel grande citoplasma dell'ovocito (Figura 3D). In rari casi, una macchia rotonda di intensa espressione EGFP potrebbe essere osservata all'interno dell'ooplasma. Questa osservazione è stata probabilmente causata dall'iniezione involontaria di DKO-faSC intatti. La mancata rottura della membrana cellulare DKO-phaESC probabilmente non è compatibile con l'ulteriore sviluppo dell'embrione e dovrebbe essere evitata. Un'ora dopo l'iniezione, gli embrioni sono stati attivati mediante trattamento con cloruro distronzio 18. Sei ore dopo l'inizio dell'attivazione, al microscopio sono stati osservati fino a 3 corpi polari(figura 3E). Questi corpi polari corrispondono al primo e al secondo corpo polare dell'ovocita e a uno pseudo corpo polare del DKO-phaESC7. Inoltre, due pronuclei sono stati osservati sotto un microscopio a contrasto di interferenza differenziale, che assomigliava allo stadio pronucleare degli zigoti dopo la normale fecondazione con lo sperma.
Per dimostrare la competenza allo sviluppo, gli embrioni semi-clonati sono stati coltivati allo stadio della blastocista(figura 5A). Inoltre, un topo a tempo pieno è stato ottenuto dopo aver trasferito embrioni semi-clonati a 2 cellule negli ovidotti di una femmina ricevente (Figura 5B). Come previsto, il topo derivato da un embrione semi-clonato era una femmina in quanto né gli ovociti né i faESC derivati dagli ovociti trasportano un cromosoma Y. Il topo semi-clonato era evidentemente normale e produceva prole sana se accoppiato con un maschio webster svizzero di tipo selvatico. Fino ad ora, il topo semi-clonato è vivo da oltre 600 giorni senza apparenti problemi di salute.
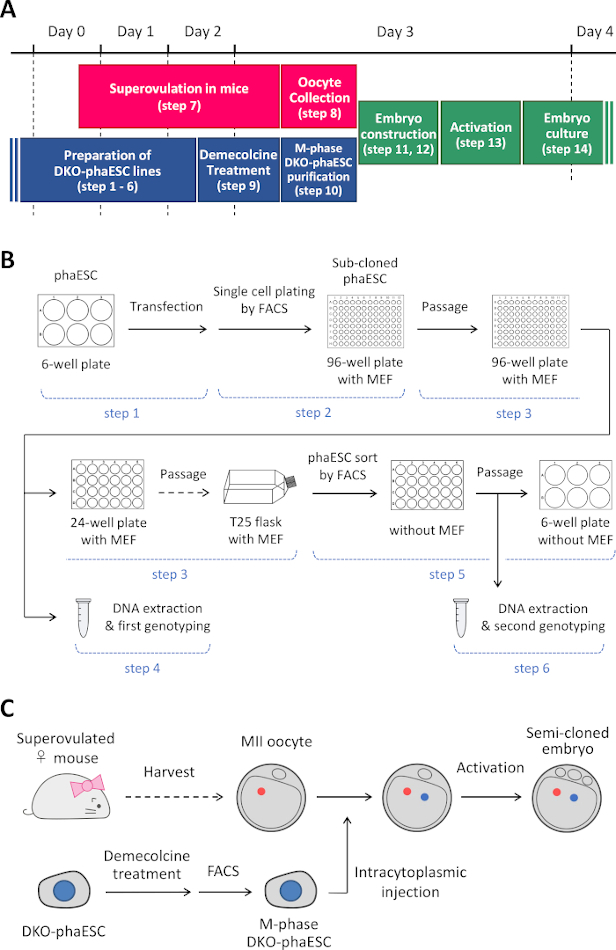
Figura 1: Panoramica dell'applicazione dei DKO-fasi come sostituzione degli spermatozoi. (A) Un lasso di tempo delle procedure del protocollo. (B) Lo schema mostra i passaggi per stabilire le linee DKO-phaESC (passaggi da 1 a 6). (C) Lo schema mostra le misure per costruire embrioni semi-clonati mediante iniezione intracitoplasmatica di un DKO-phaESC in un ovocita MII (fasi 7-14). Abbreviazioni: DKO = doppio knockout; phaESC = cellula staminale embrionale aploide partogenetica; FACS = smistamento cellulare attivato dalla fluorescenza; MEF = fibroblasto embrionale del topo. Clicca qui per visualizzare una versione più grande di questa figura.
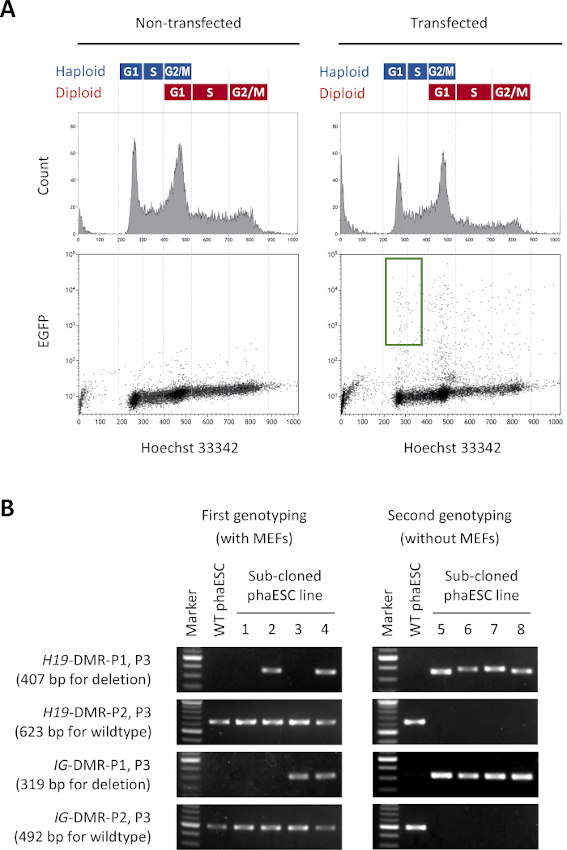
Figura 2: Definizione di linee DKO-phaESC mediante eliminazioni mediate da CRISPR/Cas9 di DLF H19e IG. (A) Analisi citometrica del flusso dei feSC dopo la trasfezione con plasmidi piggyBac per l'espressione stabile di EGFP e con 4 plasmidi CRISPR/Cas9. Le fasi non trasfette vengono visualizzate come controllo. Il profilo del DNA (in alto) mostra la distribuzione del ciclo cellulare delle cellule aploidi e diploidi. Le cellule aploidi della fase G1/S che esprimono EGFP sono indicate dal cancello verde (in basso, a destra). (B) Genotipizzazione dei sotto cloni phaESC coltivati su MEF (primo genotipizzazione) e dopo la rimozione dei MEF (secondo genotipizzazione). Le righe phaESC sotto-clonate 1, 2, 3 e 4 rappresentano cellule di tipo jolly, celle con eliminazione H19-DMR, con eliminazione IG-DMR e con eliminazioni combinate H19- e IG-DMR, rispettivamente. Le linee DKO-phaESC 5-8 possiedono sia eliminazioni H19-DMR che IG-DMR e sono libere da alleli wildtype. Abbreviazioni: DKO = doppio knockout; phaESC = cellula staminale embrionale aploide partogenetica; DMR = regione metilata differenziale; MEF = fibroblasto embrionale del topo; EGFP = proteina fluorescente verde migliorata; WT = tipo jolly. Clicca qui per visualizzare una versione più grande di questa figura.
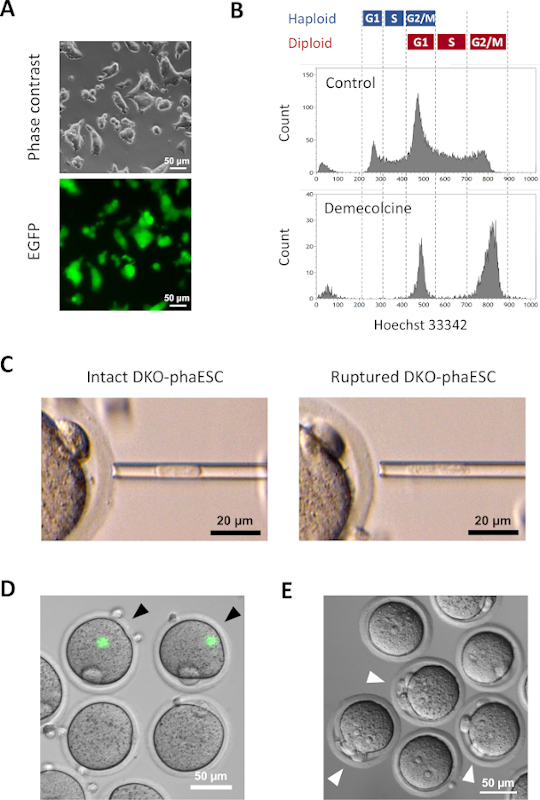
Figura 3: Iniezione di DKO-faESC arrestati mitoticamente negli ovociti MII. (A) Morfologia di una coltura DKO-phaESC che trasporta un transgene CAG-EGFP e cancellazioni dei DLF H19 e IG;; barra di scala = 50 μm. (B) L'analisi rappresentativa della citometria del flusso dei DKO-faSES dopo l'arresto con demecolicina per 8 h. DKO-fasi senza trattamento con demecolcina è indicata come controllo. (C) Morfologia dei DKO-faESC nella pipetta di microiniezione. Viene mostrato un singolo DKO-phaESC intatto prima (a sinistra) e dopo (a destra) rupturing della membrana plasma mediante pipettazione; barra di scala = 20 μm. (D) Embrioni costruiti a 1 h dopo l'iniezione di DKO-fasci negli ovociti MII. Viene mostrata un'immagine unita di fluorescenza EGFP e campo luminoso. Le punte di freccia nere indicano punti rotondi di intensa espressione EGFP dopo l'iniezione di DKO-feCS intatti, che dovrebbero essere evitati; barra di scala = 50 μm. (E) Viene mostrata un'immagine di contrasto di interferenza differenziale di embrioni semi-clonati 6 ore dopo l'inizio dell'attivazione con cloruro di stronzio. Le punte di freccia bianche indicano embrioni con 3 corpi polari tra cui uno pseudo corpo polare dal DKO-phaESC iniettato; barra di scala = 50 μm. Abbreviazioni: DKO = doppio knockout; phaESC = cellula staminale embrionale aploide partogenetica; EGFP = proteina fluorescente verde migliorata. Clicca qui per visualizzare una versione più grande di questa figura.
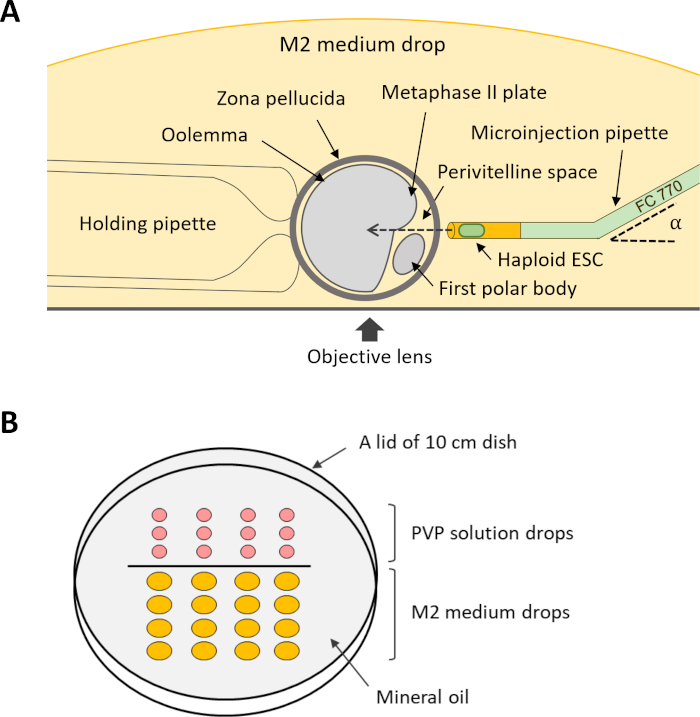
Figura 4: Schema dell'installazione per l'iniezione intracitoplasmatica di DKO-fasi in ovociti. (A) Viene mostrata la disposizione di una pipetta di iniezione, di una pipetta di tenuta e di un ovocita nella camera di iniezione. α, angolo di piegatura della pipetta di microiniezione. (B) Disposizione delle gocce nella parabola di micromanipolazione per iniezione intracitoplasmatica. Abbreviazioni: DKO = doppio knockout; phaESC = cellula staminale embrionale aploide partogenetica. Clicca qui per visualizzare una versione più grande di questa figura.
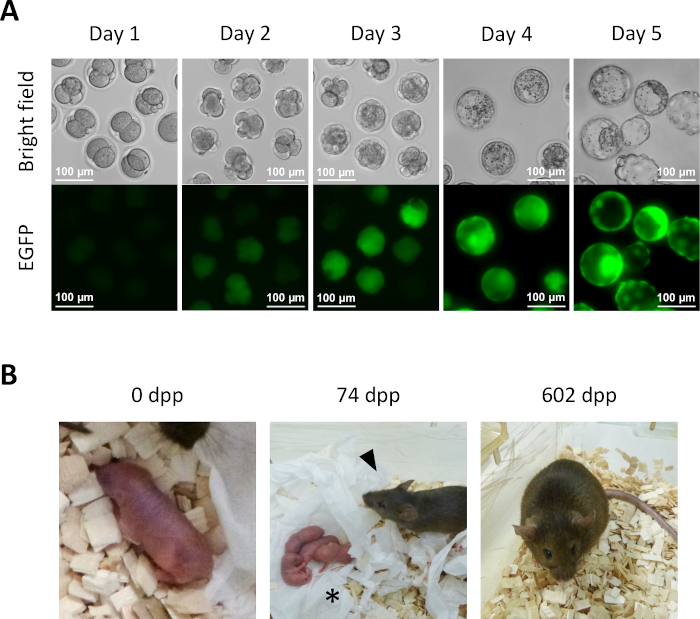
Figura 5: Sviluppo di embrioni semi-clonati. (A) Sviluppo preimpianto di embrioni semi-clonati in vitro. L'espressione EGFP è inizialmente osservata negli embrioni a quattro cellule al secondo giorno dopo l'iniezione intracitoplasmatica. Al giorno 4, l'intensa espressione EGFP può essere osservata nelle blastocsti; barra di scala = 100 μm. ( B )Untopo semi-clonato ottenuto dopo il trasferimento di embrioni a 2 cellule a una femmina ricevente. A 74 dpp, il topo semi-clonato (punta di freccia) ha consegnato i suoi primi cuccioli (asterisco) per nascita naturale dopo l'accoppiamento con un maschio webster svizzero di tipo selvatico. Gli embrioni semi-clonati e il topo semi-clonato mostrato in questa figura sono identici a quelli riportati in Aizawa etal. Abbreviazioni: EGFP = proteina fluorescente verde migliorata; .dpp, giorni dopo il parto. Clicca qui per visualizzare una versione più grande di questa figura.
| Soluzione di gelatina | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| Acqua | - | 500 mL | - |
| gelatina | - | 1 g | 0.2% |
| Mezzo aploide a cellule staminali embrionali (HaESC) | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| NDiff 227 | - | 10 mL | - |
| CHIR 99021 | 10 mM | 3 μL | 3 μM |
| PD 0325901 | 10 mM | 1 μL | 1 μM |
| Lif | 1 x 106 UI/mL | 10 μL | 1.000 UI/mL |
| Penicillina-Streptomicina | 100x | 100 μL | 1x |
| Buffer di manutenzione HaESC | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| Mezzo HaESC | - | 1 mL | - |
| Soluzione HEPES | 1 M | 20 μL | 20 mM |
| Tampone di lavaggio | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| DMEM / F-12 | - | 100 mL | - |
| Frazione BSA | 7.5% | 7,1 mL | 0.5% |
| Mef medium | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| DMEM | - | 500 mL | - |
| Fbs | - | 56 mL | 10% |
| β-mercaptoetanolo | 14,3 mol/L | 3,9 μL | 100 μM |
| Penicillina-Streptomicina | 100x | 5,6 mL | 1x |
| Buffer di Lysis | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| Acqua | - | 8,25 mL | - |
| Tris-HCl (pH 8.5) | 1 M | 1 mL | 100 mM |
| Edta | 0,5 M | 100 μL | 5 mM |
| Soluzione SDS | 10% | 200 μL | 0.2% |
| NaCl | 5 M | 400 μL | 200 mM |
| Proteinasi K | 20 mg/mL | 50 μL | 100 μg/mL |
| Mezzo di attivazione | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| KSOM | - | 1 mL | - |
| Cloruro di stronzio | 1 M | 5 μL | 5 mM |
| EGTA | 0,5 M, pH 8,0 | 4 μL | 2 mM |
| Soluzione PVP | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| Mezzo M2 | - | 5 mL | - |
| Pvp | - | 0,6 g | 12% |
| Soluzione PMSG | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| Pbs | - | 450 μL | - |
| PMSG | 500 UI/mL | 50 μL | 50 UI/mL |
| soluzione hCG | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| Pbs | - | 450 μL | - |
| HCG | 500 UI/mL | 50 μL | 50 UI/mL |
| Mezzo di ialuronidasi | |||
| componente | Concentrazione di scorte | Volume/peso | Concentrazione finale |
| Mezzo M2 | - | 380 μL | - |
| ialuronidasi | 10 mg/mL | 20 μL | 0,5 mg/mL |
| Abbreviazioni: LIF = fattore inibitorio della leucemia; MEF = fibroblasto embrionale del topo; | |||
| FBS = siero bovino fetale; DMEM = Mezzo aquila modificato di Dulbecco; BSA = albumina di siero bovino; | |||
| EDTA = acido etilendiamminatetraacetico; SDS = dodecilsolfuro di sodio; EGTA = acido tetraacetico etilene-bis(ossienenitrilo);; | |||
| PBS = salina tamponata da fosfati; PVP = polivinilpirrolidone; hCG = gonadotropina corionica umana; | |||
| PMSG = gonadotropina siere della cavalla incinta. | |||
Tabella 1: Ricetta di mezzo, tampone e soluzione.
| nome | Sequenza (da 5' a 3') | applicazione | |
| H19-DMR-P1 | GTG GTT AGT TCT ATA TGG GG | Genotipizzazione | |
| H19-DMR-P2 | AGA TGG GGT CAT TCT TTT CC | Genotipizzazione | |
| H19-DMR-P3 | TCT TAC AGT CTG GTC TTG GT | Genotipizzazione | |
| IG-DMR-P1 | TGT GCA GCA GCA AAG CTA AG | Genotipizzazione | |
| IG-DMR-P2 | CCA CAA AAA CCT CCC TTT CA | Genotipizzazione | |
| IG-DMR-P3 | ATA CGA TAC GGC AAC CAA CG | Genotipizzazione | |
| H19-DMR-gRNA1-F | CAC CCA TGA ACT CAG AAG AGA CTG | gRNA | |
| H19-DMR-gRNA1-R | AAA CCA GTC TCT TCT GAG TTC ATG | gRNA | |
| H19-DMR-gRNA2-F | CAC CAG GTG AGA ACC ACT GCT GAG | gRNA | |
| H19-DMR-gRNA2-R | AAA CCT CAG CAG TGG TTC TCA CCT | gRNA | |
| IG-DMR-gRNA1-F | CAC CCG TAC AGA GCT CCA TGG CAC | gRNA | |
| IG-DMR-gRNA1-R | AAA CGT GCC ATG GAG CTC TGT ACG | gRNA | |
| IG-DMR-gRNA2-F | CAC CCT GCT TAG AGG TAC TAC GCT | gRNA | |
| IG-DMR-gRNA2-R | AAA CAG CGT AGT ACC TCT AAG CAG | gRNA | |
Tabella 2: Elenco degli oligonucleotidi.
Discussione
La clonazione dei mammiferi mediante trasferimento nucleare di cellule somatiche (SCNT) è stata introdotta negli anni'90 19,20,21. Questi sviluppi hanno fatto seguito a studi di clonazione condotti 30 anni prima inanfibi 22. Il notevole ritardo riflette la difficoltà dell'embriologia e dell'imprinting genomico nei mammiferi. Lo sviluppo di SCNT di mammiferi è la base per l'applicazione dell'haESC per sostituire lo sperma, che è dettagliato in questo protocollo.
La sincronizzazione del ciclo cellulare è un fattore importante per il successo di SCNT23. Questo vale anche per l'iniezione di haESC in questo protocollo. L'introduzione di un genoma donatore in un ricevente richiede che le fasi del ciclo cellulare siano abbinate per evitare rotture cromosomiche o aneuploidie che abrogano lo sviluppo dell'embrione. La semiclonazione ha l'ulteriore complessità che due genomi e un citoplasto devono essere compatibili. Una precedente relazione ha dimostrato che l'iniezione di ESESC androgenetici arrestati in fase di m ha prodotto tassi di sviluppo migliori di embrioni semi-clonati rispetto all'iniezione di ESESC in fase G1 negli ovociti7. Questo rapporto suggerisce la fase M come punto di sincronizzazione adatto per la semiclonazione. Di conseguenza, i faESC sono stati arrestati mitoticamente in metafase con demecolicina e iniettati nell'ooplasma degli ovociti MII, che sono stati naturalmente arrestati nella metafase II di meiosi. È importante sottolineare che l'arresto in fase M può essere raggiunto negli ESC dei topi con alta efficienza, fornendo un'eccellente sincronizzazione tra i cicli cellulari del donatore e del ricevente.
Durante la mitosi, la membrana nucleare si rompe e si forma un mandrino a cui si attaccano i cromosomi replicati. Dopo l'iniezione di DKO-fasi M, i cromatidi sorelle vengono segregati in un corpo pseudo polare e nello zigote7 (Figura 3E). Di conseguenza, un singolo insieme di cromosomi di un DKO-phaESC contribuisce all'embrione semi-clonato. È fondamentale che le cromatidi sorelle dei cromosomi DKO-phaESC possano essere correttamente segregate dopo l'iniezione. La membrana plasmatica di un DKO-phaESC intatto previene la segregazione in un corpo pseudo polare. Abbiamo infatti osservato rari casi in cui la membrana plasmatica dei DKO-faSE non è stata interrotta e gli embrioni hanno mostrato DKO-faESC intatti nell'ooplasma dopo l'iniezione (Figura 3D). Pertanto, è necessario fare attenzione a rimuovere la membrana plasmatica dei DKO-faSCI mediante pipettazione. Durante l'iniezione, è altrettanto importante evitare l'interruzione del mandrino meiotico dell'ovocita, che potrebbe portare a difetti di segregazione cromosomici e indurre anche l'aneuploidia.
Nei mammiferi, l'imprinting genomico limita l'applicazione dei fasesci come sostituzione dello sperma. Gli haESC partenogenetici possiedono una configurazione materna delle impronte genomiche, mentre gli spermatozoi possiedono una configurazione paterna. Pertanto, la generazione di cuccioli a tempo pieno non si è verificata dopo l'iniezione di faESC di tipo selvatico come sostituzione dello sperma. Per superare questa limitazione, le eliminazioni dei DLF IG- e H19- sono progettate in fasi. La modifica dell'espressione impressa nei loci materni Igf2-H19 e Gtl2-Dlk1 è sufficiente per cambiare la configurazione delle impronte genomiche per consentire la generazione di topi semi-clonati con una frequenza superiore al 5,1%, basata su embrioni trasferiti a 2 cellule. Queste osservazioni suggeriscono che il targeting di due geni impressi commuta il genoma dei faESC in una configurazione paterna funzionale in grado di sostituire lo sperma nei topi. Tuttavia, per questa strategia è necessaria una modificazione genetica permanente dei FASC. Come strategia alternativa, si può prendere in considerazione l'haESC androgenetico. Gli haESC androgenetici derivano dal genoma dello sperma e possiedono impronte patene. Ci sono stati rapporti che gli haESC androgenetici di tipo selvatico hanno contribuito come sostituto dello sperma a generare cuccioli a lungo termine a una frequenza compresa tra l'1,3% e l'1,9%degli embrioni trasferiti 4,7,24. I cuccioli a tempo pieno sono stati ottenuti anche iniettando haESC androgenetici con deprezzazioni dei DR IG- e H19- ad una frequenza del 20,2% degli embrioni a 2 celluletrasferiti 24. L'aumento dell'efficienza della semiclonazione mediante haESC androgenetici modificati è probabilmente dovuto al fatto che le impronte possono diventare instabili nella cultura. I difetti di imprinting sono corretti dalle deiezioni genetiche dei D DMR critici.
Considerando la difficoltà di introdurre modifiche genetiche direttamente nei genomi degli ovocati o degli spermatozoi, gli ESES sono uno strumento prezioso per manipolare separatamente i genomi parentali. L'uso degli haESC come sostituto dello sperma offre un notevole vantaggio per l'editing del genoma nella linea germinale del topo. Un recente studio ha combinato l'editing del genoma basato su CRISPR / Cas9 con l'applicazione di haESC per la caratterizzazione delle regioni di imprinting che sono fondamentali per lo sviluppo embrionale12. Questo studio ha analizzato il ruolo del Rasgrf1-DMR in combinazione con i DMC H19e IGnello sviluppo di topi bimaternali e la funzione di 7 diversi DMC nello sviluppo di topi bipaternali. Il metodo per sostituire gli ESESC con lo sperma ha costituito la base per approcci di screening genetico per identificare gli amminoacidi chiave all'interno della proteina DND1 nello sviluppo primordiale delle cellule germinali e per identificare i geni nellosviluppo osseo 24,25,26. Studi sull'imprinting genomico e sullo screening genetico per identificare i fattori chiave nello sviluppo embrionale sono approcci considerevoli per l'applicazione degli eSESC come genomi gametici.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo il Dott. Giulio Di Minin per la derivazione delle linee phaESC e il Dott. Remo Freimann per il funzionamento della citometria a flusso. Riconosciamo anche la signora Michèle Schaffner e il signor Thomas M. Hennek per il supporto tecnico con il trasferimento di embrioni. Questo lavoro è stato sostenuto dalla Fondazione nazionale svizzera per la scienza (grant 31003A_152814/1).
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mLTube | Eppendorf | 0030 125.150 | haESC culture |
| 15 ml Tube | Greiner Bio-One | 188271 | haESC culture |
| 12-well plate | Thermo Scientific | 150628 | haESC culture |
| 24-well plate | Thermo Scientific | 142475 | haESC culture |
| 6-well plate | Thermo Scientific | 140675 | haESC culture |
| 96-well plate | Thermo Scientific | 167008 | haESC culture |
| β-Mercaptoethanol | Merck | M6250 | haESC culture |
| BSA fraction | Gibco | 15260-037 | haESC culture |
| CHIR 99021 | Axon | 1386 | haESC culture |
| Demecolcine solution | Merck | D1925 | haESC culture |
| DMEM | Gibco | 41965-039 | haESC culture |
| DMEM / F-12 | Gibco | 11320-033 | haESC culture |
| DR4 mouse | The Jackson Laboratory | 3208 | haESC culture |
| EDTA | Merck | E5134 | haESC culture |
| FBS | biowest | S1810-500 | haESC culture |
| Freezing medium | amsbio | 11897 | haESC culture |
| Gelatin | Merck | G1890 | haESC culture |
| Hepes solution | Gibco | 15630-056 | haESC culture |
| Irradiated MEF | Gibco | A34966 | haESC culture |
| LIF (Leukemia inhibitory factor) | (Homemade) | - | haESC culture |
| Lipofection reagent | Invitrogen | 11668019 | haESC culture |
| NDiff 227 | Takara | Y40002 | haESC culture |
| PD 0325901 | Axon | 1408 | haESC culture |
| Pen Strep | Gibco | 15140-122 | haESC culture |
| piggyBac plasmid carrying a CAG-EGFP transgene | Addgene | #40973 | haESC culture |
| piggyBac transposase plasmid | System Biosciences | PB210PA-1 | haESC culture |
| pX330 plasmid | Addgene | #42230 | haESC culture |
| pX458 plasmid | Addgene | #48138 | haESC culture |
| Trypsin | Gibco | 15090-046 | haESC culture |
| DNA polymerase | Thermo Scientific | F549S | Genotyping |
| dNTP Mix | Thermo Scientific | R0192 | Genotyping |
| Ethanol | Merck | 100983 | Genotyping |
| Hydrochloric acid | Merck | 100317 | Genotyping |
| Isopropanol | Merck | 109634 | Genotyping |
| Proteinase K | Axon | A3830 | Genotyping |
| SDS | Merck | 71729 | Genotyping |
| Sodium chloride | Merck | 106404 | Genotyping |
| ThermoMixer | Epeendorf | 5384000012 | Genotyping |
| Tris | Merck | T1503 | Genotyping |
| 5 mL Tube | Falcon | 352063 | Flow cytometry |
| 5 mL Tube with cell strainer cap | Falcon | 352235 | Flow cytometry |
| Hoechst 33342 | Invitrogen | H3570 | Flow cytometry |
| MoFlo Astrios EQ | Beckman Coulter | B25982 | Flow cytometry |
| 1 mL syringe | Codan | 62.1640 | Superovulation |
| 30-gauge 1/2 inch needle | Merck | Z192362-100EA | Superovulation |
| B6D2F1 mouse | The Jackson Laboratory | 100006 | Superovulation |
| hCG | MSD animal health | 49451 | Superovulation |
| PBS | Gibco | 10010015 | Superovulation |
| PMSG | Avivasysbio | OPPA1037 5000 IU | Superovulation |
| 10-cm dish | Thermo Scientific | 150350 | Embryo manipulation |
| 4-well plate | Thermo Scientific | 179830 | Embryo manipulation |
| 50 mL tube | Greiner Bio-One | 227261 | Embryo manipulation |
| 6 cm dish | Thermo Scientific | 150288 | Embryo manipulation |
| Center-well dish | Corning | 3260 | Embryo manipulation |
| Digital rocker | Thermo Scientific | 88880020 | Embryo manipulation |
| EGTA | AppliChem | A0878 | Embryo manipulation |
| Hyaluronidase | Merck | H4272 | Embryo manipulation |
| KSOM | Merck | MR-020P-5F | Embryo manipulation |
| M16 medium | Merck | M7292 | Embryo manipulation |
| M2 medium | Merck | M7167 | Embryo manipulation |
| Mineral oil | Merck | M8410 | Embryo manipulation |
| Glass capillary | Merck | P1049-1PAK | Embryo manipulation |
| Stereomicroscope | Nikon | SMZ745 | Embryo manipulation |
| Strontium chloride | Merck | 13909 | Embryo manipulation |
| 5ml syringe | Codan | 62.5607 | Microinjection |
| Borosilicate glass cappilary | Science product | GB100T-8P | Microinjection |
| CellTram 4r Oil | Eppendorf | 5196000030 | Microinjection |
| Fluorinert FC-770 | Merck | F3556 | Microinjection |
| Holding pipette | BioMedical Instruments | - | Microinjection |
| Inverted microscope | Nikon | Eclipse Ti-U | Microinjection |
| Microforge | Narishige | MF-900 | Microinjection |
| Microinjection pipette | BioMedical Instruments | - | Microinjection |
| Microlaoder tips | Eppendorf | 5252 956.003 | Microinjection |
| Micromanipulaor arms | Narishige | NT-88-V3 | Microinjection |
| Micropipette puller | Sutter Instrument | P-1000 | Microinjection |
| PiezoXpert | Eppendorf | 5194000016 | Microinjection |
| PVP (Polyvinylpyrrolidine) | Merck | PVP360 | Microinjection |
| Syringe filter | SARSTEDT | 83.1826.001 | Microinjection |
Riferimenti
- Ellegren, H., Galtier, N. Determinants of genetic diversity. Nature Reviews: Genetics. 17 (7), 422-433 (2016).
- Huijbers, I. J. Generating genetically modified mice: A decision guide. Methods in Molecular Biology. 1642, 1-19 (2017).
- Aizawa, E., Dumeau, C. E., Freimann, R., Di Minin, G., Wutz, A. Polyploidy of semi-cloned embryos generated from parthenogenetic haploid embryonic stem cells. PloS One. 15 (9), 0233072 (2020).
- Li, W., et al. Androgenetic haploid embryonic stem cells produce live transgenic mice. Nature. 490 (7420), 407-411 (2012).
- Li, Z., et al. Birth of fertile bimaternal offspring following intracytoplasmic injection of parthenogenetic haploid embryonic stem cells. Cell Research. 26 (1), 135-138 (2016).
- Wan, H., et al. Parthenogenetic haploid embryonic stem cells produce fertile mice. Cell Research. 23 (11), 1330-1333 (2013).
- Yang, H., et al. Generation of genetically modified mice by oocyte injection of androgenetic haploid embryonic stem cells. Cell. 149 (3), 605-617 (2012).
- Zhong, C., et al. Parthenogenetic haploid embryonic stem cells efficiently support mouse generation by oocyte injection. Cell Research. 26 (1), 131-134 (2016).
- Elling, U., et al. Forward and reverse genetics through derivation of haploid mouse embryonic stem cells. Cell Stem Cell. 9 (6), 563-574 (2011).
- Leeb, M., Wutz, A. Derivation of haploid embryonic stem cells from mouse embryos. Nature. 479 (7371), 131-134 (2011).
- Surani, M. A., Barton, S. C., Norris, M. L. Development of reconstituted mouse eggs suggests imprinting of the genome during gametogenesis. Nature. 308 (5959), 548-550 (1984).
- Li, Z. K., et al. Generation of bimaternal and bipaternal mice from hypomethylated haploid ESCs with imprinting region deletions. Cell Stem Cell. 23 (5), 665-676 (2018).
- Elling, U., et al. Derivation and maintenance of mouse haploid embryonic stem cells. Nature Protocols. 14 (7), 1991-2014 (2019).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shuai, L., et al. Generation of mammalian offspring by haploid embryonic stem cells microinjection. Current Protocols in Stem Cell Biology. 31, 1-15 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923 (2012).
- Behringer, R., Gertsenstein, M., Nagy, K. V., Nagy, A. . Manipulating the Mouse Embryo: A Laboratory Manual. , (2014).
- O'Neill, G. T., Rolfe, L. R., Kaufman, M. H. Developmental potential and chromosome constitution of strontium-induced mouse parthenogenones. Molecular Reproduction and Development. 30 (3), 214-219 (1991).
- Campbell, K. H., McWhir, J., Ritchie, W. A., Wilmut, I. Sheep cloned by nuclear transfer from a cultured cell line. Nature. 380 (6569), 64-66 (1996).
- Wakayama, T., Perry, A. C., Zuccotti, M., Johnson, K. R., Yanagimachi, R. Full-term development of mice from enucleated oocytes injected with cumulus cell nuclei. Nature. 394 (6691), 369-374 (1998).
- Wilmut, I., Schnieke, A. E., McWhir, J., Kind, A. J., Campbell, K. H. Viable offspring derived from fetal and adult mammalian cells. Nature. 385 (6619), 810-813 (1997).
- Gurdon, J. B. Adult frogs derived from the nuclei of single somatic cells. Developmental Biology. 4, 256-273 (1962).
- Campbell, K. H., Alberio, R. Reprogramming the genome: role of the cell cycle. Reproduction Supplement. 61, 477-494 (2003).
- Zhong, C., et al. CRISPR-Cas9-mediated genetic screening in mice with haploid embryonic stem cells carrying a guide RNA library. Cell Stem Cell. 17 (2), 221-232 (2015).
- Bai, M., et al. Targeted genetic screening in mice through haploid embryonic stem cells identifies critical genes in bone development. PLoS Biology. 17 (7), 3000350 (2019).
- Li, Q., et al. CRISPR-Cas9-mediated base-editing screening in mice identifies DND1 amino acids that are critical for primordial germ cell development. Nature Cell Biology. 20 (11), 1315-1325 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon