Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Mediciones basadas en fluorescencia del intercambio de fosfatidilserina/fosfatidilinositol 4-fosfato entre membranas
En este artículo
Resumen
Aquí, describimos protocolos que utilizan sensores de lípidos fluorescentes y liposomas para determinar si una proteína extrae y transporta fosfatidilserina o fosfatidilinositol 4-fosfato in vitro.
Resumen
Recientemente se ha encontrado que varios miembros de la familia de proteínas relacionadas con la proteína de unión al oxisterol (OSBP) (ORP)/homólogos OSBP (Osh) conservadas evolutivamente representan un nuevo grupo de proteínas de transferencia de lípidos (LTP) en levaduras y células humanas. Transfieren fosfatidilserina (PS) del retículo endoplásmico (ER) a la membrana plasmática (PM) a través de ciclos de intercambio PS/fosfatidilinositol 4-fosfato (PI(4)P). Este hallazgo permite una mejor comprensión de cómo el PS, que es crítico para los procesos de señalización, se distribuye por toda la célula y la investigación del vínculo entre este proceso y el metabolismo de la fosfoinosítida (PIP). El desarrollo de nuevos protocolos basados en la fluorescencia ha sido fundamental en el descubrimiento y caracterización de este nuevo mecanismo celular in vitro a nivel molecular. Este artículo describe la producción y el uso de dos sensores lipídicos etiquetados fluorescentemente, NBD-C2Lact y NBD-PHFAPP,para medir la capacidad de una proteína para extraer PS o PI(4)P y transferir estos lípidos entre membranas artificiales. En primer lugar, el protocolo describe cómo producir, etiquetar y obtener muestras de alta pureza de estas dos construcciones. En segundo lugar, este artículo explica cómo usar estos sensores con un lector de microplacas de fluorescencia para determinar si una proteína puede extraer PS o PI(4)P de liposomas, utilizando Osh6p como estudio de caso. Finalmente, este protocolo muestra cómo medir con precisión la cinética del intercambio PS/PI(4)P entre liposomas de composición lipídica definida y determinar las tasas de transferencia de lípidos por transferencia de energía de resonancia de fluorescencia (FRET) utilizando un fluorómetro estándar.
Introducción
La distribución precisa de lípidos entre diferentes membranas y dentro de las membranas de las células eucariotas1,2 tiene profundas implicaciones biológicas. Descifrar cómo funcionan los LTP es un tema importante en biología celular3,4,5,6,y los enfoques in vitro son de gran valor para abordar este problema7,8,9,10,11. Aquí, se presenta una estrategia in vitrobasada en la fluorescencia que ha sido fundamental para establecer que varias proteínas ORP/Osh afectan el intercambio PS/PI(4)P entre las membranas celulares12 y con ello constituyen una nueva clase de LTPs. PS es un glicerofosfolípido aniónico que representa el 2-10% de los lípidos totales de membrana en células eucariotas13,14,16. Se distribuye a lo largo de un gradiente entre el ER y el PM, donde representa el 5-7% y hasta el 30% de los glicerofosfolípidos, respectivamente17,18,19. Además, el PS se concentra esencialmente en la valva citosólica del PM. Esta acumulación y la partición desigual de PS en el PM son críticas para los procesos de señalización celular19. Debido a la carga negativa de las moléculas de PS, la valva citosólica del PM es mucho más ancianómica que la valva citosólica de otros orgánulos1,2,19,20. Esto permite el reclutamiento, a través de fuerzas electrostáticas, de proteínas de señalización como el sustrato de C-quinasa rico en alanina miristoilada (MARCKS)21,sarcoma (Src)22,el oncogén viral del sarcoma de Kirsten-rat (K-Ras)23y el sustrato de toxina botulínica C3 relacionada con Ras 1 (Rac1)24 que contienen un tramo de aminoácidos cargados positivamente y una cola lipídica.
PS también es reconocido por la proteína quinasa C convencional de manera estereoselectiva a través de un dominio C225. Sin embargo, PS se sintetiza en el ER26,lo que indica que debe exportarse al PM antes de que pueda desempeñar su papel. No se supo cómo se logró esto19 hasta el hallazgo de que, en la levadura, Osh6p y Osh7p transfieren PS desde el ER al PM27. Estos LTP pertenecen a una familia evolutivamente conservada en eucariotas cuyo miembro fundador es OSBP y que contiene proteínas (ORPs en humanos, proteínas Osh en levaduras) integrando un dominio relacionado con OSBP (ORD) con una bolsa para albergar una molécula lipídica. Osh6p y Osh7p consisten solo en un ORD cuyas características estructurales están adaptadas para unirse específicamente a PS y transferirlo entre membranas. Sin embargo, no estaba claro cómo estas proteínas transfirieron direccionalmente PS de la ER a la PM. Osh6p y Osh7p pueden atrapar PI(4)P como un ligando lipídico alternativo12. En levaduras, PI(4)P se sintetiza a partir de fosfatidilinositol (PI) en el Golgi y el PM por PI 4-quinasas, Pik1p y Stt4p, respectivamente. Por el contrario, no hay PI(4)P en la membrana ER, ya que este lípido es hidrolizado a PI por la fosfatasa Sac1p. Por lo tanto, existe un gradiente PI(4)P tanto en las interfaces ER/Golgi como ER/PM. Osh6p y Osh7p transfieren PS del ER al PM a través de ciclos de intercambio PS/PI(4)P utilizando el gradiente PI(4)P que existe entre estas dos membranas12.
Dentro de un ciclo, Osh6p extrae PS del ER, intercambia PS por PI(4)P en el PM y transfiere PI(4)P de vuelta al ER para extraer otra molécula de PS. Osh6p/Osh7p interactúan con Ist2p28, una de las pocas proteínas que conectan y acercan la membrana ER y el PM entre sí para crear sitios de contacto ER-PM enlevaduras 29,30,31. Además, la asociación de Osh6p con membranas cargadas negativamente se debilita tan pronto como la proteína extrae uno de sus ligandos lipídicos debido a un cambio conformacional que modifica sus características electrostáticas32. Esto ayuda a Osh6p al acortar su tiempo de permanencia en la membrana, manteniendo así la eficiencia de su actividad de transferencia de lípidos. Combinado con la unión a Ist2p, este mecanismo podría permitir que Osh6p/7p ejecute de forma rápida y precisa el intercambio de lípidos en la interfaz ER/PM. En células humanas, las proteínas ORP5 y ORP8 ejecutan el intercambio PS/PI(4)P en los sitios de contacto ER-PM a través de mecanismos distintos33. Tienen un ORD central, similar a Osh6p, pero están anclados directamente al ER a través de un segmento transmembrana C-terminal33 y se acoplen al PM a través de un dominio de homología de Pleckstrin (PH) N-terminal que reconoce PI(4)P y PI(4,5)P233,34,35. ORP5/8 utiliza PI(4)P para transferir PS, y se ha demostrado que ORP5/8 regula adicionalmente los niveles de PM PI(4,5)P2 y presumiblemente modula las vías de señalización. A su vez, una disminución en los niveles de PI(4)P y PI(4,5)P2 reduce la actividad de ORP5/ORP8 ya que estas proteínas se asocian con el PM de una manera dependiente de PIP. La síntesis anormalmente alta de PS, que conduce al síndrome de Lenz-Majewski, afecta los niveles de PI(4)P a través de ORP5/836. Cuando se bloquea la actividad de ambas proteínas, el PS se vuelve menos abundante en el PM, disminuyendo la capacidad oncogénica de las proteínas de señalización37.
Por el contrario, la sobreexpresión de ORP5 parece promover la invasión de células cancerosas y los procesos metastásicos38. Por lo tanto, las alteraciones en la actividad de ORP5/8 pueden modificar severamente el comportamiento celular a través de cambios en la homeostasis lipídica. Además, ORP5 y ORP8 ocupan sitios de contacto ER-mitocondrias y preservan algunas funciones mitocondriales, posiblemente suministrando PS39. Además, ORP5 se localiza en los sitios de contacto de gotas lipídicas ER para administrar PS a las gotas lipídicas mediante el intercambio PS/PI(4)P40. La estrategia aquí descrita para medir (i) la extracción de PS y PI(4)P de liposomas y (ii) el transporte de PS y PI(4)P entre liposomas ha sido ideada para establecer y analizar la actividad de intercambio PS/PI(4)P de Osh6p/Osh7p12,32 y utilizada por otros grupos para analizar la actividad de ORP5/ORP835 y otras LTPs10, 41. Se basa en el uso de un lector de placas de fluorescencia, un espectrofluorómetro estándar de formato L y dos sensores fluorescentes, NBD-C2Lact y NBD-PHFAPP,que pueden detectar PS y PI(4)P, respectivamente.
NBD-C2Lact corresponde al dominio C2 de la glicoproteína, la lactadherina, que fue rediseñado para incluir una cisteína única expuesta a solvente cerca del presunto sitio de unión a PS; un fluoróforo NBD (7-nitrobenz-2-oxa-1,3-diazol) sensible a la polaridad está unido covalentemente a este residuo (Figura 1A)12. Para ser más precisos, el dominio C2 de la lactadherina (Bos taurus, UniProt: Q95114, residuos 270-427) se clonó en un vector pGEX-4T3 para ser expresado en fusión con glutatión S-transferasa (GST) en Escherichia coli. La secuencia C2Lact se mutó para sustituir dos residuos de cisteína accesibles al disolvente (C270, C427) con residuos de alanina e introducir un residuo de cisteína en una región cercana al supuesto sitio de unión a PS (mutación H352C) que posteriormente se puede etiquetar con N,N'-dimetil-N-(yodoacetil)-N'-(7-nitrobenz-2-oxa-1,3-diazol-4-yl) etileno diamina (IANBD) 12. Un sitio de escisión para la trombina está presente entre la proteína GST y el N-terminal del dominio C2. Una ventaja importante es que este dominio reconoce selectivamente PS de una manera independiente de Ca2+a diferencia de otros dominios C2 conocidos o Anexina A542. NBD-PHFAPP se deriva del dominio PH de la proteína 1 humana adaptadora de cuatro fosfatos (FAPP1), que fue rediseñada para incluir una sola cisteína expuesta a solvente que puede etiquetarse con un grupo NBD cerca del sitio de unión PI(4)P(Figura 1A)43. La secuencia de nucleótidos del dominio PH de la proteína FAPP humana (UniProt: Q9HB20, segmento [1-100]) ha sido clonada en un vector pGEX-4T3 para ser expresado en tándem con una etiqueta GST. La secuencia PHFAPP ha sido modificada para insertar un residuo único de cisteína dentro de la interfaz de unión a la membrana de la proteína43. Además, se ha introducido un enlazador de nueve residuos entre el sitio de escisión de la trombina y el N-terminal del dominio PH para garantizar la accesibilidad a la proteasa.
Para medir la extracción de PS de liposomas, NBD-C2Lact se mezcla con liposomas hechos de fosfatidilcolina (PC) que contienen trazas de PS. Debido a su afinidad por PS, esta construcción se une a los liposomas, y el fluoróforo NBD experimenta un cambio en la polaridad a medida que entra en contacto con el ambiente hidrófobo de la membrana, lo que provoca un cambio de azul y un aumento en la fluorescencia. Si PS se extrae casi completamente por una cantidad estequiométrica de LTP, la sonda no se asocia con liposomas, y la señal NBD es menor (Figura 1B)32. Esta diferencia de señal se utiliza para determinar si un LTP(por ejemplo, Osh6p) extrae PS. Una estrategia similar se utiliza con NBD-PHFAPP para medir la extracción de PI(4)P(Figura 1B),como se describió anteriormente12,32. Se diseñaron dos ensayos basados en FRET para (i) medir el transporte de PS de los liposomas LA a LB, que imitan la membrana ER y el PM, respectivamente, y (ii) el transporte pi(4)P en la dirección inversa. Estos ensayos se realizan en las mismas condiciones(es decir, el mismo tampón, temperatura y concentración de lípidos) para medir el intercambio PS/PI(4)P. Para medir el transporte de PS, NBD-C2Lact se mezcla con liposomas LA compuestos de PC y se dopa con 5 mol% PS y 2 mol% de fosfatidiletanolamina (Rhod-PE)- fluorescente-marcando rodamina y liposomas LB que incorporan 5 mol% PI(4)P.
En el momento cero, FRET con Rhod-PE apaga la fluorescencia NBD. Si PS se transporta de liposomas LA a LB (por ejemplo, al inyectar Osh6p), se produce un desencanto rápido debido a la translocación de moléculas delact NBD-C2 de liposomas LA a LB (Figura 1C). Dada la cantidad de PS accesible, NBD-C2Lact permanece esencialmente en un estado unido a la membrana en el transcurso del experimento12. Por lo tanto, la intensidad de la señal NBD se correlaciona directamente con la distribución de NBD-C2Lact entre los liposomas LA y LB y se puede normalizar fácilmente para determinar cuánto PS se transfiere. Para medir la transferencia de PI(4)P en la dirección opuesta, NBD-PHFAPP se mezcla con liposomas LA y LB; dado que solo se une a liposomas LB que contienen PI(4)P, pero no Rhod-PE, su fluorescencia es alta. Si PI(4)P se transfiere a los liposomas LA, se transloca a estos liposomas, y la señal disminuye debido a FRET con Rhod-PE(Figura 1C). La señal se normaliza para determinar cuánto PI(4)P se transfiere43.
Protocolo
1. Purificación de NBD-C2Lact
NOTA: Aunque este protocolo detalla el uso de un disruptor celular para romper bacterias, se puede modificar para usar otras estrategias de lisis(por ejemplo, una prensa francesa). Al comienzo de la purificación, es obligatorio usar tampón recién desgastido, filtrado y complementado con 2 mM de ditiotiretol (TDT) para evitar la oxidación de la cisteína. Sin embargo, para el paso de etiquetado de proteínas, es crucial eliminar completamente la TDT. Muchos pasos deben llevarse a cabo en hielo o en una cámara frigorífica para evitar cualquier degradación de proteínas. Se deben recoger muestras de 30 μL de volumen en diferentes etapas del protocolo para realizar un análisis por electroforesis en gel de dodecilsulfato de sodio-poliacrilamida (SDS-PAGE) utilizando un gel de acrilamida al 15% para comprobar el progreso de la purificación. Mezcle suficiente tampón de muestra de Laemmli desnaturalizando con cada alícuota y caliente la mezcla a 95 °C. Congelar y conservar los tubos a -20 °C hasta su análisis.
- Expresión de GST-C2Lact en Escherichia coli
- Mezclar 20 μL de células competentes BL21 Gold con 18 μL de agua esterilizada. Luego, mezcle 2 μL de plásmido pGEX-C2Lact (a ~ 65 ng / μL) con las bacterias y transfórice por electroporación. Resuspend las bacterias con 150 μL de lennox Lysogeny-Broth (LB) medio en autoclave (10 g/L de triptona, 5 g/L de extracto de levadura, 5 g/L de NaCl en agua desionizada, libre de glucosa). Deje que las bacterias crezcan a 37 °C durante 1 h en un tubo de microcentrífuga de tapa a presión de 2 ml.
- Inocular 25 mL de LB medio, suplementado con 50 μg/mL de ampicilina, con 150 μL de suspensión bacteriana en un matraz Erlenmeyer estéril de 125 mL. Coloque el matraz en un agitador orbital a 37 °C y deje que las bacterias crezcan durante la noche con agitación a 185 rpm.
- Llene dos matraces estériles Erlenmeyer de 2 L con 500 ml de medio LB suplementado con 50 μg/ml de ampicilina, y agregue 5 ml de suspensión de precultivo. Deje que las bacterias crezcan a 37 °C con agitación a 220 rpm.
- Medir periódicamente la densidad óptica (OD) de la suspensión a una longitud de onda (λ) de 600 nm. Cuando el OD alcance un valor de ~0.6-0.7, agregue 500 μL de una solución stock de 1 M de isopropilo β-D-1-tiogalactopyranoside (IPTG) a cada matraz para iniciar la expresión de GST-C2Lact. Agite los matraces a 185 rpm durante 4 h a 37 °C.
- Transfiera el contenido de cada matraz a una botella centrífuga de polipropileno. Centrifugar los dos frascos durante 30 min a 4600 × g a 4 °C para peletizar las bacterias. Deseche el sobrenadante y vuelva a colocar cada gránulo en 50 ml de solución salina tamponada con fosfato en frío.
- Transfiera la suspensión bacteriana contenida en cada frasco a un tubo de centrífuga cónica de 50 ml. Centrifugar los dos tubos durante 30 min a 2300 × g a 4°C. Retire el sobrenadante y guarde los tubos, cada uno de los cuales contiene un gránulo bacteriano, a -20 °C.
- Purificación de C2Lact
- En hielo, llene dos tubos centrífugos cónicos de 50 ml con 50 ml de tampón que contengan 50 mM Tris-HCl, pH 7.4 y 150 mM NaCl (en adelante, TN buffer), previamente filtrados y desgasisados por filtración al vacío por membrana.
- Para preparar el tampón de lisis en cada tubo, disuelva una tableta de cóctel inhibidor de la proteasa sin ácido tetraacético de etilendiamina en el tampón TN mediante sonicación leve o vórtice. Añadir otros antiproteasas (10 μM de bestatina, 1 μg/ml de pepstatina A y 10 μM de fosforamidadín). Es importante complementar el búfer con 2 mM de TDT.
- Llene los dos tubos que contienen los gránulos bacterianos preparados en la etapa 1.1.6, con tampón de lisis para obtener un volumen final de 30 ml en cada tubo, y descongele lentamente los gránulos en hielo durante 10 minutos. Triturar cada pellet con una espátula de acero inoxidable, y resuspendiéndolos vórtice de los tubos y/o pipeteando la suspensión hacia adelante y hacia atrás con un controlador de pipeta y una pipeta de 25 mL hasta obtener una suspensión homogénea.
- Realice la lisis utilizando un disruptor de celda preenfriado (consulte la Tabla de Materiales)cargando 30 ml de la muestra dentro del depósito y ejecutando un ciclo de ruptura en modo continuo con una presión de 1,6 bar. Recoja el lisato en el mismo tubo, mantenga el tubo en hielo e inmediatamente agregue 250 μL de una solución stock de fluoruro de fenilmetilsulfonilo (PMSF) de 200 mM preparada en isopropanol.
- Lise la otra muestra siguiendo el mismo procedimiento. Use el resto del tampón de lisis para lavar el disruptor celular y recoja el lavado para ajustar el volumen de cada lisato (~ 30 ml) a un volumen final de 50 ml.
- Suplementar cada lisato con 5 mM MgCl2,y añadir 20 μg/mL de DNAse I para fragmentar el ADN y así reducir la viscosidad de la muestra. Incubar en hielo durante 30 min. Recoger una muestra para el análisis de gel.
- Transfiera cada lisato de 50 ml a un tubo de ultracentrífuga de policarbonato preenfriado (dos en total, consulte la Tabla de materiales). Centrifugar a 186.000 × g a 4 °C durante al menos 1 h utilizando una ultracentrífuga.
- En paralelo a la etapa de centrifugación, dispensar 1,4 ml de una suspensión que contenga glutatión acoplada a perlas de agarosa al 4% en dos tubos centrífugos cónicos de 50 ml, agregar 20 ml de tampón TN complementado con 1 mM de TDT (tampón TND) a cada tubo, centrifugar a 1200 × g durante 5 minutos y desechar el sobrenadante. Repita este paso de lavado dos veces.
- Después de la centrifugación del lisato bacteriano, retire una muestra de 30 μL del sobrenadante y transfiera el sobrenadante de cada tubo de ultracentrífuga a un tubo de centrífuga cónica de 50 ml correspondiente que contenga perlas limpias. Para el análisis de gel, vuelva a colocar el pellet de escombros en uno de los tubos de la ultracentrífuga con 50 ml de tampón TND y recoja una muestra de 30 μL.
- Coloque los tubos en un rotador durante 3-4 h a 4 °C para obtener una suspensión de perla homogénea. Acondja las suspensiones de cuentas en una columna de cromatografía vacía de 25 ml. Deje que las perlas se decanten y elimine el tampón y las proteínas no unidas por flujo de gravedad. Tome una muestra del eludo para su análisis.
- Resuspend las perlas con 20 mL de tampón TND, y recoge el eluado por flujo de gravedad. Repita este paso dos veces para lavar completamente las cuentas. Aunar los eluidos recolectados y retener una muestra de 30 μL para su posterior análisis.
NOTA: Después de una breve decantación, un volumen de ~ 2 ml de suspensión de cuentas, a la que se une GST-C2Lact, sedimenta en la parte inferior de la columna. - Agregue 1 ml de suspensión de perlas a dos tubos de microcentrífuga de tapa a presión de 2 ml. Llene cada tubo con tampón TND hasta un volumen final de 1.970 ml. Tome una muestra de 30 μL de un tubo para su posterior análisis (muestra B1). Añadir 10 μL de solución de CaCl2 de 10 mM y 25 μL de una solución stock de solución de trombina proteasa humana a 0,02 U/μL.
- Coloque los dos tubos en un rotador a 4 °C durante la noche para permitir que la trombina se sespeje la etiqueta GST del dominio C2Lact. Al día siguiente, en cada tubo, mezcle 10 μL de solución pmSF de 200 mM con la suspensión de perlas para inhibir la acción de la trombina.
- Centrifugar los tubos a 700 × g durante 5 min, y recoger el sobrenadante, que contiene el dominio soluble C2Lact, de cada tubo, sin tomar las perlas. Aconjunte los sobrenadantes en un tubo de microcentrífuga de tapa a presión de 2 ml (eluado E1) que se mantiene en hielo.
- Agregue 1 ml de tampón TND a cada tubo para resuspend las perlas y lávelas; repita el paso 1.2.14. Realiza este paso tres veces más para recuperar una cantidad máxima de proteína. Cada vez, acoja los sobrenadantes recolectados en un nuevo tubo de 2 ml (eluidos E2, E3, E4 y E5) y tome una alícuota para un análisis más detallado. Al final de los pasos de lavado, tome una alícuota de la suspensión de cuentas (alícuota B2).
- Analizar las muestras de 30 μL que se recogieron en los diferentes pasos del protocolo de purificación mediante separación SDS-PAGE en un gel de acrilamida al 15%.
- Elimine las posibles perlas contaminantes agrupando todos los sobrenadantes(es decir,~ 10 ml) recolectados durante los pasos 1.2.14 y 1.2.15 en una columna de cromatografía de 10 ml. Recoge el eluado por flujo de gravedad y retiene las cuentas en la parte inferior de la columna.
- Concentrar la muestra C2Lact utilizando una unidad de filtro centrífugo con un corte de peso molecular (MWCO) de 3 kDa y una velocidad de centrifugación de 2300 × g. Detenga el procedimiento de concentración cuando el volumen de la muestra de proteína sea de ~ 1 ml.
- Preparación y purificación de NBD-C2Lact
- Equilibrar una columna de desafinación (ver la Tabla de Materiales)con tampón TN. Cargue la columna con 1 ml de muestra concentrada de C2Lact. Permita que la muestra entre completamente en el lecho de gel, agregue 1,5 ml de tampón TN libre de TDT recién desgasificado a la columna y recoja el eluido por flujo de gravedad en un tubo de microcentrífuga de tapa a presión de 2 ml.
- Diluya 50 μL de eluido en un volumen final de 300 μL de tampón TN y registre un espectro de absorbancia de 230 a 450 nm utilizando un búfer TN puro como espacio en blanco. Determinar la concentración de C2Lact en base a la absorbancia medida a 280 nm, considerando un coeficiente de extinción ε igual a 44.920 M-1.cm-1.
- Para etiquetar el constructo C2Lact con un fluoróforo NBD, mezcle la proteína con un exceso molar diez veces mayor de N,N'-dimetil-N-(yodoacetil)-N'-(7-nitrobenz-2-oxa-1,3-diazol-4-yl)etilendiamina (amida IANBD).
- Disolver 1 mg de IANBD en dimetilformamida anhidra (DMF), teniendo en cuenta que el volumen final de DMF utilizado para etiquetar la construcción C2Lact no debe exceder el 5% (v/v) del volumen de la muestra de proteína.
- Para determinar el volumen de DMF (VDMF)para disolver IANBD, primero calcule la cantidad requerida de IANBD (m, expresada en mg) para etiquetar la proteína utilizando la fórmula 1.
m = 10.000 × C × V × MWIANBD (1)
donde C es la concentración de C2Lact medida en el paso 1.3.2, V es el volumen de la muestra de C2Lact, y MWIANBD es el peso molecular del fluoróforo (420 g/mol). - Calcular VDMF usando las fórmulas 2 (con m0= 1) y 3.
VDMF= (m0/m) × VIANBD (2)
VIANBD=0,05 × V (3)
Donde m0 es la cantidad de polvo de IANBD en mg, y VIANBD es el volumen de solución de IANBD que se agregará a la muestra de C2Lact. - Añadir volumen VIANBD de la solución IANBD recién preparada a la muestra C2Lact y agitar la mezcla de reacción a 800-900 rpm durante 30 min a 25 °C con un termomezclador protegido de la luz. Deje que la reacción proceda durante 90 minutos sobre hielo. Mientras tanto, limpie una unidad de filtro centrífugo (MWCO = 3 kDa) con 10 ml de tampón TN.
- Agregue L-cisteína (en exceso molar de 10 veces a IANBD) a la mezcla de reacción para inactivar IANBD libre.
- Agregue 15 ml de tampón TN a la solución NBD-C2Lact y transfiera la solución NBD-C2Lact a la unidad de filtro centrífugo. Concentre la muestra a 2 ml para separar la mayor parte de la NBD libre de la proteína por centrifugación a 2300 × g. Repita este paso de lavado dos veces. Centrifugar la muestra en un tubo de centrífuga de tapa a presión de 2 ml durante 10 min a 19.000 × g a 4 °C para gletizar agregados potenciales, y recoger el sobrenadante.
- Diluir 50 μL de eluido en un volumen final de 300 μL de tampón TN. Registre el espectro de absorbancia de 230 a 650 nm utilizando el eluido recogido durante el procedimiento de concentración en blanco. Determinar la concentración de NBD-C2Lact utilizando la absorbancia máxima a λ=280 y 495 nm y los coeficientes de extinción ε= 44.920 M-1.cm-1 (proteína) y 25.000 M-1.cm-1 (fluoróforo NBD).
NOTA: Si los dos valores de concentración son iguales, esto indica que la construcción C2Lact está etiquetada en una proporción de 1:1 con el grupo NBD. - Si la concentración de NBD-C2Lact estimada a partir de la medición de la absorbancia de NBD excede la concentración estimada a partir de la absorbancia de residuos de triptófano (Trp), repita el paso 1.3.5 para eliminar aún más la NBD libre.
- Agregue glicerol a la muestra para obtener una concentración final del 10% (v / v) para crioproteger la construcción de NBD-C2Lact durante la congelación instantánea. Medir la concentración final de proteína.
- Preparar alícuotas de 50 μL de proteína en tubos de microcentrífuga de tapa a presión de 0,5 ml. Congele los tubos en nitrógeno líquido y guárdelos a -80 °C.
2. Purificación de NBD-PHFAPP
NOTA: El procedimiento para producir y etiquetar PHFAPP es idéntico al de NBD-C2Lact hasta la transferencia de la solución nbd-C2Lact a una unidad de filtro centrífugo en el paso 1.3.4. A partir de este paso, siga el protocolo que se describe a continuación.
- Después del paso de concentración, mantenga 2 ml de NBD-PHFAPP a 4 °C en la oscuridad durante no más de 1 día antes de realizar la cromatografía de exclusión de tamaño. Antes de la cromatografía de exclusión de tamaño, verifique que no haya un depósito de naranja (agregación durante la concentración) en la parte inferior del tubo. Si este es el caso, centrífuga la muestra a 540.000 × g durante 10 min a 4 °C, y purificar el sobrenadante mediante cromatografía de exclusión de tamaño.
NOTA: La cromatografía de exclusión de tamaño se realiza en una columna empaquetada con copolímero de dextrano-acrilamida reticulado (consulte la Tabla de Materiales),previamente equilibrado con tampón TND, utilizando un sistema de cromatografía líquida de proteína rápida (consulte la Tabla de Materiales). La columna debe estar protegida de la luz. Se utilizó un caudal de 1 mL/min, y la elución del constructo NBD-PHFAPP fue seguida por el registro de la absorbancia a λ=280 (proteína) y 480 nm (NBD) a la salida de la columna.- Inyecte la muestra NBD-PHFAPP cargada en un bucle de inyección de 2 ml en la columna e inmediatamente recoja 2,5 ml de fracciones de eluado.
- Analizar todas las fracciones que corresponden al pico principal detectado a 280 y 480 nm en un gel SDS-PAGE al 15%. Mezcle una muestra de 25 μL de cada fracción con 15 μL de tampón de muestra Laemmli antes de calentar y cargar en el gel.
NOTA: Un pico principal, que se detecta simultáneamente en λ = 280 y 480 nm, aparece una vez que se pasa un volumen de ~ 150 mL de búfer a través de la columna. - Agrupar las fracciones que contienen exclusivamente la proteína NBD-PHFAPP (~12,2 kDa), y añadir glicerol a una concentración final del 10% (v/v). Concentrar la muestra utilizando una unidad de filtro centrífugo con un MWCO de 3 kDa a un volumen final de 1 mL utilizando una velocidad de centrifugación de 2300 × g.
- Prepare alícuotas y registre un espectro de absorbancia como se describe para NBD-C2Lact. Utilice un coeficiente de extinción ε = 29,450 M-1.cm-1 para determinar la concentración de la proteína en función de la absorbancia medida a λ = 280 nm.
3. Preparación de liposomas para ensayos de extracción o transferencia de PS y PI(4)P
NOTA: Realice todos los pasos a temperatura ambiente a menos que se especifique lo contrario. Manipule los disolventes orgánicos, el rotavapor y el nitrógeno líquido con precaución.
- Prepare hidróxido de potasio (KOH) fresco, filtrado y desgasesado 50 mM 4-(2-hidroxietil)-1-piperazinatanosulfónico (HEPES), pH 7.4, 120 mM de acetato de potasio (HK).
- Para cada tipo de liposoma, tome cantidades precisas de diferentes lípidos de soluciones de stock y mézclelos en un matraz de vidrio en forma de pera de 25 ml (Tabla 1). Agregue cloroformo puro para ajustar el volumen de cada mezcla a 1 ml. Etiquete cada matraz con el nombre del liposoma. Envuelva los matraces que contienen la mezcla de lípidos dopada con Rhod-PE con papel de aluminio.
- Coloque el matraz en un evaporador rotativo. Secar los lípidos al vacío y a 25 °C durante al menos 30 min a una velocidad de rotación de 500 rpm. Para las películas lipídicas que contienen PI(4)P, precalencer el matraz a 32-34 °C durante 5 min, bajo una rotación suave, para mezclar adecuadamente PI(4)P con los otros lípidos antes de crear vacío en el matraz para eliminar el disolvente, que dejará una película de lípidos secos en la pared del matraz.
- Desconecte el matraz del evaporador y colóquelo en una cámara de vacío durante 45 minutos para eliminar cualquier resto de disolvente. Llene el matraz con 2 ml de tampón HK y agregue algunas perlas de vidrio de 4 mm de diámetro a la solución. Vórtice suavemente el matraz durante 2 min para resuspend los lípidos y preparar vesículas lipídicas multilamelares (MV) con una concentración de lípidos de 4 mM. Preparar alícuotas de 0,5 ml de MLM en tubos de microcentrífuga de tapa de rosca de 1,5 ml.
- Congelar y descongelar los tubos 5x (utilizando nitrógeno líquido y un baño de agua a 37 °C, respectivamente). Extruir los liposomas o almacenarlos a -20 °C.
- Use una mini extrusora para preparar los liposomas(es decir, vesículas unilamelares grandes) de los MLV de acuerdo con las pautas del fabricante. Utilice un filtro de policarbonato con poros cilíndricos uniformes de 200 nm de diámetro.
- Para preparar cada tipo de liposoma, extruir al menos 250 μL de la suspensión correspondiente de MV. Almacenar los liposomas extruidos a 4 °C y en la oscuridad si contienen Rhod-PE. Use los liposomas dentro de los 2 días.
| Composición lipídica (mol/mol) | Lípido | |||||
| Nombre del liposoma | DOPC (25 mg/ml) | POPS (10 mg/ml) | 16:0 Liss Rhod-PE (1 mg/ml) | C16:0/C16:0-PI(4)P (1 mg/ml) | ||
| Ensayos de extracción | Liposoma 2 mol% PS | PC/PS 98/2 | 247 μL | 12,5 μL | ||
| Liposoma 2 mol% PI(4)P | PC/PI(4)P 98/2 | 247 μL | 153 μL | |||
| Liposoma PC | PC 100 | 252 μL | ||||
| Ensayos de transporte | LA | PC/PS/Rhod-PE 93/5/2 | 234 μL | 31,4 μL | 200 μL | |
| LA sin PS | PC/Rhod-PE 98/2 | 247 μL | 200 μL | |||
| LB | PC/PI(4)P 95/5 | 237 μL | 383 μL | |||
| LB sin PI(4)P | PC 100 | 252 μL | ||||
| LA-Eq | PC/PS/PI(4)P/Rhod-PE 93/2.5/2.5/2 | 234 μL | 15,7 μL | 200 μL | 191 μL | |
| LB-Eq | PC/PS/PI(4)P 95/2.5/2.5 | 239 μL | 15,7 μL | 191 μL | ||
Tabla 1: Volúmenes de soluciones de stock de lípidos a mezclar para la preparación de liposomas. Abreviaturas: PS= fosfatidilserina; PC = fosfatidilcolina; PI(4)P = fosfatidilinositol 4-fosfato; Rhod-PE = fosfatidilinamino marcada con rodamina; DOPC = dioleoilfosfatidilcolina; POPS= 1-palmitoil-2-oleoil-sn-glicero-3-fosfo-L-serina; 16:0 Liss Rhod-PE = 1,2-dipalmitoil-sn-glicero-3-fosfoetanolamina-N-(lisamina rodamina B sulfonilo).
4. Medición de la extracción de PS o PI(4)P
NOTA: Las mediciones deben realizarse utilizando una placa negra de 96 pomos y un lector de placas de fluorescencia equipado con monocromadores: uno para excitación de fluorescencia y otro para emisión, con un ancho de banda variable.
- Prepare un tampón de HK fresco, filtrado y desgasificación complementado con 1 mM MgCl2 (tampón HKM). Preparar liposomas PC puros y liposomas PC dopados con 2 mol% PS o 2 mol% PI(4)P (concentración lipídica final de 4 mM, ver Tabla 1).
NOTA: Mantenga los tubos llenos con la suspensión de liposomas extruidos a temperatura ambiente durante todo el experimento y mantenga las proteínas en hielo. Además, proteja los sensores de lípidos de la luz. - Para el ensayo de extracción de PS, en un pozo, mezcle liposomas que contengan 2 mol% de PS (concentración de lípidos finales de 80 μM, concentración de PS accesible de 0,8 μM) con NBD-C2Lact (concentración final de 250 nM) en un volumen final de 100 μL. Llene un segundo pozo con la misma cantidad de liposoma (80 μM, 2 mol% PS) y NBD-C2Lact (250 nM) mezclado con 3 μM LTP (Osh6p como control positivo o proteína de interés).
NOTA: Un tiempo de incubación de 5 min es suficiente para que Osh6p logre la extracción de lípidos. - Llene un tercer pozo con NBD-C2Lact (250 nM) mezclado con liposomas PC puros (80 μM). Llene un cuarto pozo solo con liposomas PC puros (80 μM). Repita los pasos 4.2-4.3 para preparar tres series adicionales de cuatro pozos.
- Para cada pozo, registre un espectro NBD de 505 a 650 nm (ancho de banda 5 nm) tras la excitación a 490 nm (ancho de banda 5 nm) a 25 °C. Para cada serie, reste el espectro registrado con solo liposomas de los otros espectros.
NOTA: F y Fmax corresponden a la intensidad a 536 nm medida con liposomas que contienen PS en presencia o ausencia de LTP, respectivamente, mientras que F0 es la intensidad en la misma longitud de onda con liposomas de PC puros. Para cada serie, el porcentaje de PS accesible que se extrae de la proteína se da mediante el uso de la siguiente fórmula.
100 × (1-(F-F0)/(Fmax-F0))) (4) - Para el ensayo de extracción PI(4)P, preparar liposomas dopados con PI(4)P al 2 mol% y realizar mediciones con la sonda NBD-PHFAPP. Realice experimentos de control y determine el porcentaje de extracción de la misma manera que se describió anteriormente.
NOTA: La concentración de liposomas y proteínas es idéntica a las utilizadas en el ensayo de extracción de PS.
5. Medición en tiempo real del transporte PS
NOTA: Se utiliza un fluorímetro estándar (formato de 90°) equipado con un soporte celular de temperatura controlada y un agitador magnético para registrar la cinética de transferencia de lípidos. Para adquirir datos con precisión, es clave mantener permanentemente la muestra a la misma temperatura (establecida entre 25 y 37 ° C dependiendo del origen de la proteína(por ejemplo,levadura o humano)) y agitarla constantemente. El protocolo que se describe a continuación es para la medición del transporte de lípidos en una muestra de 600 μL contenida en una célula cilíndrica de cuarzo.
- Prepare un tampón HKM recién desgastido y filtrado. Mantenga los tubos que contienen liposomas extruidos a temperatura ambiente. Envuelva los tubos que contienen liposomas con Rhod-PE en papel de aluminio y/o guárdelos en una caja opaca para evitar cualquier fotobleaching.
- Ajuste los monocromadores de excitación y emisión a λ = 460 nm (con un ancho de banda corto (1-3 nm)) y a λ = 530 nm (con un ancho de banda grande (≥ 10 nm)), respectivamente. Establezca el tiempo de adquisición en 25 minutos con una resolución de tiempo ≤ 1 s.
- En la cubeta de cuarzo, diluir 30 μL de la suspensión de liposomas LA y un volumen de solución de stock de NBD-C2Lact en tampón HKM precalentado para preparar una muestra de 570 μL que contiene 200 μM de lípidos totales y 250 nM NBD-C2Lact. Agregue una pequeña barra de agitación magnética y coloque la cubeta en el soporte del fluorómetro.
- Una vez que la muestra esté equilibrada térmicamente (después de 3-5 min), active la medición. Después de 1 min, agregue 30 μL de suspensión de liposoma LB (concentración final de 200 μM de lípidos totales) a la muestra. Después de 3 min, inyecte LTP en la muestra para que la concentración final del LTP sea de 200 nM, y adquiera la señal durante los 21 min restantes.
- Realizar un experimento paralelo para normalizar la señal NBD. Mezclar 30 μL de suspensión de liposomas LA-Eq con 250 nM NBD-C2Lact en tampón HKM (volumen final de 570 μL). Después de 1 min, inyecte 30 μL de suspensión de liposomas LB-Eq.
NOTA: La composición lipídica de los liposomas LA-Eq y LB-Eq es similar a la de los liposomas LA y LB utilizados en el ensayo de transferencia, excepto que cada uno de ellos contiene 2.5 mol% PS y 2.5 mol% PI(4)P. Como resultado, la señal NBD que se mide, denominada FEq,corresponde a la señal que debe medirse si PS se equilibra completamente entre los liposomas LA y LB mediante un proceso de transferencia. - Convierta las curvas cinéticas medidas con un LTP de interés para determinar la cantidad de PS (en μM) transferida de los liposomas LA a LB a lo largo del tiempo. Normalice cada punto de datos (F) de la curva mediante la siguiente fórmula.
Norma F = (F-F0)/(FEq-F0) (5)
en el que F0 corresponde a la señal NBD justo antes de la adición de un LTP, y FEq es la señal medida en el paso 5.5.
NOTA: La cantidad de PS (en μM) transferida de los liposomas LA a LB corresponde a 2,5 ×normaF, considerando que el equilibrio corresponde a una situación en la que la mitad de las moléculas de PS accesibles, contenidas en la valva externa de los liposomas LA, (es decir,correspondiente al 5 mol% de 0,5 × 200 μM de lípidos totales) se ha transferido a liposomas LB.
6. Medición en tiempo real del transporte PI(4)P
- Configure el fluorímetro (longitud de onda de excitación y emisión, ancho de banda, tiempo de adquisición, resolución de tiempo) como se hace para el ensayo de transferencia PS. Del mismo modo, use el mismo tampón, cubeta y liposomas para realizar los experimentos bajo agitación constante a la misma temperatura.
- En la cubeta, mezclar 30 μL de suspensión de liposomas LB y NBD-PHFAPP con tampón HKM precalentado para obtener un volumen final de 570 μL (200 μM de lípidos totales, 250 nM NBD- PHFAPP). Una vez alcanzado el equilibrio térmico de la muestra, inicie la medición y, después de 1 min, inyecte 30 μL de suspensión de liposomas LA. Después de 3 min, inyecte el LTP de interés (concentración final de 200 nM) y registre la señal.
- Realice un segundo experimento para normalizar la señal NBD. Mezclar 30 μL de suspensión de liposomas LB-Eq con 250 nM NBD-PHFAPP en 570 μL de tampón HKM. Después de 1 min, inyecte 30 μL de suspensión de liposomas LA-Eq.
NOTA: Aquí, la señal NBD, conocida como FEq,corresponde a la que debe medirse si PI(4)P estuviera completamente equilibrada entre los liposomas LA y LB. - Convierta las curvas cinéticas para determinar la cantidad de PI(4)P (en μM) transferida de los liposomas LB a LA a lo largo del tiempo. Cada punto de datos (F) se normaliza mediante el uso de la fórmula 5 en la que F0 corresponde a la señal NBD antes de la adición de un LTP, y FEq es la señal medida en el paso 6.3.
NOTA: La cantidad de PI(4)P (en μM) transferida de los liposomas LB a LA corresponde a 2,5 ×normaF, considerando que el equilibrio corresponde a una situación en la que la mitad del PI(4)P contenido en la valva externa de los liposomas LB (es decir, 0,5 × 5 μM) se ha transferido en liposomas LA.
7. Análisis de curvas cinéticas
- Cuantificar el grado en que un LTP es eficiente determinando la velocidad a la que este LTP transfiere lípidos de una población de liposomas a la otra en los primeros segundos después de su inyección en la cubeta.
- Realizar una regresión lineal de los primeros puntos de datos de la cinética de transferencia para obtener una pendiente. Divida el valor de pendiente por la concentración de LTP en la mezcla de reacción para determinar el número de moléculas lipídicas transferidas por proteína por unidad de tiempo (min o s).
Resultados
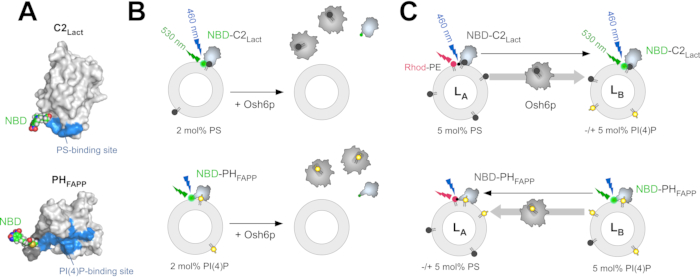
Figura 1: Descripción de los sensores lipídicos fluorescentes y ensayos in vitro. (A) Modelos tridimensionales de NBD-C2Lact y NBD-PHFAPP basados en la estructura cristalina del dominio C2 de la lactadherina bovina (PDB ID: 3BN648) y la estructura de RMN del dominio PH de la proteína ...
Discusión
Los resultados de estos ensayos dependen directamente de las señales de los sensores de lípidos fluorescentes. Por lo tanto, la purificación de estas sondas etiquetadas en una proporción de 1: 1 con NBD y sin contaminación libre de fluoróforos NBD es un paso crítico en este protocolo. También es obligatorio verificar si el LTP bajo examen está correctamente plegado y no agregado. La cantidad de LTP probada en los ensayos de extracción debe ser igual o superior a la de las moléculas accesibles de PS o PI(4)P pa...
Divulgaciones
Los autores declaran que no hay conflictos de intereses.
Agradecimientos
Estamos agradecidos a la Dra. A. Cuttriss por su cuidadosa revisión del manuscrito. Este trabajo está financiado por la beca ExCHANGE de la Agencia Nacional de Investigación de Francia (ANR-16-CE13-0006) y por el CNRS.
Materiales
| Name | Company | Catalog Number | Comments |
| L-cysteine ≥97 % (FG) | Sigma | W326305-100G | Prepare a 10 mM L-cysteine stock solution in water. Aliquots are stored at -20 °C |
| 2 mL Amber Vial, PTFE/Rub Lnr, for lipids storage in CHCL3 | Wheaton | W224681 | |
| 4 mm-diameter glass beads | Sigma | Z265934-1EA | |
| 50 mL conical centrifuge tube | Falcon | ||
| ÄKTA purifier | GE healthcare | FPLC | |
| Aluminium foil | |||
| Amicon Ultra-15 with a MWCO of 3 and 10 kDa | Merck | UFC900324, UFC901024 | |
| Amicon Ultra-4 with a MWCO of 3 and 10 kDa | Merck | UFC800324, UFC801024 | |
| Ampicillin | Prepare a 50 mg/mL stock solution with filtered and sterilized water and store it at -20 °C. | ||
| Bestatin | Sigma | B8385-10mg | |
| BL21 Gold Competent Cells | Agilent | ||
| C16:0 Liss (Rhod-PE) in CHCl3 (1 mg/mL) | Avanti Polar Lipids | 810158C-5MG | |
| C16:0/C16:0-PI(4)P | Echelon Lipids | P-4016-3 | Dissolve 1 mg of C16:0/C16:0-PI(4)P powder in 250 µL of MeOH and 250 µL of CHCl3. Then complete with CHCl3 to 1 mL. The solution must become clear. |
| C16:0/C18:1-PS (POPS) in CHCl3 (10 mg/mL) | Avanti Polar Lipids | 840034C-25mg | |
| C18:1/C18:1-PC (DOPC) in CHCl3 (25 mg/mL) | Avanti Polar Lipids | 850375C-500mg | |
| CaCl2 | Sigma | Prepare 10 mM CaCl2 stock solution in water. | |
| Cell Disruptor | Constant Dynamics | ||
| Chloroform (CHCl3) RPE-ISO | Carlo Erba | 438601 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 5056489001 | |
| Deionized (Milli-Q) water | |||
| Dimethylformamide (DMF), anhydrous, >99% pure | |||
| DNAse I Recombinant, RNAse free, in powder | Roche | 10104159001 | |
| DTT | Euromedex | EU0006-B | Prepare 1 M DTT stock solution in Milli-Q water. Prepare 1 mL aliquots and store them at -20 °C. |
| Econo-Pac chromatography columns (1.5 × 12 cm). | Biorad | 7321010 | |
| Electroporation cuvette 2 mm | Ozyme | EP102 | |
| Electroporator Eppendorf 2510 | Eppendorf | ||
| Fixed-Angle Rotor Ti45 and Ti45 tubes | Beckman | Spinning the batcerial lysates | |
| Glass-syringes (10, 25, and 50 µL) for fluorescence experiment | Hamilton | ||
| Glass-syringes (25 , 100, 250, 500, and 1000 µL) to handle lipid stock solutions | Hamilton | 1702RNR, 1710RNR, 1725RNR, 1750RN type3, 1001RN | |
| Glutathione Sepharose 4B beads | GE Healthcare | 17-0756-05 | |
| Glycerol (99% pure) | Sigma | G5516-500ML | |
| Hemolysis tubes with a cap | |||
| HEPES , >99 % pure | Sigma | H3375-500G | |
| Illustra NAP 10 desalting column | GE healthcare | GE17-0854-02 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Euromedex | EU0008-B | Prepare 1 M IPTG stock solution in Milli-Qwater. Prepare 1 mL aliquots and store them at -20 °C. |
| K-Acetate | Prolabo | 26664.293 | |
| Lennox LB Broth medium without glucose | Prepared with milli-Q water and autoclaved. | ||
| Liquid nitrogen | Linde | ||
| Methanol (MeOH) ≥99.8% | VWR | 20847.24 | |
| MgCl2 | Sigma | Prepare a 2 M MgCl2 solution. Filter the solution using a 0.45 µm filter. | |
| Microplate 96 Well PS F-Botom Black Non-Binding | Greiner Bio-one | 655900 | |
| Mini-Extruder with two 1 mL gas-tight Hamilton syringes | Avanti Polar Lipids | 610023 | |
| Monochromator-based fluorescence plate reader | TECAN | M1000 Pro | |
| N,N'-Dimethyl-N-(Iodoacetyl)-N'-(7-Nitrobenz-2-Oxa-1,3-Diazol-4-yl)Ethylenediamine) (IANBD Amide) | Molecular Probes | Dissolve 25 mg of IANBD in 2.5 mL of dimethylsulfoxide (DMSO) and prepare 25 aliquot of 100 µL in 1.5 mL screw-cap tubes. Do not completely screw the cap. Then, remove DMSO in a freeze-dryer to obtain 1 mg of dry IANBD per tube. Tubes are closed and stored at -20 °C in the dark. | |
| NaCl | Sigma | S3014-1KG | |
| PBS | 137 mM NaCl, 2.7 mM KCl, 10 mM NaH2PO4, 1.8 mM KH2PO4, autoclaved and stored at 4 °C. | ||
| Pear-shaped glass flasks (25 mL, 14/23, Duran glass) | Duran Group | ||
| Pepstatin | Sigma | p5318-25mg | |
| pGEX-C2LACT plasmid | Available on request from our lab | ||
| pGEX-PHFAPP plasmid | Available on request from our lab | ||
| Phenylmethylsulfonyl fluoride (PMSF) ≥98.5% (GC) | Sigma | P7626-25g | Prepare a 200 mM PMSF stock solution in isopropanol |
| Phosphoramidon | Sigma | R7385-10mg | |
| Polycarbonate filters (19 mm in diameter) with pore size of 0.2 µm | Avanti Polar Lipids | 610006 | |
| Poly-Prep chromatography column (with a 0-2 mL bed volume and a 10 mL reservoir) | Biorad | 7311550 | |
| Prefilters (10 mm in diameter). | Avanti Polar Lipids | 610014 | |
| PyMOL | http://pymol.org/ | Construction of the 3D models of the proteins (Figure 1A) | |
| Quartz cuvette for UV/visible fluorescence (minimum volume of 600 µL) | Hellma | ||
| Quartz cuvettes | Hellma | ||
| Refrigerated centrifuge Eppendorf 5427R | Eppendorf | ||
| Rotary evaporator | Buchi | B-100 | |
| Screw-cap microcentriguge tubes (1.5 mL) | Sarsted | ||
| Small magnetic PFTE stirring bar (5 × 2 mm) | |||
| Snap-cap microcentriguge tubes (0.5, 1, and 2 mL) | Eppendorf | ||
| SYPRO orange | fluorescent stain to detect protein in SDS-PAGE gel | ||
| Thermomixer | Starlab | ||
| THROMBIN, FROM HUMAN PLASMA | Sigma | 10602400001 | Dissolve 20 units in 1 mL of milli-Q water and prepare 25 µL aliquots in 0.5 mL Eppendorf tubes. Then freeze and store at -80 °C. |
| Tris, ultra pure | MP | 819623 | |
| Ultracentrifuge L90K | Beckman | ||
| UV/Visible absorbance spectrophotometer | SAFAS | ||
| UV/visible spectrofluorometer with a temperature-controlled cell holder and stirring device | Jasco or Shimadzu | Jasco FP-8300 or Shimadzu RF-5301PC | |
| Vacuum chamber | |||
| Water bath | Julabo | ||
| XK 16/70 column packed with Sephacryl S200HR | GE healthcare |
Referencias
- Drin, G. Topological regulation of lipid balance in cells. Annual Review of Biochemistry. 83, 51-77 (2014).
- Bigay, J., Antonny, B. Curvature, lipid packing, and electrostatics of membrane organelles: defining cellular territories in determining specificity. Developmental Cell. 23 (5), 886-895 (2012).
- Prinz, W. A. Lipid trafficking sans vesicles: where, why, how. Cell. 143 (6), 870-874 (2010).
- Holthuis, J. C., Menon, A. K. Lipid landscapes and pipelines in membrane homeostasis. Nature. 510 (7503), 48-57 (2014).
- Wong, L. H., Copic, A., Levine, T. P. Advances on the transfer of lipids by lipid transfer proteins. Trends in Biochemical Sciences. 42 (7), 516-530 (2017).
- Wong, L. H., Gatta, A. T., Levine, T. P. Lipid transfer proteins: the lipid commute via shuttles, bridges and tubes. Nature Reviews Molecular Cell Biology. 20 (2), 85-101 (2019).
- Iaea, D. B., Dikiy, I., Kiburu, I., Eliezer, D., Maxfield, F. R. STARD4 membrane interactions and sterol binding. Biochemistry. 54 (30), 4623-4636 (2015).
- Wilhelm, L. P., et al. STARD3 mediates endoplasmic reticulum-to-endosome cholesterol transport at membrane contact sites. The EMBO Journal. 36 (10), 1412-1433 (2017).
- Bian, X., Saheki, Y., De Camilli, P. Ca(2+) releases E-Syt1 autoinhibition to couple ER-plasma membrane tethering with lipid transport. The EMBO Journal. 37 (2), 219-234 (2018).
- Horenkamp, F. A., Valverde, D. P., Nunnari, J., Reinisch, K. M. Molecular basis for sterol transport by StART-like lipid transfer domains. The EMBO Journal. 37 (6), 98002 (2018).
- Jentsch, J. A., et al. Structural basis of sterol binding and transport by a yeast StARkin domain. The Journal of Biological Chemistry. 293 (15), 5522-5531 (2018).
- Moser von Filseck, J., et al. INTRACELLULAR TRANSPORT. Phosphatidylserine transport by ORP/Osh proteins is driven by phosphatidylinositol 4-phosphate. Science. 349 (6246), 432-436 (2015).
- Daum, G., et al. Systematic analysis of yeast strains with possible defects in lipid metabolism. Yeast. 15 (7), 601-614 (1999).
- Ejsing, C. S., et al. Global analysis of the yeast lipidome by quantitative shotgun mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2136-2141 (2009).
- Leidl, K., Liebisch, G., Richter, D., Schmitz, G. Mass spectrometric analysis of lipid species of human circulating blood cells. Biochimica et Biophysica Acta. 1781 (10), 655-664 (2008).
- Sampaio, J. L., et al. Membrane lipidome of an epithelial cell line. Proceedings of the National Academy of Sciences of the United States of America. 108 (5), 1903-1907 (2011).
- Vance, J. E., Steenbergen, R. Metabolism and functions of phosphatidylserine. Progress in Lipid Research. 44 (4), 207-234 (2005).
- Zinser, E., et al. Phospholipid synthesis and lipid composition of subcellular membranes in the unicellular eukaryote Saccharomyces cerevisiae. Journal of Bacteriology. 173 (6), 2026-2034 (1991).
- Leventis, P. A., Grinstein, S. The distribution and function of phosphatidylserine in cellular membranes. Annual Review of Biophysics. 39, 407-427 (2010).
- Yeung, T., et al. Membrane phosphatidylserine regulates surface charge and protein localization. Science. 319 (5860), 210-213 (2008).
- Kim, J., Shishido, T., Jiang, X., Aderem, A., McLaughlin, S. Phosphorylation, high ionic strength, and calmodulin reverse the binding of MARCKS to phospholipid vesicles. Journal of Biological Chemistry. 269 (45), 28214-28219 (1994).
- Sigal, C. T., Zhou, W., Buser, C. A., McLaughlin, S., Resh, M. D. Amino-terminal basic residues of Src mediate membrane binding through electrostatic interaction with acidic phospholipids. Proceedings of the National Academy of Sciences. 91 (25), 12253-12257 (1994).
- Gal Bivona, T., et al. PKC regulates a farnesyl-electrostatic switch on K-Ras that promotes its association with Bcl-XL on mitochondria and induces apoptosis. Molecular Cell. 21 (4), 481-493 (2006).
- Finkielstein, C. V., Overduin, M., Capelluto, D. G. Cell migration and signaling specificity is determined by the phosphatidylserine recognition motif of Rac1. The Journal of Biological Chemistry. 281 (37), 27317-27326 (2006).
- Bolsover, S. R., Gomez-Fernandez, J. C., Corbalan-Garcia, S. Role of the Ca2+/Phosphatidylserine Binding Region of the C2 Domain in the Translocation of Protein Kinase Cα to the Plasma Membrane. Journal of Biological Chemistry. 278 (12), 10282-10290 (2003).
- Vance, J. E., Tasseva, G. Formation and function of phosphatidylserine and phosphatidylethanolamine in mammalian cells. Biochimica et Biophysica Acta. 1831 (3), 543-554 (2013).
- Maeda, K., et al. Interactome map uncovers phosphatidylserine transport by oxysterol-binding proteins. Nature. 501 (7466), 257-261 (2013).
- D'Ambrosio, J. M., et al. Osh6 requires Ist2 for localization to ER-PM contacts and efficient phosphatidylserine transport in budding yeast. Journal of Cell Science. 133 (11), 243733 (2020).
- Manford, A. G., Stefan, C. J., Yuan, H. L., Macgurn, J. A., Emr, S. D. ER-to-plasma membrane tethering proteins regulate cell signaling and ER morphology. Developmental Cell. 23 (6), 1129-1140 (2012).
- Collado, J., et al. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- Hoffmann, P. C., et al. Tricalbins contribute to cellular lipid flux and form curved ER-PM contacts that are bridged by rod-shaped structures. Developmental Cell. 51 (4), 488-502 (2019).
- Lipp, N. F., et al. An electrostatic switching mechanism to control the lipid transfer activity of Osh6p. Nature Communications. 10 (1), 3926 (2019).
- Chung, J., et al. INTRACELLULAR TRANSPORT. PI4P/phosphatidylserine countertransport at ORP5- and ORP8-mediated ER-plasma membrane contacts. Science. 349 (6246), 428-432 (2015).
- Sohn, M., et al. PI(4,5)P2 controls plasma membrane PI4P and PS levels via ORP5/8 recruitment to ER-PM contact sites. The Journal of Cell Biology. 217 (5), 1797-1813 (2018).
- Ghai, R., et al. ORP5 and ORP8 bind phosphatidylinositol-4, 5-biphosphate (PtdIns(4,5)P 2) and regulate its level at the plasma membrane. Nature Communications. 8 (1), 757 (2017).
- Sohn, M., et al. Lenz-Majewski mutations in PTDSS1 affect phosphatidylinositol 4-phosphate metabolism at ER-PM and ER-Golgi junctions. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4314-4319 (2016).
- Kattan, W. E., et al. Targeting plasma membrane phosphatidylserine content to inhibit oncogenic KRAS function. Life Science Alliance. 2 (5), 00431 (2019).
- Du, X., Turner, N., Yang, H. The role of oxysterol-binding protein and its related proteins in cancer. Seminars in Cell & Developmental Biology. 81, 149-153 (2018).
- Galmes, R., et al. ORP5/ORP8 localize to endoplasmic reticulum-mitochondria contacts and are involved in mitochondrial function. EMBO reports. 17 (6), 800-810 (2016).
- Du, X., et al. ORP5 localizes to ER-lipid droplet contacts and regulates the level of PI(4)P on lipid droplets. The Journal of Cell Biology. 219 (1), 201905162 (2020).
- Wang, H., et al. ORP2 delivers cholesterol to the plasma membrane in exchange for phosphatidylinositol 4, 5-bisphosphate (PI(4,5)P2). Molecular Cell. 73 (3), 458-473 (2019).
- Kay, J. G., Grinstein, S. Sensing phosphatidylserine in cellular membranes. Sensors (Basel). 11 (2), 1744-1755 (2011).
- Moser von Filseck, J., Vanni, S., Mesmin, B., Antonny, B., Drin, G. A phosphatidylinositol-4-phosphate powered exchange mechanism to create a lipid gradient between membranes. Nature Communications. 6, 6671 (2015).
- Wills, R. C., Goulden, B. D., Hammond, G. R. V. Genetically encoded lipid biosensors. Molecular Biology of the Cell. 29 (13), 1526-1532 (2018).
- Raychaudhuri, S., Im, Y. J., Hurley, J. H., Prinz, W. A. Nonvesicular sterol movement from plasma membrane to ER requires oxysterol-binding protein-related proteins and phosphoinositides. The Journal of Cell Biology. 173 (1), 107-119 (2006).
- Lenoir, M., et al. Structural basis of wedging the Golgi membrane by FAPP pleckstrin homology domains. EMBO reports. 11 (4), 279-284 (2010).
- Liu, Y., Kahn, R. A., Prestegard, J. H. Interaction of Fapp1 with Arf1 and PI4P at a membrane surface: an example of coincidence detection. Structure. 22 (3), 421-430 (2014).
- Shao, C., Novakovic, V. A., Head, J. F., Seaton, B. A., Gilbert, G. E. Crystal structure of lactadherin C2 domain at 1.7A resolution with mutational and computational analyses of its membrane-binding motif. The Journal of Biological Chemistry. 283 (11), 7230-7241 (2008).
- Lipp, N. F., Ikhlef, S., Milanini, J., Drin, G. Lipid exchangers: cellular functions and mechanistic links with phosphoinositide metabolism. Frontiers in Cell and Developmental Biology. 8, 663 (2020).
- Venditti, R., et al. Molecular determinants of ER-Golgi contacts identified through a new FRET-FLIM system. The Journal of Cell Biology. 218 (3), 1055-1065 (2019).
- Pemberton, J. G., et al. Defining the subcellular distribution and metabolic channeling of phosphatidylinositol. The Journal of Cell Biology. 219 (3), (2020).
- Nakanishi, H., de los Santos, P., Neiman, A. M. Positive and negative regulation of a SNARE protein by control of intracellular localization. Molecular Biology of the Cell. 15 (4), 1802-1815 (2004).
- Maekawa, M., Yang, Y., Fairn, G. D. Perfringolysin O theta toxin as a tool to monitor the distribution and inhomogeneity of cholesterol in cellular membranes. Toxins. 8 (3), 67 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados