このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ホスファチジルセリン/ホスファチジルイノシトール4-膜間のリン酸交換の蛍光ベースの測定
要約
ここでは、蛍光脂質センサーおよびリポソームを用いて、タンパク質抽出物を用いてホスファチジルセリンまたはホスファチジルイノシトール4-リン酸 をインビトロで輸送するかを決定するプロトコルについて説明する。
要約
進化的に保存されたオキシステロール結合タンパク質(OSBP)関連タンパク質(ORP)/OSBPホモログ(Osh)ファミリーのいくつかのメンバーは、最近、酵母およびヒト細胞における新しい脂質移動タンパク質(LTP)群を表していることが判明した。ホスファチジルセリン(PS)をPS/ホスファチジルイノシトール4リン酸(PI)交換サイクル を介して 、小胞子(ER)から形質膜(PM)に移します。この知見により、シグナル伝達プロセスに重要なPSがどのように細胞全体に分布しているか、およびこのプロセスとホスホイノシチド(PIP)代謝との間のリンクの調査がより深く理解できる。新しい蛍光ベースのプロトコルの開発は、この新しい細胞機構の発見と特徴付け に 役立っています。本論文では、タンパク質がPSまたはPI(4)Pを抽出し、人工膜間で脂質を移動させる能力を測定するための、蛍光標識された2つの脂質センサNBD-C2ラクト およびNBD-PHFAPPの製造と使用について説明する。まず、プロトコルは、これら2つの構成体の高純度サンプルを製造、ラベル付け、取得する方法を記述する。第2に、これらのセンサーを蛍光マイクロプレートリーダーと併用して、タンパク質がPsまたはPI(4)Pをリポソームから抽出できるかどうかを判断する方法を、Osh6pをケーススタディとして使用する方法を説明する。最後に、このプロトコルは、定義された脂質組成のリポソーム間のPS/PI(4)P交換の運動量を正確に測定し、標準蛍光計を用いた蛍光共鳴エネルギー伝達(FRET)による脂質移動率を測定する方法を示す。
概要
異なる膜間および真核細胞1,2の膜内での脂質の正確な分布は、深い生物学的意味を有する。LTPsの機能の解読は、細胞生物学3、4、5、6、およびinvitroアプローチにおいて重要な問題であり、この問題7、8、9、10、11に対処する上で大きな価値がある。ここでは、細胞膜12間の複数のORP/Oshタンパク質がPS/PI(4)Pに影響を及ぼし、それによって新しいクラスのLTPsを構成することを確立するのに役立ったinvitro、蛍光ベースの戦略が提示される。 ERとPMの間の勾配に沿って分布し、グリセロリン脂質の5〜7%および最大30%をそれぞれ17、18、19に示す。また、PSは、PMの細胞体リーフレットに本質的に濃縮される。この蓄積とPMのPSの不均一なパーティションは、細胞信号プロセス19にとって重要です。PS分子の負電荷のために、PMの細胞体リーフレットは、他のオルガネラ1、2、19、20の細胞体リーフレットよりもはるかにアニオン性である。これにより、静電力を介して、ピリストアレートアラニンを豊富に含むCキナーゼ基質(MARCKS)21、肉腫(Src)22、キルステンラット肉腫ウイルス性腫瘍遺伝子(K-Ras)23、およびRas関連C3ボツリヌス毒素基質1(Rac1)24などのシグナル伝達タンパク質の採用を可能にする。
PSはまた、C2ドメイン25を介して立体選択的な方法で従来のプロテインキナーゼCによって認識される。ただし、PS は ER26で合成され、その役割を果たす前に PM にエクスポートする必要があることを示します。酵母では、Osh6pおよびOsh7pがERからPM27にPSを移すことを知るまで、これがどのように達成されたかは知られていませんでした。これらのLTPは、創設メンバーがOSBPであり、OSBP関連ドメイン(ORD)をポケットに組み込んで脂質分子をホストするタンパク質(ヒトのORP、酵母のオッシュタンパク質)を含む真核生物中の進化的に保存された家族に属しています。Osh6pとOsh7pは、構造特徴がPSを特異的に結合し、膜間で伝達するように適合したORDのみで構成されています。それにもかかわらず、これらのタンパク質がERからPMにPSをどのように方向的に移したかは不明であった。Osh6pとOsh7pは、PI(4)Pを代替脂質リガンド12としてトラップすることができます。酵母では、PI(4)Pはゴルジのホスファチジルイノシトール(PI)とPMからPI-4キナーゼ、Pik1pおよびStt4pによってそれぞれ合成される。対照的に、この脂質はSac1pホスファターゼによってPIに加水分解されるので、ER膜にはPI(4)Pはありません。したがって、PI(4)P 勾配は ER/ゴルギインターフェイスと ER/PM インターフェイスの両方に存在します。Osh6p および Osh7p は、これら 2 つの膜12の間に存在する PI(4)P 勾配を使用して、ER から PM に PS を転送します。
1サイクル内で、Osh6pはERからPSを抽出し、PMでPI(4)PにPSを交換し、PI(4)PをERに戻して別のPS分子を抽出します。Osh6p/Osh7pは、ER膜とPMを互いに近接して接続して近接させる数少ないタンパク質の1つであるIst2p28と相互作用し、酵母29、30、31にER-PMコンタクトサイトを作成する。また、負に帯電した膜とのOsh6pの関連は、その静電機能を変化させる立体構造変化によるタンパク質がその脂質リガンドの1つを抽出するとすぐに弱くなる32。これは、その膜のドウェル時間を短縮することによってOsh6pを助け、それによってその脂質移動活性の効率を維持する。Ist2pへの結合と組み合わせることで、このメカニズムにより、Osh6p/7pがER/PM界面で脂質交換を迅速かつ正確に実行することが可能になります。ヒト細胞では、ORP5およびORP8タンパク質は、ER-PM接触部位でPS/PI(4)P交換を個別のメカニズム33を介して実行する。それらは、Osh6pに似た中央ORDを有するが、C末端膜貫通セグメント33を介してERに直接固定され、PI(4)PおよびPI(4,5)P233、34、35を認識するN末端プレックストリン相同性(PH)ドメインを介してPMにドッキングする。ORP5/8はPI(4)Pを使用してPSを転送し、ORP5/8はさらにPM PI(4,5)P2レベルを調節し、シグナル伝達経路を調節することが示されています。PI(4)P および PI(4,5)P2レベルの低下は、これらのタンパク質が PIP 依存的な方法で PM に関連付けるため、ORP5/ORP8 活性を低下させます。レンツ・マジェフスキ症候群に至る異常に高いPS合成は、ORP5/836を通してPI(4)Pレベルに影響を与える。両方のタンパク質の活性が遮断されると、PSはPMであまり豊富になくなり、シグナル伝達タンパク質37の発癌能力を低下させる。
逆に、ORP5過剰発現は、癌細胞の浸潤と転移過程38を促進する。したがって、ORP5/8活性の変化は、脂質恒常性の変化を通じて細胞行動を著しく変化させることができる。また、ORP5およびORP8は、ER-ミトコンドリア接触部位を占有し、ミトコンドリア機能を保存し、PS39を供給する可能性がある。さらに、ORP5は、ER-脂質液滴接触部位に局地化し、PS/PI(4)P交換40によってPSを脂質滴に送達する。本明細書に記載されている戦略は、リポソームからの(i)PSおよびPI(4)P抽出物および(ii)リポソーム間のPSおよびPI(4)P輸送を測定し、Osh6p/Osh7p12、32のPS/PI(4)P交換活性を確立および分析するために考案され、他のグループがORP5/ORP835および他のLTPの活性を分析するために使用した 41.これは、それぞれPSとPI(4)Pを検出できる蛍光プレートリーダー、標準Lフォーマット分光蛍光計、および2つの蛍光センサ、NBD-C2ラクトとNBD-PHFAPPの使用に基づいています。
NBD-C2ラクトは、推定されるPS結合部位の近くにユニークな溶媒暴露システインを含むように再設計された糖タンパク質、ラクタドヘリンのC2ドメインに対応します。極性感受性のNBD(7-ニトロベンツ-2-オキサ-1,3-ジアゾール)フルオロフォアは、この残基に共有結合している(図1A)12。より正確に言うと、ラクタドヘリンのC2ドメイン(ボストーラス、ユニプロート:Q95114、残留物270-427)を、大腸菌中のグルタチオンS-トランスバラーゼ(GST)と融合して発現させるpGEX-4T3ベクターにクローン化した。C2ラクト配列を突然変異させ、2つの溶媒にアクセス可能なシステイン残基(C270、 C427) アラニン残基を有し、その後N,N'-ジメチル-N-(イオダセチル)-N'-n-(7-ニトロベンツ-2-オキサ-1,3-ジアゾル-4-yl)エチレンジアミン(IANB)で標識することができる推定PS結合部位(H352C突然変異)近くの領域にシステイン残基を導入する血栓の切断部位は、C2ドメインのGSTタンパク質とN末語との間に存在する。主な利点は、このドメインが他の既知のC2ドメインまたはアネキシンA542に反してCa2+独立的な方法でPSを選択的に認識することです。NBD-PHFAPPは、ヒト4リン酸アダプタータンパク質1(FAPP1)のPHドメインに由来し、PI(4)P結合部位(図1A)の近傍にNBD基で標識することができる単一の溶媒暴露システインを含むように再設計された。ヒトFAPPタンパク質のPHドメインの塩基配列(UniProt:Q9HB20、セグメント[1-100])は、GSTタグと連換えて発現されるpGEX-4T3ベクターにクローン化されている。このPHFAPP配列は、タンパク質43の膜結合界面内に特有のシステイン残基を挿入するように改変されている。さらに、プロテアーゼへのアクセス性を確保するために、9残基リンカーが、PHドメインのトロンビン切断部位とN末語との間に導入された。
リポソームからのPS抽出を測定するには、 NBD-C2Lactは、微量のPSを含むホスファチジルコリン(PC)で作られたリポソームと混合され、PSに対する親和性のために、この構造はリポソームに結合し、NBDフルオロフォアは膜の疎水性環境に接触するにつれて極性の変化を経験する。PSが量論的量のLTPによってほぼ完全に抽出された場合、プローブはリポソームと関連せず、NBD信号は低い(図1B)32。この信号の違いは、LTP(例えば、Osh6p)がPSを抽出するかどうかを決定するために使用されます。同様の戦略は、前述の12, 32のように、NBD-PHFAPPと共に PI(4)P 抽出 (図 1B) を測定するために使用されます。2つのFRETベースのアッセイは、(i)それぞれER膜とPMを模倣するLAからLBリポソームへのPS輸送を測定し、(ii)逆方向にPI(4)P輸送を測定するように設計された。これらのアッセイは、PS/PI(4)P交換を測定するために同じ条件(すなわち、同じバッファー、温度、および脂質濃度)で行われます。PS輸送を測定するために、NBD-C2ラクトは、PCで構成されるLAリポソームと混合し、5モル%のPSおよび2モル%の蛍光ローダミン標識ホスファチジルエタノールアミン(Rhod-PE)およびLBリポソームを5モルPI%PI(4)Pを組み込んだ。
時間ゼロで、ロードPEを有するFRETは、NBD蛍光をクエンチする。PSがLAからLBリポソームに輸送される場合(例えば、Osh6pを注入すると)、LAからLBリポソームへのNBD-C2Lact分子の転位に伴い、速い脱健全性が生じる(図1C)。アクセス可能なPSの量を考えると、NBD-C2のラクトは、実験12の過程で本質的に膜結合状態のままである。したがって、NBDシグナルの強度は、LAとLBリポソーム間のNBD-C2ラクトの分布と直接相関し、PSが転送される量を決定するために容易に正規化することができる。PI(4)Pの逆方向の移動を測定するために、NBD-PHFAPPはLAおよびLBリポソームと混合される。PI(4)P を含むが、ロード-PEを含まない LBリポソームにのみ結合することを考えると、その蛍光は高い。PI(4)PがLAリポソームに移された場合、これらのリポソームに転写され、Rhod-PEを用いたFRETにより信号が減少する(図1C)。信号は正規化されて、PI(4)P が転送される量が43 になります。
プロトコル
1. NBD-C2ラクトの精製
注:このプロトコルは、細菌を破壊する細胞破壊器の使用を詳述していますが、他のライシス戦略(例えばフランス のプレス)を使用するように変更することができます。精製の開始時に、システインの酸化を防ぐために、新たに脱気し、濾過し、2 mMジチオトライトール(DTT)を補充したバッファーを使用することが必須です。しかし、タンパク質ラベリングステップでは、DTTを完全に除去することが重要です。タンパク質の分解を避けるために、多くのステップを氷の上または冷たい部屋で行う必要があります。30 μLのサンプルは、15%アクリルアミドゲルを使用して、精製の進捗状況を確認するために、ドデシルスルフェート-ポリアクリルアミドゲル電気泳動(SDS-PAGE)の分析を行うために、プロトコルの異なるステップで収集する必要があります。各アリコートと十分な変性レムリサンプルバッファーを混合し、95°Cで混合物を加熱します。 チューブを凍結し、分析するまで-20 °Cで保管してください。
- 大腸菌におけるGST-C2ラクトの発現
- BL21ゴールドのコンピテントセルを20μL、殺菌水18μLと混合します。次いで、pGEX-C2ラクト プラスミド2μL(約65ng/μL)を細菌と混合し、エレクトロポレーションで変換します。150 μLのオートクレーブレノックスリソジニーブロス(LB)培地(10 g/L トリプトン、5 g/L酵母エキス、脱イオン水中5g/L NaCl、グルコースフリー)で細菌を再懸濁させます。2 mL スナップキャップマイクロ遠心チューブで細菌を1時間37°Cで増殖させます。
- LB培地25mLを接種し、50μg/mLアンピシリンを添加し、125mLの無菌エルレンマイヤーフラスコに150 μLの細菌懸濁液を含む。フラスコを37°Cの軌道シェーカーに入れ、185rpmで攪拌して一晩細菌を成長させます。
- 50 μg/mL アンピシリンを添加したLB培地500mLで2つの無菌2 Lエルレンマイヤーフラスコを充填し、5 mLの培養前懸濁液を加えます。220rpmで撹拌して37°Cで細菌を増殖させます。
- 600nmの波長(λ)で懸濁液の光学密度(OD)を定期的に測定する。ODが〜0.6-0.7の値に達したら、1 Mイソプロピルβ-D-1-チオガラクトピラノシド(IPTG)のストック溶液を500μLずつ各フラスコに加えて、GST-C2ラクトの発現を開始する。フラスコを185rpmで37°Cで4時間振ります。
- 各フラスコの内容物をポリプロピレン遠心分離器瓶に移します。4°Cで4600×gで30分間2本のボトルを遠心し、細菌をペレット化します。上清を捨て、各ペレットを50mLの冷リン酸緩衝食塩の中に再懸濁する。
- 各ボトルに含まれる細菌懸濁液を50 mL円錐形遠心分離チューブに移します。2本のチューブを4°Cで2300×gで30分間遠心分離します。上清を取り除き、細菌ペレットを含むチューブを-20°Cに保存します。
- C2の精製ラクト
- 氷上で、50 mM Tris-HCl、pH 7.4、および150 mM NaCl(以下、TNバッファと呼ばれる)を含む50 mLの緩衝管を2本50 mL充填し、膜真空濾過によって濾過および脱気する。
- 各チューブに溶解バッファーを調製するために、エチレンジアミンテトラ酢酸フリープロテアーゼ阻害剤カクテルの錠剤を軽い超音波処理または渦によってTNバッファーに溶解する。その他の抗プロテアーゼ(10 μMのベストタチン、1 μg/mL ペプスタチンA、10 μMリンカミドン)を加えます。重要なことに、2 mM DTT でバッファーを補完します。
- ステップ1.1.6で調製した細菌ペレットを含む2つのチューブを、各チューブに30mLの最終体積を得るためにリシスバッファーで充填し、氷上のペレットを10分間ゆっくりと解凍します。各ペレットをステンレス製のヘラで粉砕し、チューブをボルテックスし、ピペットコントローラと25 mLピペットで懸濁液を前後にピペット化して、均質な懸濁液が得られるまで再懸濁します。
- 予備冷却セルの破壊器( 材料表を参照)を使用して、サンプルの30 mLをリザーバ内にロードし、1.6バールの圧力で連続モードで破断サイクルを実行して、ライシスを実行します。同じチューブに溶解液を集め、チューブを氷上に保ち、イソプロパノールで調製した200 mMフェニルメチルスルホニルフッ化物(PMSF)のストック溶液から250 μLをすぐに添加します。
- 同じ手順に従って他のサンプルを lyse します。.リシスバッファーの残りの部分を使用して細胞破壊剤を洗浄し、洗浄液の体積(〜30 mL)を50mLの最終体積に調整します。
- 5 mM MgCl2で各リセートを補い、DNAを断片化し、サンプルの粘度を下げるためにDNAse Iの20 μg/mLを加えます。氷の上で30分間インキュベートします。ゲル分析用のサンプルを収集します。
- 各50 mLのリゼートをプレチルドポリカーボネート超遠心チューブに移す(合計2個、材料表を参照)。186,000×gで、超遠心分離機を使用して少なくとも1時間4°Cで遠心分離機。
- 遠心分離工程と並行して、グルタチオンを含むスラリー1.4mLを2つの50 mGaroseビーズと結合して2つの50 mL円錐遠心管に分配し、各チューブに1 mM DTT(TNDバッファー)を加えたTNバッファー20 mLを加え、1200gの遠心分離器を500gで 500g× この洗浄手順を2回繰り返します。
- 細菌のリセートの遠心分離後、上清から30μLのサンプルを取り出し、各超遠心チューブから、きれいなビーズを含む対応する50 mL円錐形遠心分離管に上清を移します。ゲル分析のために、TNDバッファーの50 mLの超遠心分離管の1つの破片ペレットを再懸濁し、30 μLサンプルを集めます。
- 4°Cで3〜4時間回転器にチューブを置き、均質なビーズ懸濁液を得る。ビード懸濁液を空の25 mLクロマトグラフィーカラムにプールします。ビーズをデカントさせ、重力流によって緩衝液および結合されていないタンパク質を除去する。分析のために溶出したサンプルを取ります。
- 20 mLのTNDバッファでビーズを再懸濁し、重力流によって溶出物を回収します。このステップを2回繰り返してビーズを完全に洗います。収集した溶出液をプールし、さらに分析するために30 μLサンプルを保持します。
注:短いデカンテーションの後、GST-C2Lact が取り付けられているビーズサスペンションの〜2 mLの体積は、列の下部に堆積物。 - 2つの2つのmLのスナップキャップのマイクロ遠心分離管に1 mLのビーズ懸濁液を加える。各チューブにTNDバッファーを充填し、最終体積1.970mLにします。さらに分析するために、1本のチューブから30μLのサンプルを取ります(B1サンプル)。0.02 U/μLでヒトトロンビンプロテアーゼ溶液のストック溶液から10 mMCaCl2 溶液と25μLの10 μLを加えます。
- 2本のチューブを一晩4°Cのローテーターに置き、トロンビンがC2ラクト ドメインからGSTタグを切断できるようにします。翌日、各チューブに、200 mM PMSF溶液の10 μLをビーズ懸濁液と混合して、トロンビン作用を抑制します。
- 700×gでチューブを5分間遠心し、ビーズを取らずに各チューブから可溶性C2ラットドメインを含む上清を回収する。上清を氷上に保つ2 mLスナップキャップマイクロ遠心チューブ(E1溶出液)にプールします。
- 各チューブに1 mLのTNDバッファを加え、ビーズを再停止し、洗浄します。ステップ 1.2.14 を繰り返します。最大量のタンパク質を回収するために、このステップをさらに3回実行する。毎回、収集した上清を新しい2mLチューブ(E2、E3、E4、E5溶出液)にプールし、さらなる分析のためにアリコートを取る。洗浄工程の終わりに、ビーズ懸濁液(アリコートB2)からアリコートを取る。
- 15%アクリルアミドゲル上のSDS-PAGE分離により精製プロトコルの異なるステップで収集した30μLサンプルを分析します。
- ステップ1.2.14と1.2.15の間に収集されたすべての上清(〜10 mL)を10 mLクロマトグラフィーカラムにプールして、潜在的な汚染ビーズを除去します。重力流で溶出物を集め、カラムの底部にビーズを保持します。
- C2Lactサンプルは、分子量カットオフ(MWCO)3 kDaの遠心フィルタユニットと2300×gの遠心分離速度を使用して濃縮します。 タンパク質試料の体積が〜1mLの場合は、濃縮手順を停止します。
- NBD-C2の調製と精製ラクト
- TN バッファーで脱塩カラム ( 材料表を参照) を平衡化します。濃縮C2ラクト サンプルの1 mLでカラムをロードします。サンプルがゲルベッドに完全に入るようにし、1.5mLの脱気剤を取り出したDTTフリーTNバッファーをカラムに加え、重力流で2mLのスナップキャップマイクロ遠心チューブに溶出物を回収します。
- 300 μL TN バッファーの最終体積で 50 μL の溶出液を希釈し、純TN バッファをブランクとして使用して 230 ~ 450 nm の吸光度スペクトルを記録します。280nmで測定した吸光度に基づいてC2ラクト 濃度を決定し、44,920 M-1.cm-1に等しいεの消光係数を考慮する。
- C2ラクトコンストラクトにNBDフルオロフォアを標識するには、タンパク質をN,N'-ジメチル-N-(イオダセチル)-N'(7-ニトロベンツ-2-オキサ-1,3-ジアゾル-4-イル)エチレンジアミン(IANBDe)と混合します。
- IANBDの1mgを無水ジメチルホルムアミド(DMF)に溶解し、C2Lact 構築物の標識に使用されるDMFの最終容積は、タンパク質サンプル量の5%(v/v)を超えてはなりません。
- IANBDを溶解するDMF(V DMF)の体積を決定するために、まず、式 1を用いてタンパク質を標識するために必要量のIANBD(m、mgで表される)を計算する。
m = 10,000 × C × V × MWIANBD (1)
ここでCはステップ1.3.2で測定したC2ラクト の濃度、VはC2ラクト サンプルの体積、MWIANBD はフルオロフォアの分子量(420g/mol)です。 - 式2 (m0= 1) と3を使用して VDMFを計算します。
VDMF= (m0/m) × VIANBD (2)
VIANBD=0.05 × V (3)
ここでm0 は、MGのIANBD粉末の量であり、VIANBD は、C2のラクト サンプルに添加されるIANBD溶液の体積である。 - C2Lactサンプルに作りたてのIANBD溶液のボリュームVIANBDを加え、光から保護されたサーモミキサーを使用して25°Cで30分間800-900 rpmで反応混合物を振ります。氷の上で90分間反応を進めます。その間に、10 mLのTNバッファで遠心フィルタユニット(MWCO= 3 kDa)をクリーニングします。
- 反応混合物にL-システイン(IANBDに10倍のモル過剰)を加えて、遊離IANBDを不活性化する。
- NBD-C2Lact溶液に15mLのTNバッファーを加え、遠心フィルターユニットにNBD-C2ラクト溶液を移します。サンプルを2mLに濃縮して、2300×gで遠心分離によってタンパク質からほとんどの遊離NBDを分離する。この洗浄手順を2回繰り返します。サンプルを4°Cで19,000×gで10分間2mLのスナップキャップ遠心管に入れてペレット電位凝集体を回収し、上清を回収します。
- 300 μL の最終体積の TN バッファーに50 μLの溶出液を希釈します。濃度手順の間に集めた溶離物をブランクとして使用して、230〜650nmの吸光度スペクトルを記録します。λ=280および495 nmの最大吸光度と、ε=44,920 M-1.cm-1(タンパク質)および25,000 M-1.cm-1(NBDフルオロフォア)で最大吸光度を使用してNBD-C2ラクト濃度を決定します。
注: 2 つの濃度値が同じ場合、これは C2Lact コンストラクトが NBD グループとの比率を 1:1 でラベル付けすることを示します。 - NBD吸光度の測定から推定されるNBD-C2ラクト 濃度がトリプトファン(Trp)残基の吸光度から推定される濃度を超える場合、ステップ1.3.5を繰り返して遊離NBDをさらに除去する。
- グリセロールをサンプルに加える場合は、フラッシュ冷凍中にNBD-C2のラクコン ストラクトを凍結保護するために、10%(v/v)の最終濃度を得る。最終的なタンパク質濃度を測定します。
- 0.5 mL スナップキャップマイクロ遠心チューブでタンパク質の50 μLアリコートを調製します。チューブを液体窒素でフラッシュフリーズし、-80°Cで保存します。
2. NBD-PHFAPPの精製
注:PHFAPPを製造し、ラベル付けする手順は、ステップ1.3.4で遠心フィルタユニットにNBD-C2Lact溶液を転送するまで、NBD-C2Lactのそれと同じです。このステップ以降、以下に説明するプロトコルに従ってください。
- 濃度ステップの後、サイズ排除クロマトグラフィーを行う前に、2 mLのNBD-PHFAPPを暗い状態で4°Cに保ちます。サイズ除外クロマトグラフィーの前に、チューブの下部にオレンジ色の沈着物(濃縮時の凝集)がないことを確認してください。この場合は、サンプルを4°Cで10分間540,000×gで遠心し、サイズ排除クロマトグラフィーで上清を精製します。
注:サイズ排除クロマトグラフィーは、高速タンパク質液体クロマトグラフィーシステムを使用して、TNDバッファーと平衡化された架橋されたデキストランアクリルアミド共重合体( 材料表を参照)を詰めたカラムに対して行われます( 材料表を参照)。列は光から保護する必要があります。流速は1mL/minを用い、NBD-PHFAPP 構築物の溶出は、カラム出口でλ=280(タンパク質)および480nm(NBD)で吸光度を記録した。- 2 mL インジェクション ループにロードされた NBD-PHFAPP サンプルをカラムに注入し、すぐに 2.5 mL の溶出部分を収集します。
- 15%SDS-PAGEゲル上で280および480 nmで検出された主ピークに対応するすべての画分を分析します。ゲルに加熱してローディングする前に、各分画の25 μLサンプルを15 μLのレムリサンプルバッファーと混合します。
注: λ= 280 と 480 nm で同時に検出されるメインピークは、カラムを通して~150 mL のバッファが通過すると現れます。 - NBD-PHFAPPタンパク質(〜12.2kDa)のみを含む分画をプールし、グリセロールを10%(v/v)の最終濃度で加えます。MWCO 3 kDaの遠心フィルターユニットを使用してサンプルを1mLの最終体積に濃縮し、遠心速度2300×gを使用します。
- アリコートを準備し、NBD-C2のラクトに説明されているように吸光度スペクトルを記録する。絶滅係数ε= 29,450 M-1.cm-1 を使用して、λ=280 nmで測定された吸光度に基づいてタンパク質の濃度を決定します。
3. PSおよびPI(4)P抽出または転写アッセイのためのリポソームの調製
注: 特に指定がない限り、室温ですべての手順を実行します。有機溶剤、ロタ蒸気、液体窒素を慎重に取り扱います。
- 新鮮な、濾過済み、脱ガス50 mM 4-(2-ヒドロキセチル)-1-ピペラジネタンスルホン酸(HEPES)-水酸化カリウム(KOH)、pH 7.4、120 mM酢酸カリウム(HK)バッファーを準備します。
- リポソームの種類ごとに、ストック溶液から異なる脂質の正確な量を取り、それらを25mL梨形ガラスフラスコに混ぜる(表1)。純粋なクロロホルムを加え、各混合物の体積を1mLに調整します。各フラスコにリポソームの名前を付けます。ロード-PEをドープした脂質混合物を含むフラスコをアルミニウム箔で包みます。
- フラスコをロータリーエバポレーターに置きます。脂質を真空下で25°Cで少なくとも30分間、回転速度500rpmで乾燥させます。PI(4)Pを含む脂質フィルムの場合、フラスコを32-34 °Cで5分間前温め、穏やかな回転下で、ピ(4)Pを他の脂質と適切に混合してから、フラスコに真空を作り出して溶媒を除去し、フラスコ壁に乾燥した脂質のフィルムを残します。
- フラスコを蒸発器から外し、45分間真空チャンバーに入れ、残りの微量の溶剤を除去します。フラスコに2 mLのHKバッファーを充填し、直径4mmのガラスビーズを溶液に数個加えます。フラスコを2分間穏やかに渦を流し、脂質を再懸濁し、4mMの脂質濃度でマルチラメラ脂質小胞(MLV)を調製する。1.5 mLスクリューキャップマイクロ遠心チューブで0.5 mLのMLVのアリコートを準備します。
- チューブを凍結解凍して5倍(液体窒素と水浴をそれぞれ37°Cで使用)。リポソームを押し出すか、-20°Cで保管してください。
- メーカーのガイドラインに従って、ミニ押出機を使用して、MLVからリポソーム(すなわち、 大きなユニラメラ小胞)を準備します。直径200nmの均一な円筒状の細孔を持つポリカーボネートフィルターを使用してください。
- 各タイプのリポソームを準備するには、対応するMLVの懸濁液の少なくとも250 μLを押し出す。2日以内にリポソームを使用してください。
| 脂質組成物(モル/モル) | 脂質 | |||||
| リポソーム名 | ドPC (25 mg/mL) | ポップ (10 mg/mL) | 16:0 リス・ロード=PE (1 mg/mL) | C16:0/C16:0-PI(4)P (1 mg/mL) | ||
| 抽出アッセイ | リポソーム 2 モル% PS | PC/PS 98/2 | 247 μL | 12.5 μL | ||
| リポソーム 2 モル% PI(4)P | PC/PI(4)P 98/2 | 247 μL | 153 μL | |||
| PCリポソーム | PC 100 | 252 μL | ||||
| 輸送アッセイ | LA | PC/PS/ロードPE 93/5/2 | 234 μL | 31.4 μL | 200 μL | |
| PsなしのL A | PC/ロードPE 98/2 | 247 μL | 200 μL | |||
| LB | PC/PI(4)P 95/5 | 237 μL | 383 μL | |||
| PI(4)P を使用しない LB | PC 100 | 252 μL | ||||
| LA-Eq | PC/PS/PI(4)P/ロードPE 93/2.5/2.5/2 | 234 μL | 15.7 μL | 200 μL | 191 μL | |
| LB-Eq | PC/PS/PI(4)P 95/2.5/2.5 | 239 μL | 15.7 μL | 191 μL | ||
表1:リポソーム製剤用に混合する脂質ストック溶液の体積。 略語: PS= ホスファチジルセリン;PC = ホスファチジルコリン;PI(4)P = ホスファチジルイノシトール 4-リン酸;ローダPE =ローダミン標識ホスファチジルエタノールアミン;DOPC = ジオレオイルホスファチジルコリン;POPS= 1-パルミトイル-2-オレオール-snグリセロ-3-ホスホ-L-セリン;16:0 リス・ロード-PE = 1,2-ジパルミトイル-sn-グリセロ-3-ホスホエタノールアミン-N-(リサミンロダミンBスルホニル).
4. PSまたはPI(4)P抽出の測定
注:測定は、黒色の96ウェルプレートとモノクローマーを装備した蛍光プレートリーダーを使用して行う必要があります:蛍光励起用と放出用、可変帯域幅を備えた1つ。
- 1 mM MgCl2 (HKM バッファー) を補充した、新鮮な、濾過された、脱ガス HK バッファーを準備します。純粋なPCリポソームとPCリポソームを2モル%PSまたは2モル%PI(4)P(4 mM最終脂質濃度、 表1参照)でドープした準備を行います。
注:実験を通して、押し出されたリポソームの懸濁液を室温で満たし、タンパク質を氷の上に置いてください。また、光から脂質センサーを保護します。 - PS抽出アッセイの場合、1つのウェルで、2モル%PS(80 μM最終脂質濃度)を含むリポソームを混合し、 0.8 μMアクセス可能PS濃度)、NBD-C2ラクト (250 nM最終濃度)を最終体積100μLで、同量のリポソーム(80μM、2モルPS)とNBD-C2ラクト (250 nM)を3μM LTP(正の対照または関心タンパク質としてオッシュ)混合して2番目のウェルを充填します。
注:5分のインキュベーション時間は、脂質抽出を達成するためにOsh6pのために十分です。 - 純粋なPCリポソーム(80 μM)と混合されたNBD-C2の授乳 (250 nM)で第3の井戸を満たします。純粋なPCリポソーム(80 μM)のみで第4の井戸を充填してください。ステップ 4.2 から 4.3 を繰り返して、3 つの追加の 4 つのウェルを準備します。
- 各ウェルについて、25 °Cで490 nm(帯域幅5 nm)で励起する場合は、505~650 nm(帯域幅5nm)のNBDスペクトルを記録します。 各系列について、他のスペクトルからリポソームのみで記録されたスペクトルを差し引きます。
注:FおよびFmax は、それぞれLTPの有無においてPS含有リポソームで測定された536nmの強度に対応し、F0 は純粋なPCリポソームと同じ波長の強度です。各系列について、タンパク質によって抽出されるアクセス可能なPSの割合は、次の式を用いて与えられる。
100 × (1-(F-F0)/(F最大-F0))(4) - PI(4)P抽出アッセイでは、2モル%PI(4)Pでドープしたリポソームを調製し、NBD-PHFAPP プローブで測定を行います。制御実験を行い、上記と同様に抽出率を決定する。
注:リポソームおよびタンパク質濃度はPS抽出アッセイで使用されるものと同一である。
5. PS輸送のリアルタイム測定
注:温度制御セルホルダーと磁気スターラーを装備した標準蛍光計(90°形式)は、脂質移動動態を記録するために使用されます。データを正確に取得するには、サンプルを同じ温度(タンパク質の起源(例えば、酵母またはヒト)に応じて25〜37°Cの間で設定)で永久に維持し、常に攪拌することが重要です。以下に説明するプロトコルは、円筒形の石英細胞に含まれる600μLサンプルにおける脂質輸送の測定に関するものである。
- 脱気し、濾過されたHKMバッファを準備します。押し出しリポソームを含むチューブを室温に保ちます。RD-PEを含むリポソームを含むチューブをアルミホイルに包み、不透明な箱に入れてフォトブリーチを防ぎます。
- λ = 460 nm(短い帯域幅(1-3 nm))、λ = 530 nm(大帯域幅(≥10 nm))で励起および放出モノクローターをそれぞれ調整します。取得時間を 25 分に設定し、時間分解能≤ 1 s にします。
- クオーツキュベットでは、LA リポソーム懸濁液の30μLとNBD-C2Lact ストック溶液の体積を事前温めHKMバッファーに希釈し、200 μM全脂質と250 μM NBD-C2ラクトを含む570 μLサンプルを調製した。小さな磁気撹拌バーを追加し、フルオロメーターホルダーにキュベットを配置します。
- サンプルが熱的に平衡化されたら(3-5分後)、測定を開始します。1分後、サンプルにLB リポソーム懸濁液(最終濃度200μM総脂質)を30μL加えます。3分後、LTPの最終濃度が200nMとなるようにサンプルにLTPを注入し、残りの21分間の信号を取得します。
- 並列実験を行い、NBD信号を正規化します。HKMバッファーに250 nM NBD-C2ラクト(最終体積570 μL)とLA-Eqリポソーム懸濁液の30 μLを混合します。1分後、LB-Eqリポソーム懸濁液を30μL注入します。
注:LA-Eq およびLB-Eq リポソームの脂質組成は、転写アッセイで使用されるLA およびLB リポソームの脂質組成と類似していますが、それぞれが2.5モル%PSおよび2.5モル%PI(4)Pを含んでいる点を除いて。その結果、測定されるNBD信号は、FEqと呼ばれ、PSが転写プロセスによってLA とLB リポソームの間で完全に平衡化された場合に測定されるべき信号に対応する。 - 目的のLTPで測定された運動曲線を変換して、時間の経過とともにLA からLB リポソームに転送されるPS(μM単位)の量を決定します。次の数式を使用して、曲線の各データポイント (F) を正規化します。
Fノルム = (F-F0)/(FEq-F0)(5)
F0 はLTPの追加直前のNBD信号に対応し、FEq はステップ5.5で測定された信号です。
注:LAからLBリポソームに転送されるPS(μM)の量は、LAリポソームの外葉に含まれるアクセス可能なPS分子の半分がLlipsomeに移された状況に対応することを考慮して、2.5×Fノルムに相当する(すなわち、0.5×200μMの総脂質の5モル%に相当する)
6. PI(4)P輸送のリアルタイム測定
- PS転写アッセイに対して行われる蛍光計(励起波長と発光波長、帯域幅、取得時間、時間分解能)を設定します。同様に、同じバッファー、キュベット、およびリポソームを使用して、同じ温度で一定撹拌下で実験を行う。
- キュベットでは、LB リポソーム懸濁液の30 μLと、プリウォームHKMバッファを使用してNBD-PHFAPP を混合し、最終容積570 μL(200 μM総脂質、250 μM NBD-PHFAPP)を得る。サンプルの熱平衡に達したら、測定を開始し、1分後に30 μLのLA リポソーム懸濁液を注入します。3分後、目的のLTP(最終濃度200nM)を注入し、信号を記録します。
- NBD信号を正規化するために2回目の実験を行います。LB-Eq リポソーム懸濁液の30 μLを、HKMバッファの570 μLに250 nM NBD-PHFAPP と混合します。1分後、LA-Eq リポソーム懸濁液を30μL注入します。
注: ここで、NBD 信号は FEqと呼ばれ、PI(4)P が LA と LB リポソームの間で完全に平衡化された場合に測定されるべきシグナルに対応します。 - 運動曲線を変換して、時間の経過に応じてLBからLAリポソームに転送されるPI(4)P(μM単位)の量を決定します。各データ点(F)は、F0がLTPの添加前にNBD信号に対応する式5を用いて正規化され、FEqはステップ6.3で測定された信号である。
注:LBからLAリポソームに転がしたPI(4)P(μM)の量は、LBリポソームの外リーフレットに含まれるPI(4)Pの半分(すなわち、0.5×5μM)がLoリポソームで転化した状況に対応することを考慮して、2.5×Fノルムに相当する。
7. 運動曲線の解析
- このLTPがキュヴェットへの注入後の最初の数秒で脂質を一つのリポソーム集団からもう一方のリポソーム集団に移す速度を決定することによって、LTPがどの程度効率的であるかを定量化する。
- 移動動力学の第1のデータ点の線形回帰を行い、傾きを得る。反応混合物中のLTP濃度で傾きの値を割り、時間単位当たりのタンパク質当たりの脂質分子の数(分またはs)を決定する。
結果
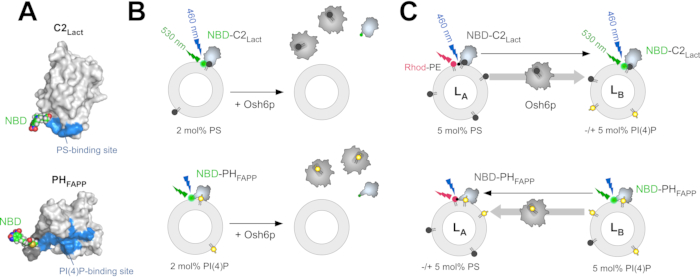
図1: 蛍光脂質センサとインビトロアッセイの説明(A) ウシラクタジンのC2ドメインの結晶構造に基づくNBD-C2ラクトおよびNBD-PHFAPPの3次元モデル(PDB ID: 3BN648)およびヒトFAPP1タンパク質のPHドメインのNMR構造(PDB ID:2KJ46)
ディスカッション
これらのアッセイの結果は、蛍光脂質センサーのシグナルに直接依存します。したがって、これらのプローブの精製は、NBDと1:1の比率で、かつ無料のNBDフルオロフォア汚染なしに標識され、このプロトコルの重要なステップです。また、検査中のLTPが適切に折り畳まれ、集計されていないかどうかを確認することも必須です。抽出アッセイで試験したLTPの量は、このLTPがこれらの脂質を効率?...
開示事項
著者らは、利益相反はないと宣言している。
謝辞
原稿を慎重に校正してくれたA.カットトリス博士に感謝しています。この研究は、フランス国家研究庁の助成金ExCHANGE(ANR-16-CE13-0006)とCNRSによって資金提供されています。
資料
| Name | Company | Catalog Number | Comments |
| L-cysteine ≥97 % (FG) | Sigma | W326305-100G | Prepare a 10 mM L-cysteine stock solution in water. Aliquots are stored at -20 °C |
| 2 mL Amber Vial, PTFE/Rub Lnr, for lipids storage in CHCL3 | Wheaton | W224681 | |
| 4 mm-diameter glass beads | Sigma | Z265934-1EA | |
| 50 mL conical centrifuge tube | Falcon | ||
| ÄKTA purifier | GE healthcare | FPLC | |
| Aluminium foil | |||
| Amicon Ultra-15 with a MWCO of 3 and 10 kDa | Merck | UFC900324, UFC901024 | |
| Amicon Ultra-4 with a MWCO of 3 and 10 kDa | Merck | UFC800324, UFC801024 | |
| Ampicillin | Prepare a 50 mg/mL stock solution with filtered and sterilized water and store it at -20 °C. | ||
| Bestatin | Sigma | B8385-10mg | |
| BL21 Gold Competent Cells | Agilent | ||
| C16:0 Liss (Rhod-PE) in CHCl3 (1 mg/mL) | Avanti Polar Lipids | 810158C-5MG | |
| C16:0/C16:0-PI(4)P | Echelon Lipids | P-4016-3 | Dissolve 1 mg of C16:0/C16:0-PI(4)P powder in 250 µL of MeOH and 250 µL of CHCl3. Then complete with CHCl3 to 1 mL. The solution must become clear. |
| C16:0/C18:1-PS (POPS) in CHCl3 (10 mg/mL) | Avanti Polar Lipids | 840034C-25mg | |
| C18:1/C18:1-PC (DOPC) in CHCl3 (25 mg/mL) | Avanti Polar Lipids | 850375C-500mg | |
| CaCl2 | Sigma | Prepare 10 mM CaCl2 stock solution in water. | |
| Cell Disruptor | Constant Dynamics | ||
| Chloroform (CHCl3) RPE-ISO | Carlo Erba | 438601 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 5056489001 | |
| Deionized (Milli-Q) water | |||
| Dimethylformamide (DMF), anhydrous, >99% pure | |||
| DNAse I Recombinant, RNAse free, in powder | Roche | 10104159001 | |
| DTT | Euromedex | EU0006-B | Prepare 1 M DTT stock solution in Milli-Q water. Prepare 1 mL aliquots and store them at -20 °C. |
| Econo-Pac chromatography columns (1.5 × 12 cm). | Biorad | 7321010 | |
| Electroporation cuvette 2 mm | Ozyme | EP102 | |
| Electroporator Eppendorf 2510 | Eppendorf | ||
| Fixed-Angle Rotor Ti45 and Ti45 tubes | Beckman | Spinning the batcerial lysates | |
| Glass-syringes (10, 25, and 50 µL) for fluorescence experiment | Hamilton | ||
| Glass-syringes (25 , 100, 250, 500, and 1000 µL) to handle lipid stock solutions | Hamilton | 1702RNR, 1710RNR, 1725RNR, 1750RN type3, 1001RN | |
| Glutathione Sepharose 4B beads | GE Healthcare | 17-0756-05 | |
| Glycerol (99% pure) | Sigma | G5516-500ML | |
| Hemolysis tubes with a cap | |||
| HEPES , >99 % pure | Sigma | H3375-500G | |
| Illustra NAP 10 desalting column | GE healthcare | GE17-0854-02 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Euromedex | EU0008-B | Prepare 1 M IPTG stock solution in Milli-Qwater. Prepare 1 mL aliquots and store them at -20 °C. |
| K-Acetate | Prolabo | 26664.293 | |
| Lennox LB Broth medium without glucose | Prepared with milli-Q water and autoclaved. | ||
| Liquid nitrogen | Linde | ||
| Methanol (MeOH) ≥99.8% | VWR | 20847.24 | |
| MgCl2 | Sigma | Prepare a 2 M MgCl2 solution. Filter the solution using a 0.45 µm filter. | |
| Microplate 96 Well PS F-Botom Black Non-Binding | Greiner Bio-one | 655900 | |
| Mini-Extruder with two 1 mL gas-tight Hamilton syringes | Avanti Polar Lipids | 610023 | |
| Monochromator-based fluorescence plate reader | TECAN | M1000 Pro | |
| N,N'-Dimethyl-N-(Iodoacetyl)-N'-(7-Nitrobenz-2-Oxa-1,3-Diazol-4-yl)Ethylenediamine) (IANBD Amide) | Molecular Probes | Dissolve 25 mg of IANBD in 2.5 mL of dimethylsulfoxide (DMSO) and prepare 25 aliquot of 100 µL in 1.5 mL screw-cap tubes. Do not completely screw the cap. Then, remove DMSO in a freeze-dryer to obtain 1 mg of dry IANBD per tube. Tubes are closed and stored at -20 °C in the dark. | |
| NaCl | Sigma | S3014-1KG | |
| PBS | 137 mM NaCl, 2.7 mM KCl, 10 mM NaH2PO4, 1.8 mM KH2PO4, autoclaved and stored at 4 °C. | ||
| Pear-shaped glass flasks (25 mL, 14/23, Duran glass) | Duran Group | ||
| Pepstatin | Sigma | p5318-25mg | |
| pGEX-C2LACT plasmid | Available on request from our lab | ||
| pGEX-PHFAPP plasmid | Available on request from our lab | ||
| Phenylmethylsulfonyl fluoride (PMSF) ≥98.5% (GC) | Sigma | P7626-25g | Prepare a 200 mM PMSF stock solution in isopropanol |
| Phosphoramidon | Sigma | R7385-10mg | |
| Polycarbonate filters (19 mm in diameter) with pore size of 0.2 µm | Avanti Polar Lipids | 610006 | |
| Poly-Prep chromatography column (with a 0-2 mL bed volume and a 10 mL reservoir) | Biorad | 7311550 | |
| Prefilters (10 mm in diameter). | Avanti Polar Lipids | 610014 | |
| PyMOL | http://pymol.org/ | Construction of the 3D models of the proteins (Figure 1A) | |
| Quartz cuvette for UV/visible fluorescence (minimum volume of 600 µL) | Hellma | ||
| Quartz cuvettes | Hellma | ||
| Refrigerated centrifuge Eppendorf 5427R | Eppendorf | ||
| Rotary evaporator | Buchi | B-100 | |
| Screw-cap microcentriguge tubes (1.5 mL) | Sarsted | ||
| Small magnetic PFTE stirring bar (5 × 2 mm) | |||
| Snap-cap microcentriguge tubes (0.5, 1, and 2 mL) | Eppendorf | ||
| SYPRO orange | fluorescent stain to detect protein in SDS-PAGE gel | ||
| Thermomixer | Starlab | ||
| THROMBIN, FROM HUMAN PLASMA | Sigma | 10602400001 | Dissolve 20 units in 1 mL of milli-Q water and prepare 25 µL aliquots in 0.5 mL Eppendorf tubes. Then freeze and store at -80 °C. |
| Tris, ultra pure | MP | 819623 | |
| Ultracentrifuge L90K | Beckman | ||
| UV/Visible absorbance spectrophotometer | SAFAS | ||
| UV/visible spectrofluorometer with a temperature-controlled cell holder and stirring device | Jasco or Shimadzu | Jasco FP-8300 or Shimadzu RF-5301PC | |
| Vacuum chamber | |||
| Water bath | Julabo | ||
| XK 16/70 column packed with Sephacryl S200HR | GE healthcare |
参考文献
- Drin, G. Topological regulation of lipid balance in cells. Annual Review of Biochemistry. 83, 51-77 (2014).
- Bigay, J., Antonny, B. Curvature, lipid packing, and electrostatics of membrane organelles: defining cellular territories in determining specificity. Developmental Cell. 23 (5), 886-895 (2012).
- Prinz, W. A. Lipid trafficking sans vesicles: where, why, how. Cell. 143 (6), 870-874 (2010).
- Holthuis, J. C., Menon, A. K. Lipid landscapes and pipelines in membrane homeostasis. Nature. 510 (7503), 48-57 (2014).
- Wong, L. H., Copic, A., Levine, T. P. Advances on the transfer of lipids by lipid transfer proteins. Trends in Biochemical Sciences. 42 (7), 516-530 (2017).
- Wong, L. H., Gatta, A. T., Levine, T. P. Lipid transfer proteins: the lipid commute via shuttles, bridges and tubes. Nature Reviews Molecular Cell Biology. 20 (2), 85-101 (2019).
- Iaea, D. B., Dikiy, I., Kiburu, I., Eliezer, D., Maxfield, F. R. STARD4 membrane interactions and sterol binding. Biochemistry. 54 (30), 4623-4636 (2015).
- Wilhelm, L. P., et al. STARD3 mediates endoplasmic reticulum-to-endosome cholesterol transport at membrane contact sites. The EMBO Journal. 36 (10), 1412-1433 (2017).
- Bian, X., Saheki, Y., De Camilli, P. Ca(2+) releases E-Syt1 autoinhibition to couple ER-plasma membrane tethering with lipid transport. The EMBO Journal. 37 (2), 219-234 (2018).
- Horenkamp, F. A., Valverde, D. P., Nunnari, J., Reinisch, K. M. Molecular basis for sterol transport by StART-like lipid transfer domains. The EMBO Journal. 37 (6), 98002 (2018).
- Jentsch, J. A., et al. Structural basis of sterol binding and transport by a yeast StARkin domain. The Journal of Biological Chemistry. 293 (15), 5522-5531 (2018).
- Moser von Filseck, J., et al. INTRACELLULAR TRANSPORT. Phosphatidylserine transport by ORP/Osh proteins is driven by phosphatidylinositol 4-phosphate. Science. 349 (6246), 432-436 (2015).
- Daum, G., et al. Systematic analysis of yeast strains with possible defects in lipid metabolism. Yeast. 15 (7), 601-614 (1999).
- Ejsing, C. S., et al. Global analysis of the yeast lipidome by quantitative shotgun mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2136-2141 (2009).
- Leidl, K., Liebisch, G., Richter, D., Schmitz, G. Mass spectrometric analysis of lipid species of human circulating blood cells. Biochimica et Biophysica Acta. 1781 (10), 655-664 (2008).
- Sampaio, J. L., et al. Membrane lipidome of an epithelial cell line. Proceedings of the National Academy of Sciences of the United States of America. 108 (5), 1903-1907 (2011).
- Vance, J. E., Steenbergen, R. Metabolism and functions of phosphatidylserine. Progress in Lipid Research. 44 (4), 207-234 (2005).
- Zinser, E., et al. Phospholipid synthesis and lipid composition of subcellular membranes in the unicellular eukaryote Saccharomyces cerevisiae. Journal of Bacteriology. 173 (6), 2026-2034 (1991).
- Leventis, P. A., Grinstein, S. The distribution and function of phosphatidylserine in cellular membranes. Annual Review of Biophysics. 39, 407-427 (2010).
- Yeung, T., et al. Membrane phosphatidylserine regulates surface charge and protein localization. Science. 319 (5860), 210-213 (2008).
- Kim, J., Shishido, T., Jiang, X., Aderem, A., McLaughlin, S. Phosphorylation, high ionic strength, and calmodulin reverse the binding of MARCKS to phospholipid vesicles. Journal of Biological Chemistry. 269 (45), 28214-28219 (1994).
- Sigal, C. T., Zhou, W., Buser, C. A., McLaughlin, S., Resh, M. D. Amino-terminal basic residues of Src mediate membrane binding through electrostatic interaction with acidic phospholipids. Proceedings of the National Academy of Sciences. 91 (25), 12253-12257 (1994).
- Gal Bivona, T., et al. PKC regulates a farnesyl-electrostatic switch on K-Ras that promotes its association with Bcl-XL on mitochondria and induces apoptosis. Molecular Cell. 21 (4), 481-493 (2006).
- Finkielstein, C. V., Overduin, M., Capelluto, D. G. Cell migration and signaling specificity is determined by the phosphatidylserine recognition motif of Rac1. The Journal of Biological Chemistry. 281 (37), 27317-27326 (2006).
- Bolsover, S. R., Gomez-Fernandez, J. C., Corbalan-Garcia, S. Role of the Ca2+/Phosphatidylserine Binding Region of the C2 Domain in the Translocation of Protein Kinase Cα to the Plasma Membrane. Journal of Biological Chemistry. 278 (12), 10282-10290 (2003).
- Vance, J. E., Tasseva, G. Formation and function of phosphatidylserine and phosphatidylethanolamine in mammalian cells. Biochimica et Biophysica Acta. 1831 (3), 543-554 (2013).
- Maeda, K., et al. Interactome map uncovers phosphatidylserine transport by oxysterol-binding proteins. Nature. 501 (7466), 257-261 (2013).
- D'Ambrosio, J. M., et al. Osh6 requires Ist2 for localization to ER-PM contacts and efficient phosphatidylserine transport in budding yeast. Journal of Cell Science. 133 (11), 243733 (2020).
- Manford, A. G., Stefan, C. J., Yuan, H. L., Macgurn, J. A., Emr, S. D. ER-to-plasma membrane tethering proteins regulate cell signaling and ER morphology. Developmental Cell. 23 (6), 1129-1140 (2012).
- Collado, J., et al. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- Hoffmann, P. C., et al. Tricalbins contribute to cellular lipid flux and form curved ER-PM contacts that are bridged by rod-shaped structures. Developmental Cell. 51 (4), 488-502 (2019).
- Lipp, N. F., et al. An electrostatic switching mechanism to control the lipid transfer activity of Osh6p. Nature Communications. 10 (1), 3926 (2019).
- Chung, J., et al. INTRACELLULAR TRANSPORT. PI4P/phosphatidylserine countertransport at ORP5- and ORP8-mediated ER-plasma membrane contacts. Science. 349 (6246), 428-432 (2015).
- Sohn, M., et al. PI(4,5)P2 controls plasma membrane PI4P and PS levels via ORP5/8 recruitment to ER-PM contact sites. The Journal of Cell Biology. 217 (5), 1797-1813 (2018).
- Ghai, R., et al. ORP5 and ORP8 bind phosphatidylinositol-4, 5-biphosphate (PtdIns(4,5)P 2) and regulate its level at the plasma membrane. Nature Communications. 8 (1), 757 (2017).
- Sohn, M., et al. Lenz-Majewski mutations in PTDSS1 affect phosphatidylinositol 4-phosphate metabolism at ER-PM and ER-Golgi junctions. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4314-4319 (2016).
- Kattan, W. E., et al. Targeting plasma membrane phosphatidylserine content to inhibit oncogenic KRAS function. Life Science Alliance. 2 (5), 00431 (2019).
- Du, X., Turner, N., Yang, H. The role of oxysterol-binding protein and its related proteins in cancer. Seminars in Cell & Developmental Biology. 81, 149-153 (2018).
- Galmes, R., et al. ORP5/ORP8 localize to endoplasmic reticulum-mitochondria contacts and are involved in mitochondrial function. EMBO reports. 17 (6), 800-810 (2016).
- Du, X., et al. ORP5 localizes to ER-lipid droplet contacts and regulates the level of PI(4)P on lipid droplets. The Journal of Cell Biology. 219 (1), 201905162 (2020).
- Wang, H., et al. ORP2 delivers cholesterol to the plasma membrane in exchange for phosphatidylinositol 4, 5-bisphosphate (PI(4,5)P2). Molecular Cell. 73 (3), 458-473 (2019).
- Kay, J. G., Grinstein, S. Sensing phosphatidylserine in cellular membranes. Sensors (Basel). 11 (2), 1744-1755 (2011).
- Moser von Filseck, J., Vanni, S., Mesmin, B., Antonny, B., Drin, G. A phosphatidylinositol-4-phosphate powered exchange mechanism to create a lipid gradient between membranes. Nature Communications. 6, 6671 (2015).
- Wills, R. C., Goulden, B. D., Hammond, G. R. V. Genetically encoded lipid biosensors. Molecular Biology of the Cell. 29 (13), 1526-1532 (2018).
- Raychaudhuri, S., Im, Y. J., Hurley, J. H., Prinz, W. A. Nonvesicular sterol movement from plasma membrane to ER requires oxysterol-binding protein-related proteins and phosphoinositides. The Journal of Cell Biology. 173 (1), 107-119 (2006).
- Lenoir, M., et al. Structural basis of wedging the Golgi membrane by FAPP pleckstrin homology domains. EMBO reports. 11 (4), 279-284 (2010).
- Liu, Y., Kahn, R. A., Prestegard, J. H. Interaction of Fapp1 with Arf1 and PI4P at a membrane surface: an example of coincidence detection. Structure. 22 (3), 421-430 (2014).
- Shao, C., Novakovic, V. A., Head, J. F., Seaton, B. A., Gilbert, G. E. Crystal structure of lactadherin C2 domain at 1.7A resolution with mutational and computational analyses of its membrane-binding motif. The Journal of Biological Chemistry. 283 (11), 7230-7241 (2008).
- Lipp, N. F., Ikhlef, S., Milanini, J., Drin, G. Lipid exchangers: cellular functions and mechanistic links with phosphoinositide metabolism. Frontiers in Cell and Developmental Biology. 8, 663 (2020).
- Venditti, R., et al. Molecular determinants of ER-Golgi contacts identified through a new FRET-FLIM system. The Journal of Cell Biology. 218 (3), 1055-1065 (2019).
- Pemberton, J. G., et al. Defining the subcellular distribution and metabolic channeling of phosphatidylinositol. The Journal of Cell Biology. 219 (3), (2020).
- Nakanishi, H., de los Santos, P., Neiman, A. M. Positive and negative regulation of a SNARE protein by control of intracellular localization. Molecular Biology of the Cell. 15 (4), 1802-1815 (2004).
- Maekawa, M., Yang, Y., Fairn, G. D. Perfringolysin O theta toxin as a tool to monitor the distribution and inhomogeneity of cholesterol in cellular membranes. Toxins. 8 (3), 67 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved