Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Membranlar Arasında Fosfataylserine/Fosfatidiylinositol 4-Fosfat Değişiminin Floresan Bazlı Ölçümleri
Bu Makalede
Özet
Burada, bir proteinin fosfatidiylserine veya fosfatidiliinositol 4-fosfat in vitro'yuayıklayıp taşımadığını belirlemek için floresan lipid sensörleri ve lipozomlar kullanarak protokolleri açıklıyoruz.
Özet
Evrimsel olarak korunmuş oksisterol bağlayıcı protein (OSBP) ile ilişkili proteinler (ORP)/OSBP homologları (Oş) ailesinin birkaç üyesinin son zamanlarda maya ve insan hücrelerinde yeni bir lipit transfer proteini (LTP) grubunu temsil ettiği bulunmuştur. Fosfatidiylinositol 4-fosfat (PI(4)P) değişim döngüleri ile endoplazmik reticulumdan (ER) plazma membranlarına (PM) fosfatidiylserine (PS) aktarırlar. Bu bulgu, sinyalizasyon işlemleri için kritik olan PS'nin hücre boyunca nasıl dağıtıldığının ve bu işlem ile fosfoinositid (PIP) metabolizması arasındaki bağlantının araştırılmasının daha iyi anlaşılmasını sağlar. Yeni floresan bazlı protokollerin geliştirilmesi, bu yeni hücresel mekanizma in vitro'nun moleküler düzeyde keşfedilmesinde ve karakterizasyonunda etkili olmuştur. Bu makale, bir proteinin PS veya PI(4)P çıkarma yeteneğini ölçmek ve bu lipitleri yapay zarlar arasında aktarmak için floresan etiketli iki lipit sensörünün, NBD-C2Lact ve NBD-PHFAPP'ınüretimini ve kullanımını açıklamaktadır. İlk olarak, protokol bu iki yapının yüksek saflıkta örneklerinin nasıl üretılacağını, etiketlenerek ve elde edilir. İkinci olarak, bu makale, bir proteinin Osh6p'yi vaka çalışması olarak kullanarak lipozomlardan PS veya PI(4)P çıkarıp çıkarabileceğini belirlemek için bu sensörlerin floresan mikro plaka okuyucu ile nasıl kullanılacağını açıklar. Son olarak, bu protokol, tanımlanmış lipid bileşiminin lipozomları arasındaki PS/PI(4)P değişiminin kinetiğinin nasıl doğru bir şekilde ölçüleceğini ve standart bir florometre kullanılarak floresan rezonans enerji transferi (FRET) ile lipid transfer oranlarının nasıl belirleneceğini göstermektedir.
Giriş
Lipitlerin farklı membranlar arasında ve ökaryotik hücrelerin zarları içinde kesin dağılımı1,2 derin biyolojik etkilere sahiptir. LTP'lerin nasıl çalıştığının şifresini çözmek hücre biyolojisinde önemli bir konudur3,4,5,6ve in vitro yaklaşımlar bu sorunu ele almada büyük değere sahiptir 7 ,8,9,10,11. Burada, birkaç ORP/Osh proteininin PS/PI(4)P hücre zarları12 arasındaki değişimi etkilediğini ve böylece yeni bir LTP sınıfı oluşturduğunu belirlemede etkili olan in vitro,floresan tabanlı bir strateji sunulmuştur. PS, ökaryotik hücrelerde toplam membran lipitlerinin% 2-10'unu temsil eden aniyonik bir gliserofsfolipiddir13,14,16. Sırasıyla17, 18,19 gliserofosfolipidlerin% 5-7'sini ve% 30'unu temsil ettiği ER ve PM arasında birgradyanboyuncadağıtılır. Ayrıca, PS esasen PM'nin sitosolik broşüründe yoğunlaşmıştır. Bu birikme ve PM'deki PS'nin düzensiz bölümü hücresel sinyal işlemleri için kritiktir19. PS moleküllerinin negatif yükü nedeniyle, PM'nin sitosolik broşürü, diğer organellerin sitozolitik broşürü 1,2,19,20'dençok daha anioniktir. Bu, elektrostatik kuvvetler aracılığıyla işe alıma olanak sağlar, miristosilat alanin bakımından zengin C-kinaz substratı (MARCKS)21, sarkom (Src)22, Kirsten-rat sarkom viral onkogen (K-Ras)23ve Ras ile ilgili C3 botulinum toksin substratı 1 (Rac1)24 gibi pozitif yüklü amino asitler ve lipidik kuyruk içeren sinyal proteinleri.
PS ayrıca geleneksel protein kinaz C tarafından stereoselektif bir şekilde C2 etki alanı25 aracılığıyla tanınır. Ancak, PS sentezlendi ER26, rolünü oynayabilmesi için PM'ye dışa aktarılması gerektiğini gösterir. Mayada, Osh6p ve Osh7p'nin PS'yi ACIL'den PM27'yeaktardığı bulguya kadar bunun nasıl19'a başarıldığı bilinmiyordu. Bu LTP'ler, kurucu üyesi OSBP olan ve bir lipid molekülü barındırmak için OSBP ile ilgili bir etki alanını (ORD) bir ceple entegre eden proteinler (insanda ORP'ler, mayadaki Oş proteinleri) içeren ökaryotlarda evrimsel olarak korunmuş bir aileye aittir. Osh6p ve Osh7p, yalnızca yapısal özellikleri PS'yi özellikle bağlamak ve membranlar arasında aktarmak için uyarlanmış bir ORD'den oluşur. Bununla birlikte, bu proteinlerin PS'yi acil servisten PM'ye nasıl aktardığı belirsizdi. Osh6p ve Osh7p, PI(4)P'yi alternatif bir lipid ligand12olarak hapsedebilir. Mayada PI(4)P, Golgi ve PM'deki fosfatidylinositol'den (PI) sırasıyla PI 4-kinaz, Pik1p ve Stt4p ile sentezlenmiştir. Buna karşılık, ER membranında PI(4)P yoktur, çünkü bu lipit Sac1p fosfataz tarafından PI'ye hidrolize edilir. Bu nedenle, hem ER/Golgi hem de ER/PM arabirimlerinde bir PI(4)P gradyanı bulunur. Osh6p ve Osh7p, bu iki membran arasında bulunan PI(4)P gradyanını kullanarak PS/PI(4)P değişim döngüleri aracılığıyla PS'den PM'ye TRANSFER12.
Bir döngü içinde, Osh6p ACIL'den PS çıkarır, PM'de PI(4)P için PS değiştirir ve başka bir PS molekülü çıkarmak için PI(4)P'yi ER'ye geri aktarır. Osh6p / Osh7p ist2pile etkileşime girer 28, ER membranını ve PM'yi birbirine bağlayan ve yakın bir hale getiren birkaç proteinden biri maya29,30,31'deER-PM temas siteleri oluşturmak için. Ek olarak, Osh6p'nin negatif yüklü membranlarla ilişkisi, protein lipid ligandlarından birini elektrostatik özelliklerini değiştiren konformasyonsal bir değişiklik nedeniyle çıkarır çıkarmaz zayıflar32. Bu, membran tutma süresini kısaltarak Osh6p'ye yardımcı olur, böylece lipid transfer aktivitesinin verimliliğini korur. Ist2p'ye bağlama ile birlikte, bu mekanizma Osh6p/7p'nin ER/PM arabiriminde lipid değişimini hem hızlı hem de doğru bir şekilde yürütmesine izin verebilir. İnsan hücrelerinde, ORP5 ve ORP8 proteinleri ER-PM temas bölgelerinde PS/PI(4)P değişimini farklı mekanizmalar aracılığıyla yürütür33. Osh6p'ye benzeyen merkezi bir ORD'ye sahiptirler, ancak doğrudan bir C terminali transmembransegmenti 33 aracılığıyla ACIL'ye bağlanırlar ve PI(4)P ve PI(4,5)P233 , 34,35'i tanıyan bir N terminali Pleckstrin homolojisi (PH) etki alanı aracılığıyla PM'ye yanaşırlar. ORP5/8, PS'yi aktarmak için PI(4)P kullanır ve ORP5/8'in PM PI(4,5)P2 seviyelerini ayrıca düzenlediği ve muhtemelen sinyal yollarını modüle ettiği gösterilmiştir. Buna karşılık, PI(4)P ve PI(4,5)P2 seviyelerindeki bir düşüş, bu proteinler PIP'e bağlı bir şekilde PM ile ilişkili olduğu için ORP5 / ORP8 aktivitesini düşürür. Lenz-Majewski sendromuna yol açan anormal derecede yüksek PS sentezi, ORP5/836aracılığıyla PI(4)P seviyelerini etkiler. Her iki proteinin aktivitesi engellendiğinde, PS PM'de daha az bol hale gelir ve proteinlerin onkojenik yeteneğini düşürür37.
Tersine, ORP5 aşırı ifade kanser hücre invazyonunu ve metastatik süreçleri teşvik ediyor gibi görünüyor38. Bu nedenle, ORP5/8 aktivitesindeki değişiklikler lipid homeostazındaki değişikliklerle hücresel davranışı ciddi şekilde değiştirebilir. Ayrıca, ORP5 ve ORP8 ER-mitokondri temas alanlarını işgal ediyor ve muhtemelen PS39sağlayarak bazı mitokondriyal işlevleri koruyor. Ayrıca ORP5, PS/PI(4)P exchange40ile LIPID damlacıklarına PS teslim etmek için ER-lipid damlacık iletişim sitelerine yerelleştirir. Burada açıklanan strateji, (i) PS ve PI(4)Lipozomlardan P ekstraksiyonunu ve (ii) LIPOMLAR ARASıNDAKI PS ve PI(4)P naklini ölçmek için Osh6p/Osh7p12,32'nin PS/PI(4)P değişim aktivitesini kurmak ve analiz etmek için tasarlanmıştır ve diğer gruplar tarafından ORP5/ORP835 ve diğer LTP10'larınaktivitesini analiz etmek için kullanılmıştır. 41. Bir floresan plaka okuyucu, standart bir L-format spektrofluorometre ve sırasıyla PS ve PI(4)P'yi algılayabilen iki floresan sensör, NBD-C2Lact ve NBD-PHFAPPkullanımına dayanmaktadır.
NBD-C2Lact, glikoprotein, lactadherin'in C2 etki alanına karşılık gelir, bu da varsayılan PS bağlama bölgesinin yakınında benzersiz bir çözücüye maruz kalan sistein içerecek şekilde yeniden tasarlanmıştır; polariteye duyarlı bir NBD (7-nitrobenz-2-oxa-1,3-diazol) floroforu bu kalıntıya yaygın olarak bağlıdır (Şekil 1A)12. Daha kesin olmak gerekirse, lactadherin C2 etki alanı (Bos taurus, UniProt: Q95114, kalıntılar 270-427) Escherichia coliglutatyon S-transferaz (GST) ile füzyon ifade etmek üzere bir pGEX-4T3 vektör içine klonlanmıştır. C2Lact dizisi daha sonra iki solventle erişilebilir sistein kalıntısını (C270, C427) alanin kalıntıları ile ve daha sonra N,N'-dimetil-ile etiketlenebilen putatif PS-bağlama bölgesinin (H352C mutasyonu) yakınındaki bir bölgeye sistein kalıntısı sokmak için N-(iodoacetyl)-N'-(7-nitrobenz-2-oxa-1,3-diazol-4-yl) etilen diamin (IANBD) 12. GST proteini ile C2 alanının N-terminusu arasında trombin için bir bölünme bölgesi bulunur. Önemli bir avantaj, bu etki alanının PS'yi bilinen diğerC2alan adlarına veya Annexin A542'yeaykırı olarak Ca 2+ bağımsız bir şekilde seçici olarak tanımasıdır. NBD-PHFAPP, PI(4)P bağlama bölgesinin yakınındaki bir NBD grubu ile etiketlenebilen tek bir solvent maruziyeti sistein içerecek şekilde yeniden tasarlanmış insan dört fosfat adaptör proteini1'in (FAPP1)PH etki alanından türetilmiştir ( Şekil 1A )43. İnsan FAPP proteininin PH etki alanının nükleotid dizisi (UniProt: Q9HB20, segment [1-100]), bir GST etiketiyle birlikte ifade edilecek bir pGEX-4T3 vektörüne klonlanmıştır. PHFAPP dizisi, protein43'ünmembran bağlayıcı arayüzüne benzersiz bir sistein kalıntısı eklemek için değiştirilmiştir. Ayrıca, proteazlara erişilebilirliği sağlamak için trombin bölünme bölgesi ile PH alanının N-terminus'u arasında dokuz kalıntılı bir bağlayıcı getirilmiştir.
Lipozomlardan PS ekstraksiyonunu ölçmek için NBD-C2Lact, eser miktarda PS içeren fosfatidiylcholine (PC) yapılmış lipozomlarla karıştırılır. PS'ye olan yakınlığı nedeniyle, bu yapı lipozomlara bağlanır ve NBD floroforu, zarın hidrofobik ortamıyla temas ettikçe polaritede bir değişiklik yaşar ve bu da mavi kayma ve floresan artışı sağlar. PS neredeyse tamamen bir stoichiometric LTP miktarı ile ayıklanırsa, prob lipozomlarla ilişkilendirmez ve NBD sinyali daha düşüktür (Şekil 1B)32. Bu sinyal farkı, bir LTP'nin(örneğin, Osh6p) PS'yi ayıklayıp ayıklamadığını belirlemek için kullanılır. Benzer bir strateji, daha önce açıklandığı gibi PI(4)P ekstraksiyonunu (Şekil 1B) ölçmek için NBD-PHFAPP ile kullanılır12,32. (i) SıRASıYLA ER membranını ve PM'yi taklit eden LA'dan LB lipozomlarına PS taşımacılığını ve (ii) PI(4)P taşımasını ters yönde ölçmek için iki FRET tabanlı test tasarlanmıştır. Bu tahliller PS/PI(4)P değişimini ölçmek için aynı koşullar(yani aynı tampon, sıcaklık ve lipid konsantrasyonu) altındagerçekleştirilir. PS taşımacılığını ölçmek için, NBD-C2Lact PC'den oluşan L A lipozomları ile karıştırılır ve 5 mol% PS ve 2 mol% 5 mol PI (4) P içeren floresan rhodamin etiketli fosfatidylethanolamine (Rhod-PE) ve LB lipozomları ile karıştırılır.
Sıfır noktasında, Rhod-PE'li FRET, NBD floresanını bastırır. PS LA'dan LB lipozomlarına taşınırsa(örneğin, Osh6p enjekte edildikten sonra), NBD-C2Lakt moleküllerinin LA'dan LB lipozomlarına translokasyonu nedeniyle hızlı bir söndürme meydana gelir (Şekil 1C). Erişilebilir PS miktarı göz önüne alındığında, NBD-C2Lact esasen deney boyunca zara bağlı bir durumda kalır12. Bu nedenle, NBD sinyalinin yoğunluğu, NBD-C2Lakt'in LA ve LB lipozomları arasındaki dağılımıyla doğrudan ilişkilidir ve ne kadar PS aktarıldığını belirlemek için kolayca normalleştirilebilir. PI(4)P'nin ters yönde transferini ölçmek için, NBD-PHFAPP LA ve LB lipozomları ile karıştırılır; Sadece PI(4)P içeren LB lipozomlarına bağlandığı, ancak Rhod-PE'ye bağlanmadığı göz önüne alındığında, floresan yüksektir. PI(4)P LA lipozomlarına transfer edilirse, bu lipozomlara doğru hareket eder ve Rhod-PE ile FRET nedeniyle sinyal azalır (Şekil 1C). Sinyal, ne kadar PI(4)P aktarıldığını belirlemek için normalleştirilir43.
Protokol
1. NBD-C2Laktının Saflaştırılması
NOT: Bu protokol bakterileri kırmak için bir hücre bozucunun kullanımını detaylandırsa da, diğer liziz stratejilerini(örneğin, bir Fransız basını) kullanmak için değiştirilebilir. Saflaştırmanın başlangıcında, sistein oksidasyonunu önlemek için taze gazdan arındırılmış, filtrelenmiş ve 2 mM dithiothreitol (DTT) ile desteklenmiş tampon kullanılması zorunludur. Bununla birlikte, protein etiketleme adımı için DTT'yi tamamen çıkarmak çok önemlidir. Herhangi bir protein bozulmasını önlemek için buzda veya soğuk bir odada birçok adım atılmalıdır. Saflaştırmanın ilerlemesini kontrol etmek için% 15 akrilamid jel kullanılarak sodyum dodecylsulfate-poliakrilamid jel elektroforez (SDS-PAGE) tarafından analiz yapmak için protokolün farklı adımlarında 30 μL hacimli numuneler toplanmalıdır. Her aliquot ile yeterince denatüre Laemmli numune tamponu karıştırın ve karışımı 95 °C'de ısıtın. Boruları dondurun ve analize kadar -20 °C'de saklayın.
- Escherichia coli'de GST-C2Lact ifadesi
- 20 μL BL21 Altın yetkin hücreleri 18 μL sterilize su ile karıştırın. Daha sonra, 2 μL pGEX-C2Lact plazmidini (~65 ng/μL'de) bakterilerle karıştırın ve elektroporasyonla dönüştürün. Bakterileri 150 μL otoklavlı Lennox Lysogeny-Broth (LB) ortamı (10 g/L tripto, 5 g/L maya özü, deiyonize suda 5 g/L NaCl, glikozsuz) ile yeniden ıslatın. Bakterilerin 2 mL snap-cap mikrosantrifüj tüpünde 1 saat boyunca 37 °C'de büyümesine izin verin.
- 50 μg/mL ampisilin ile desteklenmiş 25 mL LB ortamı aşıla, 125 mL steril Erlenmeyer şişesinde 150 μL bakteri süspansiyonu. Şişeyi 37 °C'de bir yörünge çalkalayıcıya yerleştirin ve bakterilerin 185 rpm'de ajitasyonla bir gecede büyümesine izin verin.
- İki steril 2 L Erlenmeyer şişesini 50 μg/mL ampisilin ile desteklenmiş 500 mL LB orta ile doldurun ve 5 mL ön kültür süspansiyonu ekleyin. Bakterilerin 37 °C'de 220 rpm'de ajitasyonla büyümesine izin verin.
- Süspansiyonun optik yoğunluğunu (OD) periyodik olarak 600 nm dalga boyunda (φ) ölçün. OD ~0.6-0.7 değerine ulaştığında, GST-C2Lactifadesini başlatmak için her şişeye 1 M izopropil β-D-1-tiyogalactopyranoside (IPTG) stok çözeltisinin 500 μL'sini ekleyin. Mataraları 185 rpm'de 37 °C'de 4 saat sallayın.
- Her şişenin içeriğini polipropilen santrifüj şişesine aktarın. Bakterileri peletmek için iki şişeyi 4600 × g'da 30 dakika boyunca santrifüjlayın. Süpernatantı atın ve her peletin 50 mL soğuk fosfat tamponlu salin içinde yeniden depolanın.
- Her şişede bulunan bakteriyel süspansiyonu 50 mL konik santrifüj tüpüne aktarın. İki tüpü 2300'de 30 dakika × 4 °C'de santrifüjedin. Süpernatant çıkarın ve her biri bakteriyel bir pelet içeren tüpleri -20 °C'de saklayın.
- C2'nin SaflaştırılmasıLakt
- Buz üzerinde, daha önce membran vakum filtrasyonu ile filtrelenmiş ve gazdan arındırılmış 50 mM Tris-HCl, pH 7.4 ve 150 mM NaCl (bundan böyle TN tamponu olarak adlandırılır) içeren 50 mL tampon içeren iki adet 50 mL konik santrifüj tüpü doldurun.
- Her tüpte lizis tamponunu hazırlamak için, hafif sonication veya girdap ile TN tamponunda etilenediamin tetraasetik asit içermeyen proteaz inhibitörü kokteyli tabletini çözün. Diğer antiproteazları ekleyin, (10 μM bestatin, 1 μg / mL pepstatin A ve 10 μM fosforamidon). Daha da önemlisi, tamponu 2 mM DTT ile tamamlar.
- 1.1.6 adımda hazırlanan bakteriyel peletleri içeren iki tüpü, her tüpte 30 mL'lik son bir hacim elde etmek için lizis tamponu ile doldurun ve 10 dakika boyunca buzu yavaşça buz üzerinde çözün. Her peletin paslanmaz çelik spatula ile ezin ve tüpleri girdaplayarak ve/veya süspansiyonu bir pipet denetleyicisi ve 25 mL pipet ile ileri geri pipetle sulayarak homojen bir süspansiyon elde edilene kadar yeniden depolayın.
- Numunenin 30 mL'lik kısmını rezervuarın içine yükleyerek ve 1,6 bar basınçla sürekli modda bir kırılma döngüsü çalıştırarak önceden soğulmuş bir hücre bozucu kullanarak liziz yapın (Bkz. Malzeme Tablosu). Lisatı aynı tüpte toplayın, tüpü buzda tutun ve hemen izopropanolde hazırlanan 200 mM feniltilsülfonil florür (PMSF) stok çözeltisinden 250 μL ekleyin.
- Lyse aynı prosedürü izleyen diğer örnek. Hücre bozucuyu yıkamak için liziz arabelleğin geri kalanını kullanın ve her lizatın (~30 mL) hacmini 50 mL'lik son hacme ayarlamak için yıkamayı toplayın.
- Her bir lisate 5 mM MgCl2ile takviye edin ve DNA'yı parçalamak ve böylece numunenin viskozitesini azaltmak için 20 μg / mL DNASE I ekleyin. 30 dakika boyunca buzda kuluçkaya yaslanın. Jel analizi için bir örnek toplayın.
- Her 50 mL lisate'yi ön-ön sevişmiş polikarbonat ultrasantrifüj tüpüne aktarın (toplamda iki, Malzeme Tablosunabakın). Santrifüj 186.000 × g'da 4 °C'de en az 1 saat ultracentrifuge kullanarak.
- Santrifüjleme adımına paralel olarak, iki adet 50 mL konik santrifüj tüpüne% 4 agarose boncuk ile birleştirilmiş glutatyon içeren bir bulamacın 1,4 mL'sini dağıtın, her tüpe 1 mM DTT (TND tampon) ile desteklenmiş 20 mL TN tampon ekleyin, 5 dakika boyunca 1200 × g'da santrifüj ekleyin ve süpernatantı atın. Bu yıkama adımını iki kez tekrarlayın.
- Bakteriyel lisatın santrifüjlenmesinden sonra, süperndanttan 30 μL'lik bir numune çıkarın ve süpernatantı her ultrasantuge tüpünden temiz boncuklar içeren karşılık gelen 50 mL konik santrifüj tüpüne aktarın. Jel analizi için, enkaz peletini 50 mL TND tamponlu ultrasantrifüj tüplerinden birine yeniden biriktirin ve 30 μL'lik bir örnek toplayın.
- Homojen bir boncuk süspansiyonu elde etmek için tüpleri 4 °C'de 3-4 saat boyunca bir rotator üzerine yerleştirin. Boncuk süspansiyonlarını boş bir 25 mL kromatografi sütununda birlayın. Boncukların dekante olmasına izin verin ve tamponu ve ilişkisiz proteinleri yerçekimi akışı ile çıkarın. Analiz için elüattan bir örnek alın.
- Boncukları 20 mL TND tampon ile yeniden biriktirin ve eluat'ı yerçekimi akışı ile toplayın. Boncukları tamamen yıkamak için bu adımı iki kez tekrarlayın. Toplanan eluatları bire biriktirin ve daha fazla analiz için 30 μL'lik bir numuneyi saklanın.
NOT: Kısa bir dekantasyondan sonra, GST-C2Lact'in bağlı olduğu ~2 mL'lik bir boncuk süspansiyon hacmi, sütunun altındaki çökeltiler. - İki adet 2 mL snap-cap mikrosantrifüj tüpüne 1 mL boncuk süspansiyon ekleyin. Her tüpü 1.970 mL'lik son hacme kadar TND tamponu ile doldurun. Daha fazla analiz için bir tüpten 30 μL numune alın (B1 örneği). 0,02 U/μL'de insan trombin proteaz çözeltisi stok çözeltisinden 10 μL 10 mM CaCl 2 çözeltisi ve 25 μL ekleyin.
- Trombinin C2Lact etki alanından GST etiketini kesmesine izin vermek için iki tüpü bir gecede 4 °C'de bir rotatora yerleştirin. Ertesi gün, her tüpte, trombin eylemini engellemek için boncuk süspansiyonu ile 10 μL 200 mM PMSF çözeltisini karıştırın.
- Tüpleri 5 dakika boyunca 700 × g'da santrifüj edin ve çözünür C2Lact etki alanı içeren süpernatantı boncukları almadan her tüpten toplayın. Süpernatantları buz üzerinde tutulan 2 mL snap-cap mikrosantrifüj tüpüne (E1 eluat) bir şekilde birin.
- Boncukları yeniden kullanmak için her tüpe 1 mL TND arabelleği ekleyin ve yıkayın; 1.2.14. Maksimum miktarda proteini kurtarmak için bu adımı üç kat daha fazla gerçekleştirin. Her seferinde, toplanan süpernatantları yeni bir 2 mL tüpe (E2, E3, E4 ve E5 eluates) bir şekilde bire birler ve daha fazla analiz için bir aliquot alın. Yıkama adımlarının sonunda, boncuk süspansiyonundan bir aliquot alın (aliquot B2).
- %15 akrilamid jel üzerinde SDS-PAGE ayrımı ile saflaştırma protokolünün farklı adımlarında toplanan 30 μL numuneyi analiz edin.
- 1.2.14 ve 1.2.15 adımları sırasında toplanan tüm süpernatantları(yani~10 mL) 10 mL kromatografi sütununa toplayarak potansiyel kirletici boncukları çıkarın. Eluate'i yerçekimi akışına göre toplayın ve boncukları sütunun altında tutun.
- C2Lact örneğini, 3 kDa moleküler ağırlık kesme (MWCO) ve 2300 × g santrifüjleme hızına sahip bir santrifüj filtre ünitesi kullanarak konsantre edin. Protein örneğinin hacmi ~1 mL olduğunda konsantrasyon prosedürünü durdurun.
- NBD-C2'nin hazırlanması ve saflaştırılmasıLakt
- Tuzdan arındırma sütununu (Bkz. Malzeme Tablosu)TN arabelleğiyle dengele. Sütunu 1 mL konsantre C2Lakt örneği ile yükleyin. Numunenin jel yatağına tamamen girmesine izin verin, sütuna 1,5 mL taze gazsız DTT içermeyen TN tamponu ekleyin ve eluate'i yerçekimi akışı ile 2 mL snap-cap mikrosantrifüj tüpüne toplayın.
- 50 μL elüat'ı 300 μL TN tamponun son hacminde seyreltin ve saf TN arabelleği boş olarak kullanarak 230 ila 450 nm arasında bir absorbans spektrumu kaydedin. 44.920 M -1 .cm-1'eeşit bir yok olma katsayısı ε göz önünde bulundurarak, 280 nm olarak ölçülen emiciliğe göre C2Lakt konsantrasyonu belirleyin.
- C2Lact yapıyı bir NBD floroforu ile etiketlemek için, proteini on kat azı dişi fazlalık N,N'-dimetil-N-(iodoacetyl)-N'-(7-nitrobenz-2-oxa-1,3-diazol-4-yl)etilenediamin (IANBD amide) ile karıştırın.
- C2 Lact yapısınıetiketlemek için kullanılan DMF'nin son hacminin protein numune hacminin% 5'ini (v/v) geçmemesi gerektiğini göz önünde bulundurarak, 1 mg IANBD'yi susuz dimetilformamid (DMF) içinde çözün.
- IANBD'yi çözmek içinDMF (V DMF)hacmini belirlemek için, önce formül 1kullanarak proteini etiketlemek için gerekli miktarda IANBD (m, mg ile ifade edilir) hesaplayın.
m = 10.000 × C × V × MWIANBD (1)
burada C, adım 1.3.2'de ölçülen C2Lakt konsantrasyonudur, V C2Lakt örneğinin hacmidir ve MWIANBD floroforun moleküler ağırlığıdır (420 g/mol). - 2 (m0 =1 ile) ve 3formüllerini kullanarak VDMF'yi hesaplayın.
VDMF= (m0/m) × VIANBD (2)
VIANBD=0,05 × V (3)
Burada m0 mg'daki IANBD tozu miktarıdır ve VIANBD, C2Lakt örneğine eklenecek IANBD çözeltisinin hacmidir. - C2Lact örneğine yeni hazırlanmış IANBDçözeltisinin hacim V IANBD'sini ekleyin ve reaksiyon karışımını ışıktan korunan bir termomikser kullanarak 25 °C'de 30 dakika boyunca 800-900 rpm'de sallayın. Reaksiyonun buzda 90 dakika devam etmesine izin verin. Bu arada, 10 mL TN tampon ile bir santrifüj filtre ünitesini (MWCO= 3 kDa) temizleyin.
- Serbest IANBD'yi devre dışı bırakmak için reaksiyon karışımına L-sistein (IANBD'ye 10 kat azı dişi fazlalığında) ekleyin.
- NBD-C2Lakt çözeltisine 15 mL TN tampon ekleyin ve NBD-C2Lakt çözeltisini santrifüj filtre ünitesine aktarın. Serbest NBD'nin çoğunu proteinden 2300 × g'da santrifüjleme ile ayırmak için numuneyi 2 mL'ye konsantre edin. Bu yıkama adımını iki kez tekrarlayın. Numuneyi 2 mL snap-cap santrifüj tüpünde 10 dakika boyunca 19.000 × 4 °C'de potansiyel agregaları peletlemek için santrifüjlayın ve süpernatantı toplayın.
- 50 μL eluat'ı 300 μL TN tamponun son hacminde seyreltin. Konsantrasyon prosedürü sırasında toplanan elüatları boş olarak kullanarak absorbans spektrumu 230 ila 650 nm arasında kaydedin. NBD-C2Lakt konsantrasyonunun φ=280 ve 495 nm'deki maksimum emiciliği ve yok olma katsayılarını kullanarak belirleyin ε= 44.920 M-1.cm-1 (protein) ve 25.000 M-1.cm-1 (NBD florofor).
NOT: İki konsantrasyon değeri benzerse, bu C2Lakt yapının NBD grubuyla 1:1 oranında etiketlendiğini gösterir. - NBD absorbansının ölçümünden tahmin edilen NBD-C2Lakt konsantrasyonu triptofan (Trp) kalıntılarının emiciliğinden tahmin edilen konsantrasyonu aşarsa, serbest NBD'yi daha da kaldırmak için 1.3.5 adımını tekrarlayın.
- Flaş dondurma sırasında NBD-C2Lact yapısı kriyo-korumak için% 10 (v/ v) son konsantrasyon elde etmek için numuneye gliserol ekleyin. Son protein konsantrasyonu ölçün.
- 0,5 mL snap-cap mikrosantrifüj tüplerinde 50 μL aliquots protein hazırlayın. Tüpleri sıvı nitrojende flash-dondurun ve -80 °C'de saklayın.
2. NBD-PHFAPP'ın Saflaştırılması
NOT: PHFAPP üretme ve etiketleme prosedürü, NBD-C2Lakt çözeltisinin adım 1.3.4'teki bir santrifüj filtre ünitesine aktarılmasına kadar NBD-C2Lakt ile aynıdır. Bu adımdan itibaren, aşağıda açıklanan protokolü izleyin.
- Konsantrasyon adımından sonra, boyut dışlama kromatografisi yapmadan önce 2 mL NBD-PHFAPP'ı 4 °C'de karanlıkta 1 günden fazla tutmayın. Boyut dışlama kromatografiden önce, tüpün altında turuncu birikinti (konsantrasyon sırasında toplama) olmadığını doğrulayın. Bu durumda, numuneyi 540.000 × g'da 4 °C'de 10 dakika boyunca 540.000 g'da santrifüj edin ve süpernatantı boyut dışlama kromatografisi ile arındırın.
NOT: Boyut dışlama kromatografisi, çapraz bağlı dektran-akrilamid koaloksimer (bkz. Malzeme Tablosu)ile dolu bir sütunda, daha önce TND tamponu ile dengelenmiş, hızlı proteinli sıvı kromatografi sistemi kullanılarak gerçekleştirilir (Bkz. Malzeme Tablosu). Sütun ışıktan korunmalıdır. 1 mL/dk akış hızı kullanıldı ve NBD-PHFAPP yapısın elüasyonu, emicilik sütun çıkışında φ=280 (protein) ve 480 nm (NBD) olarak kaydedildi.- 2 mL enjeksiyon döngüsüne yüklenen NBD-PHFAPP örneğini sütuna enjekte edin ve hemen 2,5 mL elüat fraksiyonu toplayın.
- %15 SDS-PAGE jel üzerinde 280 ve 480 nm olarak tespit edilen ana tepeye karşılık gelen tüm fraksiyonları analiz edin. Isıtma ve jel üzerine yüklemeden önce her fraksiyonun 25 μL'lik bir örneğini 15 μL Laemmli numune tamponu ile karıştırın.
NOT: Sütundan ~150 mL arabellek hacmi geçirildikten sonra aynı anda φ= 280 ve 480 nm olarak algılanan bir ana tepe noktası görünür. - Sadece NBD-PHFAPP proteini (~12,2 kDa) içeren fraksiyonları birnize edin ve %10 'luk (v/v) son konsantrasyonda gliserol ekleyin. Numuneyi, 3 kDa MWCO'lu bir santrifüj filtre ünitesini 2300 × g santrifüjleme hızı kullanarak 1 mL'lik son hacme kadar konsantre edin.
- Aliquots hazırlayın ve NBD-C2 Lact için açıklandığı gibi bir absorbans spektrumukaydedin. φ=280 nm olarak ölçülen emimete göre proteinin konsantrasyonunun belirlenmesi için ε= 29.450M-1 .cm-1 yok olma katsayısı kullanın.
3. PS ve PI için lipozomların hazırlanması(4)P ekstraksiyonu veya transfer tahlilleri
NOT: Aksi belirtilmedikçe tüm adımları oda sıcaklığında gerçekleştirin. Organik çözücüleri, rotavapor'ı ve sıvı nitrojeni dikkatli kullanın.
- Taze, filtrelenmiş ve gazsız 50 mM 4-(2-hidroksyetil)-1-piperazineethaneslfonic asit (HEPES)-potasyum hidroksit (KOH), pH 7.4, 120 mM potasyum asetat (HK) tampon hazırlayın.
- Her lipozom türü için, stok çözeltilerinden hassas miktarlarda farklı lipitler alın ve bunları 25 mL armut şeklindeki cam şişede karıştırın(Tablo 1). Her karışımın hacmini 1 mL'ye ayarlamak için saf kloroform ekleyin. Her şişeyi lipozom adıyla etiketle. Rhod-PE ile dopded lipid karışımı içeren şişeleri alüminyum folyo ile sarın.
- Şişeyi döner buharlaştırıcıya yerleştirin. Lipitleri vakum altında ve 25 °C'de 500 rpm dönüş hızında en az 30 dakika kurutun. PI(4)P içeren lipid filmleri için, şişe duvarında kuru lipitlerin bir filmini geride bırakacak olan çözücüyü çıkarmak için şişede vakum oluşturmadan önce PI(4)P'yi diğer lipitlerle düzgün bir şekilde karıştırmak için 5 dakika boyunca 32-34 °C'de ön ısıtma.
- Şişeyi evaporatörden çıkarın ve kalan çözücü izlerini gidermek için 45 dakika boyunca bir vakum odasına yerleştirin. Şişeyi 2 mL HK tampon ile doldurun ve çözeltiye birkaç adet 4 mm çapında cam boncuk ekleyin. Lipitleri yeniden haline getirmek ve 4 mM lipit konsantrasyonu ile çokmeller lipid veziklinleri (MLV' ler) hazırlamak için şişeyi 2 dakika boyunca hafifçe girdaplayın. 1,5 mL vidalı kapaklı mikrosantrifüj tüplerde 0,5 mL MV aliquots hazırlayın.
- Tüpleri 5x dondurun (sırasıyla sıvı azot ve 37 °C'de bir su banyosu kullanarak). Lipozomları ekstrüzyona sokun veya -20 °C'de saklayın.
- MLV'lerden gelen lipozomları(yani büyük unilamellar vesicles) üreticinin yönergelerine göre hazırlamak için mini bir ekstrüder kullanın. 200 nm çapında tek tip silindirik gözeneklere sahip bir polikarbonat filtre kullanın.
- Her lipozom türünü hazırlamak için, MLV'lerin ilgili süspansiyonunun en az 250 μL'si ekstrüde edin. Ekstrüde lipozomları 4 °C'de ve Rhod-PE içeriyorlarsa karanlıkta saklayın. Lipozomları 2 gün içinde kullanın.
| Lipid bileşimi (mol/mol) | Lipid | |||||
| Lipozom adı | DOPC (25 mg/mL) | ÇIKAR (10 mg/mL) | 16:0 Liss Rhod-PE (1 mg/mL) | C16:0/C16:0-PI(4)P (1 mg/mL) | ||
| Ekstraksiyon tahlilleri | Lipozom 2 mol% PS | PC/PS 98/2 | 247 μL | 12,5 μL | ||
| Lipozom 2 mol% PI(4)P | PC/PI(4)P 98/2 | 247 μL | 153 μL | |||
| PC lipozom | PC 100 | 252 μL | ||||
| Taşıma tahlilleri | LA | PC/PS/Rhod-PE 93/5/2 | 234 μL | 31,4 μL | 200 μL | |
| PS'siz LA | PC/Rhod-PE 98/2 | 247 μL | 200 μL | |||
| LB | PC/PI(4)P 95/5 | 237 μL | 383 μL | |||
| PI(4)P olmadan LB | PC 100 | 252 μL | ||||
| LA-Eq | PC/PS/PI(4)P/Rhod-PE 93/2.5/2.5/2 | 234 μL | 15,7 μL | 200 μL | 191 μL | |
| LB-Eq | PC/PS/PI(4)P 95/2.5/2.5 | 239 μL | 15,7 μL | 191 μL | ||
Tablo 1: Lipozom hazırlığı için karıştırılacak lipid stok çözeltilerinin hacimleri. Kısaltmalar: PS= fosfatidiylserine; PC = fosfatidilin; PI(4)P = fosfatidiylinositol 4-fosfat; Rhod-PE = rhodamin etiketli fosfatidylethanolamine; DOPC = dioleoylphosphatidylcholine; POPS= 1-palmitoyl-2-oleoyl-sn-glisro-3-fosfo-L-serine; 16:0 Liss Rhod-PE = 1,2-dipalmitoyl-sn-glisero-3-foseptosanolamin-N-(lissamine rhodamine B sulfonyl).
4. PS veya PI ölçümü(4)P ekstraksiyonu
NOT: Ölçümler siyah bir 96 kuyu plakası ve tek renklilerle donatılmış bir floresan plaka okuyucu kullanılarak yapılmalıdır: biri floresan çıkarma ve diğeri emisyon için, değişken bant genişliğine sahip.
- 1 mM MgCl2 (HKM tampon) ile desteklenmiş taze, filtrelenmiş ve gazdan arındırılmış HK tamponu hazırlayın. 2 mol% PS veya 2 mol% PI (4) P (4 mM son lipit konsantrasyonu, bkz. Tablo 1)ile doped saf PC lipozomları ve PC lipozomları hazırlayın.
NOT: Ekstrüde lipozomların süspansiyonu ile dolu tüpleri deney boyunca oda sıcaklığında tutun ve proteinleri buzda tutun. Ayrıca, lipid sensörlerini ışıktan koruyun. - PS ekstraksiyon tahlilleri için, bir kuyuda, 2 mol% PS (80 μM nihai lipid konsantrasyonu, 0,8 μM erişilebilir PS konsantrasyonu) içeren lipozomları 100 μL'lik son hacimde NBD-C2Lakt (250 nM son konsantrasyon) ile karıştırın. İkinci bir kuyuyu aynı miktarda lipozom (80 μM, %2 mol PS) ve NBD-C2Lact (250 nM) ile karıştırılmış 3 μM LTP (pozitif kontrol veya ilgi proteini olarak Osh6p) ile doldurun.
NOT: Lipid ekstraksiyonu için Osh6p için 5 dakikalık bir kuluçka süresi yeterlidir. - Üçüncü bir kuyuyu saf PC lipozomları (80 μM) ile karıştırılan NBD-C2Lact (250 nM) ile doldurun. Dördüncü bir kuyuyu sadece saf PC lipozomları (80 μM) ile doldurun. Dört kuyudan oluşan üç ek seri hazırlamak için 4.2-4.3 adımlarını tekrarlayın.
- Her kuyu için, 25 °C'de 490 nm'de (bant genişliği 5 nm) heyecanlanma üzerine 505 ila 650 nm (bant genişliği 5 nm) arasında bir NBD spektrumu kaydedin. Her seri için, diğer spektrumdan sadece lipozomlarla kaydedilen spektrumu çıkarın.
NOT: F ve Fmax, sırasıyla LTP varlığında veya yokluğunda PS içeren lipozomlarla ölçülen 536 nm'deki yoğunluğa karşılık gelirken, F0 saf PC lipozomları ile aynı dalga boyundaki yoğunluktadır. Her seri için, protein tarafından çıkarılan erişilebilir PS yüzdesi aşağıdaki formül kullanılarak verilir.
100 × (1-(((F-F0)/(Fmaks .-F0)) (4) - PI(4)P ekstraksiyon tahlilleri için% 2 mol PI(4)P ile dopdlanmış lipozomlar hazırlayın ve NBD-PHFAPP probu ile ölçümler yapın. Kontrol deneyleri yapın ve ayıklama yüzdesini yukarıda açıklandığı gibi belirleyin.
NOT: Lipozom ve protein konsantrasyonu PS ekstraksiyon testinde kullanılanlarla aynıdır.
5. PS taşımacılığının gerçek zamanlı ölçümü
NOT: Lipid transfer kinetiğini kaydetmek için sıcaklık kontrollü bir hücre tutucu ve manyetik karıştırıcı ile donatılmış standart bir florometre (90° formatında) kullanılır. Verileri doğru bir şekilde elde etmek için, numuneyi aynı sıcaklıkta kalıcı olarak tutmak (proteinin kaynağına(örneğin,maya veya insan) bağlı olarak 25 ila 37 ° C arasında ayarlayın) ve sürekli karıştırmak önemlidir. Aşağıda açıklanan protokol, silindirik kuvars hücrede bulunan 600 μL'lik bir numunede lipid taşınmasının ölçümü içindir.
- Yeni gazdan arındırılmış ve filtrelenmiş HKM tamponu hazırlayın. Ekstrüde lipozom içeren tüpleri oda sıcaklığında tutun. Lipozom içeren tüpleri alüminyum folyoya Rhod-PE ile sarın ve/veya fotobleachingi önlemek için opak bir kutuda saklayın.
- Heyecan ve emisyon monokronatörlerini sırasıyla φ = 460 nm (kısa bant genişliği (1-3 nm) ve φ = 530 nm (büyük bant genişliği (≥ 10 nm) olarak ayarlayın. 1 sn ≤ bir zaman çözünürlüğü ile alım süresini 25 dakika olarak ayarlayın.
- Kuvars cuvette, 200 μM toplam lipit ve 250 nM NBD-C2Lakt içeren 570 μL numune hazırlamak için önceden ısıtılan HKM tamponunda LA lipozom süspansiyonunun 30 μL'lik kısmını ve bir hacim NBD-C2Lakt stok çözeltisini seyreltin. Küçük bir manyetik karıştırma çubuğu ekleyin ve cuvette'i florometre tutucusuna yerleştirin.
- Numune termal olarak dengelendikten sonra (3-5 dakika sonra), ölçümü tetikleyin. 1 dakika sonra, numuneye 30 μL LB lipozom süspansiyonu (200 μM toplam lipitlerin son konsantrasyonu) ekleyin. 3 dakika sonra, LTP'nin son konsantrasyonu 200 nM olacak şekilde numuneye LTP enjekte edin ve kalan 21 dakika için sinyali alın.
- NBD sinyalini normalleştirmek için paralel bir deney gerçekleştirin. HKM tamponunda 30 μL LA-Eq lipozom süspansiyonu 250 nM NBD-C2Lact ile karıştırın (son hacim 570 μL). 1 dakika sonra, 30 μL LB-Eq lipozom süspansiyonu enjekte edin.
NOT: LA-Eq veL-Eq lipozomlarının lipid bileşimi, transfer testinde kullanılan LA ve LB lipozomlarına benzer, ancak her biri 2.5 mol% PS ve 2.5 mol% PI(4)P içerir. Sonuç olarak, FEqolarak adlandırılan NBD sinyali, PS bir aktarım işlemi ile LA ve LB lipozomları arasında tamamen dengelenmişse ölçülmesi gereken sinyale karşılık gelir. - Zaman içinde LA'dan LB lipozomlarına aktarılan PS miktarını (μM olarak) belirlemek için ltp ile ölçülen kinetik eğrileri dönüştürün. Aşağıdaki formülü kullanarak eğrinin her veri noktasını (F) normalleştirin.
FNorm = (F-F0)/(FEq-F0) (5)
F0'ın LTP'nin eklenmesinden hemen önce NBD sinyaline karşılık geldiği ve FEq'nin 5.5.
NOT: LA'dan LB lipozomlarına aktarılan PS miktarı (μM cinsinden) 2,5 × FNormunakarşılık gelir Dengenin erişilebilir PS moleküllerinin yarısının bulunduğu bir duruma karşılık geldiği düşünüldüğünde, LA lipozomlarının dış broşüründe bulunan (yani,0.5'in% 5'ine karşılık gelen × 200 μM toplam lipitler) LB lipozomlarına aktarılmıştır.
6. PI(4)P taşımacılığının gerçek zamanlı ölçümü
- PS aktarım tahlili için yapıldığı gibi flormetreyi (heyecan ve emisyon dalga boyu, bant genişliği, alım süresi, zaman çözünürlüğü) ayarlayın. Aynı şekilde, deneyleri aynı sıcaklıkta sürekli karıştırma altında gerçekleştirmek için aynı tampon, cuvette ve lipozomları kullanın.
- Cuvette, 30 μL LB lipozom süspansiyonu ve NBD-PHFAPP'ı önceden ısıtılan HKM tamponu ile karıştırarak 570 μL 'lik (200 μM toplam lipit, 250 nM NBD- PHFAPP)son bir hacim elde edin. Numunenin termal dengeye ulaştığında, ölçüme başlayın ve 1 dakika sonra 30 μL LA lipozom süspansiyonu enjekte edin. 3 dakika sonra, LTP'yi (200 nM'lik son konsantrasyon) enjekte edin ve sinyali kaydedin.
- NBD sinyalini normalleştirmek için ikinci bir deneme gerçekleştirin. 30 μL LB-Eq lipozom süspansiyonunu 570 μL HKM tamponda 250 nM NBD-PHFAPP ile karıştırın. 1 dakika sonra, 30 μL LA-Eq lipozom süspansiyonu enjekte edin.
NOT: Burada, FEqolarak adlandırılan NBD sinyali, PI(4)P'nin LA ve LB lipozomları arasında tamamen dengelenmiş olması durumunda ölçülmesi gereken sinyale karşılık gelir. - Zaman içinde LB'den LA lipozomlarına aktarılan PI(4)P (μM olarak) miktarını belirlemek için kinetik eğrileri dönüştürün. Her veri noktası (F), F0'ın LTP eklenmeden önce NBD sinyaline karşılık geldiği formül 5 kullanılarak normalleştirilir ve FEq, adım 6.3'te ölçülen sinyaldir.
NOT: LB'den LA lipozomlarına aktarılan PI(4)P (μM cinsinden) miktarı 2,5 × FNorm'akarşılık gelir Dengenin, LB lipozomlarının dış broşüründe bulunan PI(4)P'nin yarısının bulunduğu bir duruma karşılık geldiği düşünüldüğünde (yani, LA lipozomlarında 0,5 × 5 μM) aktarılmıştır.
7. Kinetik eğrilerinin analizi
- Bu LTP'nin cuvette'e enjeksiyonunu izleyen ilk birkaç saniye içinde lipitleri bir lipozom popülasyonundan diğerine transfer etme hızını belirleyerek ltp'nin ne ölçüde verimli olduğunu ölçün.
- Eğim elde etmek için aktarım kinetiğinin ilk veri noktalarının doğrusal bir gerilemesini gerçekleştirin. Zaman birimi başına protein başına aktarılan lipit moleküllerinin sayısını belirlemek için eğim değerini reaksiyon karışımındaki LTP konsantrasyonuna bölün (min veya s).
Sonuçlar
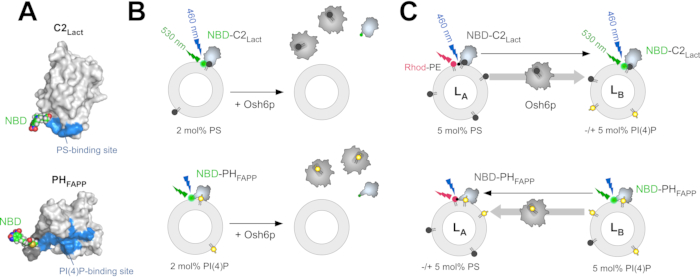
Şekil 1: Floresan lipit sensörlerinin ve in vitro tahlillerin açıklaması. (A) Sığır laktadherin C2 etki alanının kristal yapısına (PDB ID: 3BN648)ve insanFAPP1 proteininin PH alanının NMR yapısına dayanan NBD-C2Lact ve NBD-PH FAPP'ın üç boyutlu modelleri (PDB ID: 2KCJ
Tartışmalar
Bu tahlillerin sonuçları doğrudan floresan lipid sensörlerinin sinyallerine dayanır. Bu nedenle, NBD ile 1:1 oranında etiketlenen ve serbest NBD florofor kontaminasyonu olmadan etiketlenen bu probların saflaştırılması bu protokolde kritik bir adımdır. Muayene edilen LTP'nin düzgün bir şekilde katlanıp katlanmadığını ve toplanmadığını kontrol etmek de zorunludur. Bu LTP'nin bu lipitleri verimli bir şekilde çıkarıp çıkarmadığını düzgün bir şekilde ölçmek için ekstraksiyon testlerind...
Açıklamalar
Yazarlar çıkar çatışması olmadığını beyan eder.
Teşekkürler
Dr. A. Cuttriss'e el yazmasını dikkatli bir şekilde düzelttikleri için minnettarız. Bu çalışma Fransız Ulusal Araştırma Ajansı hibe ExCHANGE (ANR-16-CE13-0006) ve CNRS tarafından finanse edilmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| L-cysteine ≥97 % (FG) | Sigma | W326305-100G | Prepare a 10 mM L-cysteine stock solution in water. Aliquots are stored at -20 °C |
| 2 mL Amber Vial, PTFE/Rub Lnr, for lipids storage in CHCL3 | Wheaton | W224681 | |
| 4 mm-diameter glass beads | Sigma | Z265934-1EA | |
| 50 mL conical centrifuge tube | Falcon | ||
| ÄKTA purifier | GE healthcare | FPLC | |
| Aluminium foil | |||
| Amicon Ultra-15 with a MWCO of 3 and 10 kDa | Merck | UFC900324, UFC901024 | |
| Amicon Ultra-4 with a MWCO of 3 and 10 kDa | Merck | UFC800324, UFC801024 | |
| Ampicillin | Prepare a 50 mg/mL stock solution with filtered and sterilized water and store it at -20 °C. | ||
| Bestatin | Sigma | B8385-10mg | |
| BL21 Gold Competent Cells | Agilent | ||
| C16:0 Liss (Rhod-PE) in CHCl3 (1 mg/mL) | Avanti Polar Lipids | 810158C-5MG | |
| C16:0/C16:0-PI(4)P | Echelon Lipids | P-4016-3 | Dissolve 1 mg of C16:0/C16:0-PI(4)P powder in 250 µL of MeOH and 250 µL of CHCl3. Then complete with CHCl3 to 1 mL. The solution must become clear. |
| C16:0/C18:1-PS (POPS) in CHCl3 (10 mg/mL) | Avanti Polar Lipids | 840034C-25mg | |
| C18:1/C18:1-PC (DOPC) in CHCl3 (25 mg/mL) | Avanti Polar Lipids | 850375C-500mg | |
| CaCl2 | Sigma | Prepare 10 mM CaCl2 stock solution in water. | |
| Cell Disruptor | Constant Dynamics | ||
| Chloroform (CHCl3) RPE-ISO | Carlo Erba | 438601 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 5056489001 | |
| Deionized (Milli-Q) water | |||
| Dimethylformamide (DMF), anhydrous, >99% pure | |||
| DNAse I Recombinant, RNAse free, in powder | Roche | 10104159001 | |
| DTT | Euromedex | EU0006-B | Prepare 1 M DTT stock solution in Milli-Q water. Prepare 1 mL aliquots and store them at -20 °C. |
| Econo-Pac chromatography columns (1.5 × 12 cm). | Biorad | 7321010 | |
| Electroporation cuvette 2 mm | Ozyme | EP102 | |
| Electroporator Eppendorf 2510 | Eppendorf | ||
| Fixed-Angle Rotor Ti45 and Ti45 tubes | Beckman | Spinning the batcerial lysates | |
| Glass-syringes (10, 25, and 50 µL) for fluorescence experiment | Hamilton | ||
| Glass-syringes (25 , 100, 250, 500, and 1000 µL) to handle lipid stock solutions | Hamilton | 1702RNR, 1710RNR, 1725RNR, 1750RN type3, 1001RN | |
| Glutathione Sepharose 4B beads | GE Healthcare | 17-0756-05 | |
| Glycerol (99% pure) | Sigma | G5516-500ML | |
| Hemolysis tubes with a cap | |||
| HEPES , >99 % pure | Sigma | H3375-500G | |
| Illustra NAP 10 desalting column | GE healthcare | GE17-0854-02 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Euromedex | EU0008-B | Prepare 1 M IPTG stock solution in Milli-Qwater. Prepare 1 mL aliquots and store them at -20 °C. |
| K-Acetate | Prolabo | 26664.293 | |
| Lennox LB Broth medium without glucose | Prepared with milli-Q water and autoclaved. | ||
| Liquid nitrogen | Linde | ||
| Methanol (MeOH) ≥99.8% | VWR | 20847.24 | |
| MgCl2 | Sigma | Prepare a 2 M MgCl2 solution. Filter the solution using a 0.45 µm filter. | |
| Microplate 96 Well PS F-Botom Black Non-Binding | Greiner Bio-one | 655900 | |
| Mini-Extruder with two 1 mL gas-tight Hamilton syringes | Avanti Polar Lipids | 610023 | |
| Monochromator-based fluorescence plate reader | TECAN | M1000 Pro | |
| N,N'-Dimethyl-N-(Iodoacetyl)-N'-(7-Nitrobenz-2-Oxa-1,3-Diazol-4-yl)Ethylenediamine) (IANBD Amide) | Molecular Probes | Dissolve 25 mg of IANBD in 2.5 mL of dimethylsulfoxide (DMSO) and prepare 25 aliquot of 100 µL in 1.5 mL screw-cap tubes. Do not completely screw the cap. Then, remove DMSO in a freeze-dryer to obtain 1 mg of dry IANBD per tube. Tubes are closed and stored at -20 °C in the dark. | |
| NaCl | Sigma | S3014-1KG | |
| PBS | 137 mM NaCl, 2.7 mM KCl, 10 mM NaH2PO4, 1.8 mM KH2PO4, autoclaved and stored at 4 °C. | ||
| Pear-shaped glass flasks (25 mL, 14/23, Duran glass) | Duran Group | ||
| Pepstatin | Sigma | p5318-25mg | |
| pGEX-C2LACT plasmid | Available on request from our lab | ||
| pGEX-PHFAPP plasmid | Available on request from our lab | ||
| Phenylmethylsulfonyl fluoride (PMSF) ≥98.5% (GC) | Sigma | P7626-25g | Prepare a 200 mM PMSF stock solution in isopropanol |
| Phosphoramidon | Sigma | R7385-10mg | |
| Polycarbonate filters (19 mm in diameter) with pore size of 0.2 µm | Avanti Polar Lipids | 610006 | |
| Poly-Prep chromatography column (with a 0-2 mL bed volume and a 10 mL reservoir) | Biorad | 7311550 | |
| Prefilters (10 mm in diameter). | Avanti Polar Lipids | 610014 | |
| PyMOL | http://pymol.org/ | Construction of the 3D models of the proteins (Figure 1A) | |
| Quartz cuvette for UV/visible fluorescence (minimum volume of 600 µL) | Hellma | ||
| Quartz cuvettes | Hellma | ||
| Refrigerated centrifuge Eppendorf 5427R | Eppendorf | ||
| Rotary evaporator | Buchi | B-100 | |
| Screw-cap microcentriguge tubes (1.5 mL) | Sarsted | ||
| Small magnetic PFTE stirring bar (5 × 2 mm) | |||
| Snap-cap microcentriguge tubes (0.5, 1, and 2 mL) | Eppendorf | ||
| SYPRO orange | fluorescent stain to detect protein in SDS-PAGE gel | ||
| Thermomixer | Starlab | ||
| THROMBIN, FROM HUMAN PLASMA | Sigma | 10602400001 | Dissolve 20 units in 1 mL of milli-Q water and prepare 25 µL aliquots in 0.5 mL Eppendorf tubes. Then freeze and store at -80 °C. |
| Tris, ultra pure | MP | 819623 | |
| Ultracentrifuge L90K | Beckman | ||
| UV/Visible absorbance spectrophotometer | SAFAS | ||
| UV/visible spectrofluorometer with a temperature-controlled cell holder and stirring device | Jasco or Shimadzu | Jasco FP-8300 or Shimadzu RF-5301PC | |
| Vacuum chamber | |||
| Water bath | Julabo | ||
| XK 16/70 column packed with Sephacryl S200HR | GE healthcare |
Referanslar
- Drin, G. Topological regulation of lipid balance in cells. Annual Review of Biochemistry. 83, 51-77 (2014).
- Bigay, J., Antonny, B. Curvature, lipid packing, and electrostatics of membrane organelles: defining cellular territories in determining specificity. Developmental Cell. 23 (5), 886-895 (2012).
- Prinz, W. A. Lipid trafficking sans vesicles: where, why, how. Cell. 143 (6), 870-874 (2010).
- Holthuis, J. C., Menon, A. K. Lipid landscapes and pipelines in membrane homeostasis. Nature. 510 (7503), 48-57 (2014).
- Wong, L. H., Copic, A., Levine, T. P. Advances on the transfer of lipids by lipid transfer proteins. Trends in Biochemical Sciences. 42 (7), 516-530 (2017).
- Wong, L. H., Gatta, A. T., Levine, T. P. Lipid transfer proteins: the lipid commute via shuttles, bridges and tubes. Nature Reviews Molecular Cell Biology. 20 (2), 85-101 (2019).
- Iaea, D. B., Dikiy, I., Kiburu, I., Eliezer, D., Maxfield, F. R. STARD4 membrane interactions and sterol binding. Biochemistry. 54 (30), 4623-4636 (2015).
- Wilhelm, L. P., et al. STARD3 mediates endoplasmic reticulum-to-endosome cholesterol transport at membrane contact sites. The EMBO Journal. 36 (10), 1412-1433 (2017).
- Bian, X., Saheki, Y., De Camilli, P. Ca(2+) releases E-Syt1 autoinhibition to couple ER-plasma membrane tethering with lipid transport. The EMBO Journal. 37 (2), 219-234 (2018).
- Horenkamp, F. A., Valverde, D. P., Nunnari, J., Reinisch, K. M. Molecular basis for sterol transport by StART-like lipid transfer domains. The EMBO Journal. 37 (6), 98002 (2018).
- Jentsch, J. A., et al. Structural basis of sterol binding and transport by a yeast StARkin domain. The Journal of Biological Chemistry. 293 (15), 5522-5531 (2018).
- Moser von Filseck, J., et al. INTRACELLULAR TRANSPORT. Phosphatidylserine transport by ORP/Osh proteins is driven by phosphatidylinositol 4-phosphate. Science. 349 (6246), 432-436 (2015).
- Daum, G., et al. Systematic analysis of yeast strains with possible defects in lipid metabolism. Yeast. 15 (7), 601-614 (1999).
- Ejsing, C. S., et al. Global analysis of the yeast lipidome by quantitative shotgun mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2136-2141 (2009).
- Leidl, K., Liebisch, G., Richter, D., Schmitz, G. Mass spectrometric analysis of lipid species of human circulating blood cells. Biochimica et Biophysica Acta. 1781 (10), 655-664 (2008).
- Sampaio, J. L., et al. Membrane lipidome of an epithelial cell line. Proceedings of the National Academy of Sciences of the United States of America. 108 (5), 1903-1907 (2011).
- Vance, J. E., Steenbergen, R. Metabolism and functions of phosphatidylserine. Progress in Lipid Research. 44 (4), 207-234 (2005).
- Zinser, E., et al. Phospholipid synthesis and lipid composition of subcellular membranes in the unicellular eukaryote Saccharomyces cerevisiae. Journal of Bacteriology. 173 (6), 2026-2034 (1991).
- Leventis, P. A., Grinstein, S. The distribution and function of phosphatidylserine in cellular membranes. Annual Review of Biophysics. 39, 407-427 (2010).
- Yeung, T., et al. Membrane phosphatidylserine regulates surface charge and protein localization. Science. 319 (5860), 210-213 (2008).
- Kim, J., Shishido, T., Jiang, X., Aderem, A., McLaughlin, S. Phosphorylation, high ionic strength, and calmodulin reverse the binding of MARCKS to phospholipid vesicles. Journal of Biological Chemistry. 269 (45), 28214-28219 (1994).
- Sigal, C. T., Zhou, W., Buser, C. A., McLaughlin, S., Resh, M. D. Amino-terminal basic residues of Src mediate membrane binding through electrostatic interaction with acidic phospholipids. Proceedings of the National Academy of Sciences. 91 (25), 12253-12257 (1994).
- Gal Bivona, T., et al. PKC regulates a farnesyl-electrostatic switch on K-Ras that promotes its association with Bcl-XL on mitochondria and induces apoptosis. Molecular Cell. 21 (4), 481-493 (2006).
- Finkielstein, C. V., Overduin, M., Capelluto, D. G. Cell migration and signaling specificity is determined by the phosphatidylserine recognition motif of Rac1. The Journal of Biological Chemistry. 281 (37), 27317-27326 (2006).
- Bolsover, S. R., Gomez-Fernandez, J. C., Corbalan-Garcia, S. Role of the Ca2+/Phosphatidylserine Binding Region of the C2 Domain in the Translocation of Protein Kinase Cα to the Plasma Membrane. Journal of Biological Chemistry. 278 (12), 10282-10290 (2003).
- Vance, J. E., Tasseva, G. Formation and function of phosphatidylserine and phosphatidylethanolamine in mammalian cells. Biochimica et Biophysica Acta. 1831 (3), 543-554 (2013).
- Maeda, K., et al. Interactome map uncovers phosphatidylserine transport by oxysterol-binding proteins. Nature. 501 (7466), 257-261 (2013).
- D'Ambrosio, J. M., et al. Osh6 requires Ist2 for localization to ER-PM contacts and efficient phosphatidylserine transport in budding yeast. Journal of Cell Science. 133 (11), 243733 (2020).
- Manford, A. G., Stefan, C. J., Yuan, H. L., Macgurn, J. A., Emr, S. D. ER-to-plasma membrane tethering proteins regulate cell signaling and ER morphology. Developmental Cell. 23 (6), 1129-1140 (2012).
- Collado, J., et al. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- Hoffmann, P. C., et al. Tricalbins contribute to cellular lipid flux and form curved ER-PM contacts that are bridged by rod-shaped structures. Developmental Cell. 51 (4), 488-502 (2019).
- Lipp, N. F., et al. An electrostatic switching mechanism to control the lipid transfer activity of Osh6p. Nature Communications. 10 (1), 3926 (2019).
- Chung, J., et al. INTRACELLULAR TRANSPORT. PI4P/phosphatidylserine countertransport at ORP5- and ORP8-mediated ER-plasma membrane contacts. Science. 349 (6246), 428-432 (2015).
- Sohn, M., et al. PI(4,5)P2 controls plasma membrane PI4P and PS levels via ORP5/8 recruitment to ER-PM contact sites. The Journal of Cell Biology. 217 (5), 1797-1813 (2018).
- Ghai, R., et al. ORP5 and ORP8 bind phosphatidylinositol-4, 5-biphosphate (PtdIns(4,5)P 2) and regulate its level at the plasma membrane. Nature Communications. 8 (1), 757 (2017).
- Sohn, M., et al. Lenz-Majewski mutations in PTDSS1 affect phosphatidylinositol 4-phosphate metabolism at ER-PM and ER-Golgi junctions. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4314-4319 (2016).
- Kattan, W. E., et al. Targeting plasma membrane phosphatidylserine content to inhibit oncogenic KRAS function. Life Science Alliance. 2 (5), 00431 (2019).
- Du, X., Turner, N., Yang, H. The role of oxysterol-binding protein and its related proteins in cancer. Seminars in Cell & Developmental Biology. 81, 149-153 (2018).
- Galmes, R., et al. ORP5/ORP8 localize to endoplasmic reticulum-mitochondria contacts and are involved in mitochondrial function. EMBO reports. 17 (6), 800-810 (2016).
- Du, X., et al. ORP5 localizes to ER-lipid droplet contacts and regulates the level of PI(4)P on lipid droplets. The Journal of Cell Biology. 219 (1), 201905162 (2020).
- Wang, H., et al. ORP2 delivers cholesterol to the plasma membrane in exchange for phosphatidylinositol 4, 5-bisphosphate (PI(4,5)P2). Molecular Cell. 73 (3), 458-473 (2019).
- Kay, J. G., Grinstein, S. Sensing phosphatidylserine in cellular membranes. Sensors (Basel). 11 (2), 1744-1755 (2011).
- Moser von Filseck, J., Vanni, S., Mesmin, B., Antonny, B., Drin, G. A phosphatidylinositol-4-phosphate powered exchange mechanism to create a lipid gradient between membranes. Nature Communications. 6, 6671 (2015).
- Wills, R. C., Goulden, B. D., Hammond, G. R. V. Genetically encoded lipid biosensors. Molecular Biology of the Cell. 29 (13), 1526-1532 (2018).
- Raychaudhuri, S., Im, Y. J., Hurley, J. H., Prinz, W. A. Nonvesicular sterol movement from plasma membrane to ER requires oxysterol-binding protein-related proteins and phosphoinositides. The Journal of Cell Biology. 173 (1), 107-119 (2006).
- Lenoir, M., et al. Structural basis of wedging the Golgi membrane by FAPP pleckstrin homology domains. EMBO reports. 11 (4), 279-284 (2010).
- Liu, Y., Kahn, R. A., Prestegard, J. H. Interaction of Fapp1 with Arf1 and PI4P at a membrane surface: an example of coincidence detection. Structure. 22 (3), 421-430 (2014).
- Shao, C., Novakovic, V. A., Head, J. F., Seaton, B. A., Gilbert, G. E. Crystal structure of lactadherin C2 domain at 1.7A resolution with mutational and computational analyses of its membrane-binding motif. The Journal of Biological Chemistry. 283 (11), 7230-7241 (2008).
- Lipp, N. F., Ikhlef, S., Milanini, J., Drin, G. Lipid exchangers: cellular functions and mechanistic links with phosphoinositide metabolism. Frontiers in Cell and Developmental Biology. 8, 663 (2020).
- Venditti, R., et al. Molecular determinants of ER-Golgi contacts identified through a new FRET-FLIM system. The Journal of Cell Biology. 218 (3), 1055-1065 (2019).
- Pemberton, J. G., et al. Defining the subcellular distribution and metabolic channeling of phosphatidylinositol. The Journal of Cell Biology. 219 (3), (2020).
- Nakanishi, H., de los Santos, P., Neiman, A. M. Positive and negative regulation of a SNARE protein by control of intracellular localization. Molecular Biology of the Cell. 15 (4), 1802-1815 (2004).
- Maekawa, M., Yang, Y., Fairn, G. D. Perfringolysin O theta toxin as a tool to monitor the distribution and inhomogeneity of cholesterol in cellular membranes. Toxins. 8 (3), 67 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır