È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazioni basate sulla fluorescenza dello scambio di fosfatidilserina / fosfatidilinositolo 4-fosfato tra le membrane
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, descriviamo protocolli che utilizzano sensori lipidici fluorescenti e liposomi per determinare se una proteina estrae e trasporta fosfatidilserina o fosfatidilinositolo 4-fosfato in vitro.
Abstract
Diversi membri della famiglia delle proteine leganti l'ossisterolo (OSBP) (ORP) / OSBP (Osh) conservate evolutivamente sono stati recentemente trovati per rappresentare un nuovo gruppo di proteine di trasferimento lipidico (LTP) nel lievito e nelle cellule umane. Trasferiscono la fosfatidilserina (PS) dal reticolo endoplasmatico (ER) alla membrana plasmatica (PM) tramite cicli di scambio PS/fosfatidilinositolo 4-fosfato (PI(4)P). Questa scoperta consente una migliore comprensione di come la PS, che è fondamentale per i processi di segnalazione, è distribuita in tutta la cellula e lo studio del legame tra questo processo e il metabolismo dei fosfoinositidi (PIP). Lo sviluppo di nuovi protocolli basati sulla fluorescenza è stato determinante nella scoperta e caratterizzazione di questo nuovo meccanismo cellulare in vitro a livello molecolare. Questo documento descrive la produzione e l'uso di due sensori lipidici marcati fluorescentemente, NBD-C2Lact e NBD-PHFAPP, per misurare la capacità di una proteina di estrarre PS o PI(4)P e di trasferire questi lipidi tra membrane artificiali. In primo luogo, il protocollo descrive come produrre, etichettare e ottenere campioni ad alta purezza di questi due costrutti. In secondo luogo, questo documento spiega come utilizzare questi sensori con un lettore di micropiasche a fluorescenza per determinare se una proteina può estrarre PS o PI(4)P dai liposomi, utilizzando Osh6p come caso di studio. Infine, questo protocollo mostra come misurare con precisione la cinetica dello scambio PS/PI(4)P tra liposomi di composizione lipidica definita e determinare le velocità di trasferimento lipidico mediante trasferimento di energia di risonanza a fluorescenza (FRET) utilizzando un fluorometro standard.
Introduzione
La distribuzione precisa dei lipidi tra le diverse membrane e all'interno delle membrane delle cellule eucariotiche1,2 ha profonde implicazioni biologiche. Decifrare come funzionano le LTP è un problema importante nella biologia cellulare3,4,5,6e gli approcci in vitro sono di grande valore nell'affrontare questo problema7,8,9,10,11. Qui, viene presentata una strategia in vitro,basata sulla fluorescenza, che è stata determinante nello stabilire che diverse proteine ORP / Osh influenzano lo scambio PS / PI (4) P tra le membrane cellulari12 e quindi costituiscono una nuova classe di LTP. PS è un glicerofosfolipide anionico che rappresenta il 2-10% dei lipidi totali di membrana nelle cellule eucariotiche13,14,16. È distribuito lungo un gradiente tra l'ER e il PM, dove rappresenta il 5-7% e fino al 30% dei glicerofosfolipidi, rispettivamente17,18,19. Inoltre, la PS è essenzialmente concentrata nel foglietto citosolico del PM. Questo accumulo e la partizione irregolare di PS nel PM sono fondamentali per i processi di segnalazione cellulare19. A causa della carica negativa delle molecole di PS, il foglietto citosolico del PM è molto più anionico del foglietto citosolico di altri organelli1,2,19,20. Ciò consente il reclutamento, tramite forze elettrostatiche, di proteine di segnalazione come il substrato C-chinasi ricco di alanina miristoilata (MARCKS)21, sarcoma (Src)22, l'oncogene virale del sarcoma kirsten-ratto (K-Ras)23e il substrato della tossina botulinica C3 correlato a Ras 1 (Rac1)24 che contengono un tratto di aminoacidi caricati positivamente e una coda lipidica.
Ps è anche riconosciuta dalla proteina chinasi C convenzionale in modo stereoselettivo tramite un dominio C225. Tuttavia, PS è sintetizzato in ER26, indicando che deve essere esportato nel PM prima che possa svolgere il suo ruolo. Non si sapeva come ciò fosse stato realizzato19 fino alla scoperta che, nel lievito, Osh6p e Osh7p trasferiscono PS dal ER al PM27. Questi LTP appartengono a una famiglia evolutivamente conservata negli eucarioti il cui membro fondatore è OSBP e che contiene proteine (ORP nell'uomo, proteine dell'Osh nel lievito) che integrano un dominio correlato all'OSBP (ORD) con una tasca per ospitare una molecola lipidica. Osh6p e Osh7p sono costituiti solo da un ORD le cui caratteristiche strutturali sono adattate per legare specificamente PS e trasferirlo tra le membrane. Tuttavia, non era chiaro come queste proteine trasferivano direzionalmente il PS dall'ER al PM. Osh6p e Osh7p possono intrappolare PI(4)P come ligando lipidico alternativo12. Nel lievito, PI(4)P è sintetizzato dal fosfatidilinositolo (PI) nel Golgi e nel PM da PI 4-chinasi, Pik1p e Stt4p, rispettivamente. Al contrario, non c'è PI(4)P nella membrana ER, poiché questo lipide viene idrolizzato in PI dalla fosfatasi Sac1p. Quindi, un gradiente PI(4)P esiste in entrambe le interfacce ER/Golgi ed ER/PM. Osh6p e Osh7p trasferiscono PS dall'ER al PM tramite cicli di scambio PS/PI(4)P utilizzando il gradiente PI(4)P che esiste tra queste due membrane12.
Entro un ciclo, Osh6p estrae PS dall'ER, scambia PS per PI(4)P al PM e trasferisce PI(4)P all'ER per estrarre un'altra molecola di PS. Osh6p/Osh7p interagiscono con Ist2p28, una delle poche proteine che si connettono e portano la membrana ER e il PM in stretta vicinanza tra loro per creare siti di contatto ER-PM nel lievito29,30,31. Inoltre, l'associazione di Osh6p con membrane caricate negativamente diventa debole non appena la proteina estrae uno dei suoi ligandi lipidici a causa di un cambiamento conformazionale che modifica le sue caratteristiche elettrostatiche32. Questo aiuta Osh6p accorciando il tempo di permanenza della membrana, mantenendo così l'efficienza della sua attività di trasferimento lipidico. Combinato con il legame a Ist2p, questo meccanismo potrebbe consentire a Osh6p / 7p di eseguire rapidamente e con precisione lo scambio lipidico all'interfaccia ER / PM. Nelle cellule umane, le proteine ORP5 e ORP8 eseguono lo scambio PS/PI(4)P nei siti di contatto ER-PM attraverso meccanismi distinti33. Hanno un ORD centrale, simile a Osh6p, ma sono direttamente ancorati all'ER tramite un segmento transmembranaC-terminale 33 e si agganciano al PM tramite un dominio di omologia Pleckstrin (PH) N-terminale che riconosce PI(4)P e PI(4,5)P233,34,35. ORP5/8 utilizza PI(4)P per trasferire PS, ed è stato dimostrato che ORP5/8 regola ulteriormente i livelli di PM PI(4,5)P2 e presumibilmente modula le vie di segnalazione. A sua volta, una diminuzione dei livelli di PI(4)P e PI(4,5)P2 abbassa l'attività di ORP5/ORP8 poiché queste proteine si associano al PM in modo dipendente dal PIP. La sintesi di PS anormalmente alta, che porta alla sindrome di Lenz-Majewski, influisce sui livelli di PI(4)P attraverso ORP5/836. Quando l'attività di entrambe le proteine è bloccata, ps diventa meno abbondante al PM, abbassando la capacità oncogenica di segnalare le proteine37.
Al contrario, la sovraespressione di ORP5 sembra promuovere l'invasione delle cellule tumorali e i processi metastatici38. Pertanto, le alterazioni dell'attività di ORP5/8 possono modificare gravemente il comportamento cellulare attraverso cambiamenti nell'omeostasi lipidica. Inoltre, ORP5 e ORP8 occupano siti di contatto ER-mitocondri e conservano alcune funzioni mitocondriali, possibilmente fornendo PS39. Inoltre, ORP5 si localizza nei siti di contatto delle goccioline lipidiche ER per fornire PS alle goccioline lipidiche mediante scambio PS / PI (4) P40. La strategia qui descritta per misurare (i) l'estrazione di PS e PI(4)P dai liposomi e (ii) il trasporto di PS e PI(4)P tra liposomi è stata ideata per stabilire e analizzare l'attività di scambio PS/PI(4)P di Osh6p/Osh7p12,32 e utilizzata da altri gruppi per analizzare l'attività di ORP5/ORP835 e di altre LTP10, 41. Si basa sull'uso di un lettore di piastre a fluorescenza, uno spettrofluorometro standard in formato L e due sensori fluorescenti, NBD-C2Lact e NBD-PHFAPP, in grado di rilevare rispettivamente PS e PI (4) P.
NBD-C2Lact corrisponde al dominio C2 della glicoproteina, la lattaderina, che è stato riprogettato per includere un'unica cisteina esposta a solvente vicino al presunto sito di legame PS; un fluoroforo NBD sensibile alla polarità (7-nitrobenz-2-oxa-1,3-diazol) è legato covalentemente a questo residuo (Figura 1A)12. Per essere più precisi, il dominio C2 della lattaderina (Bos taurus, UniProt: Q95114,residui 270-427) è stato clonato in un vettore pGEX-4T3 da esprimere in fusione con la glutatione S-transferasi (GST) in Escherichia coli. La sequenza C2Lact è stata quindi mutata per sostituire due residui di cisteina accessibili con solvente (C270, C427) con residui di alanina e per introdurre un residuo di cisteina in una regione vicino al presunto sito di legame con PS (mutazione H352C) che può essere successivamente etichettato con N,N'-dimetil-N-(iodoacetil)-N'-(7-nitrobenz-2-oxa-1,3-diazol-4-il) etilene diammina (IANBD) 12. Un sito di scissione per la trombina è presente tra la proteina GST e l'N-terminus del dominio C2. Un grande vantaggio è che questo dominio riconosce selettivamente PS in modo indipendente da Ca2+contrariamente ad altri domini C2 noti o All'allegato A542. NBD-PHFAPP è derivato dal dominio PH della proteina umana 1 adattatrice di quattro fosfati (FAPP1), che è stata riprogettata per includere una singola cisteina esposta a solventi che può essere etichettata con un gruppo NBD vicino al sito di legame PI(4)P (Figura 1A)43. La sequenza nucleotidica del dominio PH della proteina FAPP umana (UniProt: Q9HB20, segmento [1-100]) è stata clonata in un vettore pGEX-4T3 da esprimere in tandem con un tag GST. La sequenza PHFAPP è stata modificata per inserire un unico residuo di cisteina all'interno dell'interfaccia di legame della membrana della proteina43. Inoltre, è stato introdotto un linker a nove residui tra il sito di scissione della trombina e l'N-terminus del dominio PH per garantire l'accessibilità alla proteasi.
Per misurare l'estrazione di PS dai liposomi, NBD-C2Lact viene miscelato con liposomi costituiti da fosfatidilcolina (PC) contenenti tracce di PS. A causa della sua affinità per PS, questo costrutto si lega ai liposomi e il fluoroforo NBD subisce un cambiamento di polarità quando entra in contatto con l'ambiente idrofobo della membrana, che provoca un blue-shift e un aumento della fluorescenza. Se ps viene estratto quasi completamente da una quantità stechiometrica di LTP, la sonda non si associa ai liposomi e il segnale NBD è inferiore (Figura 1B)32. Questa differenza di segnale viene utilizzata per determinare se un LTP(ad esempio, Osh6p) estrae PS. Una strategia simile viene utilizzata con NBD-PHFAPP per misurare l'estrazione di PI(4)P (Figura 1B), come descritto in precedenza12,32. Due saggi basati su FRET sono stati progettati per (i) misurare il trasporto di PS dai liposomi LA a LB, che imitano rispettivamente la membrana ER e il PM, e (ii) il trasporto PI(4)P nella direzione inversa. Questi test vengono eseguiti nelle stesse condizioni(cioè stesso tampone, temperatura e concentrazione lipidica) per misurare lo scambio PS/PI(4)P. Per misurare il trasporto di PS, NBD-C2Lact viene miscelato con liposomi LA composti da PC e drogati con il 5 mol% di PS e il 2 mol% di una fosfatidiletanolammina fluorescente marcata con rodamina (Rhod-PE) e liposomi LB che incorporano il 5 mol% di PI(4)P.
Al tempo zero, FRET con Rhod-PE spegne la fluorescenza NBD. Se la PS viene trasportata dai liposomi LA a LB (ad esempio, dopo l'iniezione di Osh6p), si verifica un rapido dequenching a causa della traslocazione di molecole NBD-C2Lact dai liposomi LA a LB (Figura 1C). Data la quantità di PS accessibile, NBD-C2Lact rimane essenzialmente in uno stato legato alla membrana nel corso dell'esperimento12. Pertanto, l'intensità del segnale NBD è direttamente correlata alla distribuzione di NBD-C2Lact tra i liposomi LA e LB e può essere facilmente normalizzata per determinare la quantità di PS trasferita. Per misurare il trasferimento di PI(4)P nella direzione opposta, NBD-PHFAPP viene miscelato con liposomi LA e LB; dato che si lega solo ai liposomi LB che contengono PI(4)P, ma non Rhod-PE, la sua fluorescenza è elevata. Se PI(4)P viene trasferito ai liposomi LA, si trasloca in questi liposomi e il segnale diminuisce a causa di FRET con Rhod-PE (Figura 1C). Il segnale viene normalizzato per determinare quanto PI(4)P viene trasferito43.
Protocollo
1. Purificazione dellatto NBD-C2
NOTA: Sebbene questo protocollo dettagli l'uso di un distruttore cellulare per rompere i batteri, può essere modificato per utilizzare altre strategie di lisi(ad esempio, una stampa francese). All'inizio della purificazione, è obbligatorio utilizzare un tampone appena degassato, filtrato e integrato con 2 mM ditiotreitolo (DTT) per prevenire l'ossidazione della cisteina. Tuttavia, per la fase di etichettatura delle proteine, è fondamentale rimuovere completamente la DTT. Molti passaggi devono essere eseguiti su ghiaccio o in una cella frigorifera per evitare qualsiasi degradazione delle proteine. Campioni di 30 μL di volume devono essere raccolti in diverse fasi del protocollo per eseguire un'analisi mediante elettroforesi su gel di dodecilsolfato-poliacrilammide di sodio (SDS-PAGE) utilizzando un gel di acrilammide al 15% per verificare l'avanzamento della purificazione. Mescolare sufficiente tampone per campioni Laemmli denaturante con ciascuna aliquota e riscaldare la miscela a 95 °C. Congelare e conservare i tubi a -20 °C fino all'analisi.
- Espressione di GST-C2Lact in Escherichia coli
- Mescolare 20 μL di celle competenti BL21 Gold con 18 μL di acqua sterilizzata. Quindi, mescolare 2 μL di plasmide pGEX-C2Lact (a ~ 65 ng / μL) con i batteri e trasformarli per elettroporazione. Risospendare i batteri con 150 μL di Lennox Lysogeny-Broth (LB) medio autoclavato (10 g/L triptone, 5 g/L estratto di lievito, 5 g/L NaCl in acqua deionizzata, senza glucosio). Lasciare che i batteri crescano a 37 °C per 1 ora in un tubo microcentrifuga a cappuccio a scatto da 2 ml.
- Inoculare 25 mL di LB medium, integrato con ampicillina da 50 μg/mL, con 150 μL di sospensione batterica in un matraccio Erlenmeyer sterile da 125 mL. Posizionare il matraccio in uno shaker orbitale a 37 °C e lasciare che i batteri crescano durante la notte con agitazione a 185 giri/min.
- Riempire due palloni Erlenmeyer sterili da 2 L con 500 mL di LB medium integrati con ampicillina da 50 μg/mL e aggiungere 5 mL di sospensione di precoltura. Lasciare che i batteri crescano a 37 °C con agitazione a 220 giri/min.
- Misurare periodicamente la densità ottica (OD) della sospensione a una lunghezza d'onda (λ) di 600 nm. Quando l'OD raggiunge un valore di ~0,6-0,7, aggiungere 500 μL di una soluzione stock di 1 M isopropil β-D-1-tiogalattosiranoside (IPTG) a ciascun pallone per avviare l'espressione di GST-C2Lact. Agitare i palloni a 185 giri/min per 4 ore a 37 °C.
- Trasferire il contenuto di ciascun pallone in un flacone per centrifuga in polipropilene. Centrifugare le due bottiglie per 30 min a 4600 × g a 4 °C per pellettizzare i batteri. Scartare il surnatante e ricaspenare ogni pellet in 50 ml di soluzione salina tamponata con fosfato freddo.
- Trasferire la sospensione batterica contenuta in ciascun flacone in un tubo centrifugo conico da 50 ml. Centrifugare i due tubi per 30 min a 2300 × g a 4°C. Rimuovere il surnatante e conservare i tubi, ognuno dei quali contiene un pellet batterico, a -20 °C.
- Purificazione di C2Latto
- Sul ghiaccio, riempire due tubi centrifughi conici da 50 mL con 50 mL di tampone contenente 50 mM Tris-HCl, pH 7,4 e 150 mM NaCl (di seguito chiamato tampone TN), precedentemente filtrato e degassato mediante filtrazione sottovuoto a membrana.
- Per preparare il tampone di lisi in ciascun tubo, sciogliere una compressa di cocktail inibitore della proteasi tetraacetico privo di acido etilendiamico nel tampone TN mediante lieve sonicazione o vortice. Aggiungere altre antiproteasi (10 μM di bestatina, 1 μg/mL di pepstatina A e 10 μM di fosforamidone). È importante sottolineare che integrare il buffer con 2 mM DTT.
- Riempire i due tubi che contengono i pellet batterici preparati al punto 1.1.6, con tampone di lisi per ottenere un volume finale di 30 ml in ciascun tubo e scongelare lentamente i pellet sul ghiaccio per 10 minuti. Schiacciare ogni pellet con una spatola di acciaio inossidabile e risuscigliarli facendo vortice i tubi e/o pipettando la sospensione avanti e indietro con un controller di pipetta e una pipetta da 25 ml fino ad ottenere una sospensione omogenea.
- Eseguire la lisi utilizzando un distruttore di celle preraffreddato (vedere la Tabella dei materiali)caricando 30 ml del campione all'interno del serbatoio ed eseguendo un ciclo di interruzione in modalità continua con una pressione di 1,6 bar. Raccogliere il lisito nello stesso tubo, mantenere il tubo su ghiaccio e aggiungere immediatamente 250 μL da una soluzione stock di 200 mM fenilmetilsulfonilfluoruro (PMSF) preparato in isopropanolo.
- Lisire l'altro campione seguendo la stessa procedura. Utilizzare il resto del tampone di lisi per lavare il distruttore cellulare e raccogliere il lavaggio per regolare il volume di ciascun lisi (~ 30 ml) a un volume finale di 50 ml.
- Integrare ogni lisato con 5 mM MgCl2e aggiungere 20 μg/mL di DNAse I per frammentare il DNA e quindi ridurre la viscosità del campione. Incubare sul ghiaccio per 30 min. Raccogliere un campione per l'analisi del gel.
- Trasferire ogni 50 mL di lisi in un tubo ultracentrifugato prechilled in policarbonato (due in totale, vedi la Tabella dei materiali). Centrifugare a 186.000 × g a 4 °C per almeno 1 ora utilizzando un ultracentrifuga.
- Parallelamente alla fase di centrifugazione, erogare 1,4 mL di un liquame contenente glutatione accoppiato al 4% di perle di agarosio in due tubi centrifughi conici da 50 mL, aggiungere 20 mL di tampone TN integrato con 1 mM DTT (tampone TND) a ciascun tubo, centrifugare a 1200 × g per 5 minuti ed eliminare il surnatante. Ripeti questo passaggio di lavaggio due volte.
- Dopo la centrifugazione del liscerato batterico, rimuovere un campione di 30 μL dal surnatante e trasferire il surnatante da ciascun tubo ultracentrifugato a un corrispondente tubo centrifugo conico da 50 ml che contiene perle pulite. Per l'analisi del gel, risuscise il pellet di detriti in uno dei tubi ultracentrifuga con 50 mL di tampone TND e raccogliere un campione da 30 μL.
- Posizionare i tubi su un rotatore per 3-4 ore a 4 °C per ottenere una sospensione omogenea del perla. Raggruppate le sospensioni di perle in una colonna cromatografica vuota da 25 ml. Lasciare decantare le pere e rimuovere il tampone e le proteine non legate per gravità. Prendi un campione dall'eluato per l'analisi.
- Risospendare le perle con 20 ml di tampone TND e raccogliere l'eluato dal flusso gravitazionale. Ripeti questo passaggio due volte per lavare completamente le pere. Mettere insieme gli eluati raccolti e conservare un campione da 30 μL per ulteriori analisi.
NOTA: Dopo una breve decantazione, un volume di ~ 2 mL di sospensione di perle, a cui è attaccato GST-C2Lact, sedimenti nella parte inferiore della colonna. - Aggiungere 1 mL di sospensione del tallone a due tubi microcentrifuga a scatto da 2 mL. Riempire ogni tubo con tampone TND fino a un volume finale di 1.970 ml. Prendere un campione di 30 μL da una provetta per ulteriori analisi (campione B1). Aggiungere 10 μL di soluzione di CaCl2 da 10 mM e 25 μL da una soluzione stock di soluzione di proteasi trombina umana a 0,02 U/μL.
- Posizionare i due tubi su un rotatore a 4 °C durante la notte per consentire alla trombina di staccare il tag GST dal dominio C2Lact. Il giorno successivo, in ogni tubo, mescolare 10 μL di soluzione di PMSF da 200 mM con la sospensione di perle per inibire l'azione della trombina.
- Centrifugare i tubi a 700 × g per 5 minuti e raccogliere il surnatante, che contiene il dominio solubile C2Lact, da ciascun tubo, senza prendere le perle. Impiatta i supernatanti in un tubo microcentrifuga a scatto da 2 ml (eluato E1) che viene mantenuto sul ghiaccio.
- Aggiungere 1 mL di tampone TND a ciascun tubo per risuscilare le perle e lavarle; ripetere il passaggio 1.2.14. Esegui questo passaggio tre volte di più per recuperare una quantità massima di proteine. Ogni volta, raggruppate i supernatanti raccolti in un nuovo tubo da 2 ml (eluati E2, E3, E4 ed E5) e prendete un'aliquota per ulteriori analisi. Al termine delle fasi di lavaggio, prendere un'aliquota dalla sospensione del perla (aliquota B2).
- Analizzare i campioni da 30 μL che sono stati raccolti nelle diverse fasi del protocollo di purificazione mediante separazione SDS-PAGE su un gel di acrilammide al 15%.
- Rimuovere le potenziali perle contaminanti mettendo insieme tutti i supernatanti(cioè~ 10 ml) raccolti durante i passaggi 1.2.14 e 1.2.15 in una colonna cromatografica da 10 mL. Raccogli l'eluato dal flusso gravitazionale e mantieni le perle nella parte inferiore della colonna.
- Concentrare il campione C2Lact utilizzando un'unità filtrante centrifuga con un cut-off a peso molecolare (MWCO) di 3 kDa e una velocità di centrifugazione di 2300 × g. Interrompere la procedura di concentrazione quando il volume del campione proteico è ~ 1 ml.
- Preparazione e purificazione di NBD-C2Latto
- Equilibrare una colonna di desalibtura (vedere la Tabella dei materiali)con il buffer TN. Caricare la colonna con 1 mL di campione concentrato di C2Lact. Lasciare che il campione entri completamente nel letto di gel, aggiungere 1,5 mL di tampone TN privo di DTT appena degassato alla colonna e raccogliere l'eluato per gravità in un tubo microcentrifuga a scatto da 2 ml.
- Diluire 50 μL di eluato in un volume finale di tampone TN da 300 μL e registrare uno spettro di assorbanza da 230 a 450 nm utilizzando un tampone TN puro come un bianco. Determinare la concentrazione di C2Lact in base all'assorbanza misurata a 280 nm, considerando un coefficiente di estinzione ε pari a 44.920 M-1.cm-1.
- Per etichettare il costrutto C2Lact con un fluoroforo NBD, mescolare la proteina con un eccesso molare dieci volte superiore di N,N'-dimetil-N-(iodoacetil)-N'-(7-nitrobenz-2-ossa-1,3-diazol-4-il)etilendiammina (IANBD ammide).
- Sciogliere 1 mg di IANBD in dimetilformammide anidra (DMF), tenendo presente che il volume finale di DMF utilizzato per etichettare il costrutto C2Lact non deve superare il 5% (v/v) del volume del campione proteico.
- Per determinare il volume di DMF (VDMF) per sciogliere IANBD, calcolare prima la quantità richiesta di IANBD (m, espressa in mg) per etichettare la proteina utilizzando la formula 1.
m = 10.000 × C × V × MWIANBD (1)
dove C è la concentrazione di C2Lact misurata nello step 1.3.2, V è il volume del campione C2Lact e MWIANBD è il peso molecolare del fluoroforo (420 g/mol). - Calcolare VDMF utilizzando le formule 2 (con m0=1) e 3.
VDMF= (m0/m) × VIANBD (2)
VIANBD= 0,05 × V (3)
Dove m0 è la quantità di polvere IANBD in mg e VIANBD è il volume di soluzione IANBD da aggiungere al campione C2Lact. - Aggiungere il volume VIANBD della soluzione IANBD appena preparata al campione C2Lact e agitare la miscela di reazione a 800-900 giri/min per 30 minuti a 25 °C utilizzando un bimbyxer protetto dalla luce. Lasciare che la reazione proceda per 90 minuti sul ghiaccio. Nel frattempo, pulire un'unità filtrante centrifuga (MWCO= 3 kDa) con 10 mL di tampone TN.
- Aggiungere L-cisteina (in eccesso molare 10 volte a IANBD) alla miscela di reazione per inattivare IANBD libero.
- Aggiungere 15 mL di tampone TN alla soluzione NBD-C2Lact e trasferire la soluzione NBD-C2Lact all'unità filtrante centrifuga. Concentrare il campione a 2 ml per separare la maggior parte della NBD libera dalla proteina mediante centrifugazione a 2300 × g. Ripeti questo passaggio di lavaggio due volte. Centrifugare il campione in un tubo centrifugo a tappo a scatto da 2 mL per 10 minuti a 19.000 × g a 4 °C per raccogliere gli aggregati potenziali del pellet e raccogliere il surnatante.
- Diluire 50 μL di eluato in un volume finale di 300 μL di tampone TN. Registrare lo spettro di assorbanza da 230 a 650 nm utilizzando l'eluato raccolto durante la procedura di concentrazione come un bianco. Determinare la concentrazione di NBD-C2Lact utilizzando la massima assorbanza a λ=280 e 495 nm e i coefficienti di estinzione ε= 44.920 M-1.cm-1 (proteina) e 25.000 M-1.cm-1 (fluoroforo NBD).
NOTA: se i due valori di concentrazione sono simili, ciò indica che il costrutto C2Lact è etichettato con un rapporto 1:1 con il gruppo NBD. - Se la concentrazione di NBD-C2Lact stimata dalla misurazione dell'assorbanza NBD supera la concentrazione stimata dall'assorbanza dei residui di triptofano (Trp), ripetere il passaggio 1.3.5 per rimuovere ulteriormente la NBD libera.
- Aggiungere glicerolo al campione per ottenere una concentrazione finale del 10% (v/v) per crioproteggere il costrutto NBD-C2Lact durante il flash-freezing. Misurare la concentrazione proteica finale.
- Preparare aliquote di proteine da 50 μL in tubi microcentrifuga a tappo a scatto da 0,5 mL. Congelare i tubi in azoto liquido e conservarli a -80 °C.
2. Purificazione di NBD-PHFAPP
NOTA: La procedura per produrre ed etichettare PHFAPP è identica a quella di NBD-C2Lact fino al trasferimento della soluzione NBD-C2Lact a un'unità filtrante centrifuga nella fase 1.3.4. Da questo passaggio in poi, seguire il protocollo descritto di seguito.
- Dopo la fase di concentrazione, tenere 2 mL di NBD-PHFAPP a 4 °C al buio per non più di 1 giorno prima di eseguire la cromatografia di esclusione dimensionale. Prima della cromatografia ad esclusione dimensionale, verificare che non vi sia alcun deposito di arancia (aggregazione durante la concentrazione) nella parte inferiore del tubo. In tal caso, centrifugare il campione a 540.000 × g per 10 minuti a 4 °C e purificare il surnatante mediante cromatografia ad esclusione dimensionale.
NOTA: La cromatografia di esclusione dimensionale viene eseguita su una colonna imballata con copolimero destro-acrilammide reticolato (vedi Tabella dei Materiali),precedentemente bilanciata con tampone TND, utilizzando un sistema di cromatografia liquida proteica veloce (vedi tabella dei materiali). La colonna deve essere protetta dalla luce. È stata utilizzata una portata di 1 mL/min e l'eluizione del costrutto NBD-PHFAPP è stata seguita dalla registrazione dell'assorbanza a λ=280 (proteina) e 480 nm (NBD) all'uscita della colonna.- Iniettare il campione NBD-PHFAPP caricato in un anello di iniezione da 2 ml sulla colonna e raccogliere immediatamente 2,5 mL di frazioni di eluato.
- Analizzare tutte le frazioni che corrispondono al picco principale rilevato a 280 e 480 nm su un gel SDS-PAGE al 15%. Mescolare un campione da 25 μL di ciascuna frazione con 15 μL di tampone campione Laemmli prima di riscaldare e caricare sul gel.
NOTA: un picco principale, che viene rilevato simultaneamente a λ= 280 e 480 nm, appare una volta che un volume di ~150 mL di buffer viene fatto passare attraverso la colonna. - Mettere insieme le frazioni che contengono esclusivamente la proteina NBD-PHFAPP (~12,2 kDa) e aggiungere glicerolo ad una concentrazione finale del 10% (v/v). Concentrare il campione utilizzando un'unità filtrante centrifuga con un MWCO di 3 kDa ad un volume finale di 1 mL utilizzando una velocità di centrifugazione di 2300 × g.
- Preparare aliquote e registrare uno spettro di assorbanza come descritto per NBD-C2Lact. Utilizzare un coefficiente di estinzione ε= 29.450 M-1.cm-1 per determinare la concentrazione della proteina in base all'assorbanza misurata a λ=280 nm.
3. Preparazione di liposomi per l'estrazione o il trasferimento di PS e PI(4)P
NOTA: Eseguire tutti i passaggi a temperatura ambiente se non diversamente specificato. Maneggiare solventi organici, rotavapor e azoto liquido con cautela.
- Preparare acido fresco, filtrato e degassato 50 mM 4-(2-idrossietil)-1-piperazineetansolfonico (HEPES)-idrossido di potassio (KOH), pH 7,4, tampone di acetato di potassio (HK) 120 mM.
- Per ogni tipo di liposoma, prendere quantità precise di lipidi diversi da soluzioni stock e mescolarli in un pallone di vetro a forma di pera da 25 ml (Tabella 1). Aggiungere cloroformio puro per regolare il volume di ciascuna miscela a 1 mL. Etichettare ogni pallone con il nome del liposoma. Avvolgere i palloni contenenti miscela lipidica drogata con Rhod-PE con un foglio di alluminio.
- Posizionare il pallone su un evaporatore rotante. Asciugare i lipidi sotto vuoto e a 25 °C per almeno 30 minuti ad una velocità di rotazione di 500 giri/min. Per i film lipidici che contengono PI(4)P, preriscaldare il matraccio a 32-34 °C per 5 minuti, a rotazione delicata, per mescolare correttamente PI(4)P con gli altri lipidi prima di creare vuoto nel matraccio per rimuovere il solvente, che lascerà un film di lipidi secchi sulla parete del pallone.
- Scollegare il pallone dall'evaporatore e metterlo in una camera a vuoto per 45 minuti per rimuovere eventuali tracce residue di solvente. Riempire il matraccio con 2 ml di tampone HK e aggiungere alcune perle di vetro di 4 mm di diametro alla soluzione. Ruotare delicatamente il pallone per 2 minuti per risospendare i lipidi e preparare vescicole lipidiche multilamellari (MLV) con una concentrazione lipidica di 4 mM. Preparare aliquote da 0,5 mL di MLV in tubi microcentrifuga da 1,5 mL con tappo a vite.
- Congelare-scongelare i tubi 5 volte (utilizzando azoto liquido e un bagno d'acqua a 37 °C, rispettivamente). Estrudere i liposomi o conservarli a -20 °C.
- Utilizzare un mini estrusore per preparare i liposomi(cioè grandi vescicole unilamellari) dai MLV secondo le linee guida del produttore. Utilizzare un filtro in policarbonato con pori cilindrici uniformi di 200 nm di diametro.
- Per preparare ogni tipo di liposoma, estrudere almeno 250 μL della corrispondente sospensione di MLV. Conservare i liposomi estrusi a 4 °C e al buio se contengono Rhod-PE. Utilizzare i liposomi entro 2 giorni.
| Composizione lipidica (mol/mol) | Lipide | |||||
| Nome del liposoma | DOPC (25 mg/ml) | POPS (10 mg/ml) | 16:0 Liss Rhod-PE (1 mg/ml) | C16:0/C16:0-PI(4)P (1 mg/ml) | ||
| Saggi di estrazione | Liposoma 2 mol% PS | PC/PS 98/2 | 247 μL | 12,5 μL | ||
| Liposoma 2 mol% PI(4)P | PC/PI(4)P 98/2 | 247 μL | 153 μL | |||
| Liposoma PC | PC 100 | 252 μL | ||||
| Saggi di trasporto | LA | PC/PS/Rhod-PE 93/5/2 | 234 μL | 31,4 μL | 200 μL | |
| LA senza PS | PC/Rhod-PE 98/2 | 247 μL | 200 μL | |||
| LB | PC/PI(4)P 95/5 | 237 μL | 383 μL | |||
| LB senza PI(4)P | PC 100 | 252 μL | ||||
| LA-Eq | PC/PS/PI(4)P/Rhod-PE 93/2.5/2.5/2 | 234 μL | 15,7 μL | 200 μL | 191 μL | |
| LB-Eq | PC/PS/PI(4)P 95/2.5/2.5 | 239 μL | 15,7 μL | 191 μL | ||
Tabella 1: Volumi di soluzioni lipidiche da miscelare per la preparazione dei liposomi. Abbreviazioni: PS= fosfatidilserina; PC = fosfatidilcolina; PI(4)P = fosfatidilinositolo 4-fosfato; Rhod-PE = fosfatidiletanolammina marcata con rodamina; DOPC = dioleoilfosfatidilcolina; POPS= 1-palmitoil-2-oleoil-sn-glicero-3-fosfo-L-serina; 16:0 Liss Rhod-PE = 1,2-dipalmitoil-sn-glicero-3-fosfoetanolammina-N-(lissamina rodamina B sulfonil).
4. Misurazione dell'estrazione PS o PI(4)P
NOTA: Le misurazioni devono essere effettuate utilizzando una piastra nera a 96 pozzetti e un lettore di piastre a fluorescenza dotato di monocromatori: uno per l'eccitazione a fluorescenza e uno per l'emissione, con larghezza di banda variabile.
- Preparare un tampone HK fresco, filtrato e degassato integrato con 1 mM MgCl2 (tampone HKM). Preparare liposomi PC puri e liposomi PC drogati con 2 mol% PS o 2 mol% PI(4)P (4 mM concentrazione lipidica finale, vedere Tabella 1).
NOTA: Mantenere i tubi riempiti con la sospensione di liposomi estrusi a temperatura ambiente per tutto l'esperimento e mantenere le proteine sul ghiaccio. Inoltre, proteggere i sensori lipidici dalla luce. - Per il test di estrazione PS, in un unico pozzetto, mescolare liposomi contenenti 2 mol% PS (80 μM concentrazione lipidica finale, 0,8 μM concentrazione PS accessibile) con NBD-C2Lact (250 nM concentrazione finale) in un volume finale di 100 μL. Riempire un secondo pozzo con la stessa quantità di liposoma (80 μM, 2 mol% PS) e NBD-C2Lact (250 nM) miscelato con 3 μM LTP (Osh6p come controllo positivo o proteina di interesse).
NOTA: Un tempo di incubazione di 5 minuti è sufficiente per Osh6p per ottenere l'estrazione lipidica. - Riempire un terzo pozzo con NBD-C2Lact (250 nM) miscelato con liposomi PC puri (80 μM). Riempire un quarto pozzo solo con liposomi PC puri (80 μM). Ripetere i passaggi 4.2-4.3 per preparare tre serie aggiuntive di quattro pozzi.
- Per ogni pozzo, registrare uno spettro NBD da 505 a 650 nm (larghezza di banda 5 nm) dopo l'eccitazione a 490 nm (larghezza di banda 5 nm) a 25 °C. Per ogni serie, sottrarre lo spettro registrato con i soli liposomi dagli altri spettri.
NOTA: F e Fmax corrispondono all'intensità a 536 nm misurata con liposomi contenenti PS in presenza o assenza di LTP, rispettivamente, mentre F 0 è l'intensità allastessa lunghezza d'onda con liposomi PC puri. Per ogni serie, la percentuale di PS accessibile estratta dalla proteina viene data utilizzando la seguente formula.
100 × (1-((F-F0)/(Fmax-F0))) (4) - Per il test di estrazione PI(4)P, preparare liposomi drogati con 2 mol% PI(4)P ed effettuare misurazioni con la sonda NBD-PHFAPP. Eseguire esperimenti di controllo e determinare la percentuale di estrazione nello stesso modo descritto sopra.
NOTA: la concentrazione di liposomi e proteine è identica a quella utilizzata nel test di estrazione PS.
5. Misurazione in tempo reale del trasporto PS
NOTA: per registrare la cinetica di trasferimento lipidico viene utilizzato un fluorimetro standard (formato 90°) dotato di un portacellulari a temperatura controllata e di un agitatore magnetico. Per acquisire con precisione i dati, è fondamentale mantenere il campione permanentemente alla stessa temperatura (impostata tra 25 e 37 °C a seconda dell'origine della proteina(ad esempio,lievito o uomo)) e mescolarla costantemente. Il protocollo descritto di seguito è per la misurazione del trasporto lipidico in un campione di 600 μL contenuto in una cella cilindrica di quarzo.
- Preparare il tampone HKM appena degassato e filtrato. Mantenere i tubi contenenti liposomi estrusi a temperatura ambiente. Avvolgere i tubi contenenti liposomi con Rhod-PE in un foglio di alluminio e/o conservarli in una scatola opaca per evitare qualsiasi fotosciviazione.
- Regolare i monocromatori di eccitazione ed emissione rispettivamente a λ = 460 nm (con una larghezza di banda corta (1-3 nm)) e a λ = 530 nm (con una grande larghezza di banda (≥ 10 nm)). Imposta il tempo di acquisizione a 25 minuti con una risoluzione temporale ≤ 1 s.
- Nella cuvetta di quarzo, diluire 30 μL della sospensione di liposoma LA e un volume di soluzione stock nbD-C2Lact in tampone HKM preriscaldato per preparare un campione di 570 μL contenente 200 μM di lipidi totali e 250 nM NBD-C2Lact. Aggiungere una piccola barra di agitazione magnetica e posizionare la cuvetta nel supporto del fluorometro.
- Una volta che il campione è bilanciato termicamente (dopo 3-5 minuti), attivare la misurazione. Dopo 1 min, aggiungere 30 μL di sospensione di liposoma LB (concentrazione finale di 200 μM lipidi totali) al campione. Dopo 3 minuti, iniettare LTP nel campione in modo che la concentrazione finale dell'LTP sia di 200 nM e acquisire il segnale per i restanti 21 minuti.
- Effettuare un esperimento parallelo per normalizzare il segnale NBD. Miscelare 30 μL di sospensione di liposomi LA-Eq con 250 nM NBD-C2Lact in tampone HKM (volume finale di 570 μL). Dopo 1 min, iniettare 30 μL di LB-Eq liposomi sospensione.
NOTA: La composizione lipidica dei liposomi LA-Eq e LB-Eq è simile a quella dei liposomi LA e LB utilizzati nel test di trasferimento, tranne per il fatto che ognuno di essi contiene il 2,5 mol% ps e il 2,5 mol% PI(4)P. Di conseguenza, il segnale NBD misurato, denominato FEq, corrisponde al segnale che dovrebbe essere misurato se PS fosse completamente bilanciato tra i liposomi LA e LB mediante un processo di trasferimento. - Convertire le curve cinetiche misurate con un LTP di interesse per determinare la quantità di PS (in μM) trasferita dai liposomi LA a LB nel tempo. Normalizzare ogni punto dati (F) della curva utilizzando la formula seguente.
Norma F = (F-F0)/(FEq-F0) (5)
in cui F0 corrisponde al segnale NBD poco prima dell'aggiunta di un LTP, e FEq è il segnale misurato nel passaggio 5.5.
NOTA: La quantità di PS (in μM) trasferita dai liposomi LA a LB corrisponde a 2,5 ×normaF, considerando che l'equilibrio corrisponde a una situazione in cui metà delle molecole PS accessibili, contenute nel foglietto esterno dei liposomi LA, (cioè , corrispondente al 5 mol% di 0,5 × 200 μM lipidi totali) è stata trasferita nei liposomi LB.
6. Misurazione in tempo reale del trasporto PI(4)P
- Impostare il fluorimetro (lunghezza d'onda di eccitazione ed emissione, larghezza di banda, tempo di acquisizione, risoluzione temporale) come fatto per il test di trasferimento PS. Allo stesso modo, utilizzare lo stesso tampone, cuvetta e liposomi per eseguire gli esperimenti sotto agitazione costante alla stessa temperatura.
- Nella cuvetta, mescolare 30 μL di sospensione liposomica LB e NBD-PHFAPP con tampone HKM preriscaldato per ottenere un volume finale di 570 μL (200 μM lipidi totali, 250 nM NBD- PHFAPP). Una volta raggiunto l'equilibrio termico del campione, iniziare la misurazione e, dopo 1 minuto, iniettare 30 μL di sospensione lipososo LA. Dopo 3 minuti, iniettare l'LTP di interesse (concentrazione finale di 200 nM) e registrare il segnale.
- Eseguire un secondo esperimento per normalizzare il segnale NBD. Miscelare 30 μL di sospensione liposomica LB-Eq con 250 nM NBD-PHFAPP in 570 μL di tampone HKM. Dopo 1 min, iniettare 30 μL di sospensione di liposomi LA-Eq.
NOTA: Qui, il segnale NBD, indicato come FEq, corrisponde a quello che dovrebbe essere misurato se PI(4)P fosse completamente bilanciato tra i liposomi LA e LB. - Convertire le curve cinetiche per determinare la quantità di PI(4)P (in μM) trasferita dai liposomi LB a LA nel tempo. Ogni punto dati (F) viene normalizzato utilizzando la formula 5 in cui F0 corrisponde al segnale NBD prima dell'aggiunta di un LTP e FEq è il segnale misurato nel passaggio 6.3.
NOTA: La quantità di PI(4)P (in μM) trasferita dai liposomi LB a LA corrisponde a 2,5 ×normaF , considerando che l'equilibrio corrisponde a una situazione in cui metà del PI(4)P contenuto nel foglietto esterno dei liposomi LB (cioè 0,5 × 5 μM) è stato trasferito nei liposomi LA.
7. Analisi delle curve cinetiche
- Quantificare la misura in cui un LTP è efficiente determinando la velocità con cui questo LTP trasferisce i lipidi da una popolazione di liposomi all'altra nei primi secondi successivi alla sua iniezione alla cuvetta.
- Eseguire una regressione lineare dei primi punti dati della cinetica di trasferimento per ottenere una pendenza. Dividere il valore della pendenza per la concentrazione di LTP nella miscela di reazione per determinare il numero di molecole lipidiche trasferite per proteina per unità di tempo (min o s).
Risultati
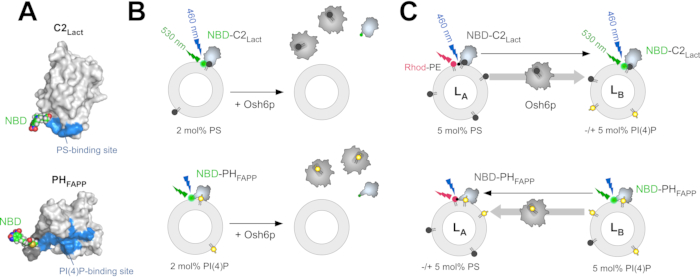
Figura 1: Descrizione dei sensori lipidici fluorescenti e saggi in vitro. (A) Modelli tridimensionali di NBD-C2Lact e NBD-PHFAPP basati sulla struttura cristallina del dominio C2 della lattaderina bovina (PDB ID: 3BN648) e sulla struttura NMR del dominio PH della proteina FAPP1 umana (PD...
Discussione
I risultati di questi test si basano direttamente sui segnali dei sensori lipidici fluorescenti. Pertanto, la purificazione di queste sonde etichettate in un rapporto 1: 1 con NBD e senza contaminazione da fluoroforo NBD libero è un passo critico in questo protocollo. È inoltre obbligatorio verificare se l'LTP in esame è correttamente piegato e non aggregato. La quantità di LTP testata nei saggi di estrazione deve essere uguale o superiore a quella delle molecole PS o PI(4)P accessibili per misurare correttamente se ...
Divulgazioni
Gli autori dichiarano che non ci sono conflitti di interesse.
Riconoscimenti
Siamo grati alla dottoressa A. Cuttriss per la sua attenta revisione del manoscritto. Questo lavoro è finanziato dalla sovvenzione ExCHANGE (ANR-16-CE13-0006) e dal CNRS.
Materiali
| Name | Company | Catalog Number | Comments |
| L-cysteine ≥97 % (FG) | Sigma | W326305-100G | Prepare a 10 mM L-cysteine stock solution in water. Aliquots are stored at -20 °C |
| 2 mL Amber Vial, PTFE/Rub Lnr, for lipids storage in CHCL3 | Wheaton | W224681 | |
| 4 mm-diameter glass beads | Sigma | Z265934-1EA | |
| 50 mL conical centrifuge tube | Falcon | ||
| ÄKTA purifier | GE healthcare | FPLC | |
| Aluminium foil | |||
| Amicon Ultra-15 with a MWCO of 3 and 10 kDa | Merck | UFC900324, UFC901024 | |
| Amicon Ultra-4 with a MWCO of 3 and 10 kDa | Merck | UFC800324, UFC801024 | |
| Ampicillin | Prepare a 50 mg/mL stock solution with filtered and sterilized water and store it at -20 °C. | ||
| Bestatin | Sigma | B8385-10mg | |
| BL21 Gold Competent Cells | Agilent | ||
| C16:0 Liss (Rhod-PE) in CHCl3 (1 mg/mL) | Avanti Polar Lipids | 810158C-5MG | |
| C16:0/C16:0-PI(4)P | Echelon Lipids | P-4016-3 | Dissolve 1 mg of C16:0/C16:0-PI(4)P powder in 250 µL of MeOH and 250 µL of CHCl3. Then complete with CHCl3 to 1 mL. The solution must become clear. |
| C16:0/C18:1-PS (POPS) in CHCl3 (10 mg/mL) | Avanti Polar Lipids | 840034C-25mg | |
| C18:1/C18:1-PC (DOPC) in CHCl3 (25 mg/mL) | Avanti Polar Lipids | 850375C-500mg | |
| CaCl2 | Sigma | Prepare 10 mM CaCl2 stock solution in water. | |
| Cell Disruptor | Constant Dynamics | ||
| Chloroform (CHCl3) RPE-ISO | Carlo Erba | 438601 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 5056489001 | |
| Deionized (Milli-Q) water | |||
| Dimethylformamide (DMF), anhydrous, >99% pure | |||
| DNAse I Recombinant, RNAse free, in powder | Roche | 10104159001 | |
| DTT | Euromedex | EU0006-B | Prepare 1 M DTT stock solution in Milli-Q water. Prepare 1 mL aliquots and store them at -20 °C. |
| Econo-Pac chromatography columns (1.5 × 12 cm). | Biorad | 7321010 | |
| Electroporation cuvette 2 mm | Ozyme | EP102 | |
| Electroporator Eppendorf 2510 | Eppendorf | ||
| Fixed-Angle Rotor Ti45 and Ti45 tubes | Beckman | Spinning the batcerial lysates | |
| Glass-syringes (10, 25, and 50 µL) for fluorescence experiment | Hamilton | ||
| Glass-syringes (25 , 100, 250, 500, and 1000 µL) to handle lipid stock solutions | Hamilton | 1702RNR, 1710RNR, 1725RNR, 1750RN type3, 1001RN | |
| Glutathione Sepharose 4B beads | GE Healthcare | 17-0756-05 | |
| Glycerol (99% pure) | Sigma | G5516-500ML | |
| Hemolysis tubes with a cap | |||
| HEPES , >99 % pure | Sigma | H3375-500G | |
| Illustra NAP 10 desalting column | GE healthcare | GE17-0854-02 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Euromedex | EU0008-B | Prepare 1 M IPTG stock solution in Milli-Qwater. Prepare 1 mL aliquots and store them at -20 °C. |
| K-Acetate | Prolabo | 26664.293 | |
| Lennox LB Broth medium without glucose | Prepared with milli-Q water and autoclaved. | ||
| Liquid nitrogen | Linde | ||
| Methanol (MeOH) ≥99.8% | VWR | 20847.24 | |
| MgCl2 | Sigma | Prepare a 2 M MgCl2 solution. Filter the solution using a 0.45 µm filter. | |
| Microplate 96 Well PS F-Botom Black Non-Binding | Greiner Bio-one | 655900 | |
| Mini-Extruder with two 1 mL gas-tight Hamilton syringes | Avanti Polar Lipids | 610023 | |
| Monochromator-based fluorescence plate reader | TECAN | M1000 Pro | |
| N,N'-Dimethyl-N-(Iodoacetyl)-N'-(7-Nitrobenz-2-Oxa-1,3-Diazol-4-yl)Ethylenediamine) (IANBD Amide) | Molecular Probes | Dissolve 25 mg of IANBD in 2.5 mL of dimethylsulfoxide (DMSO) and prepare 25 aliquot of 100 µL in 1.5 mL screw-cap tubes. Do not completely screw the cap. Then, remove DMSO in a freeze-dryer to obtain 1 mg of dry IANBD per tube. Tubes are closed and stored at -20 °C in the dark. | |
| NaCl | Sigma | S3014-1KG | |
| PBS | 137 mM NaCl, 2.7 mM KCl, 10 mM NaH2PO4, 1.8 mM KH2PO4, autoclaved and stored at 4 °C. | ||
| Pear-shaped glass flasks (25 mL, 14/23, Duran glass) | Duran Group | ||
| Pepstatin | Sigma | p5318-25mg | |
| pGEX-C2LACT plasmid | Available on request from our lab | ||
| pGEX-PHFAPP plasmid | Available on request from our lab | ||
| Phenylmethylsulfonyl fluoride (PMSF) ≥98.5% (GC) | Sigma | P7626-25g | Prepare a 200 mM PMSF stock solution in isopropanol |
| Phosphoramidon | Sigma | R7385-10mg | |
| Polycarbonate filters (19 mm in diameter) with pore size of 0.2 µm | Avanti Polar Lipids | 610006 | |
| Poly-Prep chromatography column (with a 0-2 mL bed volume and a 10 mL reservoir) | Biorad | 7311550 | |
| Prefilters (10 mm in diameter). | Avanti Polar Lipids | 610014 | |
| PyMOL | http://pymol.org/ | Construction of the 3D models of the proteins (Figure 1A) | |
| Quartz cuvette for UV/visible fluorescence (minimum volume of 600 µL) | Hellma | ||
| Quartz cuvettes | Hellma | ||
| Refrigerated centrifuge Eppendorf 5427R | Eppendorf | ||
| Rotary evaporator | Buchi | B-100 | |
| Screw-cap microcentriguge tubes (1.5 mL) | Sarsted | ||
| Small magnetic PFTE stirring bar (5 × 2 mm) | |||
| Snap-cap microcentriguge tubes (0.5, 1, and 2 mL) | Eppendorf | ||
| SYPRO orange | fluorescent stain to detect protein in SDS-PAGE gel | ||
| Thermomixer | Starlab | ||
| THROMBIN, FROM HUMAN PLASMA | Sigma | 10602400001 | Dissolve 20 units in 1 mL of milli-Q water and prepare 25 µL aliquots in 0.5 mL Eppendorf tubes. Then freeze and store at -80 °C. |
| Tris, ultra pure | MP | 819623 | |
| Ultracentrifuge L90K | Beckman | ||
| UV/Visible absorbance spectrophotometer | SAFAS | ||
| UV/visible spectrofluorometer with a temperature-controlled cell holder and stirring device | Jasco or Shimadzu | Jasco FP-8300 or Shimadzu RF-5301PC | |
| Vacuum chamber | |||
| Water bath | Julabo | ||
| XK 16/70 column packed with Sephacryl S200HR | GE healthcare |
Riferimenti
- Drin, G. Topological regulation of lipid balance in cells. Annual Review of Biochemistry. 83, 51-77 (2014).
- Bigay, J., Antonny, B. Curvature, lipid packing, and electrostatics of membrane organelles: defining cellular territories in determining specificity. Developmental Cell. 23 (5), 886-895 (2012).
- Prinz, W. A. Lipid trafficking sans vesicles: where, why, how. Cell. 143 (6), 870-874 (2010).
- Holthuis, J. C., Menon, A. K. Lipid landscapes and pipelines in membrane homeostasis. Nature. 510 (7503), 48-57 (2014).
- Wong, L. H., Copic, A., Levine, T. P. Advances on the transfer of lipids by lipid transfer proteins. Trends in Biochemical Sciences. 42 (7), 516-530 (2017).
- Wong, L. H., Gatta, A. T., Levine, T. P. Lipid transfer proteins: the lipid commute via shuttles, bridges and tubes. Nature Reviews Molecular Cell Biology. 20 (2), 85-101 (2019).
- Iaea, D. B., Dikiy, I., Kiburu, I., Eliezer, D., Maxfield, F. R. STARD4 membrane interactions and sterol binding. Biochemistry. 54 (30), 4623-4636 (2015).
- Wilhelm, L. P., et al. STARD3 mediates endoplasmic reticulum-to-endosome cholesterol transport at membrane contact sites. The EMBO Journal. 36 (10), 1412-1433 (2017).
- Bian, X., Saheki, Y., De Camilli, P. Ca(2+) releases E-Syt1 autoinhibition to couple ER-plasma membrane tethering with lipid transport. The EMBO Journal. 37 (2), 219-234 (2018).
- Horenkamp, F. A., Valverde, D. P., Nunnari, J., Reinisch, K. M. Molecular basis for sterol transport by StART-like lipid transfer domains. The EMBO Journal. 37 (6), 98002 (2018).
- Jentsch, J. A., et al. Structural basis of sterol binding and transport by a yeast StARkin domain. The Journal of Biological Chemistry. 293 (15), 5522-5531 (2018).
- Moser von Filseck, J., et al. INTRACELLULAR TRANSPORT. Phosphatidylserine transport by ORP/Osh proteins is driven by phosphatidylinositol 4-phosphate. Science. 349 (6246), 432-436 (2015).
- Daum, G., et al. Systematic analysis of yeast strains with possible defects in lipid metabolism. Yeast. 15 (7), 601-614 (1999).
- Ejsing, C. S., et al. Global analysis of the yeast lipidome by quantitative shotgun mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2136-2141 (2009).
- Leidl, K., Liebisch, G., Richter, D., Schmitz, G. Mass spectrometric analysis of lipid species of human circulating blood cells. Biochimica et Biophysica Acta. 1781 (10), 655-664 (2008).
- Sampaio, J. L., et al. Membrane lipidome of an epithelial cell line. Proceedings of the National Academy of Sciences of the United States of America. 108 (5), 1903-1907 (2011).
- Vance, J. E., Steenbergen, R. Metabolism and functions of phosphatidylserine. Progress in Lipid Research. 44 (4), 207-234 (2005).
- Zinser, E., et al. Phospholipid synthesis and lipid composition of subcellular membranes in the unicellular eukaryote Saccharomyces cerevisiae. Journal of Bacteriology. 173 (6), 2026-2034 (1991).
- Leventis, P. A., Grinstein, S. The distribution and function of phosphatidylserine in cellular membranes. Annual Review of Biophysics. 39, 407-427 (2010).
- Yeung, T., et al. Membrane phosphatidylserine regulates surface charge and protein localization. Science. 319 (5860), 210-213 (2008).
- Kim, J., Shishido, T., Jiang, X., Aderem, A., McLaughlin, S. Phosphorylation, high ionic strength, and calmodulin reverse the binding of MARCKS to phospholipid vesicles. Journal of Biological Chemistry. 269 (45), 28214-28219 (1994).
- Sigal, C. T., Zhou, W., Buser, C. A., McLaughlin, S., Resh, M. D. Amino-terminal basic residues of Src mediate membrane binding through electrostatic interaction with acidic phospholipids. Proceedings of the National Academy of Sciences. 91 (25), 12253-12257 (1994).
- Gal Bivona, T., et al. PKC regulates a farnesyl-electrostatic switch on K-Ras that promotes its association with Bcl-XL on mitochondria and induces apoptosis. Molecular Cell. 21 (4), 481-493 (2006).
- Finkielstein, C. V., Overduin, M., Capelluto, D. G. Cell migration and signaling specificity is determined by the phosphatidylserine recognition motif of Rac1. The Journal of Biological Chemistry. 281 (37), 27317-27326 (2006).
- Bolsover, S. R., Gomez-Fernandez, J. C., Corbalan-Garcia, S. Role of the Ca2+/Phosphatidylserine Binding Region of the C2 Domain in the Translocation of Protein Kinase Cα to the Plasma Membrane. Journal of Biological Chemistry. 278 (12), 10282-10290 (2003).
- Vance, J. E., Tasseva, G. Formation and function of phosphatidylserine and phosphatidylethanolamine in mammalian cells. Biochimica et Biophysica Acta. 1831 (3), 543-554 (2013).
- Maeda, K., et al. Interactome map uncovers phosphatidylserine transport by oxysterol-binding proteins. Nature. 501 (7466), 257-261 (2013).
- D'Ambrosio, J. M., et al. Osh6 requires Ist2 for localization to ER-PM contacts and efficient phosphatidylserine transport in budding yeast. Journal of Cell Science. 133 (11), 243733 (2020).
- Manford, A. G., Stefan, C. J., Yuan, H. L., Macgurn, J. A., Emr, S. D. ER-to-plasma membrane tethering proteins regulate cell signaling and ER morphology. Developmental Cell. 23 (6), 1129-1140 (2012).
- Collado, J., et al. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- Hoffmann, P. C., et al. Tricalbins contribute to cellular lipid flux and form curved ER-PM contacts that are bridged by rod-shaped structures. Developmental Cell. 51 (4), 488-502 (2019).
- Lipp, N. F., et al. An electrostatic switching mechanism to control the lipid transfer activity of Osh6p. Nature Communications. 10 (1), 3926 (2019).
- Chung, J., et al. INTRACELLULAR TRANSPORT. PI4P/phosphatidylserine countertransport at ORP5- and ORP8-mediated ER-plasma membrane contacts. Science. 349 (6246), 428-432 (2015).
- Sohn, M., et al. PI(4,5)P2 controls plasma membrane PI4P and PS levels via ORP5/8 recruitment to ER-PM contact sites. The Journal of Cell Biology. 217 (5), 1797-1813 (2018).
- Ghai, R., et al. ORP5 and ORP8 bind phosphatidylinositol-4, 5-biphosphate (PtdIns(4,5)P 2) and regulate its level at the plasma membrane. Nature Communications. 8 (1), 757 (2017).
- Sohn, M., et al. Lenz-Majewski mutations in PTDSS1 affect phosphatidylinositol 4-phosphate metabolism at ER-PM and ER-Golgi junctions. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4314-4319 (2016).
- Kattan, W. E., et al. Targeting plasma membrane phosphatidylserine content to inhibit oncogenic KRAS function. Life Science Alliance. 2 (5), 00431 (2019).
- Du, X., Turner, N., Yang, H. The role of oxysterol-binding protein and its related proteins in cancer. Seminars in Cell & Developmental Biology. 81, 149-153 (2018).
- Galmes, R., et al. ORP5/ORP8 localize to endoplasmic reticulum-mitochondria contacts and are involved in mitochondrial function. EMBO reports. 17 (6), 800-810 (2016).
- Du, X., et al. ORP5 localizes to ER-lipid droplet contacts and regulates the level of PI(4)P on lipid droplets. The Journal of Cell Biology. 219 (1), 201905162 (2020).
- Wang, H., et al. ORP2 delivers cholesterol to the plasma membrane in exchange for phosphatidylinositol 4, 5-bisphosphate (PI(4,5)P2). Molecular Cell. 73 (3), 458-473 (2019).
- Kay, J. G., Grinstein, S. Sensing phosphatidylserine in cellular membranes. Sensors (Basel). 11 (2), 1744-1755 (2011).
- Moser von Filseck, J., Vanni, S., Mesmin, B., Antonny, B., Drin, G. A phosphatidylinositol-4-phosphate powered exchange mechanism to create a lipid gradient between membranes. Nature Communications. 6, 6671 (2015).
- Wills, R. C., Goulden, B. D., Hammond, G. R. V. Genetically encoded lipid biosensors. Molecular Biology of the Cell. 29 (13), 1526-1532 (2018).
- Raychaudhuri, S., Im, Y. J., Hurley, J. H., Prinz, W. A. Nonvesicular sterol movement from plasma membrane to ER requires oxysterol-binding protein-related proteins and phosphoinositides. The Journal of Cell Biology. 173 (1), 107-119 (2006).
- Lenoir, M., et al. Structural basis of wedging the Golgi membrane by FAPP pleckstrin homology domains. EMBO reports. 11 (4), 279-284 (2010).
- Liu, Y., Kahn, R. A., Prestegard, J. H. Interaction of Fapp1 with Arf1 and PI4P at a membrane surface: an example of coincidence detection. Structure. 22 (3), 421-430 (2014).
- Shao, C., Novakovic, V. A., Head, J. F., Seaton, B. A., Gilbert, G. E. Crystal structure of lactadherin C2 domain at 1.7A resolution with mutational and computational analyses of its membrane-binding motif. The Journal of Biological Chemistry. 283 (11), 7230-7241 (2008).
- Lipp, N. F., Ikhlef, S., Milanini, J., Drin, G. Lipid exchangers: cellular functions and mechanistic links with phosphoinositide metabolism. Frontiers in Cell and Developmental Biology. 8, 663 (2020).
- Venditti, R., et al. Molecular determinants of ER-Golgi contacts identified through a new FRET-FLIM system. The Journal of Cell Biology. 218 (3), 1055-1065 (2019).
- Pemberton, J. G., et al. Defining the subcellular distribution and metabolic channeling of phosphatidylinositol. The Journal of Cell Biology. 219 (3), (2020).
- Nakanishi, H., de los Santos, P., Neiman, A. M. Positive and negative regulation of a SNARE protein by control of intracellular localization. Molecular Biology of the Cell. 15 (4), 1802-1815 (2004).
- Maekawa, M., Yang, Y., Fairn, G. D. Perfringolysin O theta toxin as a tool to monitor the distribution and inhomogeneity of cholesterol in cellular membranes. Toxins. 8 (3), 67 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon