Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Fluoreszenzbasierte Messungen des Phosphatidylserin/Phosphatidylinositol-4-Phosphat-Austauschs zwischen Membranen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir Protokolle mit fluoreszierenden Lipidsensoren und Liposomen, um festzustellen, ob ein Protein Phosphatidylserin oder Phosphatidylinositol-4-phosphat in vitroextrahiert und transportiert.
Zusammenfassung
Mehrere Mitglieder der evolutionär konservierten Oxysterol-bindenden Protein (OSBP)-verwandten Proteine (ORP) / OSBP-Homologe (Osch) Familie wurden kürzlich gefunden, um eine neuartige Lipidtransferprotein (LTP) Gruppe in Hefe und menschlichen Zellen darzustellen. Sie übertragen Phosphatidylserin (PS) aus dem endoplasmatischen Retikulum (ER) über PS/Phosphatidylinositol-4-phosphat (PI(4)P)-Austauschzyklen in die Plasmamembran (PM). Dieser Befund ermöglicht ein besseres Verständnis dafür, wie PS, das für Signalprozesse entscheidend ist, in der Zelle verteilt ist, und die Untersuchung des Zusammenhangs zwischen diesem Prozess und dem Phosphoinositidstoffwechsel (PIP). Die Entwicklung neuer fluoreszenzbasierter Protokolle war entscheidend für die Entdeckung und Charakterisierung dieses neuen zellulären Mechanismus in vitro auf molekularer Ebene. Dieser Artikel beschreibt die Herstellung und den Einsatz von zwei fluoreszierend markierten Lipidsensoren, NBD-C2Lact und NBD-PHFAPP, um die Fähigkeit eines Proteins zu messen, PS oder PI(4)P zu extrahieren und diese Lipide zwischen künstlichen Membranen zu übertragen. Zunächst beschreibt das Protokoll, wie hochreine Proben dieser beiden Konstrukte produziert, etikettiert und erhalten werden. Zweitens wird in diesem Artikel erläutert, wie diese Sensoren mit einem Fluoreszenz-Mikroplattenleser verwendet werden können, um festzustellen, ob ein Protein PS oder PI(4)P aus Liposomen extrahieren kann, wobei Osh6p als Fallstudie verwendet wird. Schließlich zeigt dieses Protokoll, wie die Kinetik des PS/PI(4)P-Austauschs zwischen Liposomen definierter Lipidzusammensetzung genau gemessen und die Lipidtransferraten durch Fluoreszenzresonanz-Energietransfer (FRET) mit einem Standardfluorometer bestimmt werden können.
Einleitung
Die genaue Verteilung der Lipide zwischen verschiedenen Membranen und innerhalb der Membranen eukaryotischer Zellen1,2 hat tiefgreifende biologische Implikationen. Die Entschlüsselung der Funktionsweise von LTPs ist ein wichtiges Thema in der Zellbiologie3,4,5,6, und In-vitro-Ansätze sind von großem Wert, um dieses Problem anzugehen7,8,9,10,11. Hier wird eine in vitro, fluoreszenzbasierte Strategie vorgestellt, die maßgeblich dazu beigetragen hat, dass mehrere ORP/Osh-Proteine den PS/PI(4)P-Austausch zwischen Zellmembranen12 bewirken und damit eine neue Klasse von LTPs darstellen. PS ist ein anionisches Glycerophospholipid, das 2-10% der gesamten Membranlipide in eukaryotischen Zellen ausmacht13,14,16. Es ist entlang eines Gradienten zwischen dem ER und dem PM verteilt, wo es 5-7% und bis zu 30% der Glycerophospholipide bzw.17,18,19darstellt. Darüber hinaus ist PS im Wesentlichen in der zytosolischen Packungsbeilage des PM konzentriert. Dieser Aufbau und die ungleichmäßige Aufteilung von PS im PM sind entscheidend für zelluläre Signalisierungsprozesse19. Aufgrund der negativen Ladung von PS-Molekülen ist das zytosolische Blättchen des PM viel anionischer als das zytosolische Blättchen anderer Organellen1,2,19,20. Dies ermöglicht die Rekrutierung von Signalproteinen wie myristoyliertem Alanin-reichem C-Kinase-Substrat (MARCKS)21, Sarkom (Src)22, Kirsten-Ratten-Sarkom-Viralonkogen (K-Ras)23und Ras-verwandtem C3-Botulinumtoxin-Substrat 1 (Rac1)24, die einen Abschnitt positiv geladener Aminosäuren und einen lipidischen Schwanz enthalten.
PS wird auch von der herkömmlichen Proteinkinase C stereoselektiv über eine C2-Domäne25erkannt. PS wird jedoch im ER26synthetisiert, was darauf hinweist, dass es in den PM exportiert werden muss, bevor es seine Rolle spielen kann. Es war nicht bekannt, wie dies erreicht wurde19 bis zu der Feststellung, dass in Hefe, Osh6p und Osh7p PS von der Notaufnahme auf die PM27übertragen. Diese LTPs gehören zu einer evolutionär konservierten Familie in Eukaryoten, deren Gründungsmitglied OSBP ist und die Proteine (ORPs im Menschen, Osch-Proteine in Hefe) enthält, die eine OSBP-verwandte Domäne (ORD) mit einer Tasche integrieren, um ein Lipidmolekül zu beherbergen. Osh6p und Osh7p bestehen nur aus einem ORD, dessen strukturelle Merkmale angepasst sind, um PS spezifisch zu binden und zwischen Membranen zu übertragen. Dennoch war unklar, wie diese Proteine PS richtungsweisend von der ER auf die PM übertragen. Osh6p und Osh7p können PI(4)P als alternativen Lipidliganden12einfangen. In Hefe wird PI(4)P aus Phosphatidylinositol (PI) im Golgi und im PM durch PI 4-Kinasen, Pik1p bzw. Stt4p synthetisiert. Im Gegensatz dazu gibt es kein PI(4)P in der ER-Membran, da dieses Lipid durch die Sac1p-Phosphatase zu PI hydrolysiert wird. Daher existiert sowohl an der ER/Golgi- als auch an der ER/PM-Schnittstelle ein PI(4)P-Gradient. Osh6p und Osh7p übertragen PS von der ER zum PM über PS/PI(4)P-Austauschzyklen unter Verwendung des PI(4)P-Gradienten, der zwischen diesen beiden Membranen besteht12.
Innerhalb eines Zyklus extrahiert Osh6p PS aus der ER, tauscht PS gegen PI(4)P am PM aus und überträgt PI(4)P zurück in die ER, um ein anderes PS-Molekül zu extrahieren. Osh6p/Osh7p interagieren mit Ist2p28, einem der wenigen Proteine, die die ER-Membran und das PM in unmittelbarer Nähe zueinander verbinden und bringen, um ER-PM-Kontaktstellen in Hefe29,30,31zu erzeugen . Darüber hinaus wird die Assoziation von Osh6p mit negativ geladenen Membranen schwach, sobald das Protein einen seiner Lipidliganden aufgrund einer Konformationsänderung extrahiert, die seine elektrostatischen Eigenschaften modifiziert32. Dies unterstützt Osh6p, indem es die Verweilzeit seiner Membran verkürzt und dadurch die Effizienz seiner Lipidtransferaktivität aufrechterhält. In Kombination mit der Bindung an Ist2p könnte dieser Mechanismus es Osh6p/7p ermöglichen, den Lipidaustausch an der ER/PM-Schnittstelle schnell und präzise auszuführen. In menschlichen Zellen führen ORP5- und ORP8-Proteine den PS/PI(4)P-Austausch an ER-PM-Kontaktstellen über unterschiedliche Mechanismenaus 33. Sie haben eine zentrale ORD, ähnlich Osh6p, sind aber über ein C-terminales Transmembransegment33 direkt an der ER verankert und docken über eine N-terminale Pleckstrin-Homologie (PH)-Domäne, die PI(4)P und PI(4,5)P233,34, 35erkennt,an den PM an. ORP5/8 verwendet PI(4)P zur Übertragung von PS, und es wurde gezeigt, dass ORP5/8 zusätzlich PM PI(4,5)P2-Pegel reguliert und vermutlich Signalwege moduliert. Im Gegenzug senkt eine Abnahme der PI(4)P- und PI(4,5)P2-Spiegel die ORP5/ORP8-Aktivität, da diese Proteine PIP-abhängig mit dem PM assoziiert werden. Eine ungewöhnlich hohe PS-Synthese, die zum Lenz-Majewski-Syndrom führt, beeinflusst die PI(4)P-Spiegel durch ORP5/836. Wenn die Aktivität beider Proteine blockiert wird, wird PS am PM weniger reichlich, was die onkogene Fähigkeit von Signalproteinen verringert37.
Umgekehrt scheint die ORP5-Überexpression die Invasion von Krebszellen und metastatische Prozesse zu fördern38. So können Veränderungen der ORP5/8-Aktivität das zelluläre Verhalten durch Veränderungen der Lipid-Homöostase stark verändern. Darüber hinaus besetzen ORP5 und ORP8 ER-Mitochondrien-Kontaktstellen und erhalten einige mitochondriale Funktionen, möglicherweise durch die Versorgung mit PS39. Darüber hinaus lokalisiert ORP5 auf ER-Lipid-Tröpfchenkontaktstellen, um PS an Lipidtröpfchen per PS/PI(4)P-Austausch40zu liefern. Die hierin beschriebene Strategie zur Messung (i) der PS- und PI(4)P-Extraktion aus Liposomen und (ii) des PS- und PI(4)P-Transports zwischen Liposomen wurde entwickelt, um die PS/PI(4)P-Austauschaktivität von Osh6p/Osh7p12,32 zu ermitteln und zu analysieren, und von anderen Gruppen zur Analyse der Aktivität von ORP5/ORP835 und anderen LTPs10verwendet. ( 41) DER PRÄSIDENT. - Nach der 41 Es basiert auf der Verwendung eines Fluoreszenzplattenlesers, eines Standard-L-Format-Spektrofluorometers und zweier Fluoreszenzsensoren, NBD-C2Lact und NBD-PHFAPP, die PS bzw. PI(4)P detektieren können.
NBD-C2Lact entspricht der C2-Domäne des Glykoproteins Lactadherin, das überarbeitet wurde, um ein einzigartiges lösungsmittelexponiertes Cystein in der Nähe der vermuteten PS-Bindungsstelle aufzunehmen; ein polaritätsempfindliches NBD-Fluorophor (7-Nitrobenz-2-oxa-1,3-diazol) ist kovalent mit diesem Rückstand verknüpft (Abbildung 1A)12. Genauer gesagt wurde die C2-Domäne von Lactadherin (Bos taurus, UniProt: Q95114, Rückstände 270-427) in einen pGEX-4T3-Vektor geklont, der in Fusion mit Glutathion-S-Transferase (GST) in Escherichia coliexprimiert wird. DieC2-Lact-Sequenz wurde dann mutiert, um zwei lösungsmittelzugängliche Cysteinreste (C270, C427) durch Alaninreste zu ersetzen und einen Cysteinrest in eine Region nahe der mutmaßlichen PS-Bindungsstelle (H352C-Mutation) einzuführen, die anschließend mit N,N'-Dimethyl-N-(iodoacetyl)-N'-(7-nitrobenz-2-oxa-1,3-diazol-4-yl)ethylendiamin (IANBD)12 markiert werden kann. Zwischen dem GST-Protein und dem N-Terminus der C2-Domäne befindet sich eine Spaltstelle für Thrombin. Ein großer Vorteil ist, dass diese Domäne PS selektiv in einer Ca2+-unabhängigen Weise im Gegensatz zu anderen bekannten C2-Domänen oder Anhang in A542erkennt. NBD-PHFAPP wird aus der PH-Domäne des humanen Vierphosphatadapterproteins 1 (FAPP1) abgeleitet, das überarbeitet wurde, um ein einzelnes lösungsmittelexponiertes Cystein zu enthalten, das mit einer NBD-Gruppe in der Nähe der PI(4)P-Bindungsstelle markiert werden kann (Abbildung 1A)43. Die Nukleotidsequenz der PH-Domäne des humanen FAPP-Proteins (UniProt: Q9HB20, Segment [1-100]) wurde in einen pGEX-4T3-Vektor geklont, der zusammen mit einem GST-Tag exprimiert wird. DiePH-FAPP-Sequenz wurde modifiziert, um einen einzigartigen Cysteinrest in die membranbindende Grenzfläche des Proteins43einzufügen. Darüber hinaus wurde ein Linker mit neun Rückständen zwischen der Thrombinspaltungsstelle und dem N-Terminus der PH-Domäne eingeführt, um den Zugang zur Protease zu gewährleisten.
Um die PS-Extraktion aus Liposomen zu messen, wird NBD-C2Lact mit Liposomen aus Phosphatidylcholin (PC) gemischt, die Spuren von PS enthalten. Aufgrund seiner Affinität zu PS bindet dieses Konstrukt an die Liposomen, und das NBD-Fluorophor erfährt eine Polaritätsänderung, wenn es mit der hydrophoben Umgebung der Membran in Kontakt kommt, was eine Blauverschiebung und eine Erhöhung der Fluoreszenz hervorruft. Wenn PS fast vollständig durch eine stöchiometrische Menge an LTP extrahiert wird, assoziiert sich die Sonde nicht mit Liposomen und das NBD-Signal ist niedriger (Abbildung 1B)32. Dieser Signalunterschied wird verwendet, um zu bestimmen, ob ein LTP(z. B. Osh6p) PS extrahiert. Eine ähnliche Strategie wird mit NBD-PHFAPP verwendet, um die PI(4)P-Extraktion zu messen (Abbildung 1B), wie zuvor12,32beschrieben. Zwei FRET-basierte Assays wurden entwickelt, um (i) den PS-Transport von LA- zuL-B-Liposomen, die die ER-Membran bzw. das PM nachahmen, und (ii) den PI(4)P-Transport in umgekehrter Richtung zu messen. Diese Assays werden unter den gleichen Bedingungen(d. h. gleiche Puffer-, Temperatur- und Lipidkonzentration) durchgeführt, um den PS/PI(4)P-Austausch zumessen. Zur Messung des PS-Transports wird NBD-C2Lact mitL-A-Liposomen gemischt, die aus PC bestehen und mit 5 mol% PS und 2 mol% eines fluoreszierenden Rhodamin-markierten Phosphatidylethanolamins (Rhod-PE)- und L B-Liposomen mit 5 mol% PI(4)P dotiert.
Zum Zeitpunkt Null löscht FRET mit Rhod-PE die NBD-Fluoreszenz ab. Wird PS von LA zu LB Liposomen transportiert(z.B. bei Der Injektion von Osh6p), kommt es durch die Translokation von NBD-C2Lact Molekülen von LA zu LB Liposomen zu einem schnellen Dequenching (Abbildung 1C). Angesichts der Menge an zugänglichem PS bleibt NBD-C2Lact im Laufe des Experiments im Wesentlichen in einem membrangebundenen Zustand12. Somit korreliert die Intensität des NBD-Signals direkt mit der Verteilung von NBD-C2Lact zwischen LA und LB Liposomen und kann leicht normalisiert werden, um zu bestimmen, wie viel PS übertragen wird. Um den Transfer von PI(4)P in die entgegengesetzte Richtung zu messen, wird NBD-PHFAPP mit LA und LB Liposomen gemischt; Da es nur anL-B-Liposomen bindet, die PI(4)P enthalten, aber nicht an Rhod-PE, ist seine Fluoreszenz hoch. Wenn PI(4)P aufL-A-Liposomen übertragen wird, transloziert es sich in diese Liposomen, und das Signal nimmt aufgrund von FRET mit Rhod-PE ab (Abbildung 1C). Das Signal wird normalisiert, um zu bestimmen, wie viel PI(4)P übertragen wird43.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Reinigung von NBD-C2Lact
HINWEIS: Obwohl dieses Protokoll die Verwendung eines Zelldisruptors zum Aufbrechen von Bakterien beschreibt, kann es modifiziert werden, um andere Lysestrategien zu verwenden(z. B. eine französische Presse). Zu Beginn der Reinigung ist es obligatorisch, einen Puffer zu verwenden, der frisch entgast, filtriert und mit 2 mM Dithiothreitol (DTT) ergänzt wird, um die Oxidation von Cystein zu verhindern. Für den Proteinmarkierungsschritt ist es jedoch entscheidend, DTT vollständig zu entfernen. Viele Schritte müssen auf Eis oder in einem kühlen Raum durchgeführt werden, um einen Proteinabbau zu vermeiden. Proben von 30 μL Volumen müssen in verschiedenen Schritten des Protokolls gesammelt werden, um eine Analyse mittels Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) mit einem 15% igen Acrylamidgel durchzuführen, um den Fortschritt der Reinigung zu überprüfen. Mischen Sie mit jedem Aliquot genügend denaturierende Laemmli-Probenpuffer und erhitzen Sie die Mischung bei 95 °C. Die Röhrchen bis zur Analyse einfrieren und bei -20 °C lagern.
- Expression von GST-C2Lact in Escherichia coli
- Mischen Sie 20 μL BL21 Gold kompetente Zellen mit 18 μL sterilisiertem Wasser. Mischen Sie dann 2 μL pGEX-C2Lact Plasmid (bei ~ 65 ng / μL) mit den Bakterien und wandeln Sie sie durch Elektroporation um. Resuspendieren Sie die Bakterien mit 150 μL autoklaviertem Lennox Lysogeny-Brühe (LB) Medium (10 g/L Trypton, 5 g/L Hefeextrakt, 5 g/L NaCl in entionisiertem Wasser, glukosefrei). Lassen Sie die Bakterien bei 37 °C für 1 h in einem 2 mL Snap-Cap-Mikrozentrifugenröhrchen wachsen.
- Impfen Sie 25 ml LB-Medium, ergänzt mit 50 μg/ml Ampicillin, mit 150 μL Bakteriensuspension in einem 125 ml sterilen Erlenmeyerkolben. Legen Sie den Kolben bei 37 °C in einen Orbitalschüttler und lassen Sie die Bakterien über Nacht mit Rührung bei 185 U/min wachsen.
- Füllen Sie zwei sterile 2 L Erlenmeyerkolben mit 500 ml LB-Medium, ergänzt mit 50 μg/ml Ampicillin, und fügen Sie 5 ml Vorkultursuspension hinzu. Lassen Sie die Bakterien bei 37 °C mit Rührung bei 220 U/min wachsen.
- Messen Sie periodisch die optische Dichte (OD) der Suspension bei einer Wellenlänge (λ) von 600 nm. Wenn der OD einen Wert von ~0,6-0,7 erreicht, fügen Sie 500 μL einer Stammlösung von 1 M Isopropyl β-D-1-thiogalactopyranosid (IPTG) in jeden Kolben hinzu, um die Expression von GST-C2Lacteinzuleiten. Schütteln Sie die Kolben bei 185 U/min für 4 h bei 37 °C.
- Den Inhalt jedes Kolbens in eine Polypropylen-Zentrifugenflasche geben. Zentrifugieren Sie die beiden Flaschen für 30 min bei 4600 × g bei 4 °C, um die Bakterien zu pelletieren. Entsorgen Sie den Überstand und resuspendieren Sie jedes Pellet in 50 ml kalter phosphatgepufferter Kochsalzlösung.
- Die in jeder Flasche enthaltene Bakteriensuspension wird in ein 50 ml konisches Zentrifugenröhrchen überführen. Zentrifugieren Sie die beiden Röhrchen für 30 min bei 2300 × g bei 4°C. Entfernen Sie den Überstand und lagern Sie die Röhrchen, die jeweils ein Bakterienpellet enthalten, bei -20 °C.
- Reinigung von C2Lakt
- Füllen Sie auf Eis zwei konische 50-ml-Zentrifugalröhrchen mit 50 ml Puffer, der 50 mM Tris-HCl, pH 7,4 und 150 mM NaCl (im Folgenden TN-Puffer genannt) enthält, die zuvor durch Membranvakuumfiltration filtriert und entgast wurden.
- Um den Lysepuffer in jedem Röhrchen vorzubereiten, lösen Sie eine Tablette Ethylendiamintetraessigsäure-freien Protease-Inhibitor-Cocktail im TN-Puffer durch milde Beschallung oder Wirbelung. Fügen Sie andere Antiproteasen hinzu (10 μM Bestatin, 1 μg/ml Pepstatin A und 10 μM Phosphoramidon). Wichtig ist, dass Sie den Puffer mit 2 mM DTT ergänzen.
- Füllen Sie die beiden Röhrchen, die die in Schritt 1.1.6 hergestellten Bakterienpellets enthalten, mit einem Lysepuffer, um ein Endvolumen von 30 ml in jedem Röhrchen zu erhalten, und tauen Sie die Pellets langsam für 10 Minuten auf Eis auf. Zerkleinern Sie jedes Pellet mit einem Edelstahlspatel und resuspendieren Sie es, indem Sie die Rohre vortexen und/oder die Suspension mit einem Pipettenregler und einer 25-ml-Pipette hin und her pipettieren, bis eine homogene Suspension erhalten ist.
- Führen Sie die Lyse mit einem vorgekühlten Zelldisruptor durch (siehe Materialtabelle),indem Sie 30 ml der Probe in das Reservoir laden und einen Bruchzyklus im kontinuierlichen Modus mit einem Druck von 1,6 bar durchlaufen. Sammeln Sie das Lysat im selben Röhrchen, halten Sie das Röhrchen auf Eis und fügen Sie sofort 250 μL aus einer Stammlösung von 200 mM Phenylmethylsulfonylfluorid (PMSF) hinzu, die in Isopropanol hergestellt wird.
- Lyse das andere Beispiel nach dem gleichen Verfahren. Verwenden Sie den Rest des Lysepuffers, um den Zelldisruptor zu waschen, und sammeln Sie die Wäsche, um das Volumen jedes Lysatsats (~ 30 ml) auf ein Endvolumen von 50 ml einzustellen.
- Ergänzen Sie jedes Lysat mit 5 mM MgCl2und fügen Sie 20 μg / ml DNAse I hinzu, um die DNA zu fragmentieren und so die Viskosität der Probe zu reduzieren. 30 Min. auf Eis inkubieren. Sammeln Sie eine Probe für die Gelanalyse.
- Je 50 ml Lysat in ein vorgeflechter Polycarbonat-Ultrazentrifugenröhrchen geben (insgesamt zwei, siehe Materialtabelle). Zentrifuge bei 186.000 × g bei 4 °C für mindestens 1 h mit einer Ultrazentrifuge.
- Parallel zum Zentrifugationsschritt 1,4 ml einer Glutathion enthaltenden Glutathion enthaltenden Glutathion, die mit 4% Agaroseperlen gekoppelt ist, in zwei konische 50 mL Zentrifugalröhrchen geben, 20 ml TN-Puffer mit 1 mM DTT (TND-Puffer) in jedes Röhrchen geben, bei 1200 × g für 5 min zentrifugieren und den Überstand entsorgen. Wiederholen Sie diesen Waschschritt zweimal.
- Nach der Zentrifugation des Bakterienlysats eine 30 μL-Probe aus dem Überstand entfernen und den Überstand aus jedem Ultrazentrifugenröhrchen in ein entsprechendes 50 mL konisches Zentrifugenröhrchen übertragen, das saubere Perlen enthält. Für die Gelanalyse resuspendieren Sie das Trümmerpellet in einem der Ultrazentrifugenröhrchen mit 50 ml TND-Puffer und sammeln Sie eine 30 μL-Probe.
- Legen Sie die Rohre für 3-4 h bei 4 °C auf einen Rotator, um eine homogene Perlensuspension zu erhalten. Die Perlensuspensionen in einer leeren 25-ml-Chromatographiesäule bündeln. Lassen Sie die Perlen dekantieren und entfernen Sie den Puffer und die ungebundenen Proteine durch Schwerkraftfluss. Entnehmen Sie eine Probe aus dem Eluat zur Analyse.
- Resuspendieren Sie die Perlen mit 20 mL TND-Puffer und sammeln Sie das Eluat durch Schwerkraftfluss. Wiederholen Sie diesen Schritt zweimal, um die Perlen vollständig zu waschen. Bündeln Sie die gesammelten Eluate und bewahren Sie eine 30 μL-Probe für die weitere Analyse auf.
HINWEIS: Nach einer kurzenDekantierung sedimentiert ein Volumen von ~2 ml Perlensuspension, an der GST-C2 Lact befestigt ist, am Boden der Säule. - Fügen Sie 1 ml Perlensuspension zu zwei 2-ml-Mikrozentrifugenröhrchen hinzu. Füllen Sie jedes Röhrchen mit TND-Puffer bis zu einem Endvolumen von 1.970 ml. Entnehmen Sie eine 30 μL-Probe aus einem Röhrchen zur weiteren Analyse (B1-Probe). 10 μL 10 mMCaCl2-Lösung und 25 μL aus einer Stammlösung humaner Thrombinproteaselösung bei 0,02 U/μL werden hinzugefügt.
- Legen Sie die beiden Röhrchen über Nacht bei 4 °C auf einen Rotator, damit Thrombin das GST-Tag von derC2-Lact-Domäne abspalten kann. Am nächsten Tag mischen Sie in jeder Tube 10 μL 200 mM PMSF-Lösung mit der Perlensuspension, um die Thrombinwirkung zu hemmen.
- Zentrifugieren Sie die Röhrchen bei 700 × g für 5 min und sammeln Sie den Überstand, der löslicheC2-Lact-Domäne enthält, aus jedem Röhrchen, ohne die Perlen zu nehmen. Die Überstände werden in einem 2 mL Snap-Cap-Mikrozentrifugenröhrchen (E1-Eluat) zusammengefasst, das auf Eis gehalten wird.
- Fügen Sie 1 ml TND-Puffer zu jedem Röhrchen hinzu, um die Perlen wieder zu besuspendieren, und waschen Sie sie; Wiederholen Sie Schritt 1.2.14. Führen Sie diesen Schritt dreimal mehr durch, um eine maximale Menge an Protein zurückzugewinnen. Jedes Mal die gesammelten Überstände in einem neuen 2-ml-Röhrchen (E2-, E3-, E4- und E5-Eluate) bündeln und ein Aliquot zur weiteren Analyse verwenden. Nehmen Sie am Ende der Waschschritte ein Aliquot aus der Perlensuspension (Aliquot B2).
- Analysieren Sie die 30 μL-Proben, die in den verschiedenen Schritten des Reinigungsprotokolls durch SDS-PAGE-Trennung auf einem 15% Acrylamidgel gesammelt wurden.
- Entfernen Sie potenziell kontaminierende Perlen, indem Sie alle Überstände(d. h.~ 10 ml), die während der Schritte 1.2.14 und 1.2.15 gesammelt wurden, in einer 10-ml-Chromatographiesäule bündeln. Sammeln Sie das Eluat durch Schwerkraftfluss und behalten Sie die Perlen am Boden der Säule.
- Konzentrieren Sie die C2Lact Probe mit einer Zentrifugalfiltereinheit mit einer Molekulargewichts-Cut-off (MWCO) von 3 kDa und einer Zentrifugationsgeschwindigkeit von 2300 × g. Stoppen Sie das Konzentrationsverfahren, wenn das Volumen der Proteinprobe ~ 1 ml beträgt.
- Herstellung und Reinigung von NBD-C2Lakt
- Gleichgewichten einer Entsalzungsspalte (siehe Materialtabelle)mit TN-Puffer. Laden Sie die Säule mit 1 ml konzentrierterC2-Lact-Probe auf. Lassen Sie die Probe vollständig in das Gelbett gelangen, fügen Sie 1,5 ml frisch entgasten DTT-freien TN-Puffer in die Säule hinzu und sammeln Sie das Eluat durch Schwerkraftfluss in einem 2-ml-Snap-Cap-Mikrozentrifugenröhrchen.
- Verdünnen Sie 50 μL Eluat in einem Endvolumen von 300 μL TN-Puffer und zeichnen Sie ein Absorptionsspektrum von 230 bis 450 nm mit reinem TN-Puffer als Rohling auf. Bestimmen Sie dieC2-Lact-Konzentration basierend auf der bei 280 nm gemessenen Absorption unter Berücksichtigung eines Extinktionskoeffizienten ε gleich 44.920 M-1,cm-1.
- Um dasC2-Lact-Konstrukt mit einem NBD-Fluorophor zu kennzeichnen, mischen Sie das Protein mit einem zehnfachen molaren Überschuss an N,N'-Dimethyl-N-(iodoacetyl)-N'-(7-Nitrobenz-2-oxa-1,3-diazol-4-yl)ethylendiamin(IANBD-Amid).
- 1 mg IANBD werden in wasserfreiem Dimethylformamid (DMF) gelöst, wobei zu beachten ist, dass das endgültige DMF-Volumen, das zur Markierung desC2-Lact-Konstrukts verwendet wird, 5% (v / v) des Proteinprobenvolumens nicht überschreiten darf.
- Um das Volumen von DMF (VDMF)zu bestimmen, um IANBD aufzulösen, berechnen Sie zunächst die erforderliche Menge an IANBD (m, ausgedrückt in mg), um das Protein mit der Formel 1zu kennzeichnen.
m = 10.000 × C × V × MWIANBD (1)
wobei C die in Schritt 1.3.2 gemessene Konzentration vonC2-Lact, V das Volumen derC2-Lact-Probe und MWIANBD das Molekulargewicht des Fluorophors (420 g/mol) ist. - Berechnen Sie VDMF mit den Formeln 2 (mit m0= 1) und 3.
VDMF= (m0/m) × VIANBD (2)
VIANBD=0,05 × V (3)
Dabei ist m0 die Menge an IANBD-Pulver in mg und VIANBD das Volumen der IANBD-Lösung, die der C2-Lact-Probe zugesetzt werden soll. - Volumen VIANBD der frisch zubereiteten IANBD-Lösung in die C2 Lact-Probe geben und das Reaktionsgemisch bei 800-900 U/min für 30 min bei 25 °C mit einemlichtgeschützten Thermomixer schütteln. Lassen Sie die Reaktion 90 Minuten auf Eis ablaufen. In der Zwischenzeit eine Zentrifugalfiltereinheit (MWCO= 3 kDa) mit 10 mL TN-Puffer reinigen.
- L-Cystein (im 10-fachen molaren Überschuss zu IANBD) wird dem Reaktionsgemisch hinzugefügt, um freies IANBD zu inaktivieren.
- Fügen Sie der NBD-C2-Lact-Lösung 15 ml TN-Puffer hinzu und übertragen Sie die NBD-C2Lact-Lösung in die Zentrifugalfiltereinheit. Konzentrieren Sie die Probe auf 2 ml, um den größten Teil des freien NBD durch Zentrifugation bei 2300 × gvom Protein zu trennen. Wiederholen Sie diesen Waschschritt zweimal. Zentrifugieren Sie die Probe in einem 2 mL Schnappkappenzentrifugenröhrchen für 10 min bei 19.000 × g bei 4 °C, um potenzielle Aggregate zu pelletieren und den Überstand zu sammeln.
- Verdünnen Sie 50 μL Eluat in einem Endvolumen von 300 μL TN-Puffer. Erfassen Sie das Absorptionsspektrum von 230 bis 650 nm mit dem während des Konzentrationsverfahrens gesammelten Eluat als Rohling. Bestimmen Sie die NBD-C2-Lact-Konzentration unter Verwendung der maximalen Absorption bei λ=280 und 495 nm und extinktionskoeffizienten ε= 44.920 M-1.cm-1 (Protein) und 25.000 M-1.cm-1 (NBD-Fluorophor).
HINWEIS: Wenn die beiden Konzentrationswerte gleich sind, bedeutet dies, dass dasC2-Lact-Konstrukt im Verhältnis 1:1 mit der NBD-Gruppe gekennzeichnet ist. - Wenn die aus der Messung der NBD-Absorption geschätzte NBD-C2-Lact-Konzentration die aus der Absorption von Tryptophan (Trp)-Rückständen geschätzte Konzentration übersteigt, wiederholen Sie Schritt 1.3.5, um die freie NBD weiter zu entfernen.
- Fügen Sie der Probe Glycerin hinzu, um eine Endkonzentration von 10% (v / v) zu erhalten, um das NBD-C2-Lact-Konstrukt während des Schockgefrierens zu kryoschützen. Messen Sie die endgültige Proteinkonzentration.
- Bereiten Sie 50 μL Aliquoten Protein in 0,5 ml Snap-Cap-Mikrozentrifugenröhrchen vor. Gefrieren Sie die Röhrchen in flüssigem Stickstoff und lagern Sie sie bei -80 °C.
2. Reinigung von NBD-PHFAPP
HINWEIS: Das Verfahren zur Herstellung und Kennzeichnung von PHFAPP ist identisch mit dem von NBD-C2Lact bis zur Übergabe der NBD-C2Lact Lösung an eine Zentrifugalfiltereinheit in Schritt 1.3.4. Befolgen Sie ab diesem Schritt das unten beschriebene Protokoll.
- Halten Sie nach dem Konzentrationsschritt 2 ml NBD-PHFAPP bei 4 °C nicht mehr als 1 Tag im Dunkeln, bevor Sie eine Größenausschlusschromatographie durchführen. Stellen Sie vor der Größenausschlusschromatographie sicher, dass sich am Boden des Röhrchens keine orange Ablagerung (Aggregation während der Konzentration) befindet. Ist dies der Fall, zentrifugieren Sie die Probe bei 540.000 × g für 10 min bei 4 °C und reinigen Sie den Überstand durch Größenausschlusschromatographie.
HINWEIS: Die Größenausschlusschromatographie wird an einer Säule durchgeführt, die mit vernetztem Dextran-Acrylamid-Copolymer (siehe Materialtabelle)gefüllt ist, die zuvor mit TND-Puffer ausgeglichen wurde, wobei ein schnelles Proteinflüssigkeitschromatographiesystem verwendet wird (siehe Materialtabelle). Die Säule muss vor Licht geschützt werden. Es wurde eine Durchflussrate von 1 ml/min verwendet, und der Elution des NBD-PH FAPP-Konstrukts folgte die Aufzeichnung der Absorption bei λ= 280 (Protein) und 480 nm (NBD) am Säulenausgang.- Injizieren Sie die NBD-PH-FAPP-Probe, die in einer 2-ml-Injektionsschleife geladen ist, auf die Säule und sammeln Sie sofort 2,5 ml Eluatfraktionen.
- Analysieren Sie alle Fraktionen, die dem bei 280 und 480 nm detektierten Hauptpeak entsprechen, auf einem 15% SDS-PAGE Gel. Mischen Sie eine 25 μL-Probe jeder Fraktion mit 15 μL Laemmli-Probenpuffer, bevor Sie das Gel erhitzen und auf das Gel laden.
HINWEIS: Ein Hauptpeak, der gleichzeitig bei λ= 280 und 480 nm detektiert wird, erscheint, sobald ein Volumen von ~150 ml Puffer durch die Säule geleitet wird. - Bündeln Sie die Fraktionen, die ausschließlich NBD-PHFAPP-Protein (~ 12,2 kDa) enthalten, und fügen Sie Glycerin in einer Endkonzentration von 10% (v / v) hinzu. Konzentrieren Sie die Probe mit einer Zentrifugalfiltereinheit mit einem MWCO von 3 kDa auf ein Endvolumen von 1 ml bei einer Zentrifugationsgeschwindigkeit von 2300 × g.
- Bereiten Sie Aliquoten vor und zeichnen Sie ein Absorptionsspektrum auf, wie für NBD-C2Lactbeschrieben. Verwenden Sie einen Extinktionskoeffizienten ε= 29.450 M-1,cm-1, um die Konzentration des Proteins basierend auf der bei λ = 280 nm gemessenen Absorption zu bestimmen.
3. Herstellung von Liposomen für PS- und PI(4)P-Extraktions- oder Transferassays
HINWEIS: Führen Sie alle Schritte bei Raumtemperatur aus, sofern nicht anders angegeben. Behandeln Sie organische Lösungsmittel, Rotavapor und flüssigen Stickstoff mit Vorsicht.
- Bereiten Sie frische, filtrierte und entgaste 50 mM 4-(2-Hydroxyethyl)-1-piperazinethansulfonsäure (HEPES)-Kaliumhydroxid (KOH), pH 7,4, 120 mM Kaliumacetat (HK) Puffer vor.
- Nehmen Sie für jede Art von Liposom genaue Mengen verschiedener Lipide aus Stammlösungen und mischen Sie sie in einem birnenförmigen Glaskolben mit 25 ml (Tabelle 1). Fügen Sie reines Chloroform hinzu, um das Volumen jeder Mischung auf 1 ml einzustellen. Beschriften Sie jeden Kolben mit dem Liposomennamen. Wickeln Sie die mit Rhod-PE dotierten Kolben mit Lipidmischung mit Aluminiumfolie um.
- Legen Sie den Kolben auf einen Rotationsverdampfer. Trocknen Sie die Lipide unter Vakuum und bei 25 °C für mindestens 30 min bei einer Drehzahl von 500 U/min. Bei Lipidfilmen, die PI(4)P enthalten, den Kolben bei 32-34 °C unter sanfter Rotation für 5 min vorwärmen, um PI(4)P ordnungsgemäß mit den anderen Lipiden zu mischen, bevor Sie im Kolben Vakuum erzeugen, um das Lösungsmittel zu entfernen, das einen Film trockener Lipide an der Kolbenwand hinterlässt.
- Trennen Sie den Kolben vom Verdampfer und legen Sie ihn für 45 Minuten in eine Vakuumkammer, um alle verbleibenden Spuren von Lösungsmitteln zu entfernen. Füllen Sie den Kolben mit 2 ml HK-Puffer und fügen Sie der Lösung einige Glasperlen mit einem Durchmesser von 4 mm hinzu. Den Kolben 2 min vorsichtig vortexen, um die Lipide wieder aufzubereiten und multilamellare Lipidvesikel (MLVs) mit einer Lipidkonzentration von 4 mM vorzubereiten. Bereiten Sie 0,5 ml Aliquoten mlvs in 1,5 ml Schraubverschluss-Mikrozentrifugenröhrchen vor.
- Gefrieren-Tauen Sie die Röhrchen 5x ein (mit flüssigem Stickstoff bzw. einem Wasserbad bei 37 °C). Extrudieren Sie die Liposomen oder lagern Sie sie bei -20 °C.
- Verwenden Sie einen Mini-Extruder, um die Liposomen(d. H. Große unilamellare Vesikel) aus den MLVs gemäß den Richtlinien des Herstellers vorzubereiten. Verwenden Sie einen Polycarbonatfilter mit gleichmäßigen zylindrischen Poren von 200 nm Durchmesser.
- Um jede Art von Liposom vorzubereiten, extrudieren Sie mindestens 250 μL der entsprechenden Suspension von MLVs. Lagern Sie die extrudierten Liposomen bei 4 °C und im Dunkeln, wenn sie Rhod-PE enthalten. Verwenden Sie die Liposomen innerhalb von 2 Tagen.
| Lipidzusammensetzung (mol/mol) | Lipid | |||||
| Liposomenname | DOPC (25 mg/ml) | KNACKGERÄUSCHE (10 mg/ml) | 16:0 Liss Rhod-PE (1 mg/ml) | C16:0/C16:0-PI(4)P (1 mg/ml) | ||
| Extraktionsassays | Liposom 2 mol% PS | PC/PS 98/2 | 247 μL | 12,5 μL | ||
| Liposom 2 mol% PI(4)P | PC/PI(4)P 98/2 | 247 μL | 153 μL | |||
| PC-Liposom | PC 100 | 252 μL | ||||
| Transport-Assays | LA | PC/PS/Rhod-PE 93/5/2 | 234 μL | 31,4 μL | 200 μL | |
| LA ohne PS | PC/Rhod-PE 98/2 | 247 μL | 200 μL | |||
| LB | PC/PI(4)P 95/5 | 237 μL | 383 μL | |||
| LB ohne PI(4)P | PC 100 | 252 μL | ||||
| LA-Eq | PC/PS/PI(4)P/Rhod-PE 93/2,5/2,5/2 | 234 μL | 15,7 μL | 200 μL | 191 μL | |
| LB-Eq | PC/PS/PI(4)P 95/2,5/2,5 | 239 μL | 15,7 μL | 191 μL | ||
Tabelle 1: Mengen an Lipidstocklösungen, die für die Liposomenzubereitung gemischt werden sollen. Abkürzungen: PS= Phosphatidylserin; PC = Phosphatidylcholin; PI(4)P = Phosphatidylinositol-4-phosphat; Rhod-PE = Rhodamin-markiertes Phosphatidylethanolamin; DOPC = Dioleoylphosphatidylcholin; POPS= 1-Palmitoyl-2-oleoyl-sn-glycero-3-phospho-L-serin; 16:0 Liss Rhod-PE = 1,2-Dipalmitoyl-sn-glycero-3-phosphoethanolamin-N-(Lissamin Rhodamin B Sulfonyl).
4. Messung der PS- oder PI(4)P-Extraktion
HINWEIS: Die Messungen müssen mit einer schwarzen 96-Well-Platte und einem Fluoreszenzplattenleser durchgeführt werden, der mit Monochromatoren ausgestattet ist: einer für die Fluoreszenzanregung und einer für die Emission mit variabler Bandbreite.
- Bereiten Sie frischen, gefilterten und entgasten HK-Puffer vor, der mit 1 mM MgCl2 (HKM-Puffer) ergänzt wird. Herstellung reiner PC-Liposomen und PC-Liposomen, die mit 2 Mol-% PS oder 2 Mol-% PI(4)P dotiert sind (4 mM Endlipidkonzentration, siehe Tabelle 1).
HINWEIS: Halten Sie die Röhrchen während des gesamten Experiments mit der Suspension extrudierter Liposomen bei Raumtemperatur gefüllt und halten Sie die Proteine auf Eis. Schützen Sie zusätzlich die Lipidsensoren vor Licht. - Für den PS-Extraktionsassay werden liposomen mit 2 mol% PS (80 μM Endlipidkonzentration, 0,8 μM zugängliche PS-Konzentration) mit NBD-C2Lact (250 nM Endkonzentration) in einem Endvolumen von 100 μL gemischt. Füllen Sie eine zweite Vertiefung mit der gleichen Menge Liposom (80 μM, 2 mol% PS) und NBD-C2Lact (250 nM) gemischt mit 3 μM LTP (Osh6p als Positivkontrolle oder protein von Interesse).
HINWEIS: Eine Inkubationszeit von 5 min ist ausreichend, damit Osh6p eine Lipidextraktion erreicht. - Füllen Sie eine dritte Vertiefung mit NBD-C2Lact (250 nM) gemischt mit reinen PC-Liposomen (80 μM). Füllen Sie eine vierte Vertiefung nur mit reinen PC-Liposomen (80 μM). Wiederholen Sie die Schritte 4.2-4.3, um drei weitere Serien mit vier Bohrungen vorzubereiten.
- Zeichnen Sie für jede Bohrung ein NBD-Spektrum von 505 bis 650 nm (Bandbreite 5 nm) bei Anregung bei 490 nm (Bandbreite 5 nm) bei 25 °C auf. Subtrahieren Sie für jede Serie das Spektrum, das nur mit Liposomen aufgezeichnet wurde, von den anderen Spektren.
HINWEIS: F und Fmax entsprechen der Intensität bei 536 nm, die mit PS-haltigen Liposomen in Gegenwart bzw. Abwesenheit von LTP gemessen wird, während F0 die Intensität bei der gleichen Wellenlänge mit reinen PC-Liposomen ist. Für jede Serie wird der Prozentsatz des zugänglichen PS, der vom Protein extrahiert wird, mit der folgenden Formel angegeben.
100 × (1-((F-F0)/(Fmax-F0))) (4) - Bereiten Sie für den PI(4)P-Extraktionsassay Liposomen vor, die mit 2 Mol%-PI(4)P dotiert sind, und führen Sie Messungen mit derNBD-PH-FAPP-Sonde durch. Führen Sie Kontrollexperimente durch und bestimmen Sie den Extraktionsprozentsatz auf die gleiche Weise wie oben beschrieben.
HINWEIS: Die Liposomen- und Proteinkonzentration ist identisch mit denen, die im PS-Extraktionsassay verwendet werden.
5. Echtzeitmessung des PS-Transports
HINWEIS: Zur Aufzeichnung der Lipidtransferkinetik wird ein Standardfluorimeter (90°-Format) verwendet, das mit einem temperaturgesteuerten Zellhalter und einem Magnetrührer ausgestattet ist. Um Daten genau zu erfassen, ist es wichtig, die Probe dauerhaft auf der gleichen Temperatur zu halten (eingestellt zwischen 25 und 37 °C je nach Herkunft des Proteins(z.B.Hefe oder Mensch)) und ständig zu rühren. Das unten beschriebene Protokoll dient der Messung des Lipidtransports in einer 600 μL-Probe, die in einer zylindrischen Quarzzelle enthalten ist.
- Frisch entgasten und gefilterten HKM-Puffer zubereiten. Bewahren Sie die Röhrchen mit extrudierten Liposomen bei Raumtemperatur auf. Wickeln Sie Röhrchen, die Liposomen mit Rhod-PE enthalten, in Aluminiumfolie und/oder lagern Sie sie in einer undurchsichtigen Box, um ein Photobleaching zu verhindern.
- Stellen Sie die Anregungs- und Emissionsmonochromatoren bei λ = 460 nm (mit einer kurzen Bandbreite (1-3 nm)) bzw. bei λ = 530 nm (mit einer großen Bandbreite (≥ 10 nm)) ein. Stellen Sie die Erfassungszeit auf 25 min mit einer Zeitauflösung ≤ 1 s ein.
- In der Quarzküvette verdünnen Sie 30 μL der L-A-Liposomensuspension und ein Volumen NBD-C2-Lact-Stammlösung in vorgewarntem HKM-Puffer, um eine 570 μL-Probe vorzubereiten, die 200 μM Gesamtlipide und 250 nM NBD-C2Lactenthält. Fügen Sie einen kleinen magnetischen Rührbalken hinzu und positionieren Sie die Küvette im Fluorometerhalter.
- Sobald die Probe thermisch ausgeglichen ist (nach 3-5 min), lösen Sie die Messung aus. Nach 1 Minute werden der Probe 30 μL LB Liposomensuspension (Endkonzentration von 200 μM Gesamtlipiden) zugefüllt. Nach 3 min injizieren Sie LTP in die Probe, so dass die Endkonzentration des LTP 200 nM beträgt, und erfassen Sie das Signal für die verbleibenden 21 min.
- Führen Sie ein paralleles Experiment durch, um das NBD-Signal zu normalisieren. Mischen Sie 30 μL LA-Eq Liposomensuspension mit 250 nM NBD-C2Lact im HKM-Puffer (Endvolumen von 570 μL). Nach 1 minute injizieren Sie 30 μL LB-Eq Liposomensuspension.
HINWEIS: Die Lipidzusammensetzung der Liposomen LA-Eq und LB-Eq ähnelt der der im Transferassay verwendeten Liposomen LA und LB, mit der Ausnahme, dass jedes von ihnen 2,5 Mol%PS und 2,5 Mol%PI(4)P enthält. Infolgedessen entspricht das gemessene NBD-Signal, das alsF-Äqbezeichnet wird, dem Signal, das gemessen werden sollte, wenn PS zwischen LA- undL-B-Liposomen durch einen Transferprozess vollständig ausgeglichen wurde. - Konvertieren Sie die kinetischen Kurven, die mit einem LTP von Interesse gemessen wurden, um die Menge an PS (in μM) zu bestimmen, die im Laufe der Zeit von LA- auf L B-Liposomen übertragen wird. Normalisieren Sie jeden Datenpunkt (F) der Kurve mithilfe der folgenden Formel.
FNorm = (F-F0)/(FEq-F0) (5)
in dem F0 dem NBD-Signal kurz vor der Addition eines LTP entspricht und FEq das in Schritt 5.5 gemessene Signal ist.
HINWEIS: Die Menge an PS (in μM), die von LA- aufL-B-Liposomen übertragen wird, entspricht 2,5 ×F-Norm,wenn man bedenkt, dass das Gleichgewicht einer Situation entspricht, in der die Hälfte der zugänglichen PS-Moleküle, die im äußeren Blättchen derL-A-Liposomen enthalten sind(d. h.5 Mol-% von 0,5 × 200 μM Gesamtlipiden), inL-B-Liposomen übertragen wurden.
6. Echtzeitmessung des PI(4)P-Transports
- Stellen Sie das Fluorimeter (Anregungs- und Emissionswellenlänge, Bandbreite, Erfassungszeit, Zeitauflösung) wie für den PS-Transferassay ein. Verwenden Sie ebenfalls den gleichen Puffer, dieselbe Küvette und dieselben Liposomen, um die Experimente unter ständigem Rühren bei der gleichen Temperatur durchzuführen.
- Mischen Sie in der Küvette 30 μL LB Liposomensuspension und NBD-PHFAPP mit vorgewärmtem HKM-Puffer, um ein Endvolumen von 570 μL (200 μM Gesamtlipide, 250 nM NBD- PHFAPP)zu erhalten. Sobald das thermische Gleichgewicht der Probe erreicht ist, beginnen Sie mit der Messung und injizieren Sie nach 1 Minute 30 μL L A-Liposomensuspension. Nach 3 Minuten injizieren Sie den interessierenden LTP (Endkonzentration von 200 nM) und zeichnen sie das Signal auf.
- Führen Sie ein zweites Experiment durch, um das NBD-Signal zu normalisieren. Mischen Sie 30 μL LB-Eq Liposomensuspension mit 250 nM NBD-PHFAPP in 570 μL HKM-Puffer. Nach 1 minute 30 μL LA-Eq Liposomensuspension injizieren.
HINWEIS: Hier entspricht das NBD-Signal, das als FEqbezeichnet wird, demjenigen, das gemessen werden sollte, wenn PI(4)P zwischen LA- undL-B-Liposomen vollständig ausgeglichen war. - Wandeln Sie die kinetischen Kurven um, um die Menge an PI(4)P (in μM) zu bestimmen, die im Laufe der Zeit von LB- aufL-A-Liposomen übertragen wird. Jeder Datenpunkt (F) wird mit Formel 5 normalisiert, in der F0 dem NBD-Signal vor der Addition eines LTP entspricht und FEq das in Schritt 6.3 gemessene Signal ist.
HINWEIS: Die Menge an PI(4)P (in μM), die von LB- aufL-A-Liposomen übertragen wird, entspricht 2,5 ×F-Norm, wenn man bedenkt, dass das Gleichgewicht einer Situation entspricht, in der die Hälfte des PI(4)P, das in der äußeren Packungsbeilage derL-B-Liposomen enthaltenist (d. h. 0,5 × 5 μM), inL-A-Liposomen übertragen wurde.
7. Analyse kinetischer Kurven
- Quantifizieren Sie das Ausmaß, in dem ein LTP effizient ist, indem Sie die Geschwindigkeit bestimmen, mit der dieses LTP Lipide in den ersten Sekunden nach seiner Injektion in die Küvette von einer Liposomenpopulation auf die andere überträgt.
- Führen Sie eine lineare Regression der ersten Datenpunkte der Transferkinetik durch, um eine Steigung zu erhalten. Dividieren Sie den Steigungswert durch die LTP-Konzentration im Reaktionsgemisch, um die Anzahl der pro Protein pro Zeiteinheit (min oder s) übertragenen Lipidmoleküle zu bestimmen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
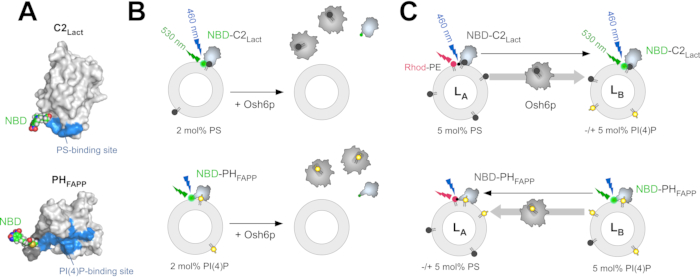
Abbildung 1: Beschreibung der fluoreszierenden Lipidsensoren und In-vitro-Assays. (A) Dreidimensionale Modelle von NBD-C2Lact und NBD-PHFAPP basierend auf der Kristallstruktur der C2-Domäne von Rinderlactadherin (PDB-ID: 3BN648) und der NMR-Struktur der PH-Domäne des menschlichen FAPP1...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Ergebnisse dieser Assays hängen direkt von den Signalen der fluoreszierenden Lipidsensoren ab. Daher ist die Reinigung dieser Sonden, die im Verhältnis 1:1 mit NBD und ohne freie NBD-Fluorophorkontamination markiert sind, ein kritischer Schritt in diesem Protokoll. Es ist auch obligatorisch zu überprüfen, ob das zu untersuchende LTP ordnungsgemäß gefaltet und nicht aggregiert ist. Die Menge an LTP, die in den Extraktionsassays getestet wurde, muss gleich oder höher als die von zugänglichen PS- oder PI(4)P-Mol...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Danksagungen
Wir danken Dr. A. Cuttriss für ihr sorgfältiges Korrekturlesen des Manuskripts. Diese Arbeit wird durch den Zuschuss der französischen Nationalen Forschungsagentur ExCHANGE (ANR-16-CE13-0006) und durch das CNRS finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| L-cysteine ≥97 % (FG) | Sigma | W326305-100G | Prepare a 10 mM L-cysteine stock solution in water. Aliquots are stored at -20 °C |
| 2 mL Amber Vial, PTFE/Rub Lnr, for lipids storage in CHCL3 | Wheaton | W224681 | |
| 4 mm-diameter glass beads | Sigma | Z265934-1EA | |
| 50 mL conical centrifuge tube | Falcon | ||
| ÄKTA purifier | GE healthcare | FPLC | |
| Aluminium foil | |||
| Amicon Ultra-15 with a MWCO of 3 and 10 kDa | Merck | UFC900324, UFC901024 | |
| Amicon Ultra-4 with a MWCO of 3 and 10 kDa | Merck | UFC800324, UFC801024 | |
| Ampicillin | Prepare a 50 mg/mL stock solution with filtered and sterilized water and store it at -20 °C. | ||
| Bestatin | Sigma | B8385-10mg | |
| BL21 Gold Competent Cells | Agilent | ||
| C16:0 Liss (Rhod-PE) in CHCl3 (1 mg/mL) | Avanti Polar Lipids | 810158C-5MG | |
| C16:0/C16:0-PI(4)P | Echelon Lipids | P-4016-3 | Dissolve 1 mg of C16:0/C16:0-PI(4)P powder in 250 µL of MeOH and 250 µL of CHCl3. Then complete with CHCl3 to 1 mL. The solution must become clear. |
| C16:0/C18:1-PS (POPS) in CHCl3 (10 mg/mL) | Avanti Polar Lipids | 840034C-25mg | |
| C18:1/C18:1-PC (DOPC) in CHCl3 (25 mg/mL) | Avanti Polar Lipids | 850375C-500mg | |
| CaCl2 | Sigma | Prepare 10 mM CaCl2 stock solution in water. | |
| Cell Disruptor | Constant Dynamics | ||
| Chloroform (CHCl3) RPE-ISO | Carlo Erba | 438601 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 5056489001 | |
| Deionized (Milli-Q) water | |||
| Dimethylformamide (DMF), anhydrous, >99% pure | |||
| DNAse I Recombinant, RNAse free, in powder | Roche | 10104159001 | |
| DTT | Euromedex | EU0006-B | Prepare 1 M DTT stock solution in Milli-Q water. Prepare 1 mL aliquots and store them at -20 °C. |
| Econo-Pac chromatography columns (1.5 × 12 cm). | Biorad | 7321010 | |
| Electroporation cuvette 2 mm | Ozyme | EP102 | |
| Electroporator Eppendorf 2510 | Eppendorf | ||
| Fixed-Angle Rotor Ti45 and Ti45 tubes | Beckman | Spinning the batcerial lysates | |
| Glass-syringes (10, 25, and 50 µL) for fluorescence experiment | Hamilton | ||
| Glass-syringes (25 , 100, 250, 500, and 1000 µL) to handle lipid stock solutions | Hamilton | 1702RNR, 1710RNR, 1725RNR, 1750RN type3, 1001RN | |
| Glutathione Sepharose 4B beads | GE Healthcare | 17-0756-05 | |
| Glycerol (99% pure) | Sigma | G5516-500ML | |
| Hemolysis tubes with a cap | |||
| HEPES , >99 % pure | Sigma | H3375-500G | |
| Illustra NAP 10 desalting column | GE healthcare | GE17-0854-02 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Euromedex | EU0008-B | Prepare 1 M IPTG stock solution in Milli-Qwater. Prepare 1 mL aliquots and store them at -20 °C. |
| K-Acetate | Prolabo | 26664.293 | |
| Lennox LB Broth medium without glucose | Prepared with milli-Q water and autoclaved. | ||
| Liquid nitrogen | Linde | ||
| Methanol (MeOH) ≥99.8% | VWR | 20847.24 | |
| MgCl2 | Sigma | Prepare a 2 M MgCl2 solution. Filter the solution using a 0.45 µm filter. | |
| Microplate 96 Well PS F-Botom Black Non-Binding | Greiner Bio-one | 655900 | |
| Mini-Extruder with two 1 mL gas-tight Hamilton syringes | Avanti Polar Lipids | 610023 | |
| Monochromator-based fluorescence plate reader | TECAN | M1000 Pro | |
| N,N'-Dimethyl-N-(Iodoacetyl)-N'-(7-Nitrobenz-2-Oxa-1,3-Diazol-4-yl)Ethylenediamine) (IANBD Amide) | Molecular Probes | Dissolve 25 mg of IANBD in 2.5 mL of dimethylsulfoxide (DMSO) and prepare 25 aliquot of 100 µL in 1.5 mL screw-cap tubes. Do not completely screw the cap. Then, remove DMSO in a freeze-dryer to obtain 1 mg of dry IANBD per tube. Tubes are closed and stored at -20 °C in the dark. | |
| NaCl | Sigma | S3014-1KG | |
| PBS | 137 mM NaCl, 2.7 mM KCl, 10 mM NaH2PO4, 1.8 mM KH2PO4, autoclaved and stored at 4 °C. | ||
| Pear-shaped glass flasks (25 mL, 14/23, Duran glass) | Duran Group | ||
| Pepstatin | Sigma | p5318-25mg | |
| pGEX-C2LACT plasmid | Available on request from our lab | ||
| pGEX-PHFAPP plasmid | Available on request from our lab | ||
| Phenylmethylsulfonyl fluoride (PMSF) ≥98.5% (GC) | Sigma | P7626-25g | Prepare a 200 mM PMSF stock solution in isopropanol |
| Phosphoramidon | Sigma | R7385-10mg | |
| Polycarbonate filters (19 mm in diameter) with pore size of 0.2 µm | Avanti Polar Lipids | 610006 | |
| Poly-Prep chromatography column (with a 0-2 mL bed volume and a 10 mL reservoir) | Biorad | 7311550 | |
| Prefilters (10 mm in diameter). | Avanti Polar Lipids | 610014 | |
| PyMOL | http://pymol.org/ | Construction of the 3D models of the proteins (Figure 1A) | |
| Quartz cuvette for UV/visible fluorescence (minimum volume of 600 µL) | Hellma | ||
| Quartz cuvettes | Hellma | ||
| Refrigerated centrifuge Eppendorf 5427R | Eppendorf | ||
| Rotary evaporator | Buchi | B-100 | |
| Screw-cap microcentriguge tubes (1.5 mL) | Sarsted | ||
| Small magnetic PFTE stirring bar (5 × 2 mm) | |||
| Snap-cap microcentriguge tubes (0.5, 1, and 2 mL) | Eppendorf | ||
| SYPRO orange | fluorescent stain to detect protein in SDS-PAGE gel | ||
| Thermomixer | Starlab | ||
| THROMBIN, FROM HUMAN PLASMA | Sigma | 10602400001 | Dissolve 20 units in 1 mL of milli-Q water and prepare 25 µL aliquots in 0.5 mL Eppendorf tubes. Then freeze and store at -80 °C. |
| Tris, ultra pure | MP | 819623 | |
| Ultracentrifuge L90K | Beckman | ||
| UV/Visible absorbance spectrophotometer | SAFAS | ||
| UV/visible spectrofluorometer with a temperature-controlled cell holder and stirring device | Jasco or Shimadzu | Jasco FP-8300 or Shimadzu RF-5301PC | |
| Vacuum chamber | |||
| Water bath | Julabo | ||
| XK 16/70 column packed with Sephacryl S200HR | GE healthcare |
Referenzen
- Drin, G. Topological regulation of lipid balance in cells. Annual Review of Biochemistry. 83, 51-77 (2014).
- Bigay, J., Antonny, B. Curvature, lipid packing, and electrostatics of membrane organelles: defining cellular territories in determining specificity. Developmental Cell. 23 (5), 886-895 (2012).
- Prinz, W. A. Lipid trafficking sans vesicles: where, why, how. Cell. 143 (6), 870-874 (2010).
- Holthuis, J. C., Menon, A. K. Lipid landscapes and pipelines in membrane homeostasis. Nature. 510 (7503), 48-57 (2014).
- Wong, L. H., Copic, A., Levine, T. P. Advances on the transfer of lipids by lipid transfer proteins. Trends in Biochemical Sciences. 42 (7), 516-530 (2017).
- Wong, L. H., Gatta, A. T., Levine, T. P. Lipid transfer proteins: the lipid commute via shuttles, bridges and tubes. Nature Reviews Molecular Cell Biology. 20 (2), 85-101 (2019).
- Iaea, D. B., Dikiy, I., Kiburu, I., Eliezer, D., Maxfield, F. R. STARD4 membrane interactions and sterol binding. Biochemistry. 54 (30), 4623-4636 (2015).
- Wilhelm, L. P., et al. STARD3 mediates endoplasmic reticulum-to-endosome cholesterol transport at membrane contact sites. The EMBO Journal. 36 (10), 1412-1433 (2017).
- Bian, X., Saheki, Y., De Camilli, P. Ca(2+) releases E-Syt1 autoinhibition to couple ER-plasma membrane tethering with lipid transport. The EMBO Journal. 37 (2), 219-234 (2018).
- Horenkamp, F. A., Valverde, D. P., Nunnari, J., Reinisch, K. M. Molecular basis for sterol transport by StART-like lipid transfer domains. The EMBO Journal. 37 (6), 98002(2018).
- Jentsch, J. A., et al. Structural basis of sterol binding and transport by a yeast StARkin domain. The Journal of Biological Chemistry. 293 (15), 5522-5531 (2018).
- Moser von Filseck, J., et al. INTRACELLULAR TRANSPORT. Phosphatidylserine transport by ORP/Osh proteins is driven by phosphatidylinositol 4-phosphate. Science. 349 (6246), 432-436 (2015).
- Daum, G., et al. Systematic analysis of yeast strains with possible defects in lipid metabolism. Yeast. 15 (7), 601-614 (1999).
- Ejsing, C. S., et al. Global analysis of the yeast lipidome by quantitative shotgun mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2136-2141 (2009).
- Leidl, K., Liebisch, G., Richter, D., Schmitz, G. Mass spectrometric analysis of lipid species of human circulating blood cells. Biochimica et Biophysica Acta. 1781 (10), 655-664 (2008).
- Sampaio, J. L., et al. Membrane lipidome of an epithelial cell line. Proceedings of the National Academy of Sciences of the United States of America. 108 (5), 1903-1907 (2011).
- Vance, J. E., Steenbergen, R. Metabolism and functions of phosphatidylserine. Progress in Lipid Research. 44 (4), 207-234 (2005).
- Zinser, E., et al. Phospholipid synthesis and lipid composition of subcellular membranes in the unicellular eukaryote Saccharomyces cerevisiae. Journal of Bacteriology. 173 (6), 2026-2034 (1991).
- Leventis, P. A., Grinstein, S. The distribution and function of phosphatidylserine in cellular membranes. Annual Review of Biophysics. 39, 407-427 (2010).
- Yeung, T., et al. Membrane phosphatidylserine regulates surface charge and protein localization. Science. 319 (5860), 210-213 (2008).
- Kim, J., Shishido, T., Jiang, X., Aderem, A., McLaughlin, S. Phosphorylation, high ionic strength, and calmodulin reverse the binding of MARCKS to phospholipid vesicles. Journal of Biological Chemistry. 269 (45), 28214-28219 (1994).
- Sigal, C. T., Zhou, W., Buser, C. A., McLaughlin, S., Resh, M. D. Amino-terminal basic residues of Src mediate membrane binding through electrostatic interaction with acidic phospholipids. Proceedings of the National Academy of Sciences. 91 (25), 12253-12257 (1994).
- Gal Bivona, T., et al. PKC regulates a farnesyl-electrostatic switch on K-Ras that promotes its association with Bcl-XL on mitochondria and induces apoptosis. Molecular Cell. 21 (4), 481-493 (2006).
- Finkielstein, C. V., Overduin, M., Capelluto, D. G. Cell migration and signaling specificity is determined by the phosphatidylserine recognition motif of Rac1. The Journal of Biological Chemistry. 281 (37), 27317-27326 (2006).
- Bolsover, S. R., Gomez-Fernandez, J. C., Corbalan-Garcia, S. Role of the Ca2+/Phosphatidylserine Binding Region of the C2 Domain in the Translocation of Protein Kinase Cα to the Plasma Membrane. Journal of Biological Chemistry. 278 (12), 10282-10290 (2003).
- Vance, J. E., Tasseva, G. Formation and function of phosphatidylserine and phosphatidylethanolamine in mammalian cells. Biochimica et Biophysica Acta. 1831 (3), 543-554 (2013).
- Maeda, K., et al. Interactome map uncovers phosphatidylserine transport by oxysterol-binding proteins. Nature. 501 (7466), 257-261 (2013).
- D'Ambrosio, J. M., et al. Osh6 requires Ist2 for localization to ER-PM contacts and efficient phosphatidylserine transport in budding yeast. Journal of Cell Science. 133 (11), 243733(2020).
- Manford, A. G., Stefan, C. J., Yuan, H. L., Macgurn, J. A., Emr, S. D. ER-to-plasma membrane tethering proteins regulate cell signaling and ER morphology. Developmental Cell. 23 (6), 1129-1140 (2012).
- Collado, J., et al. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- Hoffmann, P. C., et al. Tricalbins contribute to cellular lipid flux and form curved ER-PM contacts that are bridged by rod-shaped structures. Developmental Cell. 51 (4), 488-502 (2019).
- Lipp, N. F., et al. An electrostatic switching mechanism to control the lipid transfer activity of Osh6p. Nature Communications. 10 (1), 3926(2019).
- Chung, J., et al. INTRACELLULAR TRANSPORT. PI4P/phosphatidylserine countertransport at ORP5- and ORP8-mediated ER-plasma membrane contacts. Science. 349 (6246), 428-432 (2015).
- Sohn, M., et al. PI(4,5)P2 controls plasma membrane PI4P and PS levels via ORP5/8 recruitment to ER-PM contact sites. The Journal of Cell Biology. 217 (5), 1797-1813 (2018).
- Ghai, R., et al. ORP5 and ORP8 bind phosphatidylinositol-4, 5-biphosphate (PtdIns(4,5)P 2) and regulate its level at the plasma membrane. Nature Communications. 8 (1), 757(2017).
- Sohn, M., et al. Lenz-Majewski mutations in PTDSS1 affect phosphatidylinositol 4-phosphate metabolism at ER-PM and ER-Golgi junctions. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4314-4319 (2016).
- Kattan, W. E., et al. Targeting plasma membrane phosphatidylserine content to inhibit oncogenic KRAS function. Life Science Alliance. 2 (5), 00431(2019).
- Du, X., Turner, N., Yang, H. The role of oxysterol-binding protein and its related proteins in cancer. Seminars in Cell & Developmental Biology. 81, 149-153 (2018).
- Galmes, R., et al. ORP5/ORP8 localize to endoplasmic reticulum-mitochondria contacts and are involved in mitochondrial function. EMBO reports. 17 (6), 800-810 (2016).
- Du, X., et al. ORP5 localizes to ER-lipid droplet contacts and regulates the level of PI(4)P on lipid droplets. The Journal of Cell Biology. 219 (1), 201905162(2020).
- Wang, H., et al. ORP2 delivers cholesterol to the plasma membrane in exchange for phosphatidylinositol 4, 5-bisphosphate (PI(4,5)P2). Molecular Cell. 73 (3), 458-473 (2019).
- Kay, J. G., Grinstein, S. Sensing phosphatidylserine in cellular membranes. Sensors (Basel). 11 (2), 1744-1755 (2011).
- Moser von Filseck, J., Vanni, S., Mesmin, B., Antonny, B., Drin, G. A phosphatidylinositol-4-phosphate powered exchange mechanism to create a lipid gradient between membranes. Nature Communications. 6, 6671(2015).
- Wills, R. C., Goulden, B. D., Hammond, G. R. V. Genetically encoded lipid biosensors. Molecular Biology of the Cell. 29 (13), 1526-1532 (2018).
- Raychaudhuri, S., Im, Y. J., Hurley, J. H., Prinz, W. A. Nonvesicular sterol movement from plasma membrane to ER requires oxysterol-binding protein-related proteins and phosphoinositides. The Journal of Cell Biology. 173 (1), 107-119 (2006).
- Lenoir, M., et al. Structural basis of wedging the Golgi membrane by FAPP pleckstrin homology domains. EMBO reports. 11 (4), 279-284 (2010).
- Liu, Y., Kahn, R. A., Prestegard, J. H. Interaction of Fapp1 with Arf1 and PI4P at a membrane surface: an example of coincidence detection. Structure. 22 (3), 421-430 (2014).
- Shao, C., Novakovic, V. A., Head, J. F., Seaton, B. A., Gilbert, G. E. Crystal structure of lactadherin C2 domain at 1.7A resolution with mutational and computational analyses of its membrane-binding motif. The Journal of Biological Chemistry. 283 (11), 7230-7241 (2008).
- Lipp, N. F., Ikhlef, S., Milanini, J., Drin, G. Lipid exchangers: cellular functions and mechanistic links with phosphoinositide metabolism. Frontiers in Cell and Developmental Biology. 8, 663(2020).
- Venditti, R., et al. Molecular determinants of ER-Golgi contacts identified through a new FRET-FLIM system. The Journal of Cell Biology. 218 (3), 1055-1065 (2019).
- Pemberton, J. G., et al. Defining the subcellular distribution and metabolic channeling of phosphatidylinositol. The Journal of Cell Biology. 219 (3), (2020).
- Nakanishi, H., de los Santos, P., Neiman, A. M. Positive and negative regulation of a SNARE protein by control of intracellular localization. Molecular Biology of the Cell. 15 (4), 1802-1815 (2004).
- Maekawa, M., Yang, Y., Fairn, G. D. Perfringolysin O theta toxin as a tool to monitor the distribution and inhomogeneity of cholesterol in cellular membranes. Toxins. 8 (3), 67(2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten