JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
형광 기반 측정 인스파디딜세린/포스파디딜리노시톨 4-인산염 교환 막 간
요약
여기서, 우리는 단백질 추출물과 트랜스포파디딜리노시톨 4-인산염이 체외에서인산염을 배출하는지 여부를 결정하기 위해 형광 지질 센서와 리포좀을 사용하는 프로토콜을 설명합니다.
초록
진화적으로 보존된 옥시스테롤 결합 단백질(OSBP)-관련 단백질(ORP)/OSBP 동종학(Osh) 패밀리의 몇몇 구성원은 최근 효모 및 인간 세포에서 새로운 지질 전달 단백질(LTP) 그룹을 나타내는 것으로 밝혀졌다. 그들은 내피 성 망상 (ER)에서 PS /phosphatidylinosol 4-인산염 (PI(4)P) 교환 주기를 통해 혈장 막 (PM)으로 인산화 (PS)를 전송합니다. 이 발견은 신호 프로세스에 중요한 PS가 세포 전체에 분포되는 방법과 이 과정과 인포이노시타이드 (PIP) 대사 사이의 링크의 조사를 더 잘 이해할 수 있게 합니다. 새로운 형광 기반 프로토콜의 개발은 분자 수준에서 시험관내의 이 새로운 세포 기계장치의 발견 그리고 특성화에 중요한 역할을 했습니다. 이 논문은 두 개의 형광 표기 지질 센서, NBD-C2Lact 및 NBD-PHFAPP의생산 및 사용을 설명하며, PS 또는 PI(4)P를 추출하는 단백질의 능력을 측정하고 인공 막 사이에 이러한 지질을 전달합니다. 첫째, 프로토콜은 이 두 구문중의 고순도 샘플을 생성, 레이블 및 얻는 방법을 설명합니다. 둘째, 이 논문은 Osh6p를 사례 연구로 사용하여 단백질이 리포솜에서 PS 또는 PI(4)P를 추출할 수 있는지 여부를 결정하기 위해 형광 마이크로 플레이트 판독기와 이러한 센서를 사용하는 방법을 설명합니다. 마지막으로, 이 프로토콜은 정의된 지질 조성물의 지질성 간의 PS/PI(4)P 교환의 운동학을 정확하게 측정하고 표준 형광계를 사용하여 형광 공명 에너지 전달(FRET)에 의한 지질 전달 속도를 결정하는 방법을 보여줍니다.
서문
다른 막 사이 및 진핵 세포의 막 내지질의 정확한 분포1,2는 심오한 생물학적 연루가 있습니다. LtP함수가 세포 생물학3,4,5,6에서중요한 문제이며 시험관 내 접근 방식은 이 문제점7,8,9,10,11을해결하는 데 큰 가치가 있다. 여기서, 체외,형광계 전략은 여러 ORP/Osh 단백질이 PS/PI(4)P 교환을세포막(12)과 형성하여 새로운 종류의 LtP를 구성하는 데 중요한 역할을 하고 있다. ER과 PM 사이의 그라데이션을 따라 분포되며, 여기서각각17,18,19의글리세로포스지질의 5-7%와 최대 30%를 나타낸다. 더욱이, PS는 본질적으로 PM의 세포소 전단지에 집중된다. 이 빌드업 및 PM에서 PS의 고르지 않은 파티션은 셀룰러 신호공정(19)에매우 중요합니다. PS 분자의 음전하로 인해 PM의 세포소 전단지는 다른 세포기관1, 2,19,20의세포소 잎보다 훨씬 더 음이온적이다. 이를 통해 정전기력을 통해 채용할 수 있습니다. myristoylated alanine-rich C-kinase 기판 (MARCKS)21,육종 (Src)22,커스틴 쥐 육종 바이러스 종양 유전자 (K-Ras)23,및 Ras 관련 C3 보툴리눔 독소 기판 1 (Rac1) 기판 기판 1 (Rac1) 기판 과 같은 신호 단백질의 산산과 산의 잔디를 긍정적으로 함유하고 있습니다.
PS는 또한 C2도메인(25)을 통해 고정관시 선택적 방식으로 종래의 단백질 키나제 C에 의해 인식된다. 그러나 PS는 ER26에서합성되어 해당 역할을 하기 전에 PM으로 내보내야 한다는 것을 나타냅니다. 효모, Osh6p 및 Osh7p 전송 PS가 ER에서 PM27로전송되는 것을 발견할 때까지19이 어떻게 달성되었는지는 알려지지 않았습니다. 이 LtP는 그의 창립 멤버가 OSBP이고 단백질을 포함하는 진핵생물에 있는 진화적으로 보존된 가족에 속합니다 (인간에 있는 ORP, 효모에 있는 Osh 단백질) 지질 분자를 호스트하는 주머니와 OSBP 관련 도메인 (ORD)를 통합합니다. Osh6p 및 Osh7p는 구조적인 특징이 PS를 특별히 결합하고 멤브레인 간에 전송하도록 조정된 ORD로만 구성됩니다. 그럼에도 불구하고, 이러한 단백질이 ER에서 PM으로 PS를 방향으로 옮기는 방법은 불분명했습니다. Osh6p 및 Osh7p는 PI(4)P를 대체 지질 리간드12로트랩할 수 있다. 효모에서 PI(4)P는 골기내의 포스파디딜리노시톨(PI)과 PM에서 PI 4키나아제, 픽1p 및 Stt4p에 의해 각각 합성된다. 대조적으로, ER 멤브레인에는 PI(4)P가 없으며, 이 지질은 Sac1p 인스파타제에 의해 PI로 가수분해되기 때문이다. 따라서 ER/골기 및 ER/PM 인터페이스모두에 PI(4)P 그라데이션이 존재합니다. Osh6p 및 Osh7p 는 이 두 멤브레인(12) 사이에 존재하는 PI(4)P 그라데이션을 사용하여 PS/PI(4)P 교환 주기를 통해 ER에서 PM으로 PS를 전송한다.
한 주기 내에서, Osh6p는 ER로부터 PS를 추출하고, PM에서 PI(4)P에 대한 PS를 교환하고 PI(4)P를 ER로 다시 전송하여 다른 PS 분자를 추출한다. Osh6p/Osh7p는 Ist2p28과상호작용하며, ER 멤브레인과 PM을 서로 가깝게 연결하고 가져와서29,30,31에서ER-PM 접촉 부위를 생성한다. 또한, Osh6p의 부정적 하전 멤브레인과의 결합은 단백질이 정전기 특징을 수정하는 변형 변화로 인해 지질 리간드 중 하나를 추출하는 즉시 약해진다32. 이를 통해 Osh6p는 멤브레인 거주 시간을 단축하여 지질 전달 활동의 효율성을 유지합니다. Ist2p에 바인딩과 결합, 이 메커니즘은 빠르고 정확하게 ER / PM 인터페이스에서 지질 교환을 실행하는 Osh6p / 7p를 할 수 있습니다. 인간 세포에서, ORP5 및 ORP8 단백질은 뚜렷한 메커니즘을 통해 ER-PM 접촉 부위에서 PS/PI(4)P 교환을 실행한다33. 그들은 Osh6p와 유사한 중앙 ORD를 가지고 있지만, 직접 C 단자 막 세그먼트(33)를 통해 ER에 고정되고 PI (4)P및 PI (4,5)P233,34,35를인식하는 N 단말 플레크스트린 homology (PH) 도메인을 통해 PM에 도킹된다. ORP5/8은 PS를 전송하기 위해 PI(4)P를 사용하며, ORP5/8은 PM PI(4,5)P2 레벨을 추가로 조절하고 신호 경로를 조절하는 것으로 나타났다. 차례로, PI (4)P및 PI (4,5)P2 수준의 감소는 이러한 단백질이 PIP 의존적 방식으로 PM과 연관됨에 따라 ORP5 / ORP8 활성을 낮춥니다. 렌츠-마예스키 증후군으로 이어지는 비정상적으로 높은 PS 합성은 ORP5/836을통해 PI(4)P 수준에 영향을 미칩니다. 두 단백질의 활성이 막히면 PS가 PM에서 덜 풍부해져서단백질(37)의온코겐성 능력을 저하시한다.
반대로, ORP5 과발현은 암세포 침입 및 전이성과정(38)을촉진하는 것으로 보인다. 따라서, ORP5/8 활성에 대한 변경은 지질 항상성의 변화를 통해 세포 행동을 심각하게 수정할 수 있다. 또한, ORP5 및 ORP8은 ER-미토콘드리아 접촉 부위를 점유하고 PS39를공급함으로써 일부 미토콘드리아 기능을 보존한다. 또한, ORP5는 ER-지질 물방울 접촉 부위에 국한하여 PS/PI(4)P 교환40을통해 지질 방울에 PS를 전달한다. 본 명세서에 기재된 전략은 (i) 리포솜및 (ii) PS와 PI(4)P로부터 추출을 측정하기 위해 고안되어 Osh6p/Osh7p12,32의 PS/PI(4)P 교환활성을확립및 분석하기 위해 고안되었으며, 다른 그룹에서 ORP5/ORPP및10PS의활성을 분석하기 위해 사용되었다. 41. 각각 PS와 PI(4)P를 검출할 수 있는 형광판 판독기, 표준 L-포맷 분광계 및 2개의 형광 센서인 NBD-C2Lact 및 NBD-PHFAPP의사용을 기반으로 합니다.
NBD-C2Lact는 추정된 PS 결합 부위 근처에서 고유한 용매 노출 시스테인을 포함하도록 재설계된 당단백질, 락타데린의 C2 도메인에 대응한다. 극성 민감성 NBD(7-니트로벤즈-2-옥사-1,3-디아졸) 불소호레는 이 잔류물(도1A)12에이와 함께 연결된다. 보다 정확하게 말하자면, 락타데린의 C2 도메인(보스 타우러스, UniProt: Q95114, 잔류물 270-427)은 에셰리히아 대장균의글루타티온 S-transferase(GST)와 융합하여 발현되는 pGEX-4T3 벡터로 복제되었다. 그런 다음 C2Lact 서열은 두 개의 용매 접근 시스테인 잔류물을 대체하기 위해 돌연변이되었다(C270, C427) 알라닌 잔류물을 사용하여 이후에 N,N'-로 표시될 수 있는 퍼티제 PS 결합 부위(H352C 돌연변이) 근처의 부위에 시스테인 잔류물을 도입 디메틸-N-(요오도아세틸)-N'-(7-니트로벤츠-2-옥사-1,3-디아솔-4-yl) 에틸렌 디아민(IANBD) 12. 혈소판에 대한 분열 부위는 C2 도메인의 GST 단백질과 N-종점 사이에 존재한다. 주요 장점은 이 도메인이 다른 알려진 C2 도메인 또는 부속서 A542에반하는 Ca2+독립적인 방식으로 PS를 선택적으로 인식한다는 것입니다. NBD-PHFAPP는 인간 4-인산염 어댑터 단백질 1(FAPP1)의 PH 도메인으로부터 유래되었으며, 이는 PI(4)P 결합부위(도 1A)43근처의 NBD 그룹으로 표지될 수 있는 단일 용매 노출 시스테인을 포함하도록 재설계되었습니다. 인간 FAPP 단백질의 PH 도메인의 뉴클레오티드 서열(UniProt: Q9HB20, 세그먼트 [1-100])은 GST 태그와 함께 발현되는 pGEX-4T3 벡터로 복제되었다. PHFAPP 서열은단백질(43)의막 결합 인터페이스 내에서 고유한 시스테인 잔류물을 삽입하도록 변형되었다. 더욱이, 프로테아제에 대한 접근성을 보장하기 위해 혈전 분열 부위와 PH 도메인의 N-종자 사이에 9잔류 링커가 도입되었다.
리포솜에서 PS 추출을 측정하려면, NBD-C2Lact는 PS에 대한 친화력으로 인해 미량의 PS를 함유하는 인산염(PC)으로 만든 리포솜과 혼합되어 있으며, 이 구조는 리포솜에 결합하고, NBD 형광은 뇌의 소수성 환경과 접촉함에 따라 극성의 변화를 경험하며, 이는 조류가 증가하고 이는 조류가 증가한다. PS가 LTP의 stoichiometric 양에 의해 거의 완전히 추출되는 경우, 프로브는 리포솜과 연결되지 않으며, NBD 신호는 더낮다(도 1B)32. 신호의 이러한 차이는 LTP(예를들어, Osh6p)가 PS를 추출하는지 여부를 결정하는 데 사용됩니다. 유사한전략은 이전에 설명된 바와 같이 PI(4)P 추출(도1B)을측정하기 위해 NBD-PHFAPP와 함께사용된다. 두 개의 FRET 기반 아세약은 (i) LA에서 LB 리포솜으로 PS 수송을 측정하도록 설계되었으며, 이는 각각 ER 멤브레인과 PM을 모방하고(ii) PI(4)P를 역방향으로 수송하도록 설계되었습니다. 이러한 소는 PS/PI(4)P 교환을 측정하기 위해 동일한조건(즉, 동일한 완충, 온도 및 지질 농도)에서수행됩니다. PS 수송을 측정하기 위해, NBD-C2Lact는 PC로 구성된 LA 리포솜과 혼합되어 5 mol% PS와 2 mol%의 형광 로다민 라벨인 포스파디들레타놀라민(Rhod-PE)과 LB 리포솜이 5mol% PI(4)P를 통합한 것으로 나타났다.
시간 제로, Rhod-PE와 FRET는 NBD 형광을 담금질. PS가LA에서 LB 리포솜(예를들어, Osh6p주입 시)으로 수송되는 경우,LA에서 LB 리포좀(도1C)으로NBD-C2Lact 분자의 전역으로 인해 빠른 분해가 발생한다. 접근 가능한 PS의 양을 감안할 때, NBD-C2Lact는 실험(12)의과정을 통해 멤브레인 바운드 상태로 본질적으로 남아 있다. 따라서, NBD 신호의 강도는 LA와 LB 리포솜 사이의 NBD-C2Lact의 분포와 직접 상관관계가 있으며 쉽게 정규화되어 얼마나 많은 PS가 전달되는지 결정할 수 있다. 반대 방향으로 PI(4)P의 전송을 측정하기 위해, NBD-PHFAPP는 LA 및 LB 리포솜과 혼합된다; PI(4)P를 포함하는 LB 리포솜에만 결합하지만 Rhod-PE가 아니라는 점을 감안할 때 형광은 높습니다. PI(4)P가 LA 리포솜으로 옮겨지면 이러한 리포솜으로 배회되고, 로드-PE(도1C)를가진 FRET로 인해 신호가 감소한다. 신호는 정규화되어43로전송되는 PI(4)P양을 결정합니다.
프로토콜
1. NBD-C2수유의 정화
참고: 이 프로토콜은 박테리아를 부러뜨리기 위해 세포 파괴기의 사용을 자세히 설명하지만, 다른 용해전략(예: 프랑스 언론)을 사용하도록 수정할 수 있습니다. 정화의 시작 부분에서, 시스테인의 산화를 방지하기 위해 2mM 디티오스레이톨(DTT)으로 갓 탈착, 여과 및 보충되는 버퍼를 사용해야 한다. 그러나 단백질 라벨링 단계의 경우 DTT를 완전히 제거하는 것이 중요합니다. 단백질 분해를 피하기 위해 얼음이나 차가운 방에서 많은 단계를 수행해야 합니다. 30 μL 부피의 샘플은 15% 아크릴아미드 젤을 사용하여 나트륨 도데킬설페이트-폴리아크릴라미드 젤 전기포에 의해 분석을 수행하기 위해 프로토콜의 상이한 단계에서 수집되어야 정제의 진행 상황을 확인한다. 각 알리쿼트와 충분한 데칭 Laemmli 샘플 버퍼를 혼합하고 혼합물을 95°C에서 가열합니다. 동결 및 분석 될 때까지 -20 °C에 튜브를 저장합니다.
- 에슈리치아 대장균의 GST-C2Lact 의 표현
- BL21 골드 유능한 셀의 20 μL을 멸균 된 물 18 μL과 혼합합니다. 그런 다음 pGEX-C2Lact 플라스미드(~65 ng/μL)의 2 μL을 박테리아와 혼합하고 전기기화로 변환합니다. 오토클레이브 레녹스 리소게니-브로스(LB) 배지(10g/L 트립톤, 5g/L 효모 추출물, 탈화수, 포도당 없는 물 5g/L NaCl)의 150μL로 박테리아를 재연한다. 박테리아가 2mL 스냅 캡 마이크로 센트심분리기 튜브에서 1h로 37°C에서 자랄 수 있도록 하십시오.
- 125mL 멸균 에를렌마이어 플라스크에서 150μL의 세균 현탁액으로 50 μg/mL 암피실린으로 보충된 LB 배지25mL의 접종. 플라스크를 궤도 셰이커에 37°C로 놓고 박테리아가 185 rpm에서 교반하여 하룻밤 사이에 자도록 합니다.
- 멸균 2L Erlenmeyer 플라스크 2개를 50μg/mL 암피실린으로 보충한 LB 배지 500mL로 채우고 5mL의 프리컬쳐 서스펜션을 추가합니다. 박테리아가 220 rpm에서 동요와 37 °C에서 성장하자.
- 600nm의 파장(λ)에서 서스펜션의 광학 밀도(OD)를 주기적으로 측정한다. OD가 ~0.6-0.7의 값에 도달하면, GST-C2 Lact의 발현을 개시하기 위해 각 플라스크에 1 M 이소프로필 β-D-1-티오갈라c토피라노사이드(IPTG)의 스톡 용액의 500 μL을추가한다. 플라스크를 185 rpm에서 37°C에서 4시간 동안 흔들어 주세요.
- 각 플라스크의 내용내용을 폴리프로필렌 원심분리기 병에 옮길 수 있습니다. 4°C에서 4°C에서 30분 × 동안 2병원심분리하여 박테리아를 펠릿합니다. 상체를 버리고 각 펠릿을 50mL의 차가운 인산염 버퍼링 식염수로 재보선보세요.
- 각 병에 포함된 세균 현탁액을 50mL 원심분리기 튜브로 옮기다. 4°C에서 2300 × g에서 30분 동안 두 개의 튜브를 원심분리합니다. 상체를 제거하고 - 20 °C에서 세균 펠릿을 포함하는 각 튜브를 저장합니다.
- C2의 정화락트 (주)
- 얼음에, 50 mM Tris-HCl을 포함하는 버퍼의 50 mL와 두 개의 50 mL 원심 튜브를 채우기, pH 7.4, 150 mM NaCl (이하 TN 버퍼라고), 이전에 막 진공 여과에 의해 필터링 및 탈가스.
- 각 튜브에서 용해 버퍼를 준비하려면 가벼운 초음파 처리 또는 소용돌이에 의해 TN 버퍼에 에틸렌디아민 테트라아세트산 이없는 프로테아제 억제제 칵테일을 용해하십시오. 다른 항프로테아제(10μM 베스타틴, 1 μg/mL 펩스타틴 A, 10 μM 인스포라미드온)를 추가합니다. 중요한 것은 버퍼를 2mM DTT로 보완합니다.
- 각 튜브에서 30mL의 최종 부피를 얻기 위해 용해 버퍼로 1.1.6 단계에서 제조 된 세균 펠릿을 포함하는 두 개의 튜브를 채우고 10 분 동안 얼음에 펠릿을 천천히 해동하십시오. 스테인레스 스틸 주걱으로 각 펠릿을 분쇄하고, 튜브 및 /또는 균일 한 서스펜션이 얻을 때까지 피펫 컨트롤러와 25 mL 파이펫앞뒤로 서스펜션을 피펫하여 반복합니다.
- 저수지 내부에 샘플의 30mL를 적재하고 1.6 bar의압력으로 연속 모드에서 브레이킹 사이클을 실행하여 미리 냉각된 셀 중단기(재료 표 참조)를 사용하여 용해를 수행합니다. 동일한 튜브에서 용액을 수집하고, 튜브를 얼음 위에 보관하고, 이소프로파놀에 제조된 200m mM 페닐메틸설포닐 플루오라이드(PMSF)의 스톡 용액에서 즉시 250 μL을 추가합니다.
- 동일한 절차에 따라 다른 샘플을 lyse합니다. 나머지 용해 버퍼를 사용하여 셀 파괴기를 세척하고 세척을 수집하여 각 용액(~30mL)의 부피를 50mL의 최종 부피로 조정합니다.
- 각 용해5m MgCl2를보충하고, DNA를 단편화하고 따라서 샘플의 점도를 줄이기 위해 DNAse I의 20 μg /mL을 추가합니다. 30 분 동안 얼음에 배양. 젤 분석을 위해 샘플을 수집합니다.
- 각 50mL 리자테를 미리 냉각된 폴리카보네이트 초원심분리기 튜브로 옮춥니다(총 2개, 재료표참조). 원심분리기는 초원심분리기를 사용하여 4°C에서 186,000 × g에서 최소 1시간 동안.
- 원심분리 단계와 병행하여, 글루타티온을 함유한 슬러리 1.4mL을 50mL 원심원심튜브 2개로 4% 결합하고, 각각 튜브에 1mMM DTT(TND 버퍼)로 보충된 TN 버퍼 20mL, 1200g의 원심분리기를 1200g의 ×. 이 세탁 단계를 두 번 반복하십시오.
- 세균용 용해물의 원심분리 후, 상피체로부터 30 μL 샘플을 제거하고, 각 초원심분리기 튜브에서 깨끗한 구슬을 포함하는 50mL 원심분리기 튜브로 상전자를 이송한다. 겔 분석을 위해 TND 버퍼 50mL로 초원심분리기 튜브 중 하나에서 이물질 펠릿을 다시 중단하고 30 μL 샘플을 수집합니다.
- 동질비드 서스펜션을 얻기 위해 4°C에서 3-4h의 회전에 튜브를 놓습니다. 빈 25mL 크로마토그래피 컬럼에서 비드 서스펜션을 풀. 구슬을 데크런트하고 중력 흐름에 의해 완충제 및 결합되지 않은 단백질을 제거하십시오. 분석을 위해 용루에서 샘플을 채취하십시오.
- TND 버퍼20mL로 구슬을 재연하고 중력 흐름에 의해 용출을 수집합니다. 이 단계를 두 번 반복하여 구슬을 완전히 씻으세요. 수집된 용출을 풀, 추가 분석을 위해 30 μL 샘플을 유지합니다.
참고: 짧은 절충후 GST-C2Lact가 부착된 비드 서스펜션의 ~2mL의 부피, 열 하단의 퇴적물. - 2mL 스냅 캡 마이크로센심분리기 튜브에 1mL의 비드 서스펜션을 추가합니다. TND 버퍼로 각 튜브를 최종 부피 1.970 mL로 채웁니다. 추가 분석(B1 샘플)을 위해 한 튜브에서 30 μL 샘플을 채취하십시오. 인간 혈전 프로테아제용의 스톡 용액에서 10mM CaCl2 용액의 10 μL과 25 μL을 0.02 U/μL로 추가합니다.
- 혈소판이 C2 Lact 도메인에서 GST 태그를 갈라낼 수 있도록 하룻밤 사이에 회전에 두 개의 튜브를배치합니다. 다음 날, 각 튜브에서, 200 mM PMSF 용액의 10 μL을 비드 현탁액과 혼합하여 혈전 작용을 억제한다.
- 원심 분리튜브는 700 × g에서 5분 동안, 구슬을 복용하지 않고 각 튜브에서 수용성 C2Lact 도메인을 포함하는 상체를 수집합니다. 얼음 위에 보관되는 2mL 스냅 캡 마이크로센심분리기 튜브(E1 용출)로 수퍼네이츠를 풀.
- 각 튜브에 TND 버퍼 1mL을 추가하여 구슬을 재연하고 세척합니다. 반복 단계 1.2.14. 이 단계를 세 번 더 수행하여 최대 양의 단백질을 회수하십시오. 매번 수집된 수퍼나츠를 새로운 2mL 튜브(E2, E3, E4 및 E5 용출)로 풀을 만들고 추가 분석을 위해 알리쿼트를 사용한다. 세탁 단계가 끝나면 비드 서스펜션(알리쿼트 B2)에서 알리쿼트(aliquot B2)를 복용하십시오.
- 15% 아크릴아미드 겔에서 SDS-PAGE 분리에 의해 정제 프로토콜의 상이한 단계에서 수집된 30μL 샘플을 분석한다.
- 1.2.14 및 1.2.15단계에서 수집된 모든 수퍼나티(즉, ~10mL)를 10mL 크로마토그래피 컬럼으로 풀링하여 잠재적오염 구슬을 제거합니다. 중력 흐름에 의해 용액을 수집하고 컬럼 의 하단에 구슬을 유지합니다.
- 3kDa의 분자량 차단(MWCO)과 2300g의 원심분리 속도를 가진 원심 필터 유닛을 사용하여 C2Lact 샘플을 농축×. 단백질 샘플의 부피가 ~1mL일 때 농도 절차를 중단하십시오.
- NBD-C2의 준비 및 정화락트 (주)
- TN 버퍼를 사용하는 탈염 컬럼(재료 표참조)을 상화합니다. 1mL의 집중된 C2 Lact 샘플로 컬럼을로드합니다. 시료가 젤 침대에 완전히 들어가고, 1.5mL의 갓 탈가스DTT 없는 TN 버퍼를 컬럼에 추가하고, 중력 흐름에 의한 용출을 2mL 스냅 캡 마이크로센트심분리기 튜브로 수집한다.
- 300 μL TN 버퍼의 최종 부피에서 용출50 μL을 희석하고 순수 TN 버퍼를 공백으로 사용하여 230~450nm의 흡광스펙트럼을 기록합니다. 44,920M-1.cm-1에해당하는 멸종 계수 ε 고려하여 280nm에서 측정된 흡광도에 기초하여 C2Lact 농도를 결정한다.
- C2Lact 구조에 NBD 플루오로호레로 라벨을 부착하려면 단백질을 N,N'-디메틸-N-(이오도아세틸)-N'-(7니트로벤지-2-옥사-1,3-디아솔-4-yl)의10배 의 어과 함께 혼합한다.
- Anhydrous dimethylformamide (DMF)에 IANBD의 1 mg을 녹여, C2Lact 구조에 라벨을 붙이는 데 사용되는 DMF의 최종 볼륨이 단백질 샘플 볼륨의 5 % (v /v)를 초과해서는 안된다는 것을 명심하십시오.
- IANBD를 용해시키기 위해DMF(V DMF)의부피를 결정하기 위해, 먼저 포뮬러 1을사용하여 단백질을 라벨을 부착하기 위해 필요한 양의 IANBD(m, mg으로 발현)를 계산한다.
m = 10,000 × C × V × MWIANBD (1)
여기서 C는 1.3.2 단계에서 측정된 C2Lact의 농도이고, V는 C2Lact 샘플의 부피이며, MWIANBD는 불소호레(420 g/mol)의 분자량이다. - 수식 2(m 0=1) 및 3을사용하여 VDMF를 계산합니다.
VDMF= (m0/m)× VIANBD (2)
VIANBD=0.05 × V(3)
여기서 m0은 mg의 IANBD 분말의 양이며, VIANBD는 C2Lact 샘플에 첨가할 IANBD 용액의 부피이다. - C2Lact 샘플에 갓 준비된 IANBD 용액의 부피 VIANBD를 추가하고, 빛으로부터 보호되는 써모믹서를 사용하여 25°C에서 30분 동안 800-900 rpm에서 반응 혼합물을 흔들어 줍니다. 얼음에 90 분 동안 반응을 진행하자. 한편, TN 버퍼 10mL로 원심 필터 장치(MWCO= 3kDa)를 청소하십시오.
- L-시스테인을 첨가(IANBD에 10배 의 어과량)를 반응 혼합물에 첨가하여 무료 IANBD를 비활성화합니다.
- NBD-C2Lact 용액에 15mL의 TN 버퍼를 추가하고 NBD-C2Lact 용액을 원심 필터 단위로 전송합니다. 샘플을 2mL로 농축하여 2300g의 원심분리에 의해 단백질으로부터 대부분의 자유 NBD를 분리하는×. 이 세탁 단계를 두 번 반복하십시오. 원심분리기는 4°C에서 19,000 × g에서 10분 동안 2mL 스냅 캡 원심분리기 튜브에서 10분 동안 펠릿 잠재적 인 골재를 수집하고, 상부체를 수집한다.
- TN 버퍼의 300 μL의 최종 부피에서 용출량 50 μL을 희석시하십시오. 농도 시술 중에 수집된 용액을 공백으로 사용하여 230~650nm의 흡광도 스펙트럼을 기록한다. λ=280 및 495 nm 및 소멸 계수의 최대 흡광도를 사용하여 NBD-C2Lact 농도를 결정= ε =44,920 M-1.cm-1 (단백질) 및 25,000 M-1.cm-1 (NBD 플루오로포).
참고: 두 농도 값이 모두 인 경우 C2Lact 구문이 NBD 그룹과 1:1 비율로 레이블이 지정되어 있음을 나타냅니다. - NBD 흡광도의 측정으로부터 추정되는 NBD-C2Lact 농도가 트립토판(Trp) 잔류물의 흡광도로부터 추정되는 농도를 초과하는 경우, 반복 단계 1.3.5는 무료 NBD를 추가로 제거한다.
- 글리세롤을 시료에 추가하여 10%(v/v)의 최종 농도를 얻어 플래시 동결 시 NBD-C2Lact 구조를 보호합니다. 최종 단백질 농도를 측정합니다.
- 0.5mL 스냅 캡 마이크로센심분리기 튜브에 50 μL 알리쿼트 단백질을 준비합니다. 액체 질소에 튜브를 플래시 동결하고 -80 °C에 저장합니다.
2. NBD-PHFAPP의 정화
참고: PHFAPP를 생성하고 라벨을 부착하는 절차는 NBD-C2Lact 용액을 1.3.4단계에서 원심 필터 단위로 전송할 때까지 NBD-C2Lact의 것과 동일합니다. 이 단계부터 아래에 설명된 프로토콜을 따릅니다.
- 농도 단계 후, NBD-PHFAPP의 2mL을 어둠 속에서 4°C에서 1일 이상 크기 배제 크로마토그래피를 수행하기 전에 보관하십시오. 크기 배제 크로마토그래피 이전에는 튜브 바닥에 주황색 예금(농도 중 집계)이 없는지 확인합니다. 이 경우, 원심분리시 샘플을 540,000 g에서 4°C에서 10분 동안 ×, 크기 배제 크로마토그래피에 의해 상퍼를 정화한다.
참고: 크기 배제 크로마토그래피는 빠른 단백질 액체 크로마토그래피 시스템을 사용하여 이전에 TND 버퍼로 평형된 교차 연결된 드엑스트라아크릴아미드 공중합체(재료 표참조)로 포장된 컬럼에서 수행됩니다(재료표참조). 열은 빛으로부터 보호되어야 합니다. 1mL/min의 유속을 사용하였고, NBD-PHFAPP 구조의 용출은 기둥 출구에서 λ=280(단백질) 및 480 nm(NBD)에서 흡광도를 기록하였다.- 2mL 사출 루프에 로드된 NBD-PHFAPP 샘플을 컬럼에 주입하고 즉시 2.5mL 의 용출 분수를 수집합니다.
- 15% SDS-PAGE 젤에서 280 및 480 nm에서 검출된 주요 피크에 해당하는 모든 분획을 분석합니다. 젤에 가열 및 적재하기 전에 각 분획의 25 μL 샘플을 Laemmli 샘플 버퍼 15 μL과 혼합합니다.
참고: λ= 280 및 480 nm에서 동시에 감지되는 주 피크는 ~150mL 버퍼의 부피가 컬럼을 통과하면 나타납니다. - NBD-PHFAPP 단백질(~12.2kDa)을 독점적으로 함유한 분획을 풀고 10%(v/v)의 최종 농도로 글리세롤을 추가합니다. 3kDa의 MWCO가 있는 원심 필터 유닛을 사용하여 2300× g의원심분리 속도를 사용하여 1mL의 최종 부피에 샘플을 농축한다.
- 알리쿼트 준비, NBD-C2 Lact에 대해 설명된 대로 흡광스펙트럼을기록한다. 소멸 계수 ε= 29,450 M-1.cm-1을 사용하여 λ=280 nm에서 측정된 흡광도에 기초하여 단백질의 농도를 결정한다.
3. PS 및 PI (4)P 추출 또는 전송 분석용 리포솜 준비
참고: 달리 지정하지 않는 한 실온에서 모든 단계를 수행합니다. 유기 용매, 로타바포 및 액체 질소를 조심스럽게 처리하십시오.
- 신선하고 여과되고, 50mM 4-(2-하이드록스테틸)-1-파이프라진에탄황포닉산(HEPES)-수산화칼륨(KOH), pH 7.4, 120mMM칼륨 아세테이트(HK) 버퍼를 준비한다.
- 리포솜의 각 유형에 대해, 재고 용액에서 다른 지질의 정확한 양을 가지고, 25 mL 배 모양의 유리 플라스크(표 1)에혼합. 순수 클로로폼을 추가하여 각 혼합물의 부피를 1mL로 조정합니다. 각 플라스크에 리포솜 이름을 표시합니다. 알루미늄 호일로 Rhod-PE로 도핑된 지질 혼합물이 들어 있는 플라스크를 포장합니다.
- 플라스크를 회전 증발기위에 놓습니다. 500 rpm의 회전 속도로 적어도 30 분 동안 진공 상태에서 25 °C에서 지질을 건조시킵니다. PI(4)P를 포함하는 지질 필름의 경우, 플라스크를 32-34°C에서 5분 동안 미리 떼어낸 후, PI(4)P를 다른 지질과 적절히 혼합한 후 플라스크에서 진공을 생성하여 용매를 제거하여 플라스크 벽에 드라이 지질 필름을 남겨둡니다.
- 증발기에서 플라스크를 분리하고 45분 동안 진공 챔버에 배치하여 남은 용매 흔적을 제거합니다. 2mL의 HK 버퍼로 플라스크를 채우고 용액에 직경 4mm 의 유리 구슬을 추가합니다. 2분 동안 플라스크를 부드럽게 소용돌이쳐 지질을 재보풀시키고 지질 농도가 4m인 다각성 지질 소포(MlV)를 준비합니다. 1.5 mL 나사 캡 마이크로 센트심분리기 튜브에 MlV의 0.5 mL 알리쿼트준비.
- 튜브를 5배 동결해부림부(액체 질소 및 수조37°C의 수조 사용). 리포솜을 돌출하거나 -20 °C에 보관하십시오.
- 제조업체의 지침에 따라 MLV에서리포솜(즉, 대형 유니라멜라 소포)을 준비하기 위해 미니 압출기를 사용합니다. 직경 200nm의 균일한 원통형 모공을 가진 폴리카보네이트 필터를 사용하십시오.
- 리포솜의 각 유형을 준비하기 위하여는, MLV의 해당 현탁액의 적어도 250 μL를 돌출합니다. 2 일 이내에 리포솜을 사용하십시오.
| 지질 조성물(몰/몰) | 지질 | |||||
| 리포솜 이름 | DOPC (25 mg/mL) | 아빠 (10 mg/mL) | 16:0 리스 로드-PE (1 mg/mL) | C16:0/C16:0-PI(4)P (1 mg/mL) | ||
| 추출 분석 | 리포솜 2 몰% PS | PC/PS 98/2 | 247 μL | 12.5 μL | ||
| 리포솜 2 몰% PI(4)P | PC/PI(4)P 98/2 | 247 μL | 153 μL | |||
| PC 리포솜 | PC 100 | 252 μL | ||||
| 교통 에세이 | LA | PC/PS/로드-PE 93/5/2 | 234 μL | 31.4 μL | 200 μL | |
| PS가 없는 LA | PC/로드-PE 98/2 | 247 μL | 200 μL | |||
| LB | PC/PI(4)P 95/5 | 237 μL | 383 μL | |||
| PI가 없는 L B(4)P | PC 100 | 252 μL | ||||
| LA-Eq | PC/PS/PI(4)P/로드-PE 93/2.5/2.5/2 | 234 μL | 15.7 μL | 200 μL | 191 μL | |
| LB-Eq | PC/PS/PI(4)P 95/2.5/2.5 | 239 μL | 15.7 μL | 191 μL | ||
표 1: 립약 제제를 위해 혼합할 지질 스톡 솔루션의 볼륨. 약어: PS= 포스파디들세린; PC = 인산염 콜린; PI(4)P = 인산염 4-인산염; 론르-PE = 로다민 라벨인 포스파디들레타놀라민; DOPC = 디올릴포스파디딜콜린; POPS= 1-팔미토일-2-올레오일-sn-글리케로-3-인-L-세린; 16:0 리스 로드-PE = 1,2-디팔미토일-sn-글리세로-3-인포에탄올라민-N-(리사민 로다민 B 설포닐).
4. PS 또는 PI(4)P 추출 측정
참고: 블랙 96웰 플레이트와 단색색소가 장착된 형광판 판독기( 형광 흥분 발각용 및 방출용) 및 가변 대역폭을 사용하여 측정을 수행해야 합니다.
- 1 mM MgCl2 (HKM 버퍼)로 보충 된 신선하고 여과되고 탈가한 HK 버퍼를 준비하십시오. 순수 PC 리포솜과 PC 리포솜을 2mol% PS 또는 2mol% PI(4mM 최종 지질 농도, 표 1참조)로 도핑합니다.
참고: 실험 내내 실온에서 압출된 리포솜의 현탁액으로 채워진 튜브를 보관하고 단백질을 얼음 위에 보관하십시오. 또한 지질 센서를 빛으로부터 보호합니다. - PS 추출 분석의 경우, 1몰%PS(80 μM 최종 지질 농도)를 함유한 리포솜을 혼합하고, 0.8 μM 접근 PS 농도) NBD-C2Lact (250 nM 최종 농도)의 최종 부피 100 μL. 동일한 양의 리포솜 (80 μM, 2 mol % PS) 및 NBD-C2Lact (250 nM)가 3M μ LTP (Osh6p+ 단백질 조절)와 혼합된 두 번째 를 잘 채웁니다.
참고: Osh6p의 인큐베이션 시간은 지질 추출을 달성하기에 충분합니다. - 순수 PC 리포솜(80 μM)과 혼합된 NBD-C2 Lact(250 nM)로 세 번째 우물을 채웁니다. 순수한 PC 리포솜(80 μM)으로만 네 번째 우물을 채웁니다. 4.2-4.3 단계를 반복하여 4개의 우물 의 세 가지 추가 시리즈를 준비합니다.
- 각 우물에 대해, 25 °C에서 490 nm (대역폭 5 nm)에서 여기시 505 ~ 650 nm (대역폭 5 nm)에서 NBD 스펙트럼을 기록하십시오. 각 계열에 대해 다른 스펙트럼에서 리포솜만으로 기록된 스펙트럼을 뺍니다.
참고: F와 Fmax는 각각 LTP의 존재 또는 부재에서 PS 함유 리포솜으로 측정된 536nm의 강도에 대응하는 반면, F0은 순수 PC 리포좀이 있는 동일한 파장의 강도이다. 각 계열에 대해, 단백질에 의해 추출되는 접근 가능한 PS의 백분율은 다음 공식을 사용하여 주어집니다.
100 × (1-(F-F 0)/(F최대-F0))(4) - PI(4)P 추출 분석의 경우 2mol% PI(4)P로 도핑된 리포솜을 준비하고 NBD-PHFAPP 프로브로 측정을 수행합니다. 대조군 실험을 수행하고 위에서 설명한 것과 동일한 방식으로 추출 비율을 결정합니다.
참고: 리포솜 및 단백질 농도는 PS 추출 분석에서 사용되는 것과 동일합니다.
5. PS 수송의 실시간 측정
참고: 온도 조절 식 세포 홀더와 자기 교반기가 장착된 표준 불소계(90° 형식)는 지질 전달 운동학을 기록하는 데 사용됩니다. 데이터를 정확하게 수집하기 위해, 동일한온도(단백질(예:효모 또는 인간)의 기원에 따라 25~37°C 사이를 영구적으로 유지하고 지속적으로 저어주는 것이 핵심이다. 아래에 설명된 프로토콜은 원통형 석영 세포에 포함된 600 μL 샘플에서 지질 수송을 측정하기 위한 것입니다.
- 갓 탈바및 여과된 HKM 버퍼를 준비합니다. 압출 된 리포솜을 함유 한 튜브를 실온에서 유지하십시오. 알루미늄 호일에 Rhod-PE로 리포솜을 함유한 튜브를 감싸거나 불투명한 상자에 보관하여 광표백을 방지합니다.
- λ = 460 nm (짧은 대역폭 (1-3 nm)과 λ = 530 nm (큰 대역폭 (≥ 10 nm)에서 흥분 및 방출 단색소를 조정합니다. 1초 ≤ 시간 해상도로 획득 시간을 25분으로 설정합니다.
- 석영 큐벳에서는, LA 리포솜 서스펜션의 30 μL과 사전 온동된 HKM 버퍼에 NBD-C2Lact 스톡 용액의 부피를 희석하여 200 μM 총 지질 및 250 nM NBD-C2 Lact를 포함하는 570 μL 샘플을준비한다. 작은 자기 교반 바를 추가하고 플루오로미터 홀더에 큐벳을 배치합니다.
- 샘플이 열적으로 평형되면(3-5분 후) 측정을 트리거합니다. 1분 후, 샘플에 LB 리포솜 서스펜션(200 μM 총 지질의 최종 농도)의 30 μL을 추가합니다. 3분 후, LTP의 최종 농도가 200nM이되도록 샘플에 LTP를 주입하고, 나머지 21분 동안 신호를 획득한다.
- NBD 신호를 정규화하기 위해 병렬 실험을 수행합니다. HKM 버퍼(최종 부피 570 μL)에서 250nM NBD-C2Lact와 LA-Eq 리포솜 서스펜션 30μL을 혼합합니다. 1분 후 LB-Eq 리포솜 서스펜션 30μL를 주입합니다.
참고: LA-Eq 및 LB-Eq 리포좀의 지질 조성물은 각각 2.5 mol% PS 및 2.5 mol% PI(4)P를 포함하는 것을 제외하고는 전송 분석에 사용되는 LA 및 LB 리포솜과 유사합니다. 그 결과, FEq라고불리는 NBD 신호는 전송 프로세스에 의해 PS가 LA와 LB 리포솜 사이에서 완전히 평형화된 경우 측정되어야 하는 신호에 해당한다. - 관심 있는 LTP로 측정된 운동 곡선을 변환하여 시간이 지남에 따라 LA에서 LB 리포솜으로 전송되는 PS(μM)의 양을 결정합니다. 다음 수식을 사용하여 곡선의 각 데이터 점(F)을 정규화합니다.
F규범 = (F-F 0)/(FEq-F0)(5)
F0은 LTP를 첨가하기 직전에 NBD 신호에 대응하고, FEq는 5.5단계에서 측정된 신호이다.
참고:LA에서 LB 리포솜으로 전송되는 PS(μM)의 양은 2.5× FNorm에해당하며, 평형이LA 리포솜의 외부 전단지에 포함된 접근 가능한 PS 분자의 절반이0.5 × 200 μbm의 5mol%에 해당하는 상황에 해당한다는 점을 고려하면 2.5μm에 해당한다.
6. PI(4)P 수송의 실시간 측정
- PS 전송 분석기와 같이 불소계(여기 및 방출 파장, 대역폭, 획득 시간, 시간 해결)를 설정합니다. 마찬가지로 동일한 버퍼, 큐벳 및 리포솜을 사용하여 동일한 온도에서 일정한 교반에서 실험을 수행합니다.
- 큐벳에서,LB 리포솜 서스펜션과 NBD-PHFAPP의 30 μL을 사전 웜HKM 버퍼와 혼합하여 570 μL(200 μM 총 지질, 250nM NBD-PHFAPP)의최종 부피를 얻습니다. 시료의 열 평형에 도달하면 측정을 시작하고 1분 후에 LA 리포솜 서스펜션의 30 μL을 주입하십시오. 3분 후, 관심 있는 LTP(200nM의 최종 농도)를 주입하고 신호를 기록한다.
- NBD 신호를 정규화하기 위해 두 번째 실험을 수행합니다. LB-Eq 리포솜 서스펜션 30μL을 HKM 버퍼 570μL에 250nM NBD-PHFAPP와 혼합합니다. 1분 후 LA-Eq 리포솜 서스펜션 30μL를 주입합니다.
참고: 여기서, FEq라고불리는 NBD 신호는 PI(4)P가 LA와 LB 리포솜 사이에 완전히 평형화된 경우 측정되어야 하는 신호에 해당한다. - 운동 곡선을 변환하여 시간이 지남에 따라 LB에서 LA 리포솜으로 전송되는 PI(4)P(μM)의 양을 결정합니다. 각 데이터 포인트(F)는 F0이 LTP를 첨가하기 전에 NBD 신호에 대응하는 포뮬러 5를 사용하여 정규화되고, FEq는 6.3단계에서 측정된 신호이다.
참고:LB에서 LA로 전송되는 PI(4)P(μM)의 양은 2.5× FNorm에해당하며, 평형이 LB 리포솜의 바깥쪽 전단지에 포함된 PI(4)P의 절반이 LB 리포솜(즉,0.5 × 5 μM)의 외벽에 포함된 상황에 해당한다는 점을 고려하면Lo에서 전송되었습니다.
7. 운동 곡선 분석
- LTP가 큐벳에 주입 한 후 처음 몇 초 동안 한 지질 모집단에서 다른 지질을 전달하는 속도를 결정함으로써 LTP가 효율적인 정도를 정량화합니다.
- 경적을 얻기 위해 전송 역학의 첫 번째 데이터 포인트의 선형 회귀를 수행합니다. 반응 혼합물에서 LTP 농도로 경사 값을 나누어 시간 단위당 단백질당 전달되는 지질 분자의 수를 결정한다(min 또는 s).
결과
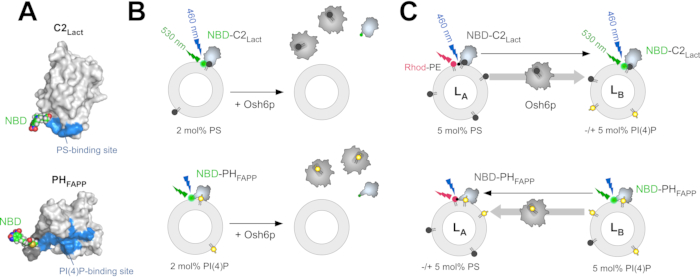
그림 1: 형광 지질 센서 및 시험관 내 작용에 대한 설명. (A)소 락타데린의 C2 도메인의 결정 구조를 기반으로 NBD-C2Lact 및 NBD-PHFAPP의 3차원 모델 (PDB ID: 3BN648)및 인간 FAPP1 단백질(PDB4K4K)의 PH 도메인의 NMR 구조(PDB4K: ID).
토론
이러한 아세의 결과는 형광 지질 센서의 신호에 직접적으로 의존합니다. 따라서, NBD를 가진 1:1 비율로 표기된 이들 프로브의 정제는 무료 NBD 불소포아 오염 없이 이 프로토콜에서 중요한 단계이다. 또한 검사 중인 LTP가 제대로 접혀 집계되지 않았는지 확인해야 합니다. 추출 분석에서 테스트된 LTP의 양은 이 LTP가 이러한 지질을 효율적으로 추출하는지 여부를 적절하게 측정하기 위해 접근 가능한 ...
공개
저자는 이해 상충이 없다고 선언합니다.
감사의 말
A. Cuttriss 박사가 원고를 주의 깊게 읽은 것에 대해 감사드립니다. 이 작품은 프랑스 국립 연구 기관 보조금 ExCHANGE (ANR-16-CE13-0006)와 CNRS에 의해 지원됩니다.
자료
| Name | Company | Catalog Number | Comments |
| L-cysteine ≥97 % (FG) | Sigma | W326305-100G | Prepare a 10 mM L-cysteine stock solution in water. Aliquots are stored at -20 °C |
| 2 mL Amber Vial, PTFE/Rub Lnr, for lipids storage in CHCL3 | Wheaton | W224681 | |
| 4 mm-diameter glass beads | Sigma | Z265934-1EA | |
| 50 mL conical centrifuge tube | Falcon | ||
| ÄKTA purifier | GE healthcare | FPLC | |
| Aluminium foil | |||
| Amicon Ultra-15 with a MWCO of 3 and 10 kDa | Merck | UFC900324, UFC901024 | |
| Amicon Ultra-4 with a MWCO of 3 and 10 kDa | Merck | UFC800324, UFC801024 | |
| Ampicillin | Prepare a 50 mg/mL stock solution with filtered and sterilized water and store it at -20 °C. | ||
| Bestatin | Sigma | B8385-10mg | |
| BL21 Gold Competent Cells | Agilent | ||
| C16:0 Liss (Rhod-PE) in CHCl3 (1 mg/mL) | Avanti Polar Lipids | 810158C-5MG | |
| C16:0/C16:0-PI(4)P | Echelon Lipids | P-4016-3 | Dissolve 1 mg of C16:0/C16:0-PI(4)P powder in 250 µL of MeOH and 250 µL of CHCl3. Then complete with CHCl3 to 1 mL. The solution must become clear. |
| C16:0/C18:1-PS (POPS) in CHCl3 (10 mg/mL) | Avanti Polar Lipids | 840034C-25mg | |
| C18:1/C18:1-PC (DOPC) in CHCl3 (25 mg/mL) | Avanti Polar Lipids | 850375C-500mg | |
| CaCl2 | Sigma | Prepare 10 mM CaCl2 stock solution in water. | |
| Cell Disruptor | Constant Dynamics | ||
| Chloroform (CHCl3) RPE-ISO | Carlo Erba | 438601 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 5056489001 | |
| Deionized (Milli-Q) water | |||
| Dimethylformamide (DMF), anhydrous, >99% pure | |||
| DNAse I Recombinant, RNAse free, in powder | Roche | 10104159001 | |
| DTT | Euromedex | EU0006-B | Prepare 1 M DTT stock solution in Milli-Q water. Prepare 1 mL aliquots and store them at -20 °C. |
| Econo-Pac chromatography columns (1.5 × 12 cm). | Biorad | 7321010 | |
| Electroporation cuvette 2 mm | Ozyme | EP102 | |
| Electroporator Eppendorf 2510 | Eppendorf | ||
| Fixed-Angle Rotor Ti45 and Ti45 tubes | Beckman | Spinning the batcerial lysates | |
| Glass-syringes (10, 25, and 50 µL) for fluorescence experiment | Hamilton | ||
| Glass-syringes (25 , 100, 250, 500, and 1000 µL) to handle lipid stock solutions | Hamilton | 1702RNR, 1710RNR, 1725RNR, 1750RN type3, 1001RN | |
| Glutathione Sepharose 4B beads | GE Healthcare | 17-0756-05 | |
| Glycerol (99% pure) | Sigma | G5516-500ML | |
| Hemolysis tubes with a cap | |||
| HEPES , >99 % pure | Sigma | H3375-500G | |
| Illustra NAP 10 desalting column | GE healthcare | GE17-0854-02 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Euromedex | EU0008-B | Prepare 1 M IPTG stock solution in Milli-Qwater. Prepare 1 mL aliquots and store them at -20 °C. |
| K-Acetate | Prolabo | 26664.293 | |
| Lennox LB Broth medium without glucose | Prepared with milli-Q water and autoclaved. | ||
| Liquid nitrogen | Linde | ||
| Methanol (MeOH) ≥99.8% | VWR | 20847.24 | |
| MgCl2 | Sigma | Prepare a 2 M MgCl2 solution. Filter the solution using a 0.45 µm filter. | |
| Microplate 96 Well PS F-Botom Black Non-Binding | Greiner Bio-one | 655900 | |
| Mini-Extruder with two 1 mL gas-tight Hamilton syringes | Avanti Polar Lipids | 610023 | |
| Monochromator-based fluorescence plate reader | TECAN | M1000 Pro | |
| N,N'-Dimethyl-N-(Iodoacetyl)-N'-(7-Nitrobenz-2-Oxa-1,3-Diazol-4-yl)Ethylenediamine) (IANBD Amide) | Molecular Probes | Dissolve 25 mg of IANBD in 2.5 mL of dimethylsulfoxide (DMSO) and prepare 25 aliquot of 100 µL in 1.5 mL screw-cap tubes. Do not completely screw the cap. Then, remove DMSO in a freeze-dryer to obtain 1 mg of dry IANBD per tube. Tubes are closed and stored at -20 °C in the dark. | |
| NaCl | Sigma | S3014-1KG | |
| PBS | 137 mM NaCl, 2.7 mM KCl, 10 mM NaH2PO4, 1.8 mM KH2PO4, autoclaved and stored at 4 °C. | ||
| Pear-shaped glass flasks (25 mL, 14/23, Duran glass) | Duran Group | ||
| Pepstatin | Sigma | p5318-25mg | |
| pGEX-C2LACT plasmid | Available on request from our lab | ||
| pGEX-PHFAPP plasmid | Available on request from our lab | ||
| Phenylmethylsulfonyl fluoride (PMSF) ≥98.5% (GC) | Sigma | P7626-25g | Prepare a 200 mM PMSF stock solution in isopropanol |
| Phosphoramidon | Sigma | R7385-10mg | |
| Polycarbonate filters (19 mm in diameter) with pore size of 0.2 µm | Avanti Polar Lipids | 610006 | |
| Poly-Prep chromatography column (with a 0-2 mL bed volume and a 10 mL reservoir) | Biorad | 7311550 | |
| Prefilters (10 mm in diameter). | Avanti Polar Lipids | 610014 | |
| PyMOL | http://pymol.org/ | Construction of the 3D models of the proteins (Figure 1A) | |
| Quartz cuvette for UV/visible fluorescence (minimum volume of 600 µL) | Hellma | ||
| Quartz cuvettes | Hellma | ||
| Refrigerated centrifuge Eppendorf 5427R | Eppendorf | ||
| Rotary evaporator | Buchi | B-100 | |
| Screw-cap microcentriguge tubes (1.5 mL) | Sarsted | ||
| Small magnetic PFTE stirring bar (5 × 2 mm) | |||
| Snap-cap microcentriguge tubes (0.5, 1, and 2 mL) | Eppendorf | ||
| SYPRO orange | fluorescent stain to detect protein in SDS-PAGE gel | ||
| Thermomixer | Starlab | ||
| THROMBIN, FROM HUMAN PLASMA | Sigma | 10602400001 | Dissolve 20 units in 1 mL of milli-Q water and prepare 25 µL aliquots in 0.5 mL Eppendorf tubes. Then freeze and store at -80 °C. |
| Tris, ultra pure | MP | 819623 | |
| Ultracentrifuge L90K | Beckman | ||
| UV/Visible absorbance spectrophotometer | SAFAS | ||
| UV/visible spectrofluorometer with a temperature-controlled cell holder and stirring device | Jasco or Shimadzu | Jasco FP-8300 or Shimadzu RF-5301PC | |
| Vacuum chamber | |||
| Water bath | Julabo | ||
| XK 16/70 column packed with Sephacryl S200HR | GE healthcare |
참고문헌
- Drin, G. Topological regulation of lipid balance in cells. Annual Review of Biochemistry. 83, 51-77 (2014).
- Bigay, J., Antonny, B. Curvature, lipid packing, and electrostatics of membrane organelles: defining cellular territories in determining specificity. Developmental Cell. 23 (5), 886-895 (2012).
- Prinz, W. A. Lipid trafficking sans vesicles: where, why, how. Cell. 143 (6), 870-874 (2010).
- Holthuis, J. C., Menon, A. K. Lipid landscapes and pipelines in membrane homeostasis. Nature. 510 (7503), 48-57 (2014).
- Wong, L. H., Copic, A., Levine, T. P. Advances on the transfer of lipids by lipid transfer proteins. Trends in Biochemical Sciences. 42 (7), 516-530 (2017).
- Wong, L. H., Gatta, A. T., Levine, T. P. Lipid transfer proteins: the lipid commute via shuttles, bridges and tubes. Nature Reviews Molecular Cell Biology. 20 (2), 85-101 (2019).
- Iaea, D. B., Dikiy, I., Kiburu, I., Eliezer, D., Maxfield, F. R. STARD4 membrane interactions and sterol binding. Biochemistry. 54 (30), 4623-4636 (2015).
- Wilhelm, L. P., et al. STARD3 mediates endoplasmic reticulum-to-endosome cholesterol transport at membrane contact sites. The EMBO Journal. 36 (10), 1412-1433 (2017).
- Bian, X., Saheki, Y., De Camilli, P. Ca(2+) releases E-Syt1 autoinhibition to couple ER-plasma membrane tethering with lipid transport. The EMBO Journal. 37 (2), 219-234 (2018).
- Horenkamp, F. A., Valverde, D. P., Nunnari, J., Reinisch, K. M. Molecular basis for sterol transport by StART-like lipid transfer domains. The EMBO Journal. 37 (6), 98002 (2018).
- Jentsch, J. A., et al. Structural basis of sterol binding and transport by a yeast StARkin domain. The Journal of Biological Chemistry. 293 (15), 5522-5531 (2018).
- Moser von Filseck, J., et al. INTRACELLULAR TRANSPORT. Phosphatidylserine transport by ORP/Osh proteins is driven by phosphatidylinositol 4-phosphate. Science. 349 (6246), 432-436 (2015).
- Daum, G., et al. Systematic analysis of yeast strains with possible defects in lipid metabolism. Yeast. 15 (7), 601-614 (1999).
- Ejsing, C. S., et al. Global analysis of the yeast lipidome by quantitative shotgun mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2136-2141 (2009).
- Leidl, K., Liebisch, G., Richter, D., Schmitz, G. Mass spectrometric analysis of lipid species of human circulating blood cells. Biochimica et Biophysica Acta. 1781 (10), 655-664 (2008).
- Sampaio, J. L., et al. Membrane lipidome of an epithelial cell line. Proceedings of the National Academy of Sciences of the United States of America. 108 (5), 1903-1907 (2011).
- Vance, J. E., Steenbergen, R. Metabolism and functions of phosphatidylserine. Progress in Lipid Research. 44 (4), 207-234 (2005).
- Zinser, E., et al. Phospholipid synthesis and lipid composition of subcellular membranes in the unicellular eukaryote Saccharomyces cerevisiae. Journal of Bacteriology. 173 (6), 2026-2034 (1991).
- Leventis, P. A., Grinstein, S. The distribution and function of phosphatidylserine in cellular membranes. Annual Review of Biophysics. 39, 407-427 (2010).
- Yeung, T., et al. Membrane phosphatidylserine regulates surface charge and protein localization. Science. 319 (5860), 210-213 (2008).
- Kim, J., Shishido, T., Jiang, X., Aderem, A., McLaughlin, S. Phosphorylation, high ionic strength, and calmodulin reverse the binding of MARCKS to phospholipid vesicles. Journal of Biological Chemistry. 269 (45), 28214-28219 (1994).
- Sigal, C. T., Zhou, W., Buser, C. A., McLaughlin, S., Resh, M. D. Amino-terminal basic residues of Src mediate membrane binding through electrostatic interaction with acidic phospholipids. Proceedings of the National Academy of Sciences. 91 (25), 12253-12257 (1994).
- Gal Bivona, T., et al. PKC regulates a farnesyl-electrostatic switch on K-Ras that promotes its association with Bcl-XL on mitochondria and induces apoptosis. Molecular Cell. 21 (4), 481-493 (2006).
- Finkielstein, C. V., Overduin, M., Capelluto, D. G. Cell migration and signaling specificity is determined by the phosphatidylserine recognition motif of Rac1. The Journal of Biological Chemistry. 281 (37), 27317-27326 (2006).
- Bolsover, S. R., Gomez-Fernandez, J. C., Corbalan-Garcia, S. Role of the Ca2+/Phosphatidylserine Binding Region of the C2 Domain in the Translocation of Protein Kinase Cα to the Plasma Membrane. Journal of Biological Chemistry. 278 (12), 10282-10290 (2003).
- Vance, J. E., Tasseva, G. Formation and function of phosphatidylserine and phosphatidylethanolamine in mammalian cells. Biochimica et Biophysica Acta. 1831 (3), 543-554 (2013).
- Maeda, K., et al. Interactome map uncovers phosphatidylserine transport by oxysterol-binding proteins. Nature. 501 (7466), 257-261 (2013).
- D'Ambrosio, J. M., et al. Osh6 requires Ist2 for localization to ER-PM contacts and efficient phosphatidylserine transport in budding yeast. Journal of Cell Science. 133 (11), 243733 (2020).
- Manford, A. G., Stefan, C. J., Yuan, H. L., Macgurn, J. A., Emr, S. D. ER-to-plasma membrane tethering proteins regulate cell signaling and ER morphology. Developmental Cell. 23 (6), 1129-1140 (2012).
- Collado, J., et al. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- Hoffmann, P. C., et al. Tricalbins contribute to cellular lipid flux and form curved ER-PM contacts that are bridged by rod-shaped structures. Developmental Cell. 51 (4), 488-502 (2019).
- Lipp, N. F., et al. An electrostatic switching mechanism to control the lipid transfer activity of Osh6p. Nature Communications. 10 (1), 3926 (2019).
- Chung, J., et al. INTRACELLULAR TRANSPORT. PI4P/phosphatidylserine countertransport at ORP5- and ORP8-mediated ER-plasma membrane contacts. Science. 349 (6246), 428-432 (2015).
- Sohn, M., et al. PI(4,5)P2 controls plasma membrane PI4P and PS levels via ORP5/8 recruitment to ER-PM contact sites. The Journal of Cell Biology. 217 (5), 1797-1813 (2018).
- Ghai, R., et al. ORP5 and ORP8 bind phosphatidylinositol-4, 5-biphosphate (PtdIns(4,5)P 2) and regulate its level at the plasma membrane. Nature Communications. 8 (1), 757 (2017).
- Sohn, M., et al. Lenz-Majewski mutations in PTDSS1 affect phosphatidylinositol 4-phosphate metabolism at ER-PM and ER-Golgi junctions. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4314-4319 (2016).
- Kattan, W. E., et al. Targeting plasma membrane phosphatidylserine content to inhibit oncogenic KRAS function. Life Science Alliance. 2 (5), 00431 (2019).
- Du, X., Turner, N., Yang, H. The role of oxysterol-binding protein and its related proteins in cancer. Seminars in Cell & Developmental Biology. 81, 149-153 (2018).
- Galmes, R., et al. ORP5/ORP8 localize to endoplasmic reticulum-mitochondria contacts and are involved in mitochondrial function. EMBO reports. 17 (6), 800-810 (2016).
- Du, X., et al. ORP5 localizes to ER-lipid droplet contacts and regulates the level of PI(4)P on lipid droplets. The Journal of Cell Biology. 219 (1), 201905162 (2020).
- Wang, H., et al. ORP2 delivers cholesterol to the plasma membrane in exchange for phosphatidylinositol 4, 5-bisphosphate (PI(4,5)P2). Molecular Cell. 73 (3), 458-473 (2019).
- Kay, J. G., Grinstein, S. Sensing phosphatidylserine in cellular membranes. Sensors (Basel). 11 (2), 1744-1755 (2011).
- Moser von Filseck, J., Vanni, S., Mesmin, B., Antonny, B., Drin, G. A phosphatidylinositol-4-phosphate powered exchange mechanism to create a lipid gradient between membranes. Nature Communications. 6, 6671 (2015).
- Wills, R. C., Goulden, B. D., Hammond, G. R. V. Genetically encoded lipid biosensors. Molecular Biology of the Cell. 29 (13), 1526-1532 (2018).
- Raychaudhuri, S., Im, Y. J., Hurley, J. H., Prinz, W. A. Nonvesicular sterol movement from plasma membrane to ER requires oxysterol-binding protein-related proteins and phosphoinositides. The Journal of Cell Biology. 173 (1), 107-119 (2006).
- Lenoir, M., et al. Structural basis of wedging the Golgi membrane by FAPP pleckstrin homology domains. EMBO reports. 11 (4), 279-284 (2010).
- Liu, Y., Kahn, R. A., Prestegard, J. H. Interaction of Fapp1 with Arf1 and PI4P at a membrane surface: an example of coincidence detection. Structure. 22 (3), 421-430 (2014).
- Shao, C., Novakovic, V. A., Head, J. F., Seaton, B. A., Gilbert, G. E. Crystal structure of lactadherin C2 domain at 1.7A resolution with mutational and computational analyses of its membrane-binding motif. The Journal of Biological Chemistry. 283 (11), 7230-7241 (2008).
- Lipp, N. F., Ikhlef, S., Milanini, J., Drin, G. Lipid exchangers: cellular functions and mechanistic links with phosphoinositide metabolism. Frontiers in Cell and Developmental Biology. 8, 663 (2020).
- Venditti, R., et al. Molecular determinants of ER-Golgi contacts identified through a new FRET-FLIM system. The Journal of Cell Biology. 218 (3), 1055-1065 (2019).
- Pemberton, J. G., et al. Defining the subcellular distribution and metabolic channeling of phosphatidylinositol. The Journal of Cell Biology. 219 (3), (2020).
- Nakanishi, H., de los Santos, P., Neiman, A. M. Positive and negative regulation of a SNARE protein by control of intracellular localization. Molecular Biology of the Cell. 15 (4), 1802-1815 (2004).
- Maekawa, M., Yang, Y., Fairn, G. D. Perfringolysin O theta toxin as a tool to monitor the distribution and inhomogeneity of cholesterol in cellular membranes. Toxins. 8 (3), 67 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유