Method Article
TGF-β mediada por la transición endotelial a mesenquimal (EndMT) y la evaluación funcional de los efectores endMT utilizando la edición de genes CRISPR / Cas9
En este artículo
Resumen
Se describen métodos para investigar TGF-β2 inducida por EndMT en células endoteliales mediante la observación de los cambios en la morfología celular y el examen de la expresión endMT relacionados con los cambios de marcador utilizando la tinción de inmunofluorescencia. La edición del gen CRISPR/Cas9 fue descrita y utilizada para agotar el gen que codifica el caracol para investigar su papel en endMT inducido por TGF-β2.
Resumen
En respuesta a señales externas específicas y la activación de ciertos factores de transcripción, las células endoteliales pueden diferenciarse en un fenotipo mesenquimal, un proceso que se denomina transición endotelial a mesenquimal (EndMT). Los resultados emergentes han sugerido que EndMT está causalmente ligado a las enfermedades humanas múltiples, tales como fibrosis y cáncer. Además, las células mesenquimales derivadas de endoteliales se pueden aplicar en procedimientos de regeneración de tejidos, ya que se pueden diferenciar aún más en varios tipos de células (por ejemplo, osteoblastos y condrocitos). Así, la manipulación selectiva de EndMT puede tener potencial clínico. Al igual que la transición epitelial-mesenquimal (EMT), EndMT puede ser fuertemente inducido por la citoquina secretada que transforma el factor de crecimiento beta (TGF-β), que estimula la expresión de los llamados factores de transcripción EndMT (EndMT-TFs), incluyendo Caracol y. Estos EndMT-TFs entonces suben y bajan los niveles de proteínas mesenquimales y endoteliales, respectivamente. Aquí, se describen los métodos para investigar TGF-β inducida endMT in vitro, incluyendo un protocolo para estudiar el papel de los TFs particulares en TGF-β inducida EndMT. Utilizando estas técnicas, proporcionamos evidencia de que TGF-β2 estimula EndMT en células endoteliales microvasculares pancreáticas murinas (células MS-1), y que el agotamiento genético de Caracol utilizando repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR)/CRISPR-proteína asociada 9 (Cas9)-mediada por la edición de genes, abroga este fenómeno. Este acercamiento puede servir como modelo para interrogar a moduladores potenciales de la biología endotelial, y se puede utilizar para realizar las pantallas genéticas o farmacológicas para identificar reguladores nuevos de EndMT, con el uso potencial en enfermedad humana.
Introducción
La transición endotelial a mesenquimal (EndMT) es un fenómeno biológico de varios pasos y dinámico que se ha relacionado con diversos procesos fisiológicos y patológicos1,2. Al final de laMT las células endoteliales pierden gradualmente sus rasgos endoteliales, mientras que adquieren propiedades mesenquimales3; así, las células endoteliales firmemente compactadas y bien organizadas se diferencian en células mesenquimales alargadas. Los cambios morfológicos en EndMT coinciden con alteraciones en la expresión de ciertos genes y proteínas. En general, la expresión de proteínas que mantienen características endoteliales, incluyendo endotelial vascular (VE)-cadherina, molécula de adhesión plaqueta/CE-1 (CD31/Pecam-1) disminuye. Simultáneamente, se acumulan proteínas relacionadas con las funciones mesenquimales, como la actina del músculo liso α (α-Sma) y la proteína muscular lisa 22α (Sm22α). Los resultados emergentes han demostrado que el EndMT postnatal contribuye al desarrollo de enfermedades humanas, como el cáncer, la fibrosis cardíaca, la hipertensión arterial pulmonar (HAP), la aterosclerosis (EA), la fibrosis de órganos,etc. 2,4,5,6,7. Una comprensión más profunda de los mecanismos subyacentes de EndMT y cómo dirigir el proceso de EndMT proporcionará nuevos métodos terapéuticos para las enfermedades relacionadas con EndMT y la medicina regenerativa.
TGF-β es uno de los principales inductores de EndMT, y otros factores involucrados conocidos incluyen Wnt / β-catenina, Muesca, y algunas citoquinas inflamatorias1. Como el contexto celular es clave para las respuestas desencadenadas por TGF-β, la interacción de TGF-β con otras señales promotoras de EndMT es relevante para que la β de TGF provoque una respuesta endMT. Sobre la activación de los receptores de la serina/de la cinasa de la serina/de la treonina del tipo de la superficie de la célula de TGF-β, se activa el camino canónico intracelular de Smad. TGF-β mediada por receptores fosforilados Smad2/3 forman complejos heteroméricos con Smad4 que se translocan en el núcleo, donde regular al alza la expresión de los factores de transcripción relacionados con EndMT. Similar a la transición epitelial-mesenquimal (EMT), los factores de transcripción como Snail, Slug, Twist, Zeb1 y Zeb2 son inducidos por la señalización de TGF-β y contribuyen a la reprogramación génica en EndMT8.
El caracol se ha identificado con frecuencia como factor dominante en EndMT. El caracol se une al promotor de genes que codifican proteínas de adhesión célula-célula y suprime su transcripción, que se ve contrarrestado por la mejora de la expresión de proteínas mesenquimales9. Las células endoteliales comprenden una población muy heterogénea y la influencia relativa de diversos estímulos extracelulares en endMT puede diferir entre contextos celulares endoteliales o tipos celulares10. Debido a sus similitudes con la EMT, algunas metodologías son útiles para investigar tanto los mecanismos EMT como endMT8. A este respecto, la Asociación Internacional de la EMT (TEMTIA) hace hincapié en la necesidad de técnicas complementarias para demostrar en última instancia la aparición de la EMT/EndMT11.
Aquí describimos un método para monitorear y visualizar el proceso de EndMT inducido por TGF-β. La tinción por inmunofluorescencia proporciona la información básica sobre los cambios de expresión en las proteínas/marcadores específicos, que se utilizan como indicadores de si se produce el proceso EndMT. Además, la tinción de inmunofluorescencia puede visualizar la localización de proteínas/marcadores y la morfología celular. Para estudiar la actividad potencial de TFs específicos (u otros reguladores aguas arriba o aguas abajo) involucrados en TGF-β mediada endMT, se describe un protocolo utilizando agrupados regularmente interespaciadas repeticiones palindrómicas cortas (CRISPR) / CRISPR-asociado a la proteína 9 (Cas9) edición de genes para agotar genes específicos de las células, utilizando el TF Caracol como ejemplo. Cas9 es una endonucleasa de ADN dual guiada por ARN que reconoce y escinde secuencias complementarias a las secuencias CRISPR en bacterias12. El sistema CRISPR/Cas9 es actualmente ampliamente utilizado porque facilita la ingeniería genética in vitro e in vivo13. Dirigido por un ARN de guía única (sgRNA), Cas9 expresado ectópicamente genera una ruptura de doble cadena en una secuencia de focalización preseleccionada en un locus genético específico. La unión final no homóloga (NHEJ) tiene lugar para reparar las roturas de hebras inducidas por Cas9, a través de inserciones o deleciones aleatorias de nucleótidos que conducen a la interrupción e inactivación del gen objetivo. Se describen en detalle los métodos para el diseño de sgRNAs selectivos y la generación de vectores lentiviral-compatibles que contienen los sgRNAs diseñados. Como resultado, las células endoteliales estables y agotadas por genes se pueden generar de una manera eficiente y confiable.
En este estudio, utilizamos células endoteliales microvasculares pancreáticas murinas (MS-1)14 como un sistema modelo para examinar el proceso endMT inducido por TGF-β2. Nuestro estudio anterior demostró que Snail es el principal factor de transcripción aumentado por TGF-β2, por el cual EndMT es inducido en células MS-115. Tras la edición del gen CRISPR/Cas9 para derogar la expresión de Snail en células MS-1, TGF-β2 no pudo mediar endMT. Este flujo de trabajo se puede aplicar para estudiar otros genes relacionados con EndMT (sospechosos).
Protocolo
1. Inducción de EndMT por TGF-β2
- Células MS-1 en el medio eagle modificado de Dulbecco (DMEM) que contiene 10% de suero bovino fetal (FBS) y 100 U/mL de penicilina/estreptomicina en una incubadora (5% de CO2,37 °C). Cubra todos los platos / platos de cultivo con gelatina al 0.1% p / v durante 10 minutos antes de su uso.
- Lave suavemente las células MS-1 con solución salina tamponada con 1x fosfato (PBS), agregue 2 mL de solución de tripsina-EDTA (0,25% de tripsina y 0,02% de EDTA) a un plato de 10 cm e incube durante 2 minutos a 37 °C para separarlas. Posteriormente, añadir 5 mL de medio de cultivo completo para apagar la reacción.
- Transfiera la suspensión de la celda a un tubo de 15 mL y centrífuga a 200 x g durante 3 min a temperatura ambiente.
- Deseche el sobrenadante y resuspend las células en 4 mL de medio fresco que contiene FBS y penicilina/estreptomicina. Cuente las celdas mediante un contador de celdas automático.
- Semilla 1 x 103 células por cm2 para cultivo posterior. Por ejemplo, semilla 9.5 x 103 celdas/pozo para placas de 6 pozos, o 1.9 x 103 celdas/pozo para placas de 24 pozos.
- Incubar las células durante la noche para permitirles adherirse y recuperarse, luego estimular las células MS-1 con TGF-β2 durante 3 días. Añadir el inhibidor de la quinasa del receptor de TGF-β SB431542 (5 μM) 30 min antes de la estimulación de TGF-β2. Tratar otras células con vehículo (DMSO).
NOTA: Disolver TGF-β2 en 4 mM de HCl que contenga al 0,1% de albúmina sérica bovina humana (BSA). Añadir la misma cantidad de tampón de ligando sin TGF-β2 al grupo control. La concentración de TGF-β2 puede adaptarse para ser de 0,1-1 ng/mL para ensayos específicos. Véanse las indicaciones en las cifras correspondientes. - Después de 3 días, examine la morfología celular con imágenes de campo brillante (con un microscopio invertido) y realice tinciones de inmunofluorescencia (consulte el paso 2) para evaluar los cambios en los marcadores relacionados con endMT. Realizar al menos tres experimentos independientes para obtener triplicados biológicos.
2. Tinción de inmunofluorescencia
- Tripsinizar las células MS-1 cultivadas (paso 1.2) y luego resebrir 1,9 x 103 células en un 0,1% w / v gelatina recubierto de 12 mm de vidrio de cubierta redonda colocado en la parte inferior de una placa de 24 pocillos.
- Después de cultivar las células durante la noche, añadir TGF-β2 (concentración final 1 ng/mL) a las células durante 3 días. Utilice el tampón de ligandos que contiene un medio como control negativo.
- Realizar pecam-1 y Sm22α tinción.
- Después de estimular las células con TGF-β2 (o Control) durante 3 días, retire el medio y lave las células con 1x PBS.
- Añadir 300 μL de formaldehído al 4% a cada pozo e incubar durante 10 min a temperatura ambiente para fijar las células. Lavar 3x con 1x PBS después de la incubación.
- Añadir 300 μL de Tritón X-100 al 0,1% en 1x PBS a cada pozo e incubar durante 10 min a temperatura ambiente para permeabilizar las células. Después de la incubación, retire la solución Triton X-100 y lave las células 3x con 1x PBS.
- Bloquee las células con albúmina sérica bovina (BSA) al 3% en 1x PBS durante 45 min a temperatura ambiente.
- Diluir los anticuerpos primarios Pecam-1 y Sm22α que reconocen las proteínas murinas 1:500 con 1x PBS. Luego incube las células fijas con anticuerpos primarios durante 45 min a temperatura ambiente.
- Después de lavar 3x con 1x PBS, incube las células con anticuerpos secundarios diluido 1000x, incluyendo el burro anti-rata Alexa 488 y el anti-conejo de cabra Alexa 594, durante 45 min a temperatura ambiente.
NOTA: Proteja las muestras de la luz durante la tinción. - Después de enjuagar 3x con 1x PBS, coloque el vidrio de la cubierta sembrado con células boca abajo en una gota de medio de montaje que contenga 4',6-diamidino-2-fenilindol (DAPI) en una diapositiva para manchar los núcleos.
- Fije la periferia del vidrio de la cubierta con esmalte de uñas transparente y guárdelo a 4 °C.
- Adquirir imágenes representativas con un microscopio confocal. Establezca las longitudes de onda del láser en 405 nm, 488 nm y 552 nm para detectar DAPI, Pecam-1 y Sm22α, respectivamente. Para cada canal, todas las imágenes se tomaron con los mismos ajustes y tiempo de exposición. Realizar al menos tres experimentos independientes para obtener triplicados biológicos.
3. Knock out of Snail usando edición CRISPR/Cas9
- Diseña dos sgRNAs independientes dirigidos al Caracolmurino.
- Diseñe sgRNAs utilizando las herramientas en línea CHOPCHOP (https://chopchop.cbu.uib.no/) y Cas-OFFinder (http://www.rgenome.net/cas-offinder/) de acuerdo con el nombre del gen y la especie objetivo.
- Predecir la actividad fuera del objetivo de los sgRNAs diseñados dirigidos a Snail con dos algoritmos independientes, incluidos Cas-OFFinder (http://www.rgenome.net/cas-offinder/) y CHOPCHOP (http://chopchop.cbu.uib.no/).
- Elija dos sgRNAs con la actividad de apagado más baja. Diseñe dos oligos de sgRNA complementarios con el sitio de corte BveI. El oligo de sentido comienza con 5'-ACCG-3' y el oligo antisentido comienza con 5'-AAAC-3'.
- Ordenar que los oligos sean sintetizados comercialmente para su uso posterior.
- Clone los oligos de ARN guía complementarios en el BveI-digerido AA19 pLKO.1-puro.U6.sgRNA. Bve I-stuffer Plásmido vectorial lentiviral para generarAA19 pLKO.1-Caracol-sgRNA16.
- Cortar el AA19 pLKO.1-puro.U6.sgRNA. Bve Plásmido I-relleno con la enzima BveI16. Mezclar 2 μg de AA19 pLKO.1-puro.U6.sgRNA. Bve I-relleno plásmido, 5 μL de 10x Buffer O, y 5 μL de la enzima BveI y añadir agua estéril para alcanzar un volumen total de 50 μL.
- Vórtice y girar brevemente hacia abajo la mezcla de reacción. Incubar la reacción a 37 °C durante 1 h.
- Cargue la mezcla de reacción en un gel de agarosa al 1% y ejecute en 1x Tris-acetato-EDTA (TAE, 50x TAE stock: 242 g de base de Tris disuelta en agua, 57,1 mL de ácido acético glacial, 100 mL de solución de EDTA de 500 mM (pH 8.0), y agregue agua a un tampón total de 1 L) hasta que se logre una buena separación.
- Corte el fragmento de la columna vertebral del gel, aíslelo con un kit de extracción de gel de acuerdo con el protocolo del fabricante (consulte la Tabla de materiales)y eleúe la columna vertebral en un tampón de elución (EB) de 40 μL.
- Mezclar 5 μL de 100 pmol/μL de sentido oligo y 5 μL de 100 pmol/μL de oligo antisentido con 1 μL de 1 M tris-HCl (pH 8.0) y añadir agua estéril para alcanzar un total de 100 μL para recocer los oligos de gRNA. Incubar la mezcla durante 5 min a 100 °C, y luego cubrir los tubos con papel de aluminio. Después de eso, enfríe lentamente la solución a temperatura ambiente para su uso posterior como un inserto.
- Para ligar los oligos de gRNA complementarios y la columna vertebral digerida por BveI, mezcle 1 μL de Bveaislado que corté AA19 pLKO.1-puro.U6.sgRNA.BveI-stuffer backbone y 2 μL de inserto (diluido 1:300) con 2 μL de tampón de ADN ligasa T4 y 1 μL de T4 ADN ligasa, y agregue agua estéril para alcanzar un total de 20 μL. Gire brevemente el tubo e incubarlo durante 4 h a temperatura ambiente para su uso posterior.
NOTA: Al realizar la ligadura, asegúrese de que se incluyan dos grupos de control. Para un grupo control, mezcle 1 μL de columna vertebral aislada, 2 μL de tampón de adn ligasa T4 y 1 μL de ADN ligasa T4 y agregue agua estéril para alcanzar un total de 20 μL, pero sin ADN oligo. Para el otro grupo control, mezclar 1 μL de columna vertebral aislada y 2 μL de tampón de ADN ligasa 10x T4, y añadir agua estéril para alcanzar un total de 20 μL, pero sin los oligos recocidos y la T4 ligasa. Estas dos ligaduras de control se utilizan para determinar el fondo de la reacción en el siguiente paso de transformación e indicar qué tan eficiente es la ligadura.
- Transformar la mezcla de reacción en TOP10 competente E. coli.
- Recoger el COMPETENTE TOP10 E. coli. células del congelador de -80 °C, y descongelarlas en el hielo.
- Añadir 2 μL de mezcla de ligadura a 50 μL de células competentes y mantener el tubo sobre hielo durante 30 min.
- Choque térmico del tubo a 42 °C durante 30 s. Poner el tubo en hielo durante 2 min.
- Añadir 950 μL de caldo de lisogenia fresca (LB) a la mezcla y agitar vigorosamente a 37 °C durante 60 min.
- Gire hacia abajo y platee las células en una placa LB de resistencia a la ampicilina caliente (100 μg/mL). Incubar la placa a 37 °C durante la noche.
- Verifique la inserción exitosa de los oligos de gRNA en el plásmido.
- Recoger 3-5 colonias en la placa en 1 mL de ampicilina (100 μg/mL) que contengan medio LB y agitar durante la noche a 30 °C.
- Aísle el ADN del plásmido con un kit de plásmidos de acuerdo con el protocolo del fabricante(Tabla de Materiales)y secuenciarlo con el cebador promotor U6 5'- GAGGGCCTATTTCCCATGATT-3' para verificar la inserción exitosa del oligo arn gRNA.
4. Generar caracol knockout MS-1 células
- Producir partículas lentivirales portadoras de Cas9 o gRNAs dirigidos a caracoles.
- Cultivo de células HEK 293T en DMEM que contienen 10% de suero bovino fetal y 100 U/mL de penicilina/estreptomicina en platos de 14,5 cm (o matraces T75) en una incubadora (5% de CO2,37 °C).
- Mezclar 9,9 μg de plásmido del gen objetivo, AA19 pLKO.1-Snail-sgRNA o pLV-Cas917 plásmido, junto con los plásmidos ayudantes 3,5 μg de pCMV-VSVG (codificando la proteína G del virus de la estomatitis vesicular, VSV-G), 6,6 μg de plásmido de elemento rev-responsive pMDLg-RRE (codificación Gag y Pol), y 5,0 μg de pRSV-REV (codificación Rev) en 500 μL de medio libre de suero. Resuspend 50 μL de polietilenmina (PEI) (2,5 mg/mL) en 500 μL de medio sérico. Mezcle suavemente plásmidos y preparaciones de PEI pipeteando hacia arriba y hacia abajo. Incubar la mezcla durante 20 min a temperatura ambiente.
- Transfecte las células HEK 293T añadiendo el medio de mezcla de la etapa 4.1.2 a las células confluentes al 80% en platos de 14,5 cm (o matraces T75) que contienen medio DMEM con FBS al 10% y penicilina/estreptomicina al 100 U/mL. Las células HEK293T se utilizan porque se transfectan fácilmente y generan altos niveles de virus18.
- Transferir células HEK 293T transfectadas a un laboratorio de bioseguridad en microbiología y biomédica (BMBL) para cultivarlas durante 24 h.
- En un laboratorio de BMBL, substituya el medio de transfección de las células HEK 293T por 12 mL de DMEM completo fresco que contenga FBS y penicilina/estreptomicina. Incubar las células durante 24 h.
- Recoja y filtre el medio con una jeringa de 20 ml y un filtro de 0,45 μm. Transfiera el medio acondicionado a un tubo de polipropileno de 15 mL.
- Añadir 12 mL de DMEM fresco completo que contenga FBS y penicilina/estreptomicina al plato de cultivo HEK 293T y al cultivo durante 24 h adicionales.
- Recoja y filtre el medio con una jeringa de 20 ml y un filtro de 0,45 μm. Transfiera el medio acondicionado a un tubo de polipropileno de 15 mL. Almacene el medio que contiene partículas lentivirales como alícuotas de 1 mL a -80 °C para su uso posterior.
- Infecte las células MS-1 con el virus pLV-Cas9.
- Semilla 1 x 105 células MS-1 por pozo en una placa de 6 pozos durante 24 h antes de la infección por lentivirus.
- Descongelar las alícuotas congeladas del virus pLV-Cas9 en un baño de agua de 37 °C.
- Mezclar 1 mL de medio virus con 1 mL de medio DMEM fresco que contenga FBS y penicilina/estreptomicina. Añadir policreno al medio (concentración final como 10 μg/mL) para aumentar la eficiencia de la infección.
- Retire el medio de la placa de 6 pocillos y reemplácelo con el medio de mezcla virus/polibreno y cultíe las células en una incubadora durante 24 h. 24 h después de la infección, reemplace el medio con un medio fresco y cultúe las células durante otras 24 h.
Nota: Mantenga siempre un pozo no infectado como un grupo de control. - Aspirar el medio del grupo infectado y del grupo control y sustituirlo por un medio DMEM con blasticidina de 4 μg/mL.
- Devolver la placa a una incubadora de 37 °C y cultivar las células durante 1 semana. Las células no infectadas morirán debido al efecto de la blasticidina. Divida las células supervivientes cuando alcancen una confluencia celular del 80% y continúe con la selección de blasticidina.
- Confirmar la expresión de Cas9 en células MS-1 por western blotting utilizando un anticuerpo contra Cas9 (el peso molecular de Cas9 es de aproximadamente 160 kDa).
- Infecte por separado las células pLV-Cas9 MS-1 con dos lentivirus independientes del gRNA.
- Semilla 1 x 105 células pLV-Cas9 MS-1 por pozo en una placa de 6 pozos durante 24 h antes de la infección.
- Siga el mismo protocolo que se describe en 4.2 para infectar por separado las células con dos lentivirus gRNA.
- Después de 24 h de infección con el virus gRNA, refresque el medio y cultúe las células durante otras 24 h.
- Substituya el medio por DMEM por 1 μg/mL de puromicina. Devolver la placa a una incubadora de 37 °C y cultivar las células durante 1 semana. Asegúrese de que las células no infectadas estén completamente muertas. Dividir las células cuando alcancen el 80% de confluencia y continuar la selección de puromicina.
- Confirme la eliminación de Snail en las células MS-1 por western blotting usando un anticuerpo contra Snail (el peso molecular de Snail es de aproximadamente 35 kDa).
Resultados
TGF-β2 induce EndMT y estimula la expresión de caracol en las células endoteliales MS-1
TGF-β es uno de los cytokines con el potencial más grande de inducir EndMT. Después de tratar las células MS-1 con TGF-β2 (1 ng/mL) durante 3 días, las células endoteliales MS-1 pierden su estructura adoquinada y se diferencian en células mesenquimales en forma de huso (Figura 1A)15. Para verificar aún más el papel de TGF-β2 en la inducción de cambios fenotípicos celulares, pretratamos las células con la pequeña molécula de activina receptor-como quinasa (ALK)4/ALK5/ALK7 inhibidor SB431542 antes de la estimulación de TGF-β219. SB431542 completamente derogado TGF-β2 inducida por cambios en la morfología celular (Figura 1A). El proceso de EndMT inducido por TGF-β2 se investigó más a fondo mediante el estudio de los cambios en la expresión de los marcadores relacionados con EndMT. Como se muestra en la Figura 1B,la proteína endotelial Pecam-1 se redujo potentemente después de la estimulación de TGF-β2, mientras que el factor mesenquimal Sm22α fue profundamente upregulated por TGF-β215. Estos datos son consistentes con la noción de que TGF-β2 desencadenó EndMT en células MS-1. A continuación, se investigaron los efectos de TGF-β2 en la expresión de caracol y. Como se muestra en la Figura 1C,el caracol fue marcadamente regulado por el TGF-β2, mientras que la expresión de slug no fue influenciada por el TGF-β2 en las células MS-115. La cuantificación de la expresión de Snail a partir de tres experimentos independientes se muestra en la Figura 1D.
Agotamiento de Caracol por CRISPR/Cas9 en células endoteliales MS-1
Como snail fue inducido por TGF-β2 y probablemente involucrado en TGF-β2-mediada por EndMT, se realizó la edición de genes CRISPR / Cas9 para agotar genéticamente la expresión de caracol en las células MS-1. Se planteó la hipótesis de que el agotamiento de Caracol sería suficiente para inhibir TGF-β2-inducida por EndMT. Como se muestra en la Figura 2A,generamos células knockout snail en dos pasos. En primer lugar, Cas9 fue expresado ectópicamente infectando las células MS-1 con un Cas9 que expresaba el lentivirus. Dado que existe un cassette de resistencia a blasticidina en la construcción de pLV-Cas9, se comprobó la expresión de Cas9 mediante el análisis de Western blot en células resistentes a blasticidina(Figura 2D). Posteriormente, introdujimos sgRNAs que se dirigieron específicamente a Snail para interrumpir su expresión de proteínas. Este procedimiento también se realizó por infección con partículas lentivirales portadoras de la construcción AA19 pLKO.1-Snail-sgRNA, que incluye un casete de expresión de puromicina. Cas9-expressing las células fueron infectadas otra vez con el gRNA que contenía lentivirus y seleccionadas más a fondo con puromycin. Se diseñaron dos oligos de sgRNA complementarios dirigidos al caracol murino con una actividad baja pronosticada fuera del objetivo (Figura 2B,C). Tras introducir dos sgRNAs de Caracol independientes en Cas9 que expresan células MS-1, la expresión de la proteína snail fue derogada(Figura 2D)15.
La Deficiencia De Caracol Inhibe TGF-β2 Inducida Por EndMT En Células MS-1
Para demostrar la función de Caracol en TGF-β2-mediada por EndMT, realizamos un ensayo endMT en células agotadas por caracol y lo comparamos con las células parentales MS-1. Como se muestra en la Figura 3A,el knockout de Snail fue suficiente para inhibir la morfología celular similar a fibroblastos impulsada por TGF-β2 en células MS-115. Además, la disminución mediada por TGF-β2 en Pecam-1 y la mejora de Sm22α se bloquearon por completo en las células MS-1 agotadas por caracol. En resumen, demostramos que snail es crítico para TGF-β2-mediada por EndMT en células MS-1(Figura 3B)15.
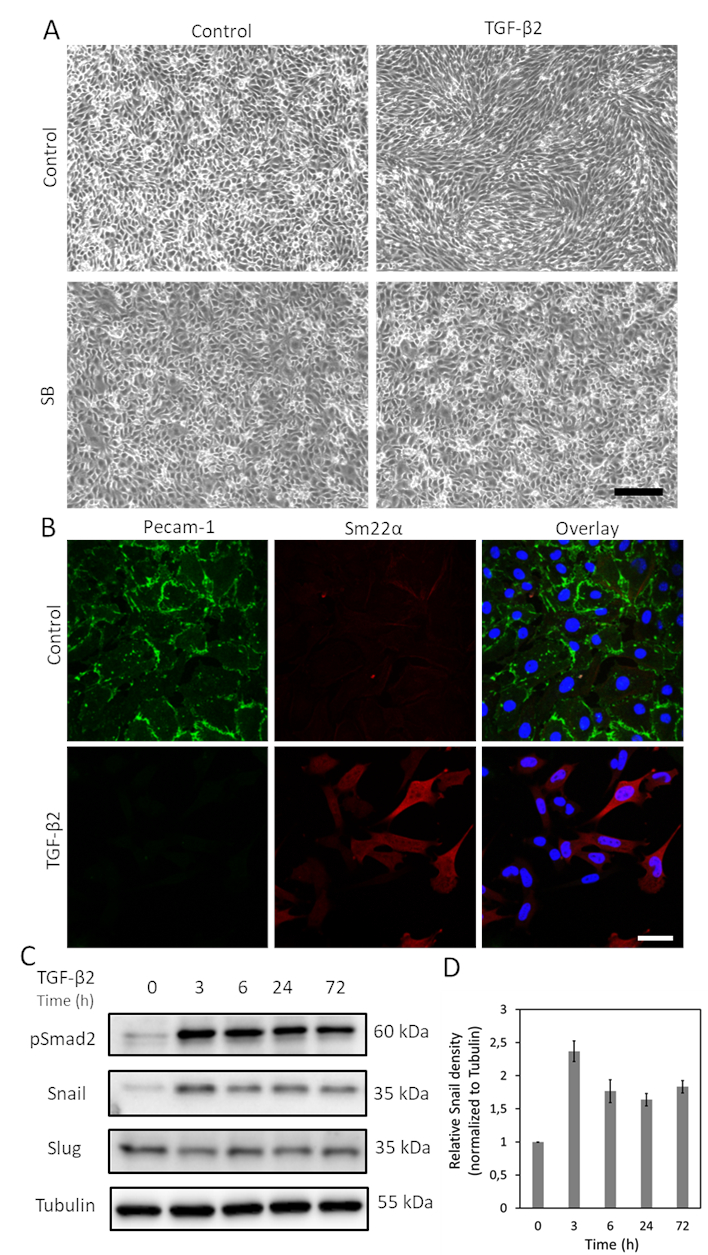
Figura 1. TGF-β2 induce endMT y la expresión de caracol en las células MS-1. A. Efectos del inhibidor de la quinasa del receptor de TGF-β2 y/o TGF-β tipo I SB-431542 sobre la morfología celular. Imágenes brillantes de células MS-1 tras el tratamiento con TGF-β2 (1 ng/mL) y/o SB-431542 (SB, 5 μM, administrado 30 min antes de TGF-β2) durante 2 días. La barra de escala representa 200 μm. B. Tinción de inmunofluorescencia de Pecam-1 (verde) y Sm22α (rojo) en células MS-1 cultivadas en medio que contiene TGF-β2 (1 ng/mL) durante 3 días. Los núcleos se visualizan en azul (DAPI). Barra de escala: 50 μm. C. Western blot con lisiado de células enteras de células TGF-β2 estimuladas ms-1. La expresión de Caracol, pero no de, fue potenciada por la estimulación de TGF-β2, como se informó anteriormente en Ma et al15. D. Cuantificación de la expresión de caracol mediante la integración de los resultados de tres experimentos independientes western blot. Haga clic aquí para ver una versión más amplia de esta figura.
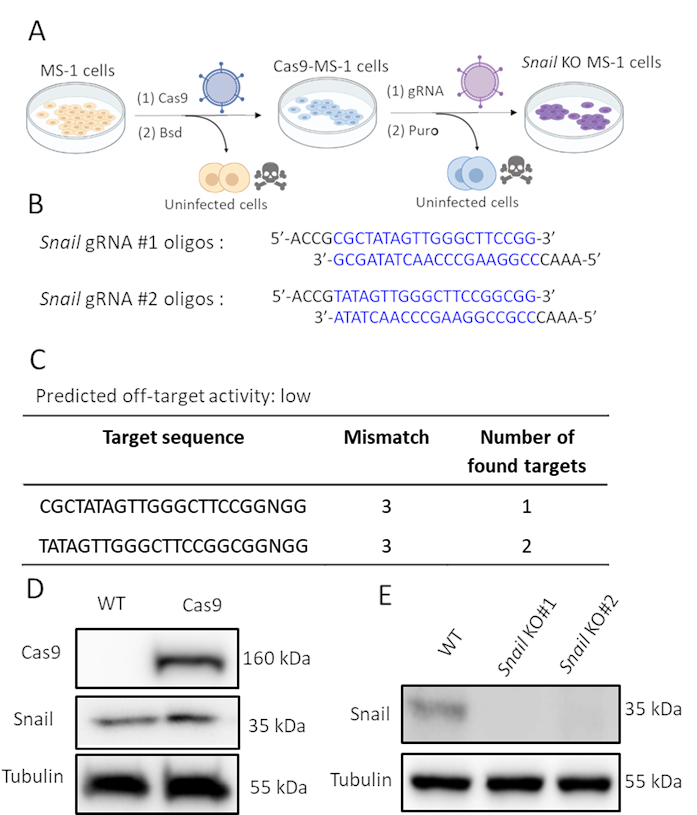
Figura 2. Agotamiento de Caracol mediante edición de genes CRISPR-Cas9. A. Esquema que representa cómo generar células knockout caracol. Bsd: Blasticidin. Puro: Puromicina. B. Oligonucleótidos de dos sgRNAs independientes dirigidos a Snail usando CHOPCHOP (http://chopchop.cbu.uib.no/) y Cas-OFFinder (http://www.rgenome.net/cas-offinder/). C. La actividad fuera del objetivo predicha de los dos gRNAs para Snail usando Cas-OFFinder (http://www.rgenome.net/cas-offinder/). D. Cas9 y la expresión de caracol en el tipo salvaje (WT) y Cas9-overexpressed MS-1 medido por el análisis de Western blot. E. Knockout de Snail con dos gRNAs independientes en células MS-1 según lo medido por el análisis de Western blot. Se reportaron resultados similares en Ma et al15. Haga clic aquí para ver una versión más amplia de esta figura.
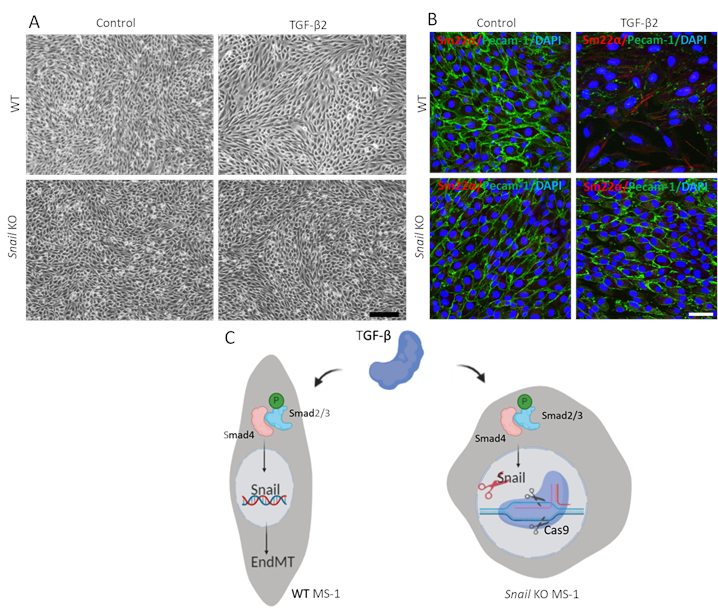
Figura 3. El agotamiento genético de Caracol inhibe TGF-β2-inducida endMT en células MS-1. A. Imágenes brillantes de células MS-1 tras el tratamiento con TGF-β2 (0,1 ng/mL) durante 3 días en células de tipo salvaje (WT, panel superior) y caracol noqueado (panel inferior). La barra de escala representa 200 μm. B. Tinción inmunofluorescente para Pecam-1 (verde), Sm22α (rojo) y núcleos (azul) de células MS-1 cultivadas en medio que contienen TGF-β2 (1 ng/mL) durante 3 días. Agotamiento de Caracol abrogado TGF-β2-inducida por la disminución de Pecam-1 y aumento de la expresión de Sm22α. Barra de escala representa 50 μm. C. Representación esquemática del efecto de la eliminación de caracol en TGF-β inducida endMT en células MS-1. TGF-β estimula la expresión de Caracol a través de la vía Smad por fosforilación Smad2/3 y más lejos impulsa EndMT. Noqueando a Snail usando edición de genes basada en CRISPR /Cas9 abrogado por TGF-β mediada por EndMT. Haga clic aquí para ver una versión más amplia de esta figura.
Discusión
Comprender el mecanismo de EndMT es fundamental para modular este proceso y dirigirse a las enfermedades relacionadas con EndMT. Aquí, se describen los métodos para realizar un ensayo de EndMT inducido por TGF-β e interrogar el papel del caracol EndMT-TF en TGF-β desencadenado EndMT, mediante la realización de CRISPR / Cas9 mediada por el agotamiento estable del gen de caracol de las células. El agotamiento de Snail utilizando el enfoque CRISPR/Cas9 abrogó con éxito el EndMT impulsado por TGF-β2 en células MS-1 (Figura 3C). Para estudiar los efectos de cualquier cytokines, como TGF-β, sobre EndMT, los ECs fueron expuestos a los cytokines y entonces el acontecimiento de EndMT fue evaluado según cambios morfológicos y cambios endoteliales y mesenquimales de la expresión del marcador en células. TGF-β2 fuertemente inducida EndMT en células MS-1 acompañado de un fuerte aumento en la expresión del factor de transcripción Caracol. El EndMT-TFs inducido por TGF-β puede diferir según la especie o el tipo de célula endotelial tejido-específica. Por ejemplo, observamos que Snail but not Slug fue significativamente upregulated por TGF-β en las células MS-1, mientras que en las células endoteliales de la vena umbilical humana (HUVECs), tanto Snail como Slug se incrementan después de la exposición a TGF-β20.
Evaluamos el grado del proceso de EndMT de dos maneras examinando cambios de la morfología de la célula y entonces investigando cambios en la expresión EndMT-relacionada de los marcadores. Después de la exposición de TGF-β por 3 días, las células experimentaron EndMT con variaciones morfológicas constantes y cambios en la expresión de marcadores EndMT-relacionados. Además de la tinción de inmunofluorescencia que realizamos aquí, las variaciones de los marcadores también pueden ser monitoreadas por western blotting a nivel de expresión de proteínas o por qRT-PCR (Real-Time Quantitative Reverse Transcription PCR) a nivel génico21. Además de estos dos métodos de ahorro de tiempo y costos que mostramos en este protocolo, hay otros métodos para examinar EndMT. Por ejemplo, la realización de análisis del transcriptoma (mediante secuenciación de ARN o qPCR) para comparar los niveles de expresión de genes relacionados con el endotelial y el mesenquimal entre las células tratadas y las células control puede evaluar con precisión EndMT22,23. Además, EndMT a menudo implica la pérdida estable de la función de barrera, que puede ser evaluada por espectroscopia de impedancia24. Además, se pueden examinar pruebas adicionales de la adquisición de propiedades similares a células madre por parte de células derivadas de EndMT. Por ejemplo, bajo condiciones específicas de cultivo, endMT mesenquimal-como las células se pueden diferenciar aún más en osteoblastos, condrocitos, adipocitos o (mio)fibroblastos. Por lo tanto, el análisis adicional para confirmar la diferenciación en diversos tipos de la célula que pertenecen al linaje del mesodermo (es decir, expresión génica y coloración de la matriz) es útil para demostrar la naturaleza multipotente de las células EndMT-derivadas. Por último, los métodos de evaluación de EndMT no se limitan a los estudios in vitro, sino que pueden extrapolarse para investigar la relación entre EndMT y algunas enfermedades in vivo o in vivo órganos. En este sentido, el uso de estrategias de rastreo de linajes específicos endoteliales se extiende ampliamente a la investigación relacionada con EndMT25.
Para investigar el papel de Snail durante EndMT, en este estudio se utilizó la edición de genes CRISPR/Cas9 para eliminar este gen. Los datos mostraron que TGF-β2 no pudo mediar endMT en células de caracol deficientes en MS-1. Esta observación demostró que el caracol es esencial para TGF-β2 inducido EndMT en células MS-1. Utilizamos un casete de expresión de sgRNA impulsado por U6 independiente para introducir sgRNAs específicos para Cas9 para apuntar a Snail. Además de este método, Ran et al.26 describieron otra estrategia para clonar la secuencia sgRNA oligos en el andamio Cas9 para generar una construcción que contenga tanto Cas9 como gRNAs. Los nuevos enfoques emergentes permiten que CRISPR/Cas incorpore funciones adicionales. Por ejemplo, se pueden lograr eliminatorias dobles o triples mediante la entrega de más sgRNAs en las células que expresan Cas927. La proteína Cas13 de ingeniería se dirige y digiere las moléculas de ARN sin interrumpir el ADN endógeno28. Además de eliminar genes con CRISPR/Cas, los ARNs de horquilla corta (shRNAs) pueden utilizarse como alternativas para derribar de forma estable la expresión génica dirigida29. Para todos los métodos de edición de genes CRISPR/Cas, siempre se debe tener en cuenta la escisión fuera del objetivo. Además, los pequeños ARN interferentes (siRNAs) silencian transitoriamente la expresión génica y la concentración de siRNA se diluye con la división celular30. Ambos métodos suprimen parcialmente la expresión génica dirigida. Por el contrario, la expresión génica ectópica también se utiliza para verificar la función génica durante EndMT/EMT31. Este enfoque puede determinar si la regulación al alza de un gen es suficiente para provocar una respuesta EndMT. Por lo tanto, actualmente hay una multitud de estrategias técnicas que se pueden utilizar para identificar y verificar posibles reguladores de EndMT. Además, el análisis transcriptómico puede ser una buena opción en la identificación y el análisis exhaustivo de los reguladores relacionados con EndMT. Recomendamos utilizar diversos acercamientos complementarios para investigar la modulación del EndMT.
En resumen, introdujimos un flujo de trabajo para identificar los factores que pueden desempeñar funciones funcionales durante el EndMT inducido por TGF-β. Este método se puede también utilizar para estudiar si otros estímulos (es decir, cytokines, factores de crecimiento, estímulos mecánicos, interacciones célula-célula) pueden modular EndMT, y la interacción de TGF-β con otros estímulos. Además, destacamos un acercamiento usando la edición del gene de CRIPSR/Cas para aclarar si cierto gen está requerido para EndMT TGF-β-inducido. Para ilustrar esta metodología, se utilizó el fuerte inductor endMT TGF-β2 en células MS-1, pero los protocolos se pueden adaptar a otras citoquinas y otros tipos de células. Esperamos que este protocolo detallado descrito sirva como trampolín para futuros estudios EndMT-relacionados.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
La investigación fue apoyada por CGC.NL y la Iniciativa de Investigación Cardiovascular de los Países Bajos: la Fundación Holandesa del Corazón, la Federación Holandesa de Centros Médicos Universitarios, la Organización Holandesa para la Investigación y el Desarrollo de la Salud y la Real Beca de la Academia de Ciencias de los Países Bajos otorgada al Phaedra-Impact (http://www.phaedraresearch.nl). JM cuenta con el apoyo del Consejo chino de becas. GSD cuenta con el apoyo de una subvención trampolín de AFM-Telethon [22379], FOP Italia y una subvención de La Fundació La Marató de TV3 (#202038).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | Pall Corporation, USA | 4614 | |
| 10× T4 DNA ligase buffer | Thermofisher Scientific, USA | B69 | |
| 12 mm round glass slice | Knittel Glass,Germany | VD10012Y1A.01 | |
| 20 mL syringe | BD Eclipse, USA | 300629 | |
| 4′,6-diamidino-2-phenylindole (DAPI) | Vector Laboratories, USA | H-1200 | VECTASHIELD Antifade Mounting Media |
| AA19_PLKO vector | Dr. M Gonçalves, Leiden University Medical Center, Netherlands | Gift | |
| Ampicillin | Serva Electrophoresis, USA | 1339903 | |
| Anti-Mouse IgG | GE Healthcare, USA | NA931 | |
| Anti-Rabbit IgG | Cell signaling, USA | 7074 | |
| Agarose | Roche, Switzerland | 11388991001 | |
| Blasticidin | Invitrogen, USA | R21001 | |
| Buffer O (10X) | Thermofisher Scientific, USA | BO5 | |
| BveI (Bspm1) | Thermofisher Scientific, USA | ER 1741 | |
| Confocal microscope | Leica Microsystems, Germany | SP8 | |
| DMEM | Thermo Fisher Scientific, USA | 11965092 | |
| Donkey anti-rat Alexa 488 | Invitrogen, USA | A21208 | |
| FBS | Thermo Fisher Scientific, USA | 16000044 | |
| Formaldehyde | Thermo Fisher Scientific, USA | 28908 | |
| Inverted microscope | Leica Microsystems, Germany | DMi8 | |
| Goat anti-rabbit Alexa 594 | Invitrogen,USA | A11012 | |
| LabNed Plasmid kit | LabNed, USA | LN2400004 | |
| MS-1 cell line | American Type Culture Collection (ATCC) | ||
| Nail polish | HEMA, Netherlands | Transparent | |
| Pecam-1 antibody | Becton Dickinson,USA | 553370 | |
| PEI | Polysciences, USA | 23966-1 | |
| PLV-Cas9 plasmid | Sigma-Aldrich, USA | Cas9BST-1EA | |
| Puromycin | Sigma-Aldrich, USA | P9620 | |
| QIAquick gel extraction kit | Qiagen, Germany | 28706 | |
| Sm22a antibody | Abcam, UK | ab14106 | |
| Snail | Cell signaling, USA | 3879 | |
| T4 DNA ligase | Thermofisher Scientific, USA | EL 0014 | |
| TC20 automated Cell Counter | Bio-Rad, USA | 1450102 | |
| Human TGF-β2 | Joachim Nickel, University of Wurzburg | Gift | Other commercial recommendation: 302-B2, R&D systems |
| Triton X-100 | Merck, USA | 1086031000 | |
| SB431542 | Tocris Bioscience, UK | 1614 | |
| Tris | Roche, Switzerland | 11814273001 |
Referencias
- Ma, J., Sanchez-Duffhues, G., Goumans, M. -J., Ten Dijke, P. TGF-β-induced endothelial to mesenchymal transition in disease and tissue engineering. Frontiers in Cell and Developmental Biology. 8, (2020).
- Piera-Velazquez, S., Jimenez, S. A. Endothelial to mesenchymal transition: role in physiology and in the pathogenesis of human diseases. Physiological Reviews. 99 (2), 1281-1324 (2019).
- Sánchez-Duffhues, G., de Vinuesa, G. A., ten Dijke, P. Endothelial-to-mesenchymal transition in cardiovascular diseases: developmental signaling pathways gone awry. Developmental Dynamics. 247 (3), 492-508 (2018).
- Evrard, S. M., et al. Endothelial to mesenchymal transition is common in atherosclerotic lesions and is associated with plaque instability. Nature Communications. 7 (1), 1-16 (2016).
- Qiao, L., et al. Endothelial fate mapping in mice with pulmonary hypertension. Circulation. 129 (6), 692-703 (2014).
- Zeisberg, E. M., et al. Endothelial-to-mesenchymal transition contributes to cardiac fibrosis. Nature Medicine. 13 (8), 952-961 (2007).
- Clere, N., Renault, S., Corre, I. Endothelial-to-mesenchymal transition in cancer. Frontiers in Cell and Developmental Biology. 8, (2020).
- Saito, A. EMT and EndMT: regulated in similar ways. The Journal of Biochemistry. 153 (6), 493-495 (2013).
- Bolós, V., et al. The transcription factor Slug represses E-cadherin expression and induces epithelial to mesenchymal transitions: a comparison with Snail and E47 repressors. Journal of Cell Science. 116 (3), 499-511 (2003).
- Aird, W. C. Endothelial cell heterogeneity. Cold Spring Harbor Perspectives in Medicine. 2 (1), 006429(2012).
- Yang, J., et al. Guidelines and definitions for research on epithelial-mesenchymal transition. Nature Reviews Molecular Cell Biology. , 1-12 (2020).
- Doudna, J. A., Charpentier, E. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), (2014).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Arbiser, J. L., et al. Oncogenic H-ras stimulates tumor angiogenesis by two distinct pathways. Proceedings of the National Academy of Sciences USA. 94 (3), 861-866 (1997).
- MA, J., et al. TGF-?-induced endothelial to mesenchymal transition results from a balance between SNAIL and ID factors. Frontiers in Cell and Developmental Biology. 182 (9), (2021).
- Chen, X., et al. Probing the impact of chromatin conformation on genome editing tools. Nucleic Acids Research. 44 (13), 6482-6492 (2016).
- Hunter, F. W., et al. Functional CRISPR and shRNA screens identify involvement of mitochondrial electron transport in the activation of evofosfamide. Molecular Pharmacology. 95 (6), 638-651 (2019).
- Mao, Y., et al. Lentiviral vectors mediate long-term and high efficiency transgene expression in HEK 293T cells. International Journal of Medical Sciences. 12 (5), 407(2015).
- Liu, S., et al. Deubiquitinase activity profiling identifies UCHL1 as a candidate oncoprotein that promotes TGFβ-induced breast cancer metastasis. Clinical Cancer Research. 26 (6), 1460-1473 (2020).
- Medici, D., et al. Conversion of vascular endothelial cells into multipotent stem-like cells. Nature Medicine. 16 (12), 1400(2010).
- Kokudo, T., et al. Snail is required for TGFβ-induced endothelial-mesenchymal transition of embryonic stem cell-derived endothelial cells. Journal of Cell Science. 121 (20), 3317-3324 (2008).
- Dong, J., et al. Single-cell RNA-seq analysis unveils a prevalent epithelial/mesenchymal hybrid state during mouse organogenesis. Genome Biology. 19 (1), 1-20 (2018).
- Oatley, M., et al. Single-cell transcriptomics identifies CD44 as a marker and regulator of endothelial to haematopoietic transition. Nature communications. 11 (1), 1-18 (2020).
- Halaidych, V., et al. Inflammatory responses and barrier function of endothelial cells derived from human induced pluripotent stem cells. Stem Cell Reports. 10 (5), 1642-1656 (2018).
- Li, Y., Lui, K. O., Zhou, B. Reassessing endothelial-to-mesenchymal transition in cardiovascular diseases. Nature Reviews Cardiology. 15 (8), 445-456 (2018).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Grav, L. M., et al. One-step generation of triple knockout CHO cell lines using CRISPR/Cas9 and fluorescent enrichment. Biotechnology Journal. 10 (9), 1446-1456 (2015).
- Abudayyeh, O. O., et al. RNA targeting with CRISPR-Cas13. Nature. 550 (7675), 280-284 (2017).
- Paddison, P. J., et al. Cloning of short hairpin RNAs for gene knockdown in mammalian cells. Nature Methods. 1 (2), 163-167 (2004).
- Reynolds, A., et al. Rational siRNA design for RNA interference. Nature Biotechnology. 22 (3), 326-330 (2004).
- Lourenço, A. R., et al. C/EBPɑ is crucial determinant of epithelial maintenance by preventing epithelial-to-mesenchymal transition. Nature Communications. 11 (1), 1-18 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados