Method Article
TGF-β-vermittelter endothelialer zu mesenchymaler Übergang (EndMT) und die funktionelle Bewertung von EndMT-Effektoren mittels CRISPR/Cas9-Gen-Editing
In diesem Artikel
Zusammenfassung
Wir beschreiben Methoden zur Untersuchung von TGF-β2-induzierter EndMT in Endothelzellen, indem wir Veränderungen der Zellmorphologie beobachten und die Expression EndMT-bedingte Markerveränderungen mittels Immunfluoreszenzfärbung untersuchen. CrispR/Cas9-Gen-Editing wurde beschrieben und verwendet, um das Gen zu erschöpfen, das für Snail kodiert, um seine Rolle bei TGF-β2-induzierter EndMT zu untersuchen.
Zusammenfassung
Als Reaktion auf spezifische externe Hinweise und die Aktivierung bestimmter Transkriptionsfaktoren können sich Endothelzellen zu einem mesenchymalen Phänotyp differenzieren, ein Prozess, der als endothelialer zu mesenchymaler Übergang (EndMT) bezeichnet wird. Neue Ergebnisse deuten darauf hin, dass EndMT ursächlich mit mehreren menschlichen Krankheiten wie Fibrose und Krebs verbunden ist. Darüber hinaus können endothelial abgeleitete mesenchymale Zellen bei Geweberegenerationsverfahren eingesetzt werden, da sie weiter in verschiedene Zelltypen (z. B. Osteoblasten und Chondrozyten) differenziert werden können. Somit kann die selektive Manipulation von EndMT klinisches Potenzial haben. Wie der epithelial-mesenchymale Übergang (EMT) kann EndMT stark durch den sezernierten Zytokin-transformierenden Wachstumsfaktor-beta (TGF-β) induziert werden, der die Expression sogenannter EndMT-Transkriptionsfaktoren (EndMT-TFs), einschließlich Schnecke und Schnecke, stimuliert. Diese EndMT-TFs regulieren dann die Spiegel von mesenchymalen bzw. endothelialen Proteinen nach oben und unten. Hier beschreiben wir Methoden zur Untersuchung von TGF-β-induzierter EndMT in vitro, einschließlich eines Protokolls zur Untersuchung der Rolle bestimmter TFs bei TGF-β-induzierter EndMT. Mit diesen Techniken liefern wir den Nachweis, dass TGF-β2 EndMT in mikrovaskulären Endothelzellen der mausen Pankrea (MS-1-Zellen) stimuliert und dass die genetische Erschöpfung von Schnecken mit geclusterten regelmäßig interspaced short palindromic repeats (CRISPR)/CRISPR-assoziiertem Protein 9 (Cas9)-vermittelter Genbearbeitung dieses Phänomen aufhebt. Dieser Ansatz kann als Modell dienen, um potenzielle Modulatoren der Endothelbiologie zu befragen, und kann verwendet werden, um genetische oder pharmakologische Screens durchzuführen, um neue Regulatoren von EndMT mit potenzieller Anwendung bei menschlichen Krankheiten zu identifizieren.
Einleitung
Endothelialer zu mesenchymaler Übergang (EndMT) ist ein mehrstufiges und dynamisches biologisches Phänomen, das mit verschiedenen physiologischen und pathologischen Prozessen in Verbindung gebracht wurde1,2. Bei EndMT verlieren Endothelzellen allmählich ihre endothelialen Merkmale, während sie mesenchymale Eigenschaftenerwerben 3; so differenzieren sich dicht verdichtete und gut organisierte Endothelzellen zu länglichen mesenchymalen Zellen. Morphologische Veränderungen in EndMT fallen mit Veränderungen in der Expression bestimmter Gene und Proteine zusammen. Im Allgemeinen nimmt die Expression von Proteinen ab, die endotheliale Eigenschaften beibehalten, einschließlich vaskulärem Endothel (VE)-Cadherin, Thrombozyten/EC-Adhäsionsmolekül-1 (CD31/Pecam-1). Gleichzeitig sammeln sich Proteine an, die mit mesenchymalen Funktionen verwandt sind, wie α-glatte Muskelaktiin (α-Sma) und glattes Muskelprotein 22α (Sm22α). Neue Ergebnisse haben gezeigt, dass postnatale EndMT zur Entwicklung menschlicher Krankheiten wie Krebs, Herzfibrose, pulmonaler arterieller Hypertonie (PAH), Atherosklerose (AS), Organfibrose usw. beiträgt2,4,5,6,7. Ein tieferes Verständnis der zugrunde liegenden Mechanismen der EndMT und wie der EndMT-Prozess zu lenken ist, wird neuartige therapeutische Methoden für EndMT-bedingte Erkrankungen und regenerative Medizin liefern.
TGF-β ist einer der wichtigsten EndMT-Induktoren, und andere bekannte beteiligte Faktoren sind Wnt / β-Catenin, Notch und einige entzündliche Zytokine1. Da der zelluläre Kontext der Schlüssel für Reaktionen ist, die durch TGF-β ausgelöst werden, ist das Zusammenspiel von TGF-β mit anderen EndMT-fördernden Signalen für TGF-β relevant, um eine EndMT-Antwort hervorzurufen. Bei Aktivierung von TGF-β Zelloberflächenrezeptoren Typ I und Typ II Serin/Threoninkinase wird der intrazelluläre kanonische Smad-Signalweg aktiviert. TGF-β rezeptorvermittelte phosphorylierte Smad2/3 bilden heteromere Komplexe mit Smad4, die in den Zellkern translozieren, wo sie die Expression von EndMT-bezogenen Transkriptionsfaktoren hochregulieren. Ähnlich wie beim epithelial-mesenchymalen Übergang (EMT) werden Transkriptionsfaktoren wie Snail, Slug, Twist, Zeb1 und Zeb2 durch TGF-β Signalisierung induziert und tragen zur Genreprogrammierung in EndMT8bei.
Schnecke wurde häufig als Schlüsselfaktor in EndMT identifiziert. Schnecke bindet an den Promotor von Genen, die für Zell-Zell-Adhäsionsproteine kodieren, und unterdrückt deren Transkription, was durch die Verstärkung der Expression von mesenchymalen Proteinen ausgeglichen wird9. Endothelzellen umfassen eine sehr heterogene Population und der relative Einfluss verschiedener extrazellulärer Reize auf EndMT kann sich zwischen endothelialen zellulären Kontexten oder Zelltypen unterscheiden10. Aufgrund seiner Ähnlichkeiten mit EMT sind einige Methoden nützlich, um die beiden Mechanismen EMT und EndMT8zu untersuchen. In diesem Zusammenhang betont die EMT International Association (TEMTIA) nachdrücklich die Notwendigkeit komplementärer Techniken, um letztendlich das Auftreten von EMT / EndMT11nachzuweisen.
Hier beschreiben wir eine Methode zur Überwachung und Visualisierung des TGF-β-induzierten EndMT-Prozesses. Die Immunfluoreszenzfärbung liefert die grundlegenden Informationen über Expressionsänderungen in gezielten Proteinen/Markern, die als Indikatoren dafür verwendet werden, ob der EndMT-Prozess stattfindet. Zusätzlich kann die Immunfluoreszenzfärbung die Lokalisation von Proteinen/Markern und die Zellmorphologie visualisieren. Um die potenzielle Aktivität spezifischer TFs (oder anderer vor- oder nachgelagerter Regulatoren) zu untersuchen, die an TGF-β vermittelten EndMT beteiligt sind, beschreiben wir ein Protokoll, das geclusterte regelmäßig interspaced short palindromic repeats (CRISPR) / CRISPR-assoziiertes Protein 9 (Cas9) Gen-Editing verwendet, um bestimmte Gene aus Zellen zu erschöpfen, am Beispiel der TF-Schnecke. Cas9 ist eine duale RNA-gesteuerte DNA-Endonuklease, die komplementäre Sequenzen zu CRISPR-Sequenzen in Bakterien erkennt und spaltet12. Das CRISPR/Cas9-System wird derzeit intensiv genutzt, da es die Gentechnik in vitro und in vivo13erleichtert. Geleitet von einer Single-Guide-RNA (sgRNA) erzeugt ektopisch exprimiertes Cas9 einen Doppelstrangbruch an einer vorausgewählten Zielsequenz in einem bestimmten Genort. Non-homologous end joining (NHEJ) findet statt, um Cas9-induzierte Strangbrüche durch zufällige Nukleotid-Insertionen oder Deletionen zu reparieren, was zur Störung und Inaktivierung des Zielgens führt. Wir beschreiben detailliert Methoden zur Gestaltung selektiver sgRNAs und zur Erzeugung lentiviral-kompatibler Vektoren, die die entworfenen sgRNAs enthalten. Dadurch können stabile genarme Endothelzellen effizient und zuverlässig erzeugt werden.
In dieser Studie verwendeten wir mikrovaskuläre Endothelzellen der mausen Pankreas (MS-1)14 als Modellsystem, um den TGF-β2-induzierten EndMT-Prozess zu untersuchen. Unsere vorherige Studie zeigte, dass Schnecke der Haupttranskriptionsfaktor ist, der durch TGF-β2 erhöht wird, wodurch EndMT in MS-1-Zellen induziert wird15. Nach der CRISPR/Cas9-Genbearbeitung zur Beendigung der Schneckenexpression in MS-1-Zellen konnte TGF-β2 EndMT nicht vermitteln. Dieser Workflow kann angewendet werden, um andere (vermutete) EndMT-bezogene Gene zu untersuchen.
Protokoll
1. Induktion von EndMT durch TGF-β2
- MS-1-Zellen in Dulbeccos modifiziertem Eagle Medium (DMEM) mit 10% fetalem Rinderserum (FBS) und 100 U/ml Penicillin/Streptomycin in einem Inkubator (5% CO2,37 °C). Alle Kulturschalen/-teller vor Gebrauch 10 min mit 0,1% w/v Gelatine beschichten.
- MS-1-Zellen vorsichtig mit 1x phosphatgepufferter Kochsalzlösung (PBS) waschen, 2 ml Trypsin-EDTA-Lösung (0,25% Trypsin und 0,02% EDTA) in eine 10 cm große Schüssel geben und 2 min bei 37 °C inkubieren, um sie zu lösen. Anschließend 5 ml vollständiges Kulturmedium hinzufügen, um die Reaktion abzuschrecken.
- Die Zellsuspension in ein 15 mL Röhrchen und eine Zentrifuge bei 200 x g für 3 min bei Raumtemperatur geben.
- Verwerfen Sie den Überstand und resuspendieren Sie die Zellen in 4 ml frischem Medium, das FBS und Penicillin/Streptomycin enthält. Zählen Sie die Zellen mit einem automatischen Zellenzähler.
- Samen Sie 1 x 103 Zellen pro cm2 für die weitere Kultur. Zum Beispiel 9,5 x 103 Zellen/Well für 6-Well-Platten oder 1,9 x 103 Zellen/Well für 24-Well-Platten aussäen.
- Inkubieren Sie die Zellen über Nacht, damit sie anhaften und sich erholen können, und stimulieren Sie dann MS-1-Zellen mit TGF-β2 für 3 Tage. Fügen Sie den TGF-β-Rezeptorkinase-Inhibitor SB431542 (5 μM) 30 min vor der TGF-β2-Stimulation hinzu. Behandeln Sie andere Zellen mit Vehicle (DMSO).
HINWEIS: TGF-β2 in 4 mM HCl auflösen, das 0,1% humanes Rinderserumalbumin (BSA) enthält. Fügen Sie der Kontrollgruppe die gleiche Menge Ligandenpuffer ohne TGF-β2 hinzu. Die TGF-β2-Konzentration kann für spezifische Assays auf 0,1-1 ng/ml angepasst werden. Siehe Hinweise in den entsprechenden Abbildungen. - Untersuchen Sie nach 3 Tagen die Zellmorphologie mit Hellfeldbildgebung (mit einem invertierten Mikroskop) und führen Sie eine Immunfluoreszenzfärbung durch (siehe Schritt 2), um EndMT-bedingte Markerveränderungen zu beurteilen. Führen Sie mindestens drei unabhängige Experimente durch, um biologische Triplikate zu erhalten.
2. Immunfluoreszenzfärbung
- Trypsinisieren Sie kultivierte MS-1-Zellen (Schritt 1.2) und säten Sie dann 1,9 x 103 Zellen auf einem 0,1% w/ v gelatinebeschichteten 12 mm runden Deckglas auf dem Boden einer 24-Well-Platte neu.
- Nachdem Sie die Zellen über Nacht kultiviert haben, fügen Sie TGF-β2 (Endkonzentration 1 ng / ml) für 3 Tage zu den Zellen hinzu. Verwenden Sie Medium, das Ligandenpuffer enthält, als Negativkontrolle.
- Führen Sie Eine Pecam-1- und Sm22α-Färbung durch.
- Nachdem Sie die Zellen 3 Tage lang mit TGF-β2 (oder Control) stimuliert haben, entfernen Sie das Medium und waschen Sie die Zellen mit 1x PBS.
- Fügen Sie 300 μL 4% Formaldehyd zu jeder Vertiefung hinzu und inkubieren Sie sie für 10 minuten bei Raumtemperatur, um die Zellen zu fixieren. Nach der Inkubation 3x mit 1x PBS waschen.
- Fügen Sie 300 μL 0,1% Triton X-100 in 1x PBS zu jeder Vertiefung hinzu und inkubieren Sie sie für 10 Minuten bei Raumtemperatur, um die Zellen zu permeabilisieren. Nach der Inkubation die Triton X-100 Lösung entfernen und die Zellen 3x mit 1x PBS waschen.
- Blockieren Sie die Zellen mit 3% Rinderserumalbumin (BSA) in 1x PBS für 45 min bei Raumtemperatur.
- Verdünnen Sie die primären Pecam-1- und Sm22α-Antikörper, die murine Proteine erkennen, 1:500 mit 1x PBS. Dann inkubieren Sie die fixierten Zellen mit primären Antikörpern für 45 min bei Raumtemperatur.
- Nach dem 3x Waschen mit 1x PBS die Zellen mit 1000x verdünnten sekundären Antikörpern, einschließlich Esel-Anti-Ratte Alexa 488 und Ziegen-Anti-Kaninchen Alexa 594, für 45 min bei Raumtemperatur inkubieren.
HINWEIS: Schützen Sie die Proben während der Fleckenfärbung vor Licht. - Nach dem 3x Spülen mit 1x PBS das mit Zellen ausgesäte Deckglas mit der Vorderseite nach unten auf einen Tropfen Montagemedium mit 4',6-Diamidino-2-phenylindol (DAPI) auf einen Objektträger legen, um die Kerne zu färben.
- Fixieren Sie die Peripherie des Deckglases mit transparentem Nagellack und lagern Sie es bei 4 °C.
- Erfassen Sie repräsentative Bilder mit einem konfokalen Mikroskop. Stellen Sie die Laserwellenlängen auf 405 nm, 488 nm und 552 nm ein, um DAPI, Pecam-1 bzw. Sm22α zu erkennen. Für jeden Kanal wurden alle Bilder mit den gleichen Einstellungen und der gleichen Belichtungszeit aufgenommen. Führen Sie mindestens drei unabhängige Experimente durch, um biologische Triplikate zu erhalten.
3. Knocken Sie die Schnecke mit CRISPR / Cas9-Bearbeitung aus
- Entwerfen Sie zwei unabhängige sgRNAs, die auf murine Schnecken abzielen.
- Entwerfen Sie sgRNAs mit den Online-Tools CHOPCHOP (https://chopchop.cbu.uib.no/) und Cas-OFFinder (http://www.rgenome.net/cas-offinder/) entsprechend dem angestrebten Gennamen und der Art.
- Prognostizieren Sie die Off-Target-Aktivität der entworfenen sgRNAs, die auf Snail abzielen, mit zwei unabhängigen Algorithmen, einschließlich Cas-OFFinder (http://www.rgenome.net/cas-offinder/) und CHOPCHOP (http://chopchop.cbu.uib.no/).
- Wählen Sie zwei sgRNAs mit der geringsten Off-Aktivität. Entwerfen Sie zwei komplementäre sgRNA-Oligos mit der BveI-Cut-Stelle. Das Sense-Oligo beginnt mit 5'-ACCG-3' und das Antisense-Oligo beginnt mit 5'-AAAC-3'.
- Ordnen Sie die Oligos an, um kommerziell für die weitere Verwendung synthetisiert zu werden.
- Klonen Sie die komplementären Leit-RNA-Oligos in die BveI-verdaute AA19 pLKO.1-puro.U6.sgRNA. Bve I-Stuffer Lentiviral vektorplasmid zur ErzeugungAA19 pLKO.1-Snail-sgRNA16.
- Schneiden Sie die AA19 pLKO.1-puro.U6.sgRNA. Bve I-Stuffer Plasmid mit dem BveI Enzym16. Mischen Sie 2 μg AA19 pLKO.1-puro.U6.sgRNA. Bve I-Stuffer-Plasmid, 5 μL 10x Buffer O und 5 μL BveI-Enzym und steriles Wasser hinzufügen, um ein Gesamtvolumen von 50 μL zu erreichen.
- Wirbel und drehen Sie kurz den Reaktionsmix herunter. Inkubieren Sie die Reaktion bei 37 °C für 1 h.
- Laden Sie die Reaktionsmischung auf ein 1% iges Agarosegel und führen Sie es in 1x Tris-Acetat-EDTA (TAE, 50x TAE-Stamm: 242 g in Wasser gelöste Tris-Base, 57,1 ml Eisessigsäure, 100 ml 500 mM EDTA (pH 8,0) Lösung und fügen Sie Wasser zu insgesamt 1 L) Puffer hinzu, bis eine gute Trennung erreicht ist.
- Schneiden Sie das Rückgratfragment aus dem Gel, isolieren Sie es mit einem Gelextraktionskit nach dem Protokoll des Herstellers (siehe Materialtabelle)und eluieren Sie das Rückgrat in 40 μL Elutionspuffer (EB).
- Mischen Sie 5 μL von 100 pmol/μL Sense-Oligo und 5 μL von 100 pmol/μL Antisense-Oligo mit 1 μL von 1 M Tris-HCl (pH 8,0) und fügen Sie steriles Wasser hinzu, um insgesamt 100 μL zu erreichen, um die gRNA-Oligos zu glühen. Inkubieren Sie die Mischung für 5 min bei 100 °C und bedecken Sie dann die Röhrchen mit Aluminiumfolie. Danach kühlen Sie die Lösung langsam auf Raumtemperatur ab, um sie weiter als Einsatz zu verwenden.
- Um die komplementären gRNA-Oligos und das BveI-verdaute Rückgrat zu ligaieren, mischen Sie 1 μL isoliertes BveI schnitt AA19 pLKO.1-puro.U6.sgRNA.BveI-Stuffer-Rückgrat und 2 μL Insert (verdünnt 1:300) mit 2 μL 10x T4 DNA-Ligase-Puffer und 1 μL T4-DNA-Ligase und fügen Sie steriles Wasser hinzu, um insgesamt 20 μL zu erreichen.
HINWEIS: Stellen Sie beim Ausführen der Ligation sicher, dass zwei Kontrollgruppen enthalten sind. Mischen Sie für eine Kontrollgruppe 1 μL isoliertes Rückgrat, 2 μL 10x T4-DNA-Ligasepuffer und 1 μL T4-DNA-Ligase und fügen Sie steriles Wasser hinzu, um insgesamt 20 μL zu erreichen, jedoch ohne Oligo-DNA. Für die andere Kontrollgruppe mischen Sie 1 μL isoliertes Rückgrat und 2 μL 10x T4-DNA-Ligase-Puffer und fügen sie steriles Wasser hinzu, um insgesamt 20 μL zu erreichen, jedoch ohne die geglühten Oligos und T4-Ligase. Diese beiden Kontrollligaturen werden verwendet, um den Reaktionshintergrund im nächsten Transformationsschritt zu bestimmen und anzuzeigen, wie effizient die Ligatur ist.
- Wandeln Sie das Reaktionsgemisch in kompetente TOP10 E. colium.
- Sammeln Sie die kompetenten TOP10 E. coli. Zellen aus dem -80 °C Gefrierschrank und auf Eis auftauen.
- Fügen Sie 2 μL Ligationsmischung zu 50 μL kompetenter Zellen hinzu und halten Sie das Röhrchen 30 Minuten lang auf Eis.
- Hitzeschocken Sie das Rohr bei 42 °C für 30 s. Legen Sie das Rohr für 2 min auf Eis.
- 950 μL frisches Lysogeniebrühemedium (LB) in die Mischung geben und bei 37 °C 60 min kräftig schütteln.
- Drehen Sie die Zellen herunter und plattieren Sie sie auf einer warmen Ampicillin-LB-Platte (100 μg/ml). Inkubieren Sie die Platte bei 37 °C über Nacht.
- Überprüfen Sie das erfolgreiche Einsetzen der gRNA-Oligos in das Plasmid.
- 3-5 Kolonien auf der Platte in 1 ml Ampicillin (100 μg/ml) mit LB-Medium aufnehmen und über Nacht bei 30 °C schütteln.
- Isolieren Sie die Plasmid-DNA mit einem Plasmid-Kit nach dem Protokoll des Herstellers (Table of Materials) und sequenzieren Sie es mit dem U6-Promotor-Primer 5'- GAGGGCCTATTTCCCATGATT -3', um die erfolgreiche Insertion des gRNA-Oligos zu überprüfen.
4. Schnecken-Knockout-MS-1-Zellen erzeugen
- Produzieren Sie lentivirale Partikel, die Cas9 oder Snail-Targeting gRNAs tragen.
- Kultur HEK 293T Zellen in DMEM mit 10% fetalem Rinderserum und 100 U/ml Penicillin/Streptomycin in 14,5 cm Geschirr (oder T75Kolben) in einem Inkubator (5% CO2,37 °C).
- Mischen Sie 9,9 μg Zielgenplasmid, AA19 pLKO.1-Snail-sgRNA oder pLV-Cas917 Plasmid zusammen mit den Helferplasmiden 3,5 μg pCMV-VSVG (kodiert für das G-Protein des vesikulären Stomatitisvirus, VSV-G), 6,6 μg drehzahlempfindliches Elementplasmid pMDLg-RRE (kodiert Gag und Pol) und 5,0 μg pRSV-REV (kodierend Rev) in 500 μL serumfreiem Medium. 50 μL Polyethylenimin (PEI) (2,5 mg/ml) in 500 μL serumfreiem Medium resuspendieren. Plasmide und PEI-Präparate durch Pipettieren auf und ab vorsichtig mischen. Inkubieren Sie die Mischung für 20 min bei Raumtemperatur.
- Transfizieren Sie HEK 293T-Zellen durch Zugabe des Mischungsmediums aus Schritt 4.1.2 bis 80% konfluenter Zellen in 14,5-cm-Schalen (oder T75-Kolben), die DMEM-Medium mit 10% FBS und 100 U / ml Penicillin / Streptomycin enthalten. HEK293T-Zellen werden verwendet, weil sie leicht transfiziert werden können und hohe Mengen an Virus18erzeugen.
- Transfizierte HEK 293T-Zellen in eine Biosicherheit im mikrobiologischen und biomedizinischen Labor (BMBL) überführen, um sie für 24 Stunden zu kulturieren.
- Ersetzen Sie in einem BMBL-Labor das Transfektionsmedium aus HEK 293T-Zellen durch 12 ml frisches vollständiges DMEM, das FBS und Penicillin/Streptomycin enthält. Inkubieren Sie die Zellen für 24 h.
- Sammeln und filtern Sie das Medium mit einer 20-ml-Spritze und einem 0,45-μm-Filter. Das konditionierte Medium in ein 15 mL Polypropylenrohr geben.
- Fügen Sie 12 ml frisches vollständiges DMEM mit FBS und Penicillin / Streptomycin in die HEK 293T-Kulturschale und -kultur für weitere 24 Stunden hinzu.
- Sammeln und filtern Sie das Medium mit einer 20-ml-Spritze und einem 0,45-μm-Filter. Das konditionierte Medium in ein 15 mL Polypropylenrohr geben. Lagern Sie das Medium, das lentivirale Partikel enthält, als 1 mL Aliquoten bei -80 °C zur weiteren Verwendung.
- MS-1-Zellen mit dem pLV-Cas9-Virus infizieren.
- Samen Sie 1 x10 5 MS-1-Zellen pro Vertiefung in einer 6-Well-Platte für 24 h vor der Lentivirus-Infektion.
- Die gefrorenen Aliquoten des pLV-Cas9-Virus in einem 37 °C-Wasserbad auftauen.
- Mischen Sie 1 ml Virusmedium mit 1 ml frischem DMEM-Medium, das FBS und Penicillin/Streptomycin enthält. Fügen Sie polybren zum Medium hinzu (Endkonzentration als 10 μg / ml), um die Infektionseffizienz zu erhöhen.
- Entfernen Sie das Medium von der 6-Well-Platte und ersetzen Sie es durch das Virus/Polybren-Mischmedium und kulturieren Sie die Zellen in einem Inkubator für 24 h. 24 h nach der Infektion, ersetzen Sie das Medium durch frisches Medium und kulturieren Sie die Zellen für weitere 24 h.
HINWEIS: Behalten Sie immer eine nicht infizierte Gut als Kontrollgruppe. - Saugen Sie das Medium aus der infizierten Gruppe und Kontrollgruppe ab und ersetzen Sie es durch DMEM-Medium mit 4 μg/ml Blasticidin.
- Die Platte in einen 37 °C-Inkubator zurückgeben und die Zellen 1 Woche lang kulturieren. Nicht infizierte Zellen sterben aufgrund der Wirkung von Blasticidin ab. Teilen Sie die überlebenden Zellen, wenn sie 80% Zellkonfluenz erreichen, und fahren Sie mit der Blasticidin-Selektion fort.
- Bestätigen Sie die Expression von Cas9 in MS-1-Zellen durch Western Blotting mit einem Antikörper gegen Cas9 (Molekulargewicht von Cas9 beträgt ca. 160 kDa).
- Infizieren Sie pLV-Cas9 MS-1-Zellen separat mit zwei unabhängigen gRNA-Lentiviren.
- 1 x 105 pLV-Cas9 MS-1 Zellen pro Vertiefung in einer 6-Well-Platte für 24 h vor der Infektion ansäpfen.
- Befolgen Sie das gleiche Protokoll wie in 4.2 beschrieben, um Zellen separat mit zwei gRNA-Lentiviren zu infizieren.
- Nach 24 Stunden Infektion mit dem gRNA-Virus erfrischen Sie das Medium und kulturieren Sie die Zellen für weitere 24 Stunden.
- Ersetzen Sie das Medium durch DMEM durch 1 μg/ml Puromycin. Die Platte in einen 37 °C-Inkubator zurückgeben und die Zellen 1 Woche lang kulturieren. Stellen Sie sicher, dass die nicht infizierten Zellen vollständig tot sind. Teilen Sie die Zellen, wenn sie 80% Konfluenz erreichen und setzen Sie die Puromycin-Selektion fort.
- Bestätigen Sie den Knock-out von Schnecke in MS-1-Zellen durch Western Blotting mit einem Antikörper gegen Snail (Molekulargewicht von Snail beträgt ca. 35 kDa).
Ergebnisse
TGF-β2 induziert EndMT und stimuliert die Schneckenexpression in MS-1-Endothelzellen
TGF-β ist eines der Zytokine mit dem größten Potenzial, EndMT zu induzieren. Nach 3-tägiger Behandlung von MS-1-Zellen mit TGF-β2 (1 ng/ml) verlieren endotheliale MS-1-Zellen ihre kopfsteinpflasterartige Struktur und differenzieren sich zu spindelförmigen mesenchymalähnlichen Zellen (Abbildung 1A)15. Um die Rolle von TGF-β2 bei der Induktion phänotypischer Zellveränderungen weiter zu überprüfen, haben wir die Zellen vor der TGF-β2-Stimulation mit der niedermolekularen Activinrezeptor-ähnlichen Kinase (ALK)4/ALK5/ALK7-Inhibitor SB431542 vorbehandelt19. SB431542 hat TGF-β2-induzierte Veränderungen der Zellmorphologie vollständig aufgehoben (Abbildung 1A). Der TGF-β2-induzierte EndMT-Prozess wurde weiter untersucht, indem Veränderungen in der Expression von EndMT-bezogenen Markern untersucht wurden. Wie in Abbildung 1Bgezeigt, war das Endothelprotein Pecam-1 nach TGF-β2-Stimulation stark erniedrigt, während der mesenchymale Faktor Sm22α durch TGF-β215stark hochreguliert wurde. Diese Daten stimmen mit der Vorstellung überein, dass TGF-β2 EndMT in MS-1-Zellen auslöste. Als nächstes untersuchten wir die Auswirkungen von TGF-β2 auf die Expression von Schnecken und Schnecken. Wie in Abbildung 1Cgezeigt, wurde Snail durch TGF-β2 deutlich hochreguliert, während die Slug-Expression in MS-1-Zellen nicht durch TGF-β2 beeinflusst wurde15. Die Quantifizierung der Schneckenexpression aus drei unabhängigen Experimenten ist in Abbildung 1D dargestellt.
Erschöpfung der Schnecke durch CRISPR/Cas9 in MS-1-Endothelzellen
Da Snail durch TGF-β2 induziert wurde und wahrscheinlich an TGF-β2-vermitteltem EndMT beteiligt war, führten wir eine CRISPR/Cas9-Genbearbeitung durch, um die Schneckenexpression in MS-1-Zellen genetisch zu erschöpfen. Wir stellten die Hypothese auf, dass die Erschöpfung der Schnecke ausreichen würde, um TGF-β2-induzierte EndMT zu hemmen. Wie in Abbildung 2Agezeigt, haben wir in zwei Schritten Schnecken-Knockout-Zellen generiert. Erstens wurde Cas9 ektopisch exprimiert, indem MS-1-Zellen mit einem Cas9-exprimierenden Lentivirus infiziert wurden. Da sich im pLV-Cas9-Konstrukt eine Blasticidin-Widerstandskassette befindet, haben wir die Expression von Cas9 durch Western-Blot-Analyse in Blasticidin-resistenten Zellen überprüft (Abbildung 2D). Anschließend haben wir sgRNAs eingeführt, die speziell auf Snail abzielten, um ihre Proteinexpression zu stören. Dieses Verfahren wurde auch durch Infektion mit lentiviralen Partikeln durchgeführt, die das AA19 pLKO.1-Snail-sgRNA-Konstrukt tragen, das eine Puromycin-Expressionskassette enthält. Cas9-exprimierende Zellen wurden erneut mit gRNA-haltigem Lentivirus infiziert und mit Puromycin weiter sele ausgewählt. Zwei komplementäre sgRNA-Oligos, die auf die murine Schnecke abzielen, wurden mit einer vorhergesagten niedrigen Off-Target-Aktivität entwickelt (Abbildung 2B, C). Nach der Einführung von zwei unabhängigen Snail sgRNAs in Cas9, die MS-1-Zellen exprimieren, wurde die Schneckenproteinexpression aufgehoben (Abbildung 2D)15.
Mangel an Schnecke hemmt TGF-β2-induzierte EndMT in MS-1-Zellen
Um die Funktion von Snail in TGF-β2-vermitteltem EndMT zu demonstrieren, führten wir einen EndMT-Assay in Schneckenzellen durch und verglichen ihn mit elterlichen MS-1-Zellen. Wie in Abbildung 3Agezeigt, war der Knockout von Snail ausreichend, um die fibroblastenähnliche Zellmorphologie zu hemmen, die durch TGF-β2 in MS-1-Zellen15angetrieben wird. Darüber hinaus wurden der TGF-β2-vermittelte Rückgang von Pecam-1 und die Erhöhung von Sm22α in Schnecken-erschöpften MS-1-Zellen vollständig blockiert. Zusammenfassend haben wir gezeigt, dass Schnecke für TGF-β2-vermittelte EndMT in MS-1-Zellen kritisch ist (Abbildung 3B)15.
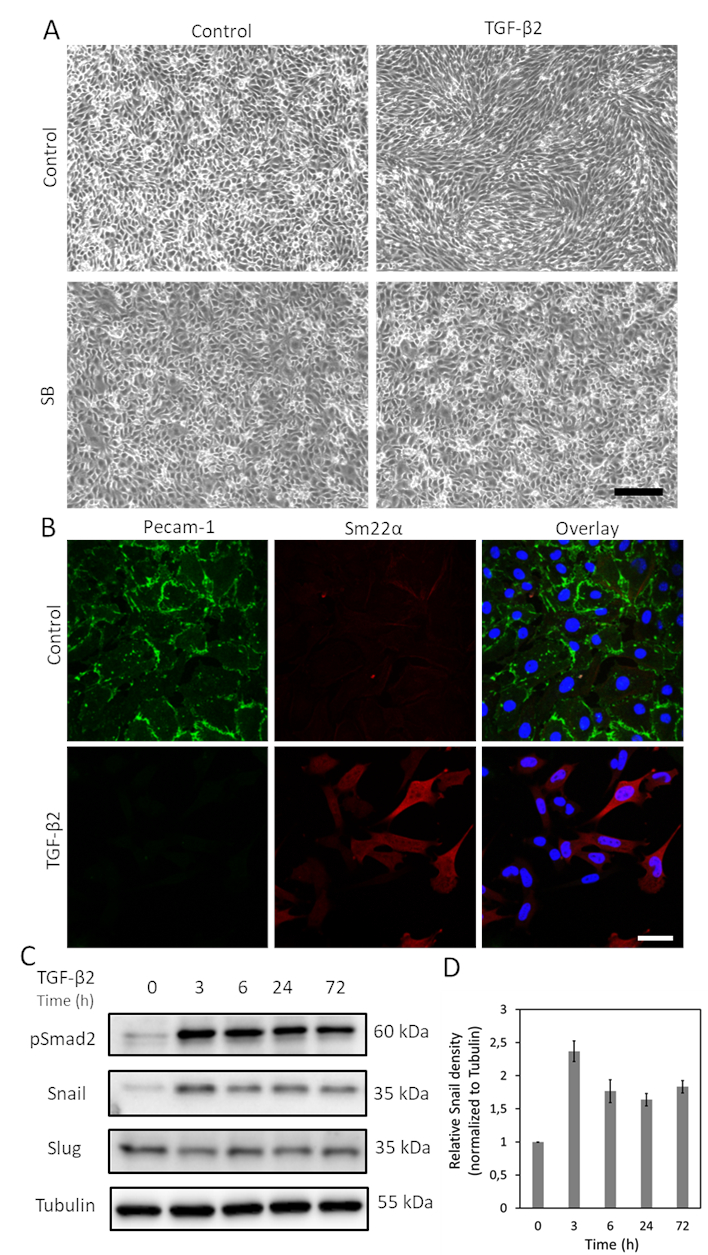
Abbildung 1. TGF-β2 induziert EndMT und Schneckenexpression in MS-1-Zellen. A. (A). Auswirkungen von TGF-β2 und/oder TGF-β Typ-I-Rezeptorkinase-Inhibitor SB-431542 auf die Zellmorphologie. Hellfeldaufnahmen von MS-1-Zellen nach Behandlung mit TGF-β2 (1 ng/ml) und/oder SB-431542 (SB, 5 μM, verabreicht 30 min vor TGF-β2) für 2 Tage. Der Maßstabsbalken steht für 200 μm. B. Immunfluoreszenzfärbung von Pecam-1 (grün) und Sm22α (rot) in MS-1-Zellen, die in einem Medium mit TGF-β2 (1 ng/ml) für 3 Tage kultiviert wurden. Kerne werden blau (DAPI) visualisiert. Maßstabsbalken: 50 μm. C. Western Blot mit ganzzelligem Lysat aus TGF-β2 stimulierten MS-1 Zellen. Die Expression von Schnecke, aber nicht von Slug, wurde durch TGF-β2-Stimulation verstärkt, wie zuvor in Ma et al15berichtet. D. Quantifizierung der Schneckenexpression durch Integration der Ergebnisse von drei unabhängigen Western-Blot-Experimenten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
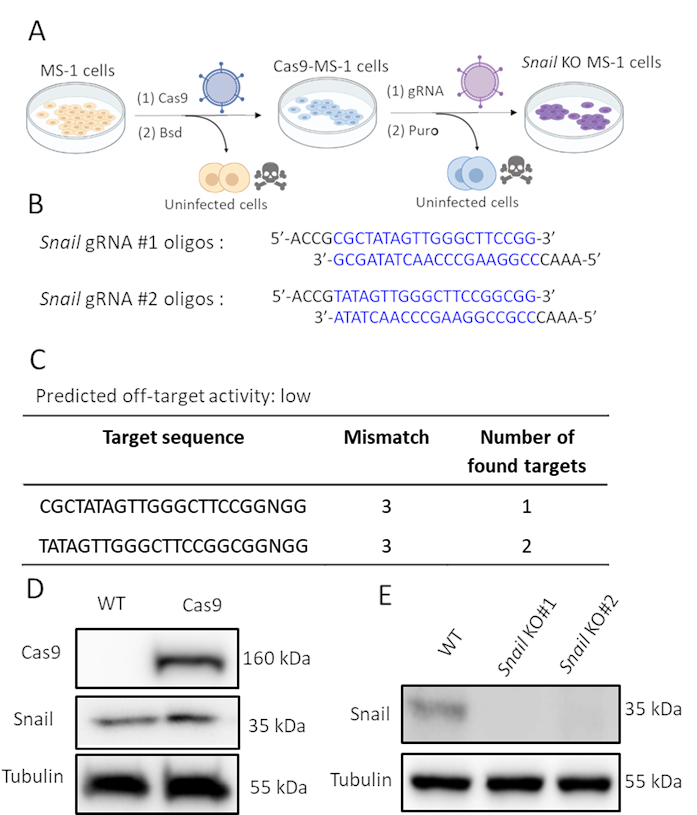
Abbildung 2. Depletion of Snail durch CRISPR-Cas9 Gen-Editing. A. (A). Schema, das zeigt, wie Schnecken-Knockout-Zellen generiert werden. Bsd: Blasticidin. Puro: Puromycin. B. Oligonukleotide von zwei unabhängigen sgRNAs, die mit CHOPCHOP (http://chopchop.cbu.uib.no/) und Cas-OFFinder (http://www.rgenome.net/cas-offinder/) auf Schnecken abzielen. C. (EN) Die vorhergesagte Off-Target-Aktivität der beiden gRNAs für Snail mit Cas-OFFinder (http://www.rgenome.net/cas-offinder/). D. Cas9- und Schneckenexpression in Wildtyp (WT) und Cas9-überexprimiertem MS-1, gemessen durch Western-Blot-Analyse. E. (D. ) Knockout von Schnecke mit zwei unabhängigen gRNAs in MS-1-Zellen, gemessen durch Western Blot-Analyse. Ähnliche Ergebnisse berichteten wir in Ma et al15. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
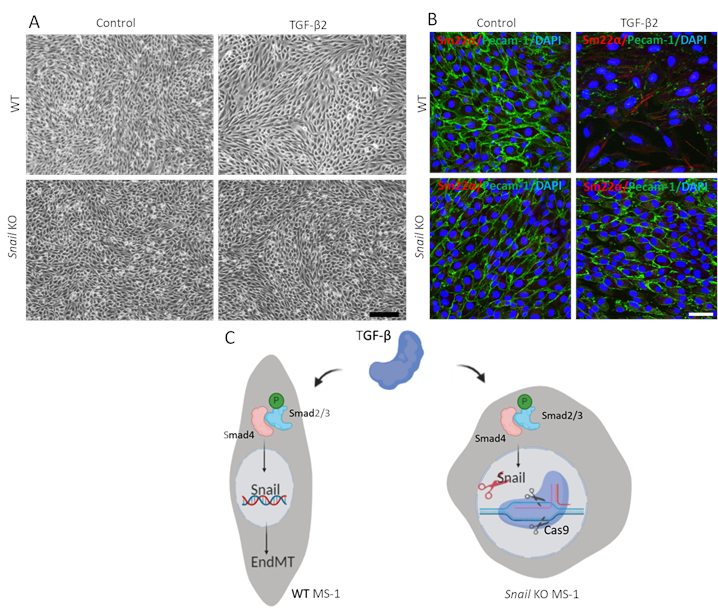
Abbildung 3. Genetische Depletion der Schnecke hemmt TGF-β2-induzierte EndMT in MS-1-Zellen. A. (A). Hellfeldaufnahmen von MS-1-Zellen nach Behandlung mit TGF-β2 (0,1 ng/ml) für 3 Tage in Wildtypzellen (WT, obere Platte) und Schnecken-Knocked-out-Zellen (untere Platte). Der Schuppenbalken stellt 200 μm dar. B. Immunfluoreszierende Färbung für Pecam-1 (grün), Sm22α (rot) und Kerne (blau) von MS-1-Zellen, die 3 Tage lang in einem Medium mit TGF-β2 (1 ng/ml) kultiviert wurden. Die Erschöpfung der Schnecke hat die TGF-β2-induzierte Abnahme von Pecam-1 und die Erhöhung der Sm22α-Expression aufgehoben. Der Skalenbalken stellt 50 μm dar. C. Schematische Darstellung der Wirkung von Schnecken-Knockout auf TGF-β-induzierte EndMT in MS-1-Zellen. TGF-β stimuliert die Expression von Snail durch Smad-Signalweg durch Phosphorylierung von Smad2/3 und treibt EndMT weiter an. Das Ausschalten von Schnecken mit CRISPR/Cas9-basierter Geneditierung hat TGF-β-vermittelte EndMT aufgehoben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Das Verständnis des Mechanismus von EndMT ist entscheidend für die Modulation dieses Prozesses und die Bekämpfung von EndMT-bedingten Krankheiten. Hier beschrieben wir Methoden, um einen TGF-β-induzierten EndMT-Assay durchzuführen und die Rolle der EndMT-TF-Schnecke bei TGF-β-ausgelöster EndMT zu untersuchen, indem wir CRISPR/Cas9-vermittelte stabile Genverarmung der Schnecke aus Zellen durchführen. Die Erschöpfung von Snail mit CRISPR/Cas9-Ansatz hat TGF-β2-getriebene EndMT in MS-1-Zellen erfolgreich aufgehoben (Abbildung 3C). Um die Auswirkungen von Zytokinen wie TGF-β auf EndMT zu untersuchen, wurden ECs Zytokinen ausgesetzt und dann wurde das Auftreten von EndMT nach morphologischen Veränderungen und endothelialen und mesenchymalen Markerexpressionsänderungen in Zellen beurteilt. TGF-β2 induzierte stark EndMT in MS-1-Zellen, begleitet von einem starken Anstieg der Expression des Transkriptionsfaktors Schnecke. Die durch TGF-β induzierten EndMT-TFs können je nach Spezies oder gewebespezifischem Endothelzelltyp unterschiedlich sein. Zum Beispiel beobachteten wir, dass Snail, aber nicht Slug durch TGF-β in MS-1-Zellen signifikant hochreguliert wurde, während in menschlichen Nabelschnurvenen-Endothelzellen (HUVECs) sowohl Snail als auch Slug nach Exposition gegenüber TGF-β20erhöht sind.
Wir bewerteten das Ausmaß des EndMT-Prozesses auf zwei Arten, indem wir Veränderungen der Zellmorphologie untersuchten und dann Veränderungen in der Expression von EndMT-bezogenen Markern untersuchten. Nach einer TGF-β Exposition für 3 Tage wurden die Zellen einer EndMT mit konsistenten morphologischen Variationen und Veränderungen in der Expression von EndMT-bezogenen Markern unterzogen. Neben der hier durchgeführten Immunfluoreszenzfärbung können Markervariationen auch durch Western Blotting auf Proteinexpressionsebene oder durch qRT-PCR (Real-Time Quantitative Reverse Transcription PCR) auf Genebene21überwacht werden. Zusätzlich zu diesen beiden zeit- und kostensparenden Methoden, die wir in diesem Protokoll gezeigt haben, gibt es weitere Methoden, um EndMT zu untersuchen. Beispielsweise kann die Durchführung einer Transkriptomanalyse (durch RNA-Sequenzierung oder qPCR) zum Vergleich der Expressionsniveaus endothelialer und mesenchymalver verwandter Gene zwischen behandelten und Kontrollzellen EndMT22,23genau beurteilen. Darüber hinaus beinhaltet EndMT oft den stabilen Verlust der Barrierefunktion, der durch Impedanzspektroskopie24beurteilt werden kann. Darüber hinaus können zusätzliche Nachweise für den Erwerb stammzellähnlicher Eigenschaften durch EndMT-abgeleitete Zellen untersucht werden. So können beispielsweise unter bestimmten Kulturbedingungen mesenchymale EndMT-Zellen weiter in Osteoblasten, Chondrozyten, Adipozyten oder (Myo-)Fibroblasten differenziert werden. Daher ist eine zusätzliche Analyse zur Bestätigung der Differenzierung in verschiedene Zelltypen, die zur Mesodermlinie gehören (d. H. Genexpression und Matrixfärbung), nützlich, um die multipotente Natur von EndMT-abgeleiteten Zellen zu demonstrieren. Schließlich sind die EndMT-Bewertungsmethoden nicht auf In-vitro-Studien beschränkt, sondern können extrapoliert werden, um den Zusammenhang zwischen EndMT und einigen Krankheiten in vivo oder in ex vivo Organen zu untersuchen. In diesem Sinne wird der Einsatz endothelspezifischer Lineage-Tracing-Strategien weitgehend auf endMT-bezogene Forschungausgeweitet 25.
Um die Rolle von Snail während EndMT zu untersuchen, wurde in dieser Studie CRISPR / Cas9-Gen-Editing verwendet, um dieses Gen auszuschalten. Die Daten zeigten, dass TGF-β2 die EndMT in Schneckenmangel-MS-1-Zellen nicht vermitteln konnte. Diese Beobachtung zeigte, dass Schnecke für TGF-β2-induzierte EndMT in MS-1-Zellen essentiell ist. Wir haben eine unabhängige U6-gesteuerte sgRNA-Expressionskassette verwendet, um spezifische sgRNAs für Cas9 einzuführen, um auf Snail abzuzielen. Zusätzlich zu dieser Methode beschrieben Ran et al.26 eine weitere Strategie zum Klonen der sgRNA-Oligos-Sequenz in das Cas9-Gerüst, um ein Konstrukt zu erzeugen, das sowohl Cas9 als auch gRNAs enthält. Neue ansätze ermöglichen es CRISPR/Cas, zusätzliche Funktionen zu integrieren. Zum Beispiel können doppelte oder dreifache Knockouts erreicht werden, indem mehr sgRNAs in Zellen geliefert werden, die Cas927exprimieren. Das entwickelte Cas13-Protein zielt auf RNA-Moleküle ab und verdaut sie, ohne die endogene DNA zu stören28. Neben dem Ausschalten von Genen mit CRISPR/Cas können kurze Haarnadel-RNAs (shRNAs) als Alternativen verwendet werden, um die gezielte Genexpression stabil zu bekämpfen29. Bei allen CRISPR/Cas-Gen-Editing-Methoden sollte immer die Off-Target-Spaltung berücksichtigt werden. Darüber hinaus schalten kleine interferierende RNAs (siRNAs) die Genexpression vorübergehend zum Schweigen und die siRNA-Konzentration wird mit Zellteilung30verdünnt. Beide Methoden unterdrücken teilweise die gezielte Genexpression. Im Gegensatz dazu wird die ektopische Genexpression auch verwendet, um die Genfunktion während EndMT/ EMT31zu überprüfen. Dieser Ansatz kann bestimmen, ob die Hochregulation eines Gens ausreicht, um eine EndMT-Reaktion hervorzurufen. Daher gibt es derzeit eine Vielzahl von technischen Strategien, mit denen potenzielle Regulatoren von EndMT identifiziert und verifiziert werden können. Außerdem kann die transkriptomische Analyse eine gute Option bei der Identifizierung und umfassenden Analyse von EndMT-bezogenen Regulatoren sein. Wir empfehlen, verschiedene komplementäre Ansätze zu verwenden, um die Modulation von EndMT zu untersuchen.
Zusammenfassend haben wir einen Workflow eingeführt, um Faktoren zu identifizieren, die während der TGF-β-induzierten EndMT eine funktionelle Rolle spielen können. Mit dieser Methode kann auch untersucht werden, ob andere Reize (z. B. Zytokine, Wachstumsfaktoren, mechanische Reize, Zell-Zell-Interaktionen) EndMT modulieren können, und das Zusammenspiel von TGF-β mit anderen Reizen. Darüber hinaus haben wir einen Ansatz mit CRIPSR/Cas-Gen-Editing hervorgehoben, um aufzuklären, ob ein bestimmtes Gen für TGF-β-induzierte EndMT benötigt wird. Um diese Methodik zu veranschaulichen, haben wir den starken EndMT-Induktor TGF-β2 in MS-1-Zellen verwendet, aber die Protokolle können an andere Zytokine und andere Zelltypen angepasst werden. Wir gehen davon aus, dass dieses detaillierte Protokoll als Sprungbrett für zukünftige EndMT-bezogene Studien dienen wird.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Die Forschung wurde von CGC.NL und der Netherlands Cardio Vascular Research Initiative unterstützt: der Dutch Heart Foundation, der Dutch Federation of University Medical Centers, der Netherlands Organization for Health Research and Development und dem Royal Netherlands Academy of Sciences Grant, der an die Phaedra-Impact (http://www.phaedraresearch.nl) vergeben wurde. JM wird vom Chinese Scholarship Council unterstützt. GSD wird durch ein Trampolin-Stipendium von AFM-Telethon [22379], FOP Italia und ein Stipendium von La Fundació La Marató de TV3 (#202038) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | Pall Corporation, USA | 4614 | |
| 10× T4 DNA ligase buffer | Thermofisher Scientific, USA | B69 | |
| 12 mm round glass slice | Knittel Glass,Germany | VD10012Y1A.01 | |
| 20 mL syringe | BD Eclipse, USA | 300629 | |
| 4′,6-diamidino-2-phenylindole (DAPI) | Vector Laboratories, USA | H-1200 | VECTASHIELD Antifade Mounting Media |
| AA19_PLKO vector | Dr. M Gonçalves, Leiden University Medical Center, Netherlands | Gift | |
| Ampicillin | Serva Electrophoresis, USA | 1339903 | |
| Anti-Mouse IgG | GE Healthcare, USA | NA931 | |
| Anti-Rabbit IgG | Cell signaling, USA | 7074 | |
| Agarose | Roche, Switzerland | 11388991001 | |
| Blasticidin | Invitrogen, USA | R21001 | |
| Buffer O (10X) | Thermofisher Scientific, USA | BO5 | |
| BveI (Bspm1) | Thermofisher Scientific, USA | ER 1741 | |
| Confocal microscope | Leica Microsystems, Germany | SP8 | |
| DMEM | Thermo Fisher Scientific, USA | 11965092 | |
| Donkey anti-rat Alexa 488 | Invitrogen, USA | A21208 | |
| FBS | Thermo Fisher Scientific, USA | 16000044 | |
| Formaldehyde | Thermo Fisher Scientific, USA | 28908 | |
| Inverted microscope | Leica Microsystems, Germany | DMi8 | |
| Goat anti-rabbit Alexa 594 | Invitrogen,USA | A11012 | |
| LabNed Plasmid kit | LabNed, USA | LN2400004 | |
| MS-1 cell line | American Type Culture Collection (ATCC) | ||
| Nail polish | HEMA, Netherlands | Transparent | |
| Pecam-1 antibody | Becton Dickinson,USA | 553370 | |
| PEI | Polysciences, USA | 23966-1 | |
| PLV-Cas9 plasmid | Sigma-Aldrich, USA | Cas9BST-1EA | |
| Puromycin | Sigma-Aldrich, USA | P9620 | |
| QIAquick gel extraction kit | Qiagen, Germany | 28706 | |
| Sm22a antibody | Abcam, UK | ab14106 | |
| Snail | Cell signaling, USA | 3879 | |
| T4 DNA ligase | Thermofisher Scientific, USA | EL 0014 | |
| TC20 automated Cell Counter | Bio-Rad, USA | 1450102 | |
| Human TGF-β2 | Joachim Nickel, University of Wurzburg | Gift | Other commercial recommendation: 302-B2, R&D systems |
| Triton X-100 | Merck, USA | 1086031000 | |
| SB431542 | Tocris Bioscience, UK | 1614 | |
| Tris | Roche, Switzerland | 11814273001 |
Referenzen
- Ma, J., Sanchez-Duffhues, G., Goumans, M. -. J., Ten Dijke, P. TGF-β-induced endothelial to mesenchymal transition in disease and tissue engineering. Frontiers in Cell and Developmental Biology. 8, (2020).
- Piera-Velazquez, S., Jimenez, S. A. Endothelial to mesenchymal transition: role in physiology and in the pathogenesis of human diseases. Physiological Reviews. 99 (2), 1281-1324 (2019).
- Sánchez-Duffhues, G., de Vinuesa, G. A., ten Dijke, P. Endothelial-to-mesenchymal transition in cardiovascular diseases: developmental signaling pathways gone awry. Developmental Dynamics. 247 (3), 492-508 (2018).
- Evrard, S. M., et al. Endothelial to mesenchymal transition is common in atherosclerotic lesions and is associated with plaque instability. Nature Communications. 7 (1), 1-16 (2016).
- Qiao, L., et al. Endothelial fate mapping in mice with pulmonary hypertension. Circulation. 129 (6), 692-703 (2014).
- Zeisberg, E. M., et al. Endothelial-to-mesenchymal transition contributes to cardiac fibrosis. Nature Medicine. 13 (8), 952-961 (2007).
- Clere, N., Renault, S., Corre, I. Endothelial-to-mesenchymal transition in cancer. Frontiers in Cell and Developmental Biology. 8, (2020).
- Saito, A. EMT and EndMT: regulated in similar ways. The Journal of Biochemistry. 153 (6), 493-495 (2013).
- Bolós, V., et al. The transcription factor Slug represses E-cadherin expression and induces epithelial to mesenchymal transitions: a comparison with Snail and E47 repressors. Journal of Cell Science. 116 (3), 499-511 (2003).
- Aird, W. C. Endothelial cell heterogeneity. Cold Spring Harbor Perspectives in Medicine. 2 (1), 006429 (2012).
- Yang, J., et al. Guidelines and definitions for research on epithelial-mesenchymal transition. Nature Reviews Molecular Cell Biology. , 1-12 (2020).
- Doudna, J. A., Charpentier, E. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), (2014).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Arbiser, J. L., et al. Oncogenic H-ras stimulates tumor angiogenesis by two distinct pathways. Proceedings of the National Academy of Sciences USA. 94 (3), 861-866 (1997).
- MA, J., et al. TGF-?-induced endothelial to mesenchymal transition results from a balance between SNAIL and ID factors. Frontiers in Cell and Developmental Biology. 182 (9), (2021).
- Chen, X., et al. Probing the impact of chromatin conformation on genome editing tools. Nucleic Acids Research. 44 (13), 6482-6492 (2016).
- Hunter, F. W., et al. Functional CRISPR and shRNA screens identify involvement of mitochondrial electron transport in the activation of evofosfamide. Molecular Pharmacology. 95 (6), 638-651 (2019).
- Mao, Y., et al. Lentiviral vectors mediate long-term and high efficiency transgene expression in HEK 293T cells. International Journal of Medical Sciences. 12 (5), 407 (2015).
- Liu, S., et al. Deubiquitinase activity profiling identifies UCHL1 as a candidate oncoprotein that promotes TGFβ-induced breast cancer metastasis. Clinical Cancer Research. 26 (6), 1460-1473 (2020).
- Medici, D., et al. Conversion of vascular endothelial cells into multipotent stem-like cells. Nature Medicine. 16 (12), 1400 (2010).
- Kokudo, T., et al. Snail is required for TGFβ-induced endothelial-mesenchymal transition of embryonic stem cell-derived endothelial cells. Journal of Cell Science. 121 (20), 3317-3324 (2008).
- Dong, J., et al. Single-cell RNA-seq analysis unveils a prevalent epithelial/mesenchymal hybrid state during mouse organogenesis. Genome Biology. 19 (1), 1-20 (2018).
- Oatley, M., et al. Single-cell transcriptomics identifies CD44 as a marker and regulator of endothelial to haematopoietic transition. Nature communications. 11 (1), 1-18 (2020).
- Halaidych, V., et al. Inflammatory responses and barrier function of endothelial cells derived from human induced pluripotent stem cells. Stem Cell Reports. 10 (5), 1642-1656 (2018).
- Li, Y., Lui, K. O., Zhou, B. Reassessing endothelial-to-mesenchymal transition in cardiovascular diseases. Nature Reviews Cardiology. 15 (8), 445-456 (2018).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Grav, L. M., et al. One-step generation of triple knockout CHO cell lines using CRISPR/Cas9 and fluorescent enrichment. Biotechnology Journal. 10 (9), 1446-1456 (2015).
- Abudayyeh, O. O., et al. RNA targeting with CRISPR-Cas13. Nature. 550 (7675), 280-284 (2017).
- Paddison, P. J., et al. Cloning of short hairpin RNAs for gene knockdown in mammalian cells. Nature Methods. 1 (2), 163-167 (2004).
- Reynolds, A., et al. Rational siRNA design for RNA interference. Nature Biotechnology. 22 (3), 326-330 (2004).
- Lourenço, A. R., et al. C/EBPɑ is crucial determinant of epithelial maintenance by preventing epithelial-to-mesenchymal transition. Nature Communications. 11 (1), 1-18 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten