Method Article
Endotelial mediado por TGF-β para transição mesenquimal (EndMT) e a avaliação funcional dos efeitos endMT usando edição de genes CRISPR/Cas9
Neste Artigo
Resumo
Descrevemos métodos para investigar o EndMT induzido pelo TGF-β2 em células endoteliais observando alterações de morfologia celular e examinando as alterações de marcadores relacionadas ao EndMT usando a coloração da imunofluorescência. A edição de genes CRISPR/Cas9 foi descrita e usada para esgotar o caracol de codificação genética para investigar seu papel no EndMT induzido pelo TGF-β2.
Resumo
Em resposta a pistas externas específicas e à ativação de certos fatores de transcrição, as células endoteliais podem se diferenciar em um fenótipo mesenquimal, um processo que é chamado de endotelial à transição mesenquimal (EndMT). Resultados emergentes sugerem que o EndMT está causalmente ligado a múltiplas doenças humanas, como fibrose e câncer. Além disso, as células mesenquimais derivadas da endotelial podem ser aplicadas em procedimentos de regeneração tecidual, pois podem ser ainda mais diferenciadas em vários tipos celulares (por exemplo, osteoblastos e condrócitos). Assim, a manipulação seletiva do EndMT pode ter potencial clínico. Como a transição epitelial-mesenquimal (EMT), endMT pode ser fortemente induzida pela citocina secreta transformando fator-beta (TGF-β), que estimula a expressão dos chamados fatores de transcrição endmt (EndMT-TFs), incluindo Snail e Slug. Estes EndMT-TFs então para cima e para baixo regulam os níveis de proteínas mesenquimais e endoteliais, respectivamente. Aqui, descrevemos métodos para investigar o EndMT induzido pelo TGF-β in vitro, incluindo um protocolo para estudar o papel de TFs específicos em EndMT induzido por TGF-β. Usando essas técnicas, fornecemos evidências de que o TGF-β2 estimula o EndMT em células microvasculares microvasculares pancreáticas murinas (células MS-1), e que o esgotamento genético do Caracol usando repetições palindrômicas curtas interespaçadas (CRISPR)/CRISPR-mediada por proteína 9 (Cas9)- revoga esse fenômeno. Essa abordagem pode servir como modelo para interrogar potenciais moduladores da biologia endotelial, e pode ser usada para realizar telas genéticas ou farmacológicas, a fim de identificar novos reguladores do EndMT, com potencial aplicação em doenças humanas.
Introdução
A transição endotelial para mesenquimal (EndMT) é um fenômeno biológico multicamuque dinâmico que tem sido ligado a diversos processos fisiológicos e patológicos1,2. Após endMT células endoteliais gradualmente perdem seus traços endoteliais, ao mesmo tempo em que adquirem propriedades mesenquimais3; assim, células endoteliais bem compactadas e bem organizadas se diferenciam em células mesenquimais alongadas. Alterações morfológicas no EndMT coincidem com alterações na expressão de certos genes e proteínas. Em geral, a expressão de proteínas que mantêm características endoteliais, incluindo endotelial vascular (VE)-cadherina, plaqueta/ce molécula-1 (CD31/Pecam-1) diminui. Simultaneamente, proteínas relacionadas às funções mesenquimais, como α-suave actina muscular (α-Sma) e proteína muscular lisa 22α (Sm22α) se acumulam. Resultados emergentes demonstraram que o EndMT pós-natal contribui para o desenvolvimento de doenças humanas, como câncer, fibrose cardíaca, hipertensão arterial pulmonar (AP), aterosclerose (AS), fibrose de órgãos, etc2,4,5,6,7. Uma compreensão mais profunda dos mecanismos subjacentes do EndMT e como dirigir o processo EndMT fornecerá novos métodos terapêuticos para doenças relacionadas ao EndMT e medicina regenerativa.
O TGF-β é um dos principais indutores endMT, e outros fatores envolvidos conhecidos incluem Wnt/β-catenin, Notch e algumas citocinas inflamatórias1. Como o contexto celular é fundamental para respostas desencadeadas pelo TGF-β, a interação do TGF-β com outros sinais de promoção do EndMT é relevante para o TGF-β provocar uma resposta EndMT. Após a ativação dos receptores de superfície de célula TGF-β tipo I e tipo II serina/threonina quinase, a via canônica intracelular Smad é ativada. TGF-β smad2/3 mediado por receptores formam complexos heteroméricos com Smad4 que se deslocam para o núcleo, onde eles regulam a expressão de fatores de transcrição relacionados ao EndMT. Semelhante à transição epitelial-mesenquimal (EMT), fatores de transcrição como Caracol, Lesma, Torção, Zeb1 e Zeb2 são induzidos pela sinalização TGF-β e contribuem para a reprogramação genética no EndMT8.
O caracol tem sido frequentemente identificado como um fator-chave no EndMT. Caracol se liga ao promotor de genes que codificam proteínas de adesão celular-célula e suprime sua transcrição, que é contrabalanceada pelo aprimoramento da expressão das proteínas mesenquimais9. As células endoteliais compreendem uma população muito heterogênea e a influência relativa de diversos estímulos extracelulares no EndMT pode diferir entre contextos celulares endoteliais ou tiposcelulares 10. Devido às suas semelhanças com o EMT, algumas metodologias são úteis para investigar tanto os mecanismos EMT quanto EndMT8. Nesse sentido, a ASSOCIAÇÃO INTERNACIONAL EMT (TEMTIA) enfatiza fortemente a necessidade de técnicas complementares para, em última instância, demonstrar a ocorrência do EMT/EndMT11.
Aqui descrevemos um método para monitorar e visualizar o processo EndMT induzido pelo TGF-β. A coloração da imunofluorescência fornece as informações básicas sobre mudanças de expressão em proteínas/marcadores direcionados, que são usados como indicadores de se o processo EndMT ocorre. Além disso, a coloração da imunofluorescência pode visualizar a localização de proteínas/marcadores e morfologia celular. Para estudar a atividade potencial de TFs específicos (ou outros reguladores a montante ou a jusante) envolvidos na edição de genes endMT mediados pelo TGF-β, descrevemos um protocolo usando repetições palindômicas curtas interespaçadas regularmente (CRISPR) /CRISPR-associated protein 9 (Cas9) para esgotar genes específicos das células, usando o Caracol TF como exemplo. Cas9 é uma endonuclease de DNA guiada por RNA dupla que reconhece e corta sequências complementares às sequências CRISPR em bactérias12. O sistema CRISPR/Cas9 é atualmente amplamente utilizado porque facilita a engenharia genética in vitro e in vivo13. Dirigido por um único guia RNA (sgRNA), o Cas9 expresso em êxtase gera uma quebra dupla de fios em uma sequência de alvos pré-selecionado em um lócus genético específico. A junção final não homólogo (NHEJ) ocorre para reparar quebras de fios induzidos pelo Cas9, através de inserções aleatórias de nucleotídeos ou exclusões, levando assim à interrupção e inativação do gene alvo. Descrevemos em detalhes métodos para projetar sgRNAs seletivos e gerar vetores compatíveis com lentiviral contendo os sgRNAs projetados. Como resultado, células endoteliais estáveis empobrecidas por genes podem ser geradas de forma eficiente e confiável.
Neste estudo, utilizamos células endoteliais microrresculares pancreáticas murinas (MS-1)14 como um sistema modelo para examinar o processo EndMT induzido pelo TGF-β2. Nosso estudo anterior demonstrou que o Caracol é o principal fator de transcrição aumentado pelo TGF-β2, pelo qual o EndMT é induzido nas células MS-115. Após a edição do gene CRISPR/Cas9 para revogar a expressão do caracol em células MS-1, o TGF-β2 não conseguiu mediar o EndMT. Este fluxo de trabalho pode ser aplicado para estudar outros (suspeitos) genes relacionados ao EndMT.
Protocolo
1. Indução de EndMT por TGF-β2
- Células MS-1 no meio águia modificada (DMEM) modificado de Dulbecco contendo 10% de soro bovino fetal (FBS) e penicilina/estreptomicina U/mL em uma incubadora (5% CO2, 37 °C). Cubra todos os pratos/pratos de cultura com 0,1% de gelatina w/v por 10 minutos antes de usar.
- Lave suavemente as células MS-1 com 1x de salina tamponada de fosfato (PBS), adicione 2 mL de solução trypsin-EDTA (0,25% trypsin e 0,02% EDTA) a um prato de 10 cm e incubar por 2 min a 37 °C para descolá-las. Posteriormente, adicione 5 mL de meio de cultura completa para saciar a reação.
- Transfira a suspensão celular para um tubo de 15 mL e centrífuga a 200 x g por 3 min a temperatura ambiente.
- Descarte o supernasciante e resuspenque as células em 4 mL de meio fresco contendo FBS e penicilina/estreptomicina. Conte as células usando um contador automático de células.
- Semente 1 x 103 células por cm2 para outra cultura. Por exemplo, sementes 9,5 x 103 células/poço para placas de 6 poços, ou 1,9 x 103 células/poço para placas de 24 poços.
- Incubar as células durante a noite para permitir que elas aderam e se recuperem, em seguida, estimular células MS-1 com TGF-β2 por 3 dias. Adicione o inibidor de quinase receptora TGF-β SB431542 (5 μM) 30 min antes da estimulação TGF-β2. Tratar outras células com veículo (DMSO).
NOTA: Dissolver TGF-β2 em 4 mM HCl contendo 0,1% de albumina de soro bovino humano (BSA). Adicione a mesma quantidade de tampão de ligante sem TGF-β2 ao grupo de controle. A concentração TGF-β2 pode ser adaptada para 0,1-1 ng/mL para ensaios específicos. Veja as indicações nos números correspondentes. - Após 3 dias, examine a morfologia celular com imagens de campo brilhante (com um microscópio invertido) e realize a coloração da imunofluorescência (ver passo 2) para avaliar as alterações de marcadores relacionadas ao EndMT. Realize pelo menos três experimentos independentes para obter triplicados biológicos.
2. Coloração de imunofluorescência
- Trippsinize células MS-1 cultivadas (passo 1.2) e, em seguida, ressarem 1,9 x 103 células em um vidro de cobertura redonda de 0,1% w/v revestido de gelatina de 12 mm colocado na parte inferior de uma placa de 24 poços.
- Depois de esculpir as células durante a noite, adicione TGF-β2 (concentração final 1 ng/mL) às células por 3 dias. Use o meio contendo tampão de ligamento como um controle negativo.
- Realize a coloração pecam-1 e Sm22α.
- Depois de estimular as células com TGF-β2 (ou Controle) por 3 dias, remova o meio e lave as células com 1x PBS.
- Adicione 300 μL de 4% de formaldeído a cada poço e incubar por 10 minutos à temperatura ambiente para fixar as células. Lave 3x com 1x PBS após a incubação.
- Adicione 300 μL de 0,1% Triton X-100 em 1x PBS a cada poço e incubar por 10 minutos à temperatura ambiente para permeabiliizar as células. Após a incubação, remova a solução Triton X-100 e lave as células 3x com 1x PBS.
- Bloqueie as células com 3% de albumina de soro bovino (BSA) em 1x PBS por 45 min a temperatura ambiente.
- Diluir os anticorpos Pecam-1 e Sm22α primários que reconhecem proteínas murinas 1:500 com 1x PBS. Em seguida, incubar as células fixas com anticorpos primários por 45 minutos à temperatura ambiente.
- Depois de lavar 3x com 1x PBS, incubar as células com anticorpos secundários diluídos de 1000x, incluindo o anti-rato de burro Alexa 488 e o anti-coelho de cabra Alexa 594, por 45 minutos à temperatura ambiente.
NOTA: Proteja as amostras contra a luz durante a coloração. - Depois de enxaguar 3x com PBS 1x, coloque o vidro de cobertura semeado com células de frente para baixo em uma gota de meio de montagem contendo 4',6-diamidino-2-fenilôndole (DAPI) em um slide para manchar os núcleos.
- Fixar a periferia do vidro da tampa com esmalte transparente e armazená-lo a 4 °C.
- Adquira imagens representativas com um microscópio confocal. Defina os comprimentos de onda laser em 405 nm, 488 nm e 552 nm para detectar DAPI, Pecam-1 e Sm22α, respectivamente. Para cada canal, todas as fotos foram tiradas com as mesmas configurações e tempo de exposição. Realize pelo menos três experimentos independentes para obter triplicados biológicos.
3. Derrube o Caracol usando edição CRISPR/Cas9
- Projete dois sgRNAs independentes visando caracolmurine .
- Design sgRNAs usando as ferramentas on-line CHOPCHOP (https://chopchop.cbu.uib.no/) e Cas-OFFinder (http://www.rgenome.net/cas-offinder/) de acordo com o nome e espécie do gene alvo.
- Prever a atividade fora do alvo das sgRNAs projetadas visando o Caracol com dois algoritmos independentes, incluindo Cas-OFFinder (http://www.rgenome.net/cas-offinder/) e CHOPCHOP (http://chopchop.cbu.uib.no/).
- Escolha dois sgRNAs com o menor fora de atividade. Projete dois oligos sgRNA complementares com o site de corte bveI. O oligo de sentido começa com 5'-ACCG-3' e o oligo antissense começa com 5'-AAAC-3'.
- Ordene que os oligos sejam sintetizados comercialmente para uso posterior.
- Clone o guia complementar RNA oligos no BveI digerido AA19 pLKO.1-puro.U6.sgRNA. Bve Plasmídeo vetorial lentiviral para gerar AA19 pLKO.1-Snail-sgRNA16.
- Corte o AA19 pLKO.1-puro.U6.sgRNA. Bve Plasmid de recheio com a enzima BveI16. Misture 2 μg de AA19 pLKO.1-puro.U6.sgRNA. Bve Plasmid de recheador I, 5 μL de 10x Buffer O, e 5 μL de enzima BveI e adicionar água estéril para atingir um volume total de 50 μL.
- Vórtice e gire brevemente a mistura de reação. Incubar a reação a 37 °C por 1 h.
- Carregue a mistura de reação em um gel de 1% de agarose e execute em 1x Tris-acetato-EDTA (TAE, 50x ESTOQUE TAE: 242 g de base Tris dissolvido em água, 57,1 mL de ácido acético glacial, 100 mL de 500 mM EDTA (pH 8.0) solução, e adicionar água a um total de 1 L) tampão até que uma boa separação seja alcançada.
- Corte o fragmento da espinha dorsal do gel, isole-o com um kit de extração de gel de acordo com o protocolo do fabricante (ver a Tabela de Materiais) e elute a espinha dorsal em 40 μL tampão de eluição (EB).
- Misture 5 μL de 100 pmol/μL sense oligo e 5 μL de 100 pmol/μL antisense oligo com 1 μL de 1 M Tris-HCl (pH 8.0) e adicione água estéril para atingir um total de 100 μL para anneal o gRNA oligos. Incubar a mistura por 5 min a 100 °C e, em seguida, cubra os tubos com papel alumínio. Depois disso, esfrie lentamente a solução para a temperatura ambiente para mais uso como uma inserção.
- Para ligar os oligos gRNA complementares e a espinha dorsal digerida por BveI, misturar 1 μL de Bveisolado corte AA19 pLKO.1-puro.U6.sgRNA.BveI-stuffer backbone e 2 μL de inserção (diluído 1:300) com 2 μL de tampão de ligase de DNA T4 10x e 1 μL de ligase de DNA T4, e adicionar água estéril para atingir um total de 20 μL. Gire brevemente o tubo e incuba-o por 4h à temperatura ambiente para uso posterior.
NOTA: Ao realizar a ligadura, certifique-se de que dois grupos de controle estejam incluídos. Para um grupo controle, misture 1 μL de espinha dorsal isolada, 2 μL de tampão de ligase de DNA T4 10x e 1 μL de liga ligase de DNA T4 e adicione água estéril para atingir um total de 20 μL, mas sem DNA oligo. Para o outro grupo controle, misture 1 μL de espinha dorsal isolada e 2 μL de tampão de ligase de DNA T4 de 10x, e adicione água estéril para atingir um total de 20 μL, mas sem os oligos resaled e t4 ligase. Essas duas ligaduras de controle são usadas para determinar o fundo de reação na próxima etapa de transformação e indicar o quão eficiente é a ligadura.
- Transforme a mistura de reação em TOP10 E. colicompetente .
- Colete o TOP10 E. colicompetente . células do congelador -80 °C, e descongelá-las no gelo.
- Adicione 2 μL de mistura de ligadura a 50 μL de células competentes e mantenha o tubo no gelo por 30 minutos.
- Choque térmico do tubo a 42 °C para 30 s. Coloque o tubo no gelo por 2 minutos.
- Adicione 950 μL de caldo de liseogenia fresco (LB) médio à mistura e agite vigorosamente a 37 °C por 60 min.
- Gire para baixo e emplaque as células em uma placa LB de resistência de ampicillina quente (100 μg/mL). Incubar a placa a 37 °C durante a noite.
- Verifique a inserção bem sucedida dos oligos gRNA no plasmid.
- Escolha 3-5 colônias na placa em 1 mL de ampicillina (100 μg/mL) contendo meio LB e agite durante a noite a 30 °C.
- Isole o DNA plasmídeo com um kit de plasmídeo de acordo com o protocolo do fabricante (Tabela de Materiais) e sequenciá-lo com o primer de promotores U6 5'- GAGGGCCTATTTCCCATGATT -3' para verificar a inserção bem sucedida do oligo gRNA.
4. Gerar células MS-1 de eliminação de caracol
- Produzir partículas lentiviras que carregam Cas9 ou GRNAs de alvo de caracol.
- Cultura HEK 293T células em DMEM contendo 10% de soro bovino fetal e penicilina/estreptomicina U/mL em pratos de 14,5 cm (ou frascos T75) em uma incubadora (5% CO2, 37 °C).
- Misture 9,9 μg de plasmídeo genético de alvo, AA19 pLKO.1-Snail-sgRNA ou pLV-Cas917 plasmid, juntamente com o plasmídeo auxiliar 3,5 μg de pCMV-VSVG (codificando a proteína G do vírus da estomatite veicular, VSV-G), 6,6 μg de elemento rev-responsivo plasmídeo pMDLg-RRE (codificação Gag and Pol) e 5,0 μg de pRSV-REV (codificação Rev) em 500 μL de meio livre de soro. Resuspend 50 μL de polietilenimina (PEI) (2,5 mg/mL) em 500 μL de meio livre de soro. Misture suavemente plasmídeos e preparações PEI por pipetar para cima e para baixo. Incubar a mistura por 20 minutos em temperatura ambiente.
- Transfect HEK 293T células adicionando o meio de mistura da etapa 4.1.2 a 80% células confluentes em pratos de 14,5 cm (ou frascos T75) que contêm mamêndulo com 10% FBS e penicilina/estreptomicina de 100 U/mL. As células HEK293T são usadas porque são facilmente transfeinadas e geram altos níveis de vírus18.
- Transfira células HEK 293T transfectadas para uma biossegurança em laboratório microbiológico e biomédico (BMBL) para cultuá-las por 24 horas.
- Em um laboratório BMBL, substitua o meio de transfecção das células HEK 293T por 12 mL de DMEM completo fresco contendo FBS e penicilina/estreptomicina. Incubar as células por 24 h.
- Colete e filtre o meio com uma seringa de 20 mL e um filtro de 0,45 μm. Transfira o meio condicionado para um tubo de polipropileno de 15 mL.
- Adicione 12 mL de DMEM completo fresco contendo FBS e penicilina/estreptomicina ao prato e cultura da cultura HEK 293T por mais 24 h.
- Colete e filtre o meio com uma seringa de 20 mL e um filtro de 0,45 μm. Transfira o meio condicionado para um tubo de polipropileno de 15 mL. Armazene o meio contendo partículas lentivirais como alíquotas de 1 mL a -80 °C para uso posterior.
- Infecte células MS-1 com o vírus pLV-Cas9.
- Semente 1 x 105 células MS-1 por poço em uma placa de 6 poços por 24 horas antes da infecção por lentivírus.
- Descongele as alíquotas congeladas do vírus pLV-Cas9 em um banho de água de 37 °C.
- Misture 1 mL de vírus médio com 1 mL de médio DMEM fresco contendo FBS e penicilina/estreptomicina. Adicione polibreno ao médio (concentração final como 10 μg/mL) para aumentar a eficiência da infecção.
- Retire o meio da placa de 6 poços e substitua-o pelo vírus/polibreno misturar meio e cultivar as células em uma incubadora por 24 h. 24 h pós-infecção, substitua o meio por meio fresco e cultue as células por mais 24 h.
NOTA: Mantenha sempre um grupo de controle não infectado. - Aspire o meio do grupo infectado e do grupo controle e substitua-o pelo meio DMEM por 4 μg/mL blasticidina.
- Retorne a placa para uma incubadora de 37 °C e cultue as células durante 1 semana. Células não infectadas morrerão devido ao efeito de blasticidina. Divida as células sobreviventes quando atingirem 80% de confluência celular e continuem com a seleção de blasticidina.
- Confirme a expressão de Cas9 em células MS-1 por manchas ocidentais usando um anticorpo contra Cas9 (peso molecular de Cas9 é de aproximadamente 160 kDa).
- Infecte separadamente células pLV-Cas9 MS-1 com dois lentivírus gRNA independentes.
- Sementes 1 x 105 pLV-Cas9 MS-1 células por poço em uma placa de 6 poços por 24 horas antes da infecção.
- Siga o mesmo protocolo descrito no 4.2 para infectar separadamente células com dois lentivírus de gRNA.
- Após 24 h de infecção com o vírus gRNA, refresque o meio e a cultura das células por mais 24 h.
- Substitua o meio por DMEM por 1 μg/mL de puramicina. Retorne a placa para uma incubadora de 37 °C e cultue as células durante 1 semana. Certifique-se de que as células não infectadas estão completamente mortas. Divida as células quando atingirem 80% de confluência e continuem a seleção de mosmicina.
- Confirme o knock-out de Snail em células MS-1 por manchas ocidentais usando um anticorpo contra Caracol (peso molecular de Caracol é de aproximadamente 35 kDa).
Resultados
TGF-β2 induz EndMT e estimula expressão de caracol em células endoteliais MS-1
TGF-β é uma das citocinas com maior potencial para induzir EndMT. Depois de tratar as células MS-1 com TGF-β2 (1 ng/mL) por 3 dias, as células Endoteliais MS-1 perdem sua estrutura semelhante a paralelepípedos e se diferenciam em células mesenquimais em forma de fuso(Figura 1A)15. Para verificar melhor o papel do TGF-β2 na indução de alterações fenotípicas celulares, pré-tratamos as células com a pequena molécula activin receptor-like quinase (ALK)4/ALK5/ALK7 inibidor SB431542 antes da estimulação TGF-β219. SB431542 completamente revogada alterações de morfologia celular induzidas por TGF-β2(Figura 1A). O processo EndMT induzido pelo TGF-β2 foi ainda mais investigado pelo estudo de mudanças na expressão de marcadores relacionados ao EndMT. Como mostrado na Figura 1B,a proteína endotelial Pecam-1 foi fortemente diminuída após a estimulação TGF-β2, enquanto o fator mesenquimal Sm22α foi profundamente regulado pelo TGF-β215. Esses dados são consistentes com a noção de que o TGF-β2 desencadeou o EndMT em células MS-1. Em seguida, investigamos os efeitos do TGF-β2 na expressão de Caracol e Lesma. Como mostrado na Figura 1C,o caracol foi marcadamente regulado pelo TGF-β2, enquanto a expressão de lesma não foi influenciada pelo TGF-β2 nas células MS-115. A quantificação da expressão de caracol a partir de três experimentos independentes é mostrada na Figura 1D.
Esgotamento do caracol por CRISPR/Cas9 em células endoteliais MS-1
Como o Caracol foi induzido pelo TGF-β2 e provavelmente envolvido no EndMT mediado pelo TGF-β2, realizamos a edição de genes CRISPR/Cas9 para esgotar geneticamente a expressão do caracol em células MS-1. Nós imaginamos que o esgotamento do Caracol seria suficiente para inibir o EndMT induzido pelo TGF-β2. Como mostrado na Figura 2A,geramos células de nocaute de caracol em duas etapas. Em primeiro lugar, Cas9 foi expresso em êxtase infectando células MS-1 com um Cas9 expressando lentivírus. Como existe uma fita de resistência blasticidina na construção pLV-Cas9, verificamos a expressão de Cas9 pela análise de manchas ocidentais em células resistentes a blasticidina(Figura 2D). Posteriormente, introduzimos sgRNAs que especificamente visavam Caracol para interromper sua expressão proteica. Este procedimento também foi realizado por infecção com partículas lentiviras que carregam a construção AA19 pLKO.1-Snail-sgRNA, que inclui um de expressão de puromicina. As células que expressam Cas9 foram novamente infectadas com gRNA contendo lentivírus e ainda selecionadas com puramicina. Dois oligos sgRNA complementares direcionados ao caracol murino foram projetados com uma atividade baixa prevista fora do alvo(Figura 2B,C). Depois de introduzir duas sgRNAs de caracol independentes em Cas9 expressando células MS-1, a expressão proteica de caracol foi revogada(Figura 2D)15.
Deficiência de Caracol inibe EndMT induzido por TGF-β2 em células MS-1
Para demonstrar a função de Caracol no EndMT mediado por TGF-β2, realizamos um ensaio EndMT em células esgotadas por caracol e comparamos com as células MS-1 parentais. Como mostrado na Figura 3A,o nocaute de Caracol foi suficiente para inibir a morfologia celular semelhante ao fibroblasto impulsionada por TGF-β2 nas células MS-115. Além disso, o declínio mediado pelo TGF-β2 no Pecam-1 e o aprimoramento do Sm22α foram completamente bloqueados em células MS-1 esgotadas por Caracol. Em resumo, demonstramos que o Caracol é fundamental para o EndMT mediado por TGF-β2 em células MS-1(Figura 3B)15.
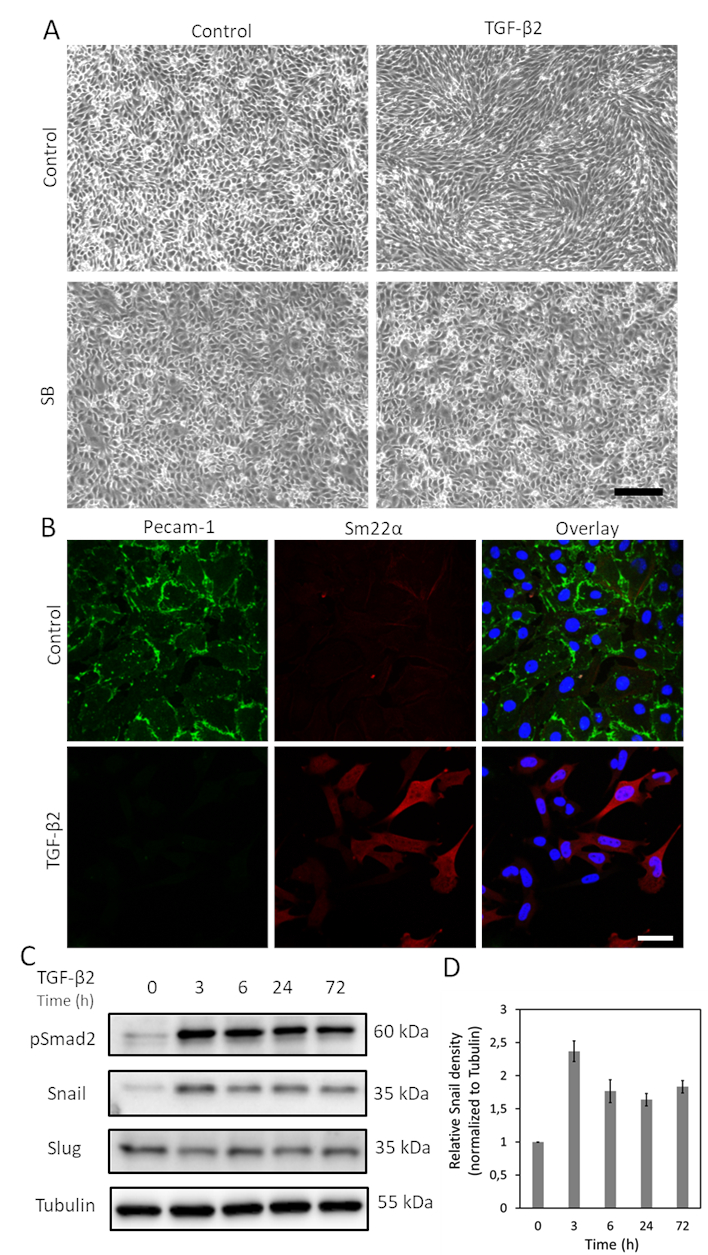
Figura 1. TGF-β2 induz a expressão EndMT e Snail em células MS-1. A. Efeitos do TGF-β2 e/ou TGF-β inibidor de quinase tipo I SB-431542 na morfologia celular. Imagens de Brightfield de células MS-1 após o tratamento com TGF-β2 (1 ng/mL) e/ou SB-431542 (SB, 5 μM, administradas 30 min antes do TGF-β2) por 2 dias. A barra de escala representa 200 μm. B. A coloração da imunofluorescência de Pecam-1 (verde) e Sm22α (vermelho) em células MS-1 cultivadas em meio contendo TGF-β2 (1 ng/mL) por 3 dias. Os núcleos são visualizados em azul (DAPI). Barra de escala: 50 μm. C. Mancha ocidental com lise celular inteira de células TGF-β2 estimuladas MS-1. A expressão de Caracol, mas não Lesma, foi reforçada pela estimulação TGF-β2, como relatado anteriormente em Ma et al15. D. Quantificação da expressão de Caracol integrando os resultados de três experimentos independentes de manchas ocidentais. Clique aqui para ver uma versão maior desta figura.
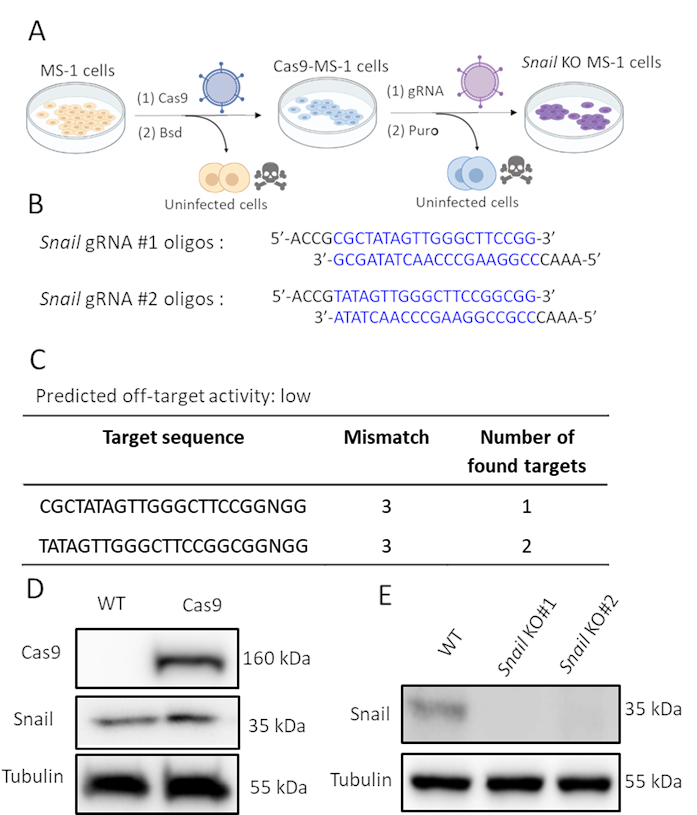
Figura 2. Esgotamento do caracol pela edição de genes CRISPR-Cas9. A. Esquema que retrata como gerar células de nocaute de Caracol. Bsd: Blasticidin. Pura: Puromicina. B. Oligonucleotídeos de dois sgRNAs independentes direcionados a Caracol usando CHOPCHOP (http://chopchop.cbu.uib.no/) e Cas-OFFinder (http://www.rgenome.net/cas-offinder/). C. A atividade fora do alvo prevista dos dois gRNAs para Caracol usando Cas-OFFinder (http://www.rgenome.net/cas-offinder/). D. Expressão cas9 e caracol em tipo selvagem (WT) e Cas9-overexpressed MS-1 medido pela análise de manchas ocidentais. E. Nocaute de Caracol com dois gRNAs independentes em células MS-1 medida pela análise de manchas ocidentais. Relatamos resultados semelhantes em Ma et al15. Clique aqui para ver uma versão maior desta figura.
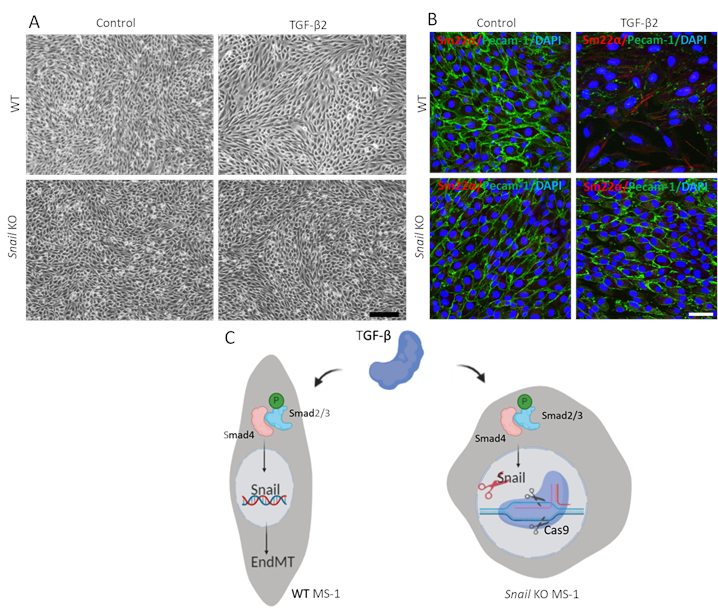
Figura 3. O esgotamento genético do Caracol inibe o EndMT induzido pelo TGF-β2 em células MS-1. A. Imagens de Brightfield de células MS-1 após o tratamento com células TGF-β2 (0,1 ng/mL) por 3 dias em células de tipo selvagem (WT, painel superior) e Caracol nocauteados (painel inferior). A barra de escala representa 200 μm. B. A coloração imunofluorescente para Pecam-1 (verde), Sm22α (vermelho) e núcleos (azul) de células MS-1 cultivadas em meio contendo TGF-β2 (1 ng/mL) por 3 dias. Esgotamento do Caracol revogado TGF-β2-induzido diminuição de Pecam-1 e aumento da expressão Sm22α. A barra de escala representa 50 μm. C. Representação esquemática do efeito do nocaute de Caracol no EndMT induzido por TGF-β em células MS-1. TGF-β estimula a expressão de Caracol através da via Smad por fosforilando Smad2/3 e outras unidades EndMT. Nocauteando o Snail usando o endMT mediado por β genes CRISPR/Cas9. Clique aqui para ver uma versão maior desta figura.
Discussão
Entender o mecanismo do EndMT é fundamental para modular esse processo e direcionar doenças relacionadas ao EndMT. Aqui, descrevemos métodos para realizar um ensaio EndMT induzido por TGF-β e interrogar o papel do Caracol EndMT-TF em EndMT-β-acionado, realizando o esgotamento genético estável mediado por CRISPR/Cas9 das células. O esgotamento do Caracol usando a abordagem CRISPR/Cas9 revogou com sucesso o EndMT conduzido pelo TGF-β2 em células MS-1(Figura 3C). Para estudar os efeitos de quaisquer citocinas, como tgf-β, no EndMT, os CE foram expostos a citocinas e, em seguida, a ocorrência de EndMT foi avaliada de acordo com alterações morfológicas e alterações de expressão de marcadores endoteliais e mesenquimais nas células. TGF-β2 induziu fortemente endMT em células MS-1 acompanhadas por um forte aumento na expressão do fator de transcrição Caracol. Os EndMT-TFs induzidos por TGF-β podem diferir de acordo com a espécie ou tipo de célula endotelial específica do tecido. Por exemplo, observamos que o caracol, mas não o lesma, foi significativamente regulado pelo TGF-β em células MS-1, enquanto nas células endoteliais da veia umbilical humana (HUVECs), tanto Caracol quanto Lesma são aumentadas após a exposição ao TGF-β20.
Avaliamos a extensão do processo EndMT de duas maneiras examinando as alterações da morfologia celular e, em seguida, investigando alterações na expressão de marcadores relacionados ao EndMT. Após a exposição tgf-β por 3 dias, as células foram submetidas ao EndMT com variações morfológicas consistentes e alterações na expressão de marcadores relacionados ao EndMT. Além da coloração da imunofluorescência que realizamos aqui, as variações de marcadores também podem ser monitoradas por manchas ocidentais no nível de expressão proteica ou por qRT-PCR (PCr de Transcrição Reversa Quantitativa em Tempo Real) nos níveis genéticos21. Além desses dois métodos de economia de custos que mostramos neste protocolo, existem outros métodos para examinar o EndMT. Por exemplo, a realização de análises de transcriptome (por sequenciamento de RNA ou qPCR) para comparar os níveis de expressão de genes relacionados à endotelial e mesenquimal entre células tratadas e de controle pode avaliar com precisão o EndMT22,23. Além disso, o EndMT envolve frequentemente a perda estável da função da barreira, que pode ser avaliada pela espectroscopia de impedância24. Além disso, podem ser examinadas provas adicionais da aquisição de propriedades semelhantes a células-tronco por células derivadas do EndMT. Por exemplo, em condições específicas de cultura, as células mesenquimais endMT podem ser ainda mais diferenciadas em osteoblastos, condrócitos, adipócitos ou (mio)fibroblastos. Portanto, análises adicionais para confirmar a diferenciação em diferentes tipos de células pertencentes à linhagem de mesoderme (ou seja, expressão genética e coloração de matriz) são úteis para demonstrar a natureza multipotente das células derivadas do EndMT. Finalmente, os métodos de avaliação endMT não se limitam a estudos in vitro, mas podem ser extrapolados para investigar a relação entre EndMT e algumas doenças in vivo ou em órgãos ex vivo. Nesse sentido, o uso de estratégias de rastreamento de linhagem específicas da endotelia é amplamente estendido à pesquisa relacionada ao EndMT25.
Para investigar o papel de Caracol durante o EndMT, neste estudo a edição de genes CRISPR/Cas9 foi usada para eliminar esse gene. Os dados mostraram que o TGF-β2 falhou em mediar o EndMT em células MS-1 deficientes de Caracol. Esta observação demonstrou que o caracol é essencial para o EndMT induzido pelo TGF-β2 em células MS-1. Usamos um de expressão sgRNA independente com u6 para introduzir sgRNAs específicos para Cas9 para atingir Snail. Além deste método, Ran et al.26 descreveram outra estratégia para clonar a sequência de oligos sgRNA no andaime Cas9 para gerar uma construção contendo cas9 e gRNAs. Abordagens novas emergentes permitem que o CRISPR/Cas incorpore funções adicionais. Por exemplo, nocautes duplos ou triplos podem ser alcançados fornecendo mais sgRNAs em células expressando Cas927. A proteína Cas13 projetada visa e digere moléculas de RNA sem interromper o DNA endógeno28. Além de eliminar genes com CRISPR/Cas, rnAs de grampo de cabelo curto (shRNAs) podem ser usados como alternativas para derrubar a expressão genética direcionada29. Para todos os métodos de edição de genes CRISPR/Cas, o decote fora do alvo deve ser sempre levado em consideração. Além disso, pequenas RNAs interferentes (siRNAs) silenciam transitoriamente a expressão genética e a concentração de siRNA é diluída com a divisãocelular 30. Ambos os métodos suprimem parcialmente a expressão genética direcionada. Em contraste, a expressão genética ectópica também é usada para verificar a função genética durante o EndMT/EMT31. Essa abordagem pode determinar se a regulação de um gene é suficiente para obter uma resposta EndMT. Portanto, atualmente existem uma infinidade de estratégias técnicas que podem ser usadas para identificar e verificar potenciais reguladores da EndMT. Além disso, a análise transcriômica pode ser uma boa opção na identificação e análise abrangente dos reguladores relacionados ao EndMT. Recomendamos o uso de diferentes abordagens complementares para investigar a modulação do EndMT.
Em resumo, introduzimos um fluxo de trabalho para identificar fatores que podem desempenhar funções funcionais durante o EndMT induzido pelo TGF-β. Este método também pode ser usado para estudar se outros estímulos (ou seja, citocinas, fatores de crescimento, estímulos mecânicos, interações célula-células) podem modular o EndMT, e a interação do TGF-β com outros estímulos. Além disso, destacamos uma abordagem usando a edição de genes CRIPSR/Cas para elucidar se um determinado gene é necessário para o EndMT induzido por β TGF. Para ilustrar essa metodologia, utilizamos o forte indutor endMT TGF-β2 em células MS-1, mas os protocolos podem ser adaptados a outros citocinas e outros tipos de células. Esperamos que este protocolo detalhado descrito sirva de trampolim para futuros estudos relacionados ao EndMT.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
A pesquisa foi apoiada pela CGC.NL e pela Iniciativa de Pesquisa Cardio Vascular dos Países Baixos: a Dutch Heart Foundation, a Federação Holandesa de Centros Médicos Universitários, a Organização Holandesa para Pesquisa e Desenvolvimento em Saúde e a Royal Netherlands Academy of Sciences Grant concedida ao Phaedra-Impact (http://www.phaedraresearch.nl). JM é apoiado pelo Conselho de Bolsas chinesa. O GSD é apoiado por uma subvenção do Trampolim da AFM-Teleton [22379], fop Italia e uma subvenção do La Fundació La Marató de TV3 (#202038).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | Pall Corporation, USA | 4614 | |
| 10× T4 DNA ligase buffer | Thermofisher Scientific, USA | B69 | |
| 12 mm round glass slice | Knittel Glass,Germany | VD10012Y1A.01 | |
| 20 mL syringe | BD Eclipse, USA | 300629 | |
| 4′,6-diamidino-2-phenylindole (DAPI) | Vector Laboratories, USA | H-1200 | VECTASHIELD Antifade Mounting Media |
| AA19_PLKO vector | Dr. M Gonçalves, Leiden University Medical Center, Netherlands | Gift | |
| Ampicillin | Serva Electrophoresis, USA | 1339903 | |
| Anti-Mouse IgG | GE Healthcare, USA | NA931 | |
| Anti-Rabbit IgG | Cell signaling, USA | 7074 | |
| Agarose | Roche, Switzerland | 11388991001 | |
| Blasticidin | Invitrogen, USA | R21001 | |
| Buffer O (10X) | Thermofisher Scientific, USA | BO5 | |
| BveI (Bspm1) | Thermofisher Scientific, USA | ER 1741 | |
| Confocal microscope | Leica Microsystems, Germany | SP8 | |
| DMEM | Thermo Fisher Scientific, USA | 11965092 | |
| Donkey anti-rat Alexa 488 | Invitrogen, USA | A21208 | |
| FBS | Thermo Fisher Scientific, USA | 16000044 | |
| Formaldehyde | Thermo Fisher Scientific, USA | 28908 | |
| Inverted microscope | Leica Microsystems, Germany | DMi8 | |
| Goat anti-rabbit Alexa 594 | Invitrogen,USA | A11012 | |
| LabNed Plasmid kit | LabNed, USA | LN2400004 | |
| MS-1 cell line | American Type Culture Collection (ATCC) | ||
| Nail polish | HEMA, Netherlands | Transparent | |
| Pecam-1 antibody | Becton Dickinson,USA | 553370 | |
| PEI | Polysciences, USA | 23966-1 | |
| PLV-Cas9 plasmid | Sigma-Aldrich, USA | Cas9BST-1EA | |
| Puromycin | Sigma-Aldrich, USA | P9620 | |
| QIAquick gel extraction kit | Qiagen, Germany | 28706 | |
| Sm22a antibody | Abcam, UK | ab14106 | |
| Snail | Cell signaling, USA | 3879 | |
| T4 DNA ligase | Thermofisher Scientific, USA | EL 0014 | |
| TC20 automated Cell Counter | Bio-Rad, USA | 1450102 | |
| Human TGF-β2 | Joachim Nickel, University of Wurzburg | Gift | Other commercial recommendation: 302-B2, R&D systems |
| Triton X-100 | Merck, USA | 1086031000 | |
| SB431542 | Tocris Bioscience, UK | 1614 | |
| Tris | Roche, Switzerland | 11814273001 |
Referências
- Ma, J., Sanchez-Duffhues, G., Goumans, M. -. J., Ten Dijke, P. TGF-β-induced endothelial to mesenchymal transition in disease and tissue engineering. Frontiers in Cell and Developmental Biology. 8, (2020).
- Piera-Velazquez, S., Jimenez, S. A. Endothelial to mesenchymal transition: role in physiology and in the pathogenesis of human diseases. Physiological Reviews. 99 (2), 1281-1324 (2019).
- Sánchez-Duffhues, G., de Vinuesa, G. A., ten Dijke, P. Endothelial-to-mesenchymal transition in cardiovascular diseases: developmental signaling pathways gone awry. Developmental Dynamics. 247 (3), 492-508 (2018).
- Evrard, S. M., et al. Endothelial to mesenchymal transition is common in atherosclerotic lesions and is associated with plaque instability. Nature Communications. 7 (1), 1-16 (2016).
- Qiao, L., et al. Endothelial fate mapping in mice with pulmonary hypertension. Circulation. 129 (6), 692-703 (2014).
- Zeisberg, E. M., et al. Endothelial-to-mesenchymal transition contributes to cardiac fibrosis. Nature Medicine. 13 (8), 952-961 (2007).
- Clere, N., Renault, S., Corre, I. Endothelial-to-mesenchymal transition in cancer. Frontiers in Cell and Developmental Biology. 8, (2020).
- Saito, A. EMT and EndMT: regulated in similar ways. The Journal of Biochemistry. 153 (6), 493-495 (2013).
- Bolós, V., et al. The transcription factor Slug represses E-cadherin expression and induces epithelial to mesenchymal transitions: a comparison with Snail and E47 repressors. Journal of Cell Science. 116 (3), 499-511 (2003).
- Aird, W. C. Endothelial cell heterogeneity. Cold Spring Harbor Perspectives in Medicine. 2 (1), 006429 (2012).
- Yang, J., et al. Guidelines and definitions for research on epithelial-mesenchymal transition. Nature Reviews Molecular Cell Biology. , 1-12 (2020).
- Doudna, J. A., Charpentier, E. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), (2014).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Arbiser, J. L., et al. Oncogenic H-ras stimulates tumor angiogenesis by two distinct pathways. Proceedings of the National Academy of Sciences USA. 94 (3), 861-866 (1997).
- MA, J., et al. TGF-?-induced endothelial to mesenchymal transition results from a balance between SNAIL and ID factors. Frontiers in Cell and Developmental Biology. 182 (9), (2021).
- Chen, X., et al. Probing the impact of chromatin conformation on genome editing tools. Nucleic Acids Research. 44 (13), 6482-6492 (2016).
- Hunter, F. W., et al. Functional CRISPR and shRNA screens identify involvement of mitochondrial electron transport in the activation of evofosfamide. Molecular Pharmacology. 95 (6), 638-651 (2019).
- Mao, Y., et al. Lentiviral vectors mediate long-term and high efficiency transgene expression in HEK 293T cells. International Journal of Medical Sciences. 12 (5), 407 (2015).
- Liu, S., et al. Deubiquitinase activity profiling identifies UCHL1 as a candidate oncoprotein that promotes TGFβ-induced breast cancer metastasis. Clinical Cancer Research. 26 (6), 1460-1473 (2020).
- Medici, D., et al. Conversion of vascular endothelial cells into multipotent stem-like cells. Nature Medicine. 16 (12), 1400 (2010).
- Kokudo, T., et al. Snail is required for TGFβ-induced endothelial-mesenchymal transition of embryonic stem cell-derived endothelial cells. Journal of Cell Science. 121 (20), 3317-3324 (2008).
- Dong, J., et al. Single-cell RNA-seq analysis unveils a prevalent epithelial/mesenchymal hybrid state during mouse organogenesis. Genome Biology. 19 (1), 1-20 (2018).
- Oatley, M., et al. Single-cell transcriptomics identifies CD44 as a marker and regulator of endothelial to haematopoietic transition. Nature communications. 11 (1), 1-18 (2020).
- Halaidych, V., et al. Inflammatory responses and barrier function of endothelial cells derived from human induced pluripotent stem cells. Stem Cell Reports. 10 (5), 1642-1656 (2018).
- Li, Y., Lui, K. O., Zhou, B. Reassessing endothelial-to-mesenchymal transition in cardiovascular diseases. Nature Reviews Cardiology. 15 (8), 445-456 (2018).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Grav, L. M., et al. One-step generation of triple knockout CHO cell lines using CRISPR/Cas9 and fluorescent enrichment. Biotechnology Journal. 10 (9), 1446-1456 (2015).
- Abudayyeh, O. O., et al. RNA targeting with CRISPR-Cas13. Nature. 550 (7675), 280-284 (2017).
- Paddison, P. J., et al. Cloning of short hairpin RNAs for gene knockdown in mammalian cells. Nature Methods. 1 (2), 163-167 (2004).
- Reynolds, A., et al. Rational siRNA design for RNA interference. Nature Biotechnology. 22 (3), 326-330 (2004).
- Lourenço, A. R., et al. C/EBPɑ is crucial determinant of epithelial maintenance by preventing epithelial-to-mesenchymal transition. Nature Communications. 11 (1), 1-18 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados