Method Article
TGF-β endoteliale mediato a transizione mesenchimale (EndMT) e la valutazione funzionale degli effettori EndMT utilizzando crispr/cas9 gene editing
In questo articolo
Riepilogo
Descriviamo metodi per studiare l'EndMT indotto da TGF-β2 nelle cellule endoteliali osservando i cambiamenti della morfologia cellulare ed esaminando i cambiamenti marcatori correlati all'endmt usando la colorazione dell'immunofluorescenza. L'editing genico CRISPR/Cas9 è stato descritto e utilizzato per esaurire il gene che codifica per Snail per indagare il suo ruolo nell'EndMT indotto da TGF-β2.
Abstract
In risposta a specifici segnali esterni e all'attivazione di alcuni fattori di trascrizione, le cellule endoteliali possono differenziarsi in un fenotipo mesenchimale, un processo che viene definito endoteliale alla transizione mesenchimale (EndMT). I risultati emergenti hanno suggerito che endmt è causalmente collegato a molteplici malattie umane, come la fibrosi e il cancro. Inoltre, le cellule mesenchimali derivate dall'endoteliale possono essere applicate nelle procedure di rigenerazione dei tessuti, in quanto possono essere ulteriormente differenziate in vari tipi di cellule (ad esempio, osteoblasti e condrociti). Pertanto, la manipolazione selettiva di EndMT può avere un potenziale clinico. Come la transizione epiteliale-mesenchimale (EMT), EndMT può essere fortemente indotta dalla citochina secreta che trasforma il fattore di crescita-beta (TGF-β), che stimola l'espressione dei cosiddetti fattori di trascrizione EndMT (EndMT-TFs), tra cui Lumaca e Lumaca. Questi EndMT-TF quindi denotino rispettivamente i livelli di proteine mesenchimali ed endoteliali. Qui descriviamo metodi per indagare endmt indotto da TGF-β in vitro, incluso un protocollo per studiare il ruolo di particolari TF nell'EndMT indotto da TGF-β. Utilizzando queste tecniche, forniamo la prova che il TGF-β2 stimola l'EndMT nelle cellule endoteliali microvascolari pancreatiche murine (cellule MS-1), e che l'esaurimento genetico della lumaca usando ripetizioni palindromiche brevi raggruppate regolarmente interspaziate (CRISPR)/crispr-associated protein 9 (Cas9) mediato editing genico, abroga questo fenomeno. Questo approccio può servire come modello per interrogare potenziali modulatori della biologia endoteliale, e può essere utilizzato per eseguire schermi genetici o farmacologici al fine di identificare nuovi regolatori di EndMT, con potenziale applicazione nelle malattie umane.
Introduzione
La transizione endoteliale a mesenchimale (EndMT) è un fenomeno biologico multistep e dinamico che è stato collegato a diversi processi fisiologici epatologici 1,2. Al fineMT le cellule endoteliali perdono gradualmente i loro tratti endoteliali, acquisendo al contempo proprietà mesenchimali3; così, le cellule endoteliali strettamente compattate e ben organizzate si differenziano in cellule mesenchimali allungate. I cambiamenti morfologici nell'EndMT coincidono con le alterazioni nell'espressione di alcuni geni e proteine. In generale, l'espressione di proteine che mantengono caratteristiche endoteliali, tra cui endoteliale vascolare (VE)-cadherina, molecola di adesione piastrinica/CE-1 (CD31/Pecam-1) diminuisce. Allo stesso tempo, si accumulano proteine legate alle funzioni mesenchimale, come l'actina muscolare α liscia (α-Sma) e la proteina muscolare liscia 22α (Sm22α). Risultati emergenti hanno dimostrato che l'EndMT postnatale contribuisce allo sviluppo di malattie umane, come il cancro, la fibrosi cardiaca, l'ipertensione arteriosa polmonare (IAP), l'aterosclerosi (AS), la fibrosi degli organi,ecc. 2,4,5,6,7. Una comprensione più profonda dei meccanismi sottostanti di EndMT e di come dirigere il processo EndMT fornirà nuovi metodi terapeutici per le malattie correlate all'EndMT e la medicina rigenerativa.
TGF-β è uno dei principali induttori endmt, e altri fattori coinvolti noti includono Wnt / β-catenina, Notch e alcune citochine infiammatorie1. Poiché il contesto cellulare è fondamentale per le risposte innescate da TGF-β, l'interazione di TGF-β con altri segnali di promozione EndMT è rilevante per TGF-β per ottenere una risposta EndMT. All'attivazione dei recettori TGF-β di superficie cellulare di tipo I e serina/treonina chinasi di tipo II, viene attivata la via Smad canonica intracellulare. TGF-β recettore mediato fosforilato Smad2/3 forma complessi eteromerici con Smad4 che si traslocano nel nucleo, dove upregolano l'espressione dei fattori di trascrizione correlati all'EndMT. Simile alla transizione epiteliale-mesenchimale (EMT), fattori di trascrizione come Lumaca, Lumaca, Torsione, Zeb1 e Zeb2 sono indotti dalla segnalazione TGF-β e contribuiscono alla riprogrammazione genica in EndMT8.
La lumaca è stata spesso identificata come un fattore chiave in EndMT. La lumaca si lega al promotore dei geni che codificano le proteine di adesione cellulare-cellula e sopprime la loro trascrizione, che è controbilanciata dal miglioramento dell'espressione delle proteine mesenchimali9. Le cellule endoteliali comprendono una popolazione molto eterogenea e l'influenza relativa di diversi stimoli extracellulari sull'EndMT può differire tra i contesti cellulari endoteliali o i tipi dicellule 10. A causa delle sue somiglianze con EMT, alcune metodologie sono utili per indagare entrambi i meccanismi EMT e EndMT8. A questo proposito, l'Associazione internazionale EMT (TEMTIA) sottolinea con forza la necessità di tecniche complementari per dimostrare in ultima analisi la presenza di EMT/EndMT11.
Qui descriviamo un metodo per monitorare e visualizzare il processo EndMT β indotto da TGF. La colorazione dell'immunofluorescenza fornisce le informazioni di base sui cambiamenti di espressione nelle proteine/marcatori mirati, che vengono utilizzati come indicatori del verificarsi del processo EndMT. Inoltre, la colorazione dell'immunofluorescenza può visualizzare la localizzazione di proteine / marcatori e la morfologia cellulare. Per studiare l'attività potenziale di specifici TF (o altri regolatori a monte o a valle) coinvolti nell'EndMT mediato TGF-β, descriviamo un protocollo utilizzando brevi ripetizioni palindromiche raggruppate regolarmente interspaziate (CRISPR) /CRISPR-associated protein 9 (Cas9) editing genico per esaurire geni specifici dalle cellule, usando la Lumaca TF come esempio. Cas9 è una doppia endonucleasi del DNA guidata dall'RNA che riconosce e scicca sequenze complementari alle sequenze CRISPR nei batteri12. Il sistema CRISPR/Cas9 è attualmente ampiamente utilizzato perché facilita l'ingegneria genetica in vitro e in vivo13. Diretto da un singolo RNA guida (sgRNA), Cas9 espresso ectopicamente genera una rottura a doppio filamento in una sequenza di targeting preselezionata in uno specifico locus genico. L'unione finale non omologa (NHEJ) avviene per riparare le rotture di filamenti indotte da Cas9, attraverso inserimenti o deezioni nucleotidiche casuali, portando così all'interruzione e all'inattivazione del gene mirato. Descriviamo in dettaglio i metodi per progettare sgRNA selettivi e generare vettori compatibili con lentivirali contenenti gli sgRNA progettati. Di conseguenza, le cellule endoteliali stabili e impoverite da geni possono essere generate in modo efficiente e affidabile.
In questo studio, abbiamo usato cellule endoteliali microvascolari pancreatiche murine (MS-1)14 come sistema modello per esaminare il processo EndMT indotto da TGF-β2. Il nostro studio precedente ha dimostrato che la lumaca è il principale fattore di trascrizione aumentato da TGF-β2, con il quale EndMT è indotto nelle cellule MS-115. Dopo l'editing genico CRISPR/Cas9 per abrogare l'espressione di Lumaca nelle cellule MS-1, il TGF-β2 non è riuscito a mediare EndMT. Questo flusso di lavoro può essere applicato per studiare altri (sospetti) geni correlati all'EndMT.
Protocollo
1. Induzione di EndMT da parte di TGF-β2
- Cellule MS-1 nel mezzo aquila modificato (DMEM) di Dulbecco contenente il 10% di siero bovino fetale (FBS) e 100 U/mL penicillina/streptomicina in un'incubatrice (5% CO2, 37 °C). Rivestire tutti i piatti/piatti di coltura con gelatina 0,1% con gelatina per 10 minuti prima dell'uso.
- Lavare delicatamente le cellule MS-1 con 1x soluzione salina tamponata dal fosfato (PBS), aggiungere 2 mL di soluzione di tripside-EDTA (0,25% tripina e 0,02% EDTA) a un piatto di 10 cm e incubare per 2 minuti a 37 °C per staccarle. Successivamente, aggiungere 5 mL di mezzo di coltura completo per spegnere la reazione.
- Trasferire la sospensione cellulare su un tubo da 15 ml e centrifugare a 200 x g per 3 minuti a temperatura ambiente.
- Scartare il supernatante e rimescolare le cellule in 4 mL di mezzo fresco contenente FBS e penicillina/streptomicina. Contare le celle utilizzando un contatore automatico di celle.
- Seme 1 x 103 cellule per cm2 per ulteriore coltura. Ad esempio, seme 9,5 x 103 celle/pozzo per piastre a 6 pozzi o 1,9 x 103 celle/pozzo per piastre a 24 pozzi.
- Incubare le cellule durante la notte per consentire loro di aderire e recuperare, quindi stimolare le cellule MS-1 con TGF-β2 per 3 giorni. Aggiungere l'inibitore della β del recettore TGF-chinasi SB431542 (5 μM) 30 min prima della stimolazione TGF-β2. Trattare altre cellule con il veicolo (DMSO).
NOTA: Sciogliere il TGF-β2 in 4 mM HCl contenente lo 0,1% di albumina di siero bovino umano (BSA). Aggiungere la stessa quantità di buffer di ligando senza TGF-β2 al gruppo di controllo. La concentrazione di TGF-β2 può essere adattata a 0,1-1 ng/mL per test specifici. Cfr. indicazioni nelle cifre corrispondenti. - Dopo 3 giorni, esaminare la morfologia cellulare con l'imaging a campo luminoso (con un microscopio invertito) ed eseguire la colorazione dell'immunofluorescenza (vedere il passaggio 2) per valutare i cambiamenti marcatori correlati alla endmt. Eseguire almeno tre esperimenti indipendenti per ottenere triplicati biologici.
2. Colorazione dell'immunofluorescenza
- Tripinare le cellule MS-1 coltivate (fase 1.2) e quindi reseed 1,9 x 103 celle su un vetro di copertura rotonda 12 mm rivestito con gelatina 0,1% w/v posto sul fondo di una piastra da 24 pozze.
- Dopo aver coltivato le cellule durante la notte, aggiungere TGF-β2 (concentrazione finale 1 ng/mL) alle cellule per 3 giorni. Utilizzare il supporto contenente il buffer ligando come controllo negativo.
- Eseguire la colorazione Pecam-1 e Sm22α.
- Dopo aver stimolato le cellule con TGF-β2 (o Controllo) per 3 giorni, rimuovere il mezzo e lavare le cellule con 1x PBS.
- Aggiungere 300 μL di formaldeide al 4% ad ogni pozzo e incubare per 10 minuti a temperatura ambiente per fissare le cellule. Lavare 3 volte con 1 PBS dopo l'incubazione.
- Aggiungere 300 μL dello 0,1% di Tritone X-100 in 1x PBS ad ogni pozzo e incubare per 10 minuti a temperatura ambiente per permeabilizzare le cellule. Dopo l'incubazione, rimuovere la soluzione Triton X-100 e lavare le cellule 3 volte con 1x PBS.
- Bloccare le cellule con albumina siere bovina al 3% (BSA) in 1x PBS per 45 minuti a temperatura ambiente.
- Diluire gli anticorpi primari Pecam-1 e Sm22α che riconoscono le proteine murine 1:500 con 1x PBS. Quindi incubare le cellule fisse con anticorpi primari per 45 minuti a temperatura ambiente.
- Dopo aver lavato 3 volte con 1x PBS, incubare le cellule con anticorpi secondari diluiti 1000x, tra cui l'anti-ratto dell'asino Alexa 488 e l'anti-coniglio di capra Alexa 594, per 45 minuti a temperatura ambiente.
NOTA: Proteggere i campioni dalla luce durante la colorazione. - Dopo aver risciacquato 3x con 1x PBS, posizionare il vetro di copertura seminato con cellule a faccia in giù su una goccia di mezzo di montaggio contenente 4',6-diamidino-2-fenilindolo (DAPI) su uno scivolo per macchiare i nuclei.
- Fissare la periferia del vetro di copertura con smalto trasparente e conservarlo a 4 °C.
- Acquisire immagini rappresentative con un microscopio confocale. Impostare le lunghezze d'onda laser a 405 nm, 488 nm e 552 nm per rilevare RISPETTIVAMENTE DAPI, Pecam-1 e Sm22α. Per ogni canale, tutte le immagini sono state scattate con le stesse impostazioni e il tempo di esposizione. Eseguire almeno tre esperimenti indipendenti per ottenere triplicati biologici.
3. Eliminare la lumaca utilizzando l'editing CRISPR / Cas9
- Progetta due sgRNA indipendenti rivolti alla lumaca murina.
- Progetta sgRNA utilizzando gli strumenti online CHOPCHOP (https://chopchop.cbu.uib.no/) e Cas-OFFinder (http://www.rgenome.net/cas-offinder/) in base al nome e alle specie del gene mirato.
- Prevedere l'attività off-target degli sgRNA progettati destinati a Snail con due algoritmi indipendenti, tra cui Cas-OFFinder (http://www.rgenome.net/cas-offinder/) e CHOPCHOP (http://chopchop.cbu.uib.no/).
- Scegliere due SgRNA con la bassa off-activity. Progetta due oligo di sgRNA complementari con il sito di taglio BveI. L'oligo di senso inizia con 5 '-ACCG-3' e l'oligo antisenso inizia con 5'-AAAC-3'.
- Ordina che gli oligi siano sintetizzati commercialmente per un ulteriore utilizzo.
- Clonare gli oligo di RNA guida complementari nell'AA19 pLKO.1-puro.U6.sgRNA di BveI. Bve Plasmide vettoriale lentivirale I-stuffer per generareAA19 pLKO.1-Lumaca-sgRNA16.
- Tagliare l'AA19 pLKO.1-puro.U6.sgRNA. Bve Plasmide I-stuffer con l'enzima BveI16. Mescolare 2 μg di AA19 pLKO.1-puro.U6.sgRNA. Bve Plasmide I-stuffer, 5 μL di 10x Buffer O, e 5 μL di enzima BveI e aggiungere acqua sterile per raggiungere un volume totale di 50 μL.
- Vortice e girare brevemente verso il basso il mix di reazione. Incubare la reazione a 37 °C per 1 h.
- Caricare la miscela di reazione su un gel di agarosio dell'1% ed eseguire in 1x Tris-acetato-EDTA (TAE, 50x stock di TAE: 242 g di base Tris sciolta in acqua, 57,1 mL di acido acetico glaciale, 100 mL di soluzione EDTA da 500 mM (pH 8.0) e aggiungere acqua a un totale di 1 L) tampone fino a raggiungere una buona separazione.
- Tagliare il frammento di spina dorsale dal gel, isolarlo con un kit di estrazione del gel secondo il protocollo del produttore (vedi tabella dei materiali)ed eludere la spina dorsale in tampone di eluizione da 40 μL (EB).
- Mescolare 5 μL di oligo di senso 100 pmol/μL e 5 μL di oligo antisenso 100 pmol/μL con 1 μL di 1 M Tris-HCl (pH 8.0) e aggiungere acqua sterile per raggiungere un totale di 100 μL per ricottura degli oligo gRNA. Incubare la miscela per 5 minuti a 100 °C, quindi coprire i tubi con un foglio di alluminio. Successivamente, raffreddare lentamente la soluzione a temperatura ambiente per un ulteriore utilizzo come inserto.
- Per ligare gli oligo gRNA complementari e la spina dorsale digerita BveI, mescolare 1 μL di BveI isolato tagliare AA19 pLKO.1-puro.U6.sgRNA.BveI-stuffer backbone e 2 μL di inserto (diluito 1:300) con 2 μL di 10x T4 DNA ligasi tampone e 1 μL di T4 DNA ligasi, e aggiungere acqua sterile per raggiungere un totale di 20 μL. Ruotare brevemente il tubo e incubarlo per 4 ore a temperatura ambiente per un ulteriore utilizzo.
NOTA: quando si esegue la legatura, assicurarsi che siano inclusi due gruppi di controllo. Per un gruppo di controllo, mescolare 1 μL di spina dorsale isolata, 2 μL di tampone di ligasi del DNA T4 10x e 1 μL di DNA ligasi T4 e aggiungere acqua sterile per raggiungere un totale di 20 μL, ma senza DNA di oligo. Per l'altro gruppo di controllo, mescolare 1 μL di spina dorsale isolata e 2 μL di tampone di ligasi del DNA T4 10x, e aggiungere acqua sterile per raggiungere un totale di 20 μL, ma senza oligo ricotto e ligasi T4. Queste due legazioni di controllo vengono utilizzate per determinare lo sfondo di reazione nella fase di trasformazione successiva e indicare quanto sia efficiente la legatura.
- Trasformare la miscela di reazione in TOP10 E. coli competente.
- Ritirare il TOP10 E. coli competente. cellule dal congelatore -80 °C e scongelarle sul ghiaccio.
- Aggiungere 2 μL di miscela di legatura a 50 μL di cellule competenti e tenere il tubo sul ghiaccio per 30 minuti.
- Shock termico del tubo a 42 °C per 30 s. Metti il tubo sul ghiaccio per 2 minuti.
- Aggiungere 950 μL di brodo di lisogenia fresca (LB) medio alla miscela e agitare vigorosamente a 37 °C per 60 min.
- Spin giù e piastrare le cellule su una piastra LB di resistenza all'ampicillina calda (100 μg/mL). Incubare la piastra a 37 °C durante la notte.
- Verificare il successo dell'inserimento degli oligo gRNA nel plasmide.
- Raccogliere 3-5 colonie sul piatto in 1 mL di ampicillina (100 μg/mL) contenente mezzo LB e agitare durante la notte a 30 °C.
- Isolare il DNA plasmide con un kit plasmide secondo il protocollo del produttore (Table of Materials) e sequenziarlo con il primer promotore U6 5'- GAGGGCCTATTTCCCATGATT -3' per verificare il successo dell'inserimento dell'oligo gRNA.
4. Genera cellule MS-1 knockout lumaca
- Produrre particelle lentivirali che trasportano GRNA a punta di Cas9 o lumaca.
- Cellule HEK 293T di coltura in DMEM contenenti il 10% di siero bovino fetale e 100 U/mL penicillina/streptomicina in piatti da 14,5 cm (o matracci T75) in un'incubatrice (5% CO2, 37 °C).
- Mescolare 9,9 μg di plasmide genico mirato, AA19 pLKO.1-Lumaca-sgRNA o pLV-Cas917 plasmide, insieme ai plasmidi aiutanti 3,5 μg di pCMV-VSVG (codifica della proteina G del virus della stomatite vescicolare, VSV-G), 6,6 μg di elemento rev-responsive plasmid pMDLg-RRE (codifica Gag e Pol), e 5,0 μg di pRSV-REV (codifica Rev) in 500 μL di mezzo privo di siero. Resopend 50 μL di polietilenimina (PEI) (2,5 mg/mL) in 500 μL di mezzo privo di siero. Mescolare delicatamente plasmidi e preparati di PEI pipettando su e giù. Incubare la miscela per 20 minuti a temperatura ambiente.
- Trasfetto le cellule HEK 293T aggiungendo il mezzo di miscela dal passo 4.1.2 all'80% di cellule confluenti in piatti da 14,5 cm (o matracci T75) che contengono mezzo DMEM con FBS al 10% e penicillina/streptomicina U/mL. Le cellule HEK293T vengono utilizzate perché sono facilmente trasfette e generano alti livelli di virus18.
- Trasferire cellule HEK 293T trasfette in una biosicurezza in laboratorio microbiologico e biomedico (BMBL) per coltarle per 24 ore.
- In un laboratorio BMBL, sostituire il mezzo di trasfezione dalle cellule HEK 293T con 12 mL di DMEM fresco completo contenente FBS e penicillina/streptomicina. Incubare le cellule per 24 ore.
- Raccogliere e filtrare il mezzo con una siringa da 20 ml e un filtro da 0,45 μm. Trasferire il mezzo condizionato in un tubo di polipropilene da 15 ml.
- Aggiungere 12 mL di DMEM fresco completo contenente FBS e penicillina/ streptomicina al piatto e alla coltura di coltura HEK 293T per ulteriori 24 ore.
- Raccogliere e filtrare il mezzo con una siringa da 20 ml e un filtro da 0,45 μm. Trasferire il mezzo condizionato in un tubo di polipropilene da 15 ml. Conservare il mezzo contenente particelle lentivirali come aliquote di 1 mL a -80 °C per un ulteriore utilizzo.
- Infettare le cellule MS-1 con il virus pLV-Cas9.
- Seme 1 x 105 cellule MS-1 per pozzo in una piastra da 6 po 'per 24 ore prima dell'infezione da lentivirus.
- Scongelare le aliquote congelate del virus pLV-Cas9 in un bagno d'acqua a 37 °C.
- Mescolare 1 mL di mezzo virale con 1 mL di mezzo DMEM fresco contenente FBS e penicillina/streptomicina. Aggiungere polibrene al mezzo (concentrazione finale come 10 μg/mL) per aumentare l'efficienza dell'infezione.
- Rimuovere il mezzo dalla piastra a 6 porri e sostituirlo con il mezzo di miscela virus/polibrene e colturare le cellule in un'incubatrice per 24 ore e 24 ore dopo l'infezione, sostituire il mezzo con mezzo fresco e colturare le cellule per altri 24 h.
NOTA: tenere sempre un non infetto e un gruppo di controllo. - Aspirare il mezzo dal gruppo infetto e dal gruppo di controllo e sostituirlo con mezzo DMEM con blasticidina da 4 μg/mL.
- Riportare la piastra in un incubatore a 37 °C e coltura delle cellule per 1 settimana. Le cellule non infette moriranno a causa dell'effetto della blasticillina. Dividi le cellule sopravvissute quando raggiungono l'80% di confluenza cellulare e continuano con la selezione della blastidina.
- Confermare l'espressione di Cas9 nelle cellule MS-1 mediante gonfiore occidentale usando un anticorpo contro Cas9 (il peso molecolare di Cas9 è di circa 160 kDa).
- Infettare separatamente le cellule pLV-Cas9 MS-1 con due lentivirus gRNA indipendenti.
- Seme 1 x 105 cellule MS-1 pLV-Cas9 per pozzo in una piastra da 6 po 'per 24 ore prima dell'infezione.
- Seguire lo stesso protocollo descritto nella 4.2 per infettare separatamente le cellule con due lentivirus gRNA.
- Dopo 24 ore di infezione dal virus del gRNA, aggiornare il mezzo e colturare le cellule per altri 24 h.
- Sostituire il mezzo con DMEM con 1 μg/mL puromicina. Riportare la piastra in un incubatore a 37 °C e coltura delle cellule per 1 settimana. Assicurarsi che le cellule non infette siano completamente morte. Dividi le cellule quando raggiungono l'80% di confluenza e continuano la selezione di puromicina.
- Confermare il knock-out di Lumaca nelle cellule MS-1 mediante gonfiore occidentale usando un anticorpo contro lumaca (il peso molecolare di Lumaca è di circa 35 kDa).
Risultati
TGF-β2 induce endmt e stimola l'espressione di lumaca nelle cellule endoteliali MS-1
TGF-β è una delle citochine con il maggior potenziale per indurre EndMT. Dopo aver trattato le cellule MS-1 con TGF-β2 (1 ng/mL) per 3 giorni, le cellule Endoteliali MS-1 perdono la loro struttura simile a ciottoli e si differenziano in cellule mesenchimali a forma di mandrino(Figura 1A)15. Per verificare ulteriormente il ruolo del TGF-β2 nell'indurre cambiamenti fenotipiche cellulari, abbiamo pretrattato le cellule con la piccola molecola di activina recettore-simile chinasi (ALK)4/ALK5/ALK7 inibitore SB431542 prima della stimolazione TGF-β219. SB431542 ha completamente abrogato i cambiamenti morfologici delle cellule indotte da TGF-β2 (Figura 1A). Il processo EndMT indotto dal TGF-β2 è stato ulteriormente studiato studiando i cambiamenti nell'espressione dei marcatori correlati all'EndMT. Come mostrato nella figura 1B, la proteina endoteliale Pecam-1 è stata potentemente diminuita dopo la stimolazione TGF-β2, mentre il fattore mesenchimale Sm22α è stato profondamente upregolato da TGF-β215. Questi dati sono coerenti con l'idea che TGF-β2 ha attivato EndMT nelle cellule MS-1. Successivamente, abbiamo studiato gli effetti di TGF-β2 sull'espressione di lumaca e lumaca. Come mostrato nella figura 1C, Lumaca è stata marcatamente upregolata da TGF-β2, mentre l'espressione di Lumaca non è stata influenzata da TGF-β2 nelle cellule MS-115. La quantificazione dell'espressione di Lumaca da tre esperimenti indipendenti è mostrata nella figura 1D.
Esaurimento della lumaca da parte di CRISPR/Cas9 nelle cellule endoteliali MS-1
Poiché Snail è stato indotto da TGF-β2 e probabilmente coinvolto in EndMT mediato da TGF-β2, abbiamo eseguito l'editing genico CRISPR/Cas9 per esaurire geneticamente l'espressione di Lumaca nelle cellule MS-1. Abbiamo ipotizzato che l'esaurimento della lumaca sarebbe stato sufficiente per inibire l'EndMT indotto da TGF-β2. Come mostrato nella figura 2A, abbiamo generato cellule knockout lumaca in due passaggi. In primo luogo, Cas9 è stato espresso ectopicamente infettando le cellule MS-1 con un Cas9 che esprimeva lentivirus. Poiché nel costrutto pLV-Cas9 è presente una cassetta di resistenza alla blastidina, abbiamo controllato l'espressione di Cas9 mediante analisi western blot in celle resistenti alla blastidina (Figura 2D). Successivamente, abbiamo introdotto sgRNA che hanno specificamente preso di mira Snail per interrompere la sua espressione proteica. Questa procedura è stata eseguita anche per infezione da particelle lentivirali che trasportano il costrutto AA19 pLKO.1-Snail-sgRNA, che include una cassetta di espressione puromicina. Le cellule che esprimono Cas9 sono state nuovamente infettate da gRNA contenente lentivirus e ulteriormente selezionate con puromicina. Due oligo di sgRNA complementari mirati alla lumaca murina sono stati progettati con una bassa attività off-target prevista (Figura 2B,C). Dopo aver introdotto due SgRNA di lumaca indipendenti in Cas9 che esprimono cellule MS-1, l'espressione della proteina Lumaca è stata abrogata (Figura 2D)15.
La carenza di lumaca inibisce l'EndMT indotto da TGF-β2 nelle cellule MS-1
Per dimostrare la funzione della lumaca nell'EndMT mediato da TGF-β2, abbiamo eseguito un test EndMT nelle cellule impoverite di lumaca e l'abbiamo confrontato con le cellule parentali MS-1. Come mostrato nella figura 3A, il knockout di Lumaca è stato sufficiente per inibire la morfologia cellulare simile ai fibroblasti guidata da TGF-β2 nelle cellule MS-115. Inoltre, il declino mediato da TGF-β2 in Pecam-1 e il miglioramento di Sm22α sono stati completamente bloccati nelle cellule MS-1 impoverite di lumaca. In sintesi, abbiamo dimostrato che Snail è fondamentale per l'EndMT mediato da TGF-β2 nelle cellule MS-1 (Figura 3B)15.
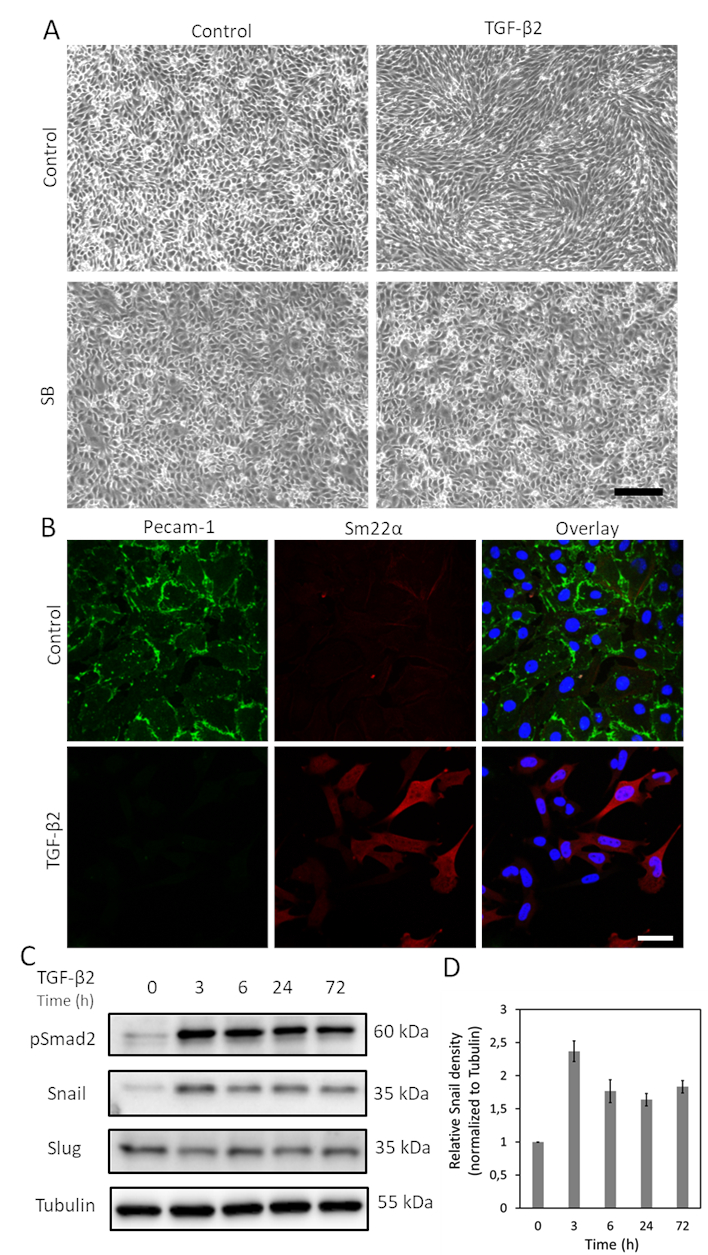
Figura 1. TGF-β2 induce l'espressione endmt e lumaca nelle cellule MS-1. A. La commissione per l'a Effetti dell'inibitore della tgf-β2 e/o β del recettore della chinasi di tipo I SB-431542 sulla morfologia cellulare. Immagini a campo luminoso delle cellule MS-1 al trattamento con TGF-β2 (1 ng/mL) e/o SB-431542 (SB, 5 μM, somministrato 30 min prima di TGF-β2) per 2 giorni. La barra di scala rappresenta 200 μm. Colorazione ad immunofluorescenza di Pecam-1 (verde) e Sm22α (rosso) in cellule MS-1 coltivate in mezzo contenenti TGF-β2 (1 ng/mL) per 3 giorni. I nuclei sono visualizzati in blu (DAPI). Barra di scala: 50 μm. C. Macchia occidentale con lisato a cellule intere di cellule MS-1 stimolate da TGF-β2. L'espressione di Lumaca, ma non Slug, è stata migliorata dalla stimolazione TGF-β2, come precedentemente riportato in Ma et al15. D. La commissione per l' Quantificazione dell'espressione di Lumaca integrando i risultati di tre esperimenti western blot indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.
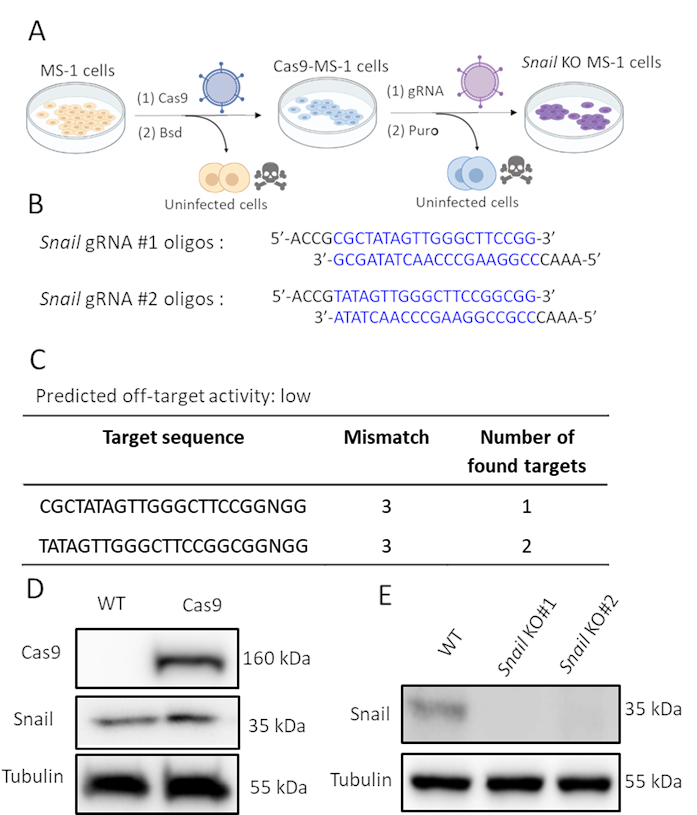
Figura 2. Esaurimento della lumaca da parte dell'editing genico CRISPR-Cas9. A. La commissione per l'a Schema che descrive come generare cellule knockout di lumaca. Bsd: Blasticidina. Puro: Puromicina. B. La commissione per l' Oligonucleotidi di due sgRNA indipendenti che prendono di mira Lumaca usando CHOPCHOP (http://chopchop.cbu.uib.no/) e Cas-OFFinder (http://www.rgenome.net/cas-offinder/). C. La commissione per l' Attività off-target prevista dei due gRNA per Lumaca utilizzando Cas-OFFinder (http://www.rgenome.net/cas-offinder/). D. La commissione per l' Espressione cas9 e lumaca in tipo selvaggio (WT) e MS-1 sovraespresso cas9 misurato dall'analisi western blot. E. La commissione per l' Knockout di Lumaca con due gRNA indipendenti nelle cellule MS-1 misurate dall'analisi western blot. Abbiamo riportato risultati simili in Ma et al15. Clicca qui per visualizzare una versione più grande di questa figura.
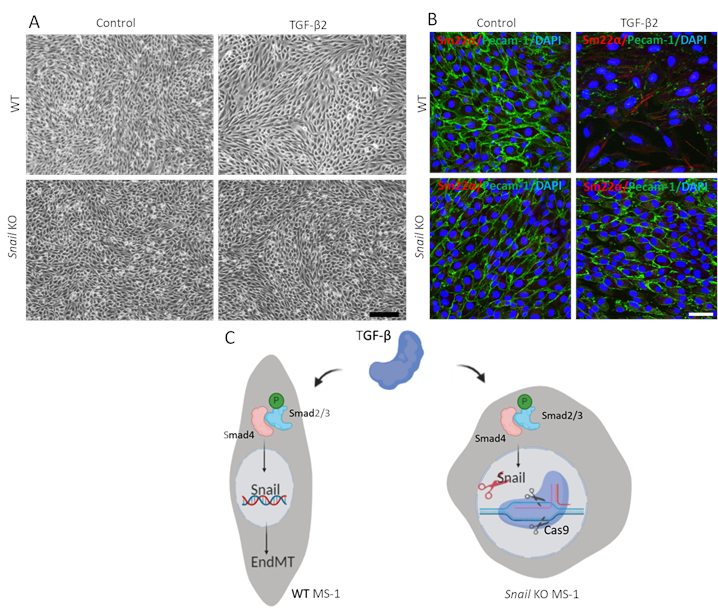
Figura 3. L'esaurimento genetico della lumaca inibisce l'EndMT indotto da TGF-β2 nelle cellule MS-1. A. La commissione per l'a Immagini brightfield di cellule MS-1 al trattamento con TGF-β2 (0,1 ng/mL) per 3 giorni in cellule wildtype (WT, pannello superiore) e Snail knocked out (pannello inferiore). La barra di scala rappresenta 200 μm. B. Colorazione immunofluorescente per Pecam-1 (verde), Sm22α (rosso) e nuclei (blu) di cellule MS-1 coltivate in mezzo contenente TGF-β2 (1 ng/mL) per 3 giorni. L'esaurimento della lumaca ha abrogato la diminuzione indotta da TGF-β2 di Pecam-1 e l'aumento dell'espressione di Sm22α. La barra di scala rappresenta 50 μ β m. TGF-β stimola l'espressione della lumaca attraverso la via Smad fosforilando Smad2/3 e guida ulteriormente EndMT. Knocking Out Snail utilizzando l'editing genico basato su CRISPR /Cas9 ha abrogato L'EndMT β mediato con TGF. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Comprendere il meccanismo dell'EndMT è fondamentale per modulare questo processo e indirizzare le malattie correlate alla endmt. Qui, abbiamo descritto i metodi per eseguire un test EndMT indotto da TGF-β e interrogare il ruolo della lumaca EndMT-TF nell'EndMT innescato da TGF-β, eseguendo l'esaurimento genico stabile mediato da CRISPR / Cas9 di Lumaca dalle cellule. L'esaurimento della lumaca mediante l'approccio CRISPR/Cas9 ha abrogato con successo l'EndMT guidato da TGF-β2 nelle cellule MS-1 (Figura 3C). Per studiare gli effetti di qualsiasi citochina, come il TGF-β, sull'EndMT, gli EC sono stati esposti alle citochine e quindi il verificarsi di EndMT è stato valutato in base a cambiamenti morfologici e cambiamenti di espressione del marcatore endoteliale e mesenchimale nelle cellule. TGF-β2 EndMT fortemente indotto nelle cellule MS-1 accompagnato da un forte aumento dell'espressione del fattore di trascrizione Lumaca. Gli EndMT-TF indotti dal TGF-β possono differire a seconda della specie o del tipo di cellula endoteliale specifica del tessuto. Ad esempio, abbiamo osservato che lumaca ma non lumaca è stata significativamente upregolata dal TGF-β nelle cellule MS-1, mentre nelle cellule endoteliali venei ombelicali umane (HUVEC), sia Lumaca che Lumaca sono aumentate dopo l'esposizione a TGF-β20.
La Corte ha valutato l'estensione del processo EndMT in due modi esaminando le modifiche della morfologia cellulare e quindi studiando le modifiche nell'espressione dei marcatori correlati all'EndMT. Dopo l'esposizione β TGF-β per 3 giorni, le cellule sono state sottoposte a EndMT con variazioni morfologiche coerenti e cambiamenti nell'espressione dei marcatori correlati alla EndMT. Oltre alla colorazione dell'immunofluorescenza che abbiamo eseguito qui, le variazioni marcatori possono anche essere monitorate dall'gonfiore occidentale a livello di espressione proteica o da qRT-PCR (Real-Time Quantitative Reverse Transcription PCR) ai livelli genico21. Oltre a questi due metodi di risparmio di tempo e costi mostrati in questo protocollo, esistono altri metodi per esaminare EndMT. Ad esempio, l'esecuzione dell'analisi del trascrittoma (mediante sequenziamento dell'RNA o qPCR) per confrontare i livelli di espressione dei geni endoteliali e mesenchimali correlati tra cellule trattate e di controllo può valutare con precisione EndMT22,23. Inoltre, EndMT comporta spesso la perdita stabile della funzione barriera, che può essere valutata mediante spettroscopia di impedenza24. Inoltre, possono essere esaminate ulteriori prove dell'acquisizione di proprietà simili a cellule staminali da parte di cellule derivate da EndMT. Ad esempio, in condizioni di coltura specifiche, le cellule mesenchimali EndMT possono essere ulteriormente differenziate in osteoblasti, condrociti, adipociti o (mio)fibroblasti. Pertanto, un'analisi aggiuntiva per confermare la differenziazione in diversi tipi di cellule appartenenti al lignaggio mesoderma (cioè l'espressione genica e la colorazione matriciale) è utile per dimostrare la natura multipotente delle cellule derivate da EndMT. Infine, i metodi di valutazione EndMT non si limitano agli studi in vitro, ma possono essere estrapolati per indagare la relazione tra EndMT e alcune malattie in vivo o in organi ex vivo. In questo senso, l'uso di strategie di tracciamento del lignaggio specifiche dell'endoteliale è ampiamente esteso alla ricerca relativa all'EndMT25.
Per indagare il ruolo di Lumaca durante EndMT, in questo studio l'editing genico CRISPR/Cas9 è stato utilizzato per eliminare questo gene. I dati hanno mostrato che il TGF-β2 non è riuscito a mediare EndMT in cellule MS-1 carenti di lumaca. Questa osservazione ha dimostrato che la lumaca è essenziale per l'EndMT indotto da TGF-β2 nelle cellule MS-1. Abbiamo utilizzato una cassetta di espressione sgRNA indipendente guidata da U6 per introdurre sgRNA specifici per Cas9 per colpire Snail. In aggiunta a questo metodo, Ran etal. I nuovi approcci emergenti consentono a CRISPR/Cas di incorporare funzioni aggiuntive. Ad esempio, è possibile ottenere ko doppi o tripli fornendo più sgRNA nelle celle che esprimono Cas927. La proteina Cas13 ingegnerizza bersagli e digerisce molecole di RNA senza interrompere il DNA endogeno28. Oltre a eliminare i geni con CRISPR/Cas, gli RNA a tornante corta (shRNA) possono essere utilizzati come alternative all'espressione genica mirata29. Per tutti i metodi di editing genico CRISPR/Cas, la scissione off-target deve sempre essere presa in considerazione. Inoltre, piccoli RNA interferenti (siRNA) silenziano transitoriamente l'espressione genica e la concentrazione di siRNA viene diluita con la divisionecellulare 30. Entrambi questi metodi sopprimono parzialmente l'espressione genica mirata. Al contrario, l'espressione genica ectopica viene utilizzata anche per verificare la funzione genica durante EndMT/EMT31. Questo approccio può determinare se l'upregulation di un gene è sufficiente per ottenere una risposta EndMT. Pertanto, attualmente ci sono una moltitudine di strategie tecniche che possono essere utilizzate per identificare e verificare potenziali regolatori di EndMT. Inoltre, l'analisi trascrittamica può essere una buona opzione nell'identificazione e nell'analisi completa dei regolatori correlati a EndMT. Si consiglia di utilizzare diversi approcci complementari per indagare la modulazione di EndMT.
In sintesi, è stato introdotto un flusso di lavoro per identificare i fattori che possono svolgere ruoli funzionali durante l'EndMT β indotto da TGF-β. Questo metodo può anche essere usato per studiare se altri stimoli (cioè citochine, fattori di crescita, stimoli meccanici, interazioni cellula-cellula) possono modulare EndMT e l'interazione di TGF-β con altri stimoli. Inoltre, abbiamo evidenziato un approccio che utilizza l'editing genico CRIPSR/Cas per chiarire se è necessario un certo gene per l'EndMT indotto da β TGF. Per illustrare questa metodologia, abbiamo usato il forte induttore EndMT TGF-β2 nelle cellule MS-1, ma i protocolli possono essere adattati ad altre citochine e altri tipi di cellule. Ci aspettiamo che questo protocollo dettagliato descritto serva da trampolino di lancio per i futuri studi relativi all'EndMT.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
La ricerca è stata sostenuta da CGC.NL e dalla Netherlands Cardio Vascolar Research Initiative: la Dutch Heart Foundation, la Dutch Federation of University Medical Centers, l'Organizzazione olandese per la ricerca e lo sviluppo sanitario e la Royal Netherlands Academy of Sciences Grant assegnata alla Phaedra-Impact (http://www.phaedraresearch.nl). JM è sostenuto dal Chinese Scholarship Council. GSD è sostenuta da una sovvenzione trampolino di AFM-Telethon [22379], FOP Italia e da una sovvenzione di La Fundació La Marató de TV3 (#202038).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | Pall Corporation, USA | 4614 | |
| 10× T4 DNA ligase buffer | Thermofisher Scientific, USA | B69 | |
| 12 mm round glass slice | Knittel Glass,Germany | VD10012Y1A.01 | |
| 20 mL syringe | BD Eclipse, USA | 300629 | |
| 4′,6-diamidino-2-phenylindole (DAPI) | Vector Laboratories, USA | H-1200 | VECTASHIELD Antifade Mounting Media |
| AA19_PLKO vector | Dr. M Gonçalves, Leiden University Medical Center, Netherlands | Gift | |
| Ampicillin | Serva Electrophoresis, USA | 1339903 | |
| Anti-Mouse IgG | GE Healthcare, USA | NA931 | |
| Anti-Rabbit IgG | Cell signaling, USA | 7074 | |
| Agarose | Roche, Switzerland | 11388991001 | |
| Blasticidin | Invitrogen, USA | R21001 | |
| Buffer O (10X) | Thermofisher Scientific, USA | BO5 | |
| BveI (Bspm1) | Thermofisher Scientific, USA | ER 1741 | |
| Confocal microscope | Leica Microsystems, Germany | SP8 | |
| DMEM | Thermo Fisher Scientific, USA | 11965092 | |
| Donkey anti-rat Alexa 488 | Invitrogen, USA | A21208 | |
| FBS | Thermo Fisher Scientific, USA | 16000044 | |
| Formaldehyde | Thermo Fisher Scientific, USA | 28908 | |
| Inverted microscope | Leica Microsystems, Germany | DMi8 | |
| Goat anti-rabbit Alexa 594 | Invitrogen,USA | A11012 | |
| LabNed Plasmid kit | LabNed, USA | LN2400004 | |
| MS-1 cell line | American Type Culture Collection (ATCC) | ||
| Nail polish | HEMA, Netherlands | Transparent | |
| Pecam-1 antibody | Becton Dickinson,USA | 553370 | |
| PEI | Polysciences, USA | 23966-1 | |
| PLV-Cas9 plasmid | Sigma-Aldrich, USA | Cas9BST-1EA | |
| Puromycin | Sigma-Aldrich, USA | P9620 | |
| QIAquick gel extraction kit | Qiagen, Germany | 28706 | |
| Sm22a antibody | Abcam, UK | ab14106 | |
| Snail | Cell signaling, USA | 3879 | |
| T4 DNA ligase | Thermofisher Scientific, USA | EL 0014 | |
| TC20 automated Cell Counter | Bio-Rad, USA | 1450102 | |
| Human TGF-β2 | Joachim Nickel, University of Wurzburg | Gift | Other commercial recommendation: 302-B2, R&D systems |
| Triton X-100 | Merck, USA | 1086031000 | |
| SB431542 | Tocris Bioscience, UK | 1614 | |
| Tris | Roche, Switzerland | 11814273001 |
Riferimenti
- Ma, J., Sanchez-Duffhues, G., Goumans, M. -. J., Ten Dijke, P. TGF-β-induced endothelial to mesenchymal transition in disease and tissue engineering. Frontiers in Cell and Developmental Biology. 8, (2020).
- Piera-Velazquez, S., Jimenez, S. A. Endothelial to mesenchymal transition: role in physiology and in the pathogenesis of human diseases. Physiological Reviews. 99 (2), 1281-1324 (2019).
- Sánchez-Duffhues, G., de Vinuesa, G. A., ten Dijke, P. Endothelial-to-mesenchymal transition in cardiovascular diseases: developmental signaling pathways gone awry. Developmental Dynamics. 247 (3), 492-508 (2018).
- Evrard, S. M., et al. Endothelial to mesenchymal transition is common in atherosclerotic lesions and is associated with plaque instability. Nature Communications. 7 (1), 1-16 (2016).
- Qiao, L., et al. Endothelial fate mapping in mice with pulmonary hypertension. Circulation. 129 (6), 692-703 (2014).
- Zeisberg, E. M., et al. Endothelial-to-mesenchymal transition contributes to cardiac fibrosis. Nature Medicine. 13 (8), 952-961 (2007).
- Clere, N., Renault, S., Corre, I. Endothelial-to-mesenchymal transition in cancer. Frontiers in Cell and Developmental Biology. 8, (2020).
- Saito, A. EMT and EndMT: regulated in similar ways. The Journal of Biochemistry. 153 (6), 493-495 (2013).
- Bolós, V., et al. The transcription factor Slug represses E-cadherin expression and induces epithelial to mesenchymal transitions: a comparison with Snail and E47 repressors. Journal of Cell Science. 116 (3), 499-511 (2003).
- Aird, W. C. Endothelial cell heterogeneity. Cold Spring Harbor Perspectives in Medicine. 2 (1), 006429 (2012).
- Yang, J., et al. Guidelines and definitions for research on epithelial-mesenchymal transition. Nature Reviews Molecular Cell Biology. , 1-12 (2020).
- Doudna, J. A., Charpentier, E. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), (2014).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Arbiser, J. L., et al. Oncogenic H-ras stimulates tumor angiogenesis by two distinct pathways. Proceedings of the National Academy of Sciences USA. 94 (3), 861-866 (1997).
- MA, J., et al. TGF-?-induced endothelial to mesenchymal transition results from a balance between SNAIL and ID factors. Frontiers in Cell and Developmental Biology. 182 (9), (2021).
- Chen, X., et al. Probing the impact of chromatin conformation on genome editing tools. Nucleic Acids Research. 44 (13), 6482-6492 (2016).
- Hunter, F. W., et al. Functional CRISPR and shRNA screens identify involvement of mitochondrial electron transport in the activation of evofosfamide. Molecular Pharmacology. 95 (6), 638-651 (2019).
- Mao, Y., et al. Lentiviral vectors mediate long-term and high efficiency transgene expression in HEK 293T cells. International Journal of Medical Sciences. 12 (5), 407 (2015).
- Liu, S., et al. Deubiquitinase activity profiling identifies UCHL1 as a candidate oncoprotein that promotes TGFβ-induced breast cancer metastasis. Clinical Cancer Research. 26 (6), 1460-1473 (2020).
- Medici, D., et al. Conversion of vascular endothelial cells into multipotent stem-like cells. Nature Medicine. 16 (12), 1400 (2010).
- Kokudo, T., et al. Snail is required for TGFβ-induced endothelial-mesenchymal transition of embryonic stem cell-derived endothelial cells. Journal of Cell Science. 121 (20), 3317-3324 (2008).
- Dong, J., et al. Single-cell RNA-seq analysis unveils a prevalent epithelial/mesenchymal hybrid state during mouse organogenesis. Genome Biology. 19 (1), 1-20 (2018).
- Oatley, M., et al. Single-cell transcriptomics identifies CD44 as a marker and regulator of endothelial to haematopoietic transition. Nature communications. 11 (1), 1-18 (2020).
- Halaidych, V., et al. Inflammatory responses and barrier function of endothelial cells derived from human induced pluripotent stem cells. Stem Cell Reports. 10 (5), 1642-1656 (2018).
- Li, Y., Lui, K. O., Zhou, B. Reassessing endothelial-to-mesenchymal transition in cardiovascular diseases. Nature Reviews Cardiology. 15 (8), 445-456 (2018).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Grav, L. M., et al. One-step generation of triple knockout CHO cell lines using CRISPR/Cas9 and fluorescent enrichment. Biotechnology Journal. 10 (9), 1446-1456 (2015).
- Abudayyeh, O. O., et al. RNA targeting with CRISPR-Cas13. Nature. 550 (7675), 280-284 (2017).
- Paddison, P. J., et al. Cloning of short hairpin RNAs for gene knockdown in mammalian cells. Nature Methods. 1 (2), 163-167 (2004).
- Reynolds, A., et al. Rational siRNA design for RNA interference. Nature Biotechnology. 22 (3), 326-330 (2004).
- Lourenço, A. R., et al. C/EBPɑ is crucial determinant of epithelial maintenance by preventing epithelial-to-mesenchymal transition. Nature Communications. 11 (1), 1-18 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon