Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Estudios de interacciones chaperona-cochaperona utilizando un ensayo homogéneo basado en cuentas
En este artículo
Resumen
Este protocolo presenta una técnica para sondear las interacciones proteína-proteína utilizando perlas de donantes ligadas al glutatión con co-chaperonas con motivos TPR fusionadas con GST y perlas aceptoras junto con un péptido derivado de Hsp90. Hemos utilizado esta técnica para detectar moléculas pequeñas para interrumpir las interacciones Hsp90-FKBP51 o Hsp90-FKBP52 e identificado inhibidores potentes y selectivos de la interacción Hsp90-FKBP51.
Resumen
Dirigirse a las interacciones proteína de choque térmico 90 (Hsp90)-cochaperona proporciona la posibilidad de regular específicamente los procesos intracelulares dependientes de Hsp90. El pentapéptido MEEVD conservado en el extremo C de Hsp90 es responsable de la interacción con el motivo de repetición tetratricopéptido (TPR) de las co-chaperonas. La proteína de unión a FK506 (FKBP) 51 y FKBP52 son dos co-chaperonas similares con motivos de TPR involucradas en enfermedades dependientes de hormonas esteroides con diferentes funciones. Por lo tanto, la identificación de moléculas que bloquean específicamente las interacciones entre Hsp90 y FKBP51 o FKBP52 proporciona un potencial terapéutico prometedor para varias enfermedades humanas. Aquí, describimos el protocolo para un ensayo homogéneo de proximidad luminiscente amplificada para sondear las interacciones entre Hsp90 y sus co-chaperonas asociadas FKBP51 y FKBP52. En primer lugar, hemos purificado las proteínas FKBP51 y FKBP52 que contienen motivos TPR en forma marcada con glutatión S-transferasa (GST). Utilizando las perlas de donantes ligadas al glutatión con proteínas con motivo tPR fusionadas con GST y las perlas aceptoras junto con un péptido C-terminal de 10 mer de Hsp90, hemos sondeado las interacciones proteína-proteína en un entorno homogéneo. Hemos utilizado este ensayo para detectar moléculas pequeñas para interrumpir las interacciones Hsp90-FKBP51 o Hsp90-FKBP52 e identificado inhibidores potentes y selectivos de la interacción Hsp90-FKBP51.
Introducción
Las chaperonas moleculares contribuyen a la homeostasis de las proteínas, incluido el plegamiento, el transporte y la degradación de las proteínas. Regulan varios procesos celulares y están vinculados a numerosas enfermedades como el cáncer y las enfermedades neurodegenerativas1. La proteína de choque térmico 90 (Hsp90) es una de las chaperonas más importantes cuya función depende de cambios conformacionales impulsados por la hidrólisis de ATP y la unión con proteínas cliente mediadas por sus co-chaperonas2. A pesar de un potencial obvio de Hsp90 como objetivo terapéutico, ajustar su función representa un gran desafío. Hay varios inhibidores de Hsp90 dirigidos a la región de unión a ATP N-terminal, que han sido evaluados en ensayos clínicos, pero ninguno de ellos ha sido aprobado para su comercialización3. Debido a la falta de un bolsillo de unión al ligando bien definido4,apuntar a la región C-terminal de Hsp90 ha tenido un éxito limitado4. Recientemente, la interrupción de las interacciones Hsp90-cochaperona por moléculas pequeñas se ha investigado como una estrategia alternativa5. Apuntar a las interacciones Hsp90-cochaperona no provocaría una respuesta general al estrés celular y brinda la posibilidad de regular específicamente varios procesos intracelulares. El pentapéptido MEEVD conservado en el extremo C de Hsp90 es responsable de la interacción con el motivo de repetición tetratricopéptido (TPR) de las co-chaperonas6. De las 736 proteínas que contienen motivos TPR anotadas en la base de datos de proteínas humanas, ~ 20 proteínas diferentes interactúan con Hsp90 a través de este péptido7. Las moléculas que compiten por la unión al péptido MEEVD interrumpirían las interacciones entre Hsp90 y las co-chaperonas que contienen un dominio TPR. El sitio de unión al péptido tiene una estructura terciaria similar, pero la homología general entre los diferentes dominios de motivos TPR es relativamente baja7,lo que brinda la oportunidad de identificar moléculas específicamente capaces de bloquear las interacciones entre Hsp90 y las co-chaperonas particulares con motivos TPR. Entre estas co-chaperonas con motivos TPR, la proteína de unión a FK506 (FKBP) 51 y FKBP52 son reguladores de la señalización del receptor de hormona esteroide (SHR) e involucradas en varias enfermedades dependientes de hormonas esteroides, incluyendo cáncer, enfermedades relacionadas con el estrés, enfermedades metabólicas y enfermedad de Alzheimer8. Aunque FKBP51 y FKBP52 comparten > similitud de secuencia del 80%, sus funciones difieren: FKBP52 es un regulador positivo de la actividad de SHR, mientras que FKBP51 es un regulador negativo en la mayoría de los casos8. Por lo tanto, la identificación de moléculas, bloqueando específicamente las interacciones entre Hsp90 y FKBP51 o FKBP52, proporciona un potencial terapéutico prometedor para enfermedades relacionadas.
Unensayo amplificado Luminescent Proximity Homogenous A(AlphaScreen) fue desarrollado por primera vez en 1994 por Ullman EF et al.9. Ahora es ampliamente utilizado para detectar diferentes tipos de interacciones biológicas, como el péptido10,la proteína11,el ADN12,el ARN13y el azúcar14. En esta técnica, hay dos tipos de cuentas (diámetro 200 nm), una es la cuenta donante y la otra es la cuenta aceptora. Las biomoléculas se inmovilizan sobre estas cuentas; sus interacciones biológicas ponen en proximidad a las cuentas donantes y aceptoras. A 680 nm, un fotosensibilizador en la cuenta del donante ilumina y convierte el oxígeno en oxígeno singlete. Debido a que el oxígeno singlete tiene una vida útil corta, solo puede difundirse hasta 200 nm. Si la perla aceptora está en proximidad, su derivado tioxeno reacciona con el oxígeno singlete generando quimioluminascencia a 370 nm. Esta energía activa aún más los fluoróforos en la misma cuenta aceptora para emitir luz a 520-620 nm15. Si las interacciones biológicas se interrumpen, la cuenta aceptora y la cuenta donante no pueden alcanzar la proximidad, lo que resulta en la descomposición del oxígeno singlete y la baja señal producida.
Aquí describimos un protocolo que utiliza esta técnica para el cribado de moléculas pequeñas que inhiben las interacciones entre Hsp90 y las co-chaperonas TPR, especialmente FKBP51 y FKBP52. Los péptidos largos de 10 aminoácidos correspondientes al extremo C extremo Hsp90 están unidos a las perlas aceptoras. Las co-chaperonas TPR purificadas marcadas con GST interactúan con las perlas de donantes ligadas al glutatión. Cuando la interacción entre los péptidos derivados de Hsp90 y las co-chaperonas con motivos TPR une las perlas, se produce una señal amplificada(Figura 1A). Si las moléculas pequeñas examinadas pueden inhibir las interacciones entre Hsp90 y las co-chaperonas con motivo tPR, esta señal amplificada disminuirá (Figura 1B). Su IC50 se puede calcular mediante medición cuantitativa. Este protocolo se puede extender a cualquier interacción chaperona - TPR-motivo co-chaperona de interés y es de gran importancia en el desarrollo de nuevas moléculas, bloqueando específicamente la interacción entre Hsp90 y FKBP51 o FKBP52.

Figura 1: El principio básico de este ensayo. (A) GST-FKBP51 purificado interactúa con cuentas de donantes ligadas al glutatión. Los péptidos largos de 10 aminoácidos correspondientes al extremo C-terminal de Hsp90 se unen a las perlas aceptoras. La interacción entre los péptidos derivados de Hsp90 y el dominio TPR de FKBP51 acerca las perlas del donante y del aceptor. A 680 nm, un fotosensibilizador en la cuenta del donante ilumina y convierte el oxígeno en oxígeno singlete. El derivado tioxeno en la perla aceptora reacciona con el oxígeno singlete y genera quimioluminescencia a 370 nm. Esta energía activa aún más los fluoróforos en la misma cuenta aceptora para emitir luz a 520-620 nm. (B) Cuando las moléculas pequeñas inhiben las interacciones entre Hsp90 y FKBP51, las perlas donante y aceptora no pueden alcanzar la proximidad. Luego, el oxígeno singlete con una vida útil corta se descompone y no se produce ninguna señal detectable. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
NOTA: En la figura 2se muestra una descripción general de este protocolo.
1. Expresión y purificación de GST-FKBP51 y GST-FKBP52 (Figura 2A)
-
Plasmidios
NOTA: Obtenga clones de ADNc para FKBP51 humano (id de clon: 5723416) y para FKBP52 humano (id de clon: 7474554) del consorcio IMAGE.- Amplificar el ADN FKBP51 humano mediante PCR con cebadores (hacia adelante; 5'GGATCCATGACTACTGATGAAGGT-3', inverso; 5'-CTCGAGCTATGCTTCTGTCTCCAC-3') que contienen voladizos BamHI y XhoI y clonar en vector pGEX6-1 en los sitios de restricción BamHI / XhoI.
- Amplificar el ADN FKBP52 humano mediante PCR con cebadores (hacia adelante; 5'-GAATTCATGACAGCCGAGGAGATG-3', reverso; 5'-CTCGAGCTATGCTTCTGTCTCCAC-3') que contienen voladizos EcoRI y XhoI y clonar en vector pGEX6-2 en sitios de restricción EcoRI / XhoI.
NOTA: La configuración y las condiciones de la reacción de PCR se muestran en la Tabla 1 y la Tabla 2. - Verifique la secuencia insertada y transforme los plásmidos en la E. coli químicamente competente de acuerdo con el protocolo de fabricación.
-
Expresión y purificación de proteínas
- Agregue 25 g de base de caldo de Luria (LB) en 1 L de agua destilada para hacer la solución de LB. Autoclave a 121 °C durante 15 min. Después del enfriamiento, agregue 50 μg / ml de ampicilina.
- Tome una colonia de bacterias que expresen GST-FKBP51 o GST-FKBP52 y mezcle con 500 μL de solución de LB en un tubo de 1,5 ml. Vórtice.
- Agregue la mezcla de "1.2.2" en 1 L de solución lb en el matraz Erlenmeyer cubierto con un papel de aluminio. Incubar el matraz Erlenmeyer en la coctelera durante la noche a 37 °C.
- Inducir la expresión de proteínas mediante la adición de 1 mM de isopropil-β-D-tiogalactósido (IPTG) al matraz de Erlenmeyer y continuar la incubación durante otras 2 h.
- Para obtener pellets celulares, centrífuga a 5.000 x g durante 15 min. Retire el sobrenadante.
NOTA: Los pellets celulares se pueden almacenar a -20 °C. - Resuspend los gránulos celulares en 40 mL de PBS y sonicar 3 x 20 s sobre hielo. Agregue 1 mM PMSF, 1 mM EDTA y cóctel inhibidor de proteasa (1 tableta) para prevenir la proteólisis.
- Centrifugar la suspensión durante 30 min 50,000 x g para eliminar los restos celulares y aplicar el sobrenadante sobre la columna de trampa GST de 5 ml.
- Después de lavar la columna con 30 mL PBS, elute GST-FKBP51 y GST-FKBP52 con 5 mL de 10 mM de glutatión en PBS.
- Concentrar proteínas en 15 mL 10.000 MWCO unidad de centrifugación. Para eliminar el glutatión libre, pase los concentrados a través de la columna PD-10 equilibrada con 0.5x PBS y concéntrese nuevamente en el dispositivo de centrífuga de filtro.
- Recolectar fracciones que contengan proteínas. Verifique las proteínas en SDS-PAGE y ajuste las concentraciones de proteínas a 1 mg/ml.
NOTA: El rendimiento típico de proteínas es de 2-5 mg / L de cultivo. La proteína se puede almacenar a -20 °C.
2. Acoplamiento del péptido C-terminal Hsp90 a las perlas aceptoras (Figura 2B)
-
Preparación de péptidos Hsp90
- Sintetizar diez aminoácidos péptidos NH2-EDASRMEEVD-COOH correspondientes a aminoácidos 714-724 de la isoforma beta Hsp90 humana (UniProt ID: P08238) mediante un servicio de síntesis de péptidos.
- Diluya el péptido Hsp90 en PBS a una concentración de 1 mg/ml.
-
Preparación de cuentas de aceptor
- Diluya las perlas aceptoras no conjugadas en PBS a una concentración de 1 mg/ml y transfiéralas a un tubo de 1,5 ml.
- Realizar el lavado por centrifugación a 16.000 x g durante 15 min. Retire con cuidado el sobrenadante.
-
Conjugación
- Establezca la proporción entre cuentas y péptidos como 10: 1. En el tubo de 1,5 ml que contiene 1 mg de pellet de perla aceptora (preparado como se describió anteriormente), agregue 1 ml de PBS (pH 7.4), 0,1 mg de péptido diluido, 1,25 μL de Tween-20, 10 μL de una solución de 400 mM de cianoborohidruro de sodio (NaBH3CN) en agua.
PRECAUCIÓN: NaBH3CN es tóxico; use una campana extractora y guantes. La solución de NaBH3CN debe estar recién preparada. - Incubar durante 24 h a 37 °C con agitación de extremo a extremo (10-20 rpm) en un agitador rotativo.
- Establezca la proporción entre cuentas y péptidos como 10: 1. En el tubo de 1,5 ml que contiene 1 mg de pellet de perla aceptora (preparado como se describió anteriormente), agregue 1 ml de PBS (pH 7.4), 0,1 mg de péptido diluido, 1,25 μL de Tween-20, 10 μL de una solución de 400 mM de cianoborohidruro de sodio (NaBH3CN) en agua.
-
Enfriamiento de reacción y lavado de cuentas
- Agregue 20 μL de solución de 1 M Tris-HCl (pH 8.0) a la reacción para bloquear sitios no reaccionados. Incubar durante 1 h a 37 °C.
- Centrifugadora a 16.000 x g (o velocidad máxima) durante 15 min a 4 °C. Retire el sobrenadante y vuelva a colocar el pellet de perlas en 1 ml de solución de Tris-HCl (100 mM, pH 8.0).
- Repita el paso de lavado tres veces.
- Después de la última centrifugación, vuelva a colocar las perlas a 1 mg/ml en tampón de almacenamiento (1 ml de 0,5 × PBS con azida de sodio al 0,01% como conservante). Almacene la solución de perla aceptora conjugada a 4 °C protegida contra la luz.
PRECAUCIÓN: La azida de sodio es tóxica; use una campana extractora y guantes.
3. El ensayo que investiga la interacción entre GST-FKBP51 o GST-FKBP52 y el péptido C-terminal Hsp90, y la inhibición con pequeños compuestos de masa molecular (Figura 2C)
-
Proteínas marcadas con GST que interactúan con las perlas de donantes de glutatión
- Configure las reacciones en placas de 384 pozos.
- Preparar la solución que contiene 10 μg/ml de las perlas donantes de glutatión en 0,5x PBS, pH 7,4.
NOTA: Después de un almacenamiento prolongado, las cuentas se asientan y necesitan ser vórtices. - Añadir GST-FKBP51 o GST-FKBP52 a una concentración final de 10 μg/ml.
- Incubar en la oscuridad a 25 °C durante 10 min.
NOTA: En este paso, las proteínas marcadas con GST interactuarán con el glutatión unido a las perlas. Para cada pozo, se utilizarán 22,5 μL de esta mezcla. La concentración de los socios vinculantes debe determinarse empíricamente. Titula GST-FKBP51 y GST-FKBP52 y elige la concentración que da la mejor señal.
-
Adición de compuestos
- Realizar diluciones seriadas de compuestos de prueba en DMSO.
NOTA: Las concentraciones utilizadas son típicamente 10, 30, 100, 300, 1,000 y 3,000 μM. - Añadir 0,25 μL de DMSO (control negativo) o péptido C-terminal Hsp90 (control positivo, 30 μM) o compuestos en DMSO a la esquina de cada pozo de la placa. Use triplicatos para cada concentración de compuestos.
- Agregue 22.5 μL de la solución que contiene perlas de donantes de glutatión con proteínas marcadas con GST a cada pozo.
- Agite el plato suavemente con la mano pero a fondo. Incubar en la oscuridad a 25 °C durante 15 min.
NOTA: Durante este tiempo, los compuestos interactuarán con el dominio TPR en el sitio de unión al péptido Hsp90 C-terminal.
- Realizar diluciones seriadas de compuestos de prueba en DMSO.
-
Adición de cuentas de aceptación
- Diluya las perlas aceptoras con el péptido C-terminal Hsp90 unido a 100 μg/mL en 0.5x PBS.
- Añadir 2,25 μL de perlas aceptoras diluidas a cada pozo.
- Mezclar suavemente pero a fondo. Incubar en la oscuridad a 25 °C durante 15 min.
NOTA: En este paso, las perlas donante y aceptora se ponen en proximidad por las interacciones proteína-péptido. El volumen final de la mezcla de reacción es de 25 μL. Por lo tanto, las concentraciones finales de compuestos oscilan entre 0,1 y 30 μM.
-
Lectura de placas
NOTA: Lea la placa utilizando un lector de placas configurado en el modo correspondiente.- Encienda el instrumento y abra el software
- Elija el protocolo correspondiente.
- Haga clic en Editar mapa de placa y seleccione el pozo que se utiliza en la placa para la medición.
- Haga clic en Siguiente para continuar y Ejecutar el protocolo seleccionado.
- Después de la medición, haga clic en Mostrar resultados para ver los resultados.
- Exporte los datos.
4. Análisis de datos
-
Factor Z' y relación señal-fondo (S/B)
- Calcule el factor Z' y la relación S/B para el ensayo utilizando la siguiente ecuación:
Z'=1-(3σpos+3σneg)/│μpos-μneg│16
S/B=μneg/μpos
donde, σ y μ representan las desviaciones estándar y las medias de los controles positivos (péptido C-terminal Hsp90, 30 μM) y negativos (DMSO), respectivamente. Un factor Z' > 0.5 asegurará que el ensayo sea lo suficientemente robusto para la detección. Para controlar la sensibilidad del ensayo, también se ha calculado la relación S/B.
- Calcule el factor Z' y la relación S/B para el ensayo utilizando la siguiente ecuación:
-
Curva dosis-respuesta y CI 50
NOTA: Utilice el análisis de regresión no lineal para ajustar los datos de los inhibidores del IBP de Hsp90-cochaperonas por software.- Cree una tabla de datos XY en el cuadro de diálogo de bienvenida y seleccione NúmerosX , e Y Escriba 3 (si triplica) replicar valores en columnas en paralelo.
- Normalizar los datos de señal de las muestras al grupo de control negativo. Importe los valores de concentración a la columna X y los valores de señal a la columna Y.
- Haga clic en Analizar y elija Concentración de transformación (X) en Transformar | Normalizar. Elija Transformar en logaritmos.
NOTA: Esto transformará la concentración en una escala logarítrica. Si su concentración inicial es cero, configálelo en un número muy pequeño que sea efectivamente cero (por ejemplo, 0.1 nM) para no perder esos valores ya que el logaritmo de cero no está definido. - Haga clic en Analizar y elija Regresión no lineal (ajuste de curva) en Análisis XY,abra la Inhibición de dosis-respuesta y elija Registro (inhibidor) vs. respuesta - Pendiente variable.
- Haga clic en Aceptar para ver los resultados (que contienen el valor IC50) y los gráficos.
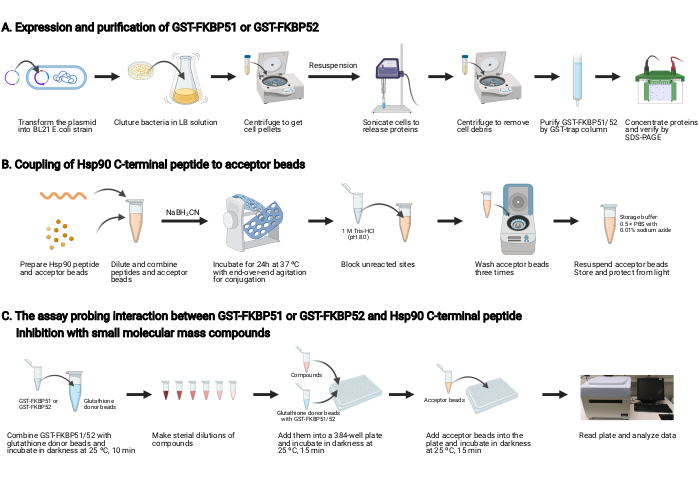
Figura 2: Esquema de este protocolo. (A) Expresión y purificación de GST-FKBP51 y GST-FKBP52. (B) Acoplamiento del péptido C-terminal Hsp90 a las perlas aceptoras. (C) El ensayo que investiga la interacción entre GST-FKBP51 o GST-FKBP52 y el péptido Hsp90 C-terminal. Inhibición con pequeños compuestos de masa molecular. Creado con BioRender.com Haga clic aquí para ver una versión más grande de esta figura.
Resultados
En nuestro ensayo, el factor Z' y la relación S/B son 0.82 y 13.35, respectivamente(Figura 3A),lo que demuestra que nuestro ensayo es robusto y confiable para el cribado de alto rendimiento. Luego lo usamos para detectar pequeños compuestos de masa molecular. La Figura 3B presenta la inhibición dependiente de la dosis de las interacciones chaperona-cochaperona con una molécula pequeña seleccionada (D10). Las curvas dosis-respuesta para D10 se generan median...
Discusión
Aquí describimos un protocolo que utiliza el ensayo para el cribado de moléculas pequeñas que inhiben las interacciones entre Hsp90 y las co-chaperonas con motivos TPR, especialmente FKBP51 y FKBP52. Su alta puntuación Z' (>0,8) demuestra la robustez y fiabilidad de un formato de alto rendimiento. Los resultados se pueden obtener en una hora, y se requieren pequeñas cantidades de perlas, proteínas y compuestos. Además, este protocolo podría extenderse fácilmente a cualquier interacción Hsp90 /Hsp70 - TPR-motif ...
Divulgaciones
Los autores no reportan conflictos de intereses.
Agradecimientos
Este estudio fue apoyado por subvenciones del Consejo Sueco de Investigación (2018-02843), la Fundación del Cerebro (Fo 2019-0140), la Fundación para Enfermedades Geriátricas en el Instituto Karolinska, la Fundación Gunvor y Josef Anérs, la Fundación Magnus Bergvalls, la Fundación Gun y Bertil Stohnes, la Fundación Tore Nilssons para la investigación médica, la Fundación Margaretha af Ugglas y la Fundación para Viejos Sirvientes.
Materiales
| Name | Company | Catalog Number | Comments |
| 384-well plates | Perkin Elmer | 6008350 | Assay volume 25 ml |
| Amicon 10.000 MWCO centrifugation unit | Millipore | UFC901008 | Concentrate protein |
| Ampicillin | Sigma | A0166 | Antibiotics |
| Bacteria shaker Unimax 1010 | Heidolph | Culture bacteria | |
| cDNA clones for human FKBP51 | Source BioScience | clone id: 5723416 | pCMV-SPORT6 vector |
| cDNA clones for human FKBP52 | Source BioScience | clone id: 7474554 | pCMV-SPORT6 vector |
| Chemically Competent E. coli | Invitrogen | C602003 | One Shot BL21 Star (DE3) |
| Data analysis software | GraphPad Prism | 9.0.0 | Analysis data and make figures |
| Data analysis software | Excel | Analysis data | |
| DMSO | Supelco | 1.02952.1000 | Dilute compounds |
| DPBS | Gibco | 14190-144 | Prepare solution |
| EDTA | Calbiochem | 344504 | Prevent proteolysis during sonication |
| Glutathione | Sigma | G-4251 | Elute GST-tagged proteins |
| Glutathione donor beads | Perkin Elmer | 6765300 | Donor bead |
| GST-trap column | Cytiva (GE Healthcare) | 17528201 | Purify GST-tagged proteins |
| Isopropyl-β-D-thiogalactoside | Thermo Fisher Scientific | R0392 | Induce protein expression |
| LB Broth (Miller) | Sigma | L3522 | Microbial growth medium |
| PCR instrument | BIO-RAD | S1000 Thermal Cycler | Amplification/PCR |
| PD-10 column | Cytiva (GE Healthcare) | 17085101 | Solution exchange |
| pGEX-6P-1 vector | Cytiva (GE Healthcare) | 28954648 | Plasmid |
| pGEX-6P-2 vector | Cytiva (GE Healthcare) | 28954650 | Plasmid |
| Plate reader | Perkin Elmer | EnSpire 2300 Multilabel Reader | Read alpha plate |
| Plate reader software | Perkin Elmer | EnSpire Manager | Plate reader software |
| Plate reader software protocol | Perkin Elmer | Alpha 384-well Low volume | Use this protocol to read plate |
| PMSF | Sigma | P7626 | Prevent proteolysis during sonication |
| protease inhibitor cocktail | Sigma | S8830 | Prevent proteolysis during sonication |
| Sodium azide | Sigma | S2002 | As a preservative |
| Sodium cyanoborohydride (NaBH3CN) | Sigma | 156159 | Activates matrix for coupling |
| Ten amino acid peptide NH2-EDASRMEEVD-COOH corresponding to amino acids 714-724 of human Hsp90 beta isoform | Peptide 2.0 inc | Synthesize Hsp90 C-terminal peptide | |
| Test-Tube Rotator | LABINCO | Make end-over-end agitation | |
| Tris-HCl | Sigma | 10708976001 | Block unreacted sites of acceptor beads |
| Tween-20 | Sigma | P1379 | Prevent beads aggregation |
| Ultra centrifuge Avanti J-20 XP | Beckman Coulter | Centrifuge to get bacteria cell pellets | |
| Ultrasonic cell disruptor | Microson | Sonicate cells to release protein | |
| Unconjugated acceptor beads | Perkin Elmer | 6762003 | Acceptor beads |
| XCell SureLock Mini-Cell and XCell II Blot Module | Invitrogen | EI0002 | SDS-PAGE |
Referencias
- Muchowski, P. J., Wacker, J. L. Modulation of neurodegeneration by molecular chaperones. Nature Reviews Neuroscience. 6 (1), 11-22 (2005).
- Eckl, J. M., Richter, K. Functions of the Hsp90 chaperone system: lifting client proteins to new heights. International Journal of Biochemistry and Molecular Biology. 4 (4), 157-165 (2013).
- Yuno, A. Clinical evaluation and biomarker profiling of Hsp90 inhibitors. Methods in Molecular Biology. 1709, 426-441 (2018).
- Dutta Gupta, S., Bommaka, M. K., Banerjee, A. Inhibiting protein-protein interactions of Hsp90 as a novel approach for targeting cancer. European Journal of Medicinal Chemistry. 178, 48-63 (2019).
- Pavlov, P. F., Hutter-Paier, B., Havas, D., Windisch, M., Winblad, B. Development of GMP-1 a molecular chaperone network modulator protecting mitochondrial function and its assessment in fly and mice models of Alzheimer's disease. Journal of Cellular and Molecular Medicine. 22 (7), 3464-3474 (2018).
- Young, J. C., Obermann, W. M., Hartl, F. U. Specific binding of tetratricopeptide repeat proteins to the C-terminal 12-kDa domain of hsp90. Journal of Biological Chemistry. 273 (29), 18007-18010 (1998).
- Scheufler, C., et al. Structure of TPR domain-peptide complexes: critical elements in the assembly of the Hsp70-Hsp90 multichaperone machine. Cell. 101 (2), 199-210 (2000).
- Storer, C. L., Dickey, C. A., Galigniana, M. D., Rein, T., Cox, M. B. FKBP51 and FKBP52 in signaling and disease. Trends in Endocrinology & Metabolism. 22 (12), 481-490 (2011).
- Ullman, E. F., et al. Luminescent oxygen channeling immunoassay: measurement of particle binding kinetics by chemiluminescence. Proceedings of the National Academy of Sciences of the United States of America. 91 (12), 5426-5430 (1994).
- Wigle, T. J., et al. Screening for inhibitors of low-affinity epigenetic peptide-protein interactions: an AlphaScreen-based assay for antagonists of methyl-lysine binding proteins. Journal of Biomolecular Screening. 15 (1), 62-71 (2010).
- Guenat, S., et al. Homogeneous and nonradioactive high-throughput screening platform for the characterization of kinase inhibitors in cell lysates. Journal of Biomolecular Screening. 11 (8), 1015-1026 (2006).
- Sabatucci, A., et al. A new methodological approach for in vitro determination of the role of DNA methylation on transcription factor binding using AlphaScreen(R) analysis: Focus on CREB1 binding at hBDNF promoter IV. Journal of Neuroscience Methods. 341, 108720 (2020).
- Mills, N. L., Shelat, A. A., Guy, R. K. Assay Optimization and Screening of RNA-Protein Interactions by AlphaScreen. Journal of Biomolecular Screening. 12 (7), 946-955 (2007).
- Huang, X., et al. A competitive alphascreen assay for detection of hyaluronan. Glycobiology. 28 (3), 137-147 (2018).
- Principles of alphascreen amplified luinescent proximmity homogenous assay. PerkinElmer Life Sciences Available from: https://www.perkinelmer.com/lab-solutions/resources/docs/APP_AlphaScreen_Principles.pdf (2021)
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados