Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Études des interactions chaperonne-cochapéroé à l’aide d’un essai homogène à base de billes
Dans cet article
Résumé
Ce protocole présente une technique pour sonder les interactions protéine-protéine à l’aide de perles de donneur liées au glutathion avec des co-chaperons à motif TPR fusionnés GST et des perles accepteurs couplées à un peptide dérivé de Hsp90. Nous avons utilisé cette technique pour dépister de petites molécules afin de perturber les interactions Hsp90-FKBP51 ou Hsp90-FKBP52 et identifié des inhibiteurs puissants et sélectifs de l’interaction Hsp90-FKBP51.
Résumé
Le ciblage des interactions protéine de choc thermique 90 (Hsp90)-cochaperone offre la possibilité de réguler spécifiquement les processus intracellulaires dépendants de Hsp90. Le pentapeptide MEEVD conservé à la terminaison C de Hsp90 est responsable de l’interaction avec le motif de répétition tétratricopeptide (TPR) des co-chaperons. FK506-binding protein (FKBP) 51 et FKBP52 sont deux co-chaperons similaires à motif TPR impliqués dans des maladies hormono-dépendantes stéroïdiennes avec des fonctions différentes. Par conséquent, l’identification de molécules bloquant spécifiquement les interactions entre Hsp90 et FKBP51 ou FKBP52 offre un potentiel thérapeutique prometteur pour plusieurs maladies humaines. Nous décrivons ici le protocole d’un test homogène de proximité luminescente amplifié pour sonder les interactions entre Hsp90 et ses co-chaperons partenaires FKBP51 et FKBP52. Tout d’abord, nous avons purifié les protéines FKBP51 et FKBP52 contenant des motifs TPR sous forme marquée glutathion S-transférase (GST). En utilisant les perles de donneur liées au glutathion avec des protéines À motif TPR fusionnées à la TPS et les perles accepteurs couplées à un peptide C-terminal 10-mer de Hsp90, nous avons sondé les interactions protéine-protéine dans un environnement homogène. Nous avons utilisé ce test pour dépister de petites molécules afin de perturber les interactions Hsp90-FKBP51 ou Hsp90-FKBP52 et identifié des inhibiteurs puissants et sélectifs de l’interaction Hsp90-FKBP51.
Introduction
Les chaperons moléculaires contribuent à l’homéostasie des protéines, y compris le repliement, le transport et la dégradation des protéines. Ils régulent plusieurs processus cellulaires et sont liés à de nombreuses maladies telles que le cancer et les maladies neurodégénératives1. La protéine de choc thermique 90 (Hsp90) est l’un des chaperons les plus importants dont la fonction dépend des changements conformationnels entraînés par l’hydrolyse de l’ATP et la liaison avec les protéines clientes médiées par ses co-chaperons2. Malgré un potentiel évident de Hsp90 en tant que cible thérapeutique, affiner sa fonction représente un grand défi. Il existe plusieurs inhibiteurs de Hsp90 ciblant la région de liaison de l’ATP N-terminal, qui ont été évalués dans des essais cliniques, mais aucun d’entre eux n’a été approuvé pour lacommercialisation 3. En raison de l’absence d’une poche de liaison au ligand4bien définie, le ciblage de la région C-terminale de Hsp90 a eu un succès limité4. Récemment, la perturbation des interactions Hsp90-cochaperone par de petites molécules a été étudiée comme stratégie alternative5. Le ciblage des interactions Hsp90-cochaperone ne provoquerait pas de réponse générale au stress cellulaire et offre la possibilité de réguler spécifiquement divers processus intracellulaires. Le pentapeptide MEEVD conservé à la terminaison C de Hsp90 est responsable de l’interaction avec le motif de répétition tétratricopeptide (TPR) des co-chaperons6. Sur les 736 protéines contenant des motifs TPR annotées dans la base de données des protéines humaines, environ 20 protéines différentes interagissent avec Hsp90 via ce peptide7. Les molécules en concurrence pour la liaison peptidique MEEVD perturberaient les interactions entre Hsp90 et les co-chaperons contenant un domaine TPR. Le site de liaison peptidique a une structure tertiaire similaire, mais l’homologie globale entre les différents domaines du motif TPR est relativement faible7,ce qui offre la possibilité d’identifier des molécules spécifiquement capables de bloquer les interactions entre Hsp90 et des co-chaperons particuliers à motif TPR. Parmi ces co-chaperons à motif TPR, la protéine de liaison FK506 (FKBP) 51 et FKBP52 sont des régulateurs de la signalisation des récepteurs hormonaux stéroïdiens (SHR) et impliqués dans plusieurs maladies hormono-dépendantes des stéroïdes, y compris le cancer, les maladies liées au stress, les maladies métaboliques et la maladie d’Alzheimer8. Bien que FKBP51 et FKBP52 partagent > similitude de séquence de 80%, leurs fonctions diffèrent: FKBP52 est un régulateur positif de l’activité SHR, tandis que FKBP51 est un régulateur négatif dans la plupart des cas8. Par conséquent, l’identification de molécules, bloquant spécifiquement les interactions entre Hsp90 et FKBP51 ou FKBP52, offre un potentiel thérapeutique prometteur pour les maladies connexes.
Unessai amplifié Luminescent Proximity Homogenous A(AlphaScreen) a été développé pour la première fois en 1994 par Ullman EF et al.9. Maintenant, il est largement utilisé pour détecter différents types d’interactions biologiques, telles que lepeptide 10,la protéine11,l’ADN12,l’ARN13et le sucre14. Dans cette technique, il existe deux types de perles (diamètre 200 nm), l’une est la perle donneuse et l’autre est la perle accepteuse. Les biomolécules sont immobilisées sur ces perles; leurs interactions biologiques rapprochent les perles donneuses et accepteuses. À 680 nm, un photosensibilisateur dans la perle du donneur éclaire et convertit l’oxygène en oxygène syté. Parce que l’oxygène syt a une courte durée de vie, il ne peut diffuser que jusqu’à 200 nm. Si la bille accepteur est à proximité, son dérivé de thioxène réagit avec l’oxygène singulet générant une chimiluminescence à 370 nm. Cette énergie active en outre les fluorophores dans la même bille accepteur pour émettre de la lumière à 520-620 nm15. Si les interactions biologiques sont perturbées, la perle accepteur et la perle donneuse ne peuvent pas atteindre la proximité, ce qui entraîne la désintégration de l’oxygène syulet et un faible signal produit.
Nous décrivons ici un protocole utilisant cette technique pour le criblage de petites molécules inhibant les interactions entre Hsp90 et les co-chaperons TPR, en particulier FKBP51 et FKBP52. Les 10 peptides longs d’acides aminés correspondant à Hsp90 extrême C-terminus sont attachés à des billes acceptrices. Les co-chaperons TPR purifiés marqués de la TPS interagissent avec les perles de donneurs liées au glutathion. Lorsque l’interaction entre les peptides dérivés de Hsp90 et les co-chaperons à motif TPR rassemble les billes, un signal amplifié est produit(Figure 1A). Si les petites molécules criblées peuvent inhiber les interactions entre Hsp90 et les co-chaperons à motif TPR, ce signal amplifié sera diminué(Figure 1B). Leur IC50 peut être calculé par mesure quantitative. Ce protocole peut être étendu à toutes les interactions chaperon -TPR-motif co-chaperon d’intérêt et est d’une grande importance dans le développement de nouvelles molécules, bloquant spécifiquement l’interaction entre Hsp90 et FKBP51 ou FKBP52.

Figure 1: Principe de base de ce test. (A) La GST-FKBP51 purifiée interagit avec les billes de donneur liées au glutathion. Les 10 peptides longs d’acides aminés correspondant à l’extrême C-terminus de Hsp90 sont attachés à des billes acceptrices. L’interaction entre les peptides dérivés de Hsp90 et le domaine TPR de FKBP51 rapproche les perles donneuses et acceptrices. À 680 nm, un photosensibilisateur dans la perle du donneur éclaire et convertit l’oxygène en oxygène syté. Le dérivé de thioxène sur la bille accepteur réagit avec l’oxygène syt et génère une chimiluminescence à 370 nm. Cette énergie active en outre les fluorophores dans la même bille accepteur pour émettre de la lumière à 520-620 nm. (B) Lorsque de petites molécules inhibent les interactions entre Hsp90 et FKBP51, les perles donneuses et acceptatrices ne peuvent pas atteindre la proximité. Ensuite, l’oxygène syt avec une courte durée de vie se désintègre et aucun signal détectable n’est produit. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Remarque : Une vue d’ensemble de ce protocole est illustrée à la figure 2.
1. Expression et purification de la TPS-FKBP51 et de la TPS-FKBP52 (Figure 2A)
-
Plasmides
REMARQUE: Obtenez des clones d’ADNc pour FKBP51 humain (id de clone: 5723416) et pour FKBP52 humain (ID de clone: 7474554) auprès du consortium IMAGE.- Amplifier l’ADN FKBP51 humain par PCR avec des amorces (en avant; 5'GGATCCATGACTACTGATGAAGGT-3', inverse; 5'-CTCGAGCTATGCTTCTCTCCAC-3') contenant des surplombs BamHI et XhoI et cloner en vecteur pGEX6-1 sur les sites de restriction BamHI / XhoI.
- Amplifier l’ADN FKBP52 humain par PCR avec des amorces (en avant; 5'-GAATTCATGACAGCCGAGGAGATG-3', inverse; 5'-CTCGAGCTATGCTTCTCTCCAC-3') contenant des surplombs EcoRI et XhoI et cloner en vecteur pGEX6-2 sur les sites de restriction EcoRI / XhoI.
REMARQUE: La configuration et les conditions de la réaction par PCR sont indiquées dans les tableaux 1 et 2. - Vérifier la séquence insérée et transformer les plasmides en E. coli chimiquement compétent selon le protocole de fabrication.
-
Expression et purification des protéines
- Ajouter 25 g de bouillon Luria (LB) dans 1 L d’eau distillée pour faire la solution LB. Autoclavez-le à 121 °C pendant 15 min. Après refroidissement, ajouter 50 μg/mL d’ampicilline.
- Prendre une colonie de bactéries exprimant GST-FKBP51 ou GST-FKBP52 et mélanger avec 500 μL de solution LB dans un tube de 1,5 mL. Tourbillon.
- Ajouter le mélange de « 1.2.2 » dans 1 L de solution LB dans la fiole Erlenmeyer recouverte d’une feuille d’aluminium. Incuber la fiole d’Erlenmeyer dans le shaker pendant la nuit à 37 °C.
- Induire l’expression des protéines en ajoutant 1 mM d’isopropyl-β-D-thiogalactoside (IPTG) à la fiole d’Erlenmeyer et poursuivre l’incubation pendant encore 2 h.
- Pour obtenir des pastilles de cellule, centrifuger à 5 000 x g pendant 15 min. Retirez le surnageant.
REMARQUE: Les granulés de cellule peuvent être stockés à -20 ° C. - Resuspendez les pastilles cellulaires dans 40 mL de PBS et soniquez 3 x 20 s sur de la glace. Ajouter 1 mM pmSF, 1 mM EDTA et un cocktail d’inhibiteurs de protéase (1 comprimé) pour prévenir la protéolyse.
- Centrifuger la suspension pendant 30 min 50 000 x g pour éliminer les débris cellulaires et appliquer le surnageant sur une colonne de piège GST de 5 mL.
- Après avoir lavé la colonne avec 30 mL de PBS, éluez GST-FKBP51 et GST-FKBP52 avec 5 mL de glutathion de 10 mM dans du PBS.
- Protéines concentrées sur une unité de centrifugation de 15 mL 10.000 MWCO. Pour éliminer le glutathion libre, passez les concentrés à travers la colonne PD-10 équilibrés avec 0,5x PBS et concentrez-vous à nouveau sur le dispositif de centrifugeuse filtrante.
- Recueillir les fractions contenant des protéines. Vérifiez les protéines dans SDS-PAGE et ajustez les concentrations de protéines à 1 mg/mL.
REMARQUE: Le rendement en protéines typique est de 2-5 mg / L de culture. La protéine peut être stockée à -20 °C.
2. Couplage du peptide C-terminal Hsp90 aux billes acceptrices (Figure 2B)
-
Préparation peptidique Hsp90
- Synthétiser dixacides aminés peptide NH 2-EDASRMEEVD-COOH correspondant aux acides aminés 714-724 de l’isoforme bêta Hsp90 humaine (UniProt ID: P08238) par un service de synthèse peptidique.
- Diluer le peptide Hsp90 dans le PBS à une concentration de 1 mg/mL.
-
Préparation des perles d’accepteur
- Diluer les billes d’accepteur non conjuguées dans du PBS à une concentration de 1 mg/mL et transférer dans un tube de 1,5 mL.
- Effectuer le lavage par centrifugation à 16 000 x g pendant 15 min. Retirez soigneusement le surnageant.
-
Conjugaison
- Définissez le rapport entre les perles et le peptide sur 10:1. Dans le tube de 1,5 mL contenant 1 mg de pastille de bille accepteur (préparé comme décrit ci-dessus), ajouter 1 mL de PBS (pH 7,4), 0,1 mg de peptide dilué, 1,25 μL de Tween-20, 10 μL d’une solution de cyanoborohydrure de sodium (NaBH3CN) de 400 mM dans l’eau.
ATTENTION : NaBH3CN est toxique; utilisez une hotte et des gants. La solution de NaBH3CN doit être fraîchement préparée. - Incuber pendant 24 h à 37 °C avec agitation bout à bout (10-20 tr/min) sur un agitateur rotatif.
- Définissez le rapport entre les perles et le peptide sur 10:1. Dans le tube de 1,5 mL contenant 1 mg de pastille de bille accepteur (préparé comme décrit ci-dessus), ajouter 1 mL de PBS (pH 7,4), 0,1 mg de peptide dilué, 1,25 μL de Tween-20, 10 μL d’une solution de cyanoborohydrure de sodium (NaBH3CN) de 400 mM dans l’eau.
-
Trempe par réaction et lavage des billes
- Ajouter 20 μL de solution de Tris-HCl (pH 8,0) de 1 M à la réaction pour bloquer les sites qui n’ont pas réagi. Incuber pendant 1 h à 37 °C.
- Centrifuger à 16 000 x g (ou vitesse maximale) pendant 15 min à 4 °C. Retirer le surnageant et resussuspener la pastille de bille dans 1 mL de solution de Tris-HCl (100 mM, pH 8,0).
- Répétez l’étape de lavage trois fois.
- Après la dernière centrifugation, remise en service des billes à 1 mg/mL dans un tampon de stockage (1 mL de 0,5 × PBS avec 0,01 % d’azide de sodium comme agent de conservation). Conserver la solution de billes accepteurs conjuguées à 4 °C à l’abri de la lumière.
ATTENTION: L’azide de sodium est toxique; utilisez une hotte et des gants.
3. Le test sondant l’interaction entre GST-FKBP51 ou GST-FKBP52 et le peptide C-terminal Hsp90, et l’inhibition avec des composés de petite masse moléculaire (Figure 2C)
-
Protéines marquées GST interagissant avec des billes de donneurs de glutathion
- Mettez en place les réactions dans des plaques de 384 puits.
- Préparer la solution contenant 10 μg/mL de billes donneuses de glutathion dans 0,5x PBS, pH 7,4.
REMARQUE: Après un stockage prolongé, les perles se déposent et doivent être tourbillondées. - Ajouter GST-FKBP51 ou GST-FKBP52 à une concentration finale de 10 μg/mL.
- Incuber dans l’obscurité à 25 °C pendant 10 min.
REMARQUE: À cette étape, les protéines marquées GST interagiront avec le glutathion attaché aux perles. Pour chaque puits, 22,5 μL de ce mélange seront utilisés. La concentration des partenaires contraignants doit être déterminée empiriquement. Titrez GST-FKBP51 et GST-FKBP52 et choisissez la concentration qui donne le meilleur signal.
-
Ajout de composé
- Effectuer des dilutions en série de composés d’essai dans le DMSO.
REMARQUE: Les concentrations utilisées sont généralement de 10, 30, 100, 300, 1 000 et 3 000 μM. - Ajouter 0,25 μL de DMSO (contrôle négatif) ou de peptide C-terminal Hsp90 (contrôle positif, 30 μM) ou de composés dans le DMSO au coin de chaque puits de la plaque. Utilisez des triples pour chaque concentration de composé.
- Ajouter 22,5 μL de la solution contenant des perles de donneur de glutathion avec des protéines marquées GST à chaque puits.
- Serrez l’assiette doucement avec la main mais à fond. Incuber dans l’obscurité à 25 °C pendant 15 min.
REMARQUE: Pendant ce temps, les composés interagiront avec le domaine TPR au site de liaison peptidique Hsp90 C-terminal.
- Effectuer des dilutions en série de composés d’essai dans le DMSO.
-
Ajout de perles d’accepteur
- Diluer les billes acceptatrices avec le peptide C-terminal Hsp90 attaché à 100 μg/mL dans 0,5x PBS.
- Ajouter 2,25 μL de billes d’accepteur diluées à chaque puits.
- Mélanger doucement mais bien. Incuber dans l’obscurité à 25 °C pendant 15 min.
REMARQUE: À cette étape, les billes du donneur et de l’accepteur sont rapprochées par les interactions protéine-peptide. Le volume final du mélange réactionnel est de 25 μL. Par conséquent, les concentrations finales de composés varient de 0,1 à 30 μM.
-
Lecture de plaques
REMARQUE: Lisez la plaque à l’aide d’un lecteur de plaque réglé dans le mode approprié.- Allumez l’instrument et ouvrez le logiciel
- Choisissez le protocole approprié.
- Cliquez sur Modifier la carte de la plaque et sélectionnez le puits utilisé dans la plaque pour la mesure.
- Cliquez sur Suivant pour continuer et exécuter le protocole sélectionné.
- Après la mesure, cliquez sur Afficher les résultats pour afficher les résultats.
- Exportez les données.
4. Analyse des données
-
Facteur Z' et rapport signal/arrière-plan (S/B)
- Calculez le facteur Z' et le rapport S/B pour le dosage à l’aide de l’équation suivante :
Z'=1-(3σpos+3σneg)/│μpos-μneg│16
S/B=μneg/μpos
où, σ et μ représentent les écarts-types et les moyennes des témoins positifs (Hsp90 C-terminal peptide, 30 μM) et négatifs (DMSO), respectivement. Un facteur Z' > 0,5 garantira que le test est suffisamment robuste pour le dépistage. Pour surveiller la sensibilité du test, le rapport S / B a également été calculé.
- Calculez le facteur Z' et le rapport S/B pour le dosage à l’aide de l’équation suivante :
-
Courbe dose-réponse et CI 50
REMARQUE: Utilisez une analyse de régression non linéaire pour ajuster les données des inhibiteurs de l’IPP Hsp90-cochaperones par logiciel.- Créez une table de données XY dans la boîte de dialogue Bienvenue et sélectionnez X Numbers, et Y Enter 3 (si triplé) réplique les valeurs dans des colonnes côte à côte.
- Normaliser les données de signal des échantillons au groupe témoin négatif. Importez les valeurs de concentration dans la colonne X et les valeurs de signal dans la colonne Y.
- Cliquez sur Analyser et choisissez Transformer la concentration (X) sous Transformer | Normaliser. Choisissez Transformer en logarithmes.
REMARQUE: Cela transformera la concentration en une échelle logarithmique. Si votre concentration de départ est nulle, réglez-la sur un très petit nombre qui est effectivement nul (par exemple, 0,1 nM) pour ne pas perdre ces valeurs puisque le logarithme de zéro n’est pas défini. - Cliquez sur Analyser et choisissez Régression non linéaire (ajustement de courbe) sous Analyses XY, ouvrez l’inhibition dose-réponse et choisissez Log(inhibiteur) vs réponse -- Pente variable.
- Cliquez sur OK pour afficher les résultats (contenant la valeur IC50) et les graphiques.
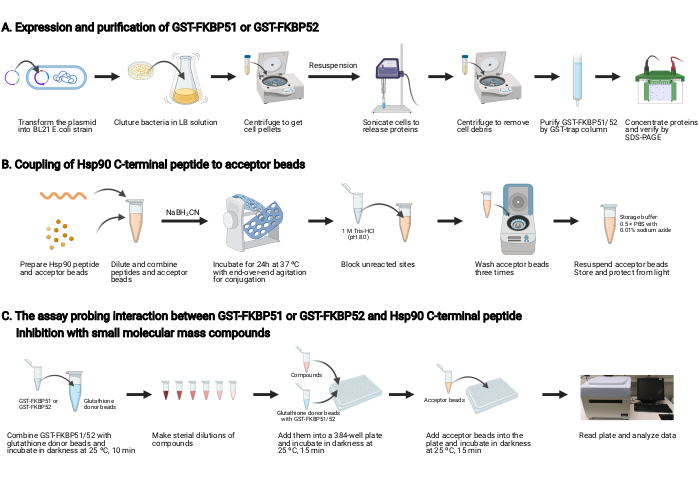
Figure 2: Schéma de ce protocole. (A) Expression et purification de la TPS-FKBP51 et de la TPS-FKBP52. (B) Couplage du peptide C-terminal Hsp90 aux billes acceptrices. (C) Le test sondant l’interaction entre GST-FKBP51 ou GST-FKBP52 et Hsp90 C-terminal peptide. Inhibition avec des composés de petite masse moléculaire. Créé avec BioRender.com Veuillez cliquer ici pour afficher une version agrandie de cette figure.
Résultats
Dans notre essai, le facteur Z' et le rapport S/B sont respectivement de 0,82 et 13,35(figure 3A),ce qui démontre que notre essai est robuste et fiable pour le criblage à haut débit. Nous l’avons ensuite utilisé pour filtrer de petits composés de masse moléculaire. La figure 3B présente l’inhibition dose-dépendante des interactions chaperon-cochaperone avec une petite molécule sélectionnée (D10). Les courbes dose-réponse pour D10 sont générées...
Discussion
Nous décrivons ici un protocole utilisant le test pour le criblage de petites molécules inhibant les interactions entre Hsp90 et les co-chaperons à motif TPR, en particulier FKBP51 et FKBP52. Son score Z' élevé (>0,8) démontre la robustesse et la fiabilité d’un format à haut débit. Les résultats peuvent être obtenus en une heure et de petites quantités de perles, de protéines et de composés sont nécessaires. De plus, ce protocole pourrait facilement être étendu à toutes les interactions de co-chaperon...
Déclarations de divulgation
Les auteurs ne signalent aucun conflit d’intérêts.
Remerciements
Cette étude a été soutenue par des subventions du Conseil suédois de la recherche (2018-02843), de la Brain Foundation (Fo 2019-0140), de la Foundation for Geriatric Diseases du Karolinska Institutet, de la Gunvor and Josef Anérs Foundation, de la Magnus Bergvalls Foundation, de la Gun and Bertil Stohnes Foundation, de la Tore Nilssons Foundation for medical research, de la Margaretha af Ugglas Foundation et de la Foundation for Old Servants.
matériels
| Name | Company | Catalog Number | Comments |
| 384-well plates | Perkin Elmer | 6008350 | Assay volume 25 ml |
| Amicon 10.000 MWCO centrifugation unit | Millipore | UFC901008 | Concentrate protein |
| Ampicillin | Sigma | A0166 | Antibiotics |
| Bacteria shaker Unimax 1010 | Heidolph | Culture bacteria | |
| cDNA clones for human FKBP51 | Source BioScience | clone id: 5723416 | pCMV-SPORT6 vector |
| cDNA clones for human FKBP52 | Source BioScience | clone id: 7474554 | pCMV-SPORT6 vector |
| Chemically Competent E. coli | Invitrogen | C602003 | One Shot BL21 Star (DE3) |
| Data analysis software | GraphPad Prism | 9.0.0 | Analysis data and make figures |
| Data analysis software | Excel | Analysis data | |
| DMSO | Supelco | 1.02952.1000 | Dilute compounds |
| DPBS | Gibco | 14190-144 | Prepare solution |
| EDTA | Calbiochem | 344504 | Prevent proteolysis during sonication |
| Glutathione | Sigma | G-4251 | Elute GST-tagged proteins |
| Glutathione donor beads | Perkin Elmer | 6765300 | Donor bead |
| GST-trap column | Cytiva (GE Healthcare) | 17528201 | Purify GST-tagged proteins |
| Isopropyl-β-D-thiogalactoside | Thermo Fisher Scientific | R0392 | Induce protein expression |
| LB Broth (Miller) | Sigma | L3522 | Microbial growth medium |
| PCR instrument | BIO-RAD | S1000 Thermal Cycler | Amplification/PCR |
| PD-10 column | Cytiva (GE Healthcare) | 17085101 | Solution exchange |
| pGEX-6P-1 vector | Cytiva (GE Healthcare) | 28954648 | Plasmid |
| pGEX-6P-2 vector | Cytiva (GE Healthcare) | 28954650 | Plasmid |
| Plate reader | Perkin Elmer | EnSpire 2300 Multilabel Reader | Read alpha plate |
| Plate reader software | Perkin Elmer | EnSpire Manager | Plate reader software |
| Plate reader software protocol | Perkin Elmer | Alpha 384-well Low volume | Use this protocol to read plate |
| PMSF | Sigma | P7626 | Prevent proteolysis during sonication |
| protease inhibitor cocktail | Sigma | S8830 | Prevent proteolysis during sonication |
| Sodium azide | Sigma | S2002 | As a preservative |
| Sodium cyanoborohydride (NaBH3CN) | Sigma | 156159 | Activates matrix for coupling |
| Ten amino acid peptide NH2-EDASRMEEVD-COOH corresponding to amino acids 714-724 of human Hsp90 beta isoform | Peptide 2.0 inc | Synthesize Hsp90 C-terminal peptide | |
| Test-Tube Rotator | LABINCO | Make end-over-end agitation | |
| Tris-HCl | Sigma | 10708976001 | Block unreacted sites of acceptor beads |
| Tween-20 | Sigma | P1379 | Prevent beads aggregation |
| Ultra centrifuge Avanti J-20 XP | Beckman Coulter | Centrifuge to get bacteria cell pellets | |
| Ultrasonic cell disruptor | Microson | Sonicate cells to release protein | |
| Unconjugated acceptor beads | Perkin Elmer | 6762003 | Acceptor beads |
| XCell SureLock Mini-Cell and XCell II Blot Module | Invitrogen | EI0002 | SDS-PAGE |
Références
- Muchowski, P. J., Wacker, J. L. Modulation of neurodegeneration by molecular chaperones. Nature Reviews Neuroscience. 6 (1), 11-22 (2005).
- Eckl, J. M., Richter, K. Functions of the Hsp90 chaperone system: lifting client proteins to new heights. International Journal of Biochemistry and Molecular Biology. 4 (4), 157-165 (2013).
- Yuno, A. Clinical evaluation and biomarker profiling of Hsp90 inhibitors. Methods in Molecular Biology. 1709, 426-441 (2018).
- Dutta Gupta, S., Bommaka, M. K., Banerjee, A. Inhibiting protein-protein interactions of Hsp90 as a novel approach for targeting cancer. European Journal of Medicinal Chemistry. 178, 48-63 (2019).
- Pavlov, P. F., Hutter-Paier, B., Havas, D., Windisch, M., Winblad, B. Development of GMP-1 a molecular chaperone network modulator protecting mitochondrial function and its assessment in fly and mice models of Alzheimer's disease. Journal of Cellular and Molecular Medicine. 22 (7), 3464-3474 (2018).
- Young, J. C., Obermann, W. M., Hartl, F. U. Specific binding of tetratricopeptide repeat proteins to the C-terminal 12-kDa domain of hsp90. Journal of Biological Chemistry. 273 (29), 18007-18010 (1998).
- Scheufler, C., et al. Structure of TPR domain-peptide complexes: critical elements in the assembly of the Hsp70-Hsp90 multichaperone machine. Cell. 101 (2), 199-210 (2000).
- Storer, C. L., Dickey, C. A., Galigniana, M. D., Rein, T., Cox, M. B. FKBP51 and FKBP52 in signaling and disease. Trends in Endocrinology & Metabolism. 22 (12), 481-490 (2011).
- Ullman, E. F., et al. Luminescent oxygen channeling immunoassay: measurement of particle binding kinetics by chemiluminescence. Proceedings of the National Academy of Sciences of the United States of America. 91 (12), 5426-5430 (1994).
- Wigle, T. J., et al. Screening for inhibitors of low-affinity epigenetic peptide-protein interactions: an AlphaScreen-based assay for antagonists of methyl-lysine binding proteins. Journal of Biomolecular Screening. 15 (1), 62-71 (2010).
- Guenat, S., et al. Homogeneous and nonradioactive high-throughput screening platform for the characterization of kinase inhibitors in cell lysates. Journal of Biomolecular Screening. 11 (8), 1015-1026 (2006).
- Sabatucci, A., et al. A new methodological approach for in vitro determination of the role of DNA methylation on transcription factor binding using AlphaScreen(R) analysis: Focus on CREB1 binding at hBDNF promoter IV. Journal of Neuroscience Methods. 341, 108720 (2020).
- Mills, N. L., Shelat, A. A., Guy, R. K. Assay Optimization and Screening of RNA-Protein Interactions by AlphaScreen. Journal of Biomolecular Screening. 12 (7), 946-955 (2007).
- Huang, X., et al. A competitive alphascreen assay for detection of hyaluronan. Glycobiology. 28 (3), 137-147 (2018).
- Principles of alphascreen amplified luinescent proximmity homogenous assay. PerkinElmer Life Sciences Available from: https://www.perkinelmer.com/lab-solutions/resources/docs/APP_AlphaScreen_Principles.pdf (2021)
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon