このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
均質ビーズベースアッセイを用いたシャペロンコカペロン相互作用の研究
要約
このプロトコルは、グルタチオン結合ドナービーズとGST融合TPR-モチーフコシャペロンおよびアクセプタービーズとHsp90由来ペプチドを結合してタンパク質とタンパク質相互作用を調べるための技術を提示する。この技術を用いて、Hsp90-FKBP51またはHsp90-FkbP52相互作用を破壊する小分子をスクリーニングし、強力で選択的なHsp90-FKBP51相互作用阻害剤を同定しました。
要約
ヒートショックタンパク質90(Hsp90)-コカペロン相互作用を標的とすると、Hsp90依存性細胞内プロセスを特異的に調節する可能性が得る。Hsp90のC末流で保存されたMEEVDペンタペプチドは、コシャペロンのテトラトリコペプチド反復(TPR)モチーフとの相互作用を担う。FK506結合タンパク質(FKBP)51およびFKBP52は、異なる機能を有するステロイドホルモン依存性疾患に関与する2つの類似したTPR-モチーフコシャペロンである。したがって、Hsp90とFKBP51またはFKBP52との間の相互作用を特異的に遮断する分子を同定することは、いくつかのヒト疾患に有望な治療可能性を提供する。ここでは、Hsp90とそのパートナーのコシャペロンFKBP51とFKBP52との相互作用をプローブするための増幅された発光近接均質アッセイのプロトコルについて説明する。まず、TPRモチーフ含有タンパク質FKBP51およびFKBP52をグルタチオンS-トランスファーゼ(GST)タグ付けされた形態で精製しました。GST融合TPR-モチーフタンパク質を有するグルタチオン結合ドナービーズと、Hsp90の10-mer C末端ペプチドと結合したアクセプタービーズを用いて、均質な環境下でタンパク質とタンパク質相互作用を調査した。このアッセイを使用して、Hsp90-FKBP51またはHsp90-FKBP52相互作用を破壊する小分子をスクリーニングし、強力で選択的なHsp90-FKBP51相互作用阻害剤を同定しました。
概要
分子シャペロンは、タンパク質のフォールディング、輸送、分解など、タンパク質ホメオスタシスに寄与します。彼らはいくつかの細胞プロセスを調節し、癌や神経変性疾患などの多数の疾患にリンクされています1.ヒートショックタンパク質90(Hsp90)は、その機能がATP加水分解によって駆動される立体構造変化に依存し、その共シャペロン2によって媒介されるクライアントタンパク質と結合する最も重要なシャペロンの1つである。治療目標としてHsp90の明らかな可能性にもかかわらず、その機能を微調整する大きな課題を表しています。N末端ATP結合領域を標的としたいくつかのHsp90阻害剤は臨床試験で評価されているが、いずれもマーケティングのために承認されていない3。明確に定義されたリガンド結合ポケット4の欠如のために、Hsp90のC末端領域を標的にすることは、限られた成功を持っていた4.近年、低分子によるHsp90-コカペロン相互作用の破壊が代替戦略5として検討されている。Hsp90-コカペロン相互作用を標的とすることは、一般的な細胞ストレス応答を引き出すものではなく、細胞内プロセスを特異的に調節する可能性を提供する。Hsp90のC末流で保存されたMEEVDペンタペプチドは、コシャペロン6のテトラトリコペプチド反復(TPR)モチーフとの相互作用を担う。ヒトタンパク質データベースにアポイントトされた736 TPRモチーフ含有タンパク質のうち、〜20種類の異なるタンパク質がこのペプチド7を介してHsp90と相互作用する。MEEVDペプチド結合を競合する分子は、TPRドメインを含むHsp90とコシャペロンとの相互作用を破壊するであろう。ペプチド結合部位は類似した三次構造を有するが、異なるTPRモチーフドメイン間の全体的な相同性は比較的低い7であり、Hsp90と特定のTPR-モチーフコシャペロンとの間の相互作用を特異的に遮断し得る分子を同定する機会を提供する。これらのTPR-モチーフコシャペロンの中でも、FK506結合タンパク質(FKBP)51およびFKBP52は、ステロイドホルモン受容体(SHR)シグナル伝達の調節因子であり、癌、ストレス関連疾患、代謝性疾患、およびアルツハイマー病8を含むいくつかのステロイドホルモン依存性疾患に関与する。FKBP51とFKBP52は80%の配列類似性>共有するが、それらの機能は異なる:FKBP52はSHR活性の肯定的な調節因子であり、FKBP51はほとんどの場合8において負の調節因子である。したがって、分子を同定し、Hsp90とFKBP51またはFKBP52との間の相互作用を特異的に遮断し、関連疾患に有望な治療可能性を提供する。
Mplified Luminescent PロキシミティH眼用Assay(アルファスクリーン)は、1994年にウルマンEFららによって最初に開発されました。現在では、ペプチド10、タンパク質11、DNA12、RNA13、糖14など、さまざまな種類の生物学的相互作用を検出するために広く使用されています。この技術では、ビーズの2種類(直径200nm)があり、一方がドナービーズ、もう1つがアクピコンドールビーズである。生体分子はこれらのビーズに固定化されています。彼らの生物学的相互作用は、ドナーとアクセクタービーズを近接させる。680 nmでは、ドナービーズの光化剤が酸素を点灯し、酸素を単一酸素に変換します。単一酸素は寿命が短いため、最大200nmまで拡散することしかできません。アクセクサビーズが近接している場合、そのチオキセン誘導体は、370nmで化学発光を生成する単一の酸素と反応する。このエネルギーは、520-620 nm15で発光する同じアクセプタビーズ内の蛍光体をさらに活性化する。生物学的相互作用が中断された場合、アクセプタービーズとドナービーズは近接に達することができず、その結果、単一の酸素崩壊および低生成信号が生じる。
ここでは、Hsp90とTPRコシャペロン、特にFKBP51およびFKBP52との相互作用を阻害する小分子をスクリーニングするためのこの技術を用いたプロトコルについて説明する。Hsp90極限C末端に対応する10アミノ酸長ペプチドは、アクセプタービーズに結合している。精製されたGSTタグ付きTPRコシャペロンは、グルタチオン結合ドナービーズと相互作用する。Hsp90由来ペプチドとTPR-モチーフコシャペロンとの相互作用がビーズを一緒に持ち込むとき、増幅シグナルが生成される(図1A)。スクリーニングされた小分子がHsp90とTPR-motifコシャペロンとの相互作用を阻害できる場合、この増幅シグナルは減少する(図1B)。それらのIC50 は、定量測定により算出することができる。このプロトコルは、任意のシャペロンに拡張することができます - TPR-モチーフコシャペロン相互作用は、関心のある、新しい分子の開発において非常に重要であり、特にHsp90とFKBP51またはFKBP52間の相互作用を遮断する。

図1: 本アッセイの基本原理( A)精製 GST-FKBP51 はグルタチオン結合ドナービーズと相互作用する。Hsp90の極度C末端に対応する10アミノ酸長ペプチドは、アクセプタービーズに結合されている。FKBP51のHsp90由来ペプチドとTPRドメインとの相互作用は、ドナーとアクセプタービーズを近接させます。680 nmでは、ドナービーズの光化剤が酸素を点灯し、酸素を単一酸素に変換します。アクセプティクサビーズ上のチオキセン誘導体は、一重の酸素と反応し、370 nmで化学発光を生成します。このエネルギーは、520-620 nmで発光するために、同じアクセプタービーズ内のフルオロフォアをさらに活性化します。(B)小分子がHsp90とFKBP51の相互作用を阻害すると、ドナーとアクセプタービーズは近接に達することができない。その後、短い寿命の単一の酸素が崩壊し、検出可能な信号が生成されません。 この図の大きなバージョンを表示するには、ここをクリックしてください。
プロトコル
注 : このプロトコルの概要を 図 2に示します。
1. GST-FKBP51およびGST-FKBP52の発現と精製 (図2A)
-
プラスミド
注:画像コンソーシアムから、ヒトFKBP51(クローンID:5723416)およびヒトFKBP52(クローンID:7474554)のcDNAクローンを取得します。- バミヒとXhoI制限部位でpGEX6-1ベクターにオーバーハングし、クローンを含むプライマー(前方;5'GGATATATACTGATGCGAAGGT-3'、逆;5'CTCCTCTATTTCTCTCTCTCTCCACCAC CAC CAC-3')を用いてヒトFKBP51 DNAを増幅する。
- プライマーを用いたPCR(フォワード;5'GAATCATGACAGGCGAGATG-3'、逆;5'-CTCGAGCTATTTCTCTCTCTCCACCAC CAC CAC-3')を含むエコRIとXhoIがオーバーハングし、エコRI/ XhoI制限部位でpGEX6-2ベクターにクローン化することにより、ヒトFKBP52 DNAを増幅する。
注: PCR 反応のセットアップと条件を 表 1 および 表 2に示します。 - 挿入されたシーケンスを確認し、製造プロトコルに従って、化学的に有能な 大腸菌にプラスミドを変換します。
-
タンパク質の発現と精製
- 1 Lの蒸留水に25gのルリアスープ(LB)ベースを加えて、LB溶液を作ります。121°Cで15分間オートクレーブします。冷却後、50 μg/mL アンピシリンを加えます。
- GST-FKBP51またはGST-FKBP52を発現する細菌のコロニーを取り、1.5 mLチューブ内のLB溶液500 μLと混合します。渦動。
- 「1.2.2」の混合物を、アルミホイルで覆われたエルレンマイヤーフラスコのLB溶液1 Lに加えます。37°Cで一晩シェーカーにエルレンマイヤーフラスコをインキュベートします。
- 1 mM イソプロピルβ-D-チオガラクトシド(IPTG)をエルレンマイヤーフラスコに添加してタンパク質発現を誘導し、さらに2時間インキュベーションを続けます。
- 細胞ペレットを得るために、遠心分離機で5,000 x g で15分間。上清を取り除く。
メモ:細胞ペレットは-20°Cで保存することができます。 - 細胞ペレットを40 mLのPBSで再懸濁し、氷の上で3 x 20 sの超音波処理を行います。1 mM PMSF、1 mM EDTA、プロテアーゼ阻害剤カクテル(1錠)を加え、タンパク質分解を防止します。
- 30分間の懸濁液を50,000 x g の遠心分離して、細胞の破片を除去し、上清を5 mL GSTトラップカラムに塗布します。
- 30 mL PBS でカラムを洗浄した後、PBS で 10 mM グルタチオンの 5 mL で GST-FKBP51 および GST-FKBP52 を溶出します。
- タンパク質を15 mL 10.000 MWCO遠心分離ユニットに濃縮します。遊離グルタチオンを除去するために、0.5x PBSで平衡化されたPD-10カラムを通して濃縮物を通過し、再度フィルター遠心装置に集中する。
- タンパク質含有分画を収集する。SDS-PAGEでタンパク質を確認し、タンパク質濃度を1mg/mLに調整します。
注:典型的なタンパク質収量は2〜5mg/ L培養です。タンパク質は-20°Cで保存することができる。
2. Hsp90 C末端ペプチドとアクセプタビーズの結合 (図2B)
-
Hsp90ペプチド製剤
- ペプチド合成サービスにより、ヒトHsp90βアイソフォームのアミノ酸714-724(ユニプロトID:P08238)に対応する10アミノ酸ペプチドNH2-EDASRMEEVD-COOHを合成する。
- Pbs中のHsp90ペプチドを1mg/mL濃度に希釈します。
-
アクソータビーズの準備
- PBSで非コンジュゲートアクシーバタービーズを1mg/mL濃度に希釈し、1.5 mLチューブに移します。
- 16,000xgで15分間遠心分離による洗浄を行います。上清を慎重に取り除いてください。
-
活用
- ビーズとペプチドの比率を10:1に設定します。1 mgのアクセプタービーズペレット(上記のように調製)を含む1.5mLチューブに、PBS(pH 7.4)を1 mL、希釈ペプチドの0.1mg、Tween-20の1.25 μL、シアノボロヒドリドナトリウム(NaBH3 CN)の400mM溶液の10μLを水中に加えます。
注意: NaBH3CN は有毒です。ヒュームフードと手袋を使用してください。NaBH3CN溶液は、新たに調製する必要があります。 - 回転式シェーカーでエンドオーバー攪拌(10-20 rpm)で37°Cで24時間インキュベートします。
- ビーズとペプチドの比率を10:1に設定します。1 mgのアクセプタービーズペレット(上記のように調製)を含む1.5mLチューブに、PBS(pH 7.4)を1 mL、希釈ペプチドの0.1mg、Tween-20の1.25 μL、シアノボロヒドリドナトリウム(NaBH3 CN)の400mM溶液の10μLを水中に加えます。
-
反応焼入れとビーズ洗浄
- 反応反応に1 M Tris-HCl(pH 8.0)溶液20μLを加え、未反応部位をブロックします。37°Cで1時間インキュベートする。
- 遠心分離機 16,000 x g (または最高速度) で 15 分 4 °C で 15 分. 上清を取り除き、トリス-HCl溶液(100 mM、pH 8.0)の1 mLでビーズペレットを再懸濁します。
- 洗浄工程を3回繰り返します。
- 最後の遠心分離の後、保存バッファー内の1 mg/mLでビーズを再懸濁します(0.5 ×PBSの1 mL、防腐剤として0.01%アジドナトリウムを使用)。コンジュゲートアクセプタービーズ溶液を4°Cの光で保護して保管してください。
注意:アジドナトリウムは有毒です。ヒュームフードと手袋を使用してください。
3. GST-FKBP51またはGST-FKBP52とHsp90 C末端ペプチドとの相互作用を調べたアッセイと、小分子質量化合物による阻害(図2C)
-
グルタチオンドナービーズと相互作用するGSTタグ付きタンパク質
- 384ウェルプレートに反応を設定します。
- 0.5x PBS、pH 7.4でグルタチオンドナービーズの10 μg/mLを含む溶液を調製します。
注:長期保存後、ビーズは落ち着き、渦を起こす必要があります。 - GST-FKBP51またはGST-FKBP52を最終濃度10 μg/mLに追加します。
- 暗闇の中で25°Cで10分間インキュベートします。
注:このステップでは、GSTタグ付きタンパク質はビーズに付着したグルタチオンと相互作用します。各ウェルに対して、この混合物の22.5 μLが使用されます。結合パートナーの濃度は経験的に決定されなければならない。GST-FKBP51とGST-FKBP52を活性化し、最良の信号を与える濃度を選択します。
-
化合物の追加
- DMSOでテスト化合物のシリアル希釈を行います.
注意:使用濃度は通常10、30、100、300、1,000、3,000μMです。 - DMSO(陰性対照)またはHsp90 C末端ペプチド(陽性対照、30μM)またはDMSO中の化合物をプレートの各ウェルの隅に0.25 μL加えます。すべての化合物濃度に三つ子を使用してください。
- グルタチオンドナービーズを含む溶液を22.5 μL添加し、各ウェルにGSTタグ付きタンパク質を添加します。
- 手でやさしくプレートを振るが、徹底的に。25°Cの暗闇の中で15分間インキュベートします。
注:この間、化合物はHsp90 C末端ペプチド結合部位でTPRドメインと相互作用する。
- DMSOでテスト化合物のシリアル希釈を行います.
-
アクソータビーズの追加
- 取り付けられたHsp90 C末端ペプチドを使用してアクセプタービーズを0.5x PBSで100 μg/mLに希釈します。
- 各ウェルに希釈アクパクタービーズ2.25 μLを加えます。
- やさしく、しかし徹底的に混ぜます。25°Cの暗闇の中で15分間インキュベートします。
注:このステップでは、ドナーとアクセプタービーズは、タンパク質とペプチドの相互作用によって近接します。反応混合物の最終容積は25μLである。したがって、化合物の最終的な濃度は0.1から30 μMの範囲です。
-
プレート読み取り
注: 関連するモードでセットされたプレートリーダーを使用してプレートを読み取ります。- 機器の電源を入れ、ソフトウェアを開きます
- 該当するプロトコルを選択します。
- [ プレート マップを編集] をクリックし、測定用のプレートで使用されているウェルを選択します。
- [ 次へ ] をクリックして続行し、選択したプロトコルを 実行 します。
- 測定後、[ 結果の表示 ] をクリックして結果を表示します。
- データをエクスポートします。
4. データ分析
-
Z'ファクターと信号対背景(S/B)比
- 次の式を使用して、アッセイのZ'係数とS/B比を計算します。
Z'=1-(3σpos+3σ neg)/│μポス-μネグ│16
S/B=μネグ/μポス
ここで、σとμは、それぞれ正の標準偏差と平均(Hsp90 C末端ペプチド、30μM)と陰性(DMSO)コントロールを表します。0.5>Z'因子は、アッセイがスクリーニングに十分に堅牢であることを保証します。アッセイ感度を監視するために、S/B比も算出されています。
- 次の式を使用して、アッセイのZ'係数とS/B比を計算します。
-
線量応答曲線とIC 50
注意:ソフトウェアによるHsp90-コカペロンPPI阻害剤のデータに適合するために、非線形回帰分析を使用してください。- [ようこそ] ダイアログで XY データ テーブルを作成し、[X番号] を選択し、[Y Enter 3]を選択します。
- サンプルの信号データを陰性制御グループに正規化します。濃度値を X 列に、シグナル値を Y 列にインポートします。
- [ 解析] をクリックし、[ 変換]|の下で[変換濃度(X)]を 選択 します。正規化.[ 対数に変換]を選択します。
注: これは、濃度を対数スケールに変換します。開始濃度がゼロの場合は、ゼロの対数が定義されていないので、これらの値を失わないように、実質的にゼロ(例えば0.1 nM)の非常に小さな数値に設定します。 - [分析] をクリックし、[XY 分析] の下で[非線形回帰 (曲線適合) ]を選択し、[線量応答阻害] を開いて[Log(阻害剤)対応答 ] - [変数勾配] を選択します。
- [OK] をクリックして[結果](IC50値を含む)とグラフを表示します。
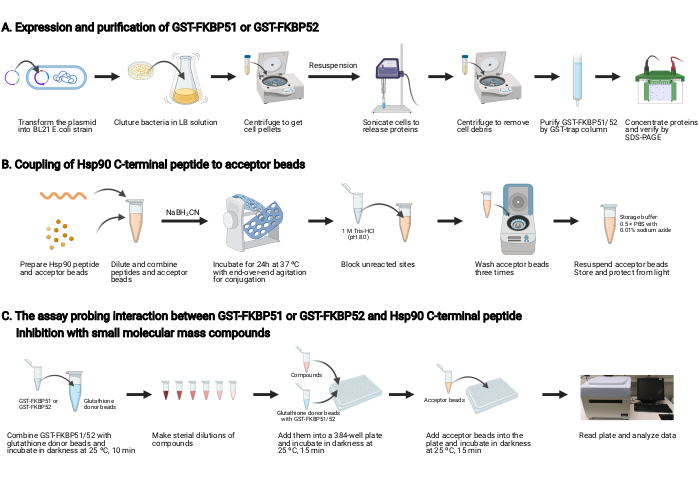
図2: 本プロトコルの概略図 (A) GST-FKBP51 および GST-FKBP52 の発現と精製(B)アクセプタビーズへのHsp90 C末端ペプチドのカップリング。(C)GST-FKBP51またはGST-FKBP52とHsp90 C末端ペプチドとの相互作用を調査するアッセイ。小分子質量化合物による阻害BioRender.com で作成された この図の大きなバージョンを表示するには、ここをクリックしてください。
結果
アッセイでは、Z'因子とS/B比はそれぞれ0.82と13.35(図3A)であり、当社のアッセイは、高スループットスクリーニングに対して堅牢で信頼性が高いことを示しています。その後、それを使用して小さな分子質量化合物をスクリーニングしました。 図3B は、選択された小分子とのシャペロンコカペロン相互作用の用量依存的阻害(D10)を示す。D10の線量応?...
ディスカッション
ここでは、Hsp90とTPR-モチーフコシャペロン、特にFKBP51およびFKBP52との相互作用を阻害する小分子をスクリーニングするためのアッセイを用いたプロトコルについて説明する。高いZ'スコア(>0.8)は、ハイスループット形式の堅牢性と信頼性を示しています。結果は1時間以内に得ることができ、少量のビーズ、タンパク質および化合物が必要とされる。さらに、このプロトコルは、関心のある任?...
開示事項
著者は利益相反を報告しません。
謝辞
この研究は、スウェーデン研究評議会(2018-02843)、ブレイン財団(Fo 2019-0140)、カロリンスカ研究所、ガンバーとヨーゼフ・アノール財団、マグヌス・ベルクヴァルス財団、ガンとベルティル・ストーネス財団、トレ・ニルソンズ医学研究財団、トレ・ニルソンズ財団からの助成金によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| 384-well plates | Perkin Elmer | 6008350 | Assay volume 25 ml |
| Amicon 10.000 MWCO centrifugation unit | Millipore | UFC901008 | Concentrate protein |
| Ampicillin | Sigma | A0166 | Antibiotics |
| Bacteria shaker Unimax 1010 | Heidolph | Culture bacteria | |
| cDNA clones for human FKBP51 | Source BioScience | clone id: 5723416 | pCMV-SPORT6 vector |
| cDNA clones for human FKBP52 | Source BioScience | clone id: 7474554 | pCMV-SPORT6 vector |
| Chemically Competent E. coli | Invitrogen | C602003 | One Shot BL21 Star (DE3) |
| Data analysis software | GraphPad Prism | 9.0.0 | Analysis data and make figures |
| Data analysis software | Excel | Analysis data | |
| DMSO | Supelco | 1.02952.1000 | Dilute compounds |
| DPBS | Gibco | 14190-144 | Prepare solution |
| EDTA | Calbiochem | 344504 | Prevent proteolysis during sonication |
| Glutathione | Sigma | G-4251 | Elute GST-tagged proteins |
| Glutathione donor beads | Perkin Elmer | 6765300 | Donor bead |
| GST-trap column | Cytiva (GE Healthcare) | 17528201 | Purify GST-tagged proteins |
| Isopropyl-β-D-thiogalactoside | Thermo Fisher Scientific | R0392 | Induce protein expression |
| LB Broth (Miller) | Sigma | L3522 | Microbial growth medium |
| PCR instrument | BIO-RAD | S1000 Thermal Cycler | Amplification/PCR |
| PD-10 column | Cytiva (GE Healthcare) | 17085101 | Solution exchange |
| pGEX-6P-1 vector | Cytiva (GE Healthcare) | 28954648 | Plasmid |
| pGEX-6P-2 vector | Cytiva (GE Healthcare) | 28954650 | Plasmid |
| Plate reader | Perkin Elmer | EnSpire 2300 Multilabel Reader | Read alpha plate |
| Plate reader software | Perkin Elmer | EnSpire Manager | Plate reader software |
| Plate reader software protocol | Perkin Elmer | Alpha 384-well Low volume | Use this protocol to read plate |
| PMSF | Sigma | P7626 | Prevent proteolysis during sonication |
| protease inhibitor cocktail | Sigma | S8830 | Prevent proteolysis during sonication |
| Sodium azide | Sigma | S2002 | As a preservative |
| Sodium cyanoborohydride (NaBH3CN) | Sigma | 156159 | Activates matrix for coupling |
| Ten amino acid peptide NH2-EDASRMEEVD-COOH corresponding to amino acids 714-724 of human Hsp90 beta isoform | Peptide 2.0 inc | Synthesize Hsp90 C-terminal peptide | |
| Test-Tube Rotator | LABINCO | Make end-over-end agitation | |
| Tris-HCl | Sigma | 10708976001 | Block unreacted sites of acceptor beads |
| Tween-20 | Sigma | P1379 | Prevent beads aggregation |
| Ultra centrifuge Avanti J-20 XP | Beckman Coulter | Centrifuge to get bacteria cell pellets | |
| Ultrasonic cell disruptor | Microson | Sonicate cells to release protein | |
| Unconjugated acceptor beads | Perkin Elmer | 6762003 | Acceptor beads |
| XCell SureLock Mini-Cell and XCell II Blot Module | Invitrogen | EI0002 | SDS-PAGE |
参考文献
- Muchowski, P. J., Wacker, J. L. Modulation of neurodegeneration by molecular chaperones. Nature Reviews Neuroscience. 6 (1), 11-22 (2005).
- Eckl, J. M., Richter, K. Functions of the Hsp90 chaperone system: lifting client proteins to new heights. International Journal of Biochemistry and Molecular Biology. 4 (4), 157-165 (2013).
- Yuno, A. Clinical evaluation and biomarker profiling of Hsp90 inhibitors. Methods in Molecular Biology. 1709, 426-441 (2018).
- Dutta Gupta, S., Bommaka, M. K., Banerjee, A. Inhibiting protein-protein interactions of Hsp90 as a novel approach for targeting cancer. European Journal of Medicinal Chemistry. 178, 48-63 (2019).
- Pavlov, P. F., Hutter-Paier, B., Havas, D., Windisch, M., Winblad, B. Development of GMP-1 a molecular chaperone network modulator protecting mitochondrial function and its assessment in fly and mice models of Alzheimer's disease. Journal of Cellular and Molecular Medicine. 22 (7), 3464-3474 (2018).
- Young, J. C., Obermann, W. M., Hartl, F. U. Specific binding of tetratricopeptide repeat proteins to the C-terminal 12-kDa domain of hsp90. Journal of Biological Chemistry. 273 (29), 18007-18010 (1998).
- Scheufler, C., et al. Structure of TPR domain-peptide complexes: critical elements in the assembly of the Hsp70-Hsp90 multichaperone machine. Cell. 101 (2), 199-210 (2000).
- Storer, C. L., Dickey, C. A., Galigniana, M. D., Rein, T., Cox, M. B. FKBP51 and FKBP52 in signaling and disease. Trends in Endocrinology & Metabolism. 22 (12), 481-490 (2011).
- Ullman, E. F., et al. Luminescent oxygen channeling immunoassay: measurement of particle binding kinetics by chemiluminescence. Proceedings of the National Academy of Sciences of the United States of America. 91 (12), 5426-5430 (1994).
- Wigle, T. J., et al. Screening for inhibitors of low-affinity epigenetic peptide-protein interactions: an AlphaScreen-based assay for antagonists of methyl-lysine binding proteins. Journal of Biomolecular Screening. 15 (1), 62-71 (2010).
- Guenat, S., et al. Homogeneous and nonradioactive high-throughput screening platform for the characterization of kinase inhibitors in cell lysates. Journal of Biomolecular Screening. 11 (8), 1015-1026 (2006).
- Sabatucci, A., et al. A new methodological approach for in vitro determination of the role of DNA methylation on transcription factor binding using AlphaScreen(R) analysis: Focus on CREB1 binding at hBDNF promoter IV. Journal of Neuroscience Methods. 341, 108720 (2020).
- Mills, N. L., Shelat, A. A., Guy, R. K. Assay Optimization and Screening of RNA-Protein Interactions by AlphaScreen. Journal of Biomolecular Screening. 12 (7), 946-955 (2007).
- Huang, X., et al. A competitive alphascreen assay for detection of hyaluronan. Glycobiology. 28 (3), 137-147 (2018).
- Principles of alphascreen amplified luinescent proximmity homogenous assay. PerkinElmer Life Sciences Available from: https://www.perkinelmer.com/lab-solutions/resources/docs/APP_AlphaScreen_Principles.pdf (2021)
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved