Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Explorando el metabolismo energético mitocondrial de esferoides de microtissue 3D individuales utilizando el análisis de flujo extracelular
En este artículo
Erratum Notice
Resumen
Estos protocolos ayudarán a los usuarios a sondear el metabolismo energético mitocondrial en esferoides derivados de líneas celulares de cáncer 3D utilizando el análisis de flujo extracelular Seahorse.
Resumen
Los agregados celulares tridimensionales (3D), denominados esferoides, se han convertido en la vanguardia del cultivo celular in vitro en los últimos años. En contraste con el cultivo de células como monocapas bidimensionales de una sola célula (cultivo 2D), el cultivo celular esferoide promueve, regula y apoya la arquitectura celular fisiológica y las características que existen in vivo, incluida la expresión de proteínas de matriz extracelular, señalización celular, expresión génica, producción de proteínas, diferenciación y proliferación. La importancia del cultivo 3D ha sido reconocida en muchos campos de investigación, incluyendo oncología, diabetes, biología de células madre e ingeniería de tejidos. Durante la última década, se han desarrollado métodos mejorados para producir esferoides y evaluar su función metabólica y destino.
Los analizadores de flujo extracelular (XF) se han utilizado para explorar la función mitocondrial en microtejidos 3D, como los esferoides, utilizando una placa de captura de islotes XF24 o una microplaca esferoide XFe96. Sin embargo, no se han descrito en detalle distintos protocolos y la optimización del sondeo del metabolismo energético mitocondrial en esferoides utilizando la tecnología XF. Este artículo proporciona protocolos detallados para sondear el metabolismo energético mitocondrial en esferoides 3D individuales utilizando microplacas esferoides con el analizador XFe96 XF. Utilizando diferentes líneas celulares de cáncer, se ha demostrado que la tecnología XF es capaz de distinguir entre la respiración celular en esferoides 3D no solo de diferentes tamaños, sino también de diferentes volúmenes, números de células, contenido y tipo de ADN.
Las concentraciones óptimas del compuesto efector mitocondrial de oligomicina, BAM15, rotenona y antimicina A se utilizan para sondear parámetros específicos del metabolismo energético mitocondrial en esferoides 3D. Este documento también discute los métodos para normalizar los datos obtenidos de los esferoides y aborda muchas consideraciones que deben considerarse al explorar el metabolismo de los esferoides utilizando la tecnología XF. Este protocolo ayudará a impulsar la investigación en modelos avanzados de esferoides in vitro .
Introducción
Los avances en los modelos in vitro en la investigación biológica han progresado rápidamente en los últimos 20 años. Tales modelos ahora incluyen modalidades de órgano en un chip, organoides y esferoides de microtejido 3D, todos los cuales se han convertido en un enfoque común para mejorar la traducción entre estudios in vitro e in vivo. El uso de modelos in vitro avanzados, particularmente esferoides, abarca varios campos de investigación, incluida la ingeniería de tejidos, la investigación con células madre, el cáncer y la biología de enfermedades 1,2,3,4,5,6,7 y las pruebas de seguridad, incluida la toxicología genética 8,9,10, la toxicología de nanomateriales 11, 12,13,14, y pruebas de seguridad y eficacia de medicamentos 8,15,16,17,18,19.
La morfología celular normal es crítica para el fenotipo y la actividad biológica. El cultivo de células en esferoides microtisulares 3D permite a las células adoptar una morfología, función fenotípica y arquitectura, más parecida a la observada in vivo pero difícil de capturar con las técnicas clásicas de cultivo celular monocapa. Tanto in vivo como in vitro, la función celular se ve directamente afectada por el microambiente celular, que no se limita a la comunicación y programación celular (por ejemplo, formaciones de unión célula-célula, oportunidades para formar nichos celulares); exposición celular a hormonas y factores de crecimiento en los entornos inmediatos (por ejemplo, exposición a citoquinas celulares como parte de una respuesta inflamatoria); composición de matrices físicas y químicas (por ejemplo, si las células se cultivan en plástico de cultivo de tejidos rígidos o en un entorno de tejido elástico); y lo más importante, cómo el metabolismo celular se ve afectado por la nutrición y el acceso al oxígeno, así como por el procesamiento de productos de desecho metabólicos como el ácido láctico.
El análisis de flujo metabólico es una forma poderosa de examinar el metabolismo celular dentro de sistemas in vitro definidos. Específicamente, la tecnología XF permite el análisis de cambios en vivo y en tiempo real en la bioenergética celular de células y tejidos intactos. Dado que muchos eventos metabólicos intracelulares ocurren del orden de segundos a minutos, los enfoques funcionales en tiempo real son primordiales para comprender los cambios en tiempo real en el flujo metabólico celular en células y tejidos intactos in vitro.
Este artículo proporciona protocolos para cultivar líneas celulares derivadas del cáncer A549 (adenocarcinoma de pulmón), HepG2/C3A (carcinoma hepatocelular), MCF-7 (adenocarcinoma de mama) y SK-OV-3 (adenocarcinoma de ovario) como modelos de esferoides 3D in vitro utilizando enfoques de agregación forzada (Figura 1). También (i) describe en detalle cómo sondear el metabolismo energético mitocondrial de esferoides 3D individuales utilizando el analizador XF Agilent XFe96, (ii) destaca formas de optimizar los ensayos XF utilizando esferoides 3D individuales, y (iii) discute consideraciones y limitaciones importantes de sondear el metabolismo de los esferoides 3D utilizando este enfoque. Lo más importante es que este artículo describe cómo se recopilan los conjuntos de datos que permiten el cálculo de la tasa de consumo de oxígeno (OCR) para determinar la fosforilación oxidativa y, por lo tanto, la función mitocondrial en los esferoides celulares. Aunque no se analiza para este protocolo, la tasa de acidificación extracelular (ECAR) es otro parámetro que se mide junto con los datos de OCR en experimentos XF. Sin embargo, ECAR a menudo se interpreta de manera deficiente o incorrecta a partir de conjuntos de datos XF. Proporcionamos un comentario sobre las limitaciones del cálculo de ECAR siguiendo los enfoques básicos del fabricante de la tecnología.
Access restricted. Please log in or start a trial to view this content.
Protocolo
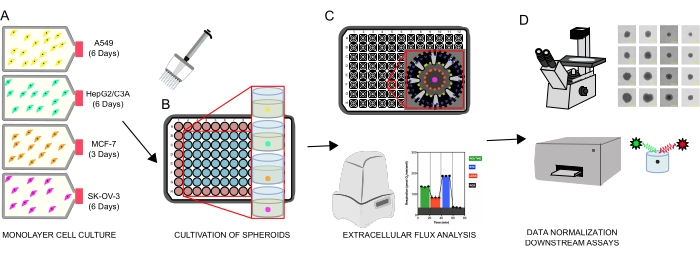
Figura 1: Flujo de trabajo gráfico para la generación de esferoides celulares, análisis de flujo extracelular y ensayos posteriores. Cuatro líneas celulares de cáncer se cultivaron selectivamente como monocapas (A), se separaron de los matraces de cultivo de tejidos y se sembraron en microplacas de 96 pocillos de unión ultrabaja para formar esferoides (B). El carcinoma de pulmón A549, el carcinoma hepático HepG2/C3A, el adenocarcinoma de ovario SK-OV-3 y las células de carcinoma de mama MCF-7 se sembraron a 1 × 103-8 × 103 células/pozo y crecieron hasta 7 días para formar esferoides individuales y optimizar la densidad de siembra de esferoides y el tiempo de cultivo mediante observación continua y mediciones planimétricas. Una vez formados, los esferoides individuales se lavaron en un medio XF libre de suero y se sembraron cuidadosamente en microplacas de ensayo de esferoides, precubiertas con poli-D-lisina (C). Los esferoides se sometieron a análisis de flujo extracelular utilizando el analizador XFe96 utilizando varios protocolos para abordar: (1) el tamaño óptimo de los esferoides para la respuesta de la respiración mitocondrial basal; (2) titulación optimizada de inhibidores respiratorios mitocondriales; (3) optimización de la colocación de esferoides dentro de pozos de microplacas. (D) Se utilizaron análisis posteriores a la XF, microscopía de contraste de fase y cuantificación del ADN esferoide para la normalización de datos y otros ensayos in vitro posteriores. Haga clic aquí para ver una versión más grande de esta figura.
1. Cultivo de líneas celulares de cáncer como esferoides 3D in vitro
| Línea celular | Descripción | Medio de cultivo | Fuente |
| A549 | Línea celular de carcinoma de pulmón | RPMI 1640 | Colección Europea de Cultivos Celulares Autenticados (ECACC) |
| Piruvato de sodio (1 mM) | |||
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| HepG2/C3A | Línea celular de carcinoma hepático, un derivado clonal de la línea celular hepG2 madre | DMEM | Colección Americana de Cultivo de Tejidos (ATCC) |
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| MCF7 | Línea celular de adenocarcinoma mamario | RPMI 1640 | Colección Europea de Cultivos Celulares Autenticados (ECACC) |
| Piruvato de sodio (1 mM) | |||
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| SK-OV-3 | Línea celular de adenocarcinoma ovárico | RPMI 1640 | Colección Europea de Cultivos Celulares Autenticados (ECACC) |
| Piruvato de sodio (1 mM) | |||
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| Componente | Medio de ensayo RPMI (volumen final de 50 ml) | ||
| Base Media | Agilent Seahorse XF RPMI, pH 7.4 | ||
| Glucosa (1 M de stock estéril) | 11 mM (solución madre de 0,55 ml) | ||
| L-glutamina (stock estéril de 200 mM) | 2 mM (0,5 ml de solución madre) | ||
| Piruvato de sodio (stock estéril de 100 mM) | 1 mM (0,5 ml de solución madre) |
Tabla 1: Medios de línea celular de cáncer y composiciones de medios XF.
- Cultive todas las líneas celulares utilizando la técnica estándar de cultivo de tejido aséptico y confirme que están libres de micoplasma utilizando un kit de ensayo adecuado.
- Cultivo de las líneas celulares en matraces de cultivo tisular T75 o equivalentes, utilizando el medio recomendado (Tabla 1). Cultivar las líneas celulares al 65-80% de confluencia y pasarlas regularmente hasta un máximo de 25 pasajes.
- Enjuague los matraces de cultivo celular dos veces en solución salina tamponada con fosfato modificado (DBPS) de Dulbecco.
- Separar las células de los matraces con 3 ml del reactivo de disociación celular (ver la Tabla de Materiales) durante 5 min a 37 °C y confirmar el desprendimiento por microscopía.
- Aspire la suspensión celular desprendida suavemente para asegurar una suspensión unicelular y desactivar el reactivo de disociación celular con 7 ml de medio de cultivo tisular completo.
- Recoger las células por centrifugación a 300 × g durante 5 min, desechar el sobrenadante y volver a suspender las células en medio completo.
- Cuente las células utilizando un hemocitómetro o un contador celular automatizado y titule a la densidad celular deseada requerida para la siembra.
NOTA: Para sembrar una placa entera de 96 pocillos a 100 μL/pocillo a 4 × 103 células/pozo, las células deben ajustarse a 4 × 104 células/ml en un volumen recomendado de 12 ml. - Decantar la suspensión celular en un reservorio estéril y dispensar 100 μL de la suspensión celular en cada pocillo de una microplaca repelente celular utilizando un pipettor multicanal.
NOTA: Solo los 60 pocillos internos de una microplaca deben sembrarse y el resto debe llenarse con DPBS. Esto formará una barrera de evaporación, asegurará la homogeneidad de los esferoides en toda la placa y minimizará los efectos del borde de la placa. - Centrifugar microplacas esferoides a 300 × g durante 15 min para forzar a las células a formar agregados sueltos.
- Incubar las placas a 37 °C, 5% CO2 durante un mínimo de 3 días para asegurar la formación de esferoides.
- Realizar microscopía de contraste de fase utilizando prácticas de laboratorio estandarizadas para monitorear el crecimiento de los esferoides. Reponga el medio de cultivo celular cada 3 días o dos veces por semana realizando un intercambio de medio volumen.
2. Sondeo del metabolismo energético mitocondrial de esferoides individuales utilizando la tecnología de flujo extracelular (XF)
- Preparación del ensayo (un día antes)
- Compruebe la viabilidad de los esferoides utilizando un microscopio de luz invertida con contraste de fase a un aumento de 4x para garantizar la estructura esferoide intacta, la morfología y la uniformidad general entre las muestras.
- Hidrate el cartucho del sensor.
- Alícuota ~ 20 ml del calibrante en un tubo cónico.
- Coloque el tubo cónico que contiene el calibrante en una incubadora sin CO2 a 37 °C durante la noche.
- Retire el contenido del kit de ensayo.
- Retire el cartucho del sensor de la placa de utilidad y colóquelo boca abajo en la encimera junto a la placa de utilidad.
- Pipetear 200 μL de ddH2O estéril en cada pocillo de la placa de utilidad del cartucho del sensor utilizando una pipeta P300 multicanal.
- Coloque el cartucho del sensor en la parte superior de la placa de utilidad.
- Compruebe que el nivel de agua en cada pozo es lo suficientemente alto como para sumergir las sondas del sensor.
- Transfiera el cartucho del sensor ensamblado a una incubadora de 37 °C sin CO2 y déjelo durante la noche.
NOTA: Este paso se puede realizar 12-72 h antes del comienzo del ensayo.
- Microplaca de ensayo de esferoides de capa
- Utilizando técnicas asépticas, añadir 30 μL/pocillo de solución estéril de poli-D-lisina (0,1 mg/ml) a la microplaca esferoide e incubarla durante 30 min a temperatura ambiente.
- Aspire la solución de cada pocillo de la microplaca esferoide, invierta la placa y tóquela firmemente sobre papel de seda para eliminar cualquier solución residual.
- Lavar la placa dos veces con 200 μL/pocillo de ddH2O estéril.
- Después del lavado final, invierta la microplaca y cúbrala firmemente sobre papel de seda para eliminar el agua residual.
- Deje que la placa se seque al aire durante 30 minutos antes de usarla o almacenarla a 4 °C para su uso futuro.
NOTA: La microplaca de ensayo de esferoides debe recubrirse con un adhesivo molecular para garantizar que los esferoides se fijen en la parte inferior de la microplaca. Sin un adhesivo molecular, los esferoides pueden desprenderse e interferir con los resultados del ensayo. Otros adhesivos moleculares también se pueden utilizar como una alternativa a la poli-D-lisina para placas de prerecubrimiento. Las placas preestucados se pueden almacenar a 4 °C, pero deben dejarse equilibrar a temperatura ambiente antes del comienzo del ensayo.
- Preparar el medio de ensayo XF
- Preparar el medio XF RPMI, como se detalla en la Tabla 1, y el filtro estéril con un filtro de jeringa de 0,22 μm
- Preparación del ensayo (1 h antes del ensayo)
- Precalentar el medio de ensayo XF RPMI suplementado a 37 °C.
- Precalentar la microplaca de ensayo de esferoides recubiertos en una incubadora sin CO2 a 37 °C o en un baño seco.
- Prepare el cartucho del sensor.
- Saque el tubo cónico que contiene el calibrante y el cartucho del sensor de la incubadora de aire.
- Retire el cartucho del sensor de la placa de utilidad y colóquelo boca abajo en la superficie de trabajo.
- Usando una pipeta multicanal P300, aspire el agua de la placa de servicio y deséchela.
- Vierta la solución de calibrante en un depósito de reactivo estéril y agregue 200 μL/pocillo del calibrante precalentado a la placa de utilidad utilizando una pipeta multicanal P300.
- Tome el cartucho del sensor y colóquelo de nuevo en la parte superior de la placa de utilidad, asegurándose de que los sensores estén bien sumergidos en el calibrante.
- Vuelva a transferir el cartucho del sensor montado a la incubadora sin CO2 a 37 °C hasta que esté listo para cargar las soluciones de inyección de puerto.
- Lave los esferoides con el medio de ensayo.
- Retire la placa de cultivo de esferoides de la incubadora de 37 °C, 5% de CO2 y observe los esferoides bajo el microscopio para garantizar su integridad antes de los pasos de transferencia de esferoides.
- Cargue todos los pocillos de la placa esferoide con 180 μL/pocillo de medio de ensayo precalentado, incluidos los pozos de corrección de fondo.
- Llene parcialmente una placa de Petri de 7 cm con 3 ml del medio de ensayo.
- Usando una pipeta multicanal cargada con puntas de pipeta de orificio ancho, transfiera los esferoides de la placa de cultivo de 96 pocillos a placas de Petri de 7 cm ajustando el pipeteador a un volumen de aspiración de 10-50 μL.
- Semillas de esferoides en la microplaca de ensayo de esferoides pre-recubierta.
- Usando un microscopio de disección y un aparato de caja de luz, transfiera los esferoides de la placa de Petri a la microplaca de ensayo de esferoides como se detalla a continuación.
- Ajuste el volumen de un pipeteador de un solo canal equipado con una punta de pipeta de orificio ancho a 20 μL y aspire cuidadosamente un solo esferoide. Coloque la punta directamente en el centro de cada pozo de la microplaca de ensayo de esferoides y permita que la gravedad eluya un solo esferoide en el centro de cada pozo, es decir, no expulse ningún medio de la punta de la pipeta y permita que la acción capilar retire el esferoide de la punta de la pipeta. Para confirmar la elución, el contenido del pipeteador se puede volver a pipetear en la placa de Petri de 7 cm bajo el microscopio.
NOTA: La elución por gravedad de un solo esferoide generalmente toma de 15 a 30 s dependiendo del tamaño / densidad del esferoide. Durante este tiempo, el pipeteador no debe ser removido. Cualquier pozo de corrección de fondo debe estar libre de esferoides y solo contener medio de ensayo. Bajo el microscopio, confirme la posición de cada esferoide. Idealmente, cada esferoide debe colocarse dentro del centro de cada pozo. - Una vez que todos los esferoides se hayan transferido a la microplaca de ensayo de esferoides, transfiera la placa a una incubadora sin CO2 a 37 ° C durante un mínimo de 1 h antes del ensayo.
- Ajuste el volumen de un pipeteador de un solo canal equipado con una punta de pipeta de orificio ancho a 20 μL y aspire cuidadosamente un solo esferoide. Coloque la punta directamente en el centro de cada pozo de la microplaca de ensayo de esferoides y permita que la gravedad eluya un solo esferoide en el centro de cada pozo, es decir, no expulse ningún medio de la punta de la pipeta y permita que la acción capilar retire el esferoide de la punta de la pipeta. Para confirmar la elución, el contenido del pipeteador se puede volver a pipetear en la placa de Petri de 7 cm bajo el microscopio.
- Usando un microscopio de disección y un aparato de caja de luz, transfiera los esferoides de la placa de Petri a la microplaca de ensayo de esferoides como se detalla a continuación.
3. Preparación y carga de compuestos en el cartucho del sensor para ensayos XF
| Estrategia de inyección | Compuesto (Puerto) | Volumen de arranque del micropocillo XFe96 (μL) | Concentración de pozo final deseada | Volumen de puerto (μL) | Volumen final de micropocillos XFe96 después de la inyección (μL) | Concentración de existencias de trabajo |
| 1 | Oligomicina (A) | 180 | 3 ug/ml | 20 | 200 | 30 μg/ml |
| Rotenona (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimicina A (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| Rotenona (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimicina A (B) | 200 | 2 μM | 20 | 220 | 22 μM |
Tabla 2: Concentraciones de compuestos mitocondriales para sondear el metabolismo energético mitocondrial de esferoides 3D individuales utilizando el analizador XFe96.
- Prepare las concentraciones de material de trabajo de cada compuesto como se indica en la Tabla 2 utilizando un medio de ensayo XF RPMI totalmente suplementado y precalentado.
- Oriente la placa del cartucho (acoplada a la placa de utilidad) en cuanto a la columna, 1-12 de izquierda a derecha.
- Si utiliza una guía de carga, colóquela encima de la placa del cartucho de acuerdo con el procedimiento de carga del pozo, por ejemplo, si el puerto A se carga primero, asegúrese de que A sea visible en la esquina superior izquierda de la guía.
- Transfiera la solución de trabajo de cada compuesto a un depósito adecuado y, utilizando una pipeta multicanal P100 calibrada, dispense 20 μL en todos los puertos correspondientes. Repita para cada compuesto en los puertos restantes.
NOTA: Si no se utiliza algún puerto en la placa del cartucho del sensor, estos pueden dejarse vacíos o llenarse con un medio de ensayo. Si sólo se utiliza una selección de una carta de puerto específica, asegúrese de que los otros puertos correspondientes a esa letra estén cargados con un medio de ensayo; de lo contrario, se inyectará aire en el pozo, comprometiendo los resultados en esos pozos. - Después de la carga en el puerto, retire las guías de carga de la placa (si se utilizan) y prepare el analizador para cargar el cartucho del sensor.
NOTA: Si el ensayo no se está ejecutando inmediatamente después de cargar los puertos, vuelva a colocar la tapa en el cartucho del sensor y vuelva a colocar la placa en la incubadora de aire a 37 °C hasta que esté lista para cargar en la máquina.
4. Diseño de ensayos, estrategias de inyección y adquisición de datos
- Ejecución del ensayo
- Encienda el analizador y conéctelo al controlador (computadora).
NOTA: Esto se puede verificar mediante el estado de conexión del instrumento en el panel de widgets del software Wave Controller. - Navegue a la página de plantillas en el software WAVE, busque el archivo de plantilla de ensayo para el experimento y haga doble clic para abrirlo.
Nota : si la plantilla de ensayo no aparece en la vista Plantillas , importe el archivo de plantilla en la carpeta de plantilla desde una unidad de red compartida o unidad flash USB. - Para iniciar el ensayo, haga clic en la ficha Ejecutar ensayo .
NOTA: Si las definiciones de grupo se han asignado correctamente dentro del mapa de la placa, el ensayo estará listo para ejecutarse como lo indica la marca verde en el lado derecho de la página. En esta etapa, cualquier información adicional se puede ingresar en la página de resumen del ensayo o en la página que se deja en blanco; continúe con el siguiente paso. Debido a la penetración retardada de moduladores mitocondriales en esferoides microtisulares 3D (Figura 2), utilice la información del protocolo de medición descrita en la Tabla 3.
- Encienda el analizador y conéctelo al controlador (computadora).
| Período de medición | Número de inyección y puerto | Detalles de la medición | Duración del período (h:min:s) |
| Calibración | No aplicable | Los analizadores XF siempre realizan esta calibración para asegurarse de que las mediciones sean precisas | 00:20:00 (esto es un promedio y puede variar entre máquinas) |
| Equillibración | No aplicable | El equilibrio ocurre después de la calibración y se recomienda. | 00:10:00 |
| Basal | No aplicable | Ciclos = 5 | 00:30:00 |
| Mezcla = 3:00 | |||
| Espera = 0:00 | |||
| Medida = 3:00 | |||
| Oligomicina / BAM15 | Inyección 1 (puerto A) | Ciclos = 10 | 01:00:00 |
| Mezcla = 3:00 | |||
| Espera = 0:00 | |||
| Medida = 3:00 | |||
| Rotenona + antimicina A | Inyección 2 (Puerto B) | Ciclos = 10 | 01:00:00 |
| Mezcla = 3:00 | |||
| Espera = 0:00 | |||
| Medida = 3:00 | |||
| Tiempo total: | 03:00:00 | ||
Tabla 3: Configuración del protocolo para sondear el metabolismo energético mitocondrial de esferoides 3D individuales utilizando el analizador XFe96.
- Haga clic en Iniciar ejecución para abrir el cuadro de diálogo Guardar ubicación .
- Introduzca la ubicación de guardado para el archivo de resultados y coloque el cartucho del sensor ensamblado en la bandeja térmica que aparece desde la puerta en el lateral del analizador. Espere a que la bandeja térmica se abra automáticamente y la pantalla muestre el mensaje De la placa de utilidad del calibrador de carga . Antes de seguir las indicaciones en pantalla, asegúrese de i) el ajuste adecuado del cartucho del sensor en la placa de utilidad, ii) la tapa se retira del cartucho del sensor y iii) la orientación correcta del cartucho del sensor en la placa de utilidad.
- Siga los comandos en pantalla para iniciar la calibración del cartucho del sensor.
NOTA: El tiempo necesario para completar la calibración es de aproximadamente 10-20 min (para ensayos a 37 °C). - Después de la calibración del cartucho del sensor, cargue la microplaca esferoide en el analizador siguiendo las instrucciones en pantalla del controlador de onda para iniciar el paso de equilibrio de 12 minutos.
NOTA: Las casillas verdes con marcas blancas indican una calibración "buena" para ese pozo. Si algún pozo no proporciona una calibración "buena", se indicará con una caja roja y una cruz blanca. Dichos pozos deben anotarse y excluirse de cualquier análisis después de que se complete el ensayo utilizando la pestaña de ensayo de modificación . - Espere a que el analizador comience automáticamente a adquirir mediciones de referencia después de que la máquina haya completado el paso de equilibrio (como se describe en el protocolo del instrumento).
- Para completar el experimento, siga los comandos en pantalla del controlador WAVE.
NOTA: Una vez que la microplaca esferoide se haya retirado del analizador, deseche el cartucho del sensor y reserve la placa esferoide para un análisis adicional si es necesario (por ejemplo, cuantificación de ADN de doble cadena (ds)). Si la microplaca no es necesaria para un análisis posterior, se puede desechar junto con el cartucho del sensor. - Espere a que aparezca el cuadro de diálogo de ensayo y vea los resultados o vuelva a la vista de plantillas .
5. Estrategias de normalización y análisis de datos: normalización posterior al ensayo y ensayos posteriores (pasos opcionales)
- Normalización de datos
- Para normalizar los datos de esferoides, consulte la serie de protocolos pertinentes a las estrategias de normalización de datos para calcular el tamaño y el volumen de los esferoides y cuantificar el dsDNA en ensayos de esferoides. Estos se han incluido como archivos complementarios; consulte el archivo complementario 1 y el archivo complementario 2.
- Análisis de datos
- Para exportar datos a uno de los generadores de análisis automatizados, siga los comandos de exportación de datos en el controlador WAVE y seleccione el generador de exportación que coincida con el tipo de ensayo. Alternativamente, exporte el archivo de datos y cárguelo en Seahorse Analytics.
NOTA: La desventaja de los generadores de informes y el análisis de Seahorse es que el análisis de datos se limita a cómo está diseñado el ensayo XF y no permite que se tomen promedios a través de los ciclos de medición. La exportación manual de conjuntos de datos desde el software del instrumento permite la preferencia del usuario a este respecto. Dado que la estrategia de inyección para evaluar la respiración mitocondrial de los esferoides 3D probablemente diferirá de la de una prueba típica de 'MitoStress', se han desarrollado una serie de plantillas de hojas de cálculo para ayudar a analizar estos conjuntos de datos, específicos para cultivos celulares 3D y se proporcionarán a pedido. Estos archivos de plantilla de datos proporcionarán datos sobre los parámetros respiratorios mitocondriales clave detallados y explicados en la Figura 2. - Para analizar los datos, exporte los datos como un informe de hoja de cálculo desde el software del controlador WAVE y utilice una plantilla de hoja de cálculo independiente para el análisis.
- Para exportar datos a uno de los generadores de análisis automatizados, siga los comandos de exportación de datos en el controlador WAVE y seleccione el generador de exportación que coincida con el tipo de ensayo. Alternativamente, exporte el archivo de datos y cárguelo en Seahorse Analytics.
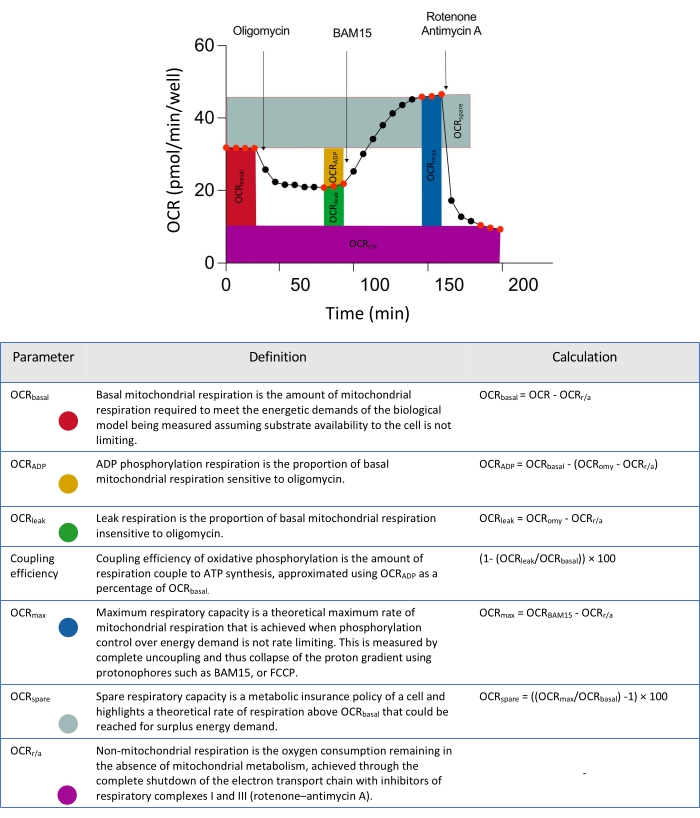
Figura 2: Descriptores esquemáticos para parámetros derivados de análisis de datos de flujo extracelular. Abreviatura: OCR = tasa de consumo de oxígeno. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para obtener esferoides compactos y bien formados, cada línea celular se optimizó individualmente para la densidad de siembra y la duración del cultivo (Figura 3). Las líneas celulares A549, HepG2/C3A y SK-OV-3 inicialmente formaron agregados sueltos que no progresaron a esferoides redondos con perímetros claramente definidos hasta después de 7 días en cultivo. Por el contrario, las células MCF-7 podrían formar esferoides en 3 días. Hubo una clara correlación entre la densidad ini...
Access restricted. Please log in or start a trial to view this content.
Discusión
Principales conclusiones y productos
Este artículo proporciona un protocolo detallado para sondear el metabolismo energético mitocondrial de esferoides 3D individuales utilizando una serie de líneas celulares derivadas del cáncer con el analizador XFe96 XF. Se desarrolla y describe un método para el cultivo rápido de esferoides celulares A549, HepG2 /C3A, MCF7 y SK-OV-3 utilizando tecnologías repelentes celulares para la agregación forzada. Este protocolo aborda muchas consideraciones sobre el...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
N.J.C fue apoyado por un premio BBSRC MIBTP CASE con Sygnature Discovery Ltd (BB/M01116X/1, 1940003)
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
Referencias
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
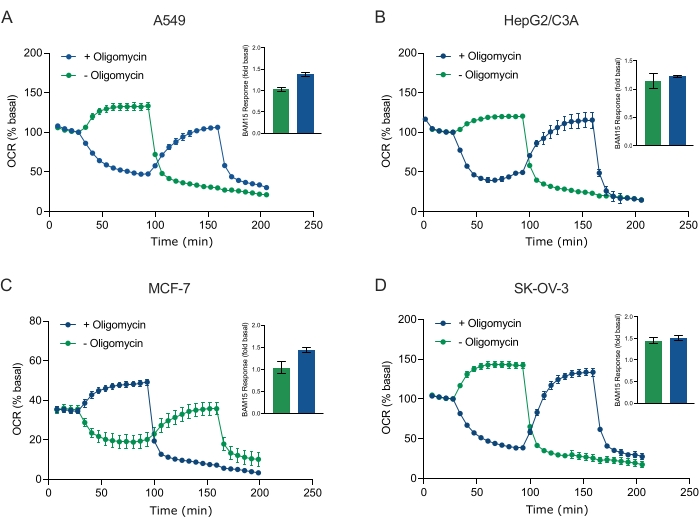
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
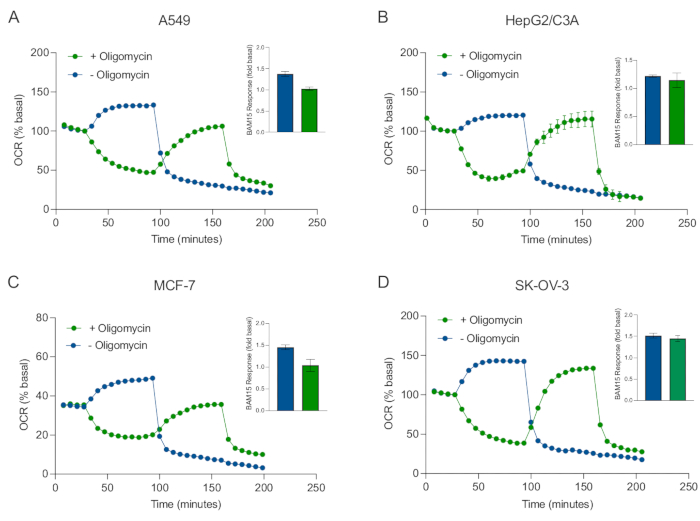
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados