Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Erforschung des mitochondrialen Energiestoffwechsels einzelner 3D-Mikrogewebesphäroide mittels extrazellulärer Flussanalyse
In diesem Artikel
Erratum Notice
Zusammenfassung
Diese Protokolle werden den Benutzern helfen, den mitochondrialen Energiestoffwechsel in 3D-Krebszelllinien-abgeleiteten Sphäroiden mithilfe der extrazellulären Flussanalyse von Seepferdchen zu untersuchen.
Zusammenfassung
Dreidimensionale (3D) zelluläre Aggregate, sogenannte Sphäroide, sind in den letzten Jahren an die Spitze der In-vitro-Zellkultur gerückt. Im Gegensatz zur Kultivierung von Zellen als zweidimensionale, einzellige Monoschichten (2D-Kultur) fördert, reguliert und unterstützt die sphäroide Zellkultur die physiologische Zellarchitektur und -merkmale, die in vivo existieren, einschließlich der Expression extrazellulärer Matrixproteine, Zellsignalisierung, Genexpression, Proteinproduktion, Differenzierung und Proliferation. Die Bedeutung der 3D-Kultur wurde in vielen Forschungsbereichen anerkannt, darunter Onkologie, Diabetes, Stammzellbiologie und Tissue Engineering. In den letzten zehn Jahren wurden verbesserte Methoden entwickelt, um Sphäroide herzustellen und ihre Stoffwechselfunktion und ihr Schicksal zu beurteilen.
Extrazelluläre Flussanalysatoren (XF) wurden verwendet, um die mitochondriale Funktion in 3D-Mikrogeweben wie Sphäroiden zu untersuchen, wobei entweder eine XF24-Insel-Capture-Platte oder eine XFe96-Sphäroid-Mikroplatte verwendet wurden. Unterschiedliche Protokolle und die Optimierung des mitochondrialen Energiestoffwechsels in Sphäroiden unter Verwendung der XF-Technologie wurden jedoch nicht im Detail beschrieben. Dieses Dokument enthält detaillierte Protokolle zur Untersuchung des mitochondrialen Energiestoffwechsels in einzelnen 3D-Sphäroiden unter Verwendung von Sphäroid-Mikrotiterplatten mit dem XFe96 XF-Analysator. Unter Verwendung verschiedener Krebszelllinien kann die XF-Technologie nachweislich zwischen der Zellatmung in 3D-Sphäroiden nicht nur unterschiedlicher Größe, sondern auch unterschiedlicher Volumina, Zellnummern, DNA-Gehalt und -Typ unterscheiden.
Die optimalen mitochondrialen Effektorverbindungskonzentrationen von Oligomycin, BAM15, Rotenon und Antimycin A werden verwendet, um spezifische Parameter des mitochondrialen Energiestoffwechsels in 3D-Sphäroiden zu untersuchen. Dieses Papier diskutiert auch Methoden zur Normalisierung von Daten, die von Sphäroiden erhalten wurden, und befasst sich mit vielen Überlegungen, die bei der Erforschung des Sphäroidstoffwechsels mit der XF-Technologie berücksichtigt werden sollten. Dieses Protokoll wird dazu beitragen, die Forschung in fortschrittlichen In-vitro-Sphäroidmodellen voranzutreiben.
Einleitung
Fortschritte bei In-vitro-Modellen in der biologischen Forschung haben in den letzten 20 Jahren rasante Fortschritte gemacht. Solche Modelle umfassen nun Organ-on-a-Chip-Modalitäten, Organoide und 3D-Mikrogewebe-Sphäroide, die alle zu einem gemeinsamen Schwerpunkt geworden sind, um die Translation zwischen In-vitro- und In-vivo-Studien zu verbessern. Die Verwendung fortschrittlicher In-vitro-Modelle, insbesondere von Sphäroiden, umfasst mehrere Forschungsbereiche, darunter Tissue Engineering, Stammzellforschung, Krebs und Krankheitsbiologie 1,2,3,4,5,6,7 und Sicherheitstests, einschließlich genetischer Toxikologie 8,9,10, Nanomaterial-Toxikologie 11, 12,13,14 und Arzneimittelsicherheits- und Wirksamkeitsprüfung 8,15,16,17,18,19.
Die normale Zellmorphologie ist entscheidend für den biologischen Phänotyp und die biologische Aktivität. Die Kultivierung von Zellen zu 3D-Mikrogewebe-Sphäroiden ermöglicht es Zellen, eine Morphologie, phänotypische Funktion und Architektur anzunehmen, die eher der in vivo beobachteten ähnelt, aber mit klassischen Monolayer-Zellkulturtechniken schwer einzufangen ist. Sowohl in vivo als auch in vitro wird die zelluläre Funktion direkt von der zellulären Mikroumgebung beeinflusst, die nicht auf die zelluläre Kommunikation und Programmierung beschränkt ist (z. B. Zell-Zell-Verbindungsbildungen, Möglichkeiten zur Bildung von Zellnischen); Zellexposition gegenüber Hormonen und Wachstumsfaktoren in der unmittelbaren Umgebung (z. B. zelluläre Zytokin-Exposition als Teil einer Entzündungsreaktion); Zusammensetzung physikalischer und chemischer Matrizen (z. B. ob Zellen in einer steifen Gewebekulturplastik oder einer elastischen Gewebeumgebung gezüchtet werden); und vor allem, wie der Zellstoffwechsel durch die Ernährung und den Zugang zu Sauerstoff sowie die Verarbeitung von Stoffwechselabfallprodukten wie Milchsäure beeinflusst wird.
Die metabolische Flussanalyse ist eine leistungsstarke Möglichkeit, den Zellstoffwechsel innerhalb definierter In-vitro-Systeme zu untersuchen. Insbesondere ermöglicht die XF-Technologie die Analyse von Live-Echtzeit-Veränderungen in der zellulären Bioenergetik intakter Zellen und Gewebe. Angesichts der Tatsache, dass viele intrazelluläre Stoffwechselereignisse in der Größenordnung von Sekunden bis Minuten auftreten, sind funktionelle Ansätze in Echtzeit von größter Bedeutung, um Echtzeitänderungen des zellulären Stoffwechselflusses in intakten Zellen und Geweben in vitro zu verstehen.
Dieses Papier enthält Protokolle für die Kultivierung von Krebs-abgeleiteten Zelllinien A549 (Lungen-Adenokarzinom), HepG2 / C3A (hepatozelluläres Karzinom), MCF-7 (Brust-Adenokarzinom) und SK-OV-3 (ovarielles Adenokarzinom) als In-vitro-3D-Sphäroidmodelle unter Verwendung von erzwungenen Aggregationsansätzen (Abbildung 1). Es beschreibt auch (i) detailliert, wie der mitochondriale Energiestoffwechsel einzelner 3D-Sphäroide mit dem Agilent XFe96 XF-Analysator untersucht werden kann, (ii) zeigt Möglichkeiten zur Optimierung von XF-Assays mit einzelnen 3D-Sphäroiden auf und (iii) diskutiert wichtige Überlegungen und Einschränkungen der Untersuchung des 3D-Sphäroidstoffwechsels mit diesem Ansatz. Am wichtigsten ist, dass dieses Papier beschreibt, wie Datensätze gesammelt werden, die die Berechnung der Sauerstoffverbrauchsrate (OCR) ermöglichen, um die oxidative Phosphorylierung und damit die mitochondriale Funktion in zellulären Sphäroiden zu bestimmen. Obwohl nicht für dieses Protokoll analysiert, ist die extrazelluläre Versauerungsrate (ECAR) ein weiterer Parameter, der neben OCR-Daten in XF-Experimenten gemessen wird. ECAR wird jedoch oft schlecht oder falsch aus XF-Datensätzen interpretiert. Wir kommentieren die Grenzen der Berechnung von ECAR nach grundlegenden Ansätzen des Technologieherstellers.
Access restricted. Please log in or start a trial to view this content.
Protokoll
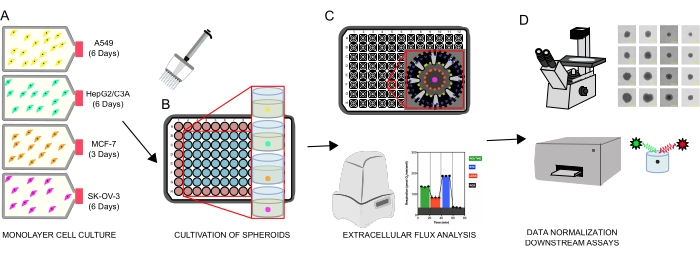
Abbildung 1: Grafischer Workflow zur Erzeugung zellulärer Sphäroide, extrazellulärer Flussanalyse und nachgelagerter Assays. Vier Krebszelllinien wurden selektiv als Monoschichten (A) kultiviert, von Gewebekulturkolben gelöst und in Ultralow-Attachment-96-Well-Mikroplatten gesät, um Sphäroide zu bilden (B). A549-Lungenkarzinom, HepG2 / C3A-Leberkarzinom, SK-OV-3-Ovarialadenokarzinom und MCF-7-Mammakarzinomzellen wurden bei 1 × 10 3-8 × 103 Zellen / gut ausgesät und bis zu 7 Tage gezüchtet, um einzelne Sphäroide zu bilden und die Sphäroidaussaatdichte und Kultivierungszeit durch kontinuierliche Beobachtung und planimetrische Messungen zu optimieren. Nach der Bildung wurden einzelne Sphäroide in ein serumfreies XF-Medium gewaschen und sorgfältig in Sphäroid-Assay-Mikrotiterplatten gesät, die mit Poly-D-Lysin (C) vorbeschichtet waren. Sphäroide wurden einer extrazellulären Flussanalyse mit dem XFe96-Analysator unter Verwendung mehrerer Protokolle unterzogen, um Folgendes zu adressieren: (1) optimale Sphäroidgröße für die basale mitochondriale Atmungsreaktion; (2) optimierte Titration von mitochondrialen Atemhemmern; (3) Optimierung der Sphäroidplatzierung in Mikroplattenvertiefungen. (D) Post-XF-Analysen, Phasenkontrastmikroskopie und Sphäroid-DNA-Quantifizierung wurden für die Datennormalisierung und andere nachgelagerte In-vitro-Assays verwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
1. Kultivierung von Krebszelllinien als 3D-In-vitro-Sphäroide
| Zelllinie | Beschreibung | Nährmedium | Quelle |
| A-549 | Lungenkarzinom-Zelllinie | RPMI 1640 | Europäische Sammlung authentifizierter Zellkulturen (ECACC) |
| Natriumpyruvat (1 mM) | |||
| Penicillin- Streptomycin - (100 U / ml - 100 mg / ml) | |||
| 10 % (v/v) FBS | |||
| HepG2/C3A | Leberkarzinom-Zelllinie, ein klonales Derivat der Elternzelllinie HepG2 | DMEM | Amerikanische Gewebekultursammlung (ATCC) |
| Penicillin- Streptomycin - (100 U / ml - 100 mg / ml) | |||
| 10 % (v/v) FBS | |||
| MCF7 | Brust-Adenokarzinom-Zelllinie | RPMI 1640 | Europäische Sammlung authentifizierter Zellkulturen (ECACC) |
| Natriumpyruvat (1 mM) | |||
| Penicillin- Streptomycin - (100 U / ml - 100 mg / ml) | |||
| 10 % (v/v) FBS | |||
| SK-OV-3 | Ovarielle Adenokarzinom-Zelllinie | RPMI 1640 | Europäische Sammlung authentifizierter Zellkulturen (ECACC) |
| Natriumpyruvat (1 mM) | |||
| Penicillin- Streptomycin - (100 U / ml - 100 mg / ml) | |||
| 10 % (v/v) FBS | |||
| Bestandteil | RPMI-Assay-Medium (50 ml Endvolumen) | ||
| Basismedium | Agilent Seepferdchen XF RPMI, pH 7,4 | ||
| Glukose (1 M steriler Vorrat) | 11 mM (0,55 mL Stammlösung) | ||
| L-Glutamin (200 mM steriler Vorrat) | 2 mM (0,5 mL Stammlösung) | ||
| Natriumpyruvat (100 mM steriler Vorrat) | 1 mM (0,5 mL Stammlösung) |
Tabelle 1: Krebszelllinienmedien und XF-Medienzusammensetzungen.
- Kultivieren Sie alle Zelllinien mit der aseptischen Standard-Gewebekulturtechnik und bestätigen Sie, dass sie frei von Mykoplasmen sind, indem Sie ein geeignetes Assay-Kit verwenden.
- Kultivieren Sie die Zelllinien in T75-Gewebekulturflaschen oder einem gleichwertigen Plasma unter Verwendung des empfohlenen Mediums (Tabelle 1). Kultivieren Sie die Zelllinien auf 65-80% Konfluenz und passieren Sie sie regelmäßig bis zu einem Maximum von 25 Passagen.
- Spülen Sie die Zellkulturkolben zweimal in Dulbeccos modifizierter phosphatgepufferter Kochsalzlösung (DBPS) ab.
- Lösen Sie die Zellen aus den Kolben mit 3 ml des Zelldissoziationsreagenzes (siehe Materialtabelle) für 5 min bei 37 °C und bestätigen Sie die Ablösung durch Mikroskopie.
- Saugen Sie die abgelöste Zellsuspension vorsichtig ab, um eine Einzelzellsuspension zu gewährleisten, und deaktivieren Sie das Zelldissoziationsreagenz mit 7 ml vollständigem Gewebekulturmedium.
- Sammeln Sie die Zellen durch Zentrifugation bei 300 × g für 5 min, verwerfen Sie den Überstand und suspendieren Sie die Zellen in vollständigem Medium.
- Zählen Sie die Zellen mit einem Hämozytometer oder einem automatisierten Zellzähler und titrieren Sie auf die gewünschte Zelldichte, die für die Aussaat erforderlich ist.
HINWEIS: Um eine ganze 96-Well-Platte bei 100 μL / Well bei 4 × 103 Zellen / Well zu säen, sollten die Zellen auf 4 × 104 Zellen / ml in einem empfohlenen Volumen von 12 ml titriert werden. - Dekantieren Sie die Zellsuspension in ein steriles Reservoir und geben Sie 100 μL der Zellsuspension mit einem Mehrkanalpipettor in jede Vertiefung einer zellabweisenden Mikrotiterplatte ab.
HINWEIS: Nur die inneren 60 Vertiefungen einer Mikrotiterplatte sollten ausgesät und der Rest mit DPBS gefüllt werden. Dies bildet eine Verdampfungsbarriere, gewährleistet die Sphäroidhomogenität über die Platte und minimiert Plattenrandeffekte. - Zentrifugieren Sie Sphäroid-Mikrotiterplatten bei 300 × g für 15 Minuten, um die Zellen in lose Aggregate zu zwingen.
- Inkubieren Sie die Platten bei 37 ° C, 5% CO2 für mindestens 3 Tage, um die Sphäroidbildung sicherzustellen.
- Führen Sie eine Phasenkontrastmikroskopie mit standardisierten Laborpraktiken durch, um das Wachstum von Sphäroiden zu überwachen. Füllen Sie das Zellkulturmedium alle 3 Tage oder zweimal wöchentlich auf, indem Sie einen halbvolumigen Medienaustausch durchführen.
2. Untersuchung des mitochondrialen Energiestoffwechsels einzelner Sphäroide mit Hilfe der extrazellulären Flussmitteltechnologie (XF)
- Assay-Vorbereitung (einen Tag vorher)
- Überprüfen Sie die Sphäroidlebensfähigkeit mit einem inversen Lichtmikroskop mit Phasenkontrast bei 4-facher Vergrößerung, um eine intakte Sphäroidstruktur, Morphologie und allgemeine Gleichmäßigkeit zwischen den Proben zu gewährleisten.
- Hydratisieren Sie die Sensorpatrone.
- Aliquot ~ 20 ml des Kalibers in ein konisches Rohr.
- Das konische Rohr, das das Kalibrant enthält, wird über Nacht in einen Nicht-CO2 37 °C-Inkubator gegeben.
- Entfernen Sie den Inhalt aus dem Assay-Kit.
- Entfernen Sie die Sensorkassette von der Gebrauchsplatte und legen Sie sie kopfüber auf die Arbeitsplatte neben der Stromplatte.
- Pipette 200 μL steriles ddH2O in jede Vertiefung der Sensorkartuschen-Utility-Platte mit einer Mehrkanal-P300-Pipette.
- Legen Sie die Sensorkassette auf die Utility-Platte.
- Überprüfen Sie, ob der Wasserstand in jedem Brunnen hoch genug ist, um die Sensorsonden einzutauchen.
- Die montierte Sensorkartusche in einen CO2-freien 37 °C-Inkubator geben und über Nacht stehen lassen.
HINWEIS: Dieser Schritt kann 12-72 h vor Beginn des Assays durchgeführt werden.
- Coat Sphäroid Assay Mikrotiterplatte
- Mit aseptischen Techniken 30 μL/Well sterile Poly-D-Lysin-Lösung (0,1 mg/ml) in die Sphäroid-Mikroplatte geben und 30 min bei Raumtemperatur inkubieren.
- Saugen Sie die Lösung aus jeder Vertiefung der Sphäroid-Mikrotiterplatte ab, invertieren Sie die Platte und klopfen Sie sie fest auf Seidenpapier, um die Restlösung zu entfernen.
- Waschen Sie die Platte zweimal mit 200 μL/Well von sterilem ddH2O.
- Kehren Sie nach der letzten Wäsche die Mikrotiterplatte um und klopfen Sie sie fest auf Seidenpapier, um Restwasser zu entfernen.
- Lassen Sie die Platte 30 Minuten an der Luft trocknen, bevor Sie sie verwenden oder bei 4 ° C für die zukünftige Verwendung lagern.
HINWEIS: Die Sphäroid-Assay-Mikrotiterplatte sollte mit einem molekularen Klebstoff beschichtet werden, um sicherzustellen, dass die Sphäroide am Boden der Mikrotiterplatte fixiert sind. Ohne einen molekularen Klebstoff können sich Sphäroide lösen und die Assay-Ergebnisse beeinträchtigen. Andere molekulare Klebstoffe können auch als Alternative zu Poly-D-Lysin für die Vorbeschichtung von Platten verwendet werden. Vorbeschichtete Platten können bei 4 °C gelagert werden, sollten aber vor Beginn des Assays auf Raumtemperatur ausgeglichen werden.
- XF Assay-Medium vorbereiten
- Vorbereiten von XF RPMI-Medium, wie in Tabelle 1 beschrieben, und Sterilfilter mit einem 0,22-μm-Spritzenfilter
- Assay-Vorbereitung (1 h vor dem Assay)
- Das ergänzte XF RPMI Assay medium auf 37 °C vorwärmen.
- Vorwärmen Sie die beschichtete Sphäroid-Assay-Mikroplatte in einem Nicht-CO2 37 °C-Inkubator oder Trockenbad.
- Bereiten Sie die Sensorkassette vor.
- Nehmen Sie das konische Rohr mit dem Kalibrant und der Sensorpatrone aus dem Luftinkubator.
- Entfernen Sie die Sensorkassette von der Gebrauchsplatte und legen Sie sie kopfüber auf die Arbeitsfläche.
- Saugen Sie mit einer P300-Mehrkanalpipette das Wasser von der Utility-Platte ab und entsorgen Sie es.
- Gießen Sie die Kalibrantlösung in ein steriles Reagenzreservoir und geben Sie 200 μL/Vertiefung des vorgewärmten Calibrats mit einer P300-Mehrkanalpipette auf die Gebrauchsplatte.
- Nehmen Sie die Sensorpatrone auf und legen Sie sie wieder auf die Gebrauchsplatte, um sicherzustellen, dass die Sensoren gut in das Kaliber eingetaucht sind.
- Bringen Sie die montierte Sensorkassette zurück in den Nicht-CO2 37 °C-Inkubator, bis sie bereit ist, die Anschlussinjektionslösungen zu laden.
- Waschen Sie die Sphäroide mit dem Assay-Medium.
- Entfernen Sie die Sphäroidkulturplatte aus dem 37 ° C, 5% CO2 -Inkubator und beobachten Sie die Sphäroide unter dem Mikroskop, um ihre Integrität vor den Sphäroidtransferschritten sicherzustellen.
- Laden Sie alle Vertiefungen der Sphäroidplatte mit 180 μL/Vertiefung des vorgewärmten Assay-Mediums, einschließlich aller Hintergrundkorrekturquellen.
- Füllen Sie eine 7 cm große Petrischale teilweise mit 3 ml des Assay-Mediums.
- Mit einer Mehrkanalpipette, die mit breiten Pipettenspitzen beladen ist, werden die Sphäroide von der 96-Well-Kulturplatte in 7 cm große Petrischalen übertragen, indem der Pipetter auf ein Aspirationsvolumen von 10-50 μL eingestellt wird.
- Samen Sie Sphäroide in die vorbeschichtete Sphäroid-Assay-Mikroplatte.
- Übertragen Sie die Sphäroide mit einem Dissektionsmikroskop und einer Lightbox-Vorrichtung von der Petrischale auf die Sphäroid-Assay-Mikroplatte, wie unten beschrieben.
- Stellen Sie das Volumen eines Einkanalpipettors mit einer breiten Pipettenspitze auf 20 μL ein und saugen Sie vorsichtig ein einzelnes Sphäroid ab. Platzieren Sie die Spitze direkt in der Mitte jeder Vertiefung der Sphäroid-Assay-Mikroplatte und lassen Sie die Schwerkraft ein einzelnes Sphäroid in die Mitte jedes Vertiefungslochs eluieren, d.h. stoßen Sie kein Medium aus der Pipettenspitze aus und lassen Sie die Kapillarwirkung das Sphäroid aus der Pipettenspitze entfernen. Um die Elution zu bestätigen, kann der Inhalt des Pipettierers unter dem Mikroskop zurück in die 7 cm große Petrischale pipettiert werden.
HINWEIS: Die Schwerkraftelution eines einzelnen Sphäroids dauert typischerweise 15-30 s, abhängig von der Größe / Dichte des Sphäroids. Während dieser Zeit sollte der Pipetter nicht entfernt werden. Alle Hintergrundkorrekturvertiefungen sollten frei von Sphäroiden sein und nur Assay-Medium enthalten. Bestätigen Sie unter dem Mikroskop die Position jedes Sphäroids. Jedes Sphäroid sollte idealerweise in der Mitte jedes Brunnens positioniert werden. - Sobald alle Sphäroide auf die Sphäroid-Assay-Mikroplatte übertragen wurden, wird die Platte vor dem Assay mindestens 1 h lang bei 37 °C in einen Nicht-CO2-Inkubator überführt.
- Stellen Sie das Volumen eines Einkanalpipettors mit einer breiten Pipettenspitze auf 20 μL ein und saugen Sie vorsichtig ein einzelnes Sphäroid ab. Platzieren Sie die Spitze direkt in der Mitte jeder Vertiefung der Sphäroid-Assay-Mikroplatte und lassen Sie die Schwerkraft ein einzelnes Sphäroid in die Mitte jedes Vertiefungslochs eluieren, d.h. stoßen Sie kein Medium aus der Pipettenspitze aus und lassen Sie die Kapillarwirkung das Sphäroid aus der Pipettenspitze entfernen. Um die Elution zu bestätigen, kann der Inhalt des Pipettierers unter dem Mikroskop zurück in die 7 cm große Petrischale pipettiert werden.
- Übertragen Sie die Sphäroide mit einem Dissektionsmikroskop und einer Lightbox-Vorrichtung von der Petrischale auf die Sphäroid-Assay-Mikroplatte, wie unten beschrieben.
3. Aufbereitung und Laden von Verbindungen in die Sensorkartusche für XF-Assays
| Injektionsstrategie | Verbindung (Port) | XFe96 Mikrowellen-Startvolumen (μL) | Gewünschte Endbohrlochkonzentration | Anschlussvolumen (μL) | Endgültiges XFe96-Mikrowellenvolumen nach der Injektion (μL) | Konzentration des Arbeitsmaterials |
| 1 | Oligomycin (A) | 180 | 3 μg/ml | 20 | 200 | 30 μg/ml |
| Rotenon (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimycin A (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| Rotenon (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimycin A (B) | 200 | 2 μM | 20 | 220 | 22 μM |
Tabelle 2: Mitochondriale Verbindungskonzentrationen zur Untersuchung des mitochondrialen Energiestoffwechsels einzelner 3D-Sphäroide mit dem XFe96 Analyzer.
- Bereiten Sie die Arbeitsstoffkonzentrationen jeder Verbindung gemäß Tabelle 2 unter Verwendung eines vollständig ergänzten, vorgewärmten XF-RPMI-Assay-Mediums vor.
- Richten Sie die Patronenplatte (gekoppelt an die Utility-Platte) spaltenweise aus, 1-12 von links nach rechts.
- Wenn Sie eine Ladeführung verwenden, legen Sie sie gemäß dem Well-Loading-Verfahren auf die Patronenplatte, z. B. wenn Port A zuerst geladen wird, stellen Sie sicher, dass A in der oberen linken Ecke der Führung sichtbar ist.
- Die Arbeitslösung jeder Verbindung in ein geeignetes Reservoir geben und mit einer kalibrierten P100-Mehrkanalpipette 20 μL in alle entsprechenden Ports dosieren. Wiederholen Sie den Vorgang für jede Verbindung in die verbleibenden Ports.
HINWEIS: Wenn keine Anschlüsse an der Sensorpatronenplatte verwendet werden, können diese leer gelassen oder mit Assay-Medium gefüllt werden. Wenn nur eine Auswahl eines bestimmten Portbuchstabens verwendet wird, stellen Sie sicher, dass die anderen Ports, die diesem Buchstaben entsprechen, mit Assay-Medium geladen sind. Andernfalls wird Luft in das Bohrloch injiziert, wodurch die Ergebnisse in diesen Bohrlöchern beeinträchtigt werden. - Entfernen Sie nach dem Laden des Anschlusses die Plattenladeführungen (falls verwendet) und bereiten Sie den Analysator für das Laden der Sensorpatrone vor.
HINWEIS: Wenn der Assay nicht sofort nach dem Laden der Anschlüsse durchgeführt wird, setzen Sie den Deckel wieder auf die Sensorkassette und legen Sie die Platte wieder in den 37 °C Luftinkubator, bis sie bereit ist, in die Maschine zu laden.
4. Assay-Design, Injektionsstrategien und Datenerfassung
- Ausführen des Assays
- Schalten Sie den Analysator ein und stellen Sie eine Verbindung zum Controller (Computer) her.
HINWEIS: Dies kann durch den Geräteverbindungsstatus im Widget-Panel der Wave Controller-Software überprüft werden. - Navigieren Sie zur Vorlagenseite in der WAVE-Software, suchen Sie die Assay-Vorlagendatei für das Experiment und doppelklicken Sie, um sie zu öffnen.
HINWEIS: Wenn die Assay-Vorlage nicht in der Vorlagenansicht angezeigt wird, importieren Sie die Vorlagendatei von einem freigegebenen Netzlaufwerk oder USB-Flashlaufwerk in den Vorlagenordner . - Um den Assay zu starten, klicken Sie auf die Registerkarte Assay ausführen .
HINWEIS: Wenn die Gruppendefinitionen innerhalb der Plattenkarte korrekt zugeordnet wurden, kann der Assay ausgeführt werden, wie durch das grüne Häkchen auf der rechten Seite der Seite angezeigt. Zu diesem Zeitpunkt können alle zusätzlichen Informationen auf der Seite mit der Zusammenfassung des Assays oder auf der leeren Seite eingegeben werden. Fahren Sie mit dem nächsten Schritt fort. Aufgrund der verzögerten Penetration von mitochondrialen Modulatoren in 3D-Mikrogewebe-Sphäroide (Abbildung 2) sind die in Tabelle 3 beschriebenen Messprotokollinformationen zu verwenden.
- Schalten Sie den Analysator ein und stellen Sie eine Verbindung zum Controller (Computer) her.
| Messzeitraum | Einspritznummer und Port | Details zur Messung | Periodendauer (h:min:s) |
| Kalibrierung | Nicht zutreffend | XF-Analysatoren führen diese Kalibrierung immer durch, um sicherzustellen, dass die Messungen genau sind | 00:20:00 Uhr (dies ist ein Durchschnitt und kann zwischen den Maschinen variieren) |
| Equillibration | Nicht zutreffend | Das Gleichgewicht tritt nach der Kalibrierung auf und wird empfohlen. | 00:10:00 |
| Basal | Nicht zutreffend | Zyklen = 5 | 00:30:00 |
| Mix = 3:00 | |||
| Wartezeit = 0:00 | |||
| Maß = 3:00 | |||
| Oligomycin / BAM15 | Einspritzung 1 (Anschluss A) | Zyklen = 10 | 01:00:00 |
| Mix = 3:00 | |||
| Wartezeit = 0:00 | |||
| Maß = 3:00 | |||
| Rotenon + Antimycin A | Einspritzung 2 (Anschluss B) | Zyklen = 10 | 01:00:00 |
| Mix = 3:00 | |||
| Wartezeit = 0:00 | |||
| Maß = 3:00 | |||
| Gesamtzeit: | 03:00:00 | ||
Tabelle 3: Protokollaufbau zur Untersuchung des mitochondrialen Energiestoffwechsels einzelner 3D-Sphäroide mit dem XFe96 Analyzer.
- Klicken Sie auf Ausführen starten , um das Dialogfeld Speicherort aufzurufen.
- Geben Sie den Speicherort für die Ergebnisdatei ein, und legen Sie die montierte Sensorkassette auf das Thermofach, das an der Tür an der Seite des Analysators angezeigt wird. Warten Sie, bis sich das Thermofach automatisch öffnet und auf dem Bildschirm die Meldung Load Calibrant Utility Plate angezeigt wird. Bevor Sie den Anweisungen auf dem Bildschirm folgen, stellen Sie sicher, dass i) die Sensorpatrone ordnungsgemäß auf der Utility-Platte sitzt, ii) der Deckel von der Sensorpatrone entfernt wird und iii) die Sensorpatrone auf der Utility-Platte richtig ausgerichtet ist.
- Befolgen Sie die Anweisungen auf dem Bildschirm, um die Kalibrierung der Sensorpatrone zu starten.
HINWEIS: Die Zeit bis zum Abschluss der Kalibrierung beträgt ca. 10-20 min (bei Assays bei 37 °C). - Laden Sie nach der Kalibrierung der Sensorkartusche die Sphäroid-Mikroplatte in den Analysator, indem Sie den Anweisungen auf dem Bildschirm auf dem Wave Controller folgen, um den 12-minütigen Gleichgewichtsschritt einzuleiten.
HINWEIS: Grüne Kästchen mit weißen Häkchen zeigen eine "gute" Kalibrierung für diesen Brunnen an. Wenn Bohrlöcher keine "gute" Kalibrierung bieten, werden sie mit einem roten Kasten und einem weißen Kreuz gekennzeichnet. Solche Vertiefungen sollten notiert und von jeder Analyse ausgeschlossen werden, nachdem der Assay mit der Registerkarte Modifikationsassay abgeschlossen wurde. - Warten Sie, bis der Analysator automatisch mit der Erfassung der Basismessungen beginnt, nachdem die Maschine den Gleichgewichtsschritt abgeschlossen hat (wie im Geräteprotokoll beschrieben).
- Um das Experiment abzuschließen, folgen Sie den Bildschirmbefehlen auf dem WAVE-Controller.
HINWEIS: Sobald die Sphäroid-Mikroplatte aus dem Analysator entfernt wurde, entsorgen Sie die Sensorpatrone und legen Sie die Sphäroidplatte für weitere Analysen beiseite, falls erforderlich (z. B. doppelsträngige (ds) DNA-Quantifizierung). Wenn die Mikrotiterplatte nicht für die weitere Analyse benötigt wird, kann sie zusammen mit der Sensorpatrone entsorgt werden. - Warten Sie, bis das Assay-Dialogfeld angezeigt wird, und zeigen Sie die Ergebnisse an oder kehren Sie zur Vorlagenansicht zurück.
5. Datennormalisierungs- und Analysestrategien - Post-Assay-Normalisierung und Downstream-Assays (optionale Schritte)
- Datennormalisierung
- Um Sphäroiddaten zu normalisieren, beziehen Sie sich auf die Reihe von Protokollen, die für Datennormalisierungsstrategien zur Berechnung der Sphäroidgröße und des Sphäroidvolumens und zur Quantifizierung von dsDNA in Sphäroidassays relevant sind. Diese wurden als ergänzende Dateien aufgenommen; siehe Zusatzdatei 1 und Ergänzungsdatei 2.
- Datenanalyse
- Um Daten in einen der automatisierten Analysegeneratoren zu exportieren, folgen Sie den Datenexportbefehlen auf dem WAVE-Controller und wählen Sie den Exportgenerator aus, der dem Assay-Typ entspricht. Alternativ können Sie die Datendatei exportieren und in Seahorse Analytics hochladen.
HINWEIS: Der Nachteil von Berichtsgeneratoren und Seahorse-Analysen besteht darin, dass die Datenanalyse auf die Art und Weise beschränkt ist, wie der XF-Assay konzipiert ist, und keine Mittelwerte über Messzyklen hinweg zulässt. Der manuelle Export von Datensätzen aus der Gerätesoftware ermöglicht diesbezüglich eine Benutzerpräferenz. Angesichts der Tatsache, dass sich die Injektionsstrategie zur Beurteilung der mitochondrialen Atmung von 3D-Sphäroiden wahrscheinlich von der eines typischen "MitoStress" -Tests unterscheidet, wurde eine Reihe von Tabellenvorlagen entwickelt, um die Analyse dieser Datensätze zu unterstützen, die für 3D-Zellkulturen spezifisch sind und auf Anfrage zur Verfügung gestellt werden. Diese Datenvorlagendateien liefern Daten zu den wichtigsten mitochondrialen Atmungsparametern, die in Abbildung 2 detailliert beschrieben und erläutert werden. - Um die Daten zu analysieren, exportieren Sie die Daten als Tabellenkalkulationsbericht aus der WAVE-Controller-Software und verwenden Sie eine unabhängige Tabellenkalkulationsvorlage für die Analyse.
- Um Daten in einen der automatisierten Analysegeneratoren zu exportieren, folgen Sie den Datenexportbefehlen auf dem WAVE-Controller und wählen Sie den Exportgenerator aus, der dem Assay-Typ entspricht. Alternativ können Sie die Datendatei exportieren und in Seahorse Analytics hochladen.
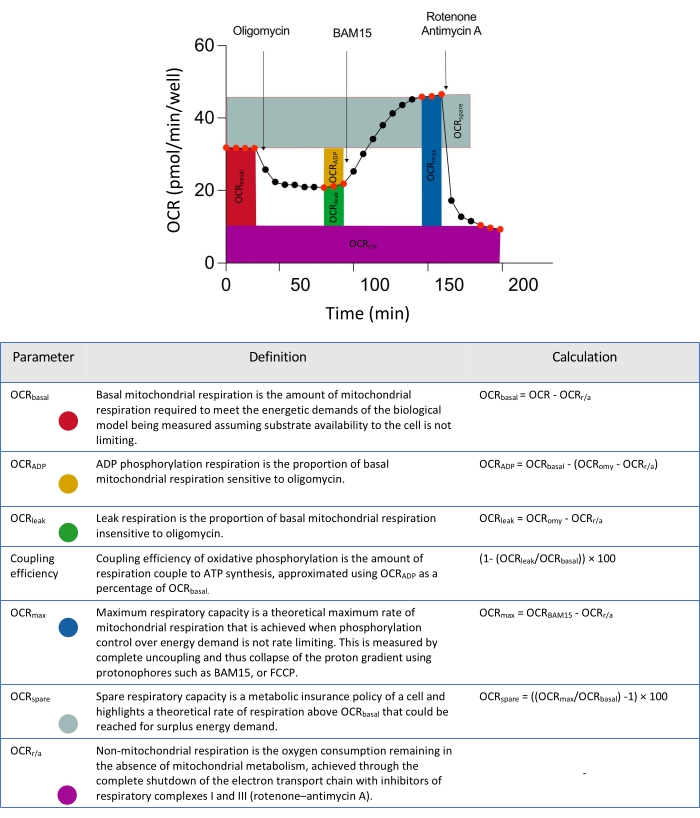
Abbildung 2: Schematische Deskriptoren für Parameter, die aus extrazellulären Flussdatenanalysen abgeleitet wurden. Abkürzung: OCR = Sauerstoffverbrauchsrate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Um wohlgeformte, kompakte Sphäroide zu erhalten, wurde jede Zelllinie einzeln für die Aussaatdichte und die Kultivierungsdauer optimiert (Abbildung 3). A549-, HepG2/C3A- und SK-OV-3-Zelllinien bildeten zunächst lose Aggregate, die erst nach 7 Tagen in Kultur zu runden Sphäroiden mit klar definierten Umfängen übergingen. Umgekehrt könnten MCF-7-Zellen innerhalb von 3 Tagen Sphäroide bilden. Es gab eine klare Korrelation zwischen der anfänglichen Zellaussaatdichte und dem Sphäroidvol...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Wichtigste Ergebnisse und Ergebnisse
Dieses Dokument enthält ein detailliertes Protokoll zur Untersuchung des mitochondrialen Energiestoffwechsels einzelner 3D-Sphäroide unter Verwendung einer Reihe von Krebszelllinien mit dem XFe96 XF Analyzer. Es wird ein Verfahren zur schnellen Kultivierung von A549, HepG2/C3A, MCF7 und SK-OV-3 zellulären Sphäroiden unter Verwendung von zellabweisenden Technologien zur erzwungenen Aggregation entwickelt und beschrieben. Dieses Protokoll befasst sich mit vielen ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte zu erklären.
Danksagungen
N.J.C wurde durch einen BBSRC MIBTP CASE Award mit Sygnature Discovery Ltd unterstützt (BB/M01116X/1, 1940003)
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
Referenzen
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
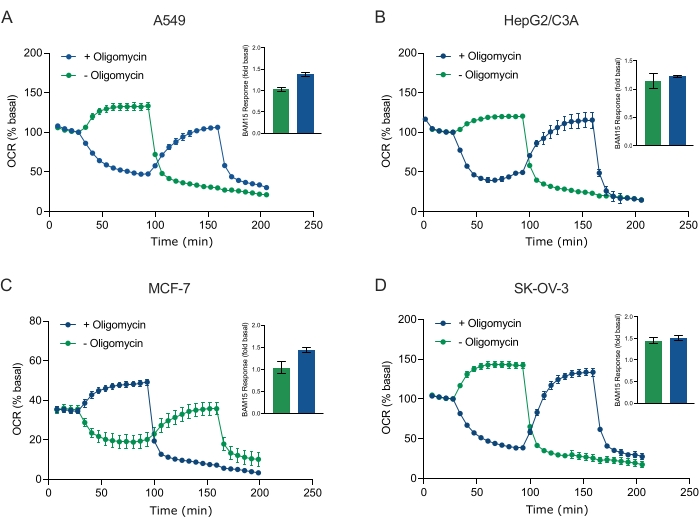
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
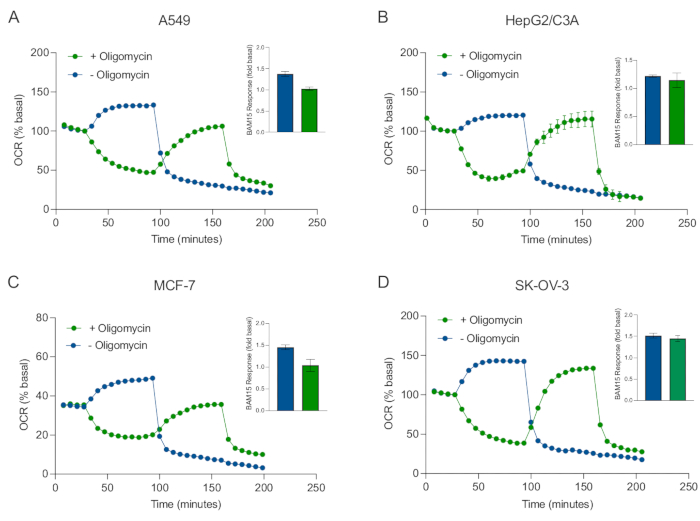
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten