È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Esplorazione del metabolismo energetico mitocondriale di singoli sferoidi microtessuati 3D utilizzando l'analisi del flusso extracellulare
In questo articolo
Erratum Notice
Riepilogo
Questi protocolli aiuteranno gli utenti a sondare il metabolismo energetico mitocondriale negli sferoidi derivati dalla linea cellulare del cancro 3D utilizzando l'analisi del flusso extracellulare di Cavalluccio marino.
Abstract
Gli aggregati cellulari tridimensionali (3D), chiamati sferoidi, sono diventati l'avanguardia della coltura cellulare in vitro negli ultimi anni. A differenza della coltura delle cellule come monostrati bidimensionali a singola cellula (coltura 2D), la coltura cellulare sferoide promuove, regola e supporta l'architettura cellulare fisiologica e le caratteristiche che esistono in vivo, compresa l'espressione di proteine della matrice extracellulare, la segnalazione cellulare, l'espressione genica, la produzione di proteine, la differenziazione e la proliferazione. L'importanza della cultura 3D è stata riconosciuta in molti campi di ricerca, tra cui oncologia, diabete, biologia delle cellule staminali e ingegneria tissutale. Nell'ultimo decennio, sono stati sviluppati metodi migliorati per produrre sferoidi e valutare la loro funzione metabolica e il loro destino.
Gli analizzatori di flusso extracellulare (XF) sono stati utilizzati per esplorare la funzione mitocondriale in microtessuti 3D come gli sferoidi utilizzando una piastra di cattura delle isole XF24 o una micropiastra sferoide XFe96. Tuttavia, protocolli distinti e l'ottimizzazione del metabolismo energetico mitocondriale di sonda negli sferoidi utilizzando la tecnologia XF non sono stati descritti in dettaglio. Questo documento fornisce protocolli dettagliati per sondare il metabolismo energetico mitocondriale in singoli sferoidi 3D utilizzando micropiastre sferoidi con l'analizzatore XF XFe96. Utilizzando diverse linee cellulari tumorali, la tecnologia XF ha dimostrato di essere in grado di distinguere tra respirazione cellulare in sferoidi 3D non solo di diverse dimensioni, ma anche di diversi volumi, numeri di cellule, contenuto e tipo di DNA.
Le concentrazioni ottimali di composti effettori mitocondriali di oligomicina, BAM15, rotenone e antimicina A vengono utilizzate per sondare parametri specifici del metabolismo energetico mitocondriale negli sferoidi 3D. Questo documento discute anche i metodi per normalizzare i dati ottenuti dagli sferoidi e affronta molte considerazioni che dovrebbero essere prese in considerazione quando si esplora il metabolismo sferoide utilizzando la tecnologia XF. Questo protocollo aiuterà a guidare la ricerca in modelli sferoidi avanzati in vitro .
Introduzione
I progressi nei modelli in vitro nella ricerca biologica sono progrediti rapidamente negli ultimi 20 anni. Tali modelli ora includono modalità organ-on-a-chip, organoidi e sferoidi microtesssuei 3D, che sono diventati tutti un obiettivo comune per migliorare la traduzione tra studi in vitro e in vivo. L'uso di modelli avanzati in vitro, in particolare sferoidi, abbraccia diversi campi di ricerca, tra cui l'ingegneria tissutale, la ricerca sulle cellule staminali, il cancro e la biologia delle malattie 1,2,3,4,5,6,7 e i test di sicurezza, tra cui la tossicologia genetica 8,9,10, la tossicologia dei nanomateriali11, 12,13,14 e test di sicurezza ed efficacia dei farmaci 8,15,16,17,18,19.
La normale morfologia cellulare è fondamentale per il fenotipo e l'attività biologica. La coltivazione di cellule in sferoidi microtissuei 3D consente alle cellule di adottare una morfologia, una funzione fenotipica e un'architettura, più simile a quella osservata in vivo ma difficile da catturare con le classiche tecniche di coltura cellulare monostrato. Sia in vivo che in vitro, la funzione cellulare è direttamente influenzata dal microambiente cellulare, che non si limita alla comunicazione e alla programmazione cellulare (ad esempio, formazioni di giunzione cellula-cellula, opportunità di formare nicchie cellulari); esposizione cellulare agli ormoni e ai fattori di crescita negli ambienti immediati (ad esempio, esposizione cellulare alle citochine come parte di una risposta infiammatoria); composizione di matrici fisiche e chimiche (ad esempio, se le cellule vengono coltivate in plastica di coltura tissutale rigida o in un ambiente di tessuto elastico); e, soprattutto, come il metabolismo cellulare è influenzato dalla nutrizione e dall'accesso all'ossigeno, nonché dalla lavorazione di prodotti di scarto metabolici come l'acido lattico.
L'analisi del flusso metabolico è un modo potente per esaminare il metabolismo cellulare all'interno di sistemi in vitro definiti. In particolare, la tecnologia XF consente l'analisi di cambiamenti vivi e in tempo reale nella bioenergetica cellulare di cellule e tessuti intatti. Dato che molti eventi metabolici intracellulari si verificano nell'ordine di secondi o minuti, gli approcci funzionali in tempo reale sono fondamentali per comprendere i cambiamenti in tempo reale nel flusso metabolico cellulare in cellule e tessuti intatti in vitro.
Questo documento fornisce protocolli per la coltivazione di linee cellulari derivate dal cancro A549 (adenocarcinoma polmonare), HepG2 / C3A (carcinoma epatocellulare), MCF-7 (adenocarcinoma mammario) e SK-OV-3 (adenocarcinoma ovarico) come modelli sferoidi 3D in vitro utilizzando approcci ad aggregazione forzata (Figura 1). Inoltre (i) descrive in dettaglio come sondare il metabolismo energetico mitocondriale di singoli sferoidi 3D utilizzando l'analizzatore Agilent XFe96 XF, (ii) evidenzia i modi per ottimizzare i saggi XF utilizzando singoli sferoidi 3D e (iii) discute importanti considerazioni e limitazioni del sondaggio del metabolismo sferoide 3D utilizzando questo approccio. Ancora più importante, questo documento descrive come vengono raccolti i set di dati che consentono il calcolo del tasso di consumo di ossigeno (OCR) per determinare la fosforilazione ossidativa e quindi la funzione mitocondriale negli sferoidi cellulari. Sebbene non sia stato analizzato per questo protocollo, il tasso di acidificazione extracellulare (ECAR) è un altro parametro che viene misurato insieme ai dati OCR negli esperimenti XF. Tuttavia, ECAR è spesso interpretato in modo errato o errato dai set di dati XF. Forniamo un commento sui limiti del calcolo dell'ECAR seguendo gli approcci di base del produttore della tecnologia.
Access restricted. Please log in or start a trial to view this content.
Protocollo
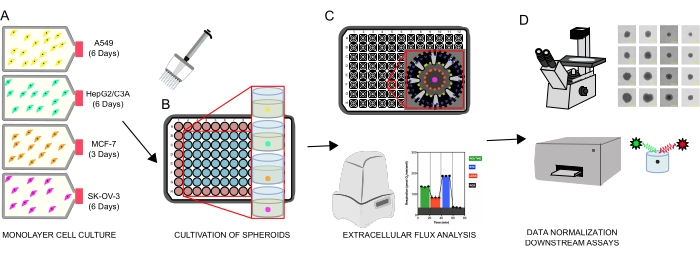
Figura 1: Flusso di lavoro grafico per la generazione di sferoidi cellulari, analisi del flusso extracellulare e saggi a valle. Quattro linee cellulari tumorali sono state coltivate selettivamente come monostrati (A), staccate dai palloni di coltura tissutale e seminate in micropiastre a 96 pozzetti di attacco ultrabasso per formare sferoidi (B). Il carcinoma polmonare A549, il carcinoma epatico HepG2 / C3A, l'adenocarcinoma ovarico SK-OV-3 e le cellule di carcinoma mammario MCF-7 sono state seminate a 1 × 103-8 × 103 cellule / pozzetto e cresciute fino a 7 giorni per formare singoli sferoidi e ottimizzare la densità di semina degli sferoidi e il tempo di coltivazione mediante osservazione continua e misurazioni planimetriche. Una volta formati, i singoli sferoidi sono stati lavati in un mezzo XF privo di siero e accuratamente seminati in micropiastre per il dosaggio sferoidale, precolate con poli-D-lisina (C). Gli sferoidi sono stati sottoposti ad analisi del flusso extracellulare utilizzando l'analizzatore XFe96 utilizzando diversi protocolli per affrontare: (1) dimensioni sferoidi ottimali per la risposta respiratoria mitocondriale basale; (2) titolazione ottimizzata degli inibitori respiratori mitocondriali; (3) ottimizzazione del posizionamento sferoidale all'interno di pozzetti di micropiastre. (D) Le analisi post XF, la microscopia a contrasto di fase e la quantificazione del DNA sferoide sono state utilizzate per la normalizzazione dei dati e altri saggi in vitro a valle. Fare clic qui per visualizzare una versione più grande di questa figura.
1. Coltivazione di linee cellulari tumorali come sferoidi 3D in vitro
| Linea cellulare | Descrizione | Terreno di coltura | Fonte |
| A549 · | Linea cellulare di carcinoma polmonare | RPMI 1640 · | Collezione europea di colture cellulari autenticate (ECACC) |
| Piruvato di sodio (1 mM) | |||
| Penicillina- Streptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| HepG2/C3A | Linea cellulare del carcinoma epatico, un derivato clonale della linea cellulare HepG2 madre | DMEM · | American Tissue Culture Collection (ATCC) |
| Penicillina- Streptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| MCF7 · | Linea cellulare di adenocarcinoma mammario | RPMI 1640 · | Collezione europea di colture cellulari autenticate (ECACC) |
| Piruvato di sodio (1 mM) | |||
| Penicillina- Streptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| SK-OV-3 · | Linea cellulare di adenocarcinoma ovarico | RPMI 1640 · | Collezione europea di colture cellulari autenticate (ECACC) |
| Piruvato di sodio (1 mM) | |||
| Penicillina- Streptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| Componente | Mezzo di saggio RPMI (volume finale di 50 ml) | ||
| Base Media | Agilent Cavalluccio marino XF RPMI, pH 7,4 | ||
| Glucosio (1 M di brodo sterile) | 11 mM (0,55 mL di soluzione madre) | ||
| L-glutammina (200 mM di stock sterile) | 2 mM (0,5 mL di soluzione madre) | ||
| Piruvato di sodio (100 mM di stock sterile) | 1 mM (0,5 mL di soluzione madre) |
Tabella 1: Supporti della linea cellulare tumorale e composizioni dei media XF.
- Coltivare tutte le linee cellulari utilizzando la tecnica standard di coltura tissutale asettica e confermare che sono prive di micoplasma utilizzando un kit di analisi adatto.
- Coltivare le linee cellulari in palloni di coltura tissutale T75 o equivalenti, utilizzando il mezzo raccomandato (Tabella 1). Coltivare le linee cellulari al 65-80% di confluenza e passarle regolarmente fino ad un massimo di 25 passaggi.
- Risciacquare due volte i palloni di coltura cellulare nella soluzione salina tamponata con fosfato modificato (DBPS) di Dulbecco.
- Staccare le cellule dai palloni con 3 mL del reagente di dissociazione cellulare (vedere la Tabella dei materiali) per 5 minuti a 37 °C e confermare il distacco al microscopio.
- Aspirare delicatamente la sospensione cellulare staccata per garantire una sospensione unicellulare e disattivare il reagente di dissociazione cellulare con 7 mL di terreno di coltura tissutale completo.
- Raccogliere le cellule per centrifugazione a 300 × g per 5 minuti, scartare il surnatante e risospescere le cellule in mezzo completo.
- Contare le cellule utilizzando un emocitometro o un contatore cellulare automatizzato e titolare la densità cellulare desiderata necessaria per la semina.
NOTA: Per seminare un'intera piastra a 96 pozzetti a 100 μL/pozzetto a 4 × 103 cellule/pozzetto, le cellule devono essere titolate a 4 × 104 cellule/mL in un volume raccomandato di 12 mL. - Decantare la sospensione cellulare in un serbatoio sterile ed erogare 100 μL della sospensione cellulare in ciascun pozzetto di una micropiastra repellente per cellule utilizzando un pipettor multicanale.
NOTA: solo i 60 pozzetti interni di una micropiastra devono essere seminati e il resto riempito con DPBS. Ciò formerà una barriera di evaporazione, garantirà l'omogeneità sferoide attraverso la piastra e ridurrà al minimo gli effetti del bordo della piastra. - Centrifugare micropiastre sferoidi a 300 × g per 15 minuti per forzare le cellule in aggregati sciolti.
- Incubare le piastre a 37 °C, 5% CO2 per un minimo di 3 giorni per garantire la formazione di sferoidi.
- Eseguire la microscopia a contrasto di fase utilizzando pratiche di laboratorio standardizzate per monitorare la crescita degli sferoidi. Ricostituire il terreno di coltura cellulare ogni 3 giorni o due volte alla settimana eseguendo uno scambio di mezzo mezzo volume.
2. Sondare il metabolismo energetico mitocondriale di singoli sferoidi utilizzando la tecnologia Extracellular Flux (XF)
- Preparazione del test (un giorno prima)
- Controllare la vitalità sferoidale utilizzando un microscopio a luce invertita con contrasto di fase con ingrandimento 4x per garantire la struttura sferoide intatta, la morfologia e l'uniformità generale tra i campioni.
- Idratare la cartuccia del sensore.
- Aliquota ~20 mL del calibrante in un tubo conico.
- Posizionare il tubo conico contenente il calibrante in un incubatore non CO2 a 37 °C durante la notte.
- Rimuovere il contenuto dal kit di analisi.
- Rimuovere la cartuccia del sensore dalla piastra di utilità e posizionarla a testa in giù sul piano di lavoro accanto alla piastra di utilità.
- Pipettare 200 μL di ddH2O sterile in ciascun pozzetto della piastra di utilità della cartuccia del sensore utilizzando una pipetta P300 multicanale.
- Posizionare la cartuccia del sensore sulla parte superiore della piastra di utilità.
- Verificare che il livello dell'acqua in ciascun pozzo sia sufficientemente alto da immergere le sonde del sensore.
- Trasferire la cartuccia del sensore assemblata in un incubatore non CO2 a 37 °C e lasciarla per una notte.
NOTA: questo passaggio può essere eseguito 12-72 ore prima dell'inizio del test.
- Micropiastra per analisi sferoide del mantello
- Utilizzando tecniche asettiche, aggiungere 30 μL/pozzetto di soluzione sterile di Poli-D-Lisina (0,1 mg/mL) alla micropiastra sferoide e incubarla per 30 minuti a temperatura ambiente.
- Aspirare la soluzione da ciascun pozzetto della micropiastra sferoidale, invertire la piastra e picchiettarla saldamente sulla carta velina per rimuovere qualsiasi soluzione residua.
- Lavare la piastra due volte con 200 μL/pozzetto di ddH2O sterile.
- Dopo il lavaggio finale, capovolgere la micropiastra e picchiettarla saldamente sulla carta velina per rimuovere l'acqua residua.
- Lasciare asciugare la piastra all'aria per 30 minuti prima di utilizzarla o conservarla a 4 °C per un uso futuro.
NOTA: La micropiastra per il dosaggio dello sferoide deve essere rivestita con un adesivo molecolare per garantire che gli sferoidi siano fissati sul fondo della micropiastra. Senza un adesivo molecolare, gli sferoidi possono essere rimossi e interferire con i risultati del test. Altri adesivi molecolari possono anche essere utilizzati come alternativa alla poli-D-lisina per le piastre di precoating. Le piastre precoate possono essere conservate a 4 °C, ma devono essere lasciate in equilibrio a temperatura ambiente prima dell'inizio del test.
- Preparare il mezzo di analisi XF
- Preparare il mezzo RPMI XF, come descritto nella Tabella 1, e il filtro sterile con un filtro a siringa da 0,22 μm
- Preparazione del test (1 ora prima del test)
- Preriscaldare il mezzo di saggio XF RPMI integrato a 37 °C.
- Preriscaldare la micropiastra di analisi sferoidale rivestita in un incubatore non CO2 a 37 °C o in un bagno secco.
- Preparare la cartuccia del sensore.
- Estrarre il tubo conico contenente il calibrante e la cartuccia del sensore dall'incubatore ad aria.
- Rimuovere la cartuccia del sensore dalla piastra di utilità e posizionarla a testa in giù sul piano di lavoro.
- Utilizzando una pipetta multicanale P300, aspirare l'acqua dalla piastra di servizio e scartarla.
- Versare la soluzione di calibrante in un serbatoio di reagente sterile e aggiungere 200 μL/pozzetto del calibrante preriscaldato alla piastra di servizio utilizzando una pipetta multicanale P300.
- Prelevare la cartuccia del sensore e riposizionarla sopra la piastra di utilità, assicurandosi che i sensori siano ben immersi nel calibrante.
- Trasferire nuovamente la cartuccia del sensore assemblata nell'incubatore non CO2 a 37 °C fino a quando non sarà pronta per caricare le soluzioni di iniezione della porta.
- Lavare gli sferoidi con il mezzo di analisi.
- Rimuovere la piastra di coltura sferoidale dall'incubatore a 37 °C, 5% CO2 e osservare gli sferoidi al microscopio per garantirne l'integrità prima delle fasi di trasferimento dello sferoide.
- Caricare tutti i pozzetti della piastra sferoidale con 180 μL/pozzetto di terreno di saggio preriscaldato, compresi eventuali pozzetti di correzione dello sfondo.
- Riempire parzialmente una capsula di Petri di 7 cm con 3 ml del mezzo di saggio.
- Utilizzando una pipetta multicanale caricata con ampie punte di pipetta orifizio, trasferire gli sferoidi dalla piastra di coltura a 96 pozzetti in piastre di Petri da 7 cm impostando il pipettor a un volume di aspirazione di 10-50 μL.
- Sferoidi di semi nella micropiastra di analisi sferoidale pre-rivestita.
- Utilizzando un microscopio di dissezione e un apparecchio lightbox, trasferire gli sferoidi dalla capsula di Petri alla micropiastra di saggio sferoidale come descritto di seguito.
- Impostare il volume di un pipettor a canale singolo dotato di un'ampia punta della pipetta dell'orifizio a 20 μL e aspirare accuratamente un singolo sferoide. Posizionare la punta direttamente al centro di ciascun pozzetto della micropiastra di saggio sferoidale e consentire alla gravità di eluire un singolo sferoide nel centro di ciascun pozzetto, ovvero non espellere alcun mezzo dalla punta della pipetta e consentire l'azione capillare per prelevare lo sferoide dalla punta della pipetta. Per confermare l'eluizione, il contenuto del pipettor può essere reintubato nella capsula di Petri da 7 cm al microscopio.
NOTA: l'eluizione gravitazionale di un singolo sferoide richiede in genere 15-30 s a seconda delle dimensioni / densità dello sferoide. Durante questo periodo, il pipettor non deve essere rimosso. Qualsiasi pozzetto di correzione dello sfondo deve essere privo di sferoidi e contenere solo un mezzo di analisi. Al microscopio, confermare la posizione di ciascun sferoide. Ogni sferoide dovrebbe idealmente essere posizionato all'interno del centro di ciascun pozzetto. - Una volta che tutti gli sferoidi sono stati trasferiti nella micropiastra di analisi sferoidale, trasferire la piastra in un incubatore non CO2 a 37 °C per un minimo di 1 ora prima del test.
- Impostare il volume di un pipettor a canale singolo dotato di un'ampia punta della pipetta dell'orifizio a 20 μL e aspirare accuratamente un singolo sferoide. Posizionare la punta direttamente al centro di ciascun pozzetto della micropiastra di saggio sferoidale e consentire alla gravità di eluire un singolo sferoide nel centro di ciascun pozzetto, ovvero non espellere alcun mezzo dalla punta della pipetta e consentire l'azione capillare per prelevare lo sferoide dalla punta della pipetta. Per confermare l'eluizione, il contenuto del pipettor può essere reintubato nella capsula di Petri da 7 cm al microscopio.
- Utilizzando un microscopio di dissezione e un apparecchio lightbox, trasferire gli sferoidi dalla capsula di Petri alla micropiastra di saggio sferoidale come descritto di seguito.
3. Preparazione e caricamento dei composti nella cartuccia del sensore per i saggi XF
| Strategia di iniezione | Compound (Porto) | Volume iniziale del microwell XFe96 (μL) | Concentrazione finale del pozzo desiderata | Volume della porta (μL) | Volume finale del microwell XFe96 post iniezione (μL) | Concentrazione delle scorte di lavoro |
| 1 | Oligomicina (A) | 180 | 3 ug/mL | 20 | 200 | 30 μg/mL |
| Rotenone (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimicina A (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| Rotenone (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimicina A (B) | 200 | 2 μM | 20 | 220 | 22 μM |
Tabella 2: Concentrazioni di composti mitocondriali per sondare il metabolismo energetico mitocondriale di singoli sferoidi 3D utilizzando l'analizzatore XFe96.
- Preparare le concentrazioni di magazzino di ciascun composto come indicato nella Tabella 2 utilizzando un mezzo di saggio RPMI XF completamente integrato e preriscaldato.
- Orientare la piastra della cartuccia (accoppiata alla piastra di utilità) in base alla colonna, 1-12 da sinistra a destra.
- Se si utilizza una guida di carico, posizionarla sopra la piastra della cartuccia in base alla procedura di caricamento del pozzo, ad esempio, se la porta A viene caricata per prima, assicurarsi che A sia visibile nell'angolo in alto a sinistra della guida.
- Trasferire la soluzione di lavoro di ciascun composto in un serbatoio adatto e, utilizzando una pipetta multicanale P100 calibrata, erogare 20 μL in tutte le porte corrispondenti. Ripetere l'operazione per ogni composto nelle porte rimanenti.
NOTA: se non vengono utilizzate porte sulla piastra della cartuccia del sensore, queste possono essere lasciate vuote o riempite con il supporto di analisi. Se viene utilizzata solo una selezione di una specifica lettera di porta, assicurarsi che le altre porte corrispondenti a tale lettera siano caricate con un supporto di analisi; in caso contrario, l'aria verrà iniettata nel pozzo, compromettendo i risultati in quei pozzi. - Dopo il caricamento della porta, rimuovere le guide di caricamento delle piastre (se utilizzate) e preparare l'analizzatore per il caricamento della cartuccia del sensore.
NOTA: se il test non viene eseguito immediatamente dopo aver caricato le porte, riposizionare il coperchio sulla cartuccia del sensore e rimettere la piastra nell'incubatore ad aria a 37 °C fino a quando non sarà pronta per essere caricata nella macchina.
4. Progettazione del test, strategie di iniezione e acquisizione dati
- Esecuzione del test
- Accendere l'analizzatore e collegarlo al controller (computer).
NOTA: questo può essere verificato dallo stato della connessione dello strumento nel pannello widget del software Wave Controller. - Passare alla pagina dei modelli nel software WAVE, trovare il file del modello di analisi per l'esperimento e fare doppio clic per aprirlo.
NOTA: se il modello di analisi non viene visualizzato nella vista Modelli , importare il file modello nella cartella del modello da un'unità di rete condivisa o da un'unità flash USB. - Per avviare il test, fare clic sulla scheda Esegui test .
NOTA: se le definizioni di gruppo sono state allocate correttamente all'interno della mappa della piastra, il test sarà pronto per essere eseguito come indicato dal segno di spunta verde sul lato destro della pagina. In questa fase, qualsiasi informazione aggiuntiva può essere inserita nella pagina di riepilogo del test o nella pagina lasciata vuota; procedere al passaggio successivo. A causa della penetrazione ritardata dei modulatori mitocondriali negli sferoidi microtissue 3D (Figura 2), utilizzare le informazioni sul protocollo di misurazione descritte nella Tabella 3.
- Accendere l'analizzatore e collegarlo al controller (computer).
| Periodo di misurazione | Numero di iniezione e porta | Dettagli di misurazione | Durata del periodo (h:min:s) |
| Taratura | Non applicabile | Gli analizzatori XF eseguono sempre questa calibrazione per assicurarsi che le misurazioni siano accurate | 00:20:00 (questa è una media e può variare tra le macchine) |
| Equillibrazione | Non applicabile | L'equilibrio avviene dopo la calibrazione ed è raccomandato. | 00:10:00 |
| Basale | Non applicabile | Cicli = 5 | 00:30:00 |
| Mix = 3:00 | |||
| Attendi = 0:00 | |||
| Misura = 3:00 | |||
| Oligomicina / BAM15 | Iniezione 1 (Porta A) | Cicli = 10 | 01:00:00 |
| Mix = 3:00 | |||
| Attendi = 0:00 | |||
| Misura = 3:00 | |||
| Rotenone + antimicina A | Iniezione 2 (Porta B) | Cicli = 10 | 01:00:00 |
| Mix = 3:00 | |||
| Attendi = 0:00 | |||
| Misura = 3:00 | |||
| Tempo totale: | 03:00:00 | ||
Tabella 3: Configurazione del protocollo per sondare il metabolismo energetico mitocondriale di singoli sferoidi 3D utilizzando l'analizzatore XFe96.
- Fare clic su Avvia esecuzione per visualizzare la finestra di dialogo Salva percorso .
- Immettere la posizione di salvataggio per il file dei risultati e posizionare la cartuccia del sensore assemblata sul vassoio termico che appare dalla porta sul lato dell'analizzatore. Attendere che il vassoio termico si apra automaticamente e che sullo schermo venga visualizzato il messaggio Load Calibrant Utility Plate . Prima di seguire le istruzioni visualizzate sullo schermo, assicurarsi i) il corretto adattamento della cartuccia del sensore sulla piastra dell'utilità, ii) che il coperchio venga rimosso dalla cartuccia del sensore e iii) il corretto orientamento della cartuccia del sensore sulla piastra dell'utilità.
- Seguire i comandi sullo schermo per avviare la calibrazione della cartuccia del sensore.
NOTA: Il tempo necessario per completare la calibrazione è di circa 10-20 minuti (per saggi a 37 °C). - Dopo la calibrazione della cartuccia del sensore, caricare la micropiastra sferoide nell'analizzatore seguendo le istruzioni visualizzate sullo schermo sul Wave Controller per avviare la fase di bilanciamento di 12 minuti.
NOTA: le caselle verdi con segni di spunta bianchi indicano una calibrazione "buona" per quel pozzo. Se alcuni pozzi non riescono a fornire una calibrazione "buona", saranno indicati con una scatola rossa e una croce bianca. Tali pozzi devono essere annotati ed esclusi da qualsiasi analisi dopo che il test è stato completato utilizzando la scheda del test di modifica . - Attendere che l'analizzatore inizi automaticamente ad acquisire le misurazioni di base dopo che la macchina ha completato la fase di equilibratura (come indicato nel protocollo dello strumento).
- Per completare l'esperimento, seguire i comandi sullo schermo sul controller WAVE.
NOTA: una volta che la micropiastra sferoidale è stata rimossa dall'analizzatore, scartare la cartuccia del sensore e mettere da parte la piastra sferoide per ulteriori analisi, se necessario (ad esempio, quantificazione del DNA a doppio filamento (ds).) Se la micropiastra non è necessaria per ulteriori analisi, può essere scartata insieme alla cartuccia del sensore. - Attendere la visualizzazione della finestra di dialogo del test e visualizzare i risultati o tornare alla visualizzazione dei modelli .
5. Strategie di normalizzazione e analisi dei dati - normalizzazione post-test e saggi a valle (passaggi facoltativi)
- Normalizzazione dei dati
- Per normalizzare i dati sferoidi, fare riferimento alla serie di protocolli pertinenti alle strategie di normalizzazione dei dati per il calcolo delle dimensioni e del volume degli sferoidi e la quantificazione del dsDNA nei saggi sferoidi. Questi sono stati inclusi come file supplementari; vedere Il file supplementare 1 e il file supplementare 2.
- Analisi dei dati
- Per esportare i dati in uno dei generatori di analisi automatizzati, seguire i comandi di esportazione dei dati sul controller WAVE e selezionare il generatore di esportazione che corrisponde al tipo di test. In alternativa, esporta il file di dati e caricalo in Seahorse Analytics.
NOTA: lo svantaggio dei generatori di report e dell'analisi Seahorse è che l'analisi dei dati è limitata al modo in cui è progettato il test XF e non consente di prendere le medie tra i cicli di misurazione. L'esportazione manuale dei set di dati dal software dello strumento consente la preferenza dell'utente a questo proposito. Dato che la strategia di iniezione per valutare la respirazione mitocondriale degli sferoidi 3D sarà probabilmente diversa da quella di un tipico test "MitoStress", sono stati sviluppati una serie di modelli di fogli di calcolo per aiutare ad analizzare questi set di dati, specifici per le colture cellulari 3D e saranno forniti su richiesta. Questi file modello di dati forniranno dati sui principali parametri respiratori mitocondriali dettagliati e spiegati nella Figura 2. - Per analizzare i dati, esportare i dati come report del foglio di calcolo dal software del controller WAVE e utilizzare un modello di foglio di calcolo indipendente per l'analisi.
- Per esportare i dati in uno dei generatori di analisi automatizzati, seguire i comandi di esportazione dei dati sul controller WAVE e selezionare il generatore di esportazione che corrisponde al tipo di test. In alternativa, esporta il file di dati e caricalo in Seahorse Analytics.
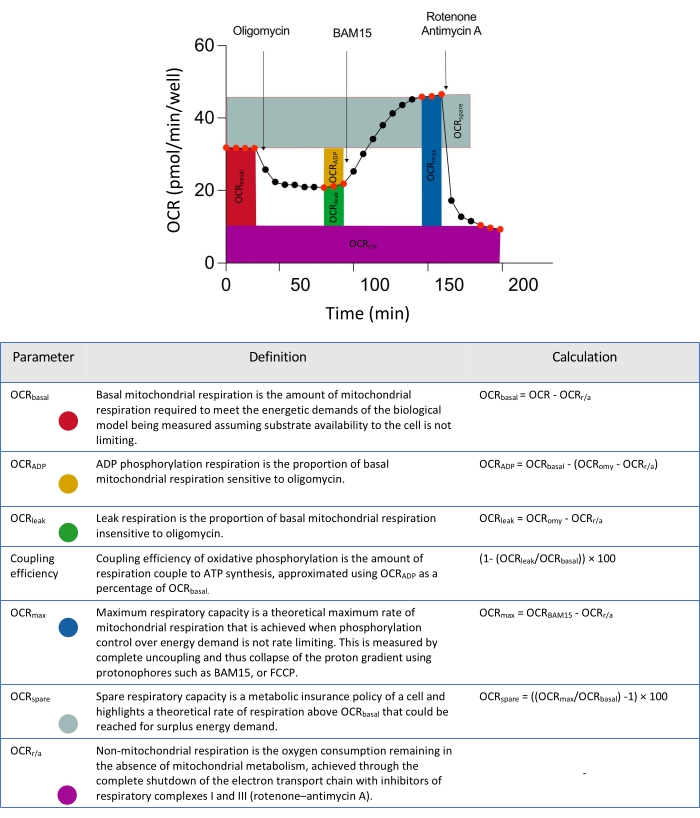
Figura 2: Descrittori schematici per parametri derivati da analisi di dati di flusso extracellulare. Abbreviazione: OCR = tasso di consumo di ossigeno. Fare clic qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per ottenere sferoidi compatti e ben formati, ogni linea cellulare è stata ottimizzata individualmente per la densità di semina e la durata della coltivazione (Figura 3). Le linee cellulari A549, HepG2/C3A e SK-OV-3 inizialmente formavano aggregati sciolti che non progredivano verso sferoidi rotondi con perimetri chiaramente definiti fino a dopo 7 giorni in coltura. Al contrario, le cellule MCF-7 potrebbero formare sferoidi entro 3 giorni. C'era una chiara correlazione tra la densità iniz...
Access restricted. Please log in or start a trial to view this content.
Discussione
Principali risultati e realizzazioni
Questo documento fornisce un protocollo dettagliato per sondare il metabolismo energetico mitocondriale di singoli sferoidi 3D utilizzando una serie di linee cellulari derivate dal cancro con l'analizzatore XF XFe96. Viene sviluppato e descritto un metodo per la rapida coltivazione di sferoidi cellulari A549, HepG2/C3A, MCF7 e SK-OV-3 utilizzando tecnologie cellorepellenti per l'aggregazione forzata. Questo protocollo affronta molte considerazioni sulla sonda del m...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
N.J.C è stato supportato da un BBSRC MIBTP CASE Award con Sygnature Discovery Ltd (BB/M01116X/1, 1940003)
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
Riferimenti
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
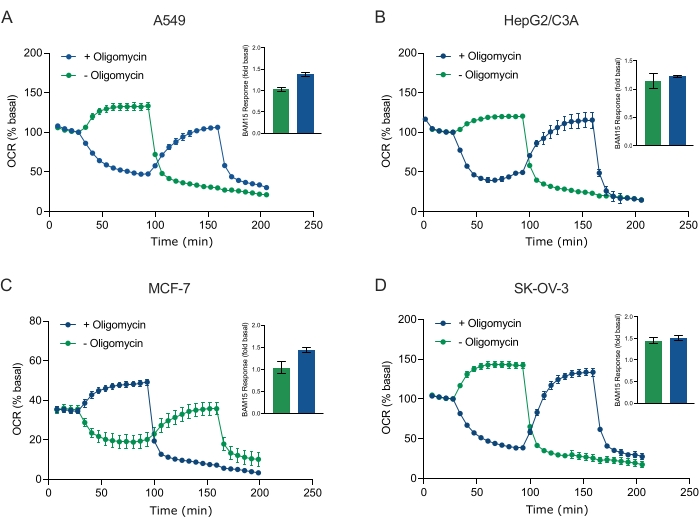
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
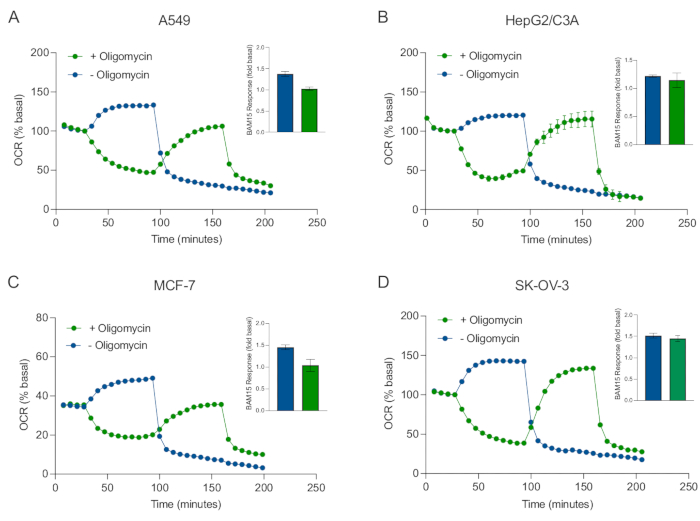
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon