É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Explorando o metabolismo de energia mitocondrial de spheróides microtissue 3D únicos usando análise de fluxo extracelular
Neste Artigo
Erratum Notice
Resumo
Esses protocolos ajudarão os usuários a sondar o metabolismo de energia mitocondrial em esferoides derivados de células cancerígenas 3D usando a análise de fluxo extracelular do Seahorse.
Resumo
Agregados celulares tridimensionais (3D), denominados esferoides, tornaram-se a vanguarda da cultura celular in vitro nos últimos anos. Em contraste com as células cultivadas como monocamadas bidimensionais e unicelulares (cultura 2D), a cultura celular esfóide promove, regula e suporta arquitetura fisiológica celular e características que existem in vivo, incluindo a expressão de proteínas de matriz extracelular, sinalização celular, expressão genética, produção de proteínas, diferenciação e proliferação. A importância da cultura 3D tem sido reconhecida em muitas áreas de pesquisa, incluindo oncologia, diabetes, biologia de células-tronco e engenharia de tecidos. Na última década, métodos aprimorados foram desenvolvidos para produzir esferoides e avaliar sua função metabólica e destino.
Analisadores de fluxo extracelular (XF) têm sido usados para explorar a função mitocondrial em microtissues 3D, como esferoides usando uma placa de captura de ilhotas XF24 ou uma microplaca esferoide XFe96. No entanto, protocolos distintos e a otimização da sondagem do metabolismo de energia mitocondrial em esferoides usando a tecnologia XF não foram descritos em detalhes. Este artigo fornece protocolos detalhados para sondar o metabolismo de energia mitocondrial em esferoides 3D únicos usando microplapes esferoides com o analisador XFe96 XF. Usando diferentes linhas de células cancerígenas, a tecnologia XF é demonstrada ser capaz de distinguir entre a respiração celular em esferoides 3D não apenas de tamanhos diferentes, mas também diferentes volumes, números celulares, conteúdo de DNA e tipo.
As concentrações compostas de efeito mitocondrial ideal de oligomicina, BAM15, rotenona e antimicina A são usadas para sondar parâmetros específicos do metabolismo de energia mitocondrial em esferoides 3D. Este artigo também discute métodos para normalizar dados obtidos a partir de esferoides e aborda muitas considerações que devem ser consideradas ao explorar o metabolismo esferoide usando a tecnologia XF. Este protocolo ajudará a impulsionar a pesquisa em modelos esferoides in vitro avançados.
Introdução
Os avanços em modelos in vitro em pesquisa biológica progrediram rapidamente nos últimos 20 anos. Tais modelos agora incluem modalidades organ-on-a-chip, organoides e esferoides microtissue 3D, todos os quais se tornaram um foco comum para melhorar a tradução entre estudos in vitro e in vivo. O uso de modelos in vitro avançados, particularmente esferoides, abrange diversos campos de pesquisa, incluindo engenharia de tecidos, pesquisa de células-tronco, câncer e biologia de doenças 1,2,3,4,5,6,7, e testes de segurança, incluindo toxicologia genética 8,9,10, toxicologia de nanomateriais11, 12,13,14, e testes de segurança e eficácia de medicamentos 8,15,16,17,18,19.
A morfologia celular normal é fundamental para o fenótipo biológico e a atividade. A colheita de células em esferoides microtissue 3D permite que as células adotem uma morfologia, função fenotípica e arquitetura, mais semelhantes às observadas in vivo , mas difíceis de capturar com técnicas clássicas de cultura celular monocamada. Tanto in vivo quanto in vitro, a função celular é diretamente impactada pelo microambiente celular, que não se limita à comunicação e programação celular (por exemplo, formações de junção celular-célula, oportunidades de formação de nichos celulares); exposição celular a hormônios e fatores de crescimento nos ambientes imediatos (por exemplo, exposição à citocina celular como parte de uma resposta inflamatória); composição de matrizes físicas e químicas (por exemplo, se as células são cultivadas em plástico de cultura tecidual rígida ou um ambiente de tecido elástico); e, mais importante, como o metabolismo celular é impactado pela nutrição e acesso ao oxigênio, bem como pelo processamento de resíduos metabólicos, como o ácido láctico.
A análise do fluxo metabólico é uma maneira poderosa de examinar o metabolismo celular dentro de sistemas in vitro definidos. Especificamente, a tecnologia XF permite a análise de mudanças ao vivo e em tempo real nos bioenergésicos celulares de células e tecidos intactos. Dado que muitos eventos metabólicos intracelulares ocorrem dentro da ordem de segundos a minutos, abordagens funcionais em tempo real são primordiais para entender mudanças em tempo real no fluxo metabólico celular em células intactas e tecidos in vitro.
Este artigo fornece protocolos para o cultivo das linhas celulares derivadas do câncer A549 (adenocarcinoma pulmonar), HepG2/C3A (carcinoma hepatocelular), MCF-7 (adenocarcinoma mamário) e SK-OV-3 (adenocarcinoma ovariano) como modelos esferoides in vitro 3D usando abordagens de agregação forçada (Figura 1). Ele também (i) descreve em detalhes como sondar o metabolismo de energia mitocondrial de esferoides 3D únicos usando o analisador Agilent XFe96 XF, (ii) destaca maneiras de otimizar os ensaios XF usando spheroids 3D únicos, e (iii) discute considerações e limitações importantes de sondagem do metabolismo esferoide 3D usando essa abordagem. Mais importante, este artigo descreve como são coletados conjuntos de dados que permitem o cálculo da taxa de consumo de oxigênio (OCR) para determinar a fosforilação oxidativa e, portanto, a função mitocondrial em esferoides celulares. Embora não seja analisada para este protocolo, a taxa de acidificação extracelular (ECAR) é outro parâmetro que é medido ao lado de dados OCR em experimentos XF. No entanto, o ECAR é muitas vezes mal ou incorretamente interpretado a partir de conjuntos de dados XF. Nós fornecemos um comentário sobre as limitações de cálculo do ECAR seguindo abordagens básicas do fabricante de tecnologia.
Access restricted. Please log in or start a trial to view this content.
Protocolo
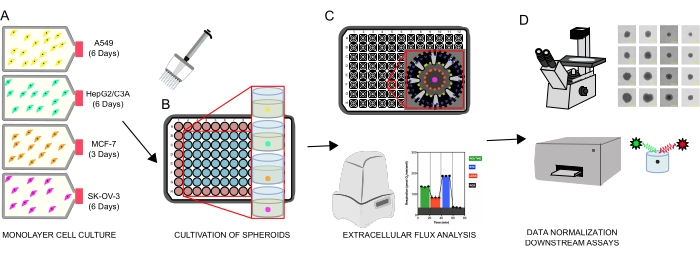
Figura 1: Fluxo de trabalho gráfico para a geração de esferoides celulares, análise de fluxo extracelular e ensaios a jusante. Quatro linhas de células cancerígenas foram seletivamente cultivadas como monocamadas (A), separadas dos frascos da cultura tecidual, e semeadas em microplacas de 96 poços para formar esferoides (B). Carcinoma pulmonar A549, carcinoma hepg2/C3A, As células de adenocarcinoma ovariano SK-OV-3 e as células de carcinoma mamário MCF-7 foram semeadas em 1 × 103-8 × 103 células/bem e cresceram até 7 dias para formar esferoides únicos e otimizar a densidade e o tempo de cultivo de sementes e cultivo e esferoide por meio de observação contínua e medidas planimétricas. Uma vez formados, os sferóides únicos foram lavados em um meio XF livre de soro e cuidadosamente semeados em microplacas de ensaio esferoide, pré-revestidas com poli-D-lysine (C). Os spheroids foram submetidos à análise de fluxo extracelular usando o analisador XFe96 usando vários protocolos para abordar: (1) tamanho esferoide ideal para resposta à respiração mitocondrial basal; (2) titulação otimizada de inibidores respiratórios mitocondriais; (3) otimização da colocação esferoide dentro de poços de microplacão. (D) Análises pós-XF, microscopia de contraste de fase e quantificação de DNA esferoide foram utilizadas para normalização de dados e outros ensaios in vitro a jusante. Clique aqui para ver uma versão maior desta figura.
1. Cultivo de linhas de células cancerosas como esferoides in vitro 3D
| Linha celular | Descrição | Meio de cultura | Fonte |
| A549 | Linha celular de carcinoma pulmonar | RPMI 1640 | Coleção Europeia de Culturas Celulares Autenticadas (ECACC) |
| Piruvato de sódio (1 mM) | |||
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| HepG2/C3A | Linha celular de carcinoma hepático, um derivado clonal da linha de células HepG2 pai | DMEM | Coleção Americana de Cultura de Tecidos (ATCC) |
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| MCF7 | Linha celular de adenocarcinoma mamário | RPMI 1640 | Coleção Europeia de Culturas Celulares Autenticadas (ECACC) |
| Piruvato de sódio (1 mM) | |||
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| SK-OV-3 | Linha celular adenocarcinoma ovariana | RPMI 1640 | Coleção Europeia de Culturas Celulares Autenticadas (ECACC) |
| Piruvato de sódio (1 mM) | |||
| Penicilina- Estreptomicina - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| Componente | Meio de ensaio RPMI (volume final de 50 mL) | ||
| Meio base | Agilent Seahorse XF RPMI, pH 7.4 | ||
| Glicose (1 M estoque estéril) | 11 mM (solução de estoque de 0,55 mL) | ||
| L-glutamina (estoque estéril de 200 mM) | 2 mM (0,5 mL de solução de estoque) | ||
| Piruvato de sódio (estoque estéril de 100 mM) | 1 mM (0,5 mL de solução de estoque) |
Tabela 1: Mídia de linha celular cancerosa e composições de mídia XF.
- Cultize todas as linhas celulares usando a técnica padrão de cultura de tecido asséptico e confirme que elas estão livres de mycoplasma usando um kit de ensaio adequado.
- Cultura as linhas celulares em frascos de cultura tecidual T75 ou equivalente, utilizando o meio recomendado (Tabela 1). Cultume as linhas celulares para 65-80% de confluência e passagem-as regularmente até um máximo de 25 passagens.
- Enxágüe os frascos de cultura celular duas vezes na solução salina modificada de fosfato (DBPS) modificada de Dulbecco.
- Retire as células dos frascos com 3 mL do reagente de dissociação celular (ver a Tabela de Materiais) por 5 min a 37 °C e confirme o descolamento por microscopia.
- Aspire suavemente a suspensão da célula isolada para garantir uma suspensão unicelular e desativar o reagente de dissociação celular com 7 mL de meio completo de cultura tecidual.
- Recolher as células por centrifugação a 300 × g por 5 min, descartar o supernascedor e resuspensar as células em meio completo.
- Conte as células usando um hemótmetro ou um contador celular automatizado e titulação à densidade celular desejada necessária para semeadura.
NOTA: Para semear uma placa inteira de 96 poços a 100 μL/well a 4 × 103 células/bem, as células devem ser tituladas a 4 × 104 células/mL em um volume recomendado de 12 mL. - Decante a suspensão da célula em um reservatório estéril e dispense 100 μL da suspensão celular em cada poço de uma microplaca repelente celular usando um pipettor multicanal.
NOTA: Apenas os 60 poços internos de uma microplaca devem ser semeados e o restante preenchido com DPBS. Isso formará uma barreira de evaporação, garantirá a homogeneidade esferoide através da placa e minimizará os efeitos da borda da placa. - Centrífugas microplatas esferoides a 300 × g por 15 minutos para forçar as células a agregados soltos.
- Incubar as placas a 37 °C, 5% de CO2 por um mínimo de 3 dias para garantir a formação de esferoides.
- Realizar microscopia de contraste de fase utilizando práticas laboratoriais padronizadas para monitorar o crescimento de esferoides. Reponha o meio de cultura celular a cada 3 dias ou duas vezes por semana, realizando uma troca média de meio volume.
2. Sondagem do metabolismo de energia mitocondrial de esferoides únicos usando tecnologia de fluxo extracelular (XF)
- Preparação de ensaios (um dia antes)
- Verifique a viabilidade esferoide usando um microscópio de luz invertida com contraste de fase na ampliação de 4x para garantir estrutura eferóide intacta, morfologia e uniformidade geral entre as amostras.
- Hidrate o cartucho do sensor.
- Alíquota ~20 mL do calibrante em um tubo cônico.
- Coloque o tubo cônico contendo o calibrante em uma incubadora não CO2 37 °C durante a noite.
- Remova o conteúdo do kit de ensaio.
- Remova o cartucho do sensor da placa do utilitário e coloque-o de cabeça para baixo na bancada ao lado da placa de utilidade.
- Pipeta 200 μL de ddH2O estéril em cada poço da placa de utilidade do cartucho do sensor usando uma pipeta P300 multicanal.
- Coloque o cartucho do sensor em cima da placa de utilidade.
- Verifique se o nível de água em cada poço é alto o suficiente para submergir as sondas do sensor.
- Transfira o cartucho do sensor montado para uma incubadora não-CO2 de 37 °C e deixe-o durante a noite.
NOTA: Esta etapa pode ser realizada 12-72 h antes do início do ensaio.
- Talo spheroid ensaio microplato
- Utilizando técnicas assépticas, adicione 30 μL/poço de solução estéril de Poli-D-Lysine (0,1 mg/mL) à microplacão e incuba-a por 30 minutos à temperatura ambiente.
- Aspire a solução de cada poço da microplacão esferoide, inverta a placa e bata-a firmemente no papel de tecido para remover qualquer solução residual.
- Lave a placa duas vezes com 200 μL/poço de ddH2O estéril.
- Após a lavagem final, inverta a microplacão e bata-a firmemente no papel de tecido para remover qualquer água residual.
- Deixe a placa secar ao ar por 30 minutos antes de usá-la ou armazená-la a 4 °C para uso futuro.
NOTA: A microplacão de ensaio esferoide deve ser revestida com um adesivo molecular para garantir que os esferoides sejam fixados na parte inferior da microplacão. Sem um adesivo molecular, os esferoides podem se desalojar e interferir com os resultados do ensaio. Outros adesivos moleculares também podem ser usados como uma alternativa à Poli-D-Lysine para pré-revestimento de placas. As placas pré-revestidas podem ser armazenadas a 4 °C, mas devem ser deixadas para equilibrar a temperatura ambiente antes do início do ensaio.
- Prepare o meio de ensaio XF
- Prepare o meio XF RPMI, conforme detalhado na Tabela 1, e filtro estéril com um filtro de seringa de 0,22 μm
- Preparação de ensaio (1h antes do ensaio)
- Pré-aquecimento do ensaio XF RPMI suplementado médio a 37 °C.
- Pré-aquecimento o ensaio esferoide revestido microplaca em uma incubadora não CO2 37 °C ou banho seco.
- Prepare o cartucho do sensor.
- Retire o tubo cônico contendo o calibrante e o cartucho do sensor da incubadora de ar.
- Remova o cartucho do sensor da placa do utilitário e coloque-o de cabeça para baixo na superfície de trabalho.
- Usando uma pipeta multicanal P300, aspire a água da placa de utilidade e descarte-a.
- Despeje a solução calibrante em um reservatório de reagente estéril e adicione 200 μL/poço do calibrante pré-caçado à placa de utilidade usando uma pipeta multicanal P300.
- Pegue o cartucho do sensor e coloque-o de volta em cima da placa de utilidade, garantindo que os sensores estejam bem submersos no calibrante.
- Transfira o cartucho de sensor montado de volta para a incubadora não-CO2 37 °C até estar pronto para carregar as soluções de injeção da porta.
- Lave os esferoides com o meio de ensaio.
- Remova a placa de cultura esferoide da incubadora de 37 °C, 5% de CO2 e observe os esferoides sob o microscópio para garantir sua integridade antes das etapas de transferência de esferoide.
- Carregue todos os poços da placa esferoide com 180 μL/poço de meio de ensaio pré-armado, incluindo quaisquer poços de correção de fundo.
- Encha parcialmente uma placa de Petri de 7 cm com 3 mL do meio de ensaio.
- Usando uma pipeta multicanal carregada com pontas de pipeta de orifício largo, transfira os esferoides da placa de cultura de 96 poços em pratos petri de 7 cm, colocando o pipettor em um volume de aspiração de 10-50 μL.
- Esferoides de sementes na microplacão de ensaio esferoide pré-revestido.
- Usando um microscópio de dissecção e um aparelho de caixa de luz, transfira os esferoides da placa de Petri para a microplaca de ensaio esferoide conforme detalhado abaixo.
- Defina o volume de um pipettor de um único canal equipado com uma ponta de pipeta de orifício largo para 20 μL e aspire cuidadosamente um único esferoide. Coloque a ponta diretamente no centro de cada poço da microplacão de ensaio esferoide e deixe que a gravidade esfolóide no centro de cada poço, ou seja, não expulse nenhum meio da ponta pipeta e permita que a ação capilar retire o esferoide da ponta pipeta. Para confirmar a eluição, o conteúdo do pipettor pode ser pipetado de volta para a placa de Petri de 7 cm sob o microscópio.
NOTA: A eluição de gravidade de um único esferoide normalmente leva 15-30 s, dependendo do tamanho/densidade esferoide. Durante este tempo, o pipettor não deve ser removido. Todos os poços de correção de fundo devem estar livres de esferoides e conter apenas o meio de ensaio. Sob o microscópio, confirme a posição de cada esferoide. Cada esferoide deve ser posicionado idealmente dentro do centro de cada poço. - Uma vez que todos os esferoides tenham sido transferidos para a microplaca de ensaio esferoide, transfira a placa para uma incubadora não-CO2 a 37 °C por um mínimo de 1h antes do ensaio.
- Defina o volume de um pipettor de um único canal equipado com uma ponta de pipeta de orifício largo para 20 μL e aspire cuidadosamente um único esferoide. Coloque a ponta diretamente no centro de cada poço da microplacão de ensaio esferoide e deixe que a gravidade esfolóide no centro de cada poço, ou seja, não expulse nenhum meio da ponta pipeta e permita que a ação capilar retire o esferoide da ponta pipeta. Para confirmar a eluição, o conteúdo do pipettor pode ser pipetado de volta para a placa de Petri de 7 cm sob o microscópio.
- Usando um microscópio de dissecção e um aparelho de caixa de luz, transfira os esferoides da placa de Petri para a microplaca de ensaio esferoide conforme detalhado abaixo.
3. Preparação e carregamento de compostos no cartucho do sensor para ensaios XF
| Estratégia de Injeção | Composto (Porta) | Volume de partida de microwell XFe96 (μL) | Concentração final de poços desejada | Volume da porta (μL) | Injeção final de microwell volume XFe96 (μL) | Concentração de estoque de trabalho |
| 1 | Oligomicina (A) | 180 | 3 ug/mL | 20 | 200 | 30 μg/mL |
| Rotenone (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimicina A (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| Rotenone (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimicina A (B) | 200 | 2 μM | 20 | 220 | 22 μM |
Tabela 2: Concentrações de compostos mitocondriais para sondar o metabolismo de energia mitocondrial de esferoides 3D únicos usando o Analisador XFe96.
- Prepare as concentrações de estoque de trabalho de cada composto, conforme observado na Tabela 2 usando um meio de ensaio XF RPMI totalmente suplementado e pré-equipado.
- Oriente a placa do cartucho (acoplado à placa de utilidade) em termos de coluna, 1-12 da esquerda para a direita.
- Se usar um guia de carga, coloque-o sobre a placa do cartucho de acordo com o procedimento de carregamento de poço, por exemplo, se a porta A for carregada primeiro, certifique-se de que A esteja visível no canto superior esquerdo da guia.
- Transfira a solução de funcionamento de cada composto para um reservatório adequado e, utilizando uma pipeta multicanal P100 calibrada, dispense 20 μL em todas as portas correspondentes. Repita cada composto nas portas restantes.
NOTA: Se não forem utilizadas as portas na placa do cartucho do sensor, estas podem ser deixadas vazias ou preenchidas com meio de ensaio. Se houver apenas uma seleção de uma letra de porta específica, certifique-se de que as outras portas correspondentes a essa letra estejam carregadas com meio de ensaio; caso contrário, o ar será injetado no poço, comprometendo os resultados nesses poços. - Após o carregamento da porta, remova as guias de carregamento da placa (se utilizada) e prepare o analisador para carregar o cartucho do sensor.
NOTA: Se o ensaio não estiver sendo executado imediatamente após o carregamento das portas, coloque a tampa de volta no cartucho do sensor e coloque a placa de volta na incubadora de ar de 37 °C até estar pronta para carregar na máquina.
4. Projeto de ensaio, estratégias de injeção e aquisição de dados
- Executando o ensaio
- Ligue o analisador e conecte-se ao controlador (computador).
NOTA: Isso pode ser verificado pelo status de conexão de instrumento no painel de widget do software Wave Controller. - Navegue até a página de modelos no software WAVE, encontre o arquivo de modelo de ensaio para o experimento e clique duas vezes para abri-lo.
NOTA: Se o modelo de ensaio não aparecer na exibição Modelos , importe o arquivo de modelo para a pasta de modelo de uma unidade de rede compartilhada ou unidade flash USB. - Para iniciar o ensaio, clique na guia Executar ensaios .
NOTA: Se as definições de grupo tiverem sido corretamente alocadas dentro do mapa da placa, o ensaio estará pronto para ser executado conforme indicado pelo carrapato verde no lado direito da página. Nesta fase, qualquer informação adicional pode ser inserção na página de resumo do ensaio ou na página deixada em branco; prossiga para o próximo passo. Devido à penetração retardada de moduladores mitocondriais em esferoides microtissue 3D (Figura 2), utilize as informações do protocolo de medição descritas na Tabela 3.
- Ligue o analisador e conecte-se ao controlador (computador).
| Período de medição | Número de injeção e porta | Detalhes de medição | Duração do período (h:min:s) |
| Calibração | Não é aplicável | Os analisadores XF sempre realizam esta calibração para garantir que as medidas sejam precisas | 00:20:00 (esta é uma média e pode variar entre máquinas) |
| Equillibração | Não é aplicável | O equilíbrio ocorre após a calibração e é recomendado. | 00:10:00 |
| Basal | Não é aplicável | Ciclos = 5 | 00:30:00 |
| Mix = 3:00 | |||
| Espera = 0:00 | |||
| Medida = 3:00 | |||
| Oligomicina / BAM15 | Injeção 1 (Porta A) | Ciclos = 10 | 01:00:00 |
| Mix = 3:00 | |||
| Espera = 0:00 | |||
| Medida = 3:00 | |||
| Rotenona + antimicina A | Injeção 2 (Porta B) | Ciclos = 10 | 01:00:00 |
| Mix = 3:00 | |||
| Espera = 0:00 | |||
| Medida = 3:00 | |||
| Tempo total: | 03:00:00 | ||
Tabela 3: Configuração do protocolo para sondagem do metabolismo de energia mitocondrial de esferoides 3D únicos usando o Analisador XFe96.
- Clique em iniciar a execução para trazer a caixa de diálogo de localização de salvamento .
- Digite o local de salvamento para o arquivo de resultado e coloque o cartucho do sensor montado na bandeja térmica que aparece da porta na lateral do analisador. Aguarde que a bandeja térmica abra automaticamente e a tela exiba a mensagem de placa de utilidade do calibrador de carga. Antes de seguir as instruções na tela, certifique-se de que i) ajuste adequado do cartucho do sensor na placa utilitário, ii) a tampa é removida do cartucho do sensor, e iii) orientação correta do cartucho do sensor na placa do utilitário.
- Siga os comandos na tela para iniciar a calibração do cartucho do sensor.
NOTA: O tempo necessário para completar a calibração é de aproximadamente 10-20 min (para ensaios a 37 °C). - Após a calibração do cartucho do sensor, carregue a microplaca esferoide no analisador seguindo as instruções na tela no Controlador de Ondas para iniciar a etapa de equilíbrio de 12 minutos.
NOTA: Caixas verdes com carrapatos brancos indicam uma calibração "boa" para esse poço. Se algum poço não fornecer uma calibração "boa", eles serão indicados com uma caixa vermelha e cruz branca. Esses poços devem ser observados e excluídos de qualquer análise após a conclusão do ensaio usando a guia de ensaio de modificação . - Aguarde que o analisador comece automaticamente a adquirir medições de linha de base após a máquina ter concluído a etapa de equilíbrio (conforme descrito no protocolo do instrumento).
- Para completar o experimento, siga os comandos na tela no controlador WAVE.
NOTA: Uma vez que a microplacã esferoide tenha sido removida do analisador, descarte o cartucho do sensor e reserve a placa esferoide para análise posterior, se necessário (por exemplo, quantificação de DNA de dupla fita). Se a microplacão não for necessária para análise suplementar, ela pode ser descartada junto com o cartucho do sensor. - Aguarde que a caixa de diálogo de ensaio apareça e visualize os resultados ou retorne à exibição de modelos .
5. Estratégias de normalização e análise de dados - normalização pós-ensaio e ensaios a jusante (etapas opcionais)
- Normalização de dados
- Para normalizar os dados esferoides, consulte a série de protocolos pertinentes às estratégias de normalização de dados para calcular o tamanho e o volume e quantificar o DSDNA em ensaios esferoides. Estes foram incluídos como arquivos suplementares; ver arquivo suplementar 1 e arquivo suplementar 2.
- Análise de dados
- Para exportar dados para um dos geradores de análise automatizados, siga os comandos de exportação de dados no controlador WAVE e selecione o gerador de exportação que corresponde ao tipo de ensaio. Alternativamente, exporte o arquivo de dados e carregue-o para análises do Seahorse.
NOTA: A desvantagem dos geradores de relatórios e da análise de Seahorse é que a análise de dados está limitada à forma como o ensaio XF é projetado e não permite que médias sejam tomadas em todos os ciclos de medição. A exportação manual de conjuntos de dados do software de instrumento permite a preferência do usuário a esse respeito. Dado que a estratégia de injeção para avaliar a respiração mitocondrial de esferoides 3D provavelmente difere do de um teste típico de 'MitoStress', uma série de modelos de planilha foram desenvolvidos para ajudar a analisar esses conjuntos de dados, específicos para culturas de células 3D e serão fornecidos mediante solicitação. Esses arquivos de modelo de dados fornecerão dados sobre os principais parâmetros respiratórios mitocondriais detalhados e explicados na Figura 2. - Para analisar os dados, exporte os dados como um relatório de planilha do software do controlador WAVE e use um modelo de planilha independente para análise.
- Para exportar dados para um dos geradores de análise automatizados, siga os comandos de exportação de dados no controlador WAVE e selecione o gerador de exportação que corresponde ao tipo de ensaio. Alternativamente, exporte o arquivo de dados e carregue-o para análises do Seahorse.
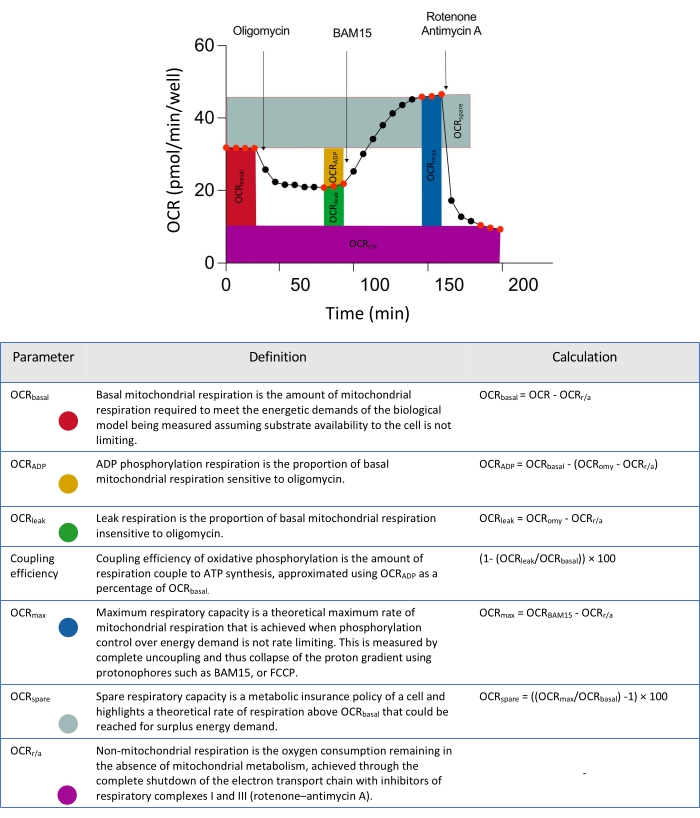
Figura 2: Descritores esquemáticos para parâmetros derivados de análises de dados de fluxo extracelular. Abreviação: OCR = taxa de consumo de oxigênio. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para obter spheróides bem formados e compactos, cada linha celular foi otimizada individualmente para a densidade de semeadura e duração do cultivo (Figura 3). As linhas celulares A549, HepG2/C3A e SK-OV-3 inicialmente formaram agregados soltos que não progrediram para esferoides redondos com perímetros claramente definidos até depois de 7 dias na cultura. Por outro lado, as células MCF-7 podem formar esferoides dentro de 3 dias. Houve uma clara correlação entre a densidade inicial ...
Access restricted. Please log in or start a trial to view this content.
Discussão
Principais descobertas e saídas
Este artigo fornece um protocolo detalhado para sondar o metabolismo de energia mitocondrial de esferoides 3D únicos usando uma série de linhas celulares derivadas do câncer com o XFe96 XF Analyzer. Um método é desenvolvido e descrito para o rápido cultivo de Esferoides celulares A549, HepG2/C3A, MCF7 e SK-OV-3 usando tecnologias repelentes celulares para agregação forçada. Este protocolo aborda muitas considerações sobre a sondagem do metabolismo esferoide ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse para declarar.
Agradecimentos
N.J.C foi apoiado por um Prêmio BBSRC MIBTP CASE com a Sygnature Discovery Ltd (BB/M01116X/1, 1940003)
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
Referências
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
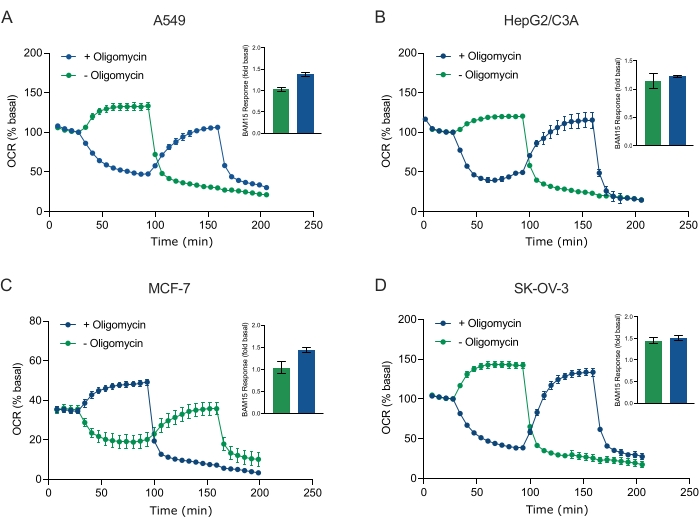
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
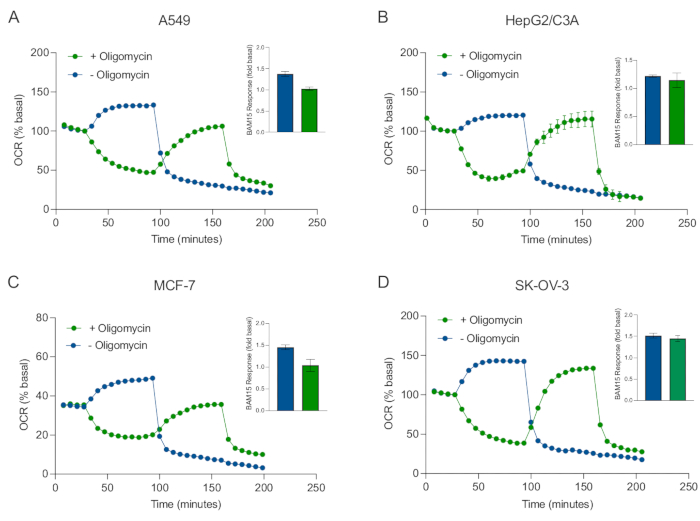
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados