Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Hücre Dışı Akı Analizi Kullanarak Tek 3D Mikrodoku Sferoidlerinin Mitokondriyal Enerji Metabolizmasını Keşfetmek
Bu Makalede
Erratum Notice
Özet
Bu protokoller, kullanıcıların Denizatı hücre dışı akı analizini kullanarak 3D kanser hücre hattı kaynaklı sferoidlerde mitokondriyal enerji metabolizmasını araştırmalarına yardımcı olacaktır.
Özet
Sferoidler olarak adlandırılan üç boyutlu (3D) hücresel agregalar, son yıllarda in vitro hücre kültürünün ön saflarında yer almaktadır. Hücreleri iki boyutlu, tek hücreli tek katmanlı (2D kültür) olarak kültürlemenin aksine, küresel hücre kültürü, hücre dışı matris proteinlerinin ekspresyonu, hücre sinyalizasyonu, gen ekspresyonu, protein üretimi, farklılaşma ve proliferasyon dahil olmak üzere in vivo olarak var olan fizyolojik hücresel mimariyi ve özellikleri teşvik eder, düzenler ve destekler. 3D kültürün önemi, onkoloji, diyabet, kök hücre biyolojisi ve doku mühendisliği dahil olmak üzere birçok araştırma alanında kabul edilmiştir. Son on yılda, sferoidler üretmek ve metabolik işlevlerini ve kaderlerini değerlendirmek için geliştirilmiş yöntemler geliştirilmiştir.
Hücre dışı akı (XF) analizörleri, bir XF24 adacık yakalama plakası veya bir XFe96 küresel mikroplaka kullanarak sferoidler gibi 3D mikro dokularda mitokondriyal fonksiyonu keşfetmek için kullanılmıştır. Bununla birlikte, farklı protokoller ve XF teknolojisini kullanarak sferoidlerde mitokondriyal enerji metabolizmasının araştırılmasının optimizasyonu ayrıntılı olarak tanımlanmamıştır. Bu yazıda, XFe96 XF analizörü ile küresel mikroplakalar kullanılarak tek bir 3D sferoidde mitokondriyal enerji metabolizmasının araştırılması için ayrıntılı protokoller sunulmaktadır. Farklı kanser hücre hatları kullanarak, XF teknolojisinin sadece farklı boyutlarda değil, aynı zamanda farklı hacimlerde, hücre sayılarında, DNA içeriğinde ve tipinde 3D sferoidlerde hücresel solunum arasında ayrım yapabildiği gösterilmiştir.
Oligomisin, BAM15, rotenon ve antimisin A'nın optimal mitokondriyal efektör bileşik konsantrasyonları, 3D sferoidlerde mitokondriyal enerji metabolizmasının spesifik parametrelerini araştırmak için kullanılır. Bu yazıda ayrıca sferoidlerden elde edilen verileri normalleştirme yöntemleri tartışılmakta ve XF teknolojisini kullanarak küresel metabolizmayı keşfederken göz önünde bulundurulması gereken birçok husus ele alınmaktadır. Bu protokol, gelişmiş in vitro sferoid modellerde araştırma yapılmasına yardımcı olacaktır.
Giriş
Biyolojik araştırmalarda in vitro modellerdeki ilerlemeler son 20 yılda hızla ilerlemiştir. Bu tür modeller artık çip üzerinde organ modalitelerini, organoidleri ve 3D mikrodoku sferoidlerini içeriyor ve bunların hepsi in vitro ve in vivo çalışmalar arasındaki çeviriyi geliştirmek için ortak bir odak noktası haline geldi. Gelişmiş in vitro modellerin, özellikle de sferoidlerin kullanımı, doku mühendisliği, kök hücre araştırması, kanser ve hastalık biyolojisi 1,2,3,4,5,6,7 ve genetik toksikoloji 8,9,10, nanomalzemeler toksikolojisi 11 dahil olmak üzere güvenlik testleri dahil olmak üzere çeşitli araştırma alanlarını kapsamaktadır. 12,13,14 ve ilaç güvenliği ve etkinliği testi8,15,16,17,18,19.
Normal hücre morfolojisi biyolojik fenotip ve aktivite için kritik öneme sahiptir. Hücreleri 3D mikrodoku sferoidlerine kültürlemek, hücrelerin in vivo olarak gözlemlenene daha çok benzeyen ancak klasik tek katmanlı hücre kültürü teknikleriyle yakalanması zor olan bir morfoloji, fenotipik fonksiyon ve mimari benimsemelerini sağlar. Hem in vivo hem de in vitro , hücresel fonksiyon, hücresel iletişim ve programlama ile sınırlı olmayan hücresel mikro çevreden doğrudan etkilenir (örneğin, hücre-hücre birleşme oluşumları, hücre nişleri oluşturma fırsatları); yakın çevrelerde hormonlara ve büyüme faktörlerine hücre maruziyeti (örneğin, enflamatuar yanıtın bir parçası olarak hücresel sitokin maruziyeti); fiziksel ve kimyasal matrislerin bileşimi (örneğin, hücrelerin sert doku kültürü plastiğinde mi yoksa elastik bir doku ortamında mı yetiştirildiği); ve en önemlisi, hücresel metabolizmanın beslenme ve oksijene erişimin yanı sıra laktik asit gibi metabolik atık ürünlerin işlenmesinden nasıl etkilendiği.
Metabolik akı analizi, tanımlanmış in vitro sistemlerdeki hücresel metabolizmayı incelemenin güçlü bir yoludur. Spesifik olarak, XF teknolojisi, bozulmamış hücrelerin ve dokuların hücresel biyoenerjetik özelliklerindeki canlı, gerçek zamanlı değişikliklerin analizine izin verir. Birçok hücre içi metabolik olayın saniyeler ila dakikalar arasında meydana geldiği göz önüne alındığında, gerçek zamanlı fonksiyonel yaklaşımlar, bozulmamış hücrelerde ve dokularda in vitro hücresel metabolik akıdaki gerçek zamanlı değişiklikleri anlamak için çok önemlidir.
Bu yazıda kanserden türetilmiş hücre hatları A549 (akciğer adenokarsinomu), HepG2/C3A (hepatosellüler karsinom), MCF-7 (meme adenokarsinomu) ve SK-OV-3 (over adenokarsinomu) hücre hatlarının in vitro 3D sferoid modeller kullanılarak zorla agregasyon yaklaşımları kullanılarak geliştirilmesi için protokoller sunulmaktadır (Şekil 1). Ayrıca, (i) Agilent XFe96 XF analizörü kullanılarak tek 3D sferoidlerin mitokondriyal enerji metabolizmasının nasıl araştırılacağını ayrıntılı olarak açıklar, (ii) tek 3D sferoidleri kullanarak XF analizlerini optimize etmenin yollarını vurgular ve (iii) bu yaklaşımı kullanarak 3D sferoid metabolizmasını araştırmanın önemli hususlarını ve sınırlamalarını tartışır. En önemlisi, bu makalede, hücresel sferoidlerde oksidatif fosforilasyonu ve dolayısıyla mitokondriyal fonksiyonu belirlemek için oksijen tüketim hızının (OCR) hesaplanmasına izin veren veri kümelerinin nasıl toplandığı açıklanmaktadır. Bu protokol için analiz edilmemiş olsa da, hücre dışı asitleşme hızı (ECAR), XF deneylerinde OCR verileriyle birlikte ölçülen başka bir parametredir. Bununla birlikte, ECAR genellikle XF veri kümelerinden kötü veya yanlış yorumlanır. Teknoloji üreticisinin temel yaklaşımlarını izleyerek ECAR'ı hesaplamanın sınırlamaları hakkında bir yorum sunuyoruz.
Access restricted. Please log in or start a trial to view this content.
Protokol
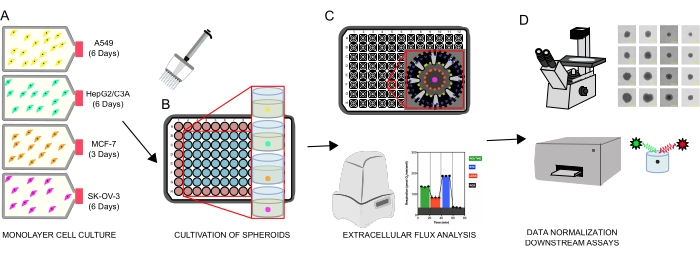
Şekil 1: Hücresel sferoidlerin oluşturulması, hücre dışı akı analizi ve aşağı akış tahlilleri için grafiksel iş akışı. Dört kanser hücresi hattı, doku kültürü şişelerinden ayrılan tek katmanlı (A) olarak seçici olarak kültürlendi ve sferoidler (B) oluşturmak için ultra düşük bağlanmalı 96 kuyucuklu mikroplakalara tohumlandı. A549 akciğer karsinomu, HepG2 / C3A karaciğer karsinomu, SK-OV-3 yumurtalık adenokarsinomu ve MCF-7 meme karsinomu hücreleri 1 × 10 3-8 × 103 hücre / kuyuda tohumlandı ve tek sferoidler oluşturmak ve sürekli gözlem ve planimetrik ölçümlerle sferoid tohumlama yoğunluğunu ve yetiştirme süresini optimize etmek için 7 güne kadar büyütüldü. Oluştuktan sonra, tek sferoidler serumsuz bir XF ortamına yıkandı ve poli-D-lizin (C) ile önceden kaplanmış sferoid tahlil mikroplakalarına dikkatlice tohumlandı. Sferoidler, XFe96 analizörü kullanılarak hücre dışı akı analizine tabi tutuldu: (1) bazal mitokondriyal solunum yanıtı için optimal küresel boyut; (2) mitokondriyal solunum inhibitörlerinin optimize edilmiş titrasyonları; (3) mikroplaka kuyuları içinde küresel yerleşimin optimizasyonu. (D) XF sonrası analizler, faz kontrast mikroskobu ve sferoid DNA nicelleştirmesi, veri normalizasyonu ve diğer aşağı akış in vitro tahlilleri için kullanılmıştır. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
1. Kanser hücre hatlarının 3D in vitro sferoidler olarak yetiştirilmesi
| Hücre hattı | Tarif | Kültür ortamı | Kaynak |
| A549 · | Akciğer karsinomu hücre hattı | RPMI 1640 Devir sayısı | Avrupa Doğrulanmış Hücre Kültürleri Koleksiyonu (ECACC) |
| Sodyum piruvat (1 mM) | |||
| Penisilin- Streptomisin - (100 U/mL – 100 mg/mL) | |||
| % 10 (v/v) FBS | |||
| HepG2/C3A | Hepatik karsinom hücre hattı, ana HepG2 hücre hattının klonal bir türevi | cesaret | Amerikan Doku Kültürü Koleksiyonu (ATCC) |
| Penisilin- Streptomisin - (100 U/mL – 100 mg/mL) | |||
| % 10 (v/v) FBS | |||
| MCF7 Serisi | Meme adenokarsinomu hücre hattı | RPMI 1640 Devir sayısı | Avrupa Doğrulanmış Hücre Kültürleri Koleksiyonu (ECACC) |
| Sodyum piruvat (1 mM) | |||
| Penisilin- Streptomisin - (100 U/mL – 100 mg/mL) | |||
| % 10 (v/v) FBS | |||
| SK-OV-3 | Yumurtalık adenokarsinomu hücre hattı | RPMI 1640 Devir sayısı | Avrupa Doğrulanmış Hücre Kültürleri Koleksiyonu (ECACC) |
| Sodyum piruvat (1 mM) | |||
| Penisilin- Streptomisin - (100 U/mL – 100 mg/mL) | |||
| % 10 (v/v) FBS | |||
| Parça | RPMI tahlil ortamı (50 mL son hacim) | ||
| Baz Orta | Çevik Denizatı XF RPMI, pH 7,4 | ||
| Glikoz (1 M steril stok) | 11 mM (0,55 mL stok çözümü) | ||
| L-glutamin (200 mM steril stok) | 2 mM (0,5 mL stok çözeltisi) | ||
| Sodyum piruvat (100 mM steril stok) | 1 mM (0,5 mL stok çözeltisi) |
Tablo 1: Kanser hücre hattı ortamı ve XF ortam bileşimleri.
- Standart aseptik doku kültürü tekniğini kullanarak tüm hücre hatlarını kültürleyin ve uygun bir tahlil kiti kullanarak mikoplazma içermediklerini onaylayın.
- Önerilen ortamı kullanarak T75 doku kültürü şişelerinde veya eşdeğerindeki hücre hatlarını kültürleyin (Tablo 1). Hücreyi% 65-80 oranında akıcılığa kadar kültürleyin ve bunları düzenli olarak en fazla 25 pasaja kadar geçirin.
- Hücre kültürü şişelerini Dulbecco'nun modifiye fosfat tamponlu salininde (DBPS) iki kez durulayın.
- Hücreleri, 37 ° C'de 5 dakika boyunca 3 mL hücre ayrışma reaktifi ile şişelerden ayırın ( Malzeme Tablosuna bakınız) ve mikroskopi ile ayrılmayı onaylayın.
- Tek hücreli bir süspansiyon sağlamak için ayrılmış hücre süspansiyonunu nazikçe aspire edin ve hücre ayrışma reaktifini 7 mL tam doku kültürü ortamı ile devre dışı bırakın.
- Hücreleri 5 dakika boyunca 300 × g'da santrifüjleme yoluyla toplayın, süpernatanı atın ve hücreleri tam ortamda yeniden askıya alın.
- Bir hemositometre veya otomatik bir hücre sayacı kullanarak hücreleri sayın ve tohumlama için gereken istenen hücre yoğunluğuna titre edin.
NOT: 96 delikli bir plakanın tamamını 4 × 103 hücre/kuyuda 100 μL/kuyucukta tohumlamak için, hücreler önerilen 12 mL'lik bir hacimde 4 × 104 hücre/mL'ye titre edilmelidir. - Hücre süspansiyonunu steril bir rezervuara boşaltın ve çok kanallı bir pipetör kullanarak hücre süspansiyonunun 100 μL'sini hücre itici bir mikro plakanın her bir kuyucuğuna dağıtın.
NOT: Bir mikroplakanın sadece iç 60 kuyucuğu tohumlanmalı ve geri kalanı DPBS ile doldurulmalıdır. Bu bir buharlaşma bariyeri oluşturacak, plaka boyunca küresel homojenliği sağlayacak ve plaka kenar etkilerini en aza indirecektir. - Hücreleri gevşek agregalara zorlamak için 300 × g'da küresel mikro plakaları 15 dakika boyunca santrifüj yapın.
- Küresel oluşumu sağlamak için plakaları en az 3 gün boyunca 37 °C'de,% 5 CO2'de inkübe edin.
- Sferoidlerin büyümesini izlemek için standartlaştırılmış laboratuvar uygulamalarını kullanarak faz kontrastlı mikroskopi gerçekleştirin. Hücre kültürü ortamını her 3 günde bir veya haftada iki kez, yarım hacimli bir ortam değişimi gerçekleştirerek doldurun.
2. Hücre Dışı Akı (XF) Teknolojisi kullanarak tek sferoidlerin mitokondriyal enerji metabolizmasının araştırılması
- Tahlil hazırlığı (bir gün önce)
- Bozulmamış küresel yapı, morfoloji ve numuneler arasında genel homojenlik sağlamak için 4x büyütmede faz kontrastına sahip ters çevrilmiş bir ışık mikroskobu kullanarak küresel canlılığı kontrol edin.
- Sensör kartuşunu nemlendirin.
- Kalibrantın ~ 20 mL'sini konik bir tüp haline getirir.
- Kalibranı içeren konik tüpü gece boyunca CO2 37 °C olmayan bir inkübatöre yerleştirin.
- İçeriği tahlil kitinden çıkarın.
- Sensör kartuşunu yardımcı plakadan çıkarın ve yardımcı plakanın yanındaki tezgahın üzerine baş aşağı yerleştirin.
- Çok kanallı P300 pipet kullanarak sensör kartuşu yardımcı plakasının her bir kuyucuğuna200μL steril ddH 2 O pipetin.
- Sensör kartuşunu yardımcı plakanın üzerine yerleştirin.
- Her bir kuyucuktaki su seviyesinin, sensör problarını suya batırmak için yeterince yüksek olup olmadığını kontrol edin.
- Takılan sensör kartuşunu CO2 olmayan 37 °C inkübatöre aktarın ve gece boyunca bırakın.
NOT: Bu adım, tahlil başlamadan 12-72 saat önce gerçekleştirilebilir.
- Ceket sferoid tahlil mikroplakası
- Aseptik teknikler kullanarak, sferoid mikroplakaya 30 μL / kuyucuk steril Poli-D-Lizin (0.1 mg / mL) çözeltisi ekleyin ve oda sıcaklığında 30 dakika boyunca inkübe edin.
- Çözeltiyi küresel mikroplakanın her bir oluğundan aspire edin, plakayı ters çevirin ve herhangi bir artık çözeltiyi çıkarmak için doku kağıdına sıkıca dokunun.
- Plakayı 200 μL/kuyucuk steril ddH2O ile iki kez yıkayın.
- Son yıkamadan sonra, mikro plakayı ters çevirin ve kalan suyu çıkarmak için kağıt mendile sıkıca dokunun.
- Kullanmadan veya ileride kullanmak üzere 4 °C'de saklamadan önce plakanın 30 dakika boyunca hava ile kurumasını bekleyin.
NOT: Sferoid tahlil mikroplakası, sferoidlerin mikroplakanın dibine sabitlenmesini sağlamak için moleküler bir yapıştırıcı ile kaplanmalıdır. Moleküler bir yapıştırıcı olmadan, sferoidler yerinden çıkabilir ve tahlil sonuçlarına müdahale edebilir. Diğer moleküler yapıştırıcılar, plakaları ön kaplamak için Poli-D-Lizin'e alternatif olarak da kullanılabilir. Önceden kaplanmış plakalar 4 ° C'de saklanabilir, ancak tahlil başlamadan önce oda sıcaklığına dengelenmelidir.
- XF Testi ortamını hazırlama
- Tablo 1'de ayrıntılı olarak açıklandığı gibi XF RPMI ortamını hazırlayın ve 0,22 μm şırınga filtreli steril filtre uygulayın
- Tahlil hazırlığı (tahlilden 1 saat önce)
- Takviyeli XF RPMI tahlil ortamını 37 ° C'ye kadar önceden ısıtın.
- Kaplanmış sferoid tahlil mikroplakasını CO2 37 °C olmayan bir inkübatörde veya kuru banyoda önceden ısıtın.
- Sensör kartuşunu hazırlayın.
- Kalibrant ve sensör kartuşunu içeren konik tüpü hava inkübatöründen çıkarın.
- Sensör kartuşunu yardımcı plakadan çıkarın ve çalışma yüzeyine baş aşağı yerleştirin.
- P300 çok kanallı pipet kullanarak, suyu kullanım plakasından aspire edin ve atın.
- Kalibrant çözeltisini steril bir reaktif haznesine dökün ve P300 çok kanallı pipet kullanarak ön ısıtılmış kalibrantın 200 μL/kuyucuğuna yardımcı plakaya ekleyin.
- Sensör kartuşunu alın ve tekrar yardımcı plakanın üzerine yerleştirin, böylece sensörlerin kalipranta iyice batırıldığından emin olun.
- Montajlı sensör kartuşunu, port enjeksiyon çözeltilerini yüklemeye hazır olana kadar CO2 37 °C olmayan inkübatöre geri aktarın.
- Sferoidleri tahlil ortamı ile yıkayın.
- Küresel kültür plakasını 37 °C,% 5 CO2 inkübatöründen çıkarın ve sferoid transfer adımlarından önce bütünlüklerini sağlamak için sferoidleri mikroskop altında gözlemleyin.
- Küresel plakanın tüm kuyularını, herhangi bir arka plan düzeltme kuyusu da dahil olmak üzere 180 μL / önceden ısıtılmış tahlil ortamı kuyusu ile yükleyin.
- 7 cm'lik bir Petri kabını 3 mL tahlil ortamı ile kısmen doldurun.
- Geniş delikli pipet uçlarıyla yüklü çok kanallı bir pipet kullanarak, pipetleyiciyi 10-50 μL'lik bir aspirasyon hacmine ayarlayarak sferoidleri 96 delikli kültür plakasından 7 cm Petri kaplarına aktarın.
- Önceden kaplanmış sferoid tahlil mikroplakasına tohum sferoidleri.
- Bir diseksiyon mikroskobu ve bir ışık kutusu aparatı kullanarak, sferoidleri Petri kabından aşağıda ayrıntılı olarak açıklandığı gibi sferoid tahlil mikroplakasına aktarın.
- Geniş delikli pipet ucuyla donatılmış tek kanallı pipetörün ses seviyesini 20 μL'ye ayarlayın ve tek bir sferoidi dikkatlice aspire edin. Ucu doğrudan sferoid tahlil mikroplakasının her bir kuyucuğunun ortasına yerleştirin ve yerçekiminin her bir kuyucuğun merkezine tek bir sferoid salınmasına izin verin, yani pipet ucundan herhangi bir ortamı dışarı atmayın ve kılcal hareketin sferoidi pipet ucundan çekmesine izin verin. Elüsyonu doğrulamak için, pipetleyicinin içeriği mikroskop altında 7 cm'lik Petri kabına geri pipetlenebilir.
NOT: Tek bir sferoidin yerçekimi elüsyonu, küresel boyuta / yoğunluğa bağlı olarak tipik olarak 15-30 s sürer. Bu süre zarfında pipetleyici çıkarılmamalıdır. Herhangi bir arka plan düzeltme kuyusu sferoidlerden arındırılmış olmalı ve sadece tahlil ortamı içermelidir. Mikroskop altında, her sferoidin konumunu onaylayın. Her küre ideal olarak her bir kuyucuğun ortasına yerleştirilmelidir. - Tüm sferoidler sferoid tahlil mikroplakasına aktarıldıktan sonra, plakayı tahlilden en az 1 saat önce 37 ° C'de CO2 olmayan bir inkübatöre aktarın.
- Geniş delikli pipet ucuyla donatılmış tek kanallı pipetörün ses seviyesini 20 μL'ye ayarlayın ve tek bir sferoidi dikkatlice aspire edin. Ucu doğrudan sferoid tahlil mikroplakasının her bir kuyucuğunun ortasına yerleştirin ve yerçekiminin her bir kuyucuğun merkezine tek bir sferoid salınmasına izin verin, yani pipet ucundan herhangi bir ortamı dışarı atmayın ve kılcal hareketin sferoidi pipet ucundan çekmesine izin verin. Elüsyonu doğrulamak için, pipetleyicinin içeriği mikroskop altında 7 cm'lik Petri kabına geri pipetlenebilir.
- Bir diseksiyon mikroskobu ve bir ışık kutusu aparatı kullanarak, sferoidleri Petri kabından aşağıda ayrıntılı olarak açıklandığı gibi sferoid tahlil mikroplakasına aktarın.
3. XF testleri için bileşiklerin hazırlanması ve sensör kartuşuna yüklenmesi
| Enjeksiyon Stratejisi | Bileşik (Bağlantı Noktası) | XFe96 mikro kuyucuk başlangıç hacmi (μL) | İstenilen nihai kuyu konsantrasyonu | Bağlantı Noktası Hacmi (μL) | Son XFe96 mikrowell hacimli enjeksiyon sonrası (μL) | Çalışma stoğu konsantrasyonu |
| 1 | Oligomisin (A) | 180 | 3 ug/mL | 20 | 200 | 30 μg/mL |
| Rotenon (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimisin A (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| Rotenon (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimisin A (B) | 200 | 2 μM | 20 | 220 | 22 μM |
Tablo 2: XFe96 Analizörü kullanılarak tek 3D sferoidlerin mitokondriyal enerji metabolizmasını araştırmak için mitokondriyal bileşik konsantrasyonları.
- Tamamen takviyeli, önceden ısıtılmış XF RPMI tahlil ortamı kullanarak Tablo 2'de belirtildiği gibi her bileşiğin çalışma stok konsantrasyonlarını hazırlayın.
- Kartuş plakasını (yardımcı plakaya bağlanmış) sütun yönünde, soldan sağa 1-12 olacak şekilde yönlendirin.
- Yükleme kılavuzu kullanıyorsanız, iyi yükleme prosedürüne göre kartuş plakasının üzerine yerleştirin, örneğin önce A bağlantı noktası yüklenecekse, kılavuzun sol üst köşesinde A'nın görünür olduğundan emin olun.
- Her bileşiğin çalışma çözeltisini uygun bir hazneye aktarın ve kalibre edilmiş bir P100 çok kanallı pipet kullanarak 20 μL'yi ilgili tüm portlara dağıtın. Her bileşik için kalan bağlantı noktalarına tekrarlayın.
NOT: Sensör kartuşu plakasında herhangi bir bağlantı noktası kullanılmazsa, bunlar boş bırakılabilir veya tahlil ortamıyla doldurulabilir. Yalnızca belirli bir port harfinin seçimi kullanılıyorsa, bu mektuba karşılık gelen diğer portların tahlil ortamı ile yüklendiğinden emin olun; aksi takdirde, kuyuya hava enjekte edilecek ve bu kuyulardaki sonuçlardan ödün verilecektir. - Bağlantı noktası yüklemesinden sonra, plaka yükleme kılavuzlarını (kullanılıyorsa) çıkarın ve analizörü sensör kartuşunu yüklemek üzere hazırlayın.
NOT: Bağlantı noktaları yüklendikten hemen sonra tahlil yapılmıyorsa, kapağı tekrar sensör kartuşuna yerleştirin ve makineye yüklenmeye hazır olana kadar plakayı 37 °C hava inkübatörüne geri koyun.
4. Tahlil tasarımı, enjeksiyon stratejileri ve veri toplama
- Tahlilin yürütülmesi
- Analizörü açın ve denetleyiciye (bilgisayar) bağlayın.
NOT: Bu, Wave Controller yazılımının widget panelindeki cihaz bağlantı durumu ile doğrulanabilir. - WAVE yazılımında şablonlar sayfasına gidin, denemenin tahlil şablonu dosyasını bulun ve çift tıklayarak açın.
NOT: Tahlil şablonu Şablonlar görünümünde görünmüyorsa, şablon dosyasını paylaşılan ağ sürücüsünden veya USB flash sürücüden şablon klasörüne alın. - Tahlili başlatmak için, Tahlil Çalıştır sekmesini tıklatın.
NOT: Grup tanımları plaka haritası içinde doğru şekilde ayrılmışsa, tahlil sayfanın sağ tarafındaki yeşil onay işaretiyle belirtildiği gibi çalışmaya hazır olacaktır. Bu aşamada, herhangi bir ek bilgi tahlil özeti sayfasına veya boş bırakılan sayfaya girilebilir; bir sonraki adıma geçin. 3D mikrodoku sferoidlerinde mitokondriyal modülatörlerin gecikmiş penetrasyonu nedeniyle (Şekil 2), Tablo 3'te açıklanan ölçüm protokolü bilgilerini kullanın.
- Analizörü açın ve denetleyiciye (bilgisayar) bağlayın.
| Ölçüm Süresi | Enjeksiyon Numarası ve Portu | Ölçüm Detayları | Süre (h:min:s) |
| Kalibrasyon | Uygulanamaz | XF analizörleri, ölçümlerin doğru olduğundan emin olmak için her zaman bu kalibrasyonu gerçekleştirir | 00:20:00 (bu ortalamadır ve makineler arasında değişebilir) |
| Equillibration | Uygulanamaz | Dengeleme Kalibrasyondan sonra gerçekleşir ve tavsiye edilir. | 00:10:00 |
| Bazal | Uygulanamaz | Döngüler = 5 | 00:30:00 |
| Karışım = 3:00 | |||
| Bekle = 0:00 | |||
| Ölçü = 3:00 | |||
| Oligomisin / BAM15 | Enjeksiyon 1 (Port A) | Döngüler = 10 | 01:00:00 |
| Karışım = 3:00 | |||
| Bekle = 0:00 | |||
| Ölçü = 3:00 | |||
| Rotenon + antimisin A | Enjeksiyon 2 (Port B) | Döngüler = 10 | 01:00:00 |
| Karışım = 3:00 | |||
| Bekle = 0:00 | |||
| Ölçü = 3:00 | |||
| Toplam Süre: | 03:00:00 | ||
Tablo 3: XFe96 Analizörü kullanılarak tek 3D sferoidlerin mitokondriyal enerji metabolizmasını araştırmak için protokol kurulumu.
- Konumu kaydet iletişim kutusunu açmak için çalıştırmayı başlat'ı tıklatın.
- Sonuç dosyasının kaydetme konumunu girin ve monte edilmiş sensör kartuşunu, analizörün yan tarafındaki kapıdan görünen termal tepsiye yerleştirin. Termal tepsinin otomatik olarak açılmasını ve ekranın Kalibrant Yükle Yardımcı Plakası mesajını görüntülemesini bekleyin. Ekrandaki talimatları izlemeden önce, i) sensör kartuşunun Yardımcı plakaya düzgün oturduğundan, ii) kapağın sensör kartuşundan çıkarıldığından ve iii) yardımcı plakadaki sensör kartuşunun doğru yönlendirildiğinden emin olun.
- Sensör kartuşu kalibrasyonunu başlatmak için ekrandaki komutları izleyin.
NOT: Kalibrasyonu tamamlamak için geçen süre yaklaşık 10-20 dakikadır (37 °C'de tahliller için). - Sensör kartuşu kalibrasyonundan sonra, 12 dakikalık dengeleme adımını başlatmak için Dalga Kontrol Cihazındaki ekrandaki talimatları izleyerek küresel mikro plakayı analizöre yükleyin.
NOT: Beyaz onay işaretli yeşil kutular, bu kuyu için 'iyi' bir kalibrasyonu gösterir. Herhangi bir kuyucuk 'iyi' bir kalibrasyon sağlayamazsa, kırmızı bir kutu ve beyaz haç ile belirtilecektir. Bu tür kuyular, değişiklik testi sekmesi kullanılarak tahlil tamamlandıktan sonra not edilmeli ve herhangi bir analizden çıkarılmalıdır. - Makine dengeleme adımını tamamladıktan sonra analizörün otomatik olarak taban çizgisi ölçümlerini almaya başlamasını bekleyin (cihaz protokolünde belirtildiği gibi).
- Denemeyi tamamlamak için WAVE denetleyicisindeki ekrandaki komutları izleyin.
NOT: Küresel mikro plaka analizörden çıkarıldıktan sonra, sensör kartuşunu atın ve gerekirse daha fazla analiz için küresel plakayı bir kenara koyun (örneğin, çift sarmallı (ds) DNA nicelemesi). Mikro plaka daha fazla analiz için gerekli değilse, sensör kartuşu ile birlikte atılabilir. - Tahlil iletişim kutusunun görünmesini bekleyin ve sonuçları görüntüleyin veya şablonlar görünümüne dönün.
5. Veri normalleştirme ve analiz stratejileri - tahlil sonrası normalizasyon ve aşağı akış tahlilleri (isteğe bağlı adımlar)
- Veri normalleştirme
- Küresel verileri normalleştirmek için, küresel boyut ve hacmi hesaplamak ve sferoid tahlillerde dsDNA'yı ölçmek için veri normalleştirme stratejileriyle ilgili protokoller dizisine bakın. Bunlar ek dosyalar olarak eklenmiştir; Ek dosya 1 ve Ek dosya 2'ye bakın.
- Veri analizi
- Verileri otomatik analiz üreteçlerinden birine aktarmak için, WAVE denetleyicisindeki veri dışa aktarma komutlarını izleyin ve tahlil türüyle eşleşen dışa aktarma oluşturucuyu seçin. Alternatif olarak, veri dosyasını dışa aktarın ve Seahorse analitiğine yükleyin.
NOT: Rapor üreteçlerinin ve Seahorse analitiğinin dezavantajı, veri analizinin XF testinin nasıl tasarlandığıyla sınırlı olması ve ölçüm döngüleri boyunca ortalamaların alınmasına izin vermemesidir. Veri kümelerinin cihaz yazılımından manuel olarak dışa aktarılması, bu konuda kullanıcı tercihine izin verir. 3D sferoidlerin mitokondriyal solunumunu değerlendirmek için enjeksiyon stratejisinin tipik bir 'MitoStress' testinden farklı olacağı göz önüne alındığında, 3D hücre kültürlerine özgü bu veri kümelerini analiz etmeye yardımcı olmak için bir dizi elektronik tablo şablonu geliştirilmiştir ve talep üzerine sağlanacaktır. Bu veri şablonu dosyaları, Şekil 2'de ayrıntılı ve açıklanan temel mitokondriyal solunum parametreleri hakkında veri sağlayacaktır. - Verileri analiz etmek için, verileri WAVE denetleyici yazılımından elektronik tablo raporu olarak dışa aktarın ve analiz için bağımsız bir elektronik tablo şablonu kullanın.
- Verileri otomatik analiz üreteçlerinden birine aktarmak için, WAVE denetleyicisindeki veri dışa aktarma komutlarını izleyin ve tahlil türüyle eşleşen dışa aktarma oluşturucuyu seçin. Alternatif olarak, veri dosyasını dışa aktarın ve Seahorse analitiğine yükleyin.
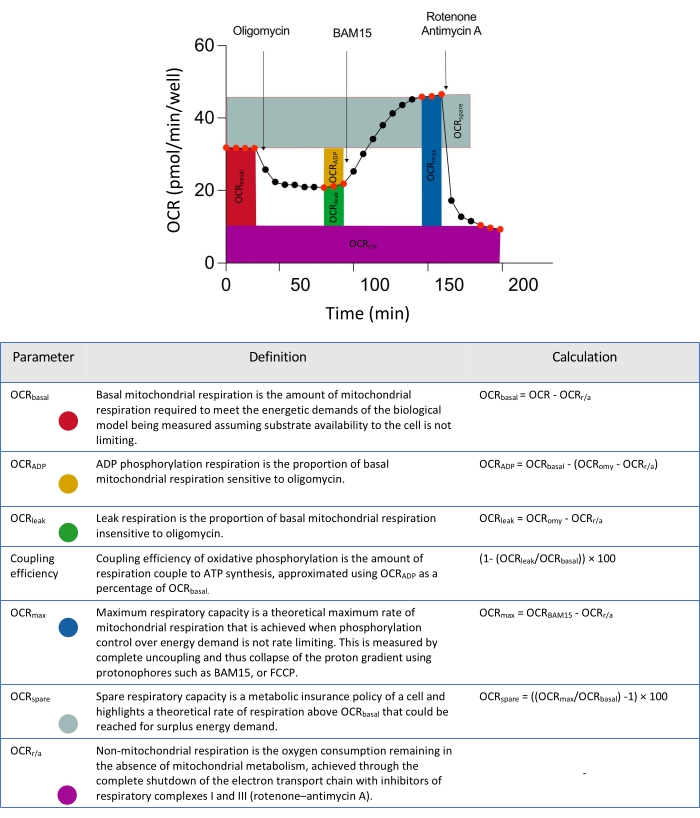
Şekil 2: Hücre dışı akı veri analizlerinden türetilen parametreler için şematik tanımlayıcılar. Kısaltma: OCR = oksijen tüketim oranı. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
İyi biçimlendirilmiş, kompakt sferoidler elde etmek için, her hücre hattı tohumlama yoğunluğu ve ekim süresi için ayrı ayrı optimize edilmiştir (Şekil 3). A549, HepG2 / C3A ve SK-OV-3 hücre hatları başlangıçta, kültürde 7 gün sonrasına kadar açıkça tanımlanmış çevrelere sahip yuvarlak sferoidlere ilerlemeyen gevşek agregalar oluşturdu. Tersine, MCF-7 hücreleri 3 gün içinde sferoidler oluşturabilir. Tüm sferoid modeller için kültür periyodundan sonra il...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Ana bulgular ve çıktılar
Bu makale, XFe96 XF Analyzer ile bir dizi kanser kaynaklı hücre hattı kullanarak tek 3D sferoidlerin mitokondriyal enerji metabolizmasını araştırmak için ayrıntılı bir protokol sunmaktadır. Zorla toplama için hücre kovucu teknolojiler kullanılarak A549, HepG2 / C3A, MCF7 ve SK-OV-3 hücresel sferoidlerin hızlı bir şekilde yetiştirilmesi için bir yöntem geliştirilmiş ve tanımlanmıştır. Bu protokol, (1) küresel kültür protokollerinin optimizasyon...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların beyan edecekleri herhangi bir çıkar çatışması yoktur.
Teşekkürler
N.J.C, Sygnature Discovery Ltd ile BBSRC MIBTP CASE Award tarafından desteklenmiştir (BB/M01116X/1, 1940003)
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
Referanslar
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
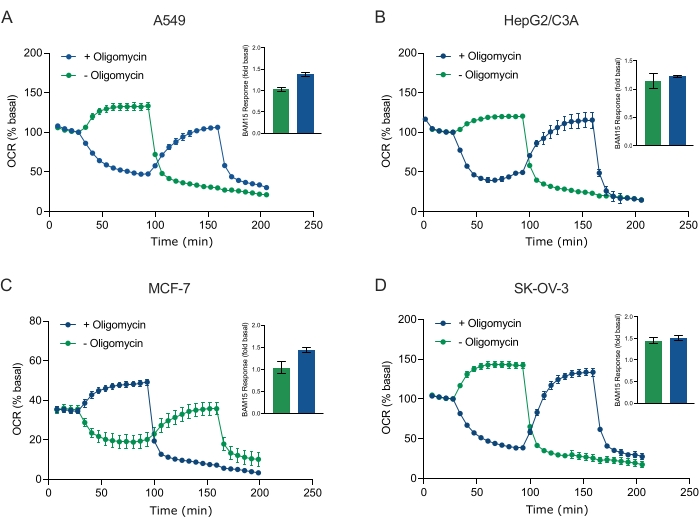
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
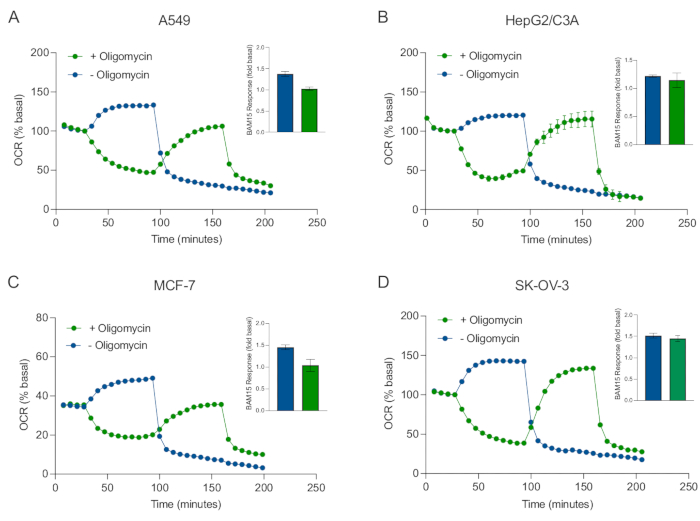
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır