Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Exploration du métabolisme énergétique mitochondrial des sphéroïdes de microtissu 3D uniques à l’aide de l’analyse du flux extracellulaire
Dans cet article
Erratum Notice
Résumé
Ces protocoles aideront les utilisateurs à sonder le métabolisme énergétique mitochondrial dans les sphéroïdes dérivés de lignées cellulaires cancéreuses 3D à l’aide de l’analyse du flux extracellulaire Seahorse.
Résumé
Les agrégats cellulaires tridimensionnels (3D), appelés sphéroïdes, sont devenus l’avant-garde de la culture cellulaire in vitro ces dernières années. Contrairement à la culture de cellules en tant que monocouches unicellulaires bidimensionnelles (culture 2D), la culture de cellules sphéroïdes favorise, régule et soutient l’architecture cellulaire physiologique et les caractéristiques qui existent in vivo, y compris l’expression des protéines de matrice extracellulaire, la signalisation cellulaire, l’expression génique, la production de protéines, la différenciation et la prolifération. L’importance de la culture 3D a été reconnue dans de nombreux domaines de recherche, notamment l’oncologie, le diabète, la biologie des cellules souches et le génie tissulaire. Au cours de la dernière décennie, des méthodes améliorées ont été développées pour produire des sphéroïdes et évaluer leur fonction métabolique et leur devenir.
Les analyseurs de flux extracellulaire (XF) ont été utilisés pour explorer la fonction mitochondriale dans des microtissus 3D tels que les sphéroïdes à l’aide d’une plaque de capture d’îlot XF24 ou d’une microplaque sphéroïde XFe96. Cependant, des protocoles distincts et l’optimisation du métabolisme énergétique mitochondrial sondant chez les sphéroïdes à l’aide de la technologie XF n’ont pas été décrits en détail. Cet article fournit des protocoles détaillés pour sonder le métabolisme énergétique mitochondrial dans des sphéroïdes 3D uniques à l’aide de microplaques sphéroïdes avec l’analyseur XFe96 XF. En utilisant différentes lignées cellulaires cancéreuses, il est démontré que la technologie XF est capable de distinguer la respiration cellulaire dans les sphéroïdes 3D de différentes tailles, mais aussi de différents volumes, numéros de cellules, contenu et type d’ADN.
Les concentrations optimales de composé effecteur mitochondrial d’oligomycine, de BAM15, de roténone et d’antimycine A sont utilisées pour sonder des paramètres spécifiques du métabolisme énergétique mitochondrial dans les sphéroïdes 3D. Cet article traite également des méthodes permettant de normaliser les données obtenues à partir de sphéroïdes et aborde de nombreuses considérations qui devraient être prises en compte lors de l’exploration du métabolisme des sphéroïdes à l’aide de la technologie XF. Ce protocole aidera à stimuler la recherche sur des modèles avancés de sphéroïdes in vitro .
Introduction
Les progrès des modèles in vitro dans la recherche biologique ont progressé rapidement au cours des 20 dernières années. Ces modèles incluent désormais des modalités d’organe sur puce, des organoïdes et des sphéroïdes de microtissus 3D, qui sont tous devenus un objectif commun pour améliorer la traduction entre les études in vitro et in vivo. L’utilisation de modèles in vitro avancés, en particulier les sphéroïdes, couvre plusieurs domaines de recherche, notamment l’ingénierie tissulaire, la recherche sur les cellules souches, le cancer et la biologie des maladies 1,2,3,4,5,6,7, et les tests de sécurité, y compris la toxicologie génétique 8,9,10, la toxicologie des nanomatériaux 11, 12,13,14, et tests d’innocuité et d’efficacité des médicaments 8,15,16,17,18,19.
La morphologie cellulaire normale est essentielle au phénotype et à l’activité biologiques. Cultiver des cellules en sphéroïdes de microtissus 3D permet aux cellules d’adopter une morphologie, une fonction phénotypique et une architecture, plus proches de celles observées in vivo mais difficiles à capturer avec les techniques classiques de culture cellulaire monocouche. Tant in vivo qu’in vitro, la fonction cellulaire est directement affectée par le microenvironnement cellulaire, qui ne se limite pas à la communication et à la programmation cellulaires (p. ex., formations de jonctions cellule-cellule, possibilités de former des niches cellulaires); l’exposition cellulaire aux hormones et aux facteurs de croissance dans les environnements immédiats (p. ex., exposition aux cytokines cellulaires dans le cadre d’une réponse inflammatoire); la composition des matrices physiques et chimiques (p. ex., si les cellules sont cultivées dans un plastique de culture tissulaire rigide ou dans un environnement tissulaire élastique); et surtout, comment le métabolisme cellulaire est affecté par la nutrition et l’accès à l’oxygène ainsi que par le traitement des déchets métaboliques tels que l’acide lactique.
L’analyse du flux métabolique est un moyen puissant d’examiner le métabolisme cellulaire dans des systèmes in vitro définis. Plus précisément, la technologie XF permet d’analyser les changements en direct et en temps réel dans la bioénergétique cellulaire des cellules et des tissus intacts. Étant donné que de nombreux événements métaboliques intracellulaires se produisent dans l’ordre de quelques secondes à quelques minutes, les approches fonctionnelles en temps réel sont primordiales pour comprendre les changements en temps réel du flux métabolique cellulaire dans les cellules et les tissus intacts in vitro.
Cet article fournit des protocoles pour la culture des lignées cellulaires dérivées du cancer A549 (adénocarcinome pulmonaire), HepG2/C3A (carcinome hépatocellulaire), MCF-7 (adénocarcinome du sein) et SK-OV-3 (adénocarcinome ovarien) en tant que modèles sphéroïdes 3D in vitro utilisant des approches d’agrégation forcée (Figure 1). Il décrit également en détail comment sonder le métabolisme énergétique mitochondrial de sphéroïdes 3D uniques à l’aide de l’analyseur Agilent XFe96 XF, (ii) met en évidence les moyens d’optimiser les tests XF à l’aide de sphéroïdes 3D uniques, et (iii) discute des considérations et des limites importantes de la sonde du métabolisme des sphéroïdes 3D à l’aide de cette approche. Plus important encore, cet article décrit comment sont collectés des ensembles de données qui permettent le calcul du taux de consommation d’oxygène (OCR) pour déterminer la phosphorylation oxydative et donc la fonction mitochondriale dans les sphéroïdes cellulaires. Bien qu’il ne soit pas analysé pour ce protocole, le taux d’acidification extracellulaire (ECAR) est un autre paramètre mesuré aux côtés des données OCR dans les expériences XF. Cependant, ECAR est souvent mal ou mal interprété à partir des jeux de données XF. Nous fournissons un commentaire sur les limites du calcul de l’ECAR en suivant les approches de base du fabricant de la technologie.
Access restricted. Please log in or start a trial to view this content.
Protocole
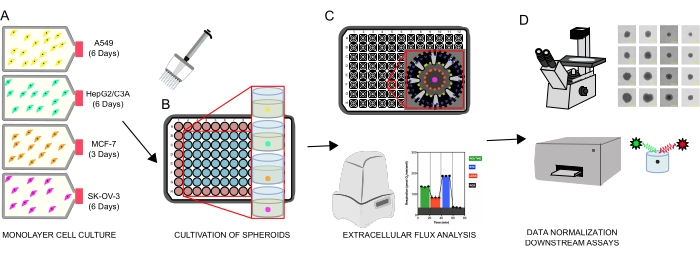
Figure 1 : Flux de travail graphique pour la génération de sphéroïdes cellulaires, l’analyse du flux extracellulaire et les essais en aval. Quatre lignées cellulaires cancéreuses ont été cultivées sélectivement sous forme de monocouches (A), détachées de flacons de culture tissulaire et ensemencées dans des microplaques ultrabasses de 96 puits pour former des sphéroïdes (B). Le carcinome pulmonaire A549, le carcinome hépatique HepG2/C3A, l’adénocarcinome ovarien SK-OV-3 et le carcinome du sein MCF-7 ont été ensemencés à 1 × 103-8 × 103 cellules/ puits et ont grandi jusqu’à 7 jours pour former des sphéroïdes uniques et optimiser la densité d’ensemencement des sphéroïdes et le temps de culture par observation continue et mesures planimétriques. Une fois formés, les sphéroïdes simples ont été lavés dans un milieu XF sans sérum et soigneusement ensemencés dans des microplaques de dosage sphéroïde, pré-enduites de poly-D-lysine (C). Les sphéroïdes ont été soumis à une analyse du flux extracellulaire à l’aide de l’analyseur XFe96 utilisant plusieurs protocoles pour traiter: (1) la taille optimale des sphéroïdes pour la réponse respiratoire mitochondriale basale; (2) titrage optimisé des inhibiteurs respiratoires mitochondriaux; (3) optimisation du placement des sphéroïdes dans les puits de microplaques. (D) Les analyses post-XF, la microscopie à contraste de phase et la quantification de l’ADN sphéroïde ont été utilisées pour la normalisation des données et d’autres essais in vitro en aval. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Culture de lignées cellulaires cancéreuses sous forme de sphéroïdes in vitro 3D
| Lignée cellulaire | Description | Milieu de culture | Source |
| L’A549 | Lignée cellulaire de carcinome pulmonaire | RPMI 1640 | Collection européenne de cultures cellulaires authentifiées (ECACC) |
| Pyruvate de sodium (1 mM) | |||
| Pénicilline- Streptomycine - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| HepG2/C3A | Lignée cellulaire de carcinome hépatique, un dérivé clonal de la lignée cellulaire parente HepG2 | Le | Collection américaine de culture tissulaire (ATCC) |
| Pénicilline- Streptomycine - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| MCF7 | Lignée cellulaire d’adénocarcinome du sein | RPMI 1640 | Collection européenne de cultures cellulaires authentifiées (ECACC) |
| Pyruvate de sodium (1 mM) | |||
| Pénicilline- Streptomycine - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| SK-OV-3 | Lignée cellulaire de l’adénocarcinome ovarien | RPMI 1640 | Collection européenne de cultures cellulaires authentifiées (ECACC) |
| Pyruvate de sodium (1 mM) | |||
| Pénicilline- Streptomycine - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| Composant | Milieu d’essai RPMI (volume final de 50 mL) | ||
| Milieu de base | Agilent Seahorse XF RPMI, pH 7,4 | ||
| Glucose (1 M de stock stérile) | 11 mM (solution mère de 0,55 mL) | ||
| L-glutamine (stock stérile de 200 mM) | 2 mM (0,5 mL de solution mère) | ||
| Pyruvate de sodium (stock stérile de 100 mM) | 1 mM (0,5 mL de solution mère) |
Tableau 1 : Compositions des milieux de lignées cellulaires cancéreuses et des milieux XF.
- Cultivez toutes les lignées cellulaires à l’aide de la technique de culture tissulaire aseptique standard et confirmez qu’elles sont exemptes de mycoplasmes à l’aide d’un kit de dosage approprié.
- Cultiver les lignées cellulaires dans des flacons de culture tissulaire T75 ou l’équivalent, en utilisant le milieu recommandé (tableau 1). Cultivez les lignées cellulaires à 65-80% de confluence et passez-les régulièrement jusqu’à un maximum de 25 passages.
- Rincez les flacons de culture cellulaire deux fois dans la solution saline tamponnée au phosphate (DBPS) modifiée de Dulbecco.
- Détacher les cellules des flacons avec 3 mL de réactif de dissociation cellulaire (voir le tableau des matériaux) pendant 5 min à 37 °C et confirmer le détachement par microscopie.
- Aspirer doucement la suspension cellulaire détachée pour assurer une suspension unicellulaire et désactiver le réactif de dissociation cellulaire avec 7 mL de milieu de culture tissulaire complet.
- Prélever les cellules par centrifugation à 300 × g pendant 5 min, jeter le surnageant et remettre les cellules en suspension dans un milieu complet.
- Comptez les cellules à l’aide d’un hémocytomètre ou d’un compteur cellulaire automatisé et titrez à la densité cellulaire souhaitée requise pour l’ensemencement.
REMARQUE: Pour ensemencer une plaque entière de 96 puits à 100 μL / puits à 4 × 103 cellules / puits, les cellules doivent être titrées à 4 × 104 cellules / mL dans un volume recommandé de 12 mL. - Décanter la suspension cellulaire dans un réservoir stérile et distribuer 100 μL de la suspension cellulaire dans chaque puits d’une microplaque répulsive cellulaire à l’aide d’un pipettor multicanal.
REMARQUE: Seuls les 60 puits intérieurs d’une microplaque doivent être ensemencés et le reste rempli de DPBS. Cela formera une barrière d’évaporation, assurera l’homogénéité des sphéroïdes sur la plaque et minimisera les effets de bord de la plaque. - Centrifuger les microplaques sphéroïdes à 300 × g pendant 15 min pour forcer les cellules en agrégats lâches.
- Incuber les plaques à 37 °C, 5% de CO2 pendant au moins 3 jours pour assurer la formation de sphéroïdes.
- Effectuer une microscopie à contraste de phase en utilisant des pratiques de laboratoire normalisées pour surveiller la croissance des sphéroïdes. Reconstituer le milieu de culture cellulaire tous les 3 jours ou deux fois par semaine en effectuant un échange de milieu d’un demi-volume.
2. Sonder le métabolisme énergétique mitochondrial des sphéroïdes simples à l’aide de la technologie de flux extracellulaire (XF)
- Préparation du test (un jour avant)
- Vérifiez la viabilité des sphéroïdes à l’aide d’un microscope à lumière inversée avec contraste de phase à un grossissement de 4x pour assurer une structure sphéroïde intacte, une morphologie et une uniformité globale entre les échantillons.
- Hydratez la cartouche du capteur.
- Aliquote ~20 mL du calibrant dans un tube conique.
- Placer le tube conique contenant l’étrier dans un incubateur sans CO2 37 °C pendant la nuit.
- Retirez le contenu du kit d’analyse.
- Retirez la cartouche du capteur de la plaque utilitaire et placez-la à l’envers sur le plan de travail à côté de la plaque utilitaire.
- Pipette 200 μL de ddH2O stérile dans chaque puits de la plaque d’utilité de la cartouche du capteur à l’aide d’une pipette P300 multicanal.
- Placez la cartouche du capteur sur le dessus de la plaque utilitaire.
- Vérifiez que le niveau d’eau dans chaque puits est suffisamment élevé pour submerger les sondes du capteur.
- Transférez la cartouche de capteur assemblée dans un incubateur sans CO2 37 °C et laissez-la toute la nuit.
REMARQUE: Cette étape peut être effectuée 12 à 72 heures avant le début du test.
- Microplaque de dosage sphéroïde de couche
- À l’aide de techniques aseptiques, ajouter 30 μL/puits de poly-D-lysine stérile (0,1 mg/mL) à la microplaque sphéroïde et l’incuber pendant 30 min à température ambiante.
- Aspirer la solution de chaque puits de la microplaque sphéroïde, inverser la plaque et la tapoter fermement sur le papier de soie pour éliminer toute solution résiduelle.
- Laver la plaque deux fois avec 200 μL/puits de ddH2O stérile.
- Après le lavage final, inverser la microplaque et la tapoter fermement sur du papier de soie pour éliminer toute eau résiduelle.
- Laisser sécher la plaque à l’air libre pendant 30 min avant de l’utiliser ou de la stocker à 4 °C pour une utilisation future.
REMARQUE: La microplaque de dosage des sphéroïdes doit être recouverte d’un adhésif moléculaire pour s’assurer que les sphéroïdes sont fixés au bas de la microplaque. Sans adhésif moléculaire, les sphéroïdes peuvent se déloger et interférer avec les résultats des tests. D’autres adhésifs moléculaires peuvent également être utilisés comme alternative à la poly-D-lysine pour les plaques de prérevêtement. Les plaques prélaquées peuvent être conservées à 4 °C, mais doivent être laissées à la température ambiante avant le début du dosage.
- Préparer le milieu du test XF
- Préparer le milieu XF RPMI, comme indiqué dans le tableau 1, et le filtre stérile avec un filtre à seringue de 0,22 μm
- Préparation du test (1 h avant le test)
- Préchauffer le milieu d’essai XF RPMI supplémenté jusqu’à 37 °C.
- Préchauffer la microplaque de dosage sphéroïde revêtue dans un incubateur ou un bain sec sans CO2 à 37 °C.
- Préparez la cartouche du capteur.
- Retirez le tube conique contenant l’étrier et la cartouche du capteur de l’incubateur à air.
- Retirez la cartouche du capteur de la plaque utilitaire et placez-la à l’envers sur la surface de travail.
- À l’aide d’une pipette multicanal P300, aspirez l’eau de la plaque de service public et jetez-la.
- Versez la solution d’étriquant dans un réservoir de réactif stérile et ajoutez 200 μL/puits du calibrant préavertissé à la plaque utilitaire à l’aide d’une pipette multicanal P300.
- Prenez la cartouche du capteur et replacez-la sur la plaque d’utilité, en vous assurant que les capteurs sont bien immergés dans l’étrier.
- Transférer la cartouche de capteur assemblée dans l’incubateur sans CO2 37 °C jusqu’à ce qu’elle soit prête à charger les solutions d’injection de orifice.
- Lavez les sphéroïdes avec le milieu d’essai.
- Retirez la plaque de culture sphéroïde de l’incubateur à 37 °C, 5 % de CO2 et observez les sphéroïdes au microscope pour assurer leur intégrité avant les étapes de transfert des sphéroïdes.
- Charger tous les puits de la plaque sphéroïde avec 180 μL/puits de milieu d’essai préavertis, y compris tous les puits de correction de fond.
- Remplissez partiellement une boîte de Petri de 7 cm avec 3 mL du milieu d’essai.
- À l’aide d’une pipette multicanal chargée de larges pointes de pipette à orifice, transférez les sphéroïdes de la plaque de culture à 96 puits dans des boîtes de Petri de 7 cm en réglant le pipetteur à un volume d’aspiration de 10 à 50 μL.
- Ensemencez les sphéroïdes dans la microplaque de dosage des sphéroïdes pré-enduits.
- À l’aide d’un microscope à dissection et d’un appareil lightbox, transférez les sphéroïdes de la boîte de Petri à la microplaque de dosage des sphéroïdes, comme indiqué ci-dessous.
- Réglez le volume d’un pipetteur monocanal équipé d’une large pointe de pipette à orifice à 20 μL et aspirez soigneusement un seul sphéroïde. Placez la pointe directement au centre de chaque puits de la microplaque d’essai sphéroïde et laissez la gravité éluer un seul sphéroïde au centre de chaque puits, c’est-à-dire n’expulsez aucun milieu de la pointe de la pipette et laissez l’action capillaire retirer le sphéroïde de la pointe de la pipette. Pour confirmer l’élution, le contenu du pipetteur peut être pipeté dans la boîte de Petri de 7 cm au microscope.
REMARQUE: L’élution par gravité d’un seul sphéroïde prend généralement 15 à 30 s en fonction de la taille / densité du sphéroïde. Pendant ce temps, le pipetteur ne doit pas être retiré. Tout puits de correction de fond doit être exempt de sphéroïdes et ne contenir que du milieu d’essai. Sous le microscope, confirmez la position de chaque sphéroïde. Chaque sphéroïde devrait idéalement être positionné au centre de chaque puits. - Une fois que tous les sphéroïdes ont été transférés sur la microplaque d’essai des sphéroïdes, transférer la plaque dans un incubateur sans CO2 à 37 °C pendant au moins 1 h avant le test.
- Réglez le volume d’un pipetteur monocanal équipé d’une large pointe de pipette à orifice à 20 μL et aspirez soigneusement un seul sphéroïde. Placez la pointe directement au centre de chaque puits de la microplaque d’essai sphéroïde et laissez la gravité éluer un seul sphéroïde au centre de chaque puits, c’est-à-dire n’expulsez aucun milieu de la pointe de la pipette et laissez l’action capillaire retirer le sphéroïde de la pointe de la pipette. Pour confirmer l’élution, le contenu du pipetteur peut être pipeté dans la boîte de Petri de 7 cm au microscope.
- À l’aide d’un microscope à dissection et d’un appareil lightbox, transférez les sphéroïdes de la boîte de Petri à la microplaque de dosage des sphéroïdes, comme indiqué ci-dessous.
3. Préparation et chargement des composés dans la cartouche du capteur pour les tests XF
| Stratégie d’injection | Composé (port) | Volume de démarrage du micropuit XFe96 (μL) | Concentration finale souhaitée dans le puits | Volume du port (μL) | Volume final du micropuit XFe96 après injection (μL) | Concentration du stock de travail |
| 1 | Oligomycine (A) | 180 | 3 ug/mL | 20 | 200 | 30 μg/mL |
| Roténone (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimycine A (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| Roténone (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| Antimycine A (B) | 200 | 2 μM | 20 | 220 | 22 μM |
Tableau 2 : Concentrations de composés mitochondriaux pour sonder le métabolisme énergétique mitochondrial de sphéroïdes 3D uniques à l’aide de l’analyseur XFe96.
- Préparer les concentrations sur le stock de travail de chaque composé, comme indiqué dans le tableau 2 , à l’aide d’un milieu d’essai XF RPMI préavertissé entièrement complété.
- Orientez la plaque de cartouche (couplée à la plaque utilitaire) en colonne, 1-12 de gauche à droite.
- Si vous utilisez un guide de chargement, placez-le sur la plaque de la cartouche conformément à la procédure de chargement du puits, par exemple, si le port A est chargé en premier, assurez-vous que A est visible dans le coin supérieur gauche du guide.
- Transférer la solution de travail de chaque composé dans un réservoir approprié et, à l’aide d’une pipette multicanal P100 étalonnée, distribuer 20 μL dans tous les ports correspondants. Répétez l’opération pour chaque composé dans les ports restants.
REMARQUE: Si aucun port n’est utilisé sur la plaque de la cartouche du capteur, ceux-ci peuvent être laissés vides ou remplis de support de dosage. Si seule une sélection d’une lettre de port spécifique est utilisée, assurez-vous que les autres ports correspondant à cette lettre sont chargés avec un support d’essai; sinon, de l’air sera injecté dans le puits, compromettant les résultats dans ces puits. - Après le chargement du port, retirez les guides de chargement de la plaque (le cas échéant) et préparez l’analyseur pour le chargement de la cartouche du capteur.
REMARQUE: Si le test n’est pas exécuté immédiatement après le chargement des ports, replacez le couvercle sur la cartouche du capteur et remettez la plaque dans l’incubateur à air à 37 ° C jusqu’à ce qu’elle soit prête à charger dans la machine.
4. Conception des essais, stratégies d’injection et acquisition de données
- Exécution du test
- Mettez l’analyseur sous tension et connectez-le au contrôleur (ordinateur).
REMARQUE: Cela peut être vérifié par l’état de la connexion de l’instrument dans le panneau de widget du logiciel Wave Controller. - Accédez à la page des modèles dans le logiciel WAVE, recherchez le fichier de modèle de test de l’expérience et double-cliquez dessus pour l’ouvrir.
Remarque : Si le modèle de test n’apparaît pas dans la vue Modèles , importez le fichier de modèle dans le dossier de modèle à partir d’un lecteur réseau partagé ou d’un lecteur flash USB. - Pour démarrer le test, cliquez sur l’onglet Exécuter le test .
REMARQUE: Si les définitions de groupe ont été correctement attribuées dans la carte des plaques, le test sera prêt à s’exécuter comme indiqué par la coche verte sur le côté droit de la page. À ce stade, toute information supplémentaire peut être saisie sur la page de résumé de l’essai ou sur la page laissée vide; passez à l’étape suivante. En raison de la pénétration retardée des modulateurs mitochondriaux dans les sphéroïdes de microtissus 3D (Figure 2), utilisez les informations du protocole de mesure décrites dans le tableau 3.
- Mettez l’analyseur sous tension et connectez-le au contrôleur (ordinateur).
| Période de mesure | Numéro d’injection et port | Détails de la mesure | Durée de la période (h:min:s) |
| Étalonnage | Sans objet | Les analyseurs XF effectuent toujours cet étalonnage pour s’assurer que les mesures sont précises | 00:20:00 (il s’agit d’une moyenne qui peut varier d’une machine à l’autre) |
| Équillibration | Sans objet | L’équilibrage a lieu après l’étalonnage et il est recommandé. | 00:10:00 |
| Basal | Sans objet | Cycles = 5 | 00:30:00 |
| Mélange = 3:00 | |||
| Attendre = 0:00 | |||
| Mesure = 3:00 | |||
| Oligomycine / BAM15 | Injection 1 (Port A) | Cycles = 10 | 01:00:00 |
| Mélange = 3:00 | |||
| Attendre = 0:00 | |||
| Mesure = 3:00 | |||
| Roténone + antimycine A | Injection 2 (Port B) | Cycles = 10 | 01:00:00 |
| Mélange = 3:00 | |||
| Attendre = 0:00 | |||
| Mesure = 3:00 | |||
| Temps total: | 03:00:00 | ||
Tableau 3 : Configuration du protocole pour sonder le métabolisme énergétique mitochondrial de sphéroïdes 3D uniques à l’aide de l’analyseur XFe96.
- Cliquez sur Démarrer pour afficher la boîte de dialogue Emplacement d’enregistrement .
- Entrez l’emplacement d’enregistrement du fichier de résultats et placez la cartouche de capteur assemblée sur le plateau thermique qui apparaît de la porte sur le côté de l’analyseur. Attendez que le plateau thermique s’ouvre automatiquement et que l’écran affiche le message Load Calibrant Utility Plate . Avant de suivre les instructions à l’écran, assurez-vous i) que la cartouche du capteur est bien ajustée sur la plaque utilitaire, ii) que le couvercle est retiré de la cartouche du capteur et iii) que la cartouche du capteur est correctement orientée sur la plaque utilitaire.
- Suivez les commandes à l’écran pour lancer l’étalonnage de la cartouche du capteur.
REMARQUE: Le temps nécessaire pour terminer l’étalonnage est d’environ 10-20 min (pour les essais à 37 ° C). - Après l’étalonnage de la cartouche du capteur, chargez la microplaque sphéroïde dans l’analyseur en suivant les instructions à l’écran sur le contrôleur d’ondes pour lancer l’étape d’équilibrage de 12 minutes.
REMARQUE: Les cases vertes avec des coches blanches indiquent un « bon » étalonnage pour ce puits. Si des puits ne fournissent pas un « bon » étalonnage, ils seront indiqués par une boîte rouge et une croix blanche. Ces puits doivent être notés et exclus de toute analyse une fois que l’essai est terminé à l’aide de l’onglet de l’essai de modification . - Attendez que l’analyseur commence automatiquement à acquérir des mesures de base une fois que la machine a terminé l’étape d’équilibrage (comme indiqué dans le protocole de l’instrument).
- Pour terminer l’expérience, suivez les commandes à l’écran sur le contrôleur WAVE.
REMARQUE: Une fois que la microplaque sphéroïde a été retirée de l’analyseur, jetez la cartouche du capteur et mettez de côté la plaque sphéroïde pour une analyse plus approfondie si nécessaire (par exemple, quantification de l’ADN double brin (ds)). Si la microplaque n’est pas nécessaire pour une analyse plus approfondie, elle peut être jetée avec la cartouche du capteur. - Attendez que la boîte de dialogue d’analyse s’affiche et affichez les résultats ou revenez à la vue des modèles .
5. Stratégies de normalisation et d’analyse des données - normalisation post-essai et essais en aval (étapes facultatives)
- Normalisation des données
- Pour normaliser les données sphéroïdes, reportez-vous à la série de protocoles pertinents pour les stratégies de normalisation des données pour calculer la taille et le volume des sphéroïdes et quantifier l’ADNds dans les essais sphéroïdes. Ceux-ci ont été inclus en tant que fichiers supplémentaires; voir Fichier supplémentaire 1 et Fichier supplémentaire 2.
- Analyse des données
- Pour exporter des données dans l’un des générateurs d’analyse automatisés, suivez les commandes d’exportation de données sur le contrôleur WAVE et sélectionnez le générateur d’exportation qui correspond au type de test. Vous pouvez également exporter le fichier de données et le télécharger dans Seahorse Analytics.
REMARQUE: L’inconvénient des générateurs de rapports et de l’analyse Seahorse est que l’analyse des données est limitée à la façon dont le test XF est conçu et ne permet pas de prendre des moyennes à travers les cycles de mesure. L’exportation manuelle des jeux de données à partir du logiciel de l’instrument permet à l’utilisateur de préférer à cet égard. Étant donné que la stratégie d’injection pour évaluer la respiration mitochondriale des sphéroïdes 3D sera probablement différente de celle d’un test typique « MitoStress », une série de modèles de feuilles de calcul ont été développés pour aider à analyser ces ensembles de données, spécifiques aux cultures cellulaires 3D et seront fournis sur demande. Ces fichiers de modèles de données fourniront des données sur les principaux paramètres respiratoires mitochondriaux détaillés et expliqués à la figure 2. - Pour analyser les données, exportez les données sous forme de rapport de feuille de calcul à partir du logiciel de contrôleur WAVE et utilisez un modèle de feuille de calcul indépendant pour l’analyse.
- Pour exporter des données dans l’un des générateurs d’analyse automatisés, suivez les commandes d’exportation de données sur le contrôleur WAVE et sélectionnez le générateur d’exportation qui correspond au type de test. Vous pouvez également exporter le fichier de données et le télécharger dans Seahorse Analytics.
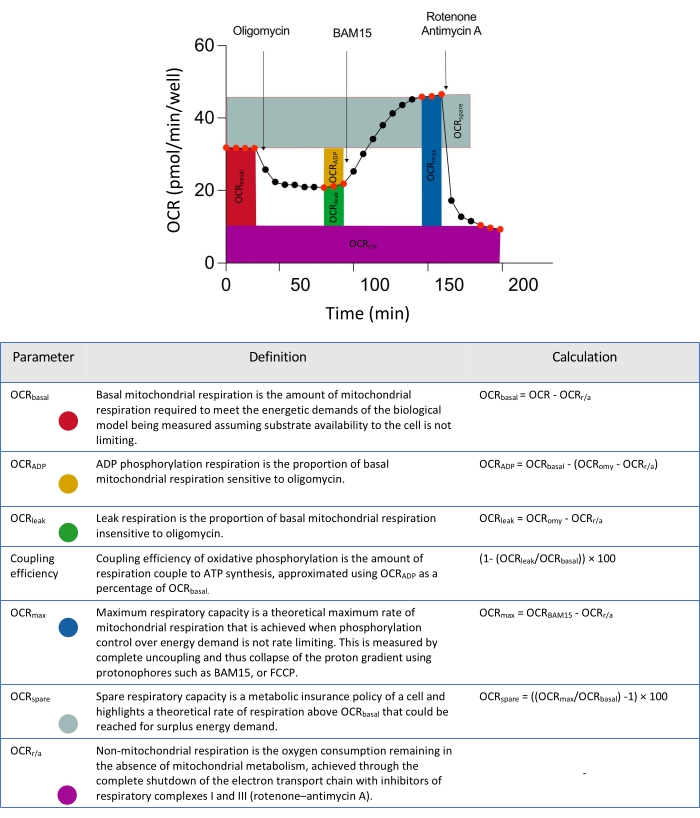
Figure 2 : Descripteurs schématiques pour les paramètres dérivés des analyses de données de flux extracellulaire. Abréviation : OCR = taux de consommation d’oxygène. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Pour obtenir des sphéroïdes compacts et bien formés, chaque lignée cellulaire a été optimisée individuellement pour la densité d’ensemencement et la durée de culture (figure 3). Les lignées cellulaires A549, HepG2/C3A et SK-OV-3 ont initialement formé des agrégats lâches qui n’ont progressé vers des sphéroïdes ronds avec des périmètres clairement définis qu’après 7 jours de culture. Inversement, les cellules MCF-7 pourraient former des sphéroïdes dans les 3 jours....
Access restricted. Please log in or start a trial to view this content.
Discussion
Principales constatations et extrants
Cet article fournit un protocole détaillé pour sonder le métabolisme énergétique mitochondrial de sphéroïdes 3D uniques à l’aide d’une série de lignées cellulaires dérivées du cancer avec l’analyseur XFe96 XF. Une méthode est développée et décrite pour la culture rapide des sphéroïdes cellulaires A549, HepG2/C3A, MCF7 et SK-OV-3 en utilisant des technologies d’hydrofuge cellulaire pour l’agrégation forcée. Ce protocole aborde de nombr...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
N.J.C a reçu un BBSRC MIBTP CASE Award avec Sygnature Discovery Ltd (BB/M01116X/1, 1940003)
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
Références
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
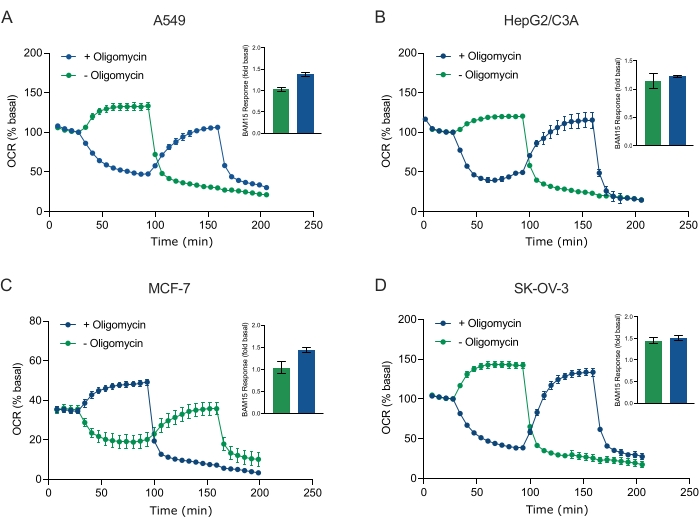
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
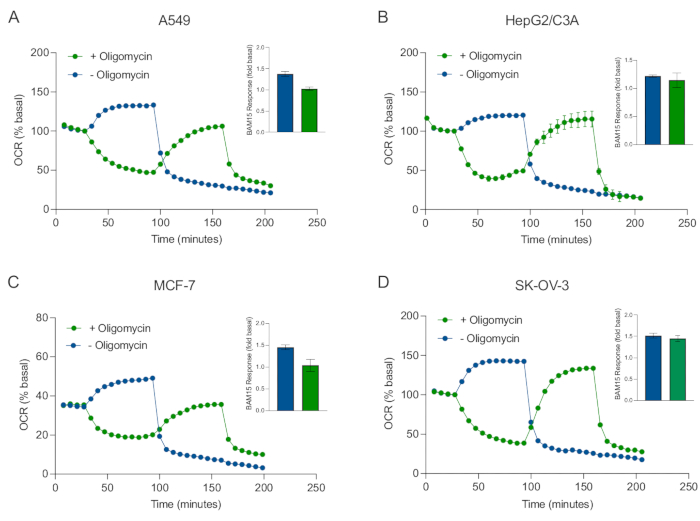
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon