このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細胞外フラックス解析を用いた単一3D微細組織スフェロイドのミトコンドリアエネルギー代謝の探索
Erratum Notice
要約
これらのプロトコルは、タツノオトシゴの細胞外フラックス分析を使用して、3Dがん細胞株由来スフェロイドにおけるミトコンドリアエネルギー代謝を調べるのに役立ちます。
要約
スフェロイドと呼ばれる三次元(3D)細胞凝集体は、近年、 in vitro 細胞培養の最前線となっている。細胞を2次元の単一細胞単層(2D培養)として培養するのとは対照的に、スフェロイド細胞培養は、細胞外マトリックスタンパク質の発現、細胞シグナル伝達、遺伝子発現、タンパク質産生、分化、および増殖を含む、 生体内に存在する生理学的細胞アーキテクチャおよび特性を促進し、調節し、サポートする。3D培養の重要性は、腫瘍学、糖尿病、幹細胞生物学、組織工学など、多くの研究分野で認識されています。過去10年間で、スフェロイドを産生し、その代謝機能と運命を評価するための改良された方法が開発されました。
細胞外フラックス(XF)分析装置は、XF24膵島捕捉プレートまたはXFe96スフェロイドマイクロプレートのいずれかを使用して、スフェロイドなどの3Dマイクロ組織におけるミトコンドリア機能を探索するために使用されてきました。しかしながら、XF技術を用いたスフェロイドにおけるミトコンドリアエネルギー代謝のプロービングの明確なプロトコルおよび最適化は、詳細には記載されていない。この論文では、XFe96 XFアナライザーを搭載したスフェロイドマイクロプレートを使用して、単一の3Dスフェロイドのミトコンドリアエネルギー代謝を調べるための詳細なプロトコルを提供します。異なるがん細胞株を使用して、XF技術は、異なるサイズだけでなく、異なる体積、細胞数、DNA含量およびタイプの3Dスフェロイドにおける細胞呼吸を区別できることが実証されている。
オリゴマイシン、BAM15、ロテノン、およびアンチマイシンAの最適なミトコンドリアエフェクター化合物濃度は、3Dスフェロイドにおけるミトコンドリアエネルギー代謝の特異的パラメータをプローブするために使用される。この論文では、回転楕円体から得られたデータを正規化する方法についても説明し、XF技術を使用して回転楕円体代謝を探索する際に考慮すべき多くの考慮事項について説明します。このプロトコルは、高度な in vitro スフェロイドモデルの研究を促進するのに役立ちます。
概要
生物学的研究におけるin vitroモデルの進歩は、過去20年間で急速に進歩しました。このようなモデルには、臓器オンチップモダリティ、オルガノイド、および3Dマイクロ組織スフェロイドが含まれ、これらはすべて、in vitroおよびin vivo研究間の翻訳を改善するための共通の焦点となっている。高度なin vitroモデル、特にスフェロイドの使用は、組織工学、幹細胞研究、癌、および疾患生物学1,2,3,4,5,6,7、および遺伝毒物学8,9,10、ナノ材料毒物学11を含む安全性試験を含むいくつかの研究分野にまたがり、 12、13、14、および薬物安全性および有効性試験8、15、16、17、18、19。
正常な細胞形態は、生物学的表現型および活性にとって重要である。細胞を3Dマイクロティッシュスフェロイドに培養することで、細胞は形態、表現型機能、およびアーキテクチャを採用することができ、 これは生体内で 観察されるものに似ていますが、古典的な単層細胞培養技術では捕捉が困難です。 インビボ および インビトロ の両方で、細胞機能は、細胞のコミュニケーションおよびプログラミング(例えば、細胞 - 細胞接合の形成、細胞ニッチを形成する機会)に限定されない細胞微小環境によって直接影響を受ける。即時環境におけるホルモンおよび成長因子への細胞曝露(例えば、炎症反応の一部としての細胞サイトカイン曝露);物理的および化学的マトリックスの組成(例えば、細胞が硬い組織培養プラスチックまたは弾性組織環境で増殖されるかどうか);そして最も重要なのは、細胞代謝が栄養と酸素へのアクセス、ならびに乳酸などの代謝性老廃物の処理によってどのように影響を受けるかということです。
代謝フラックス分析は、定義された in vitro システム内の細胞代謝を調べるための強力な方法です。具体的には、XF技術は、無傷の細胞および組織の細胞生体エネルギーの生きたリアルタイム変化の分析を可能にする。多くの細胞内代謝事象が数秒から数分のオーダーで起こることを考えると、イン ビトロで無傷の細胞および組織における細胞代謝フラックスのリアルタイム変化を理解するためには、リアルタイムの機能的アプローチが最も重要である。
本論文では、強制凝集アプローチを用いた in vitro 3Dスフェロイドモデルとして、がん由来細胞株A549(肺腺がん)、HepG2/C3A(肝細胞がん)、MCF-7(乳腺がん)、SK-OV-3(卵巣腺がん)を培養するためのプロトコルを提供する(図1)。また、(i)Agilent XFe96 XFアナライザーを使用して単一の3Dスフェロイドのミトコンドリアエネルギー代謝を調べる方法を詳細に説明し、(ii)単一の3Dスフェロイドを使用してXFアッセイを最適化する方法を強調し、(iii)このアプローチを使用して3Dスフェロイド代謝をプローブする重要な考慮事項と制限について説明します。最も重要なのは、この論文では、酸素消費速度(OCR)の計算が酸化的リン酸化を決定し、したがって細胞スフェロイドのミトコンドリア機能を決定することを可能にするデータセットがどのように収集されるかを説明する。このプロトコールについては分析されていないが、細胞外酸性化率(ECAR)は、XF実験においてOCRデータとともに測定される別のパラメータである。ただし、ECAR は XF データセットから不適切または誤って解釈されることがよくあります。テクノロジーメーカーの基本的なアプローチに従ってECARを計算することの限界について解説します。
Access restricted. Please log in or start a trial to view this content.
プロトコル
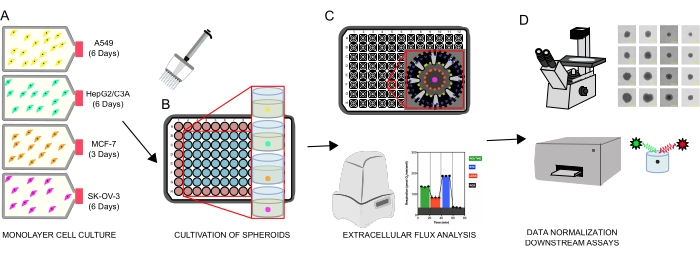
図1:細胞スフェロイドの生成、細胞外フラックス分析、ダウンストリームアッセイのグラフィカルワークフロー。4つの癌細胞株を単層(A)として選択的に培養し、組織培養フラスコから剥離し、超低付着96ウェルマイクロプレートに播種してスフェロイド(B)を形成した。A549肺癌、HepG2/C3A肝癌、SK-OV-3卵巣腺癌、およびMCF-7乳癌細胞を1×103-8×103細胞/ウェルで播種し、7日間まで増殖させて単一のスフェロイドを形成し、連続観察および平面測定によりスフェロイドの播種密度および培養時間を最適化した。一旦形成されると、単一のスフェロイドを無血清XF培地に洗浄し、ポリD-リジン(C)でプレコートしたスフェロイドアッセイマイクロプレートに慎重に播種した。スフェロイドは、いくつかのプロトコルを使用してXFe96アナライザーを使用して細胞外フラックス分析を行い、(1)基底ミトコンドリア呼吸応答に最適なスフェロイドサイズ;(2)ミトコンドリア呼吸器阻害剤の最適化された滴定;(3)マイクロプレートウェル内の回転楕円体配置の最適化。(d)XF後分析、位相差顕微鏡、およびスフェロイドDNA定量を、データ正規化および他の下流のインビトロアッセイに使用した。この図の拡大版を表示するには、ここをクリックしてください。
1. がん細胞株を3D in vitro スフェロイドとして培養
| 細胞株 | 形容 | 培地 | 源 |
| A549 · | 肺癌細胞株 | RPMI 1640 | 欧州認証細胞培養物コレクション(ECACC) |
| ピルビン酸ナトリウム (1 mM) | |||
| ペニシリン - ストレプトマイシン - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| HepG2/C3A | 肝癌細胞株は、親HepG2細胞株のクローン誘導体である | ティッカー | アメリカンティッシュカルチャーコレクション(ATCC) |
| ペニシリン - ストレプトマイシン - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| ティッカー | 乳房腺癌細胞株 | RPMI 1640 | 欧州認証細胞培養物コレクション(ECACC) |
| ピルビン酸ナトリウム (1 mM) | |||
| ペニシリン - ストレプトマイシン - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| SK-OV-3 | 卵巣腺癌細胞株 | RPMI 1640 | 欧州認証細胞培養物コレクション(ECACC) |
| ピルビン酸ナトリウム (1 mM) | |||
| ペニシリン - ストレプトマイシン - (100 U/mL – 100 mg/mL) | |||
| 10 % (v/v) FBS | |||
| コンポーネント | RPMIアッセイ培地(50mL最終容量) | ||
| ベースメディア | アジレントタツノオトシゴXF RPMI、pH 7.4 | ||
| グルコース (1 M 滅菌ストック) | 11 mM (0.55 mL ストック ソリューション) | ||
| L-グルタミン(200 mM滅菌ストック) | 2 mM (0.5 mL の原液) | ||
| ピルビン酸ナトリウム(100 mM滅菌ストック) | 1 mM (0.5 mL の原液) |
表1:癌細胞株培地およびXF培地組成物。
- 標準的な無菌組織培養技術を用いて全ての細胞株を培養し、適切なアッセイキットを用いてマイコプラズマを含まないことを確認する。
- T75組織培養フラスコまたは同等の細胞株を、推奨培地を用いて培養する(表1)。細胞株を65〜80%のコンフルエント度まで培養し、最大25継代まで定期的に継代する。
- 細胞培養フラスコをダルベッコの修飾リン酸緩衝生理食塩水(DBPS)で2回すすいでください。
- 3mLの細胞解離試薬( 材料表参照)を入れたフラスコから細胞を37°Cで5分間剥離し、顕微鏡観察により剥離を確認した。
- 剥離した細胞懸濁液を穏やかに吸引して単一細胞懸濁液を確保し、7mLの完全組織培養培地で細胞解離試薬を失活させる。
- 300 × g で5分間遠心分離して細胞を集め、上清を捨て、細胞を完全培地に再懸濁した。
- 血球計数器または自動セルカウンターを使用して細胞をカウントし、播種に必要な所望の細胞密度まで滴定する。
注: 96 ウェルプレート全体を 100 μL/ウェルで 4 ~103 × ウェルで播種するには、推奨容量 12 mL で細胞を 4 ×104 細胞/mL まで滴定する必要があります。 - 細胞懸濁液を滅菌リザーバーにデカントし、マルチチャンネルピペッターを使用して細胞撥水性マイクロプレートの各ウェルに細胞懸濁液100μLを分配する。
注:マイクロプレートの内側60ウェルのみを播種し、残りをDPBSで満たしてください。これにより、蒸発障壁が形成され、プレート全体で回転楕円体の均質性が確保され、プレートエッジの影響が最小限に抑えられます。 - スフェロイドマイクロプレートを300 × g で15分間遠心分離し、細胞を緩い凝集体に強制した。
- プレートを37°C、5%CO2 で最低3日間インキュベートし、スフェロイド形成を確実にします。
- 標準化された実験室の実践を使用して位相差顕微鏡法を実行し、スフェロイドの成長を監視します。細胞培養培地を3日ごとまたは週2回、半容量培地交換を行って補充する。
2. 細胞外フラックス(XF)技術を用いた単一スフェロイドのミトコンドリアエネルギー代謝の解明
- アッセイ調製(1日前)
- 倍率4倍の位相差を備えた倒立光学顕微鏡を使用して回転楕円体の生存率を確認し、無傷の回転楕円体構造、形態、およびサンプル間の全体的な均一性を確保します。
- センサーカートリッジをハイドレートします。
- キャリブラントのアリコート〜20mLを円錐形のチューブに入れる。
- キャリブラントを入れた円錐管を非CO2 37°Cインキュベーターに入れ、一晩置いた。
- アッセイキットから内容物を取り除きます。
- センサーカートリッジをユーティリティー・プレートから取り外し、ユーティリティー・プレートの横にあるワークトップに逆さまに置きます。
- 200 μLの滅菌ddH2OをマルチチャンネルP300ピペットを用いてセンサーカートリッジユーティリティプレートの各ウェルにピペット。
- センサーカートリッジをユーティリティープレートの上に置きます。
- 各井戸の水位がセンサープローブを水没させるのに十分な高さであることを確認します。
- 組み立てたセンサーカートリッジを非CO2 37°Cインキュベーターに移し、一晩放置します。
注:このステップは、アッセイ開始の12〜72時間前に実行できます。
- コートスフェロイドアッセイマイクロプレート
- 無菌技術を使用して、30 μL/ウェルの滅菌ポリD-リジン(0.1 mg/mL)溶液をスフェロイドマイクロプレートに加え、室温で30分間インキュベートします。
- 回転楕円体マイクロプレートの各ウェルから溶液を吸引し、プレートを反転させ、ティッシュペーパーにしっかりと叩いて残留溶液を除去します。
- プレートを200 μL/ウェルの滅菌ddH 2 Oで2回洗浄します。
- 最後の洗浄後、マイクロプレートを反転させ、ティッシュペーパーにしっかりと叩いて残留水を取り除きます。
- プレートを30分間風乾してから使用または保管し、将来の使用に備えて4°Cで保管してください。
注:スフェロイドアッセイマイクロプレートは、スフェロイドがマイクロプレートの底部に固定されていることを確認するために、分子接着剤でコーティングする必要があります。分子接着剤がないと、スフェロイドが外れてアッセイ結果を妨げる可能性があります。他の分子接着剤も、プレコートプレート用のポリ-D-リジンの代替として使用することができる。プレコートプレートは4°Cで保存できますが、アッセイ開始前に室温に平衡化するようにしておく必要があります。
- XF アッセイ培地の調製
- 表1に詳述されているように、XF RPMI培地を調製し、0.22μmシリンジフィルターを備えた滅菌フィルターを調製する
- アッセイ調製(アッセイの1時間前)
- 補充したXF RPMIアッセイ培地を37°Cに予温する。
- コーティングしたスフェロイドアッセイマイクロプレートを非CO2 37°Cインキュベーターまたは乾燥浴中で予温する。
- センサーカートリッジを準備します。
- キャリブラントとセンサーカートリッジが入った円錐管をエアインキュベーターから取り出します。
- センサーカートリッジをユーティリティプレートから取り外し、作業台に逆さまに置きます。
- P300マルチチャンネルピペットを使用して、ユーティリティプレートから水を吸引し、廃棄します。
- キャリブラント溶液を滅菌試薬リザーバに注ぎ、P300マルチチャンネルピペットを使用して、予熱したキャリブラントをユーティリティプレートに200μL/ウェル加えます。
- センサーカートリッジを拾い上げてユーティリティプレートの上に置き、センサーがキャリブラントにしっかりと沈んでいることを確認します。
- 組み立てたセンサーカートリッジを、ポート注入溶液をロードする準備ができるまで、非CO2 37°Cインキュベーターに戻します。
- スフェロイドをアッセイ培地で洗浄する。
- 37°C、5%CO2 インキュベーターからスフェロイド培養プレートを取り出し、スフェロイド転写ステップの前にそれらの完全性を確実にするために顕微鏡下でスフェロイドを観察した。
- スフェロイドプレートのすべてのウェルに、バックグラウンド補正ウェルを含む180 μL/ウェルの予温アッセイ培地をロードします。
- 7cmのシャーレに3mLのアッセイ培地を部分的に充填する。
- 広いオリフィスピペットチップをロードしたマルチチャンネルピペットを使用して、ピペッターを10〜50μLの吸引容量に設定して、96ウェル培養プレートから7cmペトリ皿にスフェロイドを移します。
- スフェロイドを予めコーティングしたスフェロイドアッセイマイクロプレートにシードする。
- 解剖顕微鏡およびライトボックス装置を用いて、以下に詳述するように、スフェロイドをシャーレからスフェロイドアッセイマイクロプレートに移す。
- 広いオリフィスピペットチップを取り付けたシングルチャンネルピペッターの容量を20μLに設定し、単一のスフェロイドを慎重に吸引します。先端をスフェロイドアッセイマイクロプレートの各ウェルの中央に直接置き、重力が単一のスフェロイドを各ウェルの中心に溶出させる、すなわち、ピペットチップから媒体を排出せず、毛細管現象がピペットチップからスフェロイドを引き出すことを可能にする。溶出を確認するために、ピペッターの内容物を顕微鏡下で7cmのペトリ皿に戻すことができます。
注:単一の回転楕円体の重力溶出は、通常、回転楕円体のサイズ/密度に応じて15〜30秒かかります。この間、ピペッターは取り外さないでください。バックグラウンド補正ウェルにはスフェロイドがなく、アッセイ媒体のみを含む必要があります。顕微鏡下で、各回転楕円体の位置を確認する。各回転楕円体は、理想的には各ウェルの中心内に配置する必要があります。 - すべてのスフェロイドがスフェロイドアッセイマイクロプレートに移されたら、アッセイの前にプレートを37°Cの非CO2 インキュベーターに最低1時間移します。
- 広いオリフィスピペットチップを取り付けたシングルチャンネルピペッターの容量を20μLに設定し、単一のスフェロイドを慎重に吸引します。先端をスフェロイドアッセイマイクロプレートの各ウェルの中央に直接置き、重力が単一のスフェロイドを各ウェルの中心に溶出させる、すなわち、ピペットチップから媒体を排出せず、毛細管現象がピペットチップからスフェロイドを引き出すことを可能にする。溶出を確認するために、ピペッターの内容物を顕微鏡下で7cmのペトリ皿に戻すことができます。
- 解剖顕微鏡およびライトボックス装置を用いて、以下に詳述するように、スフェロイドをシャーレからスフェロイドアッセイマイクロプレートに移す。
3. XFアッセイ用のセンサーカートリッジへの化合物の調製と装填
| インジェクション戦略 | コンパウンド(ポート) | XFe96 マイクロウェル開始容量 (μL) | 所望の最終井戸濃度 | ポート容量(μL) | 最終XFe96マイクロウェル容量ポストインジェクション(μL) | 作業在庫の集中 |
| 1 | オリゴマイシン (A) | 180 | 3 ug/mL | 20 | 200 | 30 μg/mL |
| ロテノン (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| アンチマイシンA(B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| ロテノン (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| アンチマイシンA(B) | 200 | 2 μM | 20 | 220 | 22 μM |
表2:XFe96アナライザーを使用して単一3Dスフェロイドのミトコンドリアエネルギー代謝を調べるためのミトコンドリア化合物濃度。
- 完全に補充された、予め温められたXF RPMIアッセイ培地を用いて、 表2 に記すように各化合物の作業ストック濃度を調製する。
- カートリッジプレート(ユーティリティプレートに結合)を列方向に左から右に1~12方向に向けます。
- ローディングガイドを使用する場合は、ウェルローディング手順に従ってカートリッジプレートの上に置きます(たとえば、ポートAを最初にロードする場合は、ガイドの左上隅に A が見えるようにします)。
- 各化合物の作用溶液を適切なリザーバに移し、較正されたP100マルチチャネルピペットを使用して、対応するすべてのポートに20μLを分配する。残りのポートへの各コンパウンドについて、この手順を繰り返します。
メモ:センサーカートリッジプレートに使用されていないポートがある場合は、これらのポートを空のままにするか、アッセイ媒体で満たすことができます。特定のポートレターの選択のみが使用されている場合は、そのレターに対応する他のポートにアッセイ媒体がロードされていることを確認してください。さもなければ、空気は井戸に注入され、それらの井戸の結果を損なうでしょう。 - ポート・ロード後、プレート・ローディング・ガイド (使用されている場合) を取り外し、センサー・カートリッジをロードするためのアナライザーを準備します。
メモ:ポートをロードした直後にアッセイが実行されない場合は、センサーカートリッジに蓋を戻し、マシンにロードできる状態になるまでプレートを37°Cのエアインキュベーターに戻します。
4. アッセイ設計、注入戦略、データ取得
- アッセイの実行
- アナライザの電源を入れ、コントローラ(コンピュータ)に接続します。
注:これは、Wave Controllerソフトウェアのウィジェットパネルの計測器接続ステータスによって確認できます。 - WAVE ソフトウェアのテンプレートページに移動し、実験のアッセイ テンプレート ファイルを見つけてダブルクリックして開きます。
メモ: アッセイテンプレートが [ テンプレート] ビューに表示されない場合は、共有ネットワークドライブまたは USB フラッシュドライブからテンプレートファイルをテンプレートフォルダにインポートします。 - アッセイを開始するには、[ アッセイの実行] タブをクリックします。
注: プレートマップ内でグループ定義が正しく割り当てられている場合、ページの右側にある緑色のチェックマークで示すようにアッセイを実行する準備が整います。この段階では、アッセイサマリーページまたは空白のままのページに追加情報を入力できます。次の手順に進みます。3D微小組織スフェロイドにおけるミトコンドリアモジュレーターの浸透遅延(図2)のために、 表3に記載の測定プロトコル情報を使用する。
- アナライザの電源を入れ、コントローラ(コンピュータ)に接続します。
| 測定期間 | インジェクション番号とポート | 測定の詳細 | 期間 (h:min:s) |
| キャリブレーション | 該当なし | XFアナライザは常にこのキャリブレーションを実行して、測定値が正確であることを確認します | 00:20:00 (これは平均であり、マシンによって異なる場合があります) |
| エクイリブレーション | 該当なし | 平衡化はキャリブレーション後に行われ、推奨されます。 | 00:10:00 |
| 基底 | 該当なし | サイクル = 5 | 00:30:00 |
| ミックス = 3:00 | |||
| 待機 = 0:00 | |||
| メジャー = 3:00 | |||
| オリゴマイシン / BAM15 | インジェクション 1(ポート A) | サイクル = 10 | 01:00:00 |
| ミックス = 3:00 | |||
| 待機 = 0:00 | |||
| メジャー = 3:00 | |||
| ロテノン+アンチマイシンA | インジェクション 2(ポート B) | サイクル = 10 | 01:00:00 |
| ミックス = 3:00 | |||
| 待機 = 0:00 | |||
| メジャー = 3:00 | |||
| 合計時間: | 03:00:00 | ||
表3:XFe96アナライザーを使用して、単一の3Dスフェロイドのミトコンドリアエネルギー代謝をプローブするためのプロトコル設定。
- [ 実行の開始 ] をクリックして、[ 保存場所 ] ダイアログ ボックスを表示します。
- 結果ファイルの 保存場所 を入力し、組み立てたセンサーカートリッジを分析器側面のドアから表示されるサーマルトレイに置きます。サーマルトレイが自動的に開き、画面に キャリブラントユーティリティプレートのロード メッセージが表示されるまで待ちます。画面の指示に従う前に、i) センサーカートリッジがユーティリティー・プレートに正しくフィットしていること、ii) センサー・カートリッジから蓋が取り外されていること、および iii) センサー・カートリッジがユーティリティー・プレート上のセンサー・カートリッジの向きが正しいことを確認してください。
- 画面上のコマンドに従って、センサーカートリッジのキャリブレーションを開始します。
メモ:キャリブレーションの完了にかかる時間は約10~20分です(37°Cでのアッセイの場合)。 - センサーカートリッジのキャリブレーション後、Wave Controllerの画面上の指示に従ってスフェロイドマイクロプレートをアナライザーにロードし、12分間の平衡化ステップを開始します。
注:白いチェックマークの付いた緑色のボックスは、その井戸の「良い」キャリブレーションを示します。いずれかの井戸が「良い」校正を提供しない場合、それらは赤い箱と白い十字で示されます。このようなウェルは、 修飾 アッセイタブを使用してアッセイが完了した後に、いかなる分析からも除外されるべきであることに留意されたい。 - 機械が平衡化ステップを完了した後、分析器が自動的にベースライン測定値の取得を開始するのを待ちます(機器プロトコルで概説されているように)。
- 実験を完了するには、WAVE コントローラーの画面上のコマンドに従います。
メモ:スフェロイドマイクロプレートをアナライザーから取り外したら、センサーカートリッジを廃棄し、必要に応じてさらに分析(例えば、二本鎖(ds)DNA定量)のためにスフェロイドプレートを脇に置いておきます。マイクロプレートがさらなる分析に必要ない場合は、センサーカートリッジと一緒に廃棄することができます。 - アッセイダイアログが表示されるのを待って結果を表示するか、 テンプレート ビューに戻ります。
5. データ正規化および分析戦略 - ポストアッセイ正規化および下流アッセイ(オプションステップ)
- データの正規化
- スフェロイドデータを正規化するには、スフェロイドサイズと体積を計算し、スフェロイドアッセイでdsDNAを定量するためのデータ正規化戦略に関連する一連のプロトコルを参照してください。これらは補足ファイルとして含まれています。 補足ファイル 1 および 補足ファイル 2 を参照してください。
- データ解析
- 自動分析ジェネレーターの 1 つにデータをエクスポートするには、WAVE コントローラーのデータエクスポートコマンドに従い、アッセイタイプに一致するエクスポートジェネレーターを選択します。または、データファイルをエクスポートして、Seahorse 分析にアップロードします。
注: レポートジェネレーターとタツノオトシゴ分析の欠点は、データ分析がXFアッセイの設計方法に限定され、測定サイクル全体で平均を取得できないことです。計測器ソフトウェアからデータセットを手動でエクスポートすると、この点でユーザの好みが可能になります。3Dスフェロイドのミトコンドリア呼吸を評価するための注入戦略が典型的な「MitoStress」テストのそれとは異なる可能性が高いことを考えると、3D細胞培養に固有のこれらのデータセットを分析するのに役立つ一連のスプレッドシートテンプレートが開発されており、要求に応じて提供されます。これらのデータテンプレートファイルは、 図2で詳細かつ説明されている主要なミトコンドリア呼吸パラメータに関するデータを提供します。 - データを分析するには、WAVEコントローラソフトウェアからデータをスプレッドシートレポートとしてエクスポートし、独立したスプレッドシートテンプレートを使用して分析します。
- 自動分析ジェネレーターの 1 つにデータをエクスポートするには、WAVE コントローラーのデータエクスポートコマンドに従い、アッセイタイプに一致するエクスポートジェネレーターを選択します。または、データファイルをエクスポートして、Seahorse 分析にアップロードします。
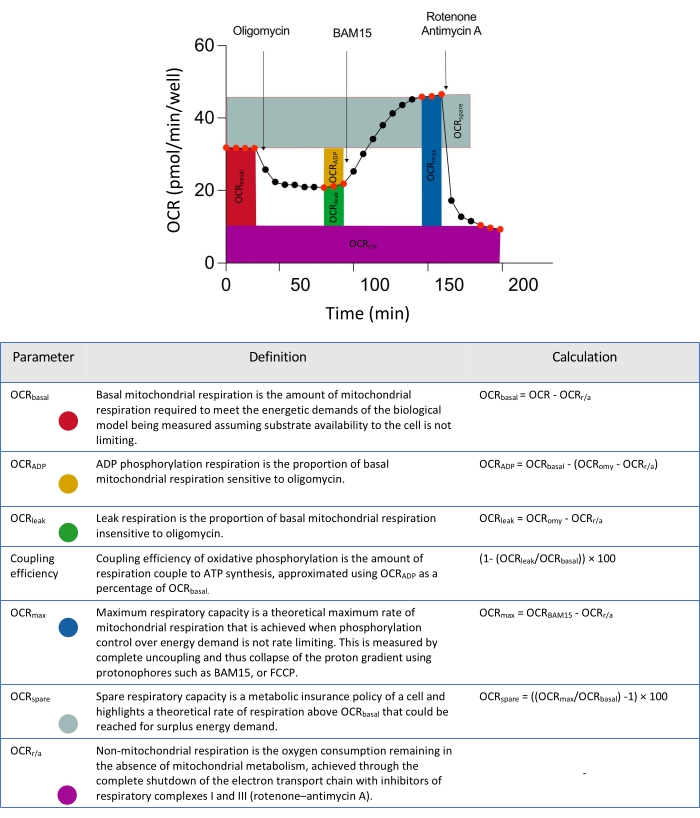
図2:細胞外フラックスデータ解析から導出されたパラメータの概略記述子。 略語: OCR = 酸素消費率。 この図の拡大版を表示するには、ここをクリックしてください。
Access restricted. Please log in or start a trial to view this content.
結果
十分に形成されたコンパクトなスフェロイドを得るために、各細胞株を播種密度および培養期間について個別に最適化した(図3)。A549、HepG2/C3A、およびSK-OV-3細胞株は、最初に緩い凝集体を形成し、培養7日後まで周囲が明確に定義された丸いスフェロイドに進行しなかった。逆に、MCF-7細胞は3日以内にスフェロイドを形成することができた。すべてのスフェロイドモデル?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
主な調査結果と成果
この論文では、XFe96 XF Analyzerを搭載した一連のがん由来細胞株を使用して、単一の3Dスフェロイドのミトコンドリアエネルギー代謝を調べるための詳細なプロトコルを提供します。強制凝集のための細胞忌避技術を用いて、A549、HepG2/C3A、MCF7、およびSK-OV-3細胞スフェロイドの迅速な培養のための方法が開発され、記述されている。このプロトコルは、(1)スフ...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は、宣言する利益相反を持っていません。
謝辞
N.J.C.は、Sygnature Discovery Ltd (BB/M01116X/1, 1940003) と共に BBSRC MIBTP CASE Award の支援を受けました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
参考文献
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
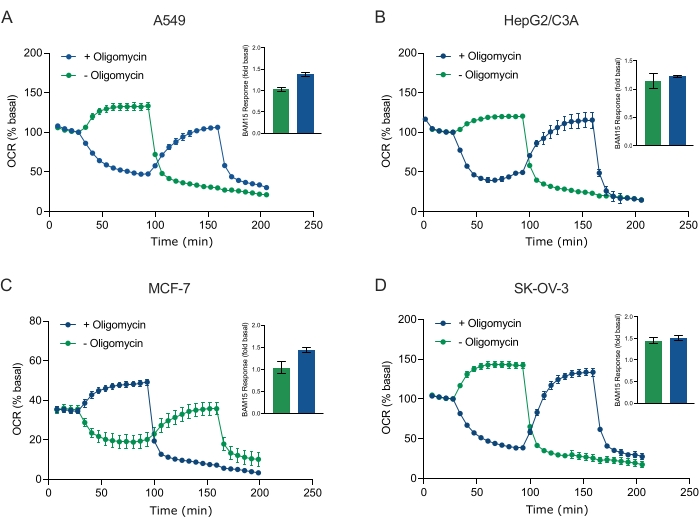
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
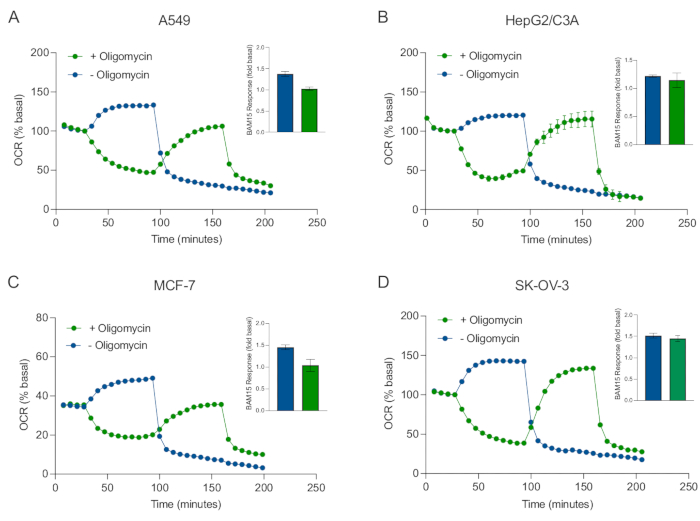
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved