Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Identificación de la actividad hemolítica y fosfolipasa en extractos crudos de anémonas de mar mediante bioensayos sencillos
En este artículo
Resumen
Aquí, describimos un protocolo para obtener extracto de veneno crudo de la anémona de mar y detectar su actividad hemolítica y fosfolipasa.
Resumen
La composición del veneno de anémona de mar incluye moléculas polipeptídicas y no proteicas. Los componentes citolíticos tienen un alto potencial biotecnológico y biomédico para diseñar nuevas herramientas moleculares. El veneno de anémona de mar se localiza en las células glandulares del ectodermo y las estructuras subcelulares llamadas nematocistos, las cuales se distribuyen por todo el cuerpo de la anémona de mar. Esta característica implica desafíos porque las células y el nematocisto deben ser lisados para liberar los componentes del veneno con otras moléculas no tóxicas. Por lo tanto, primero, el veneno se deriva de un extracto crudo (mezcla de moléculas diferentes y diversas y restos de tejido). El siguiente paso es detectar polipéptidos con bioactividades específicas. Aquí, describimos una estrategia eficiente para obtener el extracto crudo de anémona de mar y bioensayo para identificar la presencia de citolisinas. El primer paso implica técnicas económicas y sencillas (ciclo agitado y de congelación-descongelación) para liberar citolisinas. Obtuvimos la mayor actividad citolítica y proteína (~500 mg de proteína de 20 g de peso seco). A continuación, se analizó la complejidad polipeptídica del extracto mediante el gel SDS-PAGE detectando proteínas con pesos moleculares entre 10 kDa y 250 kDa. En el ensayo hemolítico, utilizamos glóbulos rojos de oveja y determinamos HU50 (11,1 ± 0,3 μg/mL). Por el contrario, la presencia de fosfolipasas en el extracto crudo se determinó utilizando yema de huevo como sustrato en un medio sólido con agarosa. En general, este estudio utiliza un protocolo eficiente y económico para preparar el extracto crudo y aplica bioensayos replicables para identificar citolisinas, moléculas con intereses biotecnológicos y biomédicos.
Introducción
Los animales marinos son una rica fuente de compuestos biológicamente activos. En las últimas décadas, la composición del veneno de anémona de mar ha atraído la atención científica ya que comprende una diversidad de polipéptidos con actividad hemolítica, citotóxica, enzimática (fosfolipasa, proteasa, quitinasa) y neurotóxica y efectos inhibitorios sobre la actividad proteolítica1. Además, estos polipéptidos son fuentes potenciales para el desarrollo de herramientas moleculares en uso biotecnológico y terapéutico 2,3.
Hay pocos informes sobre el veneno de anémona de mar y sus componentes moleculares debido a la complejidad de obtener el veneno, incluso el aislamiento y la caracterización de toxinas. Los métodos de extracción utilizados en los informes implicaron la lisis y el vaciado del contenido de las células que están relacionadas y no relacionadas con la producción de veneno1.
Una característica particular en todos los cnidarios es la ausencia de un sistema para la producción y liberación del veneno centralizado en una sola región anatómica. En cambio, los nematocistos son estructuras que mantienen el veneno 4,5. Otros tipos de células, llamadas células de la glándula epidérmica, también secretan toxinas y también se distribuyen por todo el cuerpo de las anémonas de mar6.
El primer y más crucial reto en la obtención del veneno es la generación de un extracto con suficiente manipulación en procesos posteriores, sin la inactivación o degradación de proteínas lábiles. A continuación, las células deben ser lisadas, y los componentes, en este caso, los polipéptidos deben extraerse de manera eficiente y rápida, evitando la proteólisis y la hidrólisis mientras se eliminan otros componentes celulares7.
Se utilizan diferentes métodos para obtener el extracto crudo de una anémona de mar; algunos implican sacrificar el organismo, mientras que otros permiten que se mantenga vivo. Los métodos que implican el uso de todo el cuerpo del organismo permiten la liberación de la mayoría de las toxinas del veneno8, en comparación con los métodos que mantienen vivos a los organismos, que extraen solo algunos componentes del veneno9. La preparación de un extracto requiere evaluar la presencia y potencia de una sustancia de interés a través de un bioensayo específico, que incluye estrategias para observar los efectos farmacológicos por métodos in vivo o in vitro 10.
El veneno de anémona de mar contiene polipéptidos citolíticos, toxinas formadoras de poros (PFT)11 y fosfolipasas12; estas moléculas son modelos en el estudio de la interacción proteína-lípido, herramientas moleculares en la terapia del cáncer y biosensores basados en nanoporos3. La clasificación de los PFT de anémona de mar se realiza según su tamaño o peso molecular, de 5 kDa a 80 kDa. El PFT de 20 kDa, el más estudiado y conocido como actinoporinas11, es de particular interés por su potencial biomédico en el desarrollo de herramientas moleculares para posibles aplicaciones como biosensores anticancerígenos, antimicrobianos y basados en nanoporos. Otra citolisina, incluidas las fosfolipasas, específicamente la fosfolipasa A2 (PLA2)13, libera un ácido graso debido e hidroliza los fosfolípidos, desestabilizando la membrana celular. Debido a este mecanismo de acción, PLA2 promete ser un modelo esencial para el estudio y las aplicaciones en enfermedades inflamatorias. Podría servir como modelo para estudios del comportamiento lipídico en la membrana celular14.
Aquí, describimos un protocolo eficiente para obtener el extracto crudo de anémona de mar Anthopleura dowii Verrill, 1869, y detectar hemolisinas y fosfolipasas. Ambas son toxinas relevantes que podrían usarse como plantilla para diseñar nuevas herramientas moleculares.
Protocolo
Las anémonas de mar fueron recolectadas de acuerdo a lineamientos de la Comisión Nacional de Acuacultura, Pesca y Alimentación del Gobierno Federal de México (número de permiso PPF / DGOPTA 07332.250810.4060). El Comité de Bioética del Instituto de Biotecnología de la Universidad Nacional Autónoma de México aprobó todos los experimentos con anémonas de mar. La muestra de sangre ovina fue adquirida en el Centro de Enseñanza Práctica e Investigación en Producción y Sanidad Animal (CEPIPSA, Universidad Nacional Autónoma de México).
1. Colección de organismos
- Recoger anémona de mar A. dowii.
NOTA: Aquí, los organismos fueron recolectados durante los períodos de marea baja en la zona intermareal frente a la costa de Ensenada Baja, California, México (Figura 1A, B). - Transportar los organismos al laboratorio en contenedores con agua de mar (Figura 1C).
- En el laboratorio, limpie los organismos con agua destilada a mano para eliminar las grandes partículas de sustrato que se adhieren al cuerpo.
- Congelar inmediatamente los organismos a -80 °C en un ultracongelador durante 72 h.
- Coloque los organismos en vasos especiales para liofilización, con condiciones de liofilización de -20 ° C, 0.015 psi, durante 48 h.
- Conservar los organismos liofilizados a -20 °C hasta su uso.
2. Hidratación tisular
- Reconstituir 20 g de peso seco de organismos liofilizados con 60 ml (1/3 p/v) de tampón de fosfato de sodio de 50 mM, pH 7.5 y 1 tableta de cóctel inhibidor (Tabla de materiales).
- En un agitador magnético, revuelva continuamente la muestra a ~800 rpm, 4 °C, durante 12 h.
3. Liberación de toxinas
- Para inducir la lisis celular y la descarga de nematocistos, congele la muestra a -20 °C durante 12 h y luego coloque el recipiente en agua a temperatura ambiente (RT) hasta que se descongele hasta un 90%. Repita este procedimiento tres veces.
NOTA: La muestra no se descongela al 100%; es crucial mantener la muestra a ~4 °C para evitar la degradación de proteínas. - Observe el nematocisto descargado bajo un microscopio confocal. Tome 10 μL de la muestra en un portaobjetos y coloque un cubrehojas sobre él. Observe bajo un microscopio confocal a un aumento de 100x y 60x. Aquí no era necesario un tinte.
- Procesar las imágenes capturadas en el microscopio confocal en el software ImageJ (versión 1.53c, Wayne Rasband, National Institutes of Health, USA, https://imagej.nih.gov/ij).
- Si el túbulo dentro del nematocisto se desenrolla y está afuera, los nematocistos se descargan; luego, continúe con el siguiente paso. De lo contrario, solo realice un ciclo más de congelación-descongelación.
- Para clarificar el extracto, centrifugar la muestra a 25.400 x g durante 40 min, 4 °C, recuperar el sobrenadante y centrifugarlo de nuevo. Repita este paso 3-4 veces.
- Recupera el sobrenadante y desecha el pellet. El sobrenadante o extracto crudo contiene los componentes del veneno (toxinas) y otras moléculas celulares, como lípidos, carbohidratos y ácidos nucleicos.
4. Cuantificación de la proteína total
- Determinar la concentración de proteínas del extracto crudo utilizando un kit comercial de ensayo colorimétrico Bradford15 (Tabla de Materiales).
- Diluya el reactivo de colorante (una parte de tinte con cuatro partes de agua destilada).
- Preparar la solución de albúmina sérica bovina (BSA) Fracción V (Tabla de Materiales) a una concentración de 1 mg/ml en tampón de fosfato (50 mM de fosfato de sodio, pH 7.5).
- Prepare las soluciones enumeradas en la Tabla 1 por triplicado.
- Mezcle vigorosamente cada solución en un agitador durante 4 s.
- Incubar las muestras durante 5 min en RT, sin agitar.
- Medir la absorbancia (UA) a 595 nm.
- Trazar los promedios de las absorbancias BSA de la muestra (AU vs. concentración de proteínas), y determinar la ecuación del gráfico (Ecuación 1).
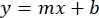 Ecuación 1
Ecuación 1
donde y es la absorbancia, m es la pendiente de la línea, x es la concentración de proteína y b es la intersección y. Para calcular la concentración de extracto crudo, resuelva para la x de la siguiente manera: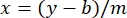 Ecuación 2
Ecuación 2
5. Determinar la complejidad del veneno polipeptídico
- Ensamble el recipiente de vidrio para la electroforesis vertical en gel de acuerdo con el manual.
- Preparar mezcla de acrilamida (30%): 29,2 g de acrilamida, 0,8 g de bis-acrilamida, el volumen final de 100 mL. Filtre la solución utilizando una membrana de 0,45 μm. Guarde la solución en un frasco oscuro a 4 °C. Luego, prepare la mezcla para el gel de resolución (Tabla 2).
- El gel SDS-PAGE consiste en un gel de resolución y un gel de apilamiento. Preparar los geles según los volúmenes de solución definidos en la Tabla 2. Durante la preparación, asegúrese de mantener cada mezcla a 4 °C para reducir el tiempo de polimerización.
NOTA: El volumen de cada mezcla varía dependiendo de la marca del equipo utilizado; para este protocolo, los volúmenes corresponden a la preparación de gel de acrilamida al 15%, de 0,75 mm de espesor, para una cámara de electroforesis vertical (Tabla de Materiales). - Después de que la acrilamida se haya polimerizado, ~ 10 min, agregue la mezcla de gel de apilamiento.
- Prepare la mezcla para el gel de apilamiento, agréguela inmediatamente a las placas de vidrio y coloque un peine para formar los pozos para cargar las muestras.
- Una vez que el gel de apilamiento se haya polimerizado (~ 15 min), retire el peine y coloque el recipiente de vidrio en una cámara para electroforesis vertical.
- Coloque 200 ml de tampón de electrodo (0,25 M Tris, 0,192 M de glicina, 0,1% SDS) en la cámara de electroforesis.
- Analizar el extracto crudo: Mezclar 30 μg de extracto crudo con 5 μL de tampón de carga de proteínas (25% glicerol, 15% dodecil sulfato de sodio, 25% 2-mercaptoetanol, 0.125 mM Tris pH 7, bromofenol azul 0.1 mg/mL) para desnaturalizar proteínas. El volumen final debe ser de <50 μL.
- Calentar a 90 °C durante 5 min, enfriar y centrifugar durante 3 s a 1.400 x g.
- Cargue las muestras en los pozos del gel de apilamiento. Cargue una escalera de peso molecular estándar.
- Ejecutar electroforesis a 25 mA constante durante 1 h 30 min.
- Retire los recipientes de vidrio de la cámara de electroforesis para proceder a la tinción de las proteínas del gel.
6. Tinción de proteínas
- Enjuague el gel en 50 ml de agua destilada para eliminar los desechos del electrodo tampón.
- Deseche el agua.
- Para evitar la difusión de las proteínas del gel, agregue la solución de fijación (60 ml de etanol, 20 ml de ácido acético y 20 ml de agua destilada, volumen final 100 ml). Incubar a RT durante 40 min con agitación a 80 rpm. Decantar la solución.
- Añadir la solución de reticulación de proteínas (15 mL de etanol, 0,25 mL de glutaraldehído, 50 mL de agua destilada, volumen final 65,25 mL), e incubar durante 30 min en RT con agitación a 80 rpm. Retire la solución.
- Lave el gel con 100 ml de agua destilada durante 5 minutos y retire el agua. Repita este procedimiento cuatro veces.
- Realizar tinción de proteínas con 50 mL de solución filtrada Coomassie azul brillante R-250 (40% metanol, 10% ácido acético, 50% agua destilada, 0.1% colorante, volumen final 100 mL), agitando a 60 rpm en un oscilador rotativo de laboratorio a RT durante 15 min.
- Recupere la solución, que se puede utilizar para manchar otros geles.
- Para eliminar el exceso de colorante, agregue una solución destintiva (40 ml de metanol, 10 ml de ácido acético, 50 ml de agua destilada). Mantenga la agitación (80 rpm) en RT hasta que las bandas (proteínas teñidas) se visualicen.
- Mantenga el gel en 50 ml de agua destilada y escanee la imagen en un sistema de documentación de gel.
7. Preparar solución de glóbulos rojos
- Extraer 1 ml de sangre de la vena yugular de una oveja.
- Retire la aguja de la jeringa y drene inmediatamente la sangre en un tubo con 50 ml de solución de Alsever (0,002 M de ácido cítrico, 0,07 M de NaCl, 0,1 M de dextrosa y 0,027 M de citrato de sodio como anticoagulante, pH 7,4).
- Invierta lentamente la solución tres veces.
- Conservar a 4 °C para su traslado al laboratorio.
- Centrifugar a 804 x g durante 5 min a 4 °C y desechar el sobrenadante.
- Resuspend el pellet en 30 ml de solución de Alsever. Agregue la solución deslizándola a lo largo de las paredes internas del tubo y, utilizando una pipeta de plástico Pasteur, resuspenda lentamente las células. Centrifugar de nuevo a 804 x g durante 5 min, 4 °C. Repita este procedimiento hasta que se aclare el sobrenadante.
- Añadir 15 ml de solución de Alsever; resuspend el pellet lentamente con una pipeta de plástico Pasteur.
- Mantener el volumen final de la solución de Alsever para lograr una concentración de 2 x 106 células/ml, aproximadamente. Luego, use una cámara neubauer o un hemocitómetro para contar las células.
NOTA: Cuente los eritrocitos en la diapositiva del hemacitómetro (Neubauer mejorado). La corredera es una corredera de 30 mm x 70 mm x 4 mm. El centro tiene dos estribos plateados (Figura 2A) (una rejilla superior y otra inferior), cada una de 3 mm x 3 mm, y dos canales a los lados de la rejilla. El conteo de celdas está en los cuadrados de esquina y centro (cuadrados rojos en la Figura 2B). La concentración óptima de células para contar debe ser del orden de 106 células. La caja central tiene una superficie de 0,1 cm2, lo que equivale a 0,0001 ml. - Coloque un cubrebocas sobre la diapositiva.
- Coloque 10 μL de la muestra entre la diapositiva y el cubrebocas y espere a que la muestra se desembolse.
- En un microscopio óptico (aumento 60x), realice el recuento en la primera caja cuadrada. Cuente solo las celdas de la cuadrícula central y las que tocan el margen superior o izquierdo del cuadro.
- Determine la concentración celular utilizando la siguiente ecuación:
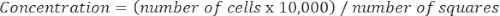 Ecuación 3
Ecuación 3
Si la muestra está muy concentrada y se requiere dilución, use la siguiente ecuación: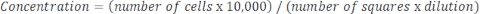 Ecuación 4
Ecuación 4
NOTA: Al tomar la solución de eritrocitos para la prueba de hemólisis, homogeneice lentamente la solución con una pipeta pasteur de plástico. - Realice los pasos por triplicado para reducir el error.
8. Ensayo de hemólisis
- Preparar las mezclas de solución (por triplicados) indicadas en la Tabla 3 en placas cónicas de 96 pocillos.
NOTA: Mezclar los eritrocitos lentamente para mantener una suspensión homogénea. - Incubar durante 1 h a 37 °C, sin agitar.
- Centrifugar la placa a 804 x g durante 5 min a 4 °C.
- Recupere el sobrenadante en otra placa de 96 pocillos con fondo plano.
- Si el extracto crudo contiene citolisinas, los eritrocitos lisarán y liberarán hemoglobina. Lea la absorbancia a 415 nm.
- Calcule el porcentaje de hemólisis, utilizando la siguiente ecuación:
% Hemólisis = ((Extracto de acróduro-tampón AAlsever) / (AH2O-AAlsever)) x 100
NOTA: El extracto de acródura, el tampón AAlsever y AH2O corresponden a la absorbancia de eritrocitos con el extracto crudo, la absorbancia de la solución de Alsever y la absorbancia del agua destilada, respectivamente. - Realice una prueba de hemólisis antes y después de cada ciclo de congelación y descongelación con 50 μg de proteína total del extracto crudo.
- Determinar el número de ciclos de congelación y descongelación necesarios para alcanzar el 100% de actividad hemolítica.
- Calcular la cantidad de extracto que produce hemólisis en el 50% de los eritrocitos (HU50); seguir el mismo protocolo descrito en la Tabla 3, aumentar la cantidad de extracto crudo de anémona de mar y diseñar la parcela con ajustes sigmoidales utilizando el software apropiado (por ejemplo, Origen, Tabla de Materiales).
9. Ensayo de fosfolipasa
- Lave un huevo de gallina con 1% de SDS en agua destilada.
- Separe la yema de huevo de la clara de huevo en condiciones estériles.
- Prepare 50 ml de solución de NaCl al 0,86% y filtre a través de un filtro de 0,22 μm. Luego, prepare la Solución A: Agregue 12 ml de yema de huevo y 36 ml de solución de NaCl al 0.86%.
- Preparar la Solución B: Mezclar 300 mg de agarosa en 50 mL de tampón (50 mM Tris, pH 7.5), filtrar a través de un filtro de 0.22 μm. Calentar la solución en un microondas hasta que hierva. Enfríe en agua tibia para alcanzar los 43-45 °C.
- Prepare la solución C: Prepare 10 mM CaCl2 y filtre con un filtro de 0,22 μm.
- Preparar la Solución D: 10 mg de rodamina 6G en 1 mL de agua destilada.
- En condiciones estériles (campana de flujo laminar), añadir 500 μL de soluciones A y C a la solución B y 100 μL de solución D, mezclar y verter 25 mL en cada placa de Petri (90 x 15 mm).
- Espere a que la solución se solidifique en condiciones estériles (30 min).
- Haga pocillos (~ 2-3 mm de diámetro) con un tubo delgado.
- Añadir un total de 20 μL de tampón de fosfato (control negativo) en un pocillo y 20 μL de una fosfolipasa determinada (control positivo) en otro pozo.
- Coloque diferentes cantidades de la proteína del extracto crudo, 5, 15, 25, 35, 45 μg, en los pocillos restantes, cada uno en un volumen final de 20 μL.
- Espere a que el agar adsorba todas las muestras (30 min).
- Incubar a 37 °C durante 20 h.
- Si se forma un halo alrededor del pozo, esto indica actividad fosfolipasa. Mide el halo formado con una pinza vernier.
NOTA: Realice el experimento por triplicado (pasos de 9.1 a 9.14).
Resultados
Los resultados representativos del protocolo utilizado para obtener el extracto crudo de anémona de mar mostraron que la combinación de dos técnicas (agitación y ciclos de congelación y descongelación) produjo una descarga eficiente de nematocistos, y la cantidad total de proteína fue de 500 mg (8 mg/ml) (Figura 3).
La complejidad proteica del extracto crudo se pudo observar a partir de 10 kDa y superior a 250 kDa a través de la electroforesis SDS-PAGE. Ad...
Discusión
La alta demanda de nuevos compuestos con aplicaciones en diferentes campos de la ciencia y la industria ha llevado al estudio del veneno. El veneno representa una rica fuente de moléculas que sirve como plantilla para generar nuevas herramientas moleculares. Sin embargo, la complejidad de estos venenos requiere la implementación y combinación de varios métodos para obtenerlos y estudiarlos.
Aquí, mostramos un método para obtener y analizar el veneno de la anémona de mar Anthopleura ...
Divulgaciones
Los autores declaran que no tienen ningún interés en competencia en relación con la publicación de este artículo.
Agradecimientos
Este trabajo fue apoyado por el Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT), con número de subvención IT200819. Los autores agradecen a Tom Musselman, Rock Paper Editing, LLC, por revisar la gramática inglesa de este manuscrito; y la asistencia técnica de Samanta Jiménez (CICESE, Ensenada), y Juan Manuel Barbosa Castillo (Instituto de Fisiología Celular, UNAM). También agradecemos al Dr. Augusto César Lizarazo Chaparro (CEPIPSA) por obtener sangre de oveja. Agradecemos especialmente al Dr. José Saniger Blesa, ICAT-UNAM, por las instalaciones de su laboratorio para la grabación de video.
Materiales
| Name | Company | Catalog Number | Comments |
| 15 mL conical centrifuge tube | Corning | 430766 | |
| 2-Bromophenol blue | Sigma | B75808 | |
| 2-mercaptoetanol | Sigma-Aldrich | M6250-100ML | |
| 50 mL conical centrifuge tubes | Corning | 430828 | |
| Acetic Acid Glacial | J.T. Baker | 9515-03 | |
| Acrylamide | Promega | V3115 | |
| Agarose | Promega | V3125 | |
| Bisacrylamide | Promega | V3143 | |
| Bovine Serum Albumin Fraction V | Sigma | A3059-100G | |
| Bradford Protein Assays | Bio-Rad | 5000006 | |
| Calcium chloride | Sigma-Aldrich | C3306 | |
| Cell culture plates 96 well, V-bottom | Corning | 3894 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge tubes | Corning | CLS430829 | |
| ChemiDoc MP system | Bio-Rad | 1708280 | |
| Citric acid | Sigma-Aldrich | 251275 | |
| Clear flat.bottom 96-Well Plates | Thermo Scientific | 3855 | |
| Coomassie Brilliant Blue G-250 | Bio-Rad | #1610406 | |
| Coomassie brilliant blue R-250 | Bio-Rad | 1610400 | |
| Dextrose | J.T. Baker | 1916-01 | |
| Ductless Enclosure | Labconco | Vertical | https://imagej.nih.gov/ij ImageJ 1.53c |
| Gel Doc EZ | Bio Rad. | Gel Documentation System | |
| Glycerol | Sigma-Aldrich | G5516-4L | |
| Hemocytometer | Marienfeld | 650030 | |
| ImageJ (Software) | NIH, USA | Version 1.53c | |
| Incubator 211 | Labnet | I5211 DS | |
| Methanol | J.T. Baker | 9049-03 | |
| Mini-PROTEAN tetra cell | Bio-Rad | 1658000EDU | |
| Na2HPO4 | J.T. Baker | 3824-01 | |
| NaCl | J.T. Baker | 3624-01 | |
| NaH2PO4.H2O | J.T. Baker | 3818-05 | |
| Origin software | version 9 | To design the plot with sigmoidal adjustments | |
| Petridish | Falcon | 351007 | |
| Pipetman kit | Gilson | F167380 | |
| Precast mini gel | BioRad | 1658004 | |
| Prestained Protein Ladder | Thermo Scientific | 26620 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protein Assay Dye Reagent Concentrate | Bio-Rad | 5000006 | |
| Rhodamine 6G | Sigma-Aldrich | 252433 | |
| SDS | Sigma-Aldrich | L4509 | |
| Sodium citrate dihydrate | JT Baker | 3646-01 | |
| Spectrophotometer | THERMO SCIENTIFIC | G10S UV-VIS | |
| Tris Base | Sigma-Aldrich | 77-86-1 | |
| Volt Power Supply | Hoefer | PS300B |
Referencias
- Frazão, B., Vasconcelos, V., Antunes, A. Sea anemone (Cnidaria, Anthozoa, Actiniaria) toxins: an overview. Marine Drugs. 10 (8), 1812-1851 (2012).
- Jayathilake, J. M. N. J., Gunathilake, K. V. K. Cnidarian toxins: recent evidences for potential therapeutic uses. The European Zoological Journal. 87 (1), 708-713 (2020).
- Ramírez-Carreto, S., Miranda-Zaragoza, B., Rodríguez-Almazán, C. Actinoporins: From the structure and function to the generation of biotechnological and therapeutic tools. Biomolecules. 10 (4), 539 (2020).
- Fautin, D. G. Structural diversity, systematics, and evolution of cnidae. Toxicon. Official Journal of the International Society on Toxinology. 54 (8), 1054-1064 (2009).
- Moran, Y., et al. Analysis of soluble protein contents from the nematocysts of a model sea anemone sheds light on venom evolution. Marine Biotechnology. 15 (3), 329-339 (2013).
- Moran, Y., et al. Neurotoxin localization to ectodermal gland cells uncovers an alternative mechanism of venom delivery in sea anemones. Proceedings of the Royal Society B: Biological Sciences. 279 (1732), 1351-1358 (2012).
- Grabski, A. C. Advances in preparation of biological extracts for protein purification. Methods in Enzymology. 463, 285-303 (2009).
- Bulati, M., et al. Partially purified extracts of sea anemone Anemonia viridis affect the growth and viability of selected tumour cell lines. BioMed Research International. 2016, 3849897 (2016).
- Orts, D. J. B., et al. Biochemical and electrophysiological characterization of two sea anemone type 1 potassium toxins from a geographically distant population of Bunodosoma caissarum. Marine Drugs. 11 (3), 655-679 (2013).
- Hader, D., Erzinger, G. . Bioassays: Advanced Methods and Applications. , (2017).
- Anderluh, G., Macek, P. Cytolytic peptide and protein toxins from sea anemones (Anthozoa: Actiniaria). Toxicon: Official Journal of the International Society on Toxinology. 40 (2), 111-124 (2002).
- Nevalainen, T. J., et al. Phospholipase A2 in cnidaria. Comparative Biochemistry and Physiology. Part B, Biochemistry & Molecular Biology. 139 (4), 731-735 (2004).
- Razpotnik, A., et al. A new phospholipase A2 isolated from the sea anemone Urticina crassicornis - its primary structure and phylogenetic classification. The FEBS Journal. 277 (12), 2641-2653 (2010).
- Dennis, E. A., Cao, J., Hsu, Y. -. H., Magrioti, V., Kokotos, G. Phospholipase A2 enzymes: Physical structure, biological function, disease implication, chemical inhibition, and therapeutic intervention. Chemical Reviews. 111 (10), 6130-6185 (2011).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Kwon, Y. -. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, 8663 (2015).
- Eno, A. E., Konya, R. S., Ibu, J. O. Biological properties of a venom extract from the sea anemone, Bunodosoma cavernata. Toxicon: Official Journal of the International Society on Toxinology. 36 (12), 2013-2020 (1998).
- Morales-Landa, J. L., et al. Antimicrobial, antiprotozoal, and toxic activities of Cnidarian extracts from the Mexican Caribbean Sea. Pharmaceutical Biology. 45 (1), 37-43 (2007).
- Sánchez-Rodríguez, J., Cruz-Vazquez, K. Isolation and biological characterization of neurotoxic compounds from the sea anemone Lebrunia danae (Duchassaing and Michelotti, 1860). Archives of Toxicology. 80 (7), 436-441 (2006).
- de Oliveira, J. S., et al. Caissarolysin I (Bcs I), a new hemolytic toxin from the Brazilian sea anemone Bunodosoma caissarum: purification and biological characterization. Biochimica Et Biophysica Acta. 1760 (3), 453-461 (2006).
- Norton, R. S., et al. Purification and characterisation of proteins with cardiac stimulatory and haemolytic activity from the anemone Actinia tenebrosa. Toxicon: Official Journal of the International Society on Toxinology. 28 (1), 29-41 (1990).
- Gondran, M., Eckeli, A. L., Migues, P. V., Gabilan, N. H., Rodrigues, A. L. S. The crude extract from the sea anemone, Bunodosoma caissarum elicits convulsions in mice: possible involvement of the glutamatergic system. Toxicon: Official Journal of the International Society on Toxinology. 40 (12), 1667-1674 (2002).
- Dion, A. S., Pomenti, A. A. Ammoniacal silver staining of proteins: mechanism of glutaraldehyde enhancement. Analytical Biochemistry. 129 (2), 490-496 (1983).
- Ramírez-Carreto, S., et al. Identification of a pore-forming protein from sea anemone Anthopleura dowii Verrill (1869) venom by mass spectrometry. The Journal of Venomous Animals and Toxins Including Tropical Diseases. 25, 147418 (2019).
- Nelson, G. J. Studies on the lipids of sheep red blood cells. I. Lipid composition in low and high potassium red cells. Lipids. 2 (1), 64-71 (1967).
- Ramírez-Carreto, S., et al. Transcriptomic and proteomic analysis of the tentacles and mucus of Anthopleura dowii Verrill, 1869. Marine Drugs. 17 (8), 436 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados