Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Identification de l’activité hémolytique et phospholipase dans les extraits bruts d’anémones de mer par des essais biologiques simples
Dans cet article
Résumé
Ici, nous décrivons un protocole pour obtenir un extrait de venin brut de l’anémone de mer et détecter son activité hémolytique et phospholipase.
Résumé
La composition du venin d’anémone de mer comprend des molécules de polypeptides et de non-protéines. Les composants cytolytiques ont un potentiel biotechnologique et biomédical élevé pour la conception de nouveaux outils moléculaires. Le venin d’anémone de mer se trouve dans les cellules glandulaires des structures ectodermiques et subcellulaires appelées nématocystes, qui sont toutes deux réparties dans tout le corps de l’anémone de mer. Cette caractéristique implique des défis car les cellules et le nématocyste doivent être lysés pour libérer les composants du venin avec d’autres molécules non toxiques. Par conséquent, tout d’abord, le venin est dérivé d’un extrait brut (mélange de molécules différentes et diverses et de débris tissulaires). L’étape suivante consiste à détecter les polypeptides avec des bioactivités spécifiques. Ici, nous décrivons une stratégie efficace pour obtenir l’extrait brut d’anémone de mer et un essai biologique pour identifier la présence de cytolysines. La première étape implique des techniques simples et peu coûteuses (cycle agité et congélation-décongélation) pour libérer des cytolysines. Nous avons obtenu l’activité cytolytique et les protéines les plus élevées (~ 500 mg de protéines à partir de 20 g de poids sec). Ensuite, la complexité polypeptidique de l’extrait a été analysée par le gel SDS-PAGE détectant des protéines de poids moléculaire compris entre 10 kDa et 250 kDa. Dans le test hémolytique, nous avons utilisé des globules rouges de mouton et déterminé HU50 (11,1 ± 0,3 μg / mL). En revanche, la présence de phospholipases dans l’extrait brut a été déterminée en utilisant du jaune d’œuf comme substrat dans un milieu solide avec de l’agarose. Dans l’ensemble, cette étude utilise un protocole efficace et peu coûteux pour préparer l’extrait brut et applique des essais biologiques reproductibles pour identifier les cytolysines, des molécules ayant des intérêts biotechnologiques et biomédicaux.
Introduction
Les animaux marins sont une riche source de composés biologiquement actifs. Au cours des dernières décennies, la composition du venin d’anémone de mer a attiré l’attention scientifique car elle comprend une diversité de polypeptides ayant une activité hémolytique, cytotoxique, enzymatique (phospholipase, protéase, chitinase), une activité neurotoxique et des effets inhibiteurs sur l’activité protéolytique1. De plus, ces polypeptides sont des sources potentielles pour le développement d’outils moléculaires à usage biotechnologique et thérapeutique 2,3.
Il existe peu de rapports sur le venin d’anémone de mer et ses composants moléculaires en raison de la complexité de l’obtention du venin, même de l’isolement et de la caractérisation des toxines. Les méthodes d’extraction utilisées dans les rapports impliquaient la lyse et la vidange du contenu des cellules qui sont liées et non liées à la production de venin1.
Une caractéristique particulière chez tous les cnidaires est l’absence d’un système de production et de libération du venin centralisé dans une seule région anatomique. Au lieu de cela, les nématocystes sont des structures qui gardent le venin 4,5. D’autres types de cellules, appelées cellules de la glande épidermique, sécrètent également des toxines et sont également distribuées dans tout le corps des anémones de mer6.
Le premier et le plus crucial défi dans l’obtention du venin est la génération d’un extrait avec une manipulation suffisante dans les processus ultérieurs, sans l’inactivation ou la dégradation des protéines labiles. Ensuite, les cellules doivent être lysées, et les composants - dans ce cas, les polypeptides doivent être extraits efficacement et rapidement, en évitant la protéolyse et l’hydrolyse tout en éliminant les autres composants cellulaires7.
Différentes méthodes sont utilisées pour obtenir l’extrait brut d’une anémone de mer; certains impliquent de sacrifier l’organisme tandis que d’autres permettent de le maintenir en vie. Les méthodes qui impliquent l’utilisation de tout le corps de l’organisme permettent la libération de la plupart des toxines du venin8, par rapport aux méthodes qui maintiennent les organismes en vie, qui n’extraient que certains composants du venin9. La préparation d’un extrait nécessite d’évaluer la présence et la puissance d’une substance d’intérêt par le biais d’un essai biologique spécifique, qui comprend des stratégies pour observer les effets pharmacologiques par des méthodes in vivo ou in vitro 10.
Le venin d’anémone de mer contient des polypeptides cytolytiques, des toxines formant des pores (PFT)11 et des phospholipases12; ces molécules sont des modèles dans l’étude de l’interaction protéine-lipide, des outils moléculaires dans le traitement du cancer et des biocapteurs basés sur le nanopore3. La classification des PFT d’anémone de mer est effectuée en fonction de leur taille ou de leur poids moléculaire, de 5 kDa à 80 kDa. Le PFT de 20 kDa, le plus étudié et le plus connu sous le nom d’actinoporines11, présente un intérêt particulier pour son potentiel biomédical dans le développement d’outils moléculaires pour des applications possibles comme biocapteurs anticancéreux, antimicrobiens et à base de nanopores. Une autre cytolysine, y compris les phospholipases, en particulier la phospholipase A2 (PLA2)13, libère un acide gras dû et hydrolyse les phospholipides, déstabilisant la membrane cellulaire. En raison de ce mécanisme d’action, PLA2 promet d’être un modèle essentiel pour l’étude et les applications dans les maladies inflammatoires. Il pourrait servir de modèle pour les études du comportement lipidique dans la membrane cellulaire14.
Ici, nous décrivons un protocole efficace pour obtenir l’extrait brut de l’anémone de mer Anthopleura dowii Verrill, 1869, et détecter les hémolysines et les phospholipases. Les deux sont des toxines pertinentes qui pourraient être utilisées comme modèle pour concevoir de nouveaux outils moléculaires.
Protocole
Les anémones de mer ont été collectées conformément aux directives de la Commission nationale de l’aquaculture, de la pêche et de l’alimentation du gouvernement fédéral du Mexique (numéro de permis PPF / DGOPTA 07332.250810.4060). Le Comité de bioéthique de l’Institut de biotechnologie de l’Université nationale autonome du Mexique a approuvé toutes les expériences avec des anémones de mer. L’échantillon de sang de mouton a été acheté au Centre d’enseignement pratique et de recherche en production et santé animales (CEPIPSA, Université nationale autonome du Mexique).
1. Collecte d’organismes
- Collecter l’anémone de mer A. dowii.
NOTE: Ici, les organismes ont été collectés pendant les périodes de marée basse dans la zone intertidale au large de la côte d’Ensenada Baja, Californie, Mexique (Figure 1A, B). - Transporter les organismes au laboratoire dans des récipients contenant de l’eau de mer (figure 1C).
- En laboratoire, nettoyez les organismes avec de l’eau distillée à la main pour éliminer les grosses particules de substrat qui adhèrent au corps.
- Congeler immédiatement les organismes à -80 °C dans un ultra-congélateur pendant 72 h.
- Placer les organismes dans des verres spéciaux pour la lyophilisation, avec des conditions de lyophilisation de -20 °C, 0,015 psi, pendant 48 h.
- Conserver les organismes lyophilisés à -20 °C jusqu’à leur utilisation.
2. Hydratation des tissus
- Reconstituer 20 g de poids sec d’organismes lyophilisés avec 60 mL (1/3 p/v) de tampon phosphate de sodium de 50 mM, pH 7,5, et 1 comprimé cocktail inhibiteur (Table des matières).
- Dans un agitateur magnétique, remuer continuellement l’échantillon à ~800 tr/min, 4 °C, pendant 12 h.
3. Libération de toxines
- Pour induire la lyse cellulaire et la décharge de nématocystes, congeler l’échantillon à -20 °C pendant 12 h, puis placer le récipient dans de l’eau à température ambiante (RT) jusqu’à ce qu’il dégèle jusqu’à 90%. Répétez cette procédure trois fois.
REMARQUE: L’échantillon ne dégèle pas à 100%; il est crucial de maintenir l’échantillon à ~4 °C pour prévenir la dégradation des protéines. - Observez le nématocyste déchargé sous un microscope confocal. Prélever 10 μL de l’échantillon sur une lame et placer un couvercle dessus. Observez au microscope confocal à un grossissement de 100x et 60x. Ici, un colorant n’était pas nécessaire.
- Traiter les images capturées au microscope confocal dans le logiciel ImageJ (version 1.53c, Wayne Rasband, National Institutes of Health, USA, https://imagej.nih.gov/ij).
- Si le tubule à l’intérieur du nématocyste se désenclenche et se trouve à l’extérieur, les nématocystes sont évacués; ensuite, passez à l’étape suivante. Sinon, n’effectuez qu’un seul cycle de gel-dégel de plus.
- Pour clarifier l’extrait, centrifugez l’échantillon à 25 400 x g pendant 40 min, 4 °C, récupérez le surnageant et centrifugez-le à nouveau. Répétez cette étape 3 à 4 fois.
- Récupérez le surnageant et jetez la pastille. L’extrait surnageant ou brut contient les composants du venin (toxines) et d’autres molécules cellulaires, telles que les lipides, les glucides et les acides nucléiques.
4. Quantification des protéines totales
- Déterminer la concentration en protéines de l’extrait brut à l’aide d’un kit commercial bradford colorimétrique15 (Table des matériaux).
- Diluer le réactif colorant (une partie du colorant avec quatre parties d’eau distillée).
- Préparer une solution d’albumine sérique bovine (BSA) Fraction V (Table des matériaux) à une concentration de 1 mg/mL dans un tampon phosphate (50 mM de phosphate de sodium, pH 7,5).
- Préparez les solutions répertoriées dans le tableau 1 en trois exemplaires.
- Mélanger vigoureusement chaque solution dans un shaker pendant 4 s.
- Incuber les échantillons pendant 5 min à TA, sans secouer.
- Mesurer l’absorbance (UA) à 595 nm.
- Tracez les moyennes des absorbances BSA de l’échantillon (UA vs concentration en protéines) et déterminez l’équation du graphique (équation 1).
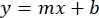 Équation 1
Équation 1
où y est l’absorbance, m est la pente de la ligne, x est la concentration en protéines et b est l’interception y. Pour calculer la concentration d’extrait brut, résolvez pour le x comme suit: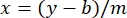 Équation 2
Équation 2
5. Déterminer la complexité du venin polypeptidique
- Assemblez le récipient en verre pour l’électrophorèse verticale sur gel conformément au manuel.
- Préparer le mélange d’acrylamide (30%) : 29,2 g d’acrylamide, 0,8 g de bis-acrylamide, le volume final de 100 mL. Filtrer la solution à l’aide d’une membrane de 0,45 μm. Conserver la solution dans une bouteille sombre à 4 °C. Ensuite, préparez le mélange pour le gel résolvant (tableau 2).
- Le gel SDS-PAGE se compose d’un gel résolvant et d’un gel empilable. Préparer les gels selon les volumes de solution définis dans le tableau 2. Pendant la préparation, assurez-vous de maintenir chaque mélange à 4 °C pour réduire le temps de polymérisation.
NOTE: Le volume de chaque mélange varie en fonction de la marque d’équipement utilisé; pour ce protocole, les volumes correspondent à la préparation d’un gel d’acrylamide à 15%, de 0,75 mm d’épaisseur, pour une chambre d’électrophorèse verticale (Table des matières). - Une fois l’acrylamide polymérisé, ~10 min, ajouter le mélange de gel empilable.
- Préparez le mélange pour le gel d’empilage, ajoutez-le immédiatement aux plaques de verre et placez un peigne pour former les puits pour charger les échantillons.
- Une fois que le gel empilable a polymérisé (~15 min), retirez le peigne et placez le récipient en verre dans une chambre pour l’électrophorèse verticale.
- Placer 200 mL de tampon d’électrode (0,25 M Tris, 0,192 M glycine, 0,1% SDS) dans la chambre d’électrophorèse.
- Analyser l’extrait brut : Mélanger 30 μg d’extrait brut avec 5 μL de tampon de charge protéique (25 % de glycérol, 15 % de dodécylsulfate de sodium, 25 % de 2-mercaptoéthanol, 0,125 mM de Tris pH 7, bleu de bromophénol 0,1 mg/mL) pour dénaturer les protéines. Le volume final doit être de <50 μL.
- Chauffer à 90 °C pendant 5 min, refroidir et centrifuger pendant 3 s à 1 400 x g.
- Chargez les échantillons dans les puits du gel empilable. Chargez une échelle de poids moléculaire standard.
- Exécuter l’électrophorèse à 25 mA constant pendant 1 h 30 min.
- Retirez les récipients en verre de la chambre d’électrophorèse pour procéder à la coloration des protéines du gel.
6. Coloration protéique
- Rincez le gel dans 50 mL d’eau distillée pour éliminer les débris de l’électrode tampon.
- Jetez l’eau.
- Pour éviter la diffusion des protéines du gel, ajouter la solution de fixation (60 mL d’éthanol, 20 mL d’acide acétique et 20 mL d’eau distillée, volume final 100 mL). Incuber à RT pendant 40 min en secouant à 80 tr/min. Décantez la solution.
- Ajouter la solution de réticulation des protéines (15 mL d’éthanol, 0,25 mL de glutaraldéhyde, 50 mL d’eau distillée, volume final 65,25 mL) et incuber pendant 30 min à RT en agitant à 80 tr/min. Supprimez la solution.
- Lavez le gel avec 100 mL d’eau distillée pendant 5 min et retirez l’eau. Répétez cette procédure quatre fois.
- Effectuer la coloration protéique avec 50 mL de solution filtrée Coomassie bleu brillant R-250 (40% de méthanol, 10% d’acide acétique, 50% d’eau distillée, 0,1% de colorant, volume final 100 mL), en remuant à 60 tr/min dans un oscillateur rotatif de laboratoire à RT pendant 15 min.
- Récupérez la solution, qui peut être utilisée pour tacher d’autres gels.
- Pour éliminer l’excès de colorant, ajouter une solution de conservation (40 mL de méthanol, 10 mL d’acide acétique, 50 mL d’eau distillée). Continuez à remuer (80 tr/min) à RT jusqu’à ce que les bandes (protéines colorées) visualisent.
- Conservez le gel dans 50 mL d’eau distillée et numérisez l’image dans un système de documentation Gel.
7. Préparer une solution de globules rouges
- Extraire 1 mL de sang de la veine jugulaire d’un mouton.
- Retirer l’aiguille de la seringue et drainer immédiatement le sang dans un tube contenant 50 mL de solution d’Alsever (0,002 M d’acide citrique, 0,07 M de NaCl, 0,1 M de dextrose et 0,027 M de citrate de sodium comme anticoagulant, pH 7,4).
- Inversez lentement la solution trois fois.
- Conserver à 4 °C pour le transfert au laboratoire.
- Centrifuger à 804 x g pendant 5 min à 4 °C et jeter le surnageant.
- Remettre en suspension la pastille dans 30 mL de solution d’Alsever. Ajoutez la solution en la faisant glisser le long des parois internes du tube et, à l’aide d’une pipette en plastique Pasteur, remettez lentement les cellules en suspension. Centrifuger à nouveau à 804 x g pendant 5 min, 4 °C. Répétez cette procédure jusqu’à ce que le surnageant soit clarifié.
- Ajouter 15 mL de solution d’Alsever; remettre en suspension lentement le granulé avec une pipette en plastique Pasteur.
- Maintenir le volume final de la solution d’Alsever pour atteindre une concentration de 2 x 106 cellules/mL, environ. Ensuite, utilisez une chambre Neubauer ou un hémocytomètre pour compter les cellules.
REMARQUE: Comptez les érythrocytes dans la lame de l’hémacytomètre (Neubauer amélioré). La diapositive est une diapositive de 30 mm x 70 mm x 4 mm. Le centre a deux plaques de pied argentées (Figure 2A) (une grille supérieure et une grille inférieure), chacune de 3 mm x 3 mm, et deux canaux sur les côtés de la grille. Le comptage des cellules se fait dans les coins et les carrés centraux (carrés rouges à la figure 2B). La concentration optimale de cellules pour le comptage doit être de l’ordre de 10à 6 cellules. La boîte centrale a une surface de 0,1 cm2, ce qui équivaut à 0,0001 mL. - Placez un couvercle sur la diapositive.
- Placez 10 μL de l’échantillon entre la lame et le couvercle et attendez que l’échantillon soit déboursé.
- Dans un microscope optique (grossissement 60x), effectuez le comptage dans la première case carrée. Comptez uniquement les cellules de la grille centrale et celles qui touchent la marge supérieure ou gauche de la boîte.
- Déterminer la concentration cellulaire à l’aide de l’équation suivante :
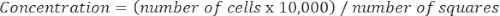 Équation 3
Équation 3
Si l’échantillon est très concentré et qu’une dilution est nécessaire, utilisez l’équation suivante: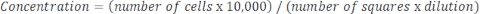 Équation 4
Équation 4
REMARQUE: Lors de la prise de la solution érythrocytaire pour le test d’hémolyse, homogénéiser lentement la solution avec une pipette Pasteur en plastique. - Effectuez les étapes en trois exemplaires pour réduire les erreurs.
8. Essai d’hémolyse
- Préparer les mélanges de solution (en trois exemplaires) indiqués dans le tableau 3 dans des plaques coniques de 96 puits.
REMARQUE: Mélanger les érythrocytes lentement pour maintenir une suspension homogène. - Incuber pendant 1 h à 37 °C, sans secouer.
- Centrifuger la plaque à 804 x g pendant 5 min à 4 °C.
- Récupérez le surnageant dans une autre plaque de 96 puits avec un fond plat.
- Si l’extrait brut contient des cytolysines, les érythrocytes vont lyser et libérer de l’hémoglobine. Lisez l’absorbance à 415 nm.
- Calculez le pourcentage d’hémolyse à l’aide de l’équation suivante :
% Hémolyse = ((Extrait d’Acrude-AAlsever buffer) / (AH2O-AAlsever)) x 100
REMARQUE: L’extrait d’acrude, le tampon AAlsever et AH2O correspondent respectivement à l’absorbance des érythrocytes avec l’extrait brut, à l’absorbance de la solution d’Alsever et à l’absorbance de l’eau distillée. - Effectuer un test d’hémolyse avant et après chaque cycle de congélation et de décongélation avec 50 μg de protéines totales provenant de l’extrait brut.
- Déterminer le nombre de cycles de congélation et de décongélation nécessaires pour atteindre une activité hémolytique de 100 %.
- Calculer la quantité d’extrait qui produit l’hémolyse dans 50% des érythrocytes (HU50); suivre le même protocole décrit dans le tableau 3, augmenter la quantité d’extrait brut d’anémone de mer et concevoir la placette avec des ajustements sigmoïdaux à l’aide d’un logiciel approprié (p. ex., origine, table des matières).
9. Dosage de la phospholipase
- Laver un œuf de poule avec 1% de FDS dans de l’eau distillée.
- Séparez le jaune d’œuf du blanc d’œuf dans des conditions stériles.
- Préparer 50 mL de solution de NaCl à 0,86 % et filtrer à travers un filtre de 0,22 μm. Ensuite, préparez la solution A: Ajouter 12 mL de jaune d’œuf et 36 mL de solution de NaCl à 0,86%.
- Préparer la solution B : Mélanger 300 mg d’agarose dans 50 mL de tampon (50 mM Tris, pH 7,5), filtrer à travers un filtre de 0,22 μm. Chauffer la solution au micro-ondes jusqu’à ébullition. Refroidir dans de l’eau tiède pour atteindre 43-45°C.
- Préparer la solution C : Préparez 10 mM de CaCl2 et filtrez-la avec un filtre de 0,22 μm.
- Préparer la solution D : 10 mg de rhodamine 6G dans 1 mL d’eau distillée.
- Dans des conditions stériles (hotte à écoulement laminaire), ajouter 500 μL de solutions A et C à la solution B et 100 μL de solution D, mélanger et verser 25 mL dans chaque boîte de Pétri (90 x 15 mm).
- Attendez que la solution se solidifie dans des conditions stériles (30 min).
- Faites des puits (~ 2-3 mm de diamètre) avec un tube mince.
- Ajouter un total de 20 μL de tampon phosphate (témoin négatif) dans un puits et 20 μL d’une phospholipase déterminée (témoin positif) dans un autre puits.
- Placer différentes quantités de protéines d’extrait brutes, 5, 15, 25, 35, 45 μg, dans les puits restants, chacun dans un volume final de 20 μL.
- Attendez que la gélose adsorbe tous les échantillons (30 min).
- Incuber à 37 °C pendant 20 h.
- Si un halo se forme autour du puits, cela indique une activité de la phospholipase. Mesurez le halo formé avec un étrier vernier.
REMARQUE: Effectuez l’expérience en trois exemplaires (étapes de 9.1 à 9.14).
Résultats
Les résultats représentatifs du protocole utilisé pour obtenir l’extrait brut d’anémone de mer ont montré que la combinaison de deux techniques (agitation et cycles de congélation et de décongélation) produisait un rejet efficace de nématocystes, et la quantité totale de protéines était de 500 mg (8 mg/mL) (figure 3).
La complexité protéique de l’extrait brut a pu être observée à partir de 10 kDa et de plus de 250 kDa par électrophorèse SD...
Discussion
La forte demande de nouveaux composés ayant des applications dans différents domaines de la science et de l’industrie a conduit à l’étude du venin. Venom représente une riche source de molécules qui sert de modèle pour générer de nouveaux outils moléculaires. Cependant, la complexité de ces venins nécessite la mise en œuvre et la combinaison de diverses méthodes pour les obtenir et les étudier.
Ici, nous montrons une méthode pour obtenir et analyser le venin de l’anémone...
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun intérêt concurrent en ce qui concerne la publication de cet article.
Remerciements
Ce travail a été soutenu par Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT), avec le numéro de subvention IT200819. Les auteurs remercient Tom Musselman, Rock Paper Editing, LLC, d’avoir vérifié la grammaire anglaise de ce manuscrit; et l’assistance technique de Samanta Jiménez (CICESE, Ensenada) et Juan Manuel Barbosa Castillo (Instituto de Fisiología Celular, UNAM). Nous remercions également le Dr Augusto César Lizarazo Chaparro (CEPIPSA) pour l’obtention de sang de mouton. Nous remercions tout particulièrement le Dr José Saniger Blesa, ICAT-UNAM, pour les installations de son laboratoire pour l’enregistrement vidéo.
matériels
| Name | Company | Catalog Number | Comments |
| 15 mL conical centrifuge tube | Corning | 430766 | |
| 2-Bromophenol blue | Sigma | B75808 | |
| 2-mercaptoetanol | Sigma-Aldrich | M6250-100ML | |
| 50 mL conical centrifuge tubes | Corning | 430828 | |
| Acetic Acid Glacial | J.T. Baker | 9515-03 | |
| Acrylamide | Promega | V3115 | |
| Agarose | Promega | V3125 | |
| Bisacrylamide | Promega | V3143 | |
| Bovine Serum Albumin Fraction V | Sigma | A3059-100G | |
| Bradford Protein Assays | Bio-Rad | 5000006 | |
| Calcium chloride | Sigma-Aldrich | C3306 | |
| Cell culture plates 96 well, V-bottom | Corning | 3894 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge tubes | Corning | CLS430829 | |
| ChemiDoc MP system | Bio-Rad | 1708280 | |
| Citric acid | Sigma-Aldrich | 251275 | |
| Clear flat.bottom 96-Well Plates | Thermo Scientific | 3855 | |
| Coomassie Brilliant Blue G-250 | Bio-Rad | #1610406 | |
| Coomassie brilliant blue R-250 | Bio-Rad | 1610400 | |
| Dextrose | J.T. Baker | 1916-01 | |
| Ductless Enclosure | Labconco | Vertical | https://imagej.nih.gov/ij ImageJ 1.53c |
| Gel Doc EZ | Bio Rad. | Gel Documentation System | |
| Glycerol | Sigma-Aldrich | G5516-4L | |
| Hemocytometer | Marienfeld | 650030 | |
| ImageJ (Software) | NIH, USA | Version 1.53c | |
| Incubator 211 | Labnet | I5211 DS | |
| Methanol | J.T. Baker | 9049-03 | |
| Mini-PROTEAN tetra cell | Bio-Rad | 1658000EDU | |
| Na2HPO4 | J.T. Baker | 3824-01 | |
| NaCl | J.T. Baker | 3624-01 | |
| NaH2PO4.H2O | J.T. Baker | 3818-05 | |
| Origin software | version 9 | To design the plot with sigmoidal adjustments | |
| Petridish | Falcon | 351007 | |
| Pipetman kit | Gilson | F167380 | |
| Precast mini gel | BioRad | 1658004 | |
| Prestained Protein Ladder | Thermo Scientific | 26620 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protein Assay Dye Reagent Concentrate | Bio-Rad | 5000006 | |
| Rhodamine 6G | Sigma-Aldrich | 252433 | |
| SDS | Sigma-Aldrich | L4509 | |
| Sodium citrate dihydrate | JT Baker | 3646-01 | |
| Spectrophotometer | THERMO SCIENTIFIC | G10S UV-VIS | |
| Tris Base | Sigma-Aldrich | 77-86-1 | |
| Volt Power Supply | Hoefer | PS300B |
Références
- Frazão, B., Vasconcelos, V., Antunes, A. Sea anemone (Cnidaria, Anthozoa, Actiniaria) toxins: an overview. Marine Drugs. 10 (8), 1812-1851 (2012).
- Jayathilake, J. M. N. J., Gunathilake, K. V. K. Cnidarian toxins: recent evidences for potential therapeutic uses. The European Zoological Journal. 87 (1), 708-713 (2020).
- Ramírez-Carreto, S., Miranda-Zaragoza, B., Rodríguez-Almazán, C. Actinoporins: From the structure and function to the generation of biotechnological and therapeutic tools. Biomolecules. 10 (4), 539 (2020).
- Fautin, D. G. Structural diversity, systematics, and evolution of cnidae. Toxicon. Official Journal of the International Society on Toxinology. 54 (8), 1054-1064 (2009).
- Moran, Y., et al. Analysis of soluble protein contents from the nematocysts of a model sea anemone sheds light on venom evolution. Marine Biotechnology. 15 (3), 329-339 (2013).
- Moran, Y., et al. Neurotoxin localization to ectodermal gland cells uncovers an alternative mechanism of venom delivery in sea anemones. Proceedings of the Royal Society B: Biological Sciences. 279 (1732), 1351-1358 (2012).
- Grabski, A. C. Advances in preparation of biological extracts for protein purification. Methods in Enzymology. 463, 285-303 (2009).
- Bulati, M., et al. Partially purified extracts of sea anemone Anemonia viridis affect the growth and viability of selected tumour cell lines. BioMed Research International. 2016, 3849897 (2016).
- Orts, D. J. B., et al. Biochemical and electrophysiological characterization of two sea anemone type 1 potassium toxins from a geographically distant population of Bunodosoma caissarum. Marine Drugs. 11 (3), 655-679 (2013).
- Hader, D., Erzinger, G. . Bioassays: Advanced Methods and Applications. , (2017).
- Anderluh, G., Macek, P. Cytolytic peptide and protein toxins from sea anemones (Anthozoa: Actiniaria). Toxicon: Official Journal of the International Society on Toxinology. 40 (2), 111-124 (2002).
- Nevalainen, T. J., et al. Phospholipase A2 in cnidaria. Comparative Biochemistry and Physiology. Part B, Biochemistry & Molecular Biology. 139 (4), 731-735 (2004).
- Razpotnik, A., et al. A new phospholipase A2 isolated from the sea anemone Urticina crassicornis - its primary structure and phylogenetic classification. The FEBS Journal. 277 (12), 2641-2653 (2010).
- Dennis, E. A., Cao, J., Hsu, Y. -. H., Magrioti, V., Kokotos, G. Phospholipase A2 enzymes: Physical structure, biological function, disease implication, chemical inhibition, and therapeutic intervention. Chemical Reviews. 111 (10), 6130-6185 (2011).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Kwon, Y. -. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, 8663 (2015).
- Eno, A. E., Konya, R. S., Ibu, J. O. Biological properties of a venom extract from the sea anemone, Bunodosoma cavernata. Toxicon: Official Journal of the International Society on Toxinology. 36 (12), 2013-2020 (1998).
- Morales-Landa, J. L., et al. Antimicrobial, antiprotozoal, and toxic activities of Cnidarian extracts from the Mexican Caribbean Sea. Pharmaceutical Biology. 45 (1), 37-43 (2007).
- Sánchez-Rodríguez, J., Cruz-Vazquez, K. Isolation and biological characterization of neurotoxic compounds from the sea anemone Lebrunia danae (Duchassaing and Michelotti, 1860). Archives of Toxicology. 80 (7), 436-441 (2006).
- de Oliveira, J. S., et al. Caissarolysin I (Bcs I), a new hemolytic toxin from the Brazilian sea anemone Bunodosoma caissarum: purification and biological characterization. Biochimica Et Biophysica Acta. 1760 (3), 453-461 (2006).
- Norton, R. S., et al. Purification and characterisation of proteins with cardiac stimulatory and haemolytic activity from the anemone Actinia tenebrosa. Toxicon: Official Journal of the International Society on Toxinology. 28 (1), 29-41 (1990).
- Gondran, M., Eckeli, A. L., Migues, P. V., Gabilan, N. H., Rodrigues, A. L. S. The crude extract from the sea anemone, Bunodosoma caissarum elicits convulsions in mice: possible involvement of the glutamatergic system. Toxicon: Official Journal of the International Society on Toxinology. 40 (12), 1667-1674 (2002).
- Dion, A. S., Pomenti, A. A. Ammoniacal silver staining of proteins: mechanism of glutaraldehyde enhancement. Analytical Biochemistry. 129 (2), 490-496 (1983).
- Ramírez-Carreto, S., et al. Identification of a pore-forming protein from sea anemone Anthopleura dowii Verrill (1869) venom by mass spectrometry. The Journal of Venomous Animals and Toxins Including Tropical Diseases. 25, 147418 (2019).
- Nelson, G. J. Studies on the lipids of sheep red blood cells. I. Lipid composition in low and high potassium red cells. Lipids. 2 (1), 64-71 (1967).
- Ramírez-Carreto, S., et al. Transcriptomic and proteomic analysis of the tentacles and mucus of Anthopleura dowii Verrill, 1869. Marine Drugs. 17 (8), 436 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon