É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Identificação da Atividade Hemolítica e Fosfolipase em Extratos Brutos de Anêmonas do Mar por Bioensaões Simples
Neste Artigo
Resumo
Aqui, descrevemos um protocolo para obter extrato de veneno bruto da anêmona do mar e detectar sua atividade hemolítica e fosfolipase.
Resumo
A composição do veneno de anêmona do mar inclui moléculas de polipeptídeos e não-proteínas. Os componentes citólticos têm um alto potencial biotecnológico e biomédico para projetar novas ferramentas moleculares. O veneno de anêmona marinha se localiza em células glandulares a partir de ectoderme e estruturas subcelulares chamadas nematocistos, ambas distribuídas por todo o corpo de anêmona do mar. Essa característica implica desafios porque as células e o nematócito devem ser lícitos para liberar os componentes do veneno com outras moléculas não tóxicas. Portanto, primeiro, o veneno é derivado de um extrato bruto (mistura de moléculas diferentes e diversas e detritos teciduais). O próximo passo é detectar polipeptídeos com bioatividades específicas. Aqui, descrevemos uma estratégia eficiente para obter o extrato bruto de anêmona do mar e bioensaio para identificar a presença de citolises. O primeiro passo envolve técnicas baratas e simples (ciclo agitado e degelo) para liberar citolísinas. Obtivemos a maior atividade citolítica e proteína (~500 mg de proteína a partir de 20 g de peso seco). Em seguida, a complexidade do polipeptídeo do extrato foi analisada pelo gel SDS-PAGE detectando proteínas com pesos moleculares entre 10 kDa e 250 kDa. No ensaio hemollítico, utilizamos glóbulos vermelhos ovelhas e determinamos hu50 (11,1 ± 0,3 μg/mL). Em contraste, a presença de fosfolipases no extrato bruto foi determinada utilizando gema de ovo como substrato em um meio sólido com agarose. No geral, este estudo utiliza um protocolo eficiente e barato para preparar o extrato bruto e aplica bioensações replicáveis para identificar citolysinas, moléculas com interesses biotécnicos e biomédicos.
Introdução
Animais marinhos são uma rica fonte de compostos biologicamente ativos. Nas últimas décadas, a composição do veneno de anêmona marinha tem atraído atenção científica, pois compreende uma diversidade de polipeptídeos com hemolítica, citotóxica, enzimática (fosfolipase, protease, quitinase) e atividade neurotóxica e efeitos inibitórios na atividade proteolítica1. Além disso, esses polipeptídeos são fontes potenciais para o desenvolvimento de ferramentas moleculares no uso biotecnológico e terapêutico 2,3.
Há poucos relatos sobre o veneno de anêmona marinha e seus componentes moleculares devido à complexidade da obtenção do veneno, até mesmo do isolamento e caracterização de toxinas. Os métodos de extração utilizados nos relatórios envolveram a lise e o esvaziamento do conteúdo das células relacionadas e não relacionadas à produçãode veneno 1.
Uma característica particular em todos os cnidários é a ausência de um sistema de produção e liberação do veneno centralizado em uma única região anatômica. Em vez disso, os nematocistos são estruturas que mantêm o veneno 4,5. Outros tipos de células, chamadas células da glândula epidérmica, também secretam toxinas e também são distribuídas por todo o corpo de anêmonas do mar6.
O primeiro e mais crucial desafio na obtenção do veneno é a geração de um extrato com manipulação suficiente em processos subsequentes, sem a inativação ou degradação de proteínas labile. Em seguida, as células devem ser liseadas, e os componentes - neste caso, os polipeptídeos devem ser extraídos de forma eficiente e rápida, evitando a proteólise e a hidrólise ao eliminar outros componentes celulares7.
Diferentes métodos são utilizados para obter o extrato bruto de uma anêmona marinha; alguns envolvem sacrificar o organismo, enquanto outros permitem que ele seja mantido vivo. Métodos que implicam o uso de todo o corpo do organismo permitem a liberação da maioria das toxinas do veneno8, em comparação com métodos que mantêm os organismos vivos, que extraem apenas alguns componentes do veneno9. A elaboração de um extrato requer avaliar a presença e potência de uma substância de interesse por meio de um bioensaio específico, que inclui estratégias para observar os efeitos farmacológicos pelos métodos in vivo ou in vitro 10.
O veneno de anêmona do mar contém polipeptídeos citólíticos, toxinas formadoras de poros (PFTs)11 e fosfolipases12; essas moléculas são modelos no estudo da interação proteína-lipídica, ferramentas moleculares na terapia do câncer e biosensores à base de nanoporos3. A classificação de PFTs de anêmona marinha é realizada de acordo com seu tamanho ou peso molecular, de 5 kDa a 80 kDa. O PFT de 20 kDa, o mais estudado e conhecido como actinoporinas11, é de particular interesse por seu potencial biomédico no desenvolvimento de ferramentas moleculares para possíveis aplicações como biosensores anticancerígenos, antimicrobianos e à base de nanoporos. Outra citolisina, incluindo fosfolipases, especificamente fosfolipase A2 (PLA2)13, libera um ácido graxo devido e hidrolisa fosfolipídios, desestabilizando a membrana celular. Devido a esse mecanismo de ação, o PLA2 promete ser um modelo essencial para o estudo e aplicações em doenças inflamatórias. Poderia servir de modelo para estudos de comportamento lipídudo na membranacelular 14.
Aqui, descrevemos um protocolo eficiente para a obtenção do extrato bruto da anêmona anthopleura dowii Verrill, 1869, e a detecção de hemolises e fosfolipases. Ambas são toxinas relevantes que poderiam ser usadas como modelo para projetar novas ferramentas moleculares.
Protocolo
As anêmonas do mar foram coletadas de acordo com as diretrizes da Comissão Nacional de Aquicultura, Pesca e Alimentação do Governo Federal do México (número de licença PPF/DGOPTA 07332.250810.4060). O Comitê de Bioética do Instituto de Biotecnologia da Universidade Nacional Autônoma do México aprovou todos os experimentos com anêmonas do mar. A amostra de sangue de ovelha foi adquirida no Centro de Ensino Prático e Pesquisa em Produção e Saúde Animal (CEPIPSA, Universidade Nacional Autônoma do México).
1. Coleta de organismos
- Coletar anêmona do mar A. dowii.
NOTA: Aqui, os organismos foram coletados durante períodos de maré baixa na zona intertidal ao largo da costa de Ensenada Baja, Califórnia, México (Figura 1A,B). - Transporte os organismos para o laboratório em recipientes com água do mar (Figura 1C).
- Em laboratório, limpe os organismos com água destilada à mão para remover as grandes partículas de substrato que aderiram ao corpo.
- Congele imediatamente os organismos a -80 °C em um ultracongelante por 72 h.
- Coloque os organismos em copos especiais para secagem congelante, com condições de liofilização de -20 °C, 0,015 psi, por 48 h.
- Armazene os organismos liofilizados a -20 °C até o uso.
2. Hidratação tecidual
- Reconstitua 20 g de peso seco de organismos liofilizados com 60 mL (1/3 w/v) de tampão fosfato de sódio de 50 mM, pH 7,5 e 1 comprimido de coquetel inibidor (Tabela de Materiais).
- Em um agitador magnético, mexa continuamente a amostra a ~800 rpm, 4 °C, por 12 h.
3. Liberação de toxinas
- Para induzir a lise celular e a descarga do nematócito, congele a amostra a -20 °C por 12 h e, em seguida, coloque o recipiente na água em temperatura ambiente (RT) até descongelar até 90%. Repita este procedimento três vezes.
NOTA: A amostra não descongela 100%; é crucial manter a amostra a ~4 °C para evitar a degradação da proteína. - Observe o nematocísto descarregado sob um microscópio confocal. Pegue 10 μL da amostra em um slide e coloque uma mancha sobre ela. Observe sob um microscópio confocal a 100x e 60x de ampliação. Aqui um corante não era necessário.
- Processe as imagens capturadas no microscópio confocal no software ImageJ (versão 1.53c, Wayne Rasband, National Institutes of Health, EUA, https://imagej.nih.gov/ij).
- Se o túbulo dentro do nematocisto se descarregar e estiver fora, os nematocistos são descarregados; então, continue com o próximo passo. Caso contrário, só conduza mais um ciclo de congelamento.
- Para esclarecer o extrato, centrifugar a amostra a 25.400 x g por 40 min, 4 °C, recuperar o sobrenante e centrífusá-la novamente. Repita esta etapa 3-4 vezes.
- Recupere o supernatante e descarte a pelota. O extrato supernacante ou bruto contém os componentes do veneno (toxinas) e outras moléculas celulares, como lipídios, carboidratos e ácidos nucleicos.
4. Quantitação da proteína total
- Determine a concentração proteica do extrato bruto utilizando um kit comercial de ensaio colorimétrico Bradford15 (Tabela de Materiais).
- Diluir o reagente de corante (uma parte do corante com quatro partes de água destilada).
- Prepare a solução de albumina de soro bovino (BSA) Fraction V (Tabela de Materiais) a 1 mg/mL de concentração em tampão fosfato (fosfato de sódio de 50 mM, pH 7.5).
- Prepare as soluções listadas na Tabela 1 em triplicado.
- Misture vigorosamente cada solução em um shaker para 4 s.
- Incubar as amostras por 5 minutos no RT, sem tremer.
- Medir a absorvância (UA) a 595 nm.
- Plote as médias das absorvências BSA da amostra (concentração de UA vs. proteína), e determine a equação do gráfico (Equação 1).
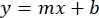 Equação 1
Equação 1
onde y é a absorvância, m é a inclinação da linha, x é a concentração de proteína, e b é o y-intercept. Para calcular a concentração de extrato bruto, resolva para o x da seguinte forma: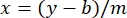 Equação 2
Equação 2
5. Determine a complexidade do veneno de polipeptídeo
- Monte o recipiente de vidro para eletroforese de gel vertical de acordo com o manual.
- Prepare a mistura de acrilamida (30%): 29,2 g de acrilamida, 0,8 g de bis-acrilamida, o volume final de 100 mL. Filtre a solução usando uma membrana de 0,45 μm. Armazene a solução em uma garrafa escura a 4 °C. Em seguida, prepare a mistura para o gel de resolução (Tabela 2).
- O gel SDS-PAGE consiste em um gel de resolução e um gel de empilhamento. Prepare os géis de acordo com os volumes de solução definidos na Tabela 2. Durante a preparação, certifique-se de manter cada mistura a 4 °C para reduzir o tempo de polimerização.
NOTA: O volume de cada mistura varia dependendo da marca de equipamentos utilizados; para este protocolo, os volumes correspondem à preparação de gel de acrilamida de 15%, 0,75 mm de espessura, para uma câmara de eletroforese vertical (Tabela de Materiais). - Depois que a acrilamida tiver polimerizado, ~10 min, adicione a mistura de gel de empilhamento.
- Prepare a mistura para o gel de empilhamento, adicione-a imediatamente às placas de vidro e coloque um pente para formar os poços para carregar as amostras.
- Uma vez que o gel de empilhamento tenha polimerizado (~15 min), remova o pente e coloque o recipiente de vidro em uma câmara para eletroforese vertical.
- Coloque 200 mL de tampão de eletrodos (0,25 M Tris, 0,192 M de glicina, 0,1% SDS) na câmara de eletroforese.
- Analise o extrato bruto: Misture 30 μg de extrato bruto com 5 μL de tampão de carga de proteína (25% glicerol, 15% sulfato de dodecil de sódio, 25% 2-mercaptoetanol, 0,125 mM Tris pH 7, azul bromofenol 0,1 mg/mL) a proteínas de desnaturais. O volume final deve ser de <50 μL.
- Aqueça a 90 °C por 5 min, esfrie e centrífugue por 3 s a 1.400 x g.
- Carregue as amostras nos poços do gel de empilhamento. Carregue uma escada de peso molecular padrão.
- Executar eletroforese a 25 mA constante por 1 h 30 min.
- Remova os recipientes de vidro da câmara de eletroforese para prosseguir com a coloração das proteínas de gel.
6. Coloração de proteínas
- Enxágüe o gel em 50 mL de água destilada para remover detritos do eletrodo tampão.
- Descarte a água.
- Para evitar a difusão das proteínas de gel, adicione a solução de fixação (60 mL de etanol, 20 mL de ácido acético e 20 mL de água destilada, volume final de 100 mL). Incubar na RT por 40 min com agitação a 80 rpm. Decante a solução.
- Adicione a solução de crosslinking de proteína (15 mL de etanol, 0,25 mL de glutaraldeído, 50 mL de água destilada, volume final de 65,25 mL) e incubar por 30 minutos na RT com agitação a 80 rpm. Remova a solução.
- Lave o gel com 100 mL de água destilada por 5 minutos e remova a água. Repita este procedimento quatro vezes.
- Realize a coloração proteica com 50 mL de solução filtrada R-250 azul brilhante coomassie (40% de metanol, 10% ácido acético, 50% água destilada, 0,1% corante, volume final de 100 mL), mexendo a 60 rpm em um oscilador rotativo de laboratório na RT por 15 min.
- Recupere a solução, que pode ser usada para manchar outros géis.
- Para eliminar o excesso de corante, adicione uma solução de desestabilização (40 mL de metanol, 10 mL de ácido acético, 50 mL de água destilada). Continue mexendo (80 rpm) no RT até que as bandas (proteínas manchadas) visualizem.
- Mantenha o gel em 50 mL de água destilada e escaneie a imagem em um sistema de documentação gel.
7. Prepare a solução de glóbulos vermelhos
- Extrair 1 mL de sangue da veia jugular de uma ovelha.
- Remova a agulha da seringa e drene imediatamente o sangue em um tubo com 50 mL de solução Alsever (ácido cítrico de 0,002 M, 0,07 M NaCl, 0,1 M dextrose e 0,027 M de sódio como anticoagulante, pH 7,4).
- Inverter lentamente a solução três vezes.
- Mantenha a 4 °C para transferência para o laboratório.
- Centrifugar a 804 x g por 5 min a 4 °C e descartar o supernatante.
- Resuspense a pelota em 30 mL de solução Alsever. Adicione a solução deslizando-a ao longo das paredes internas do tubo e, usando uma pipeta de plástico Pasteur, lentamente resuspenda as células. Centrifugar novamente a 804 x g para 5 min, 4 °C. Repita este procedimento até que o sobrenatante seja esclarecido.
- Adicionar 15 mL de solução Alsever; resuspenda a pelota lentamente com uma pipeta de plástico Pasteur.
- Mantenha o volume final da solução Alsever para alcançar uma concentração de 2 x 106 células/mL, aproximadamente. Em seguida, use uma câmara de Neubauer ou hemótmetro para contar as células.
NOTA: Conte os eritrócitos no slide hemacytómetro (Neubauer Melhorado). O slide é um slide de 30 mm x 70 mm x 4 mm. O centro tem duas placas prateadas (Figura 2A) (uma grade superior e uma inferior), cada uma com 3 mm x 3 mm, e dois canais nas laterais da grade. A contagem de células fica no canto e nas praças centrais (quadrados vermelhos na Figura 2B). A concentração ideal de células para contagem deve ser na ordem de 106 células. A caixa central tem uma superfície de 0,1 cm2, o que equivale a 0,0001 mL. - Coloque uma mancha de cobertura sobre o slide.
- Coloque 10 μL da amostra entre o slide e o deslizamento e espere que a amostra seja desembolsada.
- Em um microscópio óptico (ampliação 60x), realize a contagem na primeira caixa quadrada. Conte apenas as células da grade central e aquelas que tocam a margem superior ou esquerda da caixa.
- Determine a concentração celular usando a seguinte equação:
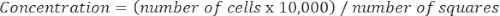 Equação 3
Equação 3
Se a amostra estiver muito concentrada e a diluição for necessária, use a seguinte equação: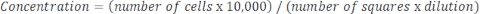 Equação 4
Equação 4
NOTA: Ao tomar a solução de eritrócito para o teste de hemólise, homogeneize lentamente a solução com uma pipeta Pasteur de plástico. - Execute as etapas em triplicado para reduzir o erro.
8. Ensaio de hemólise
- Prepare as misturas de solução (em triplicados) indicadas na Tabela 3 em placas cônicas de 96 poços.
NOTA: Misture lentamente os eritrócitos para manter uma suspensão homogênea. - Incubar por 1h a 37 °C, sem tremer.
- Centrifugar a placa a 804 x g por 5 min a 4 °C.
- Recupere o supernatante em outra placa de 96 poços com um fundo plano.
- Se o extrato bruto contiver citolises, os eritrócitos irão lise e liberarão hemoglobina. Leia a absorvência em 415 nm.
- Calcular porcentagem de hemólise, usando a seguinte equação:
% Hemolise = ((Extrato de Acrude-Tampão AAlsever) / (AH2O-AAlsever)) x 100
NOTA: Extrato de acrude, tampão AAlsever e AH2O correspondem à absorção de eritrócitos com o extrato bruto, absorção da solução Alsever e absorvância de água destilada, respectivamente. - Realize um teste de hemólise antes e depois de cada ciclo de congelamento e descongelamento com 50 μg de proteína total do extrato bruto.
- Determine o número de ciclos de congelamento e descongelamento necessários para atingir atividade 100% hemolítica.
- Calcular a quantidade de extrato que produz hemólise em 50% dos eritrócitos (HU50); seguir o mesmo protocolo descrito na Tabela 3, aumentar a quantidade de extrato bruto de anêmona do mar e projetar o lote com ajustes sigmoidal utilizando software apropriado (por exemplo, Origem, Tabela de Materiais).
9. Ensaio de fosfolipase
- Lave um ovo de galinha com 1% de SDS em água destilada.
- Separe a gema de ovo da clara de ovo em condições estéreis.
- Prepare 50 mL de solução NaCl de 0,86% e filtre através de um filtro de 0,22 μm. Em seguida, prepare a Solução A: Adicione 12 mL de gema de ovo e 36 mL de solução naCl de 0,86%.
- Prepare a solução B: Misture 300 mg de agarose em 50 mL de tampão (50 mM Tris, pH 7.5), filtro através de um filtro de 0,22 μm. Aqueça a solução em um micro-ondas até ferver. Esfrie em água morna para atingir 43-45°C.
- Prepare a solução C: Prepare 10 mM CaCl2 e filtre-o com um filtro de 0,22 μm.
- Preparar a solução D: 10 mg de rhodamina 6G em 1 mL de água destilada.
- Em condições estéreis (capô de fluxo laminar), adicione 500 μL de soluções A e C à solução B e 100 μL de solução D, misture e despeje 25 mL em cada placa de Petri (90 x 15 mm).
- Aguarde a solução para solidificar em condições estéreis (30 min).
- Faça poços (~2-3 mm de diâmetro) com um tubo fino.
- Adicione um total de 20 μL de tampão fosfato (controle negativo) em um poço e 20 μL de uma fosfolipase determinada (controle positivo) em outro poço.
- Coloque diferentes quantidades da proteína extrato bruto, 5, 15, 25, 35, 45 μg, nos poços restantes, cada um em um volume final de 20 μL.
- Aguarde o ágar para adsorb todas as amostras (30 min).
- Incubar a 37 °C por 20 h.
- Se um halo se formar ao redor do poço, isso indica atividade de fosfolipase. Meça a auréola formada com uma pinça vernier.
NOTA: Realize o experimento em triplicado (passos de 9.1-9.14).
Resultados
Os resultados representativos do protocolo utilizado para a obtenção do extrato bruto da anêmona do mar mostraram que a combinação de duas técnicas (agitação e ciclos de congelamento e descongelamento) produziu uma descarga eficiente de nematocistos, e a quantidade total de proteína foi de 500 mg (8 mg/mL) (Figura 3).
A complexidade proteica do extrato bruto poderia ser observada a partir de 10 kDa e maior que 250 kDa através da eletroforese SDS-PAGE. Al...
Discussão
A alta demanda por novos compostos com aplicações em diferentes campos da ciência e da indústria levou ao estudo do veneno. O veneno representa uma rica fonte de moléculas que serve como modelo para a geração de novas ferramentas moleculares. No entanto, a complexidade desses venenos requer a implementação e combinação de vários métodos para obtê-los e estudá-los.
Aqui, mostramos um método para obter e analisar o veneno da anêmona do mar Anthopleura dowii, Verrill 186...
Divulgações
Os autores declaram que não têm interesse em relação à publicação deste artigo.
Agradecimentos
Este trabalho foi apoiado pelo Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT), com um número de subvenção IT200819. Os autores reconhecem a Tom Musselman, Edição de Papel de Rock, LLC, por verificar a gramática inglesa deste manuscrito; e assistência técnica de Samanta Jiménez (CICESE, Ensenada) e Juan Manuel Barbosa Castillo (Instituto de Fisiología Celular, UNAM). Agradecemos também ao Dr. Augusto César Lizarazo Chaparro (CEPIPSA) pela obtenção de sangue de ovelha. Agradecemos especialmente ao Dr. José Saniger Blesa, ICAT-UNAM, pelas instalações em seu laboratório para a gravação de vídeo.
Materiais
| Name | Company | Catalog Number | Comments |
| 15 mL conical centrifuge tube | Corning | 430766 | |
| 2-Bromophenol blue | Sigma | B75808 | |
| 2-mercaptoetanol | Sigma-Aldrich | M6250-100ML | |
| 50 mL conical centrifuge tubes | Corning | 430828 | |
| Acetic Acid Glacial | J.T. Baker | 9515-03 | |
| Acrylamide | Promega | V3115 | |
| Agarose | Promega | V3125 | |
| Bisacrylamide | Promega | V3143 | |
| Bovine Serum Albumin Fraction V | Sigma | A3059-100G | |
| Bradford Protein Assays | Bio-Rad | 5000006 | |
| Calcium chloride | Sigma-Aldrich | C3306 | |
| Cell culture plates 96 well, V-bottom | Corning | 3894 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge tubes | Corning | CLS430829 | |
| ChemiDoc MP system | Bio-Rad | 1708280 | |
| Citric acid | Sigma-Aldrich | 251275 | |
| Clear flat.bottom 96-Well Plates | Thermo Scientific | 3855 | |
| Coomassie Brilliant Blue G-250 | Bio-Rad | #1610406 | |
| Coomassie brilliant blue R-250 | Bio-Rad | 1610400 | |
| Dextrose | J.T. Baker | 1916-01 | |
| Ductless Enclosure | Labconco | Vertical | https://imagej.nih.gov/ij ImageJ 1.53c |
| Gel Doc EZ | Bio Rad. | Gel Documentation System | |
| Glycerol | Sigma-Aldrich | G5516-4L | |
| Hemocytometer | Marienfeld | 650030 | |
| ImageJ (Software) | NIH, USA | Version 1.53c | |
| Incubator 211 | Labnet | I5211 DS | |
| Methanol | J.T. Baker | 9049-03 | |
| Mini-PROTEAN tetra cell | Bio-Rad | 1658000EDU | |
| Na2HPO4 | J.T. Baker | 3824-01 | |
| NaCl | J.T. Baker | 3624-01 | |
| NaH2PO4.H2O | J.T. Baker | 3818-05 | |
| Origin software | version 9 | To design the plot with sigmoidal adjustments | |
| Petridish | Falcon | 351007 | |
| Pipetman kit | Gilson | F167380 | |
| Precast mini gel | BioRad | 1658004 | |
| Prestained Protein Ladder | Thermo Scientific | 26620 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protein Assay Dye Reagent Concentrate | Bio-Rad | 5000006 | |
| Rhodamine 6G | Sigma-Aldrich | 252433 | |
| SDS | Sigma-Aldrich | L4509 | |
| Sodium citrate dihydrate | JT Baker | 3646-01 | |
| Spectrophotometer | THERMO SCIENTIFIC | G10S UV-VIS | |
| Tris Base | Sigma-Aldrich | 77-86-1 | |
| Volt Power Supply | Hoefer | PS300B |
Referências
- Frazão, B., Vasconcelos, V., Antunes, A. Sea anemone (Cnidaria, Anthozoa, Actiniaria) toxins: an overview. Marine Drugs. 10 (8), 1812-1851 (2012).
- Jayathilake, J. M. N. J., Gunathilake, K. V. K. Cnidarian toxins: recent evidences for potential therapeutic uses. The European Zoological Journal. 87 (1), 708-713 (2020).
- Ramírez-Carreto, S., Miranda-Zaragoza, B., Rodríguez-Almazán, C. Actinoporins: From the structure and function to the generation of biotechnological and therapeutic tools. Biomolecules. 10 (4), 539 (2020).
- Fautin, D. G. Structural diversity, systematics, and evolution of cnidae. Toxicon. Official Journal of the International Society on Toxinology. 54 (8), 1054-1064 (2009).
- Moran, Y., et al. Analysis of soluble protein contents from the nematocysts of a model sea anemone sheds light on venom evolution. Marine Biotechnology. 15 (3), 329-339 (2013).
- Moran, Y., et al. Neurotoxin localization to ectodermal gland cells uncovers an alternative mechanism of venom delivery in sea anemones. Proceedings of the Royal Society B: Biological Sciences. 279 (1732), 1351-1358 (2012).
- Grabski, A. C. Advances in preparation of biological extracts for protein purification. Methods in Enzymology. 463, 285-303 (2009).
- Bulati, M., et al. Partially purified extracts of sea anemone Anemonia viridis affect the growth and viability of selected tumour cell lines. BioMed Research International. 2016, 3849897 (2016).
- Orts, D. J. B., et al. Biochemical and electrophysiological characterization of two sea anemone type 1 potassium toxins from a geographically distant population of Bunodosoma caissarum. Marine Drugs. 11 (3), 655-679 (2013).
- Hader, D., Erzinger, G. . Bioassays: Advanced Methods and Applications. , (2017).
- Anderluh, G., Macek, P. Cytolytic peptide and protein toxins from sea anemones (Anthozoa: Actiniaria). Toxicon: Official Journal of the International Society on Toxinology. 40 (2), 111-124 (2002).
- Nevalainen, T. J., et al. Phospholipase A2 in cnidaria. Comparative Biochemistry and Physiology. Part B, Biochemistry & Molecular Biology. 139 (4), 731-735 (2004).
- Razpotnik, A., et al. A new phospholipase A2 isolated from the sea anemone Urticina crassicornis - its primary structure and phylogenetic classification. The FEBS Journal. 277 (12), 2641-2653 (2010).
- Dennis, E. A., Cao, J., Hsu, Y. -. H., Magrioti, V., Kokotos, G. Phospholipase A2 enzymes: Physical structure, biological function, disease implication, chemical inhibition, and therapeutic intervention. Chemical Reviews. 111 (10), 6130-6185 (2011).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Kwon, Y. -. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, 8663 (2015).
- Eno, A. E., Konya, R. S., Ibu, J. O. Biological properties of a venom extract from the sea anemone, Bunodosoma cavernata. Toxicon: Official Journal of the International Society on Toxinology. 36 (12), 2013-2020 (1998).
- Morales-Landa, J. L., et al. Antimicrobial, antiprotozoal, and toxic activities of Cnidarian extracts from the Mexican Caribbean Sea. Pharmaceutical Biology. 45 (1), 37-43 (2007).
- Sánchez-Rodríguez, J., Cruz-Vazquez, K. Isolation and biological characterization of neurotoxic compounds from the sea anemone Lebrunia danae (Duchassaing and Michelotti, 1860). Archives of Toxicology. 80 (7), 436-441 (2006).
- de Oliveira, J. S., et al. Caissarolysin I (Bcs I), a new hemolytic toxin from the Brazilian sea anemone Bunodosoma caissarum: purification and biological characterization. Biochimica Et Biophysica Acta. 1760 (3), 453-461 (2006).
- Norton, R. S., et al. Purification and characterisation of proteins with cardiac stimulatory and haemolytic activity from the anemone Actinia tenebrosa. Toxicon: Official Journal of the International Society on Toxinology. 28 (1), 29-41 (1990).
- Gondran, M., Eckeli, A. L., Migues, P. V., Gabilan, N. H., Rodrigues, A. L. S. The crude extract from the sea anemone, Bunodosoma caissarum elicits convulsions in mice: possible involvement of the glutamatergic system. Toxicon: Official Journal of the International Society on Toxinology. 40 (12), 1667-1674 (2002).
- Dion, A. S., Pomenti, A. A. Ammoniacal silver staining of proteins: mechanism of glutaraldehyde enhancement. Analytical Biochemistry. 129 (2), 490-496 (1983).
- Ramírez-Carreto, S., et al. Identification of a pore-forming protein from sea anemone Anthopleura dowii Verrill (1869) venom by mass spectrometry. The Journal of Venomous Animals and Toxins Including Tropical Diseases. 25, 147418 (2019).
- Nelson, G. J. Studies on the lipids of sheep red blood cells. I. Lipid composition in low and high potassium red cells. Lipids. 2 (1), 64-71 (1967).
- Ramírez-Carreto, S., et al. Transcriptomic and proteomic analysis of the tentacles and mucus of Anthopleura dowii Verrill, 1869. Marine Drugs. 17 (8), 436 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados