Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Identifizierung der hämolytischen und Phospholipase-Aktivität in Rohextrakten aus Seeanemonen durch einfache Bioassays
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein Protokoll zur Gewinnung von Rohgiftextrakt aus Seeanemone und zum Nachweis seiner hämolytischen und Phospholipase-Aktivität.
Zusammenfassung
Die Zusammensetzung des Seeanemonengifts umfasst Polypeptid- und Nichtproteinmoleküle. Zytolytische Komponenten haben ein hohes biotechnologisches und biomedizinisches Potenzial für die Entwicklung neuer molekularer Werkzeuge. Seeanemonengift lokalisiert sich in Drüsenzellen aus Ektoderm und subzellulären Strukturen, die Nematozysten genannt werden, die beide im gesamten Meeresanemonenkörper verteilt sind. Diese Eigenschaft bringt Herausforderungen mit sich, da die Zellen und die Nematozyste lysiert werden müssen, um die Giftkomponenten mit anderen ungiftigen Molekülen freizusetzen. Daher wird das Gift zunächst aus einem Rohextrakt (Mischung aus verschiedenen und vielfältigen Molekülen und Gewebeablagerungen) gewonnen. Der nächste Schritt besteht darin, Polypeptide mit spezifischen Bioaktivitäten nachzuweisen. Hier beschreiben wir eine effiziente Strategie zur Gewinnung des Ozeananemonen-Rohölextrakts und des Bioassays, um das Vorhandensein von Cytolysinen zu identifizieren. Der erste Schritt beinhaltet kostengünstige und unkomplizierte Techniken (Rühr- und Frost-Tau-Zyklus) zur Freisetzung von Cytolysinen. Wir erhielten die höchste zytolytische Aktivität und das höchste Protein (~ 500 mg Protein aus 20 g Trockengewicht). Als nächstes wurde die Polypeptidkomplexität des Extrakts durch SDS-PAGE-Gel analysiert, das Proteine mit Molekulargewichten zwischen 10 kDa und 250 kDa nachweist. Im hämolytischen Assay verwendeten wir rote Blutkörperchen von Schafen und bestimmten HU50 (11,1 ± 0,3 μg/ml). Im Gegensatz dazu wurde das Vorhandensein von Phospholipasen im Rohextrakt unter Verwendung von Eigelb als Substrat in einem festen Medium mit Agarose bestimmt. Insgesamt verwendet diese Studie ein effizientes und kostengünstiges Protokoll zur Herstellung des Rohextrakts und wendet replizierbare Bioassays an, um Cytolysine, Moleküle mit biotechnologischen und biomedizinischen Interessen, zu identifizieren.
Einleitung
Meerestiere sind eine reiche Quelle biologisch aktiver Verbindungen. In den letzten Jahrzehnten hat die Zusammensetzung des Seeanemonengifts wissenschaftliche Aufmerksamkeit erregt, da es eine Vielzahl von Polypeptiden mit hämolytischer, zytotoxischer, enzymatischer (Phospholipase, Protease, Chitinase) und neurotoxischer Aktivität und hemmenden Wirkungen auf die proteolytische Aktivitätumfasst 1. Darüber hinaus sind diese Polypeptide potenzielle Quellen für die Entwicklung molekularer Werkzeuge im biotechnologischen und therapeutischen Einsatz 2,3.
Es gibt nur wenige Berichte über Seeanemonengift und seine molekularen Komponenten aufgrund der Komplexität der Gewinnung des Giftes, sogar der Isolierung und Charakterisierung von Toxinen. Die in den Berichten verwendeten Extraktionsmethoden beinhalteten die Lyse und Entleerung des Inhalts von Zellen, die mit der Giftproduktion zusammenhängen und nicht damit zusammenhängen1.
Ein besonderes Merkmal bei allen Nesseltieren ist das Fehlen eines Systems zur Produktion und Freisetzung des Giftes, das in einer einzigen anatomischen Region zentralisiert ist. Stattdessen sind die Nematozysten Strukturen, die das Gift 4,5 halten. Andere Arten von Zellen, sogenannte epidermale Drüsenzellen, sezernieren ebenfalls Toxine und sind auch im ganzen Körper der Seeanemonenverteilt 6.
Die erste und wichtigste Herausforderung bei der Gewinnung des Giftes ist die Erzeugung eines Extrakts mit ausreichender Manipulation in nachfolgenden Prozessen, ohne die Inaktivierung oder den Abbau von labilen Proteinen. Als nächstes müssen die Zellen lysiert werden, und die Komponenten - in diesem Fall Polypeptide - müssen effizient und schnell extrahiert werden, wobei Proteolyse und Hydrolyse vermieden werden, während andere zelluläre Komponenten eliminiertwerden 7.
Verschiedene Methoden werden verwendet, um den Rohextrakt einer Seeanemone zu erhalten; Einige beinhalten das Opfern des Organismus, während andere es erlauben, ihn am Leben zu erhalten. Methoden, die die Verwendung des gesamten Körpers des Organismus implizieren, ermöglichen die Freisetzung der meisten Toxine aus dem Gift8, verglichen mit Methoden, die Organismen am Leben erhalten, die nur einige Bestandteile des Giftesextrahieren 9. Die Herstellung eines Extrakts erfordert die Bewertung des Vorhandenseins und der Wirksamkeit einer Substanz von Interesse durch einen spezifischen Bioassay, der Strategien zur Beobachtung der pharmakologischen Wirkungen durch In-vivo- oder In-vitro-Methoden 10 umfasst.
Seeanemonengift enthält zytolytische Polypeptide, porenbildende Toxine (PFTs)11 und Phospholipasen12; Diese Moleküle sind Modelle bei der Untersuchung der Protein-Lipid-Interaktion, molekulare Werkzeuge in der Krebstherapie und Biosensoren auf der Grundlage von Nanopore3. Die Klassifizierung von Seeanemonen-PFTs erfolgt nach ihrer Größe oder ihrem Molekulargewicht, von 5 kDa bis 80 kDa. Das 20 kDa PFT, das am meisten untersuchte und als Actinoporine11 bekannte PFT, ist von besonderem Interesse wegen seines biomedizinischen Potenzials bei der Entwicklung molekularer Werkzeuge für mögliche Anwendungen wie krebshemmende, antimikrobielle und nanoporenbasierte Biosensoren. Ein anderes Cytolysin, einschließlich Phospholipasen, insbesondere Phospholipase A2 (PLA2)13, setzt eine Fettsäure frei und hydrolysiert Phospholipide, wodurch die Zellmembran destabilisiert wird. Aufgrund dieses Wirkmechanismus verspricht PLA2 ein wesentliches Modell für die Erforschung und Anwendung bei entzündlichen Erkrankungen zu sein. Es könnte als Modell für Untersuchungen des Lipidverhaltens in der Zellmembrandienen 14.
Hier beschreiben wir ein effizientes Protokoll zur Gewinnung des Rohölextrakts aus der Seeanemone Anthopleura dowii Verrill, 1869, und zum Nachweis von Hämolysinen und Phospholipasen. Beide sind relevante Toxine, die als Vorlage für die Entwicklung neuer molekularer Werkzeuge verwendet werden könnten.
Protokoll
Die Seeanemonen wurden nach Richtlinien der Nationalen Kommission für Aquakultur, Fischerei und Ernährung der mexikanischen Bundesregierung (Genehmigungsnummer PPF / DGOPTA 07332.250810.4060) gesammelt. Die Bioethikkommission des Instituts für Biotechnologie der Nationalen Autonomen Universität von Mexiko genehmigte alle Experimente mit Seeanemonen. Die Schafblutprobe wurde am Zentrum für praktische Lehre und Forschung in der Tierproduktion und -gesundheit (CEPIPSA, Nationale Autonome Universität von Mexiko) gekauft.
1. Sammlung von Organismen
- Sammle Seeanemone A. dowii.
HINWEIS: Hier wurden die Organismen während der Ebbe in der Gezeitenzone vor der Küste von Ensenada Baja, Kalifornien, Mexiko gesammelt (Abbildung 1A, B). - Transportieren Sie die Organismen in Behältern mit Meerwasser ins Labor (Abbildung 1C).
- Reinigen Sie die Organismen im Labor mit destilliertem Wasser von Hand, um die großen Substratpartikel zu entfernen, die am Körper haften.
- Die Organismen sofort bei -80 °C in einem Ultra-Gefrierschrank für 72 h einfrieren.
- Geben Sie die Organismen in spezielle Gläser zur Gefriertrocknung mit Lyophilisationsbedingungen von -20 ° C, 0,015 psi für 48 h.
- Lagern Sie die lyophilisierten Organismen bei -20 °C bis zur Verwendung.
2. Hydratation des Gewebes
- Rekonstituieren Sie 20 g Trockengewicht lyophilisierter Organismen mit 60 ml (1/3 w/v) 50 mM Natriumphosphatpuffer, pH 7,5 und 1 Inhibitor-Cocktailtablette (Materialtabelle).
- Rühren Sie die Probe in einem Magnetrührer bei ~800 U/min, 4 °C, 12 h lang kontinuierlich um.
3. Freisetzung von Toxinen
- Um Zelllyse und Nematozystenausfluss zu induzieren, frieren Sie die Probe bei -20 ° C für 12 h ein und legen Sie den Behälter dann bei Raumtemperatur (RT) in Wasser, bis er bis zu 90% auftaut. Wiederholen Sie diesen Vorgang dreimal.
HINWEIS: Die Probe taut nicht zu 100% auf; Es ist wichtig, die Probe bei ~ 4 ° C zu halten, um den Proteinabbau zu verhindern. - Beobachten Sie die Nematozyste, die unter einem konfokalen Mikroskop entladen wird. Nehmen Sie 10 μL der Probe auf einen Objektträger und legen Sie ein Deckglas darüber. Beobachten Sie unter einem konfokalen Mikroskop bei 100-facher und 60-facher Vergrößerung. Hier war ein Farbstoff nicht notwendig.
- Verarbeiten Sie die im konfokalen Mikroskop aufgenommenen Bilder in der ImageJ-Software (Version 1.53c, Wayne Rasband, National Institutes of Health, USA, https://imagej.nih.gov/ij).
- Wenn sich der Tubulus in der Nematozyste abwickelt und sich außerhalb befindet, werden die Nematozysten entladen; Fahren Sie dann mit dem nächsten Schritt fort. Andernfalls führen Sie nur noch einen weiteren Frost-Tau-Zyklus durch.
- Um den Extrakt zu klären, zentrifugieren Sie die Probe bei 25.400 x g für 40 min, 4 °C, gewinnen Sie den Überstand und zentrifugieren Sie ihn erneut. Wiederholen Sie diesen Schritt 3-4 Mal.
- Holen Sie sich den Überstand und entsorgen Sie das Pellet. Der Überstand oder Rohextrakt enthält die Giftbestandteile (Toxine) und andere Zellmoleküle wie Lipide, Kohlenhydrate und Nukleinsäuren.
4. Quantifizierung des Gesamtproteins
- Bestimmen Sie die Proteinkonzentration des Rohextrakts mit einem kommerziellen Bradford colorimetrischen Assay15-Kit (Table of Materials).
- Verdünnen Sie das Farbstoffreagenz (ein Teil des Farbstoffs mit vier Teilen destilliertem Wasser).
- Herstellung der Lösung für Rinderserumalbumin (BSA) Fraktion V (Tabelle der Materialien) in einer Konzentration von 1 mg/ml im Phosphatpuffer (50 mM Natriumphosphat, pH 7,5).
- Bereiten Sie die in Tabelle 1 aufgeführten Lösungen in dreifacher Ausfertigung vor.
- Mischen Sie jede Lösung kräftig in einem Shaker für 4 s.
- Inkubieren Sie die Proben für 5 Minuten bei RT, ohne zu schütteln.
- Messen Sie die Absorption (AU) bei 595 nm.
- Zeichnen Sie die Mittelwerte der BSA-Absorptionsraten der Probe (AU vs. Proteinkonzentration) auf und bestimmen Sie die Gleichung des Diagramms (Gleichung 1).
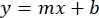 Gleichung 1
Gleichung 1
wobei y die Absorption, m die Steigung der Linie, x die Proteinkonzentration und b der y-Abschnitt ist. Um die Rohölextraktkonzentration zu berechnen, lösen Sie für das x wie folgt: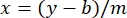 Gleichung 2
Gleichung 2
5. Polypeptid-Giftkomplexität bestimmen
- Montieren Sie den Glasbehälter für die vertikale Gelelektrophorese gemäß dem Handbuch.
- Acrylamidmischung vorbereiten (30%): 29,2 g Acrylamid, 0,8 g Bis-Acrylamid, das endgültige Volumen von 100 ml. Filtern Sie die Lösung mit einer 0,45 μm Membran. Lagern Sie die Lösung in einer dunklen Flasche bei 4 °C. Bereiten Sie dann die Mischung für das auflösende Gel vor (Tabelle 2).
- Das SDS-PAGE Gel besteht aus einem auflösenden Gel und einem Stapelgel. Bereiten Sie die Gele gemäß den in Tabelle 2 definierten Lösungsvolumina vor. Achten Sie während der Zubereitung darauf, dass jede Mischung bei 4 °C gehalten wird, um die Polymerisationszeit zu verkürzen.
HINWEIS: Das Volumen jeder Mischung variiert je nach Marke der verwendeten Ausrüstung; Für dieses Protokoll entsprechen die Volumina der Herstellung von 15% Acrylamid Gel, 0,75 mm dick, für eine vertikale Elektrophoresekammer (Materialverzeichnis). - Nachdem das Acrylamid polymerisiert ist, ~ 10 min, fügen Sie die Mischung aus Stapelgel hinzu.
- Bereiten Sie die Mischung für das Stapelgel vor, fügen Sie sie sofort zu den Glasplatten hinzu und legen Sie einen Kamm, um die Vertiefungen zum Laden der Proben zu bilden.
- Sobald das Stapelgel polymerisiert ist (~ 15 min), entfernen Sie den Kamm und legen Sie den Glasbehälter in eine Kammer für die vertikale Elektrophorese.
- Geben Sie 200 ml Elektrodenpuffer (0,25 M Tris, 0,192 M Glycin, 0,1% SDS) in die Elektrophoresekammer.
- Analysieren Sie den Rohextrakt: Mischen Sie 30 μg Rohextrakt mit 5 μL Proteinladepuffer (25% Glycerin, 15% Natriumdodecylsulfat, 25% 2-Mercaptoethanol, 0,125 mM Tris pH 7, Bromphenolblau 0,1 mg / ml), um Proteine zu denaturieren. Das Endvolumen muss <50 μL betragen.
- Bei 90 °C 5 min erhitzen, abkühlen und 3 s bei 1.400 x g zentrifugieren.
- Laden Sie die Proben in die Vertiefungen des Stapelgels. Laden Sie eine Standard-Molekulargewichtsleiter.
- Elektrophorese mit einer Konstante von 25 mA für 1 h 30 min.
- Entfernen Sie die Glasbehälter aus der Elektrophoresekammer, um mit der Färbung der Gelproteine fortzufahren.
6. Proteinfärbung
- Spülen Sie das Gel in 50 ml destilliertem Wasser ab, um Ablagerungen von der Pufferelektrode zu entfernen.
- Entsorgen Sie das Wasser.
- Um die Diffusion der Gelproteine zu vermeiden, fügen Sie die Fixierlösung hinzu (60 ml Ethanol, 20 ml Essigsäure und 20 ml destilliertes Wasser, Endvolumen 100 ml). Inkubieren Sie bei RT für 40 min mit Schütteln bei 80 U / min. Dekantieren Sie die Lösung.
- Fügen Sie die Proteinvernetzungslösung hinzu (15 ml Ethanol, 0,25 ml Glutaraldehyd, 50 ml destilliertes Wasser, Endvolumen 65,25 ml) und inkubieren Sie für 30 min bei RT mit Schütteln bei 80 U / min. Entfernen Sie die Lösung.
- Waschen Sie das Gel mit 100 ml destilliertem Wasser für 5 min und entfernen Sie das Wasser. Wiederholen Sie diesen Vorgang viermal.
- Führen Sie eine Proteinfärbung mit 50 ml Coomassie brilliant blue R-250-gefilterter Lösung durch (40% Methanol, 10% Essigsäure, 50% destilliertes Wasser, 0,1% Farbstoff, Endvolumen 100 ml), Rühren bei 60 U / min in einem Labordrehoszillator bei RT für 15 min.
- Stellen Sie die Lösung wieder her, mit der andere Gele gefärbt werden können.
- Um den überschüssigen Farbstoff zu entfernen, fügen Sie eine Färbelösung hinzu (40 ml Methanol, 10 ml Essigsäure, 50 ml destilliertes Wasser). Rühren Sie weiter (80 U / min) bei RT, bis sich die Banden (gefärbte Proteine) sichtbar machen.
- Bewahren Sie das Gel in 50 ml destilliertem Wasser auf und scannen Sie das Bild in einem Gel-Dokumentationssystem.
7. Rote Blutkörperchenlösung vorbereiten
- Extrahieren Sie 1 ml Blut aus der Halsvene eines Schafes.
- Entfernen Sie die Nadel aus der Spritze und lassen Sie das Blut sofort in ein Röhrchen mit 50 ml Alsing-Lösung (0,002 M Zitronensäure, 0,07 M NaCl, 0,1 M Dextrose und 0,027 M Natriumcitrat als Antikoagulans, pH 7,4) ab.
- Kehren Sie die Lösung dreimal langsam um.
- Für den Transfer ins Labor bei 4 °C aufbewahren.
- Bei 804 x g für 5 min bei 4 °C zentrifugieren und den Überstand entsorgen.
- Resuspendiert das Pellet in 30 ml Alver-Lösung. Fügen Sie die Lösung hinzu, indem Sie sie entlang der Innenwände des Schlauches schieben und mit einer Pasteur-Kunststoffpipette die Zellen langsam aussetzen. Erneut bei 804 x g für 5 min, 4 °C zentrifugieren. Wiederholen Sie diesen Vorgang, bis der Überstand geklärt ist.
- Fügen Sie 15 ml Alver-Lösung hinzu; Das Pellet wird langsam mit einer Pasteur-Kunststoffpipette resuspendiert.
- Halten Sie das Endvolumen der Alver-Lösung aufrecht, um eine Konzentration von ca. 2 x 106 Zellen/ml zu erreichen. Verwenden Sie dann eine Neubauer-Kammer oder ein Hämozytometer, um die Zellen zu zählen.
HINWEIS: Zählen Sie die Erythrozyten im Hämacytometer-Objektträger (Verbesserter Neubauer). Der Schlitten ist ein 30 mm x 70 mm x 4 mm Schlitten. Die Mitte hat zwei silberne Fußplatten (Abbildung 2A) (ein oberes und ein unteres Gitter), jeweils 3 mm x 3 mm, und zwei Kanäle an den Seiten des Gitters. Das Zählen von Zellen befindet sich in den Eck- und Mittelquadraten (rote Quadrate in Abbildung 2B). Die optimale Konzentration der Zellen zum Zählen sollte in der Größenordnung von 10bis 6 Zellen liegen. Die zentrale Box hat eine Oberfläche von 0,1 cm2, was 0,0001 ml entspricht. - Legen Sie ein Deckglas über die Folie.
- Legen Sie 10 μL der Probe zwischen den Objektträger und das Deckglas und warten Sie, bis die Probe ausgezahlt ist.
- Führen Sie in einem Lichtmikroskop (Vergrößerung 60x) die Zählung in der ersten quadratischen Box durch. Zählen Sie nur die Zellen im mittleren Raster und diejenigen, die den oberen oder linken Rand des Felds berühren.
- Bestimmen Sie die Zellkonzentration mit der folgenden Gleichung:
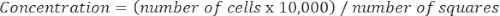 Gleichung 3
Gleichung 3
Wenn die Probe sehr konzentriert ist und eine Verdünnung erforderlich ist, verwenden Sie die folgende Gleichung: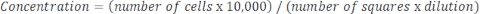 Gleichung 4
Gleichung 4
HINWEIS: Wenn Sie die Erythrozytenlösung für den Hämolysetest einnehmen, homogenisieren Sie die Lösung langsam mit einer Pasteur-Pipette aus Kunststoff. - Führen Sie die Schritte dreifach aus, um Fehler zu reduzieren.
8. Hämolyse-Assay
- Die in Tabelle 3 angegebenen Lösungsgemische (in dreifacher Ausfertigung) sind in konischen 96-Well-Platten herzustellen.
HINWEIS: Mischen Sie die Erythrozyten langsam, um eine homogene Suspension aufrechtzuerhalten. - 1 h bei 37 °C inkubieren, ohne zu schütteln.
- Zentrifen Sie die Platte bei 804 x g für 5 min bei 4 °C.
- Stellen Sie den Überstand in einer weiteren 96-Well-Platte mit einem flachen Boden wieder her.
- Wenn der Rohextrakt Cytolysine enthält, werden Erythrozyten lysieren und Hämoglobin freisetzen. Lesen Sie die Absorption bei 415 nm.
- Berechnen Sie den Hämolyseanteil mit der folgenden Gleichung:
% Hämolyse = ((Acrude extract-AAlsever buffer) / (AH2O-AAlsever)) x 100
HINWEIS: Rohextrakt, AAlsever-Puffer und AH2O entsprechen der Absorption von Erythrozyten mit dem Rohextrakt, der Absorption von Alsever-Lösung bzw. der Absorption von destilliertem Wasser. - Führen Sie vor und nach jedem Gefrier- und Auftauzyklus einen Hämolysetest mit 50 μg Gesamtprotein aus dem Rohextrakt durch.
- Bestimmen Sie die Anzahl der Gefrier- und Auftauzyklen, die erforderlich sind, um eine 100% ige hämolytische Aktivität zu erreichen.
- Berechnen Sie die Menge an Extrakt, die Hämolyse in 50% der Erythrozyten (HU50) produziert; Befolgen Sie das gleiche Protokoll, das in Tabelle 3 beschrieben ist, erhöhen Sie die Menge an Seeanemonenrohextrakt und entwerfen Sie das Diagramm mit sigmoidalen Anpassungen unter Verwendung geeigneter Software (z. B. Herkunft, Materialverzeichnis).
9. Phospholipase-Assay
- Waschen Sie ein Hühnerei mit 1% SDS in destilliertem Wasser.
- Trennen Sie das Eigelb unter sterilen Bedingungen vom Eiweiß.
- Bereiten Sie 50 ml 0,86%ige NaCl-Lösung vor und filtern Sie durch einen 0,22-μm-Filter. Dann bereiten Sie Lösung A vor: Fügen Sie 12 ml Eigelb und 36 ml 0,86% ige NaCl-Lösung hinzu.
- Bereiten Sie Lösung B vor: Mischen Sie 300 mg Agarose in 50 ml Puffer (50 mM Tris, pH 7,5), filtrieren Sie durch einen 0,22 μm Filter. Erhitzen Sie die Lösung in einer Mikrowelle bis zum Sieden. In warmem Wasser auf 43-45°C abkühlen.
- Lösung C vorbereiten: Bereiten Sie 10 mM CaCl2 vor und filtern Sie es mit einem 0,22 μm Filter.
- Bereiten Sie Lösung D vor: 10 mg Rhodamin 6G in 1 ml destilliertem Wasser.
- Unter sterilen Bedingungen (Laminar-Flow-Haube) 500 μL der Lösungen A und C zu Lösung B und 100 μL Lösung D geben, mischen und 25 ml in jede Petrischale (90 x 15 mm) gießen.
- Warten Sie, bis die Lösung unter sterilen Bedingungen (30 min) erstarrt ist.
- Machen Sie Vertiefungen (~ 2-3 mm Durchmesser) mit einem dünnen Rohr.
- Insgesamt 20 μL Phosphatpuffer (Negativkontrolle) in eine Vertiefung und 20 μL einer bestimmten Phospholipase (Positivkontrolle) in eine andere Vertiefung geben.
- Legen Sie unterschiedliche Mengen des Rohextraktproteins, 5, 15, 25, 35, 45 μg, in die verbleibenden Vertiefungen, jeweils in einem Endvolumen von 20 μL.
- Warten Sie, bis der Agar alle Proben adsorbiert hat (30 min).
- 20 h bei 37 °C inkubieren.
- Wenn sich ein Halo um das Bohrloch bildet, deutet dies auf eine Phospholipase-Aktivität hin. Messen Sie den Halo, der mit einem Vernier-Bremssattel gebildet wird.
HINWEIS: Führen Sie das Experiment dreifach durch (Schritte von 9.1-9.14).
Ergebnisse
Die repräsentativen Ergebnisse des Protokolls, das zur Gewinnung des Rohölextrakts aus Seeanemone verwendet wurde, zeigten, dass die Kombination von zwei Techniken (Rühren und Zyklen des Einfrierens und Auftauens) zu einer effizienten Entladung von Nematozysten führte und die Gesamtmenge an Protein 500 mg (8 mg / ml) betrug (Abbildung 3).
Die Proteinkomplexität des Rohextrakts konnte von 10 kDa und größer als 250 kDa durch SDS-PAGE-Elektrophorese beobachtet...
Diskussion
Die hohe Nachfrage nach neuen Verbindungen mit Anwendungen in verschiedenen Bereichen der Wissenschaft und Industrie hat zur Erforschung von Gift geführt. Venom stellt eine reiche Quelle von Molekülen dar, die als Vorlage für die Generierung neuer molekularer Werkzeuge dient. Die Komplexität dieser Gifte erfordert jedoch die Implementierung und Kombination verschiedener Methoden, um sie zu erhalten und zu untersuchen.
Hier zeigen wir eine Methode zur Gewinnung und Analyse des Giftes der
Offenlegungen
Die Autoren erklären, dass sie kein konkurrierendes Interesse an der Veröffentlichung dieses Papiers haben.
Danksagungen
Diese Arbeit wurde von Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT) mit der Fördernummer IT200819 unterstützt. Die Autoren danken Tom Musselman, Rock Paper Editing, LLC, für die Überprüfung der englischen Grammatik dieses Manuskripts; und die technische Hilfe von Samanta Jiménez (CICESE, Ensenada) und Juan Manuel Barbosa Castillo (Instituto de Fisiología Celular, UNAM). Wir danken auch Dr. Augusto César Lizarazo Chaparro (CEPIPSA) für die Gewinnung von Schafblut. Wir danken insbesondere Dr. José Saniger Blesa, ICAT-UNAM, für die Einrichtungen in seinem Labor für die Videoaufzeichnung.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL conical centrifuge tube | Corning | 430766 | |
| 2-Bromophenol blue | Sigma | B75808 | |
| 2-mercaptoetanol | Sigma-Aldrich | M6250-100ML | |
| 50 mL conical centrifuge tubes | Corning | 430828 | |
| Acetic Acid Glacial | J.T. Baker | 9515-03 | |
| Acrylamide | Promega | V3115 | |
| Agarose | Promega | V3125 | |
| Bisacrylamide | Promega | V3143 | |
| Bovine Serum Albumin Fraction V | Sigma | A3059-100G | |
| Bradford Protein Assays | Bio-Rad | 5000006 | |
| Calcium chloride | Sigma-Aldrich | C3306 | |
| Cell culture plates 96 well, V-bottom | Corning | 3894 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge tubes | Corning | CLS430829 | |
| ChemiDoc MP system | Bio-Rad | 1708280 | |
| Citric acid | Sigma-Aldrich | 251275 | |
| Clear flat.bottom 96-Well Plates | Thermo Scientific | 3855 | |
| Coomassie Brilliant Blue G-250 | Bio-Rad | #1610406 | |
| Coomassie brilliant blue R-250 | Bio-Rad | 1610400 | |
| Dextrose | J.T. Baker | 1916-01 | |
| Ductless Enclosure | Labconco | Vertical | https://imagej.nih.gov/ij ImageJ 1.53c |
| Gel Doc EZ | Bio Rad. | Gel Documentation System | |
| Glycerol | Sigma-Aldrich | G5516-4L | |
| Hemocytometer | Marienfeld | 650030 | |
| ImageJ (Software) | NIH, USA | Version 1.53c | |
| Incubator 211 | Labnet | I5211 DS | |
| Methanol | J.T. Baker | 9049-03 | |
| Mini-PROTEAN tetra cell | Bio-Rad | 1658000EDU | |
| Na2HPO4 | J.T. Baker | 3824-01 | |
| NaCl | J.T. Baker | 3624-01 | |
| NaH2PO4.H2O | J.T. Baker | 3818-05 | |
| Origin software | version 9 | To design the plot with sigmoidal adjustments | |
| Petridish | Falcon | 351007 | |
| Pipetman kit | Gilson | F167380 | |
| Precast mini gel | BioRad | 1658004 | |
| Prestained Protein Ladder | Thermo Scientific | 26620 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protein Assay Dye Reagent Concentrate | Bio-Rad | 5000006 | |
| Rhodamine 6G | Sigma-Aldrich | 252433 | |
| SDS | Sigma-Aldrich | L4509 | |
| Sodium citrate dihydrate | JT Baker | 3646-01 | |
| Spectrophotometer | THERMO SCIENTIFIC | G10S UV-VIS | |
| Tris Base | Sigma-Aldrich | 77-86-1 | |
| Volt Power Supply | Hoefer | PS300B |
Referenzen
- Frazão, B., Vasconcelos, V., Antunes, A. Sea anemone (Cnidaria, Anthozoa, Actiniaria) toxins: an overview. Marine Drugs. 10 (8), 1812-1851 (2012).
- Jayathilake, J. M. N. J., Gunathilake, K. V. K. Cnidarian toxins: recent evidences for potential therapeutic uses. The European Zoological Journal. 87 (1), 708-713 (2020).
- Ramírez-Carreto, S., Miranda-Zaragoza, B., Rodríguez-Almazán, C. Actinoporins: From the structure and function to the generation of biotechnological and therapeutic tools. Biomolecules. 10 (4), 539 (2020).
- Fautin, D. G. Structural diversity, systematics, and evolution of cnidae. Toxicon. Official Journal of the International Society on Toxinology. 54 (8), 1054-1064 (2009).
- Moran, Y., et al. Analysis of soluble protein contents from the nematocysts of a model sea anemone sheds light on venom evolution. Marine Biotechnology. 15 (3), 329-339 (2013).
- Moran, Y., et al. Neurotoxin localization to ectodermal gland cells uncovers an alternative mechanism of venom delivery in sea anemones. Proceedings of the Royal Society B: Biological Sciences. 279 (1732), 1351-1358 (2012).
- Grabski, A. C. Advances in preparation of biological extracts for protein purification. Methods in Enzymology. 463, 285-303 (2009).
- Bulati, M., et al. Partially purified extracts of sea anemone Anemonia viridis affect the growth and viability of selected tumour cell lines. BioMed Research International. 2016, 3849897 (2016).
- Orts, D. J. B., et al. Biochemical and electrophysiological characterization of two sea anemone type 1 potassium toxins from a geographically distant population of Bunodosoma caissarum. Marine Drugs. 11 (3), 655-679 (2013).
- Hader, D., Erzinger, G. . Bioassays: Advanced Methods and Applications. , (2017).
- Anderluh, G., Macek, P. Cytolytic peptide and protein toxins from sea anemones (Anthozoa: Actiniaria). Toxicon: Official Journal of the International Society on Toxinology. 40 (2), 111-124 (2002).
- Nevalainen, T. J., et al. Phospholipase A2 in cnidaria. Comparative Biochemistry and Physiology. Part B, Biochemistry & Molecular Biology. 139 (4), 731-735 (2004).
- Razpotnik, A., et al. A new phospholipase A2 isolated from the sea anemone Urticina crassicornis - its primary structure and phylogenetic classification. The FEBS Journal. 277 (12), 2641-2653 (2010).
- Dennis, E. A., Cao, J., Hsu, Y. -. H., Magrioti, V., Kokotos, G. Phospholipase A2 enzymes: Physical structure, biological function, disease implication, chemical inhibition, and therapeutic intervention. Chemical Reviews. 111 (10), 6130-6185 (2011).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Kwon, Y. -. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, 8663 (2015).
- Eno, A. E., Konya, R. S., Ibu, J. O. Biological properties of a venom extract from the sea anemone, Bunodosoma cavernata. Toxicon: Official Journal of the International Society on Toxinology. 36 (12), 2013-2020 (1998).
- Morales-Landa, J. L., et al. Antimicrobial, antiprotozoal, and toxic activities of Cnidarian extracts from the Mexican Caribbean Sea. Pharmaceutical Biology. 45 (1), 37-43 (2007).
- Sánchez-Rodríguez, J., Cruz-Vazquez, K. Isolation and biological characterization of neurotoxic compounds from the sea anemone Lebrunia danae (Duchassaing and Michelotti, 1860). Archives of Toxicology. 80 (7), 436-441 (2006).
- de Oliveira, J. S., et al. Caissarolysin I (Bcs I), a new hemolytic toxin from the Brazilian sea anemone Bunodosoma caissarum: purification and biological characterization. Biochimica Et Biophysica Acta. 1760 (3), 453-461 (2006).
- Norton, R. S., et al. Purification and characterisation of proteins with cardiac stimulatory and haemolytic activity from the anemone Actinia tenebrosa. Toxicon: Official Journal of the International Society on Toxinology. 28 (1), 29-41 (1990).
- Gondran, M., Eckeli, A. L., Migues, P. V., Gabilan, N. H., Rodrigues, A. L. S. The crude extract from the sea anemone, Bunodosoma caissarum elicits convulsions in mice: possible involvement of the glutamatergic system. Toxicon: Official Journal of the International Society on Toxinology. 40 (12), 1667-1674 (2002).
- Dion, A. S., Pomenti, A. A. Ammoniacal silver staining of proteins: mechanism of glutaraldehyde enhancement. Analytical Biochemistry. 129 (2), 490-496 (1983).
- Ramírez-Carreto, S., et al. Identification of a pore-forming protein from sea anemone Anthopleura dowii Verrill (1869) venom by mass spectrometry. The Journal of Venomous Animals and Toxins Including Tropical Diseases. 25, 147418 (2019).
- Nelson, G. J. Studies on the lipids of sheep red blood cells. I. Lipid composition in low and high potassium red cells. Lipids. 2 (1), 64-71 (1967).
- Ramírez-Carreto, S., et al. Transcriptomic and proteomic analysis of the tentacles and mucus of Anthopleura dowii Verrill, 1869. Marine Drugs. 17 (8), 436 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten