Method Article
Análisis espacio-temporal de la actividad microglial de Ca2+ a resolución de una sola célula
En este artículo
Resumen
En este trabajo, describimos un protocolo para la obtención de imágenes in vivo de la actividad microglial de Ca2+ y el posterior análisis de su dinámica espacio-temporal. Este método permite una caracterización exhaustiva de cómo la microglía responde a los cambios en el entorno cerebral, capturando adecuadamente las escalas espacio-temporales finas en las que ocurren tales eventos.
Resumen
La microglía es la única célula inmunitaria residente en el sistema nervioso central. Su morfología es muy plástica, cambiando en función de su actividad. En condiciones homeostáticas, la microglía posee una morfología muy ramificada. Esto facilita su monitoreo del entorno circundante a través de la extensión y retracción continuas de sus procesos. Sin embargo, durante la lesión cerebral y la inflamación, la microglía se activa y sufre cambios morfológicos dramáticos, retrayendo sus procesos ramificados e hinchando su cuerpo celular. Esto facilita actividades como la migración y la fagocitosis, que la microglía realiza para navegar por el entorno cerebral a un estado menos patológico.
Esta estrecha relación entre la morfología de la microglía y los cambios en su actividad ha permitido conocer considerablemente diversas funciones de la microglía. Sin embargo, tales cambios morfológicos y de actividad son en sí mismos fenómenos que pueden resultar de cualquier número de vías de señalización intracelular. Además, el desfase temporal entre el estímulo y la respuesta, así como la morfología altamente compartimentada de la microglía, dificultan el aislamiento de los mecanismos causales que sustentan la función. Para resolver este problema, desarrollamos una línea de ratones modificados genéticamente en la que una proteína indicadora fluorescente Ca2+ altamente sensible se expresa específicamente en la microglía.
Después de describir los métodos para la obtención de imágenes microgliales in vivo de Ca2+ , este artículo presenta un enfoque de análisis estructurado que clasifica esta actividad de Ca2+ en regiones subcelulares definidas racionalmente, asegurando así que las dimensiones espaciales y temporales de la información codificada se extraigan de manera significativa. Creemos que este enfoque proporcionará una comprensión detallada de las reglas de señalización intracelular que gobiernan la diversa gama de actividades microgliales asociadas con las funciones cerebrales superiores y las condiciones patológicas.
Introducción
La microglía son las células inmunitarias residentes en el sistema nervioso central (SNC) y desempeñan un papel importante en el mantenimiento de un entorno cerebral homeostático y en la regulación de la formación de circuitos neuronales durante el desarrollo del cerebro 1,2. Una característica única de la microglía en el SNC es que su morfología es altamente plástica; sin embargo, distintos fenotipos morfológicos pueden asociarse con funciones particulares. Además, la transformación entre fenotipos morfológicos es altamente dinámica, ocurriendo en escalas de tiempo rápidas en respuesta a cambios en el entorno circundante 3,4.
En condiciones fisiológicas homeostáticas, la microglía asume una morfología muy ramificada, con múltiples procesos que se irradian hacia afuera en todas las direcciones. Estos procesos ramificados por sí mismos demuestran una alta motilidad, extendiéndose y retrayéndose continuamente 3,4. Dicha actividad se dirige principalmente hacia el contacto periódico con las sinapsis neuronales, axones y somas para monitorear la actividad neuronal 5,6,7,8,9. Sin embargo, cuando el cerebro se lesiona, la microglía detecta rápidamente esta anormalidad y, como primer paso en su respuesta adaptativa, dirige la extensión de sus procesos hacia el lugar correspondiente 3,4. Cuando se requiere que las microglías realicen la fagocitosis de las células muertas y los metabolitos, asumen una morfología similar a la de los ameboides, acortando sus procesos y agrandando sus cuerpos celulares, como parte de su transición al fenotipo inmunoactivado10,11.
Sin embargo, mientras que los cambios morfológicos dramáticos de los procesos microgliales se detectan fácilmente, los cambios a escala más fina del soma celular son significativamente más difíciles de capturar, especialmente a una resolución temporal que es fisiológicamente relevante. Además, los cambios morfológicos en sí mismos solo representan el resultado integrado de cualquier número de vías de señalización intracelular. Esto es problemático para un objetivo de seguimiento de la actividad funcional y vincular mecánicamente un estímulo con la respuesta final que provoca.
Dado su papel generalizado como segundo mensajero, el examen de la dinámica intracelular de Ca2+ capta mejor la información espacio-temporal asociada cuando se estudian los procesos celulares dinámicos. Este enfoque es aplicable a la microglía dado que expresan una variedad de receptores ionotrópicos y metabotrópicos vinculados a la elevación intracelular de Ca2+ aguas abajo. De hecho, las imágenes in vivo de Ca2+ se han utilizado para caracterizar los aspectos espacio-temporales de las actividades microgliales en tiempo real, correlacionando con éxito los cambios en la actividad de Ca2+ de la microglía con lesiones cerebrales, inflamación e hiperactividad e hipoactividad en las neuronas 12,13,14,15,16. Por ejemplo, las elevaciones de Ca2+ asociadas con la extensión del proceso microglial en respuesta a la actividad neuronal hiper/hipoactiva probablemente reflejan el proceso subyacente de polimerización de actina dependiente de Ca2+ 16. Además, las imágenes in vivo de Ca2+ también pueden combinarse fácilmente con enfoques farmacológicos. Por ejemplo, mientras que la microglía expresa receptores P2X (ionotrópicos) y P2Y (metabotrópicos), la aplicación local de agonistas P2Y imita y posteriormente desensibiliza la respuesta microglialCa2+ a las neuronas vecinas dañadas13, lo que implica una mayor relevancia de la señalización de P2Y para la detección de daños neuronales.
Hasta la fecha, los informes anteriores que examinan la actividad microglial de Ca2+ han empleado métodos de análisis basados en la región de interés (ROI). Un inconveniente de estos enfoques es que todavía son demasiado toscos para poder resolver la dinámica espacio-temporal de la actividad del Ca2+ a nivel de los procesos microgliales individuales. Por lo tanto, este protocolo describe tanto los métodos convencionales basados en el ROI para analizar la actividad microglial de Ca2+ como los enfoques más nuevos basados en eventos, que pueden extraer eventos individuales de Ca2+ en procesos microgliales. Antes de esto, proporcionamos una guía general para la obtención de imágenes in vivo de dos fotones para capturar adecuadamente la actividad microglial de Ca2+ para un análisis detallado.
Protocolo
Todos los experimentos con animales fueron aprobados por los Comités de Investigación Animal del Instituto Nacional de Ciencias Fisiológicas y estuvieron de acuerdo con las pautas de los Institutos Nacionales de Salud. Para todos los experimentos, se criaron ratones machos de 8 a 10 semanas de edad bajo un ciclo de luz/oscuridad de 12/12 h con acceso ad libitum a alimentos y agua. Para visualizar la actividad de Ca2+ en la microglía, se cruzaron ratones ionizados de la molécula adaptadora de unión a Ca2+ 1 (Iba1)-transactivador de tetraciclina (Iba1-tTA) con ratones operador de tetraciclina-GCaMP6 (tetO-GCaMP6)17,18. Por lo tanto, en ausencia de suplementación con análogos de tetraciclina, el promotor Iba1 impulsa la expresión de GCaMP6, exclusivamente en la microglía. Para todos los experimentos, la suplementación dietética con doxiciclina se suspendió a las 6 semanas después del nacimiento. Al final de todos los experimentos, los ratones fueron sacrificados por sobredosis de isoflurano seguida de dislocación cervical. Consulte la Tabla de materiales para obtener detalles relacionados con todos los materiales, animales y reactivos utilizados en este protocolo.
1. Preparación quirúrgica de ratones para la obtención de imágenes in vivo de dos fotones; Día 1
- Realice todos los procedimientos quirúrgicos dentro de una cabina de flujo de aire laminar para mantener las condiciones de trabajo estériles. Antes de comenzar la cirugía, esterilice el interior del gabinete con luz ultravioleta durante 5 minutos.
- Esterilice todas las superficies de trabajo, el marco quirúrgico y los instrumentos estereotáxicos limpiándolos con etanol al 70%.
- Esterilice todos los instrumentos quirúrgicos (tijeras, fórceps, cuchilla de afeitar, pinzas) y la placa de cabeza hecha a medida para fijarla al cráneo del ratón sumergiéndolos en una solución de gluconato de clorhexidina al 1%.
- Anestesiar al ratón con ketamina (7,4 mg kg-1, intraperitoneal [i.p.]) y xilacina (10 mg kg-1, i.p.). Devuélvalo a su jaula de origen hasta que la anestesia haga efecto. Confirmar la inducción completa de la anestesia por pérdida del reflejo de pellizco del dedo del pie.
- Esterilizar el cuero cabelludo con gluconato de clorhexidina al 1%. Afeita el pelaje con una cuchilla de afeitar.
- Asegure el ratón dentro del marco de la cirugía mediante instrumentos estereotáxicos.
- Aplique ungüento veterinario en los ojos para evitar la sequedad mientras está bajo anestesia.
- Aplique jalea de xilocaína al 2% en el cuero cabelludo para controlar el dolor. Espere 5 min.
- Retire el cuero cabelludo con unas tijeras y exponga el cráneo. Limpie el periostio y seque la superficie expuesta del cráneo frotando con hisopos de algodón.
NOTA: Las áreas expuestas del cráneo deben estar completamente secas para asegurar una fuerte unión con la placa de cabeza hecha a medida. - Asegure la placa de cabeza hecha a medida al cráneo con cemento dental.
- Una vez que el cemento se haya endurecido, rellene los espacios entre la superficie del cráneo y los bordes de la placa de cabeza hecha a medida con cemento dental adicional.
- Impermeabilice las superficies del cemento y del cráneo mediante la aplicación de cemento de resina adhesiva dental a base de acrílico.
- Regrese el ratón a su jaula de origen, colocándolo en una almohadilla térmica. Vigile al ratón hasta que recupere la conciencia suficiente para mantener la decúbito esternal (dentro de las 2 h).
NOTA: Los ratones deben estar completamente recuperados al día siguiente y luego pueden ser alojados con otros animales.
2. Preparación quirúrgica de ratones para la obtención de imágenes in vivo de dos fotones; Día 2
- Realice todos los procedimientos quirúrgicos dentro de una cabina de flujo de aire laminar para mantener las condiciones de trabajo estériles. Antes de comenzar la cirugía, esterilice el interior del gabinete con luz ultravioleta durante 5 minutos.
- Laminar dos cubreobjetos de vidrio de diferentes dimensiones (vidrio superior: 3 mm × 3 mm; vidrio inferior: 2 mm × 2 mm) con resina de grado óptico curable UV.
NOTA: El cubreobjetos doble proporciona protección a largo plazo a la región del cerebro expuesta por la ventana craneal, al tiempo que permite el acceso óptico crónico. Por lo tanto, sus dimensiones pueden modificarse para adaptarse a la región del cerebro que se va a obtener la imagen. - Esterilice todas las superficies de trabajo y el marco de la cirugía limpiándolos con etanol al 70%.
- Esterilice todos los instrumentos quirúrgicos (taladro de acero, pinzas, gancho de aguja quirúrgica) sumergiéndolos en una solución de gluconato de clorhexidina al 1%.
- Anestesiar al ratón con isoflurano (4% de inducción, 1,2%-1,5% de mantenimiento). Asegure el ratón dentro del marco de la cirugía a través de su placa de cabeza.
- Para hacer una ventana craneal sobre la corteza motora primaria, marque un cuadrado con dimensiones de 2 mm × 2 mm centrado 0,2 mm anterior y 1 mm lateral del hito del cráneo de Bregma.
- Adelgaza el cráneo a lo largo del borde del cuadrado marcado con el taladro de acero.
NOTA: Cuando estén cerca del grosor deseado, las áreas adelgazadas del cráneo aparecerán transparentes cuando se humedezcan con solución salina y comenzarán a aparecer grietas finas. - Después de confirmar que todo el borde del cuadrado marcado se ha adelgazado adecuadamente, inserte con cuidado el gancho de la aguja quirúrgica justo debajo de la superficie del cráneo, orientando su punta hacia el centro del cuadrado. Levanta suavemente la pieza cuadrada del cráneo con el gancho y usa unas pinzas para despegarla del resto del cráneo. Si se produce sangrado, lave continuamente la superficie expuesta del cerebro con solución salina hasta que desaparezca por completo.
- Coloque el cubreobjetos doble sobre la superficie expuesta del cerebro, orientando el lado con el cubreobjetos más pequeño hacia el cerebro. Asegúrese de que los bordes del cubreobjetos más grande estén en contacto con los bordes de la ventana craneal.
- Usando una varilla de vidrio con punta de silicona montada en un manipulador, presione suavemente hacia abajo el cubreobjetos doble para asegurarse de que haga un buen contacto con la superficie del cerebro.
- Rellene el espacio entre el cubreobjetos doble, el cráneo y la superficie del cerebro con resina curable por UV e irradie con luz ultravioleta hasta que se endurezca (~20 s). Levante lentamente la varilla de vidrio con punta de silicona para separarla del cubreobjetos doble.
- Deje que el mouse se recupere, colocándolo sobre una almohadilla térmica. Vigile al ratón hasta que recupere la conciencia suficiente para mantener la decúbito esternal (30 min).
- Continúe con el paso 3 o devuelva el ratón a su jaula de inicio.
NOTA: Después de una cirugía competente, la duramadre y todos los vasos sanguíneos subyacentes estarán completamente intactos sin sangrado. En este caso, la inflamación es mínima y es posible proceder inmediatamente al paso 3. Si no está seguro, espere de 1 a 3 semanas para asegurarse de que cualquier inflamación se haya resuelto por completo.
3. Recopilación de datos mediante imágenes de dos fotones in vivo
- Encienda la configuración de imágenes con anticipación para asegurarse de que el láser tenga tiempo suficiente para calentarse y estabilizarse. Ajuste el láser para que emita a una longitud de onda de 920 nm, que es el espectro de dos fotones que excita de manera óptima el fluoróforo GCaMP6.
- Coloque el ratón debajo de una lente de objetivo de microscopio de 25x y acostúmbrelo durante 30 minutos. Si es necesario, anestesiar al ratón con isoflurano durante la obtención de imágenes (4% de inducción, 1,2%-1,5% de mantenimiento).
NOTA: El isoflurano puede no ser apropiado para algunas aplicaciones de investigación, ya que afecta la motilidad del proceso microglial y la actividad de Ca2+ 19,20,21. - Establezca el zoom en 1x y, a continuación, busque la microglía que expresa GCaMP6 en el área de la ventana craneal. Busca a una profundidad entre 100 μm y 300 μm por debajo de la superficie del cerebro.
- Una vez que se haya encontrado una microglía que exprese GCaMP6 adecuada, maximice el zoom para que la actividad de Ca2+ se pueda capturar con una resolución de una sola célula.
- Confirme visualmente que la actividad de Ca2+ se captura claramente y ajuste la potencia del láser y la ganancia de imagen si es necesario.
NOTA: La potencia del láser debe minimizarse tanto como sea posible para evitar el fotoblanqueo y las lesiones a la microglía. - Realice la adquisición de imágenes en cuatro dimensiones (4D) de la siguiente manera (pasos 3.6.1-3.6.3; Figura 1B):
- Establezca las dimensiones de la imagen en área XY = 512 × 512 píxeles, 0,25 μm/píxel; Área Z = cinco planos z, paso z de 3 μm (Figura 1B).
- Establezca la velocidad de adquisición en 2,5 fotogramas/s.
NOTA: El escaneo Z a esta velocidad se ve facilitado por un sistema de nanoposicionamiento piezoeléctrico. - Adquiera datos hasta que se observen al menos 10 eventos individuales de Ca2+ (generalmente 10 min).
4. Preparación para el análisis (corrección de movimiento, proyección z media/máxima)
- Antes de proceder a analizar la actividad microglial de Ca2+ , corrija las imágenes 4D para artefactos relacionados con el movimiento (registro de imágenes) utilizando el algoritmo de alineación de imágenes ECC22 dentro del entorno de programación MATLAB (R2020a). Descarga el código desde: https://www.mathworks.com/matlabcentral/fileexchange/27253-ecc-image-alignment-algorithm-image-registration.
- Cree una imagen 3D de referencia registrando todas las imágenes dentro del primer período de tiempo utilizando la operación imregister de MATLAB (caja de herramientas de procesamiento de imágenes estándar).
- Registre todas las imágenes subsiguientes, haciéndolas coincidir con el plano z correspondiente de la imagen 3D de referencia utilizando la función ecc (biblioteca de algoritmos de alineación de imágenes ECC).
- Genere proyecciones z de intensidad media a partir de las imágenes registradas utilizando la operación media de MATLAB. Si la señal de imagen es demasiado débil, genere proyecciones z de intensidad máxima en su lugar utilizando la operación max de MATLAB.
NOTA: La relación señal/ruido es peor cuando se utilizan proyecciones z de intensidad máxima. - Continúe con el paso 5 o 6. Utilice las proyecciones z generadas en el paso 4 como objetivo de análisis para mapear la dinámica espacio-temporal de la actividad de Ca2+ en los pasos 5 y 6.
5. Análisis basado en el ROI
- Utilizando las proyecciones z generadas en el paso 4, identifique los procesos microgliales que mantienen un perfil de área estable (área estable) durante todo el período de imagen para el análisis posterior de la actividad de Ca2+ de la siguiente manera (pasos 5.3-5.6):
- Genere proyecciones t de intensidad máxima separadas de las imágenes 4D registradas a partir de muestras de 2 minutos tomadas al principio y al final del período de imágenes utilizando la operación max MATLAB.
- Binarice las proyecciones t para generar polígonos de la morfología microglial correspondientes al inicio y al final del período de obtención de imágenes mediante la operación imbinarize de MATLAB. Utilice el umbral predeterminado establecido automáticamente.
NOTA: Si la señal de Ca2+ es débil durante el inicio y el final del período de imagen, la binarización puede generar polígonos con bordes faltantes. En este caso, dibuje manualmente los bordes que faltan con la herramienta de lápiz de ImageJ. - Superponga los polígonos de proyección t mediante la operación imadd de MATLAB. Las regiones superpuestas representan el área estable (Figura 2B).
- Dentro de las áreas estables identificadas, utilice la operación de MATLAB drawpolygon para definir y trazar manualmente los ROI de cada uno de los procesos microgliales primarios y cualquier subrama obvia de segundo orden.
- Realice un seguimiento de las intensidades de fluorescencia absolutas promediadas en un ROI individual durante todos los períodos de tiempo (Figura 2C, D).
- A partir de las series temporales de intensidades absolutas de fluorescencia, calcular el cambio relativo en las series temporales de intensidad de fluorescencia (ΔF/F) de acuerdo con la ecuación (1). Esta serie temporal representa la dinámica normalizada de Ca2+ de la microglía a nivel de los ROI individuales.
ΔF/F = (F(t) - F0) / F0 (1)
Donde F(t) es la intensidad de fluorescencia registrada en un momento dado y F0 esel percentil 10 de la intensidad de fluorescencia en todos los marcos de tiempo (Figura 2E). - Identifique los eventos de disparo de Ca2+ candidatos que ocurren en un ROI individual determinado de la siguiente manera (pasos 5.5.1-5.5.3):
- Aplique un filtrado de paso bajo de respuesta a impulsos finitos (FIR) de tipo I en la serie temporal ΔF/F mediante la operación fir1 de MATLAB. Establezca la frecuencia de corte en el valor de Nyquist (la mitad de la frecuencia de muestreo).
- Inspeccione visualmente la traza filtrada para confirmar que los picos de traza individuales corresponden a ráfagas de actividad de Ca2+ en las imágenes 4D registradas (Figura 2F).
- Identifique los eventos de disparo Ca2+ candidatos como inflexiones cóncavas hacia abajo en la traza de la serie temporalΔF/F . Defina un umbral de referencia como el valor medio de ΔF/F dentro de los límites máximos e inferiores que excluyen las amplitudes máxima y mínima del 10% en todos los períodos de tiempo. Defina un umbral de detección de tres SD por encima del umbral de referencia (Figura 2F, H).
- Identifique los verdaderos eventos de disparo de Ca2+ de los candidatos de la siguiente manera (pasos 5.5.1-5.5.2):
- Calcule la pendiente de cada evento candidato diferenciando entre los correspondientes fotogramas de series temporales ΔF/F filtradas mediante la operación de gradiente numérico de MATLAB.
- Posteriormente, identifique los eventos verdaderos de Ca2+ en función del tiempo de subida del perfil. Defina un umbral de línea base como el promedio de los valores de pendiente en todos los eventos candidatos. Defina un umbral de detección de tres SD por encima del umbral de referencia (Figura 2G, H).
- Caracterice los eventos Ca2+ verdaderos de la siguiente manera (pasos 5.6.1-5.6.3):
NOTA: La siguiente no es una lista exhaustiva de parámetros que se pueden utilizar para caracterizar los eventos de Ca2+ microglial. Los parámetros de interés dependen del propósito del estudio.- Derive la amplitud máxima de un evento de disparo Ca2+ verdadero como el valor ΔF/F del marco de tiempo de la serie temporal ΔF/F correspondiente.
- Derive la amplitud media de un evento de disparo Ca2+ verdadero como el valor promedio de ΔF/F en todo el subconjunto de tramas de la serie temporal ΔF/F correspondiente.
- Derive la frecuencia de eventos Ca2+ verdaderos como el número de eventos dividido por el tiempo de imagen.
6. Análisis basado en eventos
- Realice análisis basados en eventos en las proyecciones z del paso 4 utilizando la biblioteca AQuA dentro del entorno de programación de MATLAB. Descarga el código desde: https://github.com/yu-lab-vt/AQuA.
NOTA: Un tutorial de uso general para la biblioteca AQuA está disponible en: https://drive.google.com/file/d/1a3lhe0dUth-5J1-S2fZlPOCZlPbeuvUr/view. La documentación detallada de la biblioteca AQuA está disponible en: https://drive.google.com/file/d/1CckDLbrkw16b7MPlOQdYpZciIz80Snm_/view. - Después de iniciar MATLAB, cambie de la carpeta de directorio de trabajo predeterminada a la carpeta de directorio de trabajo designada de AQuA mediante la operación cd .
- Cargue las imágenes 4D registradas en el canal de análisis AQuA de la siguiente manera (pasos 6.3.1-6.3.2):
- Inicie la GUI de AQuA. Escriba aqua_gui en la ventana de comandos de MATLAB en la carpeta del directorio de trabajo designado de AQuA.
- Dentro de la GUI de AQuA, haga clic en nuevo proyecto y seleccione las imágenes registradas que se van a analizar. Especifique el tipo de datos (GCaMPInVivo_cyto_Lck_) y los parámetros de imagen (resolución temporal segundos por fotograma = 1,993; resolución espacial μm por píxel = 0,25; excluir píxeles más cortos que esta distancia al borde = 5). Haga clic en abrir para cargar los datos.
- Defina puntos de referencia para las canalizaciones de análisis posteriores mediante el trazado de áreas de interés. Por lo general, los puntos de referencia se basan en el límite de la célula y el área del cuerpo de la célula.
- Posteriormente, detecte los eventos candidatos Ca2+ mediante la ejecución de las canalizaciones de análisis automatizado para los siguientes parámetros: señal activa, supervóxel, detección de eventos, eventos limpios y eventos de combinación.
NOTA: En el tutorial de AQuA se proporciona una explicación detallada sobre cada uno de estos parámetros y sus resultados de puntuación candidatos. Brevemente, estos parámetros ajustan el umbral de detección de eventos Ca2+ de la siguiente manera: señal activa = amplitud de fluorescencia, supervóxel = agrupación de fluorescencia en el espacio 3D, detección de eventos = cinética de aumento/decaimiento, eventos limpios = relación señal/ruido y eventos de fusión = separación temporal de eventos. - Inspeccione visualmente los eventos Ca2+ detectados (superpuestos automáticamente a las imágenes registradas originales). Si es necesario, ajuste la configuración de parámetros de las canalizaciones de análisis teniendo en cuenta la calidad de la imagen en términos de los parámetros anteriores; Si la calidad de imagen de un parámetro determinado es buena, se puede establecer un umbral más alto y viceversa.
- Una vez que todos los eventos de Ca2+ se hayan detectado adecuadamente, caractérelos dentro del entorno principal de MATLAB de la siguiente manera (pasos 6.8-6.14):
- Exporte los archivos de salida del análisis AQuA.
NOTA: En la documentación de AQuA se proporciona una explicación detallada sobre los archivos de salida, las características extraídas que se utilizan para definir eventos Ca2+ y los parámetros subyacentes de estas características extraídas. - (Opcional) Clasifique todos los eventos de Ca2+ en dos grupos: 1) eventos que comienzan en el soma y 2) eventos que comienzan en los procesos.
- Acceda a la amplitud de los eventos individuales de Ca2+ dentro de la estructura res.dffMat de MATLAB del archivo de salida de análisis AQuA (archivo .mat).
- Derive la frecuencia de eventos Ca2+ individuales utilizando la operación res.fts.loc.x3D de MATLAB (biblioteca AQuA).
- Derive la duración de eventos Ca2+ individuales mediante la operación res.fts.curve.width11 de MATLAB (biblioteca AQuA).
- Derive el área de eventos Ca2+ individuales mediante la operación de MATLAB res.fts.basic.area (biblioteca AQuA).
- Categoriza todos los eventos Ca2+ como: 1) eventos locales, 2) eventos propagativos que viajan hacia el soma, y 3) eventos propagativos que se alejan del soma. Para ello, utilice las operaciones de MATLAB res.fts.region.landmarkDir.chgToward y res.fts.region.landmarkDir.chgAway (biblioteca AQuA) (Figura 3C).
Resultados
En ratones transgénicos que expresan exclusivamente GCaMP6 (proteína fluorescente sensible al Ca2+) en la microglía, normalmente observamos diversos patrones de actividad microglial de Ca2+ (Figura 2A). Es importante destacar que, incluso dentro de una sola microglía, los patrones de actividad de Ca2+ pueden diferir drásticamente entre procesos.
Para cuantificar tales diferencias de proceso a proceso en la dinámica espacio-temporal de la actividad de Ca2+ de la microglía, primero se deben identificar áreas estables y luego dividirlas en ROI finamente segmentadas (Figura 2B, C). Para cada ROI, se deben derivar y cuantificar los parámetros de la actividad de Ca2+, como la amplitud y la frecuencia, extrayendo características como las amplitudes locales y las pendientes de las trazas de la serie temporal de intensidad de fluorescencia (Figura 2D-G).
A continuación, se deben examinar los eventos individuales de Ca2+ aplicando el algoritmo de cuantificación preciso AQuA (Figura 3A). A partir de estos análisis basados en eventos, se observan típicamente grandes diferencias en las características de origen, amplitud, duración, ubicación y dirección del flujo de los eventos individuales de Ca2+ (Figura 3B). Si nos centramos en analizar la dinámica de la actividad del Ca2+ en los procesos microgliales, es informativo un esquema de clasificación de los eventos locales, los eventos que viajan hacia el soma y los eventos que se alejan del soma (Figura 3C).
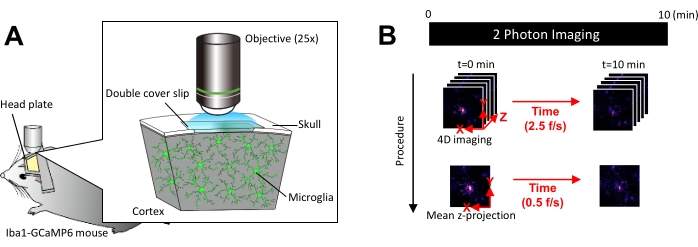
Figura 1: Configuración experimental para la obtención de imágenes microgliales Ca2+ in vivo. (A) Configuración experimental. Un ratón Iba1-tTA × tetO-GCaMP6 con expresión GCaMP6 específica de la microglía. Al insertar una ventana craneal en el cráneo del ratón, la actividad microglial de Ca2+ se puede observar in vivo utilizando microscopía de dos fotones. (B) Cronograma experimental y procedimiento de análisis. Las imágenes 4D se adquieren durante al menos 10 minutos como pilas z de cinco fotogramas. La velocidad de adquisición de fotogramas es de 2,5 fotogramas/s. Antes de analizar la actividad microglial de Ca2+, las pilas z de cinco fotogramas se convierten en proyecciones z 2D tomando la intensidad media (o máxima). La velocidad de reproducción de la proyección z es de 0,5 fotogramas/s. Haga clic aquí para ver una versión más grande de esta figura.
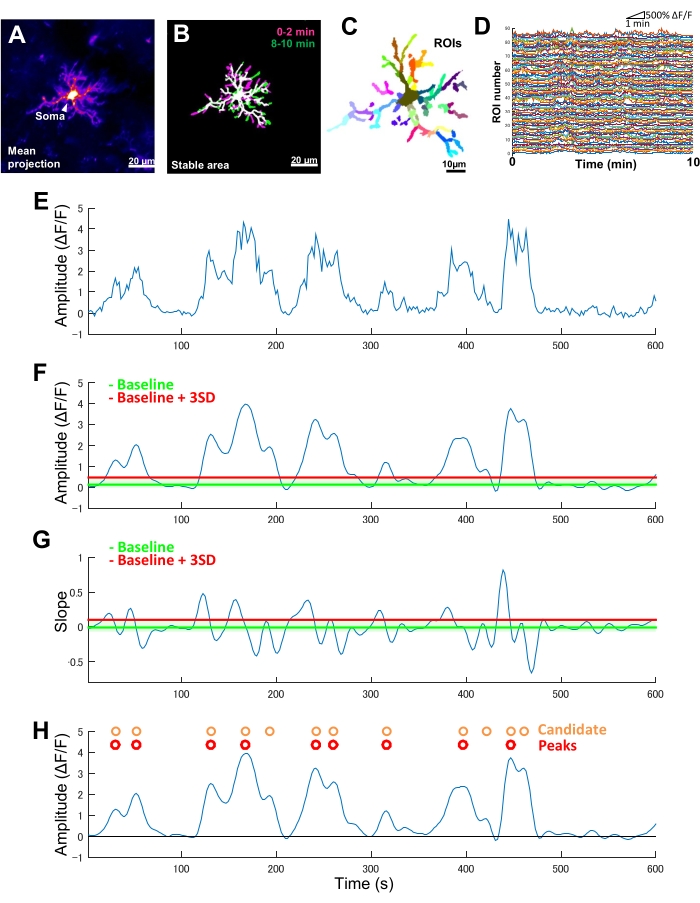
Figura 2: Análisis basado en el ROI para la actividad microglial de Ca2+ . (A) Proyección media de la intensidad de GCaMP6 durante 10 min para una microglía individual. (B) Las áreas estables (blanco) se definen por una superposición de proyecciones t binarizadas de máxima intensidad GCaMP6 derivadas de muestras de 2 minutos tomadas al principio (magenta) y al final (verde) de un período de imágenes. (C) Las áreas estables se segmentan aún más en ROI regionales. Los colores individuales indican el retorno de la inversión individual. (D) Trazas ΔF/F de todos los ROI individuales en C. Tenga en cuenta la variación en los patrones de actividad entre los ROI. (E) Traza original de una serie temporal ΔF/F derivada de valores de intensidad absoluta para un solo ROI. (F) La misma serie de tiempo ΔF/F después del filtrado de paso bajo. Los eventos candidatos Ca2+ se detectan mediante un umbral de corte de amplitud (línea roja) definido como línea base + tres SD. La línea base (línea verde) se define como el valor mediano en toda la serie temporal ΔF/F dentro de un límite máximo e inferior que excluye el 10% máximo y mínimo de los valores ΔF/F . (G) La traza de pendiente derivada de la serie temporal ΔF/F filtrada en F. Los eventos Ca2+ verdaderos se ordenan a partir de los eventos Ca2+ candidatos en función de un umbral de corte de pendiente (línea roja) definido como línea base + tres SD. La línea base (línea verde) se define como el valor promedio a lo largo de toda la serie temporal de pendiente dentro de un techo superior e inferior que excluye el 10% máximo y mínimo de los valores de pendiente. (H) Los eventos candidatos Ca2+ identificados por criterios de amplitud en F se indican en naranja. Eventos Ca2+ verdaderos ordenados a partir de eventos candidatos Ca2+ por criterios de pendiente en G en rojo. Tenga en cuenta que algunos eventos candidatos Ca2+ se han fusionado en función de los criterios de pendiente. Las series temporales ΔF/F filtradas correspondientes se superponen a continuación como referencia. La línea negra indica amplitud cero (ΔF/F). La amplitud media y máxima de los eventos Ca2+ verdaderos se derivan como la media y el máximo de sus picos correspondientes en las series temporales ΔF/F filtradas. La frecuencia (eventos/min) se deriva como el número de eventos de disparo de Ca2+ verdaderos dividido por el período de imagen (10 min). Barras de escala = 20 μm (A,B), 10 μm (C). Abreviatura: ROI = región de interés. Haga clic aquí para ver una versión más grande de esta figura.
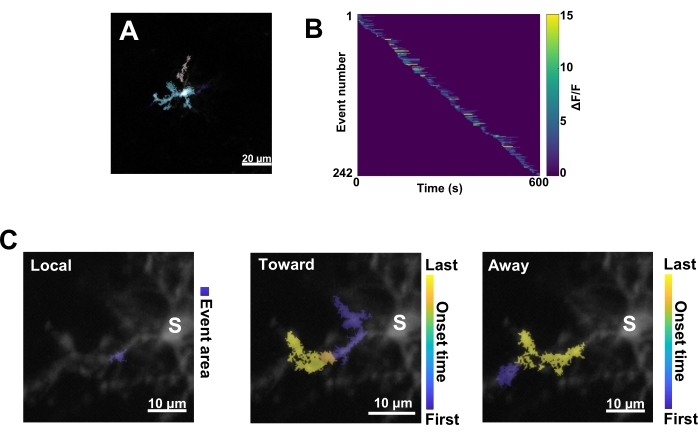
Figura 3: Análisis basado en eventos para la actividad microglial de Ca2+. (A) Imágenes representativas de eventos detectados utilizando el algoritmo AQuA. Los colores indican áreas de eventos individuales detectadas en un punto de tiempo determinado. (B) Actividad Ca2+ normalizada representativa (ΔF/F) en eventos individuales ordenados por orden de inicio. La barra de la derecha indica ΔF/F indicado en color. (C) Huellas de actividad representativas de eventos que se propagan hacia y desde el soma o eventos locales. En el caso de los eventos locales, la forma del evento se muestra en azul. En el caso de los eventos propagativos, el tiempo de inicio del evento se indica mediante la escala azul-amarilla. Dado que AQuA detecta inicialmente eventos Ca2+ individualmente, los eventos propagativos se identifican posteriormente en función de las ubicaciones espaciales superpuestas y las series temporales de múltiples eventos Ca2+ individuales. Tenga en cuenta que esta es la misma rama dendrítica utilizada para el análisis de ROI en la Figura 2E-H. Barras de escala = 20 μm (A), 10 μm (C). Abreviatura: ROI = región de interés; S = soma. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este artículo presenta un enfoque mejorado para obtener imágenes de la actividad microglial Ca2+ con una alta resolución espacio-temporal. Este método es lo suficientemente sensible como para detectar diferentes tipos de actividad microglial de Ca2+ a nivel de procesos ramificados individuales, distinguiendo fácilmente entre eventos locales y propagativos.
En el método general para la obtención de imágenes in vivo de dos fotones de la actividad microglial Ca2+ , se debe prestar especial atención a los siguientes puntos para maximizar la calidad de la imagen. En primer lugar, dado que la microglía es extremadamente sensible a las lesiones, es importante minimizar el contacto directo con la superficie del cerebro con herramientas quirúrgicas durante la cirugía. Las indicaciones clave de que la cirugía se ha realizado de manera competente son los vasos sanguíneos y la duramadre intactos y un sangrado mínimo durante la cirugía. En segundo lugar, la fijación segura de la placa de la cabeza al cráneo del ratón y el buen contacto entre el doble cubreobjetos y la superficie del cerebro reducen en gran medida los artefactos relacionados con el movimiento durante la obtención de imágenes. Esto es especialmente importante cuando se obtienen imágenes con altas resoluciones espacio-temporales y en ratones completamente despiertos. Si bien la tubería de análisis compensa de manera confiable los artefactos relacionados con el movimiento que surgen de los latidos del corazón, la respiración y la deriva general, es menos robusta cuando maneja distorsiones geométricas significativas que surgen de movimientos grandes repentinos.
Los dos métodos de análisis descritos aquí ofrecen diferentes ventajas y son adecuados para diferentes preguntas de investigación. En el análisis basado en el ROI, el usuario predefine el ROI (como los procesos individuales), lo que permite extraer la dinámica agregada de la actividad de Ca2+ de este ROI. Por lo tanto, es más adecuado para situaciones en las que se espera que los fenómenos se localicen en un área subcelular que tenga límites morfológicos bien definidos y un área relativamente grande (es decir, una rama del proceso). En el análisis basado en eventos, los eventos individuales se definen en función de la dinámica espacio-temporal de la propia actividad de la microglía Ca2+ y luego deben colocarse en el contexto de puntos de referencia definidos por el usuario dentro de la microglía para que se interprete su función. Por lo tanto, es más adecuado para situaciones en las que no se pueden hacer suposiciones sobre la localización de los fenómenos o en las que el área de interés es relativamente pequeña (es decir, una punta de proceso). Como tal, el análisis basado en eventos ofrece una resolución espacio-temporal mejorada al caracterizar la actividad microglial de Ca2+ en comparación con los métodos anteriores.
En estos ratones, el único marcador fluorescente expresado por la microglía es el indicador Ca2+ GCaMP6. Por lo tanto, en regiones donde la actividad de Ca2+ es baja, la morfología microglial debe extraerse combinando múltiples marcos de tiempo, lo que puede degradar la resolución temporal. Sin embargo, esta limitación puede superarse mediante la expresión de una proteína roja fluorescente estable separada en la microglía. En particular, recientemente se han descrito nuevos virus adenoasociados capaces de transfectar la microglía 23,24,25.
La forma en que la actividad microglial de Ca2+ se ve alterada por el entorno circundante es un tema de interés emergente. En particular, la actividad microglial de Ca2+ parece mostrar correlaciones significativas con la actividad neuronal, aunque la importancia funcional de esto aún no se ha caracterizado completamente. Por lo tanto, la combinación de la manipulación de la actividad neuronal con los métodos de imagen y análisis de la actividad microglial Ca2+ presentados aquí debería proporcionar nuevos conocimientos sobre la fisiología microglial y avanzar aún más en nuestra comprensión de las funciones que desempeña la microglía en los estados fisiológicos y patológicos.
Divulgaciones
Los autores declaran no tener conflictos de intereses asociados con este manuscrito.
Agradecimientos
Agradecemos al Prof. Kenji Tanaka (Universidad de Keio, Tokio, Japón) por proporcionar los ratones Iba1-tTA y los ratones tetO-GCaMP6. Este trabajo contó con el apoyo de Grants-in-Aid for Young Scientists (B) [16K19001 (a S.H.)], Grant-in-Aid for Early-Career Scientists [18K14825 (a S.H.)], Grant-in-Aid for Scientific Research (B) [21H03027 (a S.H.)], Grant-in-Aid for Transformative Research Areas (A) [21H05639 (a S.H.)], Grant-in-Aid for Scientific Research (A) [17H01530, 20H00500 (a J.N.)], y JST CREST Grant [JPMJCR1755 (a J.N.)], Japón.
Materiales
| Name | Company | Catalog Number | Comments |
| 2% xylocaine jelly | AstraZeneca, UK | ||
| B6(129S6)-Tg(Aif1-tTA)54Kftnk | RIKEN RBC, Japan | RBRC05769 | Iba1-tTA mice |
| B6;129-Actb(tm3.1(tetO-GCaMP6)Kftnk) | RIKEN RBC, Japan | RBRC09552 | tetO-GCaMP6 mice |
| Forceps | Fine science tools, US | 13008-12 | |
| G-CEM ONE | GC corporation, Japan | ||
| Glass capillary | Narishige, Japan | GDC-1 | |
| ImageJ | NIH, US | ||
| Isofulrane | Pfizer, US | ||
| Ketamin | Daiichi-Sankyo, Japan | ||
| Kwik-sil | World Precision Instruments, US | KWIK-SIL | |
| MATLAB, 2020a | MathWorks, US | ||
| Micro cover glass (2 x 2 mm, No.3) | Matsunami, Japan | custum-made | Bottom glass for cranial window |
| Micro cover glass (3 x 3 mm, No.0) | Matsunami, Japan | custum-made | Upper glass for cranial window |
| N25X-APO-MP | Nikon, Japan | N25X-APO-MP | Objective lens (25x) |
| Norland optical adhesive | Edmund optics, US | 6101 | |
| Piezo nano-positioning system, Nano-Drive | Mao City Labs, US | ||
| Razor blade | Feather, Japan | FA-10 | |
| Scissors | Fine science tools, US | 14060-11 | |
| Steel drill | Minitor, Japan | BS1201 | |
| Stereotaxic instruments | Narishige, Japan | SR-5M-HT | |
| Super-bond (C&B kit) | Sun Medical, Japan | 4560227797382 | |
| Surgical needle hook | Fine science tools, US | 10065-15 | |
| Ti:Sappire laser, MaiTai DeepSee | Spectra Physics, US | Mai Tai eHP DS | |
| Tweezers | Fine science tools, US | 11051-10 | |
| Tweezers | Fine science tools, US | 11255-20 | |
| Two-photon microscope | Nikon, Japan | A1R-MP | |
| UV craft resin | Kiyohara, Japan | UVR | |
| Xylazine | Bayer, Germany |
Referencias
- Miyamoto, A., et al. Microglia contact induces synapse formation in developing somatosensory cortex. Nature Communications. 7, 12540 (2016).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Davalos, D., et al. ATP mediates rapid microglial response to local brain injury in vivo. Nature Neuroscience. 8 (6), 752-758 (2005).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Science. 308 (5726), 1314-1318 (2005).
- Cserep, C., et al. Microglia monitor and protect neuronal function through specialized somatic purinergic junctions. Science. 367 (6477), 528-537 (2020).
- Kato, G., et al. Microglial contact prevents excess depolarization and rescues neurons from excitotoxicity. eNeuro. 3 (3), (2016).
- Li, Y., Du, X. -. F., Liu, C. -. S., Wen, Z. -. L., Du, J. -. L. Reciprocal regulation between resting microglial dynamics and neuronal activity in vivo. Developmental Cell. 23 (6), 1189-1202 (2012).
- Tremblay, M. -. E., Lowery, R. L., Majewska, A. K. Microglial interactions with synapses are modulated by visual experience. Plos Biology. 8 (11), 1000527 (2010).
- Wake, H., Moorhouse, A. J., Jinno, S., Kohsaka, S., Nabekura, J. Resting microglia directly monitor the functional state of synapses in vivo and determine the fate of ischemic terminals. The Journal of Neuroscience. 29 (13), 3974-3980 (2009).
- Oppenheim, R. W. Cell death during development of the nervous system. Annual Review of Neuroscience. 14 (1), 453-501 (1991).
- Sierra, A., Abiega, O., Shahraz, A., Neumann, H. Janus-faced microglia: beneficial and detrimental consequences of microglial phagocytosis. Frontiers in Cellular Neuroscience. 7, 6 (2013).
- Brawek, B., et al. A new approach for ratiometric in vivo calcium imaging of microglia. Scientific Reports. 7 (1), 6030 (2017).
- Eichhoff, G., Brawek, B., Garaschuk, O. Microglial calcium signal acts as a rapid sensor of single neuron damage in vivo. Biochimica et Biophysica Acta. 1813 (5), 1014-1024 (2011).
- Liu, L., et al. Microglial calcium waves during the hyperacute phase of ischemic stroke. Stroke. 52 (1), 274-283 (2021).
- Pozner, A., et al. Intracellular calcium dynamics in cortical microglia responding to focal laser injury in the PC::G5-tdT reporter mouse. Frontiers in Molecular Neuroscience. 8, 12 (2015).
- Umpierre, A. D., et al. Microglial calcium signaling is attuned to neuronal activity in awake mice. Elife. 9, e56502 (2020).
- Ohkura, M., et al. Genetically encoded green fluorescent Ca2+ indicators with improved detectability for neuronal Ca2+ signals. Plos One. 7 (12), e51286 (2012).
- Tanaka, K. F., et al. Expanding the repertoire of optogenetically targeted cells with an enhanced gene expression system. Cell Reports. 2 (2), 397-406 (2012).
- Liu, Y. U., et al. Neuronal network activity controls microglial process surveillance in awake mice via norepinephrine signaling. Nature Neuroscience. 22 (11), 1771-1781 (2019).
- Stowell, R. D., et al. Noradrenergic signaling in the wakeful state inhibits microglial surveillance and synaptic plasticity in the mouse visual cortex. Nature Neuroscience. 22 (11), 1782-1792 (2019).
- Sun, W. L., et al. In vivo two-photon imaging of anesthesia-specific alterations in microglial surveillance and photodamage-directed motility in mouse cortex. Frontiers in Neuroscience. 13, 421 (2019).
- Evangelidis, G. D., Psarakis, E. Z. Parametric image alignment using enhanced correlation coefficient maximization. IEEE Transactions on Pattern Analysis and Machine Intelligence. 30 (10), 1858-1865 (2008).
- Rosario, A. M., et al. Microglia-specific targeting by novel capsid-modified AAV6 vectors. Molecular Therapy. Methods & Clinical Development. 3, 16026 (2016).
- Maes, M. E., Wogenstein, G. M., Colombo, G., Casado-Polanco, R., Siegert, S. Optimizing AAV2/6 microglial targeting identified enhanced efficiency in the photoreceptor degenerative environment. Molecular Therapy. Methods & Clinical Development. 23, 210-224 (2021).
- Lin, R., et al. Directed evolution of adeno-associated virus for efficient gene delivery to microglia. Nature Methods. 19 (8), 976-985 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados