Method Article
Analyse spatio-temporelle de l’activité microgliale Ca2+ à une résolution de cellule unique
Dans cet article
Résumé
Dans cet article, nous décrivons un protocole d’imagerie in vivo de l’activité microgliale Ca2+ et l’analyse ultérieure de sa dynamique spatio-temporelle. Cette méthode permet une caractérisation approfondie de la façon dont les microglies réagissent aux changements de l’environnement cérébral, en capturant de manière appropriée les fines échelles spatio-temporelles auxquelles de tels événements se produisent.
Résumé
Les microglies sont les seules cellules immunitaires résidentes du système nerveux central. Leur morphologie est très plastique, changeant en fonction de leur activité. Dans des conditions homéostatiques, les microglies possèdent une morphologie très ramifiée. Cela facilite leur surveillance de l’environnement environnant grâce à l’extension et à la rétraction continues de leurs processus. Cependant, lors de lésions cérébrales et d’inflammation, les microglies s’activent et subissent des changements morphologiques spectaculaires, rétractant leurs processus ramifiés et gonflant leur corps cellulaire. Cela facilite des activités telles que la migration et la phagocytose, que les microglies entreprennent pour naviguer dans l’environnement cérébral vers un état moins pathologique.
Cette relation étroite entre la morphologie microgliale et les changements dans leur activité a permis de mieux comprendre diverses fonctions microgliales. Cependant, de tels changements morphologiques et d’activité sont eux-mêmes des phénomènes qui peuvent résulter d’un certain nombre de voies de signalisation intracellulaires. De plus, le décalage temporel entre le stimulus et la réponse, ainsi que la morphologie fortement compartimentée de la microglie, rendent difficile l’isolement des mécanismes causaux qui sous-tendent la fonction. Pour résoudre ce problème, nous avons développé une lignée de souris génétiquement modifiée dans laquelle une protéine indicatrice fluorescente Ca2+ très sensible est spécifiquement exprimée dans la microglie.
Après avoir décrit les méthodes d’imagerie microgliale Ca 2+ in vivo, cet article présente une approche d’analyse structurée qui classe cette activité Ca2+ dans des régions subcellulaires rationnellement définies, garantissant ainsi que les dimensions spatiales et temporelles de l’information codée sont extraites de manière significative. Nous croyons que cette approche fournira une compréhension détaillée des règles de signalisation intracellulaire qui régissent la diversité des activités microgliales associées à la fois aux fonctions cérébrales supérieures et aux conditions pathologiques.
Introduction
Les microglies sont les cellules immunitaires résidentes du système nerveux central (SNC) et jouent un rôle important dans le maintien d’un environnement cérébral homéostatique et dans la régulation de la formation des circuits neuronaux au cours du développement cérébral 1,2. Une caractéristique unique des microglies dans le SNC est que leur morphologie est très plastique ; Cependant, des phénotypes morphologiques distincts peuvent être associés à des fonctions particulières. De plus, la transformation entre les phénotypes morphologiques est très dynamique, se produisant sur des échelles de temps rapides en réponse aux changements de l’environnement environnant 3,4.
Dans des conditions physiologiques homéostatiques, la microglie adopte une morphologie très ramifiée, avec de multiples processus rayonnant vers l’extérieur dans toutes les directions. Ces processus ramifiés présentent eux-mêmes une motilité élevée, s’étendantet se rétractant continuellement3,4. Une telle activité est principalement dirigée vers le contact périodique avec les synapses neuronales, les axones et les somas pour surveiller l’activité neuronale 5,6,7,8,9. Cependant, lorsque le cerveau est blessé, les microglies détectent rapidement cette anomalie et, comme première étape de leur réponse adaptative, dirigent l’extension de leurs processus vers la région correspondante 3,4. Lorsque les microglies sont nécessaires pour entreprendre la phagocytose des cellules mortes et des métabolites, elles adoptent une morphologie semblable à celle des amiboïdes, raccourcissant leurs processus et élargissant leurs corps cellulaires, dans le cadre de leur transition vers le phénotype10,11 activé par l’immunologie.
Cependant, alors que les changements morphologiques spectaculaires des processus microgliaux sont facilement détectés, les changements d’échelle plus fins du soma cellulaire sont nettement plus difficiles à capturer, en particulier à une résolution temporelle physiologiquement pertinente. De plus, les changements morphologiques eux-mêmes ne représentent que le résultat intégré d’un certain nombre de voies de signalisation intracellulaires. Ceci est problématique dans le but de suivre l’activité fonctionnelle et de relier mécaniquement un stimulus à la réponse finale qu’il provoque.
Compte tenu de son rôle répandu en tant que second messager, l’examen de la dynamique intracellulaire du Ca2+ permet de mieux saisir les informations spatio-temporelles associées lors de l’étude des processus cellulaires dynamiques. Une telle approche est applicable aux microglies étant donné qu’elles expriment une variété de récepteurs ionotropes et métabotropiques liés à l’élévation intracellulaire en aval de Ca2+. En effet, l’imagerie in vivo Ca2+ a été utilisée pour caractériser les aspects spatio-temporels des activités microgliales en temps réel, corrélant avec succès les changements de l’activité microgliale Ca2+ avec les lésions cérébrales, l’inflammation et l’hyperactivité et l’hypoactivité dans les neurones 12,13,14,15,16. Par exemple, les élévations de Ca2+ associées à l’extension du processus microglial en réponse à une activité neuronale hyper/hypoactive reflètent probablement le processus de polymérisation de l’actine dépendant du Ca2+ 16. De plus, l’imagerie in vivo Ca2+ peut également être facilement combinée avec des approches pharmacologiques. Par exemple, alors que les microglies expriment à la fois les récepteurs P2X (ionotropiques) et P2Y (métabotropiques), l’application locale d’agonistes P2Y imite et désensibilise par la suite la réponse microgliale Ca2+ aux neurones voisins endommagés13, ce qui implique une plus grande pertinence de la signalisation P2Y pour la détection des lésions neuronales.
À ce jour, des rapports antérieurs examinant l’activité microgliale du Ca2+ ont utilisé des méthodes d’analyse basées sur la région d’intérêt (ROI). Un inconvénient de ces approches est qu’elles sont encore trop grossières pour pouvoir résoudre la dynamique spatio-temporelle de l’activité de Ca2+ au niveau des processus microgliaux individuels. Ainsi, ce protocole décrit à la fois les méthodes conventionnelles basées sur le retour sur investissement pour analyser l’activité microgliale Ca2+ et les nouvelles approches basées sur les événements, qui peuvent extraire des événements individuels Ca2+ dans les processus microgliaux. Avant cela, nous fournissons un guide général pour l’imagerie à deux photons in vivo afin de capturer de manière appropriée l’activité microgliale Ca2+ pour une analyse détaillée.
Protocole
Toutes les expériences sur les animaux ont été approuvées par les comités de recherche animale de l’Institut national des sciences physiologiques et étaient conformes aux directives des National Institutes of Health. Pour toutes les expériences, des souris mâles âgées de 8 à 10 semaines ont été élevées sous un cycle lumière/obscurité de 12/12 h avec un accès ad libitum à la nourriture et à l’eau. Pour visualiser l’activité du Ca2+ dans la microglie, des souris ionisées Iba1-tTA ont été croisées avec des souris 17,18 de l’opérateur de tétracycline GCaMP6 (tetO-GCaMP6). Ainsi, en l’absence de supplémentation en analogue de la tétracycline, le promoteur d’Iba1 pilote l’expression de GCaMP6, exclusivement dans la microglie. Pour toutes les expériences, la supplémentation alimentaire en doxycycline a été arrêtée à 6 semaines après la naissance. À la fin de toutes les expériences, les souris ont été euthanasiées par surdosage d’isoflurane suivi d’une luxation cervicale. Voir le tableau des matériaux pour plus de détails sur tous les matériaux, animaux et réactifs utilisés dans ce protocole.
1. Préparation chirurgicale de souris pour l’imagerie in vivo à deux photons ; Jour 1
- Effectuez toutes les interventions chirurgicales à l’intérieur d’une armoire à flux d’air laminaire pour maintenir des conditions de travail stériles. Avant de commencer l’opération, stérilisez l’intérieur de l’armoire avec de la lumière UV pendant 5 minutes.
- Stérilisez toutes les surfaces de travail, le cadre du cabinet et les instruments stéréotaxiques en les essuyant avec de l’éthanol à 70 %.
- Stérilisez tous les instruments chirurgicaux (ciseaux, pinces, lame de rasoir, pince à épiler) et la plaque frontale sur mesure à fixer sur le crâne de la souris en les immergeant dans une solution de gluconate de chlorhexidine à 1 %.
- Anesthésier la souris avec de la kétamine (7,4 mg kg−1, par voie intrapéritonéale [i.p.]) et de la xylazine (10 mg kg−1, i.p.). Remettez-le dans sa cage d’origine jusqu’à ce que l’anesthésie s’installe. Confirmer l’induction complète de l’anesthésie par la perte du réflexe de pincement des orteils.
- Stérilisez le cuir chevelu avec du gluconate de chlorhexidine à 1 %. Rasez la fourrure avec une lame de rasoir.
- Fixez la souris dans le cadre chirurgical à l’aide d’instruments stéréotaxiques.
- Appliquez une pommade vétérinaire sur les yeux pour prévenir la sécheresse sous anesthésie.
- Appliquez de la gelée de xylocaïne à 2 % sur le cuir chevelu pour la gestion de la douleur. Attendez 5 min.
- Retirez le cuir chevelu avec des ciseaux et exposez le crâne. Nettoyez le périoste et séchez la surface exposée du crâne en frottant avec des cotons-tiges.
REMARQUE : Les zones exposées du crâne doivent être complètement sèches pour assurer une liaison solide avec la plaque de tête sur mesure. - Fixez la plaque de tête sur mesure au crâne avec du ciment dentaire.
- Une fois que le ciment a pris, remplissez tous les espaces entre la surface du crâne et les bords de la plaque de tête sur mesure avec du ciment dentaire supplémentaire.
- Imperméabilisez les surfaces du ciment et du crâne en appliquant du ciment résine adhésif dentaire à base d’acrylique.
- Remettez la souris dans sa cage d’origine, en la plaçant sur un coussin chauffant. Surveillez la souris jusqu’à ce qu’elle retrouve une conscience suffisante pour maintenir le décubitus sternal (dans les 2 heures).
REMARQUE : Les souris doivent être complètement rétablies le lendemain et peuvent ensuite être hébergées avec d’autres animaux.
2. Préparation chirurgicale de souris pour l’imagerie in vivo à deux photons ; Jour 2
- Effectuez toutes les interventions chirurgicales à l’intérieur d’une armoire à flux d’air laminaire pour maintenir des conditions de travail stériles. Avant de commencer l’opération, stérilisez l’intérieur de l’armoire avec de la lumière UV pendant 5 minutes.
- Laminez ensemble deux lamelles en verre de dimensions différentes (verre supérieur : 3 mm × 3 mm ; verre inférieur : 2 mm × 2 mm) avec de la résine de qualité optique durcissable aux UV.
REMARQUE : La double lamelle offre une protection à long terme à la région du cerveau exposée par la fenêtre crânienne tout en permettant un accès optique chronique. Ainsi, ses dimensions peuvent être modifiées pour s’adapter à la région du cerveau à imager. - Stérilisez toutes les surfaces de travail et le cadre du cabinet en les essuyant avec de l’éthanol à 70 %.
- Stérilisez tous les instruments chirurgicaux (perceuse en acier, pince à épiler, crochet à aiguille chirurgicale) en les immergeant dans une solution de gluconate de chlorhexidine à 1 %.
- Anesthésier la souris avec de l’isoflurane (4 % d’induction, 1,2 % à 1,5 % d’entretien). Fixez la souris dans le cadre de l’opération via sa plaque de tête.
- Pour créer une fenêtre crânienne sur le cortex moteur primaire, marquez un carré de 2 mm × 2 mm centré à 0,2 mm en avant et à 1 mm latéralement du point de repère du crâne de Bregma.
- Amincissez le crâne le long du bord du carré marqué à l’aide de la perceuse en acier.
REMARQUE : Lorsqu’ils sont proches de l’épaisseur souhaitée, les zones du crâne amincies apparaîtront transparentes lorsqu’elles seront mouillées avec une solution saline, et des fissures capillaires commenceront à apparaître. - Après avoir vérifié que toute la bordure du carré marqué a été correctement amincie, insérez soigneusement le crochet de l’aiguille chirurgicale juste sous la surface du crâne, en orientant sa pointe vers le centre du carré. Soulevez doucement le morceau carré du crâne avec le crochet et utilisez une pince à épiler pour le décoller du reste du crâne. En cas de saignement, lavez continuellement la surface du cerveau exposée avec une solution saline jusqu’à ce qu’elle disparaisse complètement.
- Placez la lamelle double sur la surface exposée du cerveau, en orientant le côté avec la plus petite lamelle vers le cerveau. Assurez-vous que les bords de la plus grande lamelle sont en contact avec les bords de la fenêtre crânienne.
- À l’aide d’une tige de verre à pointe de silicium montée dans un manipulateur, appuyez doucement sur la lamelle double pour vous assurer qu’elle entre bien en contact avec la surface du cerveau.
- Remplissez l’espace entre la double lamelle, le crâne et la surface du cerveau avec de la résine durcissable aux UV et irradiez avec de la lumière UV jusqu’à ce qu’elle durcisse (~20 s). Soulevez lentement la tige de verre à pointe de silicium pour l’éloigner de la lamelle double.
- Laissez la souris récupérer en la plaçant sur un coussin chauffant. Surveiller la souris jusqu’à ce qu’elle retrouve une conscience suffisante pour maintenir sa position couchée sternale (30 min).
- Passez à l’étape 3 ou remettez la souris dans sa cage d’origine.
REMARQUE : Après une intervention chirurgicale efficace, la dure-mère et tous les vaisseaux sanguins sous-jacents seront complètement intacts sans saignement. Dans ce cas, l’inflammation est minime et il est possible de passer immédiatement à l’étape 3. En cas de doute, attendez 1 à 3 semaines pour vous assurer que toute inflammation est complètement résolue.
3. Collecte de données à l’aide de l’imagerie in vivo à deux photons
- Allumez la configuration d’imagerie à l’avance pour vous assurer que le laser a suffisamment de temps pour se réchauffer et se stabiliser. Réglez le laser pour qu’il émette à une longueur d’onde de 920 nm, qui est le spectre à deux photons qui excite de manière optimale le fluorophore GCaMP6.
- Placez la souris sous l’objectif d’un microscope 25x et habituez-la pendant 30 min. Si nécessaire, anesthésier la souris avec de l’isoflurane pendant l’imagerie (4 % d’induction, 1,2 % à 1,5 % d’entretien).
REMARQUE : L’isoflurane peut ne pas convenir à certaines applications de recherche, car il affecte la motilité du processus microglial et l’activité Ca2+ 19,20,21. - Réglez le zoom sur 1x, puis recherchez les microglies exprimant GCaMP6 dans la zone de la fenêtre crânienne. Cherchez à une profondeur comprise entre 100 et 300 m sous la surface du cerveau.
- Une fois qu’une microglie exprimant GCaMP6 appropriée a été trouvée, maximisez le zoom afin que l’activité de Ca2+ puisse être capturée avec une résolution de cellule unique.
- Vérifiez visuellement que l’activité Ca2+ est clairement capturée et ajustez la puissance du laser et le gain d’image si nécessaire.
REMARQUE : La puissance du laser doit être minimisée autant que possible pour éviter le photoblanchiment et les lésions de la microglie. - Effectuez l’acquisition d’images quadridimensionnelles (4D) comme suit (étapes 3.6.1-3.6.3 ; Figure 1B) :
- Réglez les dimensions de l’image sur la zone XY = 512 × 512 pixels, 0,25 μm/pixel ; Aire Z = cinq plans z, pas z de 3 μm (Figure 1B).
- Réglez le taux d’acquisition sur 2,5 images/s.
REMARQUE : Le balayage en Z à cette vitesse est facilité par un système de nano-positionnement piézoélectrique. - Acquérir des données jusqu’à ce qu’au moins 10 événements individuels Ca2+ soient observés (généralement 10 min).
4. Préparation à l’analyse (correction de mouvement, projection z moyenne/max)
- Avant de procéder à l’analyse de l’activité microgliale Ca2+ , corrigez les images 4D pour détecter les artefacts liés au mouvement (recalage d’images) à l’aide de l’algorithme d’alignement d’images ECC22 dans l’environnement de programmation MATLAB (R2020a). Téléchargez le code à partir de : https://www.mathworks.com/matlabcentral/fileexchange/27253-ecc-image-alignment-algorithm-image-registration.
- Créez une image 3D de référence en enregistrant toutes les images dans la première période à l’aide de l’opération imregister MATLAB (boîte à outils de traitement d’images standard).
- Enregistrez toutes les images suivantes, en les faisant correspondre avec le plan z correspondant de l’image 3D de référence à l’aide de la fonction ecc (bibliothèque d’algorithmes d’alignement d’images ECC).
- Générez des projections z d’intensité moyenne à partir des images enregistrées à l’aide de l’opération MATLAB moyenne . Si le signal d’imagerie est trop faible, générez plutôt des projections z d’intensité maximale en utilisant l’opération MATLAB max .
REMARQUE : Le rapport signal/bruit est moins bon lors de l’utilisation de projections z d’intensité maximale. - Passez à l’étape 5 ou 6. Utilisez les projections z générées à l’étape 4 comme cible d’analyse pour cartographier la dynamique spatio-temporelle de l’activité Ca2+ aux étapes 5 et 6.
5. Analyse basée sur le retour sur investissement
- À l’aide des projections z générées à l’étape 4, identifiez les processus microgliaux qui maintiennent un profil de zone stable (zone stable) tout au long de la période d’imagerie pour une analyse ultérieure de l’activité Ca2+ comme suit (étapes 5.3-5.6) :
- Générez des projections t d’intensité maximale distinctes des images 4D enregistrées à partir d’échantillons de 2 minutes prélevés au début et à la fin de la période d’imagerie à l’aide de l’opération MATLAB max .
- Binariser les projections t pour générer des polygones de la morphologie microgliale correspondant au début et à la fin de la période d’imagerie à l’aide de l’opération imbinariser MATLAB. Utilisez le seuil par défaut défini automatiquement.
REMARQUE : Si le signal Ca2+ est faible au début et à la fin de la période d’imagerie, la binarisation peut générer des polygones avec des bordures manquantes. Dans ce cas, dessinez manuellement les bordures manquantes à l’aide de l’outil crayon d’ImageJ. - Superposez les polygones de projection t à l’aide de l’opération MATLAB imadd . La ou les régions qui se chevauchent représentent la zone stable (figure 2B).
- Dans les zones stables identifiées, utilisez l’opération MATLAB drawpolygon pour définir et tracer manuellement les retours sur investissement de chacun des processus microgliaux primaires et de toutes les sous-branches de second ordre évidentes.
- Suivez les intensités de fluorescence absolues moyennes sur un retour sur investissement individuel sur toutes les périodes (Figure 2C,D).
- À partir de la série chronologique des intensités absolues de fluorescence, calculer la variation relative de l’intensité de fluorescence (ΔF/F) selon l’équation (1). Cette série chronologique représente la dynamique microgliale normalisée du Ca2+ au niveau des ROI individuels.
ΔF/F = (F(t) - F0) / F0 (1)
Où F(t) est l’intensité de fluorescence enregistrée à un moment donné et F0 est le 10e centile de l’intensité de fluorescence dans toutes les périodes de temps (Figure 2E). - Identifiez les événements de déclenchement Ca2+ candidats qui se produisent dans un retour sur investissement individuel donné comme suit (étapes 5.5.1-5.5.3) :
- Appliquez un filtrage passe-bas à réponse impulsionnelle finie (FIR) de type I sur les séries temporelles ΔF/F à l’aide de l’opération MATLAB fir1 . Réglez la fréquence de coupure sur la valeur de Nyquist (la moitié de la fréquence d’échantillonnage).
- Inspectez visuellement la trace filtrée pour confirmer que les pics de trace individuels correspondent à des rafales d’activité Ca2+ dans les images 4D enregistrées (Figure 2F).
- Identifiez les événements de décharge Ca2+ candidats en tant qu’inflexions descendantes concaves dans la trace de la série chronologiqueΔF/F. Définissez un seuil de référence comme la valeur médiane ΔF/F dans les plafonds supérieur et inférieur qui excluent les amplitudes maximale et minimale de 10 % sur toutes les périodes. Définissez un seuil de détection de trois écarts-types au-dessus du seuil de référence (Figure 2F,H).
- Identifiez les véritables événements de tir Ca2+ chez les candidats comme suit (étapes 5.5.1-5.5.2) :
- Calculez la pente de chaque événement candidat en dérivant les images de séries temporelles filtrées ΔF/F correspondantes à l’aide de l’opération MATLAB à gradient numérique .
- Identifiez ensuite les véritables événements Ca2+ en fonction du temps de montée du profil. Définissez un seuil de référence comme la moyenne des valeurs de pente pour tous les événements candidats. Définissez un seuil de détection de trois écarts-types au-dessus du seuil de référence (Figure 2G,H).
- Caractérisez les événements Ca2+ vrais comme suit (étapes 5.6.1-5.6.3) :
REMARQUE : Ce qui suit n’est pas une liste exhaustive des paramètres qui peuvent être utilisés pour caractériser les événements microgliaux Ca2+ . Les paramètres d’intérêt dépendent de l’objectif de l’étude.- Dérivez l’amplitude maximale d’un véritable événement de décharge Ca2+ comme la valeur ΔF/F de la trame de série chronologique ΔF/F correspondante.
- Dérivez l’amplitude moyenne d’un véritable événement de décharge Ca2+ comme la valeur moyenne de ΔF/F sur l’ensemble du sous-ensemble de séries temporelles ΔF/F correspondant.
- Dérivez la fréquence des événements Ca2+ vrais en divisant le nombre d’événements par le temps d’imagerie.
6. Analyse basée sur les événements
- Effectuez une analyse basée sur les événements sur les projections z à partir de l’étape 4 à l’aide de la bibliothèque AQuA dans l’environnement de programmation MATLAB. Téléchargez le code à partir de : https://github.com/yu-lab-vt/AQuA.
REMARQUE : Une procédure pas à pas d’utilisation générale de la bibliothèque AQuA est disponible à l’adresse suivante : https://drive.google.com/file/d/1a3lhe0dUth-5J1-S2fZlPOCZlPbeuvUr/view. La documentation détaillée de la bibliothèque AQuA est disponible à l’adresse suivante : https://drive.google.com/file/d/1CckDLbrkw16b7MPlOQdYpZciIz80Snm_/view. - Après avoir lancé MATLAB, passez du dossier de répertoire de travail par défaut au dossier de répertoire de travail désigné par AQuA à l’aide de l’opération cd .
- Chargez les images 4D enregistrées dans le pipeline d’analyse AQuA comme suit (étapes 6.3.1-6.3.2) :
- Lancez l’interface graphique d’AQuA. Saisissez aqua_gui dans la fenêtre de commande MATLAB du dossier de répertoire de travail désigné d’AQuA.
- Dans l’interface graphique d’AQuA, cliquez sur nouveau projet et sélectionnez les images enregistrées à analyser. Spécifiez le type de données (GCaMPInVivo_cyto_Lck_) et les paramètres d’imagerie (résolution temporelle secondes par image = 1,993 ; résolution spatiale μm par pixel = 0,25 ; exclure les pixels plus courts que cette distance à la bordure = 5). Cliquez sur Ouvrir pour charger les données.
- Définissez des points de repère pour les pipelines d’analyse suivants en traçant les zones d’intérêt. Habituellement, les points de repère sont basés sur la limite cellulaire et l’aire du corps cellulaire.
- Par la suite, détectez les événements candidats Ca2+ en exécutant les pipelines d’analyse automatisés pour les paramètres suivants : signal actif, super voxel, détection d’événements, événements propres et événements de fusion.
REMARQUE : Une explication détaillée de chacun de ces paramètres et de leurs résultats de score candidat est fournie dans la procédure pas à pas AQuA. En bref, ces paramètres ajustent le seuil de détection d’événements Ca2+ comme suit : signal actif = amplitude de fluorescence, super voxel = regroupement de fluorescence dans l’espace 3D, détection d’événements = cinétique de montée/décroissance, événements propres = rapport signal/bruit et événements de fusion = séparation temporelle des événements. - Inspectez visuellement les événements Ca2+ détectés (automatiquement superposés aux images enregistrées d’origine). Si nécessaire, ajustez les paramètres des pipelines d’analyse en tenant compte de la qualité de l’image en fonction des paramètres ci-dessus ; Si la qualité d’image d’un paramètre donné est bonne, un seuil plus élevé peut être défini, et vice versa.
- Une fois que tous les événements Ca2+ ont été détectés de manière appropriée, caractérisez ces événements dans l’environnement MATLAB principal comme suit (étapes 6.8-6.14) :
- Exportez les fichiers de sortie de l’analyse AQuA.
REMARQUE : La documentation AQuA fournit une explication détaillée des fichiers de sortie, des entités extraites utilisées pour définir les événements Ca2+ et des paramètres sous-jacents de ces entités extraites. - (facultatif) Catégorisez tous les événements Ca2+ en deux groupes : 1) les événements commençant au soma et 2) les événements commençant au niveau des processus.
- Accédez à l’amplitude des événements Ca2+ individuels dans la structure MATLAB res.dffMat du fichier de sortie d’analyse AQuA (fichier .mat).
- Dérivez la fréquence des événements Ca2+ individuels à l’aide de l’opération MATLAB res.fts.loc.x3D (bibliothèque AQuA).
- Dérivez la durée des événements Ca2+ individuels à l’aide de l’opération MATLAB res.fts.curve.width11 (bibliothèque AQuA).
- Dérivez l’aire d’événements Ca2+ individuels à l’aide de l’opération MATLAB res.fts.basic.area (bibliothèque AQuA).
- Catégorisez tous les événements Ca2+ comme suit : 1) événements locaux, 2) événements de propagation voyageant vers le soma et 3) événements de propagation s’éloignant du soma. Pour ce faire, utilisez les opérations MATLAB res.fts.region.landmarkDir.chgToward et res.fts.region.landmarkDir.chgAway (bibliothèque AQuA) (Figure 3C).
Résultats
Chez les souris transgéniques exprimant exclusivement GCaMP6 (protéine fluorescente sensible au Ca2+) dans la microglie, nous observons généralement divers modèles d’activité microgliale du Ca2+ (Figure 2A). Il est important de noter que même au sein d’une seule microglie, les modèles d’activité de Ca2+ peuvent différer considérablement entre les processus.
Pour quantifier de telles différences de processus à processus dans la dynamique spatio-temporelle de l’activité microgliale Ca2+, les zones stables doivent d’abord être identifiées, puis divisées en ROI finement segmentés (Figure 2B,C). Pour chaque ROI, des paramètres d’activité Ca2+ doivent être dérivés et quantifiés, tels que l’amplitude et la fréquence, en extrayant des caractéristiques telles que les amplitudes locales et les pentes de traces de la série temporelle d’intensité de fluorescence (Figure 2D-G).
Ensuite, les événements individuels Ca2+ doivent être examinés en appliquant l’algorithme de quantification précis AQuA (Figure 3A). À partir de ces analyses basées sur les événements, de grandes différences sont généralement observées dans l’origine, l’amplitude, la durée, l’emplacement et les caractéristiques de direction d’écoulement des événements Ca2+ individuels (Figure 3B). Si l’on se concentre sur l’analyse de la dynamique de l’activité de Ca2+ dans les processus microgliaux, un schéma de classification des événements locaux, des événements se déplaçant vers le soma et des événements s’éloignant du soma est informatif (Figure 3C).
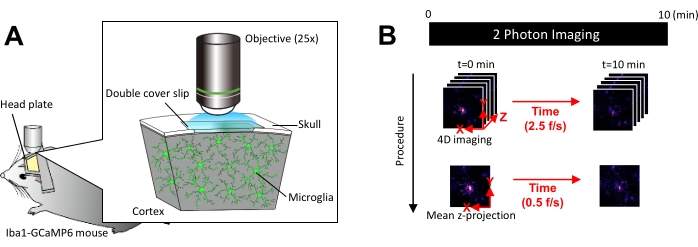
Figure 1 : Dispositif expérimental d’imagerie microgliale Ca2+ in vivo. (A) Dispositif expérimental. Une souris Iba1-tTA × tetO-GCaMP6 avec expression de GCaMP6 spécifique à la microglie. En insérant une fenêtre crânienne dans le crâne de la souris, l’activité microgliale Ca2+ peut être observée in vivo à l’aide de la microscopie à deux photons. (B) Calendrier expérimental et procédure d’analyse. Les images 4D sont acquises pendant au moins 10 minutes sous forme de piles Z de cinq images. Le taux d’acquisition d’images est de 2,5 images/s. Avant d’analyser l’activité microgliale de Ca2+, les empilements z à cinq images sont convertis en projections z 2D en prenant l’intensité moyenne (ou maximale). La vitesse de lecture de la projection z est de 0,5 image/s. Veuillez cliquer ici pour voir une version agrandie de cette figure.
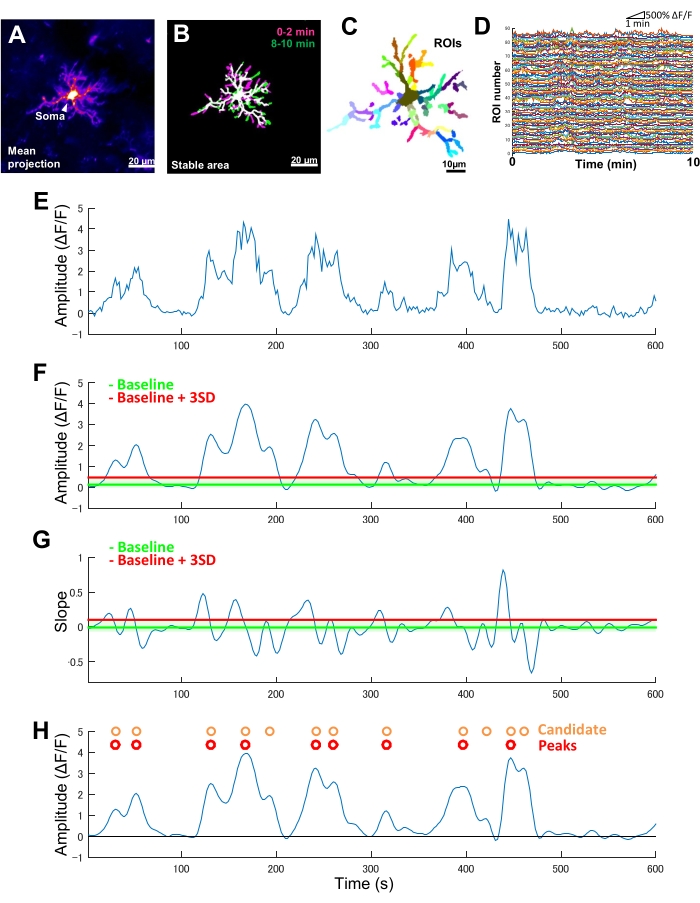
Figure 2 : Analyse basée sur le retour sur investissement de l’activité microgliale Ca2+. (A) Projection d’intensité moyenne de GCaMP6 sur 10 min pour une microglie individuelle. (B) Les zones stables (en blanc) sont définies par une superposition de projections t binarisées d’intensité maximale de GCaMP6 dérivées d’échantillons de 2 minutes prélevés au début (magenta) et à la fin (en vert) d’une période d’imagerie. (C) Les zones stables sont davantage segmentées en ROI régionaux. Les couleurs individuelles indiquent les retours sur investissement individuels. (D) Traces ΔF/F de tous les retours sur investissement individuels en C. Notez la variation des modèles d’activité entre les retours sur investissement. (E) Trace originale d’une série chronologique ΔF/F dérivée des valeurs d’intensité absolues pour un seul ROI. (F) La même série chronologique ΔF/F après filtrage passe-bas. Les événements candidats Ca2+ sont détectés par un seuil de coupure d’amplitude (ligne rouge) défini comme la ligne de base + trois écarts-types. La ligne de référence (ligne verte) est définie comme la valeur médiane de l’ensemble de la série chronologique ΔF/F à l’intérieur d’un plafond supérieur et inférieur qui exclut les valeurs maximales et minimales de 10 % des valeurs ΔF/F. (G) La trace de pente dérivée de la série temporelle filtrée ΔF/F en F. Les événements Ca2+ vrais sont triés à partir des événements Ca2+ candidats en fonction d’un seuil de coupure de pente (ligne rouge) défini comme référence + trois écarts-types. La ligne de référence (ligne verte) est définie comme la valeur moyenne sur l’ensemble de la série chronologique de pente à l’intérieur d’un plafond supérieur et inférieur qui exclut les valeurs maximales et minimales de 10 % des valeurs de pente. (H) Les événements candidats Ca2+ identifiés par des critères d’amplitude en F sont indiqués en orange. Véritables épreuves Ca2+ classées à partir des épreuves candidates Ca2+ par critères de pente en G en rouge. Notez que certaines épreuves candidates de Ca2+ ont été fusionnées sur la base des critères de pente. Les séries chronologiques filtrées ΔF/F correspondantes sont superposées ci-dessous à titre de référence. La ligne noire indique une amplitude nulle (ΔF/F). L’amplitude moyenne et maximale des événements Ca2+ vrais est dérivée comme la moyenne et le maximum de leurs pics correspondants dans la série chronologique filtrée ΔF/F. La fréquence (événements/min) est calculée en divisant le nombre d’événements de décharge Ca2+ réels par la période d’imagerie (10 min). Barres d’échelle = 20 μm (A,B), 10 μm (C). Abréviation : ROI = région d’intérêt. Veuillez cliquer ici pour voir une version agrandie de cette figure.
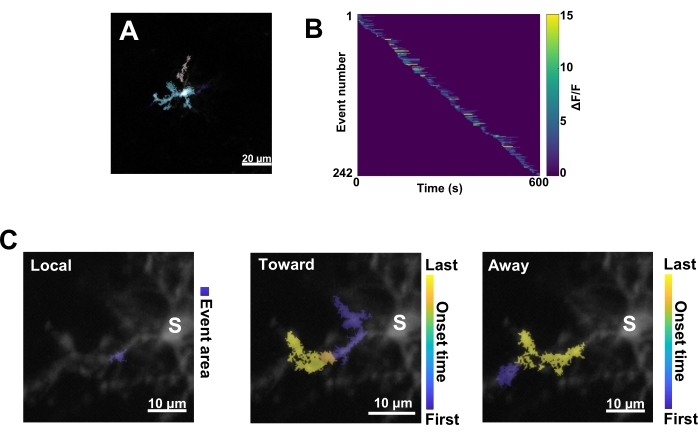
Figure 3 : Analyse basée sur les événements pour l’activité microgliale Ca2+. (A) Images représentatives des événements détectés à l’aide de l’algorithme AQuA. Les couleurs indiquent les zones d’événements individuelles détectées à un moment donné. (B) Activité Ca2+ normalisée représentative (ΔF/F) dans les événements individuels triés par ordre d’apparition. La barre de droite indique la couleur ΔF/F. (C) Empreintes d’activité représentatives des événements se propageant vers et loin du soma ou des événements locaux. Pour les événements locaux, la forme de l’événement est indiquée en bleu. Pour les événements de multiplication, l’heure d’apparition de l’événement est indiquée par une échelle bleu-jaune. Étant donné que l’AQuA détecte initialement les événements Ca2+ individuellement, les événements de propagation sont identifiés par la suite en fonction des emplacements spatiaux qui se chevauchent et des séries chronologiques de plusieurs événements individuels Ca2+. Notez qu’il s’agit de la même branche dendritique utilisée pour l’analyse du retour sur investissement dans la figure 2E-H. Barres d’échelle = 20 μm (A), 10 μm (C). Abréviation : ROI = région d’intérêt ; S = soma. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Cet article présente une approche améliorée pour l’imagerie de l’activité microgliale Ca2+ avec une résolution spatio-temporelle élevée. Cette méthode est suffisamment sensible pour détecter différents types d’activité microgliale Ca2+ au niveau des processus ramifiés uniques, en distinguant facilement les événements locaux et les événements de propagation.
Dans la méthode générale d’imagerie à deux photons in vivo de l’activité microgliale Ca2+ , une attention particulière doit être portée aux points suivants pour maximiser la qualité de l’imagerie. Tout d’abord, comme les microglies sont extrêmement sensibles aux blessures, il est important de minimiser le contact direct avec la surface du cerveau avec des outils chirurgicaux pendant la chirurgie. Les principales indications que la chirurgie a été pratiquée avec compétence sont des vaisseaux sanguins et une dure-mère intacts et des saignements très minimes pendant la chirurgie. Deuxièmement, la fixation sécurisée de la plaque frontale au crâne de la souris et un bon contact entre la double lamelle et la surface du cerveau réduisent considérablement les artefacts liés au mouvement pendant l’imagerie. Ceci est particulièrement important lors de l’imagerie avec des résolutions spatio-temporelles élevées et chez des souris complètement éveillées. Alors que le pipeline d’analyse compense de manière fiable les artefacts liés au mouvement résultant du rythme cardiaque, de la respiration et de la dérive générale, il est moins robuste lorsqu’il s’agit de gérer des distorsions géométriques importantes résultant de mouvements brusques et importants.
Les deux méthodes d’analyse décrites ici offrent des avantages différents et sont adaptées à des questions de recherche différentes. Dans l’analyse basée sur le ROI, l’utilisateur prédéfinit le ROI (comme les processus individuels), ce qui permet d’extraire la dynamique agrégée de l’activité Ca2+ de ce ROI. Ainsi, il est le plus adapté aux situations où l’on s’attend à ce que les phénomènes soient localisés dans une zone subcellulaire qui a à la fois des limites morphologiques bien définies et une zone relativement grande (c’est-à-dire une branche de processus). Dans l’analyse basée sur les événements, les événements individuels sont définis en fonction de la dynamique spatio-temporelle de l’activité microgliale Ca2+ elle-même et doivent ensuite être placés dans le contexte de points de repère définis par l’utilisateur au sein de la microglie pour que leur fonction soit interprétée. Ainsi, il est le plus adapté aux situations où des hypothèses sur la localisation des phénomènes ne peuvent pas être faites ou lorsque la zone d’intérêt est relativement petite (c’est-à-dire une pointe de processus). En tant que telle, l’analyse basée sur les événements offre une meilleure résolution spatio-temporelle lors de la caractérisation de l’activité microgliale Ca2+ par rapport aux méthodes précédentes.
Chez ces souris, le seul marqueur fluorescent exprimé par la microglie est l’indicateur Ca2+ GCaMP6. Ainsi, dans les régions où l’activité de Ca2+ est faible, la morphologie microgliale doit être extraite en combinant plusieurs cadres temporels, ce qui peut dégrader la résolution temporelle. Cependant, cette limitation peut être surmontée en exprimant une protéine rouge distincte à fluorescence stable dans la microglie. Notamment, de nouveaux virus adéno-associés capables de transfecter la microglie ont récemment été décrits 23,24,25.
La façon dont l’activité microgliale Ca2+ est modifiée par l’environnement environnant est un sujet d’intérêt émergent. En particulier, l’activité microgliale Ca2+ semble montrer des corrélations significatives avec l’activité neuronale, bien que la signification fonctionnelle de celle-ci n’ait pas encore été complètement caractérisée. Ainsi, la combinaison de la manipulation de l’activité neuronale avec les méthodes d’imagerie et d’analyse de l’activité microgliale Ca2+ présentées ici devrait apporter de nouvelles connaissances sur la physiologie microgliale et faire progresser notre compréhension des rôles que jouent les microglies dans les états physiologiques et pathologiques.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts associé à ce manuscrit.
Remerciements
Nous sommes reconnaissants au Prof. Kenji Tanaka (Keio University, Tokyo, Japon) de nous avoir fourni des souris Iba1-tTA et des souris tetO-GCaMP6. Ce travail a été soutenu par les subventions pour les jeunes scientifiques (B) [16K19001 (à S.H.)], les subventions pour les scientifiques en début de carrière [18K14825 (à H.H.)], les subventions pour la recherche scientifique (B) [21H03027 (à S.H.)], les subventions pour les domaines de recherche transformateurs (A) [21H05639 (à S.H.)], les subventions pour la recherche scientifique (A) [17H01530, 20H00500 (à J.N.)] et la subvention JST CREST [JPMJCR1755 (à J.N.)], Japon.
matériels
| Name | Company | Catalog Number | Comments |
| 2% xylocaine jelly | AstraZeneca, UK | ||
| B6(129S6)-Tg(Aif1-tTA)54Kftnk | RIKEN RBC, Japan | RBRC05769 | Iba1-tTA mice |
| B6;129-Actb(tm3.1(tetO-GCaMP6)Kftnk) | RIKEN RBC, Japan | RBRC09552 | tetO-GCaMP6 mice |
| Forceps | Fine science tools, US | 13008-12 | |
| G-CEM ONE | GC corporation, Japan | ||
| Glass capillary | Narishige, Japan | GDC-1 | |
| ImageJ | NIH, US | ||
| Isofulrane | Pfizer, US | ||
| Ketamin | Daiichi-Sankyo, Japan | ||
| Kwik-sil | World Precision Instruments, US | KWIK-SIL | |
| MATLAB, 2020a | MathWorks, US | ||
| Micro cover glass (2 x 2 mm, No.3) | Matsunami, Japan | custum-made | Bottom glass for cranial window |
| Micro cover glass (3 x 3 mm, No.0) | Matsunami, Japan | custum-made | Upper glass for cranial window |
| N25X-APO-MP | Nikon, Japan | N25X-APO-MP | Objective lens (25x) |
| Norland optical adhesive | Edmund optics, US | 6101 | |
| Piezo nano-positioning system, Nano-Drive | Mao City Labs, US | ||
| Razor blade | Feather, Japan | FA-10 | |
| Scissors | Fine science tools, US | 14060-11 | |
| Steel drill | Minitor, Japan | BS1201 | |
| Stereotaxic instruments | Narishige, Japan | SR-5M-HT | |
| Super-bond (C&B kit) | Sun Medical, Japan | 4560227797382 | |
| Surgical needle hook | Fine science tools, US | 10065-15 | |
| Ti:Sappire laser, MaiTai DeepSee | Spectra Physics, US | Mai Tai eHP DS | |
| Tweezers | Fine science tools, US | 11051-10 | |
| Tweezers | Fine science tools, US | 11255-20 | |
| Two-photon microscope | Nikon, Japan | A1R-MP | |
| UV craft resin | Kiyohara, Japan | UVR | |
| Xylazine | Bayer, Germany |
Références
- Miyamoto, A., et al. Microglia contact induces synapse formation in developing somatosensory cortex. Nature Communications. 7, 12540 (2016).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Davalos, D., et al. ATP mediates rapid microglial response to local brain injury in vivo. Nature Neuroscience. 8 (6), 752-758 (2005).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Science. 308 (5726), 1314-1318 (2005).
- Cserep, C., et al. Microglia monitor and protect neuronal function through specialized somatic purinergic junctions. Science. 367 (6477), 528-537 (2020).
- Kato, G., et al. Microglial contact prevents excess depolarization and rescues neurons from excitotoxicity. eNeuro. 3 (3), (2016).
- Li, Y., Du, X. -. F., Liu, C. -. S., Wen, Z. -. L., Du, J. -. L. Reciprocal regulation between resting microglial dynamics and neuronal activity in vivo. Developmental Cell. 23 (6), 1189-1202 (2012).
- Tremblay, M. -. E., Lowery, R. L., Majewska, A. K. Microglial interactions with synapses are modulated by visual experience. Plos Biology. 8 (11), 1000527 (2010).
- Wake, H., Moorhouse, A. J., Jinno, S., Kohsaka, S., Nabekura, J. Resting microglia directly monitor the functional state of synapses in vivo and determine the fate of ischemic terminals. The Journal of Neuroscience. 29 (13), 3974-3980 (2009).
- Oppenheim, R. W. Cell death during development of the nervous system. Annual Review of Neuroscience. 14 (1), 453-501 (1991).
- Sierra, A., Abiega, O., Shahraz, A., Neumann, H. Janus-faced microglia: beneficial and detrimental consequences of microglial phagocytosis. Frontiers in Cellular Neuroscience. 7, 6 (2013).
- Brawek, B., et al. A new approach for ratiometric in vivo calcium imaging of microglia. Scientific Reports. 7 (1), 6030 (2017).
- Eichhoff, G., Brawek, B., Garaschuk, O. Microglial calcium signal acts as a rapid sensor of single neuron damage in vivo. Biochimica et Biophysica Acta. 1813 (5), 1014-1024 (2011).
- Liu, L., et al. Microglial calcium waves during the hyperacute phase of ischemic stroke. Stroke. 52 (1), 274-283 (2021).
- Pozner, A., et al. Intracellular calcium dynamics in cortical microglia responding to focal laser injury in the PC::G5-tdT reporter mouse. Frontiers in Molecular Neuroscience. 8, 12 (2015).
- Umpierre, A. D., et al. Microglial calcium signaling is attuned to neuronal activity in awake mice. Elife. 9, e56502 (2020).
- Ohkura, M., et al. Genetically encoded green fluorescent Ca2+ indicators with improved detectability for neuronal Ca2+ signals. Plos One. 7 (12), e51286 (2012).
- Tanaka, K. F., et al. Expanding the repertoire of optogenetically targeted cells with an enhanced gene expression system. Cell Reports. 2 (2), 397-406 (2012).
- Liu, Y. U., et al. Neuronal network activity controls microglial process surveillance in awake mice via norepinephrine signaling. Nature Neuroscience. 22 (11), 1771-1781 (2019).
- Stowell, R. D., et al. Noradrenergic signaling in the wakeful state inhibits microglial surveillance and synaptic plasticity in the mouse visual cortex. Nature Neuroscience. 22 (11), 1782-1792 (2019).
- Sun, W. L., et al. In vivo two-photon imaging of anesthesia-specific alterations in microglial surveillance and photodamage-directed motility in mouse cortex. Frontiers in Neuroscience. 13, 421 (2019).
- Evangelidis, G. D., Psarakis, E. Z. Parametric image alignment using enhanced correlation coefficient maximization. IEEE Transactions on Pattern Analysis and Machine Intelligence. 30 (10), 1858-1865 (2008).
- Rosario, A. M., et al. Microglia-specific targeting by novel capsid-modified AAV6 vectors. Molecular Therapy. Methods & Clinical Development. 3, 16026 (2016).
- Maes, M. E., Wogenstein, G. M., Colombo, G., Casado-Polanco, R., Siegert, S. Optimizing AAV2/6 microglial targeting identified enhanced efficiency in the photoreceptor degenerative environment. Molecular Therapy. Methods & Clinical Development. 23, 210-224 (2021).
- Lin, R., et al. Directed evolution of adeno-associated virus for efficient gene delivery to microglia. Nature Methods. 19 (8), 976-985 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon