Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Descifrando el mecanismo molecular y la función de las toxinas formadoras de poros usando Leishmania major
En este artículo
Resumen
Aquí se presenta un protocolo que utiliza promastigotes mayores de Leishmania para determinar la unión, citotoxicidad y señalización inducida por toxinas formadoras de poros. Se proporciona una prueba de concepto con estreptolisina O. Otras toxinas también se pueden utilizar para aprovechar los mutantes genéticos disponibles en L. major para definir nuevos mecanismos de resistencia a las toxinas.
Resumen
Comprender la función y el mecanismo de las toxinas formadoras de poros (PFT) es un desafío porque las células resisten el daño de la membrana causado por las PFT. Si bien los enfoques biofísicos ayudan a comprender la formación de poros, a menudo se basan en enfoques reduccionistas que carecen del complemento completo de lípidos y proteínas de membrana. Las células humanas cultivadas proporcionan un sistema alternativo, pero su complejidad y redundancias en los mecanismos de reparación dificultan la identificación de mecanismos específicos. En contraste, el patógeno protozoario humano responsable de la leishmaniasis cutánea, Leishmania major, ofrece un equilibrio óptimo entre complejidad y relevancia fisiológica. L. major es genéticamente tratable y puede ser cultivado a alta densidad in vitro, y cualquier impacto de las perturbaciones en la infección puede medirse en modelos murinos establecidos. Además, L. major sintetiza lípidos distintos de sus homólogos mamíferos, lo que podría alterar la dinámica de la membrana. Estas alteraciones en la dinámica de la membrana se pueden probar con PFT de la familia de toxinas mejor caracterizada, las citolisinas dependientes del colesterol (CDC). Los CDC se unen al ergosterol en la membrana de Leishmania y pueden matar los promastigotes de L. major, lo que indica que L. major es un sistema modelo adecuado para determinar los mecanismos celulares y moleculares de la función de PFT . Este trabajo describe métodos para probar la función de PFT en promastigotes de L. major , incluido el cultivo de parásitos, herramientas genéticas para evaluar la susceptibilidad a los lípidos, ensayos de unión a la membrana y ensayos de muerte celular. Estos ensayos permitirán el uso rápido de L. major como un poderoso sistema modelo para comprender la función de PFT en una gama de organismos evolutivamente diversos y puntos en común en la organización de lípidos.
Introducción
Las toxinas formadoras de poros (PFT) son la familia más grande de toxinas bacterianas1, pero los mecanismos por los cuales perforan y destruyen las células son poco conocidos. La familia mejor estudiada de toxinas formadoras de poros es la de las citolisinas dependientes del colesterol (CDC). Los CDC son sintetizados principalmente por bacterias grampositivas, incluido el agente causal de la fascitis necrotizante, Streptococcus pyogenes2. S. pyogenes secreta la estreptolisina O (SLO) de los CDC, que se une a los esteroles en la membrana plasmática de las células huésped como monómeros, oligomeriza e inserta poros ~20-30 nm en la membrana1. El papel que juegan los lípidos en este proceso sigue estando poco determinado.
Un enfoque para estudiar las interacciones lípido-CDC es el uso de liposomas químicamente definidos. Si bien los liposomas definidos proporcionan información sobre los umbrales necesarios de lípidos para mantener la unión a toxinas y la formación de poros3,4, no recapitulan completamente las funciones celulares. Por ejemplo, los liposomas reconstituidos carecen de la asimetría lipídica de los huéspedes mamíferos y de las modificaciones lipídicas en respuesta a las toxinas5. Una alternativa a los liposomas es utilizar líneas celulares de mamíferos. Si bien estas líneas celulares son más relevantes fisiológicamente, existe un alto grado de redundancia en los mecanismos de detección y resistencia a toxinas2. Como consecuencia, las vías de reparación utilizadas para resistir los CDC siguen estando mal determinadas. En particular, la afluencia de Ca2+ es el principal activador de la reparación de la membrana1. Aguas abajo de la afluencia de Ca2+, se activan múltiples vías, incluida una reparación dependiente de ceramida 6,7 y una vía de reparación dependiente de MEK6. Estas vías interactúan con otros efectores proteicos, incluyendo el complejo de clasificación endosomal requerido para el transporte (ESCRT)8, y las anexinas 6,9,10. La disección de estas vías en células de mamíferos es un desafío debido a la redundancia, que confunde la interpretación de los datos.
Una forma de equilibrar la complejidad con la simplicidad para diseccionar las vías de reparación es el uso de organismos más simples, como los patógenos protozoarios del género Leishmania. Leishmania sp. causa leishmaniasis en humanos y otros animales. La leishmaniasis varía desde la leishmaniasis cutánea (lesiones cutáneas autolimitadas) hasta la leishmaniasis visceral fatal (hepatoesplenomegalia), dependiendo de la especie y otros factores11. La Leishmania mayor, el agente causal de la leishmaniasis cutánea, se transmite a los humanos a través de un vector de flebótomo y se utiliza para comprender la función y la infección de Leishmania 12. Además, Leishmania sp. son digénicos12. Existen como parásitos macrófagos mamíferos intracelulares denominados amastigotes y como promastigotes flagelados que nadan libremente en el flebótomo12. Los promastigotes de L. major pueden cultivarse en medios suplementados con suero como M199 a alta densidad13. Los promastigotes también son genéticamente tratables; Existen muchos knockouts genéticos, incluidos los dirigidos a las vías de biosíntesis de lípidos13. Estos knockouts pueden ser evaluados para el crecimiento y las diferencias en la infectividad y el desarrollo de lesiones a través de la infección de ratones Balb/c13.
Además de la relativa facilidad del cultivo de Leishmania y la gama de knockouts de biosíntesis de lípidos, el parásito tiene un genoma más simple que los mamíferos. La especie mejor caracterizada de Leishmania es L. major, que tiene muchas herramientas genéticas existentes, como mutantes con metabolismo lipídico defectuoso14. En particular, muchas proteínas de reparación están ausentes. L. major no tiene homólogos identificados hasta la fecha para proteínas clave de reparación de mamíferos como las anexinas. Esto permite la caracterización de vías de reparación conservadas evolutivamente sin la complejidad de los sistemas de mamíferos. Sin embargo, las vías de reparación no se han caracterizado en Leishmania hasta la fecha. Al mismo tiempo, las vías de señalización clave involucradas en la reparación, como la vía MEK6, se conservan en Leishmania sp.15,16, aunque los homólogos deben ser validados. La vía de la proteína quinasa activada por mitógenos (MAPK) está bien estudiada en L. mexicana, donde contribuye a la supervivencia intracelular y la termoestabilidad en células de mamíferos y controla la metaciclogénesis16. En Leishmania sp., 10 de los 15 MAPK han sido caracterizados17. Se predice que LmMAPK9 y LmMAPK13 serán los más similares a ERK1/2 de mamíferos según la identidad en la secuencia labial de fosforilación conservada. La secuencia de fosforilación del labio es TEY tanto para ERK1/2 de mamíferos como para LmMAPK9 y LmMAPK13. Sin embargo, ocho de los MAPK de Leishmania tienen un motivo de fosforilación TDY15. Se han identificado al menos dos homólogos de MEK en Leishmania sp., LmxMKK18 y quinasa relacionada con MEKK (MRK1)19. Esto sugiere que las ideas identificadas en Leishmania podrían traducirse a sistemas de mamíferos. Cuando no se traducen en sistemas de mamíferos, representan dianas terapéuticas para tratar la leishmaniasis.
Para utilizar L. major promastigotes para estudiar la reparación de la membrana y las interacciones con toxinas, se necesitan técnicas de rendimiento medio. Si bien las imágenes de células vivas de alta resolución permiten la visualización de proteínas y membranas marcadas en tiempo real, es de bajo rendimiento y es posible que no mida la supervivencia celular. Los ensayos de viabilidad de rendimiento medio incluyen la absorción de colorantes medida por citometría de flujo, la medición de la actividad mitocondrial o la liberación de proteínas celulares como la lactato deshidrogenasa (LDH). En células de mamíferos, los ensayos de LDH no miden cuantitativamente la muerte celular20. Además, los ensayos basados en la población, como la liberación de LDH o la actividad mitocondrial, no permiten un análisis robusto de una sola célula o multiparamétrico20. Por el contrario, los ensayos basados en citometría de flujo permiten el análisis multiparamétrico unicelular20. Sin embargo, estos ensayos no se han aplicado para comprender la biología de las toxinas o las respuestas a las toxinas en los promastigotes de L. major.
En este estudio, SLO se utiliza como una herramienta para comprender la perturbación de la membrana plasmática del mutante nulo esfingolípido de L. major en dos tampones diferentes: el medio M199 utilizado rutinariamente para cultivar promastigotes de L. major y el tampón de Tyrode, más simple . Se describe un ensayo de citometría de flujo de rendimiento medio que se utiliza para generar curvas dosis-respuesta de toxinas. Los datos del ensayo citométrico de flujo se modelan en una curva logística para determinar los valores de CL50 . Con esta información, se puede determinar una dosis sublítica de SLO para que los anticuerpos MAPK puedan validarse mediante Western blotting.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Se emplearon todas las directrices apropiadas y las prácticas microbiológicas, de seguridad y de cultivo celular estándar para el uso y manejo del patógeno RG2 Leishmania major y el ADN recombinante. Todos los experimentos con L. major vivo se realizaron en un gabinete de bioseguridad en un laboratorio certificado BSL-2. El trabajo fue supervisado por el Comité Institucional de Bioseguridad de la Universidad Tecnológica de Texas.
NOTA: Desde una perspectiva de seguridad, los promastigotes vivos de L. major son patógenos del Grupo de Riesgo 2. Manejar el uso de la contención adecuada, las precauciones y la supervisión del Comité Institucional de Bioseguridad (IBC). Manejar sustancias tóxicas y productos químicos de acuerdo con los procedimientos institucionales para sustancias tóxicas. Si se utilizan toxinas recombinantes, la aprobación y supervisión de IBC puede ser necesaria para el trabajo de ADN recombinante.
1. Cultivo y preparación de promastigotes de L. major
- Obtener, o fabricar y validar, mutantes genéticos principales de L. como se describió anteriormente utilizando métodos de recombinación homólogos o basados en CRISPR13,21. Use knockouts complementados con el gen agregado de nuevo en un plásmido para garantizar la especificidad del knockout.
- Cultivo silvestre tipo L. major y promastigotes spt2- a 27 °C en medio M199 completo. Cultivar las células episomales addback (spt2-/+SPT2) en M199 completo más 10 μg/mL G418 (ver Tabla 1 y Tabla de materiales).
NOTA: Toda la configuración experimental que involucra experimentos con células principales de L . debe realizarse en un gabinete de bioseguridad certificado BSL2. - Cultivar los promastigotes en M199 mediocompleto 22 hasta que alcancen la fase logarítmica (2-8 x 106 células/mL), según lo determinado por los ensayos de curva de crecimiento mayor de L . realizados previamente23. Planifique 1 x 10 5 células para cada pocillo para la citotoxicidad, más 5 x 105 células para el control de la tinción. Para Western blot, planifique 2 x 107 celdas por pocillo.
NOTA: Realice el ensayo de citotoxicidad con dos réplicas técnicas. - Para verificar la densidad celular adecuada, mezclar una alícuota (10-40 μL) de promastigotes con un volumen igual de fijador (3,7% de paraformaldehído en 1x PBS). Cargue 10 μL de la muestra fija en cada lado del hemacitómetro.
PRECAUCIÓN: El formaldehído es un producto químico tóxico. Manejar de acuerdo con las políticas institucionales para productos químicos peligrosos. - Realice el recuento de células con un microscopio con un aumento de 20x. Cuente todas las células en los 25 cuadrados pequeños en el centro del hemacitómetro. Repita para los cuadrados en ambos lados y promedie los recuentos.
NOTA: Si la variación entre recuentos es >10, recuento y promedio. Si el recuento promedio es <10 o >100, modifique la dilución y el recuento y, a continuación, calcule la densidad de cultivo utilizando la siguiente fórmula: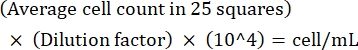 (Ec. 1)
(Ec. 1)
Por ejemplo, si hay en promedio 250 Leishmania en 25 cuadrados, la densidad de cultivo es de 5 x 106 células/ml. - Después de contar, transfiera 5 x 106 células a un tubo cónico de 15 ml y centrifugar a 1.500 x g durante 8 minutos a temperatura ambiente para granular las células.
- Deseche el sobrenadante con una pipeta de 10 ml y vortice brevemente el pellet celular. Agregue 5 ml de 1x PBS al mismo tubo y lave las células invirtiendo suavemente 3-6x. Centrifugar a 1.500 x g durante 8 min a temperatura ambiente para granular las células.
- Desechar el sobrenadante con una pipeta de 10 ml y resuspender el pellet de 5 x 106 células en 5 ml de medio (por ejemplo, M199 o 1x tampón de Tyrode) utilizado para los experimentos con una pipeta de 5 ml para obtener una concentración final de 1 x 106 células/ml.
2. Ensayo de citotoxicidad
- Preparación experimental
- Purificar la toxina como se describió anteriormente24, o comprar la toxina a un proveedor. Alícuota en alícuotas de un solo uso y conservar −80 °C durante un máximo de 1 año. Evite múltiples ciclos de congelación-descongelación.
- Determinar la actividad hemolítica de cada toxina utilizando glóbulos rojos humanos (ver Tabla de Materiales)24.
NOTA: La actividad hemolítica se utiliza porque controla las diferencias en la actividad debido a la purificación, mutaciones, etc. La elección de la especie de eritrocitos puede alterar la actividad hemolítica (por ejemplo, la intermedilisina requiere glóbulos rojos humanos). - Planifique dos réplicas técnicas para cada condición, siete diluciones para la curva dosis-respuesta y un control sin toxinas.
NOTA: Con los promastigotes de tipo salvaje (WT), spt2 y spt2-/+SPT2, se pueden probar dos tratamientos en una placa de 96 pocillos con fondo en V. Por ejemplo, se podría comparar la sensibilidad a los medios (Figura 1). En lugar de una placa inferior en V, se pueden usar tubos de microtitulación de 1,2 ml (consulte la Tabla de materiales). Debido a los tiempos de adquisición en el citómetro, no se recomienda ejecutar más de una placa a la vez. - Determine qué tampón de ensayo utilizar en función de las condiciones de prueba necesarias y el propósito del experimento.
NOTA: En este ejemplo, se comparan dos tampones de ensayo: M199 y tampón de Tyrode suplementado con el colorante de viabilidad yoduro de propidio (PI). El colesterol en suero interferirá con la actividad de los CDC24. - Calcular la cantidad de toxina necesaria en función de las condiciones y el número de genotipos tratados. Asegúrese de que se produzca una lisis específica del 50% a mitad de camino de la curva de dilución.
NOTA: Para los CDC, una dilución en serie doble dará un buen rango para el modelado logístico posterior. Para los promastigotes spt2- , 4.000 HU/mL SLO es la dilución inicial recomendada. Cuando se utilizan toxinas inactivas, se puede utilizar una masa equivalente a la dosis más alta en su lugar. - Planifique un volumen final de 200 μL por pocillo y agregue un pequeño volumen adicional (50-100 μL) para tener en cuenta el error de pipeta.
NOTA: Con tres genotipos, cada uno hecho por duplicado, habrá seis muestras para la dilución en serie. - Determine la cantidad total de toxina necesaria utilizando la siguiente fórmula:
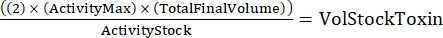 (Punto 2)
(Punto 2)
donde ActivityMax es la concentración más alta utilizada (HU/mL); TotalFinalVolume es el volumen total (para seis muestras, 200 x 6 + 100 = 1.300 μL); ActividadStock es la actividad de la reserva de toxinas (HU/mL); y VolStockToxin es el volumen de stock de toxinas necesario. - Prepare suficiente tampón de ensayo necesario para el experimento. Complemente el medio basal con un colorante de viabilidad y cualquier Ca 2+ o EGTA necesario para controlar los niveles de Ca2+. Vórtice para mezclar.
NOTA: Por ejemplo, por cada tampón de 10 ml de Tyrode, añadir 50 μL de 2 mg/ml PI y 200 μL de 100 mM CaCl 2, dando concentraciones finales de 10 μg/ml y2 mM, respectivamente. - Asegúrese de que el colorante de viabilidad PI no entre en conflicto con ninguna otra sonda utilizada, como para ensayos de unión fluorescente utilizando toxinas conjugadas Cy5 o AlexaFluor64720,25.
- Planifique diluciones de toxinas para hacer una solución 2x de toxina. Agregue tampón de ensayo a los tubos de centrífuga de 1,5 ml y enfríe con hielo. Solo agregue la toxina inmediatamente antes de comenzar el ensayo.
NOTA: En este ejemplo, se agregarían 1,3 ml de tampón de ensayo para la dilución superior, y se agregarían 650 μL para diluciones seriadas para preparar la solución de toxina 2x.
- Experimento
- Reserve 0,5 ml de promastigotes procesados del paso 1.8 en un tubo separado como "Control sin teñir".
NOTA: Esta muestra se utilizará para configurar la activación en el citómetro de flujo. - Añadir 2 mg/ml PI a una concentración final de 10 μg/ml a los promastigotes restantes. Vórtice durante 3 s.
NOTA: Después de la adición de IP a los promastigotes procesados, estas células solo se pueden usar durante un período de tiempo de 2.5 h. Después de 2.5 h, las células comienzan a morir y los resultados se vuelven erróneos. - Agregue 1 x 105 (en 100 μL/pocillo) promastigotes procesados a cada pocillo de una placa de 96 pocillos con fondo en V o tubos de microtitulación de 1,2 ml (Figura 1). Coloque la placa o la rejilla de tubos sobre hielo en un ángulo de aproximadamente 45° desde la visión. Realizar el trabajo en un armario de bioseguridad al manipular promastigotes de Leishmania .
- Añadir 100 μL de tampón de ensayo con IP a cada control sin toxinas (última fila). Verifique que el control se agregó correctamente identificando visualmente los tubos con un volumen total de 200 μL que aparecen de color más oscuro.
- Retire las alícuotas de toxinas de −80 ° C, descongele en hielo y acumule según sea necesario. Añadir el volumen de toxina calculado en el paso 2.1.5 a la dilución más alta (preparada en el paso 2.1.10). Luego, diluya en serie la toxina (Figura 1). Pipetear hacia arriba y hacia abajo al menos 8 veces para asegurar la mezcla.
NOTA: Realice sobre hielo porque los CDC se inactivan rápidamente a temperatura ambiente. - Comenzando desde la concentración más baja de toxina, agregue rápidamente 100 μL de toxina a la fila correcta (Figura 1 y Figura 2) y continúe hasta que toda la toxina se haya agregado a las células.
- Selle la placa con cinta de sellado. Incubar a 37 °C durante 30 min. Después del período de incubación, empaque y transporte la placa al citómetro de flujo.
- Reserve 0,5 ml de promastigotes procesados del paso 1.8 en un tubo separado como "Control sin teñir".
- Adquisición de datos
- Configure el citómetro de flujo (consulte la Tabla de materiales) y el software de adquisición de acuerdo con las instrucciones del fabricante y la política de la instalación. No realice el procedimiento de citometría de flujo sin capacitación previa en el citómetro.
NOTA: En este ejemplo, se utilizó un Attune NxT de 4 láseres. PI se recogió en el canal YL-1 (excitado por un láser de 561 nm, pasado a través de un LP 577, reflejado desde 600 DLP y filtrado a través de 585/16 bandpass), aunque el amplio espectro de PI permite la recolección en otros canales. - Usando la muestra de promastigotte de L. major sin teñir, configure las puertas para la dispersión hacia adelante y hacia los lados y los parámetros fluorescentes iniciales según los tintes elegidos.
- Incluya un parámetro adicional si lo desea para los diagramas de puntos para verificar la autofluorescencia (por ejemplo, "sin mancha") (Figura 3).
NOTA: En este ejemplo, se utilizó el canal BL-1 (excitado por un láser de 488 nm, pasado a través de 495 DLP y 503 LP, reflejado desde 555 DLP y filtrado a través de 530/30 bandpass). - Usando controles de tinción única, establezca las puertas para el tinte de viabilidad (PI en este estudio) y cualquier toxina marcada con fluorescencia. Monitoree la dispersión hacia adelante frente al tiempo para micro-obstrucciones.
NOTA: PI teñirá ligeramente todas las celdas por encima de los controles no teñidos. Las células muertas serán fácilmente separables, con células permeabilizadas transitoriamente entre poblaciones. - Adquiera >10,000 eventos cerrados para cada muestra en el citómetro.
NOTA: Se recomienda leer de más sensible a menos sensible, pero el orden de adquisición se puede invertir para determinar cualquier impacto del orden de lectura en los resultados de la muestra. - Guarde los datos y expórtelos según sea necesario para el análisis.
- Configure el citómetro de flujo (consulte la Tabla de materiales) y el software de adquisición de acuerdo con las instrucciones del fabricante y la política de la instalación. No realice el procedimiento de citometría de flujo sin capacitación previa en el citómetro.
- Análisis de datos
NOTA: En este estudio, Excel con el complemento Solver (consulte Tabla de materiales) se utilizó para el análisis de datos (véase Archivo complementario 1).- Promastigotes de L . major de una sola célula total mediante la dispersión hacia adelante y hacia los lados y el tiempo según sea necesario (Figura 3). Use la altura o el área según lo recomendado para el citómetro de flujo.
NOTA: Para el citómetro de flujo utilizado aquí, la altura es el parámetro recomendado en lugar del área. - Identifique y bloquee las celdas muertas como "PI alto". Puerta de celdas intermedias como "PI bajo". Las células PI altas son células muertas, mientras que las células bajas PI son permeabilizadas transitoriamente26.
NOTA: Las celdas altas de PI suelen mostrar un desplazamiento logarítmico de 2-3 de las celdas negativas. - Exporte los datos a Excel. Obtenga el nombre/ID de la muestra y %PI alto para determinar la muerte.
NOTA: Si se usaran toxinas fluorescentes, se necesitará la intensidad fluorescente mediana (MFI) de poblaciones vivas, transitoriamente permeabilizadas y negativas. %PI bajo puede exportarse para permeabilización transitoria. - Determine el % PI promedio alto para cada condición entre las dos réplicas técnicas.
NOTA: Si se necesita una IMF promedio para las toxinas fluorescentes, calcule esto también. - Calcule %Lisis específica a partir del %PI alto usando la siguiente fórmula24,25:
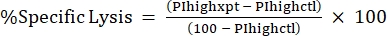 (Punto 3)
(Punto 3)
donde PIhighxpt es el % PI alto para la condición experimental; y PIhighctl es el % PI alto para el control sin toxinas. - Gráfico %Lisis específica contra la concentración de toxinas para la curva dosis-respuesta (Figura 4).
- Organice la curva dosis-respuesta en Excel para el modelado logístico. Incluya la concentración de toxinas y el %Lisis Específico promedio junto con detalles experimentales y/o cálculos altos de %PI brutos (Tabla 2).
- Compruebe que el complemento Solver está habilitado.
NOTA: Para habilitar el Solver en la versión de escritorio de Excel, vaya a Opciones de > de archivos > complementos y active la casilla Solver . Reinicie Excel. - Etiquete cuatro columnas más como "modelado", "residuos", "parámetros" y "valores de parámetros". Verifique que las primeras columnas correspondan a los parámetros experimentales, la concentración de toxinas y la %Lisis específica (Tabla 2).
- Agregue los siguientes parámetros en la columna "parámetros": L, k, c, SUM y LC50. Inicialice los parámetros L, k y c introduciendo los siguientes valores en la columna "valores de parámetros": 100, 0,05, 1.000.
- En la columna "modelado", cree el modelo logístico mediante la siguiente fórmula:
 (Punto 4)
(Punto 4)
Establezca L, k y c en las celdas que contienen esos parámetros en la columna "valores de parámetros".
Establezca x en la celda que contiene la concentración de toxina.
NOTA: Para la Tabla 2, celda G4, la fórmula es la siguiente: =$J$3/(1+EXP(-$J$4*(D4-$J$5))) - Aplique esta ecuación a todos los valores de %Lisis específica excepto el control sin toxinas.
- En la columna "residuos", calcule el cuadrado de la diferencia entre el número modelado y la lisis específica real usando la siguiente ecuación:
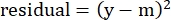 (Ec. 5)
(Ec. 5)
donde y es la %lisis específica experimental; y m es el valor correspondiente en la columna "modelado" calculado en los pasos 2.4.10-2.4.11. - En la columna "valores de parámetros" junto a "SUMA", suma todos los valores de la columna de residuos.
- En la columna "valores de parámetros" junto a "LC50", inicialice la ecuación para calcular la LC50 a partir de los valores determinados. Esto es resolver Eq 4 para x cuando m = 50.
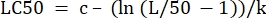 (Ec. 6)
(Ec. 6)
Para la Tabla 2, la fórmula de Excel es la siguiente: =J5-(LN(J3/(50)-1))/J4 - Abra el Solver desde la pestaña Datos. Seleccione Definir objetivo para que sea la celda que contiene la suma de los residuos calculados. Configúrelo en Min.
- Cambie las celdas variables para los valores de parámetro de L, k y c.
NOTA: El solucionador puede comportarse mejor si se deja marcada la opción "Hacer variables no negativas". - Para valores k negativos, modifique Eq 4 y Eq 6 factorizando −1 de k para cambiar k a positivo. Utilice el método de resolución no lineal GRG. Haga clic en Resolver.
- Compruebe la curva y que el LC50 se calcule automáticamente utilizando la Ecu 6 (Tabla 2). Verifique el ajuste trazando gráficamente tanto %Lisis específica como modelando contra la concentración de toxinas.
NOTA: También se puede comprobar calculando la R2 para la curva.
- Promastigotes de L . major de una sola célula total mediante la dispersión hacia adelante y hacia los lados y el tiempo según sea necesario (Figura 3). Use la altura o el área según lo recomendado para el citómetro de flujo.
3. Análisis proteico de promastigotes de L. major desafiados por toxinas
- Preparar los promastigotes de L. major como se describe en la sección 1.
- Resuspender 2 x 107 WT, spt2-, y spt2-/+SPT2 L. promastigotes mayores en 2 ml del tampón de ensayo deseado utilizando una pipeta de 5 ml (p. ej., M199 sin suero). Añadir la toxina a una concentración sublítica final e incubar a 37 °C durante 30 min.
NOTA: Por ejemplo, una dosis sublítica de SLO para promastigotes spt2-es de 500 HU/mL. - Incluir otros genotipos y controles sin toxinas.
- Centrifugar los promastigotes a 1.500 x g durante 10 min para granular las células. Deseche el sobrenadante con una pipeta de 10 ml. Pase el tubo cerrado que contiene el pellet celular rápidamente tres veces a través de una superficie irregular, como la parrilla del gabinete de bioseguridad, para romper el pellet celular.
NOTA: La bolita celular es casi invisible a simple vista. - Reconstituir 1x tampón de muestra SDS-PAGE con 2-mercaptoetanol inmediatamente antes de su uso y calentar a 95 °C durante 10 min antes de añadirlo al pellet celular. Resuspenda el pellet de la celda en un tampón de muestra SDS-PAGE caliente 1x y mezcle bien pipeteando hacia arriba y hacia abajo. Calentar el pellet de celda resuspendido en 1x tampón de muestra a 95 °C durante 10 min.
NOTA: Después de la solubilización en tampón de muestra, almacenar las muestras a largo plazo a -20 °C si es necesario. - Prepare el gel resolutivo. Desgasificar todos los componentes excepto el persulfato de amonio (APS) y TEMED durante 15 min.
- Agregue APS y TEMED inmediatamente antes de fundir el gel. Superponga cuidadosamente el gel de resolución con agua. Deje que el gel de resolución polimerice, ~ 30-45 min.
- Decantar el agua y preparar el gel de apilamiento. Añadir el gel de apilamiento, teniendo cuidado de evitar burbujas. Inserte un peine con el número relevante de pocillos y deje polimerizar durante 5 min. Monitoree la polimerización usando cualquier gel de apilamiento sobrante.
- Ensamble el gel para ejecutar SDS-PAGE y agregue un tampón de depósito a la cámara.
- Cargar 10 μL de cada muestra por pocillo, u 8 μL de la escalera de proteínas. Ejecute a 180 V hasta que las muestras entren en el gel de resolución, y luego reduzca el voltaje a ~ 160 V y ejecute hasta que el frente del tinte esté a ~0.5 cm del borde de la placa.
NOTA: El tiempo que tardan las muestras en llegar al fondo puede variar entre 1-1,5 h. El tiempo puede alargarse reduciendo el voltaje. Nunca reduzca el voltaje a cero. Los voltajes más altos pueden aumentar la "sonrisa" del gel y agrietar las placas. - Transfiera el gel a la tinción de Coomassie (para la tinción de proteínas) o a un tampón de transferencia 1x (para Western blotting). Para la tinción de Coomassie, manche durante la noche y luego demiñe, imagen y seque.
- Para Western blot, prepare el sistema de transferencia de acuerdo con las instrucciones del fabricante.
- Para una transferencia húmeda, use tampón de transferencia 1x en frío y almohadillas prehúmedas, papel de filtro y nitrocelulosa. Para el sistema Bio-rad Protean III (consulte la Tabla de materiales) utilizado aquí, el papel de filtro se puede cortar a 10 cm x 7,5 cm. Cortar la nitrocelulosa a 9 cm x 6,75 cm.
NOTA: La nitrocelulosa es altamente inflamable. Evite las llamas abiertas y otras fuentes potenciales de ignición. - Coloque el casete de transferencia con almohadillas, papel de filtro y nitrocelulosa. Despliega las burbujas de aire. Agregue el gel con cuidado.
- Agregue el papel de filtro y extienda las burbujas de aire. Agregue la almohadilla, cierre el cassette e insértela en el soporte en la orientación correcta (asegúrese de que la nitrocelulosa mire hacia el terminal rojo). Agregue una barra de agitación y una bolsa de hielo a un lado, y complete el depósito con un tampón de transferencia frío 1x. Transfiera a 110 V durante 90 min.
NOTA: El calor generado durante la transferencia puede afectar negativamente a la transferencia. Para garantizar una buena transferencia, utilice siempre un búfer de transferencia 1x en frío. - Retire la nitrocelulosa y manche con la solución de Ponceau durante ~ 5 min. Enjuagar con agua ultrapura. Marque la escalera de proteínas a lápiz y recorte la mancha según sea necesario. Destain la mancha usando el búfer de transferencia sobrante.
NOTA: Ponceau puede ser reutilizado muchas veces. - Bloquear la nitrocelulosa en 25 ml de BSA al 5% en 1x TBST a 4 °C, con agitación, durante la noche. Luego, deseche la solución de bloqueo y agregue el anticuerpo primario (1: 1,000) en BSA al 1% en 1x TBST. Agitar a 4 °C durante la noche.
NOTA: El anticuerpo primario puede guardarse a -20 °C y reutilizarse varias veces. - Lave la nitrocelulosa 3 veces durante 10 minutos cada una en 1x TBST con agitación. Deseche el lavado y agregue 10 ml de anticuerpo secundario conjugado con HRP (1:10.000) en BSA al 1% en 1x TBST. Agitar a temperatura ambiente durante 1 h. Lave la nitrocelulosa 3 veces durante 10 minutos cada una en 1x TBST con agitación.
- Prepare el reactivo ECL inmediatamente antes de obtener imágenes de la nitrocelulosa. Inmediatamente antes de la imagen, decantar el TBST y añadir el reactivo ECL a la nitrocelulosa. Agitar durante 1 min. Imagen del gel.
Access restricted. Please log in or start a trial to view this content.
Resultados
Aumento de la sensibilidad del promastigote a SLO en el tampón de Tyrode en comparación con M199
La sensibilidad SLO de los promastigotes de L. major se comparó entre diferentes tampones de ensayo. Los promastigotes de tipo salvaje, spt2- y spt2-/+SPT2 se desafiaron con SLO en M199 sin suero o tampón de Tyrode suplementado con 2 mM CaCl2 durante 30 min antes del análisis en un citómetro de flujo. Los parásitos adecuados para el anális...
Access restricted. Please log in or start a trial to view this content.
Discusión
En este estudio, se describieron métodos para estudiar los mecanismos moleculares y las funciones de las PFT, utilizando el patógeno humano Leishmania major como sistema modelo. Se desarrolló un ensayo de citotoxicidad basado en citometría de flujo de rendimiento medio para medir la viabilidad de una sola célula. La viabilidad es cuantitativa a nivel poblacional porque los valores de CL50 se pueden calcular a partir de la curva dosis-respuesta utilizando modelos logísticos. Como prueba de princi...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Este trabajo fue apoyado por el Instituto Nacional de Alergias y Enfermedades Infecciosas de los Institutos Nacionales de Salud subvención R21AI156225 a PAK y KZ (co-I) y R01AI139198 a KZ (co-I). CH desea agradecer al Departamento de Ciencias Biológicas por la Ayudantía de Enseñanza proporcionada durante el tiempo de este estudio.
Los organismos de financiación no participaron en el diseño del estudio; en la recopilación, análisis o interpretación de datos; en la redacción del manuscrito; ni en la decisión de publicar los resultados. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente las opiniones oficiales de las agencias de financiación. Los autores declaran que no tienen conflictos de intereses en competencia.
Agradecimientos
Los autores desean agradecer a los miembros de los laboratorios Keyel y Zhang por su revisión crítica del manuscrito. Los autores agradecen a la Facultad de Artes y Ciencias Microscopía por el uso de las instalaciones.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.2 mL microtiter (Marsh) tubes | Fisher | 02-681-376 | Cytotoxicity assay |
| 1.5 mL microcentrifuge tube | Fisher | 05-408-129 | Toxin dilutions |
| 15 mL centrifuge tube | Avantor VWR (Radnor, PA) | 89039-666 | To hold cells and media |
| 1x Phosphate buffered saline (PBS) | Fisher | BP399 | For cell processing |
| 3% H2O2 | Walmart (Fayetteville, AR) | N/A | For ECL |
| 5x M199 | Cell-gro | 11150067 | Basal growth media for L. major promastigotes |
| Biosafety cabinet | Baker | To culture cells in sterile conditions | |
| Bovine serum albumin (BSA) | Fisher | BP1605-100 | Fraction V acceptable purity |
| CaCl2 | Fisher | BP510-100 | Stock concentration 100 mM |
| Centrifuge | Thermo Fisher | Heraeus Megafuge 40R | To pellet the cells from culture |
| Cy5 Mono-reactive dye pack | Cytiva (Marlborough, MA) | PA25031 | Fluorophore label for toxins |
| Digital dry bath | Benchmark | BSH1002 | To denature protein samples |
| EGTA | Amresco | 0732-100G | Stock concentration 0.5 M |
| Excel | Microsoft (Redmond, VA) | Data analysis software | |
| Flow cytometer (4-laser Attune NxT) | Fisher | Cytometer for data acquisition | |
| FlowJo | BD (Ashland, OR) | Software | |
| Formaldehyde | Fisher | BP531-500 | Fixative for counting cells |
| G418 | Fisher | BP673-1 | Selection agent for cells |
| Hellmanex III | Sigma | Z805939 | Dilute 1:4 for cleaning cytometer |
| Hemacytometer | Fisher | 0267151B | For counting cells |
| Human red blood cells | Zen-bio (Durham, NC) | SER-10MLRBC | To validate toxin activity |
| Ice bucket | |||
| Light microscope | Nikon | Eclipse 55i | To visualize cells |
| Nitrocellulose | Fisher | 88018 | For probing proteins via antibodies |
| Pipettors and tips | Avantor VWR | To dispense reagents | |
| Power supply | Bio-Rad | To run SDS-PAGE and transfers | |
| Propidium iodide | Biotium | 40016 | Stock concentration 2 mg/mL in water |
| Protein ladder | Bio-Rad | 161-0373 | To determine molecular weight of proteins |
| SDS-PAGE Running Apparatus (Mini Protean III) | Bio-Rad | 165-3302 | To separate proteins based on their size |
| Sealing tape | R&D | DY992 | To seal plates with cells |
| Streptolysin O C530A plasmid insert | Cloned into pBAD-gIII vector (Reference: 7) | ||
| Streptolysin O C530A toxin | Lab purified | Specific activity 4.34 x 105 HU/mg | |
| Swinging bucket rotor | Thermo Fisher | 75003607 | To centrifuge cells |
| V-bottom plate | Greiner Bio-one | 651206 | For cytotoxicity assay |
| Vortex | Benchmark | BV1000 | To mix cells |
| Western blot imaging system (Chemi-doc) | Bio-Rad | To visualize proteins by western blot | |
| Western Blot Transfer Apparatus (Mini Protean III) | Bio-Rad | 170-3930 | Transfer proteins to nitrocellulose |
| Whatman Filter paper | GE Healthcare Life Sciences | 3030-700 | Used in transfer of proteins to nitrocellulose |
| Antibody | |||
| Anti-ERK antibody | Cell Signaling Technologies | Cat# 9102S | Rabbit (1:1000 dilution) |
| Anti-lipophosphoglycan (LPG) antibody | CreativeBioLabs | Cat# WIC79.3 | Mouse (1: 1000) |
| Anti-MEK antibody | Cell Signaling Technologies | Cat# 9122L | Rabbit (1:1000) |
| Anti-mouse IgG, HRP conjugate | Jackson Immunoresearch | Cat#715-035-151 | Donkey (1:10000) |
| Anti-phosphoERK antibody | Cell Signaling Technologies | Cat# 9101S | Rabbit (1:1000) |
| Anti-pMEK antibody | Cell Signaling Technologies | Cat# 9121S | Rabbit (1:1000) |
| Anti-rabbit IgG, HRP conjugate | Jackson Immunoresearch | Cat#711-035-152 | Donkey (1:10000) |
| Anti-tubulin antibody | Sigma | Cat# T5168 | Mouse (1: 2000) |
| Leishmania major Genotypes | Reference: 13 | ||
| Episomal addback (spt2-/+SPT2) | Δspt2::HYG/Δspt2:PAC/+pXG-SPT2 | ||
| Serine palmitoyltransferase subunit 2 knockout (spt2-) | Δspt2::HYG/Δspt2::PAC | ||
| Wild type (WT) | LV39 clone 5 (Rho/SU/59/P) |
Referencias
- Thapa, R., Ray, S., Keyel, P. A. Interaction of macrophages and cholesterol-dependent cytolysins: The impact on immune response and cellular survival. Toxins. 12 (9), 531(2020).
- Limbago, B., Penumalli, V., Weinrick, B., Scott, J. R. Role of streptolysin O in a mouse model of invasive group A streptococcal disease. Infection & Immunity. 68 (11), 6384-6390 (2000).
- Farrand, A. J., et al. The cholesterol-dependent cytolysin membrane-binding interface discriminates lipid environments of cholesterol to support beta-barrel pore insertion. Journal of Biological Chemistry. 290 (29), 17733-17744 (2015).
- Soltani, C. E., Hotze, E. M., Johnson, A. E., Tweten, R. K. Structural elements of the cholesterol-dependent cytolysins that are responsible for their cholesterol-sensitive membrane interactions. Proceedings of the National Academy of Sciences of the United States of America. 104 (51), 20226-20231 (2007).
- Schoenauer, R., et al. Down-regulation of acid sphingomyelinase and neutral sphingomyelinase-2 inversely determines the cellular resistance to plasmalemmal injury by pore-forming toxins. FASEB Journal. 33 (1), 275-285 (2019).
- Ray, S., Roth, R., Keyel, P. A. Membrane repair triggered by cholesterol-dependent cytolysins is activated by mixed lineage kinases and MEK. Science Advances. 8 (11), (2022).
- Babiychuk, E. B., Monastyrskaya, K., Draeger, A. Fluorescent annexin A1 reveals dynamics of ceramide platforms in living cells. Traffic. 9 (10), 1757-1775 (2008).
- Jimenez, A. J., et al. ESCRT machinery is required for plasma membrane repair. Science. 343 (6174), 1247136(2014).
- Demonbreun, A. R., et al. An actin-dependent annexin complex mediates plasma membrane repair in muscle. Journal of Cell Biology. 213 (6), 705-718 (2016).
- Wolfmeier, H., et al. Ca(2)(+)-dependent repair of pneumolysin pores: A new paradigm for host cellular defense against bacterial pore-forming toxins. Biochimica et Biophysica Acta. 1853 (2), 2045-2054 (2015).
- Bravo, F., Sanchez, M. R. New and re-emerging cutaneous infectious diseases in Latin America and other geographic areas. Dermatologic Clinics. 21 (4), 655-668 (2003).
- Manfredi, M., Iuliano, S. Cutaneous leishmaniasis with long duration and bleeding ulcer. Clinical Microbiology Open Access. 05, 2-6 (2016).
- Zhang, K., et al. Sphingolipids are essential for differentiation but not growth in Leishmania. EMBO Journal. 22 (22), 6016-6026 (2003).
- Zhang, K. Balancing de novo synthesis and salvage of lipids by Leishmania amastigotes. Current Opinions in Microbiology. 63, 98-103 (2021).
- Kaur, P., Goyal, N. Pathogenic role of mitogen activated protein kinases in protozoan parasites. Biochimie. 193, 78-89 (2022).
- Wiese, M. Leishmania MAP kinases--Familiar proteins in an unusual context. International Journal of Parasitology. 37 (10), 1053-1062 (2007).
- Brumlik, M. J., Pandeswara, S., Ludwig, S. M., Murthy, K., Curiel, T. J. Parasite mitogen-activated protein kinases as drug discovery targets to treat human protozoan pathogens. Journal of Signal Transduction. 2011, 971968(2011).
- Wiese, M., Kuhn, D., Grunfelder, C. G. Protein kinase involved in flagellar-length control. Eukaryotic Cell. 2 (4), 769-777 (2003).
- Agron, P. G., Reed, S. L., Engel, J. N. An essential, putative MEK kinase of Leishmania major. Molecular Biochemistry of Parasitology. 142 (1), 121-125 (2005).
- Ray, S., Thapa, R., Keyel, P. A. Multiple parameters beyond lipid binding affinity drive cytotoxicity of cholesterol-dependent cytolysins. Toxins. 11 (1), (2018).
- Beneke, T., et al. A CRISPR Cas9 high-throughput genome editing toolkit for kinetoplastids. Royal Society Open Science. 4 (5), 170095(2017).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular and Cellular Biology. 10 (3), 1084-1094 (1990).
- Moitra, S., Pawlowic, M. C., Hsu, F. F., Zhang, K. Phosphatidylcholine synthesis through cholinephosphate cytidylyltransferase is dispensable in Leishmania major. Scientific Reports. 9, 7602(2019).
- Keyel, P. A., Heid, M. E., Watkins, S. C., Salter, R. D. Visualization of bacterial toxin induced responses using live cell fluorescence microscopy. Journal of Visualized Experiments. (68), 4227(2012).
- Romero, M., et al. Intrinsic repair protects cells from pore-forming toxins by microvesicle shedding. Cell Death & Differentiation. 24 (5), 798-808 (2017).
- Keyel, P. A., et al. Streptolysin O clearance through sequestration into blebs that bud passively from the plasma membrane. Journal of Cell Science. 124, 2414-2423 (2011).
- Dong, Z., Patel, Y., Saikumar, P., Weinberg, J. M., Venkatachalam, M. A. Development of porous defects in plasma membranes of adenosine triphosphate-depleted Madin-Darby canine kidney cells and its inhibition by glycine. Laboratory Investigations. 78 (6), 657-668 (1998).
- Loomis, W. P., den Hartigh, A. B., Cookson, B. T., Fink, S. L. Diverse small molecules prevent macrophage lysis during pyroptosis. Cell Death & Disease. 10 (4), 326(2019).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados