È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Decifrare il meccanismo molecolare e la funzione delle tossine che formano i pori usando la Leishmania major
In questo articolo
Riepilogo
Presentato qui è un protocollo che utilizza Leishmania major promastigotes per determinare il legame, la citotossicità e la segnalazione indotta dalle tossine che formano i pori. Viene fornita una prova di concetto con streptolisina O. Altre tossine possono anche essere utilizzate per sfruttare i mutanti genetici disponibili in L. major per definire nuovi meccanismi di resistenza alle tossine.
Abstract
Comprendere la funzione e il meccanismo delle tossine che formano i pori (PFT) è difficile perché le cellule resistono al danno di membrana causato dai PFT. Mentre gli approcci biofisici aiutano a comprendere la formazione dei pori, spesso si basano su approcci riduzionisti privi del pieno complemento di lipidi e proteine di membrana. Le cellule umane coltivate forniscono un sistema alternativo, ma la loro complessità e ridondanza nei meccanismi di riparazione rendono difficile l'identificazione di meccanismi specifici. Al contrario, il patogeno protozoo umano responsabile della leishmaniosi cutanea, Leishmania major, offre un equilibrio ottimale tra complessità e rilevanza fisiologica. L. major è geneticamente trattabile e può essere coltivato ad alta densità in vitro, e qualsiasi impatto delle perturbazioni sull'infezione può essere misurato in modelli murini stabiliti. Inoltre, L. major sintetizza lipidi distinti dalle loro controparti dei mammiferi, che potrebbero alterare la dinamica della membrana. Queste alterazioni nella dinamica della membrana possono essere sondate con PFT della famiglia di tossine meglio caratterizzata, le citolisine colesterolo-dipendenti (CDC). I CDC si legano all'ergosterolo nella membrana della Leishmania e possono uccidere L. major promastigotes, indicando che L. major è un sistema modello adatto per determinare i meccanismi cellulari e molecolari della funzione PFT . Questo lavoro descrive i metodi per testare la funzione PFT in L. major promastigotes, tra cui coltura parassitaria, strumenti genetici per valutare la suscettibilità ai lipidi, saggi di legame della membrana e saggi di morte cellulare. Questi saggi consentiranno l'uso rapido di L. major come potente sistema modello per comprendere la funzione PFT in una serie di organismi evolutivamente diversi e punti in comune nell'organizzazione dei lipidi.
Introduzione
Le tossine che formano i pori (PFT) sono la più grande famiglia di tossine batteriche1, ma i meccanismi con cui perforano e distruggono le cellule sono poco conosciuti. La famiglia meglio studiata di tossine che formano i pori è quella delle citolisine colesterolo-dipendenti (CDC). I CDC sono sintetizzati principalmente da batteri gram-positivi, incluso l'agente eziologico della fascite necrotizzante, Streptococcus pyogenes2. S. pyogenes secerne la streptolisina O CDC (SLO), che si lega agli steroli nella membrana plasmatica delle cellule ospiti come monomeri, oligomerizza e inserisce ~ 20-30 nm pori nella membrana1. Il ruolo che i lipidi svolgono in questo processo rimane scarsamente determinato.
Un approccio allo studio delle interazioni lipidi-CDC è l'uso di liposomi chimicamente definiti. Mentre i liposomi definiti forniscono informazioni sulle soglie necessarie dei lipidi per sostenere il legame delle tossine e la formazione dei pori3,4, non ricapitolano completamente le funzioni cellulari. Ad esempio, i liposomi ricostituiti mancano dell'asimmetria lipidica degli ospiti dei mammiferi e delle modificazioni lipidiche in risposta alle tossine5. Un'alternativa ai liposomi è quella di utilizzare linee cellulari di mammifero. Mentre queste linee cellulari sono fisiologicamente più rilevanti, c'è un ampio grado di ridondanza nei meccanismi di rilevamento e resistenza delle tossine2. Di conseguenza, i percorsi di riparazione utilizzati per resistere ai CDC rimangono scarsamente determinati. In particolare, l'afflusso di Ca2+ è l'attivatore primario della riparazione della membrana1. A valle dell'afflusso di Ca2+, sono impegnati più percorsi, tra cui un 6,7 di riparazione dipendente dalla ceramide e unpercorso di riparazione 6 dipendente da MEK. Queste vie interagiscono con altri effettori proteici, incluso il complesso di selezione endosomiale richiesto per il trasporto (ESCRT)8 e le annessine 6,9,10. Sezionare questi percorsi nelle cellule di mammifero è difficile a causa della ridondanza, che confonde l'interpretazione dei dati.
Un modo per bilanciare la complessità con la semplicità per sezionare i percorsi di riparazione è l'uso di organismi più semplici, come i patogeni protozoari del genere Leishmania. Leishmania sp. causa leishmaniosi negli esseri umani e in altri animali. La leishmaniosi varia dalla leishmaniosi cutanea (lesioni cutanee autolimitate) alla leishmaniosi viscerale fatale (epatosplenomegalia), a seconda della specie e di altri fattori11. La Leishmania major, l'agente eziologico della leishmaniosi cutanea, viene trasmessa agli esseri umani attraverso un vettore di sandfly e viene utilizzata per comprendere la funzione e l'infezione della Leishmania 12. Inoltre, Leishmania sp. sono digenici12. Esistono come parassiti macrofagi intracellulari dei mammiferi chiamati amastigoti e come promastigoti flagellati che nuotano liberamente nel sandfly12. L. i promastigoti maggiori possono essere coltivati in terreni integrati con siero come M199 ad alta densità13. I promastigoti sono anche geneticamente trattabili; Esistono molti knockout genici, compresi quelli che prendono di mira le vie di biosintesi lipidica13. Questi knockout possono essere valutati per la crescita e le differenze nell'infettività e nello sviluppo delle lesioni attraverso l'infezione di topi Balb / c13.
Oltre alla relativa facilità della coltura di Leishmania e alla gamma di knockout di biosintesi lipidica, il parassita ha un genoma più semplice rispetto ai mammiferi. La specie meglio caratterizzata di Leishmania è L. major, che ha molti strumenti genetici esistenti, come mutanti con metabolismo lipidico difettoso14. In particolare, molte proteine di riparazione sono assenti. L. major non ha omologhi identificati fino ad oggi per le proteine chiave di riparazione dei mammiferi come le annessine. Ciò consente la caratterizzazione di percorsi di riparazione evolutivamente conservati senza la complessità dei sistemi dei mammiferi. Tuttavia, i percorsi di riparazione non sono stati caratterizzati in Leishmania fino ad oggi. Allo stesso tempo, le vie di segnalazione chiave coinvolte nella riparazione, come la via MEK6, sono conservate in Leishmania sp.15,16, anche se gli omologhi devono essere convalidati. La via della proteina chinasi attivata da mitogeni (MAPK) è ben studiata in L. mexicana, dove contribuisce alla sopravvivenza intracellulare e alla termostabilità nelle cellule di mammifero e controlla la metaciclogenesi16. In Leishmania sp., 10 dei 15 MAPK sono stati caratterizzati17. Si prevede che LmMAPK9 e LmMAPK13 siano i più simili a ERK1/2 dei mammiferi in base all'identità nella sequenza labiale di fosforilazione conservata. La sequenza labiale di fosforilazione è TEY sia per ERK1/2 che per LmMAPK9 e LmMAPK13. Tuttavia, otto dei MAPK di Leishmania hanno un motivo di fosforilazione TDY15. Almeno due omologhi di MEK sono stati identificati in Leishmania sp., LmxMKK18 e MEKK-related kinase (MRK1)19. Ciò suggerisce che le intuizioni identificate in Leishmania potrebbero tradursi in sistemi di mammiferi. Dove non si traducono in sistemi di mammiferi, rappresentano bersagli terapeutici per il trattamento della leishmaniosi.
Per utilizzare L. major promastigotes per studiare la riparazione della membrana e le interazioni con le tossine, sono necessarie tecniche di media produttività. Mentre l'imaging di cellule vive ad alta risoluzione consente la visualizzazione di proteine e membrane marcate in tempo reale, è a basso rendimento e potrebbe non misurare la sopravvivenza cellulare. I saggi di vitalità a medio rendimento includono l'assorbimento del colorante misurato mediante citometria a flusso, la misurazione dell'attività mitocondriale o il rilascio di proteine cellulari come la lattato deidrogenasi (LDH). Nelle cellule di mammifero, i saggi LDH non misurano quantitativamente la morte cellulare20. Inoltre, i saggi basati sulla popolazione come il rilascio di LDH o l'attività mitocondriale non consentono una solida analisi monocellulare o multiparametrica20. Al contrario, i saggi basati sulla citometria a flusso consentono l'analisi multiparametrica di singole cellule20. Tuttavia, questi test non sono stati applicati alla comprensione della biologia delle tossine o delle risposte alle tossine in L. major promastigotes.
In questo studio, SLO è usato come strumento per comprendere la perturbazione della membrana plasmatica del mutante nullo sfingolipide di L. major in due diversi tamponi: il mezzo M199 abitualmente utilizzato per la coltura di L. major promastigotes e il più semplice tampone di Tyrode . Viene descritto un test di citometria a flusso a medio rendimento che viene utilizzato per generare curve dose-risposta delle tossine. I dati del saggio citometrico a flusso sono modellati su una curva logistica per determinare i valori di LC50 . Con queste informazioni, è possibile determinare una dose sublitica di SLO in modo che gli anticorpi MAPK possano essere convalidati utilizzando il western blotting.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Sono state impiegate tutte le linee guida appropriate e le pratiche standard microbiologiche, di sicurezza e di coltura cellulare per l'uso e la gestione del DNA principale e ricombinante del patogeno RG2 Leishmania . Tutti gli esperimenti con L. major vivo sono stati eseguiti in un armadio di biosicurezza in un laboratorio certificato BSL-2. Il lavoro è stato supervisionato dal Comitato istituzionale per la biosicurezza della Texas Tech University.
NOTA: Dal punto di vista della sicurezza, i proplastigoti vivi di L. major sono patogeni del gruppo di rischio 2. Gestire utilizzando il contenimento appropriato, le precauzioni e la supervisione del Comitato istituzionale per la biosicurezza (IBC). Maneggiare sostanze tossiche e sostanze chimiche in conformità con le procedure istituzionali per le sostanze tossiche. Se vengono utilizzate tossine ricombinanti, l'approvazione e la supervisione dell'IBC possono essere necessarie per il lavoro sul DNA ricombinante.
1. Coltivazione e preparazione di L. major promastigotes
- Ottenere, o creare e convalidare, L. mutanti genetici maggiori come precedentemente descritto utilizzando metodi di ricombinazione omologa o basati su CRISPR13,21. Utilizzare knockout integrati con il gene aggiunto su un plasmide per garantire la specificità del knockout.
- Coltura selvatica tipo L. major e spt2- promastigotes a 27 °C in mezzo M199 completo. Coltura delle cellule episomiali addback (spt2-/+SPT2) in M199 completo più 10 μg/mL G418 (vedere Tabella 1 e Tabella dei materiali).
NOTA: L'intera configurazione sperimentale che prevede esperimenti con cellule L. major deve essere eseguita in un armadio di biosicurezza certificato BSL2. - Coltivare i promastigoti in M199 completo mezzo22 fino a quando non raggiungono la fase logaritmica (2-8 x 106 cellule / ml), come determinato dai saggi della curva di crescita maggiore L. fatti in precedenza23. Pianificare 1 x 10 5 cellule per ogni pozzetto per citotossicità, più 5 x 105 cellule per il controllo della colorazione. Per la macchia occidentale, pianificare 2 x 107 celle per pozzetto.
NOTA: Eseguire il test di citotossicità con due repliche tecniche. - Per verificare la corretta densità cellulare, mescolare un'aliquota (10-40 μL) di promastigoti con un uguale volume di fissativo (3,7% paraformaldeide in 1x PBS). Caricare 10 μL del campione fisso su ciascun lato dell'ematocitometro.
ATTENZIONE: La formaldeide è una sostanza chimica tossica. Gestire in conformità con le politiche istituzionali per le sostanze chimiche pericolose. - Eseguire il conteggio delle cellule utilizzando un microscopio con ingrandimento 20x. Conta tutte le cellule nei 25 quadratini al centro dell'ematocitometro. Ripeti per i quadrati su entrambi i lati e fai la media dei conteggi.
NOTA: se la variazione tra i conteggi è >10, riconteggio e media. Se il conteggio medio è <10 o >100, modificare la diluizione e ricontare, quindi calcolare la densità di coltura utilizzando la seguente formula: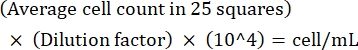 (Eq 1)
(Eq 1)
Ad esempio, se ci sono in media 250 Leishmania in 25 quadrati, la densità di coltura è 5 x 106 cellule / ml. - Dopo il conteggio, trasferire 5 x 106 celle in un tubo conico da 15 ml e centrifugare a 1.500 x g per 8 minuti a temperatura ambiente per pellettare le celle.
- Eliminare il surnatante utilizzando una pipetta da 10 ml e far vortice brevemente il pellet cellulare. Aggiungere 5 ml di 1x PBS allo stesso tubo e lavare le celle invertendo delicatamente 3-6x. Centrifugare a 1.500 x g per 8 minuti a temperatura ambiente per pellettare le celle.
- Eliminare il surnatante utilizzando una pipetta da 10 mL e risospendere il pellet 5 x 10a 6 cellule in 5 mL di terreno (ad esempio, M199 o 1x tampone di Tyrode) utilizzato per gli esperimenti con una pipetta da 5 mL per ottenere una concentrazione finale di 1 x 106 cellule/ml.
2. Saggio di citotossicità
- Preparazione sperimentale
- Purificare la tossina come descritto in precedenza24 o acquistare la tossina da un fornitore. Aliquote monouso e conservare -80 °C per un massimo di 1 anno. Evitare cicli multipli di congelamento-scongelamento.
- Determinare l'attività emolitica per ciascuna tossina utilizzando globuli rossi umani (vedere Tabella dei materiali)24.
NOTA: L'attività emolitica viene utilizzata perché controlla le differenze di attività dovute a purificazione, mutazioni, ecc. La scelta degli eritrociti da parte della specie può alterare l'attività emolitica (ad esempio, l'intermedilisina richiede globuli rossi umani). - Pianificare due repliche tecniche per ogni condizione, sette diluizioni per la curva dose-risposta e un controllo senza tossine.
NOTA: Con promastigoti wild-type (WT), spt2- e spt2-/+SPT2, è possibile testare due trattamenti in una piastra a 96 pozzetti con fondo a V. Ad esempio, è possibile confrontare la sensibilità ai supporti (Figura 1). Invece di una piastra inferiore a V, possono essere utilizzati tubi da microtitolazione da 1,2 mL (vedi Tabella dei materiali). A causa dei tempi di acquisizione sul citometro, non è consigliabile eseguire più di una piastra alla volta. - Determinare quale buffer di analisi utilizzare in base alle condizioni di test necessarie e allo scopo dell'esperimento.
NOTA: In questo esempio, vengono confrontati due tamponi di analisi: M199 e tampone di Tyrode integrato con lo ioduro di propidio (PI) del colorante di vitalità. Il colesterolo nel siero interferirà con l'attività CDC24. - Calcolare la quantità di tossina necessaria in base alle condizioni e al numero di genotipi trattati. Assicurarsi che il 50% di lisi specifica avvenga a metà della curva di diluizione.
NOTA: Per i CDC, una doppia diluizione seriale fornirà una buona gamma per la successiva modellazione logistica. Per i promastigoti spt2, 4.000 HU/mL SLO è la diluizione iniziale raccomandata. In caso di utilizzo di tossine inattive, può essere utilizzata una massa equivalente alla dose più elevata. - Pianificare un volume finale di 200 μL per pozzetto e aggiungere un piccolo volume aggiuntivo (50-100 μL) per tenere conto dell'errore della pipetta.
NOTA: Con tre genotipi, ciascuno fatto in duplicato, ci saranno sei campioni per la diluizione seriale. - Determinare la quantità totale di tossina necessaria utilizzando la seguente formula:
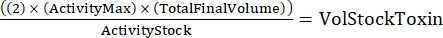 (Eq 2)
(Eq 2)
dove ActivityMax è la concentrazione massima utilizzata (HU/ml); TotalFinalVolume è il volume totale (per sei campioni, 200 x 6 + 100 = 1.300 μL); ActivityStock è l'attività dello stock di tossine (HU/ml); e VolStockToxin è il volume di tossine necessarie. - Preparare un buffer di analisi sufficiente per l'esperimento. Integrare il mezzo basale con un colorante di vitalità e qualsiasi Ca 2+ o EGTA necessario per controllare i livelli di Ca2+. Vortice da mescolare.
NOTA: Ad esempio, per ogni tampone Tyrode da 10 ml, aggiungere 50 μL di 2 mg/mL di PI e 200 μL di 100 mM CaCl 2, ottenendo concentrazioni finali rispettivamente di 10 μg/mL e2 mM. - Assicurarsi che il colorante di vitalità PI non sia in conflitto con altre sonde utilizzate, ad esempio per saggi di legame fluorescenti che utilizzano tossine coniugate Cy5 o AlexaFluor64720,25.
- Pianificare le diluizioni delle tossine per ottenere una soluzione 2x di tossina. Aggiungere il tampone di dosaggio alle provette da centrifuga da 1,5 ml e raffreddare sul ghiaccio. Aggiungere la tossina solo immediatamente prima di iniziare il test.
NOTA: in questo esempio, verranno aggiunti 1,3 ml di tampone di dosaggio per la diluizione superiore e 650 μL per le diluizioni seriali per preparare la soluzione di tossina 2x.
- Rsperimento
- Riservare 0,5 mL di promastigoti trasformati dal punto 1.8 in una provetta separata come "Controllo non colorato".
NOTA: questo campione verrà utilizzato per impostare il gating sul citometro a flusso. - Aggiungere 2 mg/mL di PI ad una concentrazione finale di 10 μg/mL ai promastagoti. Vortice per 3 s.
NOTA: Dopo l'aggiunta di PI ai promastigoti elaborati, queste cellule possono essere utilizzate solo per un periodo di tempo di 2,5 ore. Dopo 2,5 ore, le cellule iniziano a morire e i risultati diventano errati. - Aggiungere 1 x 105 (in 100 μL/pozzetto) promastigoti lavorati a ciascun pozzetto di una piastra a 96 pozzetti con fondo a V o tubi di microtitolazione da 1,2 mL (Figura 1). Posizionare la piastra o il portatubi sul ghiaccio con un angolo di circa 45° rispetto alla visualizzazione. Eseguire il lavoro in un armadio di biosicurezza quando si maneggia Leishmania promastigotes.
- Aggiungere 100 μL di tampone di analisi con PI a ciascun controllo senza tossine (ultima riga). Verificare che il controllo sia stato aggiunto correttamente identificando visivamente i tubi con un volume totale di 200 μL che appaiono di colore più scuro.
- Rimuovere le aliquote di tossina da -80°C, scongelarle sul ghiaccio e raggruppare se necessario. Aggiungere il volume di tossina calcolato al punto 2.1.5 alla diluizione massima (preparata al punto 2.1.10). Quindi, diluire serialmente la tossina (Figura 1). Pipettare su e giù almeno 8 volte per garantire la miscelazione.
NOTA: Eseguire su ghiaccio perché i CDC si inattivano rapidamente a temperatura ambiente. - Partendo dalla concentrazione di tossine più bassa, aggiungere rapidamente 100 μL di tossina alla riga corretta (Figura 1 e Figura 2) e continuare fino a quando tutta la tossina è stata aggiunta alle cellule.
- Sigillare la piastra con nastro sigillante. Incubare a 37 °C per 30 min. Dopo il periodo di incubazione, imballare e trasportare la piastra al citometro a flusso.
- Riservare 0,5 mL di promastigoti trasformati dal punto 1.8 in una provetta separata come "Controllo non colorato".
- Acquisizione dati
- Impostare il citometro a flusso (vedere la tabella dei materiali) e il software di acquisizione in base alle istruzioni del produttore e alla politica dell'impianto. Non eseguire la procedura di citometria a flusso senza una precedente formazione sul citometro.
NOTA: in questo esempio, è stato utilizzato un Attune NxT a 4 laser. PI è stato raccolto sul canale YL-1 (eccitato da un laser a 561 nm, passato attraverso un LP 577, riflesso da 600 DLP e filtrato attraverso 585/16 passa banda), sebbene l'ampio spettro di PI consenta la raccolta su altri canali. - Utilizzando il campione di L. major promastigote non colorato, impostare le porte per la dispersione anteriore e laterale e i parametri fluorescenti iniziali in base ai coloranti scelti.
- Includere un parametro aggiuntivo, se lo si desidera, per i grafici a punti per verificare l'autofluorescenza (ad esempio, "nessuna macchia") (Figura 3).
NOTA: in questo esempio, è stato utilizzato il canale BL-1 (eccitato da un laser a 488 nm, passato attraverso 495 DLP e 503 LP, riflesso da 555 DLP e filtrato tramite passa-banda 530/30). - Utilizzando controlli a colorazione singola, impostare le porte per il colorante di vitalità (PI in questo studio) e qualsiasi tossina etichettata con fluorescenza. Monitorare la dispersione in avanti rispetto al tempo per i micro-zoccoli.
NOTA: PI macchierà debolmente tutte le celle sopra i controlli non macchiati. Le cellule morte saranno facilmente separabili, con cellule transitoriamente permeabilizzate tra le popolazioni. - Acquisire >10.000 eventi gated per ogni campione sul citometro.
NOTA: si consiglia di leggere dal più sensibile al meno sensibile, ma l'ordine di acquisizione può essere invertito per determinare qualsiasi impatto dell'ordine di lettura sui risultati del campione. - Salvare i dati ed esportarli secondo necessità per l'analisi.
- Impostare il citometro a flusso (vedere la tabella dei materiali) e il software di acquisizione in base alle istruzioni del produttore e alla politica dell'impianto. Non eseguire la procedura di citometria a flusso senza una precedente formazione sul citometro.
- Analisi dei dati
Nota : in questo studio, Excel con plug-in Risolutore (vedere Tabella dei materiali) è stato utilizzato per l'analisi dei dati (vedi File supplementare 1).- Gate totale, singola cella L. maggiori promastigotes mediante gating su forward e side scatter e tempo secondo necessità (Figura 3). Utilizzare l'altezza o l'area come raccomandato per il citometro a flusso.
NOTA: per il citometro a flusso utilizzato qui, l'altezza è il parametro consigliato anziché l'area. - Identifica e cancella le cellule morte come "PI alto". Gate celle intermedie come "PI basso". Le cellule alte PI sono cellule morte, mentre le cellule basse PI sono transitoriamente permeabilizzate26.
NOTA: le celle PI alte mostrano in genere uno spostamento di 2-3 log dalle celle negative. - Esportare i dati in Excel. Ottenere il nome/ID del campione e %PI high per determinare l'uccisione.
NOTA: Se sono state utilizzate tossine fluorescenti, sarà necessaria l'intensità fluorescente mediana (MFI) delle popolazioni vive, transitoriamente permeabilizzate e negative. %PI basso può essere esportato per la permeabilizzazione transitoria. - Determinare l'elevato % PI medio per ogni condizione tra le due repliche tecniche.
NOTA: Se è necessaria una MFI media per le tossine fluorescenti, calcolare anche questo. - Calcolare %Lisi specifica dal %PI alto usando la seguente formula24,25:
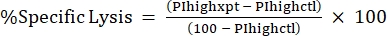 (Eq 3)
(Eq 3)
dove PIhighxpt è il %PI alto per la condizione sperimentale; e PIhighctl è l'alto %PI per il controllo senza tossine. - Grafico %Lisi specifica contro la concentrazione di tossine per la curva dose-risposta (Figura 4).
- Organizzare la curva dose-risposta in Excel per la modellazione logistica. Includere la concentrazione di tossine e la percentuale media di lisi specifica insieme a dettagli sperimentali e/o calcoli grezzi %PI elevati (Tabella 2).
- Verificare che il componente aggiuntivo Risolutore sia abilitato.
Nota : per attivare il Risolutore nella versione desktop di Excel, passare a Opzioni > file > componenti aggiuntivi e selezionare la casella Risolutore . Riavviare Excel. - Etichettare altre quattro colonne come "modellato", "residui", "parametri" e "valori di parametro". Verificare che le prime colonne corrispondano ai parametri sperimentali, alla concentrazione di tossine e alla %Lisi specifica (Tabella 2).
- Aggiungere i seguenti parametri nella colonna "parametri": L, k, c, SUM e LC50. Inizializzare i parametri L, k e c immettendo i seguenti valori nella colonna "valori dei parametri": 100, 0,05, 1.000.
- Nella colonna "modellato", creare il modello logistico utilizzando la seguente formula:
 (Eq 4)
(Eq 4)
Impostare L, k e c sulle celle contenenti tali parametri nella colonna "valori dei parametri".
Impostare x sulla cellula contenente la concentrazione di tossine.
Nota : per la tabella 2, cella G4, la formula è la seguente: =$J$3/(1+EXP(-$J$4*(D4-$J$5))) - Applicare questa equazione a tutti i valori %Specific Lysis ad eccezione del controllo senza tossine.
- Nella colonna "residui", calcolare il quadrato della differenza tra il numero modellato e la lisi specifica effettiva utilizzando la seguente equazione:
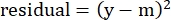 (Eq. 5)
(Eq. 5)
dove y è la %Lisi specifica sperimentale; e m è il valore corrispondente nella colonna "modellato" calcolato nei passaggi 2.4.10-2.4.11. - Nella colonna "valori dei parametri" accanto a "SOMMA", sommare tutti i valori nella colonna dei residui.
- Nella colonna "valori dei parametri" accanto a "LC50", inizializzare l'equazione per calcolare la LC50 dai valori determinati. Questo risolve Eq 4 per x quando m = 50.
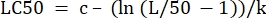 (Eq 6)
(Eq 6)
Per la tabella 2, la formula di Excel è la seguente: =J5-(LN(J3/(50)-1))/J4 - Aprire il Risolutore dalla scheda Dati. Selezionare Imposta obiettivo come cella contenente la somma dei residui calcolati. Impostalo su Min.
- Modificare le celle delle variabili per i valori dei parametri L, k e c.
NOTA: il risolutore potrebbe comportarsi meglio se "Rendi variabili non vincolate non negative" è lasciato selezionato. - Per i valori k negativi, modificare Eq 4 ed Eq 6 estraendo −1 da k per cambiare k in positivo. Utilizzare il metodo di risoluzione non lineare GRG. Fare clic su Risolvi.
- Controllare la curva e che la LC50 venga calcolata automaticamente utilizzando l'Eq 6 (Tabella 2). Verificare l'adattamento tracciando graficamente sia %Lisi specifica che modellando rispetto alla concentrazione di tossine.
NOTA: Può anche essere controllato calcolando la curva R2 .
- Gate totale, singola cella L. maggiori promastigotes mediante gating su forward e side scatter e tempo secondo necessità (Figura 3). Utilizzare l'altezza o l'area come raccomandato per il citometro a flusso.
3. Analisi proteica di L . major promastigotes con tossina
- Preparare L. major promastigotes come descritto nel paragrafo 1.
- Risospendere 2 x 107 WT, spt2- e spt2-/+SPT2 L. promastigoti maggiori in 2 mL del tampone di analisi desiderato utilizzando una pipetta da 5 mL (ad esempio, M199 senza siero). Aggiungere la tossina ad una concentrazione sublitica finale e incubare a 37 °C per 30 minuti.
NOTA: Ad esempio, una dose sublitica di SLO per spt2- promastigotes è 500 HU / ml. - Includere altri genotipi e controlli senza tossine.
- Centrifugare i promastigoti a 1.500 x g per 10 minuti per pellettare le cellule. Eliminare il surnatante utilizzando una pipetta da 10 ml. Far scorrere rapidamente il tubo chiuso contenente il pellet cellulare tre volte su una superficie irregolare, come la griglia dell'armadio di biosicurezza, per rompere il pellet cellulare.
NOTA: Il pellet cellulare è quasi invisibile ad occhio nudo. - Ricostituire 1x tampone campione SDS-PAGE con 2-mercaptoetanolo immediatamente prima dell'uso e riscaldare a 95 °C per 10 minuti prima dell'aggiunta al pellet cellulare. Risospendere il pellet di cella in 1x tampone campione SDS-PAGE caldo e mescolare bene pipettando su e giù. Riscaldare il pellet cellulare risospeso in 1x tampone campione a 95 °C per 10 minuti.
NOTA: Dopo la solubilizzazione in tampone del campione, conservare i campioni a lungo termine a -20 °C, se necessario. - Preparare il gel risolutivo. Degasare tutti i componenti tranne il persolfato di ammonio (APS) e TEMED per 15 minuti.
- Aggiungere APS e TEMED immediatamente prima di colare il gel. Sovrapporre con cura il gel risolutivo con acqua. Lasciare polimerizzare il gel risolunte, ~30-45 min.
- Decantare l'acqua e preparare il gel impilabile. Aggiungere il gel impilante, avendo cura di evitare bolle. Inserire un pettine con il relativo numero di pozzetti e lasciare polimerizzare per 5 minuti. Monitorare la polimerizzazione utilizzando qualsiasi gel impilabile avanzato.
- Assemblare il gel per l'esecuzione di SDS-PAGE e aggiungere il buffer del serbatoio alla camera.
- Caricare 10 μL di ciascun campione per pozzetto o 8 μL della scala proteica. Eseguire a 180 V fino a quando i campioni entrano nel gel risolutivo, quindi ridurre la tensione a ~ 160 V e correre fino a quando il fronte del colorante è ~ 0,5 cm dal bordo della piastra.
NOTA: Il tempo necessario affinché i campioni raggiungano il fondo può variare tra 1-1,5 ore. Il tempo può essere allungato riducendo la tensione. Non ridurre mai la tensione a zero. Tensioni più elevate possono aumentare il gel "sorridente" e rompere le piastre. - Trasferire il gel sulla colorazione Coomassie (per la colorazione delle proteine) o sul tampone di trasferimento 1x (per il western blotting). Per la colorazione di Coomassie, colorare durante la notte, quindi decolorare, visualizzare e asciugare.
- Per western blot, preparare il sistema di trasferimento secondo le istruzioni del produttore.
- Per un trasferimento a umido, utilizzare tampone di trasferimento freddo 1x e tamponi pre-bagnati, carta da filtro e nitrocellulosa. Per il sistema Bio-rad Protean III (vedi Tabella dei materiali) qui utilizzato, la carta da filtro può essere tagliata a 10 cm x 7,5 cm. Tagliare la nitrocellulosa a 9 cm x 6,75 cm.
NOTA: La nitrocellulosa è altamente infiammabile. Evitare fiamme libere e altre potenziali fonti di accensione. - Disporre la cassetta di trasferimento con tamponi, carta da filtro e nitrocellulosa. Stendere le bolle d'aria. Aggiungere il gel con cura.
- Aggiungere la carta da filtro e stendere le bolle d'aria. Aggiungere il tampone, chiudere il cassetto e inserirlo nel supporto con l'orientamento corretto (assicurarsi che la nitrocellulosa sia rivolta verso il terminale rosso). Aggiungere una barra di agitazione e un impacco di ghiaccio sul lato e riempire il serbatoio con tampone di trasferimento freddo 1x. Trasferimento a 110 V per 90 min.
NOTA: Il calore generato durante il trasferimento può influire negativamente sul trasferimento. Per garantire un buon trasferimento, utilizzare sempre un buffer di trasferimento freddo 1x. - Rimuovere la nitrocellulosa e colorare con la soluzione di Ponceau per ~5 min. Risciacquare con acqua ultrapura. Segna la scala proteica a matita e taglia la macchia secondo necessità. Conservare la macchia utilizzando il buffer di trasferimento rimanente.
NOTA: Ponceau può essere riutilizzato molte volte. - Bloccare la nitrocellulosa in 25 ml di BSA al 5% in 1x TBST a 4 °C, agitando, durante la notte. Quindi, scartare la soluzione di blocco e aggiungere l'anticorpo primario (1:1.000) in 1% BSA in 1x TBST. Agitare a 4 °C per una notte.
NOTA: L'anticorpo primario può essere salvato a -20 °C e riutilizzato più volte. - Lavare la nitrocellulosa 3x per 10 minuti ciascuno in 1x TBST agitando. Eliminare il lavaggio e aggiungere 10 ml di anticorpo secondario coniugato HRP (1:10.000) in BSA all'1% in 1x TBST. Agitare a temperatura ambiente per 1 h. Lavare la nitrocellulosa 3x per 10 minuti ciascuno in 1x TBST agitando.
- Preparare il reagente ECL immediatamente prima di eseguire l'imaging della nitrocellulosa. Immediatamente prima dell'imaging, decantare il TBST e aggiungere il reagente ECL alla nitrocellulosa. Agitare per 1 min. Immagina il gel.
Access restricted. Please log in or start a trial to view this content.
Risultati
Maggiore sensibilità alla promastigote allo SLO nel buffer di Tyrode rispetto a M199
La sensibilità SLO di L. major promastigotes è stata confrontata tra diversi buffer di analisi. I promastigoti wild-type, spt2- e spt2-/+SPT2 sono stati sfidati con SLO in M199 privo di siero o tampone di Tyrode integrato con 2 mM CaCl2 per 30 minuti prima dell'analisi su un citometro a flusso. I parassiti adatti per l'analisi erano singole cellule identif...
Access restricted. Please log in or start a trial to view this content.
Discussione
In questo studio sono stati descritti metodi per studiare i meccanismi molecolari e le funzioni dei PFT, utilizzando il patogeno umano Leishmania major come sistema modello. È stato sviluppato un saggio di citotossicità basato sulla citometria a flusso a media produttività per misurare la vitalità di una singola cellula. La vitalità è quantitativa a livello di popolazione perché i valori di LC50 possono essere calcolati dalla curva dose-risposta utilizzando la modellazione logistica. Come prova...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Questo lavoro è stato sostenuto dal National Institute Of Allergy And Infectious Diseases del National Institutes of Health grant R21AI156225 a PAK e KZ (co-I) e R01AI139198 a KZ (co-I). CH desidera ringraziare il Dipartimento di Scienze Biologiche per l'assistentato didattico fornito durante il periodo di questo studio.
Le agenzie di finanziamento non hanno avuto alcun ruolo nella progettazione dello studio; nella raccolta, analisi o interpretazione dei dati; nella stesura del manoscritto; né nella decisione di pubblicare i risultati. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente il punto di vista ufficiale delle agenzie di finanziamento. Gli autori dichiarano di non avere conflitti di interesse concorrenti.
Riconoscimenti
Gli autori desiderano ringraziare i membri dei laboratori Keyel e Zhang per la loro revisione critica del manoscritto. Gli autori ringraziano il College of Arts and Sciences Microscopy per l'uso delle strutture.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.2 mL microtiter (Marsh) tubes | Fisher | 02-681-376 | Cytotoxicity assay |
| 1.5 mL microcentrifuge tube | Fisher | 05-408-129 | Toxin dilutions |
| 15 mL centrifuge tube | Avantor VWR (Radnor, PA) | 89039-666 | To hold cells and media |
| 1x Phosphate buffered saline (PBS) | Fisher | BP399 | For cell processing |
| 3% H2O2 | Walmart (Fayetteville, AR) | N/A | For ECL |
| 5x M199 | Cell-gro | 11150067 | Basal growth media for L. major promastigotes |
| Biosafety cabinet | Baker | To culture cells in sterile conditions | |
| Bovine serum albumin (BSA) | Fisher | BP1605-100 | Fraction V acceptable purity |
| CaCl2 | Fisher | BP510-100 | Stock concentration 100 mM |
| Centrifuge | Thermo Fisher | Heraeus Megafuge 40R | To pellet the cells from culture |
| Cy5 Mono-reactive dye pack | Cytiva (Marlborough, MA) | PA25031 | Fluorophore label for toxins |
| Digital dry bath | Benchmark | BSH1002 | To denature protein samples |
| EGTA | Amresco | 0732-100G | Stock concentration 0.5 M |
| Excel | Microsoft (Redmond, VA) | Data analysis software | |
| Flow cytometer (4-laser Attune NxT) | Fisher | Cytometer for data acquisition | |
| FlowJo | BD (Ashland, OR) | Software | |
| Formaldehyde | Fisher | BP531-500 | Fixative for counting cells |
| G418 | Fisher | BP673-1 | Selection agent for cells |
| Hellmanex III | Sigma | Z805939 | Dilute 1:4 for cleaning cytometer |
| Hemacytometer | Fisher | 0267151B | For counting cells |
| Human red blood cells | Zen-bio (Durham, NC) | SER-10MLRBC | To validate toxin activity |
| Ice bucket | |||
| Light microscope | Nikon | Eclipse 55i | To visualize cells |
| Nitrocellulose | Fisher | 88018 | For probing proteins via antibodies |
| Pipettors and tips | Avantor VWR | To dispense reagents | |
| Power supply | Bio-Rad | To run SDS-PAGE and transfers | |
| Propidium iodide | Biotium | 40016 | Stock concentration 2 mg/mL in water |
| Protein ladder | Bio-Rad | 161-0373 | To determine molecular weight of proteins |
| SDS-PAGE Running Apparatus (Mini Protean III) | Bio-Rad | 165-3302 | To separate proteins based on their size |
| Sealing tape | R&D | DY992 | To seal plates with cells |
| Streptolysin O C530A plasmid insert | Cloned into pBAD-gIII vector (Reference: 7) | ||
| Streptolysin O C530A toxin | Lab purified | Specific activity 4.34 x 105 HU/mg | |
| Swinging bucket rotor | Thermo Fisher | 75003607 | To centrifuge cells |
| V-bottom plate | Greiner Bio-one | 651206 | For cytotoxicity assay |
| Vortex | Benchmark | BV1000 | To mix cells |
| Western blot imaging system (Chemi-doc) | Bio-Rad | To visualize proteins by western blot | |
| Western Blot Transfer Apparatus (Mini Protean III) | Bio-Rad | 170-3930 | Transfer proteins to nitrocellulose |
| Whatman Filter paper | GE Healthcare Life Sciences | 3030-700 | Used in transfer of proteins to nitrocellulose |
| Antibody | |||
| Anti-ERK antibody | Cell Signaling Technologies | Cat# 9102S | Rabbit (1:1000 dilution) |
| Anti-lipophosphoglycan (LPG) antibody | CreativeBioLabs | Cat# WIC79.3 | Mouse (1: 1000) |
| Anti-MEK antibody | Cell Signaling Technologies | Cat# 9122L | Rabbit (1:1000) |
| Anti-mouse IgG, HRP conjugate | Jackson Immunoresearch | Cat#715-035-151 | Donkey (1:10000) |
| Anti-phosphoERK antibody | Cell Signaling Technologies | Cat# 9101S | Rabbit (1:1000) |
| Anti-pMEK antibody | Cell Signaling Technologies | Cat# 9121S | Rabbit (1:1000) |
| Anti-rabbit IgG, HRP conjugate | Jackson Immunoresearch | Cat#711-035-152 | Donkey (1:10000) |
| Anti-tubulin antibody | Sigma | Cat# T5168 | Mouse (1: 2000) |
| Leishmania major Genotypes | Reference: 13 | ||
| Episomal addback (spt2-/+SPT2) | Δspt2::HYG/Δspt2:PAC/+pXG-SPT2 | ||
| Serine palmitoyltransferase subunit 2 knockout (spt2-) | Δspt2::HYG/Δspt2::PAC | ||
| Wild type (WT) | LV39 clone 5 (Rho/SU/59/P) |
Riferimenti
- Thapa, R., Ray, S., Keyel, P. A. Interaction of macrophages and cholesterol-dependent cytolysins: The impact on immune response and cellular survival. Toxins. 12 (9), 531(2020).
- Limbago, B., Penumalli, V., Weinrick, B., Scott, J. R. Role of streptolysin O in a mouse model of invasive group A streptococcal disease. Infection & Immunity. 68 (11), 6384-6390 (2000).
- Farrand, A. J., et al. The cholesterol-dependent cytolysin membrane-binding interface discriminates lipid environments of cholesterol to support beta-barrel pore insertion. Journal of Biological Chemistry. 290 (29), 17733-17744 (2015).
- Soltani, C. E., Hotze, E. M., Johnson, A. E., Tweten, R. K. Structural elements of the cholesterol-dependent cytolysins that are responsible for their cholesterol-sensitive membrane interactions. Proceedings of the National Academy of Sciences of the United States of America. 104 (51), 20226-20231 (2007).
- Schoenauer, R., et al. Down-regulation of acid sphingomyelinase and neutral sphingomyelinase-2 inversely determines the cellular resistance to plasmalemmal injury by pore-forming toxins. FASEB Journal. 33 (1), 275-285 (2019).
- Ray, S., Roth, R., Keyel, P. A. Membrane repair triggered by cholesterol-dependent cytolysins is activated by mixed lineage kinases and MEK. Science Advances. 8 (11), (2022).
- Babiychuk, E. B., Monastyrskaya, K., Draeger, A. Fluorescent annexin A1 reveals dynamics of ceramide platforms in living cells. Traffic. 9 (10), 1757-1775 (2008).
- Jimenez, A. J., et al. ESCRT machinery is required for plasma membrane repair. Science. 343 (6174), 1247136(2014).
- Demonbreun, A. R., et al. An actin-dependent annexin complex mediates plasma membrane repair in muscle. Journal of Cell Biology. 213 (6), 705-718 (2016).
- Wolfmeier, H., et al. Ca(2)(+)-dependent repair of pneumolysin pores: A new paradigm for host cellular defense against bacterial pore-forming toxins. Biochimica et Biophysica Acta. 1853 (2), 2045-2054 (2015).
- Bravo, F., Sanchez, M. R. New and re-emerging cutaneous infectious diseases in Latin America and other geographic areas. Dermatologic Clinics. 21 (4), 655-668 (2003).
- Manfredi, M., Iuliano, S. Cutaneous leishmaniasis with long duration and bleeding ulcer. Clinical Microbiology Open Access. 05, 2-6 (2016).
- Zhang, K., et al. Sphingolipids are essential for differentiation but not growth in Leishmania. EMBO Journal. 22 (22), 6016-6026 (2003).
- Zhang, K. Balancing de novo synthesis and salvage of lipids by Leishmania amastigotes. Current Opinions in Microbiology. 63, 98-103 (2021).
- Kaur, P., Goyal, N. Pathogenic role of mitogen activated protein kinases in protozoan parasites. Biochimie. 193, 78-89 (2022).
- Wiese, M. Leishmania MAP kinases--Familiar proteins in an unusual context. International Journal of Parasitology. 37 (10), 1053-1062 (2007).
- Brumlik, M. J., Pandeswara, S., Ludwig, S. M., Murthy, K., Curiel, T. J. Parasite mitogen-activated protein kinases as drug discovery targets to treat human protozoan pathogens. Journal of Signal Transduction. 2011, 971968(2011).
- Wiese, M., Kuhn, D., Grunfelder, C. G. Protein kinase involved in flagellar-length control. Eukaryotic Cell. 2 (4), 769-777 (2003).
- Agron, P. G., Reed, S. L., Engel, J. N. An essential, putative MEK kinase of Leishmania major. Molecular Biochemistry of Parasitology. 142 (1), 121-125 (2005).
- Ray, S., Thapa, R., Keyel, P. A. Multiple parameters beyond lipid binding affinity drive cytotoxicity of cholesterol-dependent cytolysins. Toxins. 11 (1), (2018).
- Beneke, T., et al. A CRISPR Cas9 high-throughput genome editing toolkit for kinetoplastids. Royal Society Open Science. 4 (5), 170095(2017).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular and Cellular Biology. 10 (3), 1084-1094 (1990).
- Moitra, S., Pawlowic, M. C., Hsu, F. F., Zhang, K. Phosphatidylcholine synthesis through cholinephosphate cytidylyltransferase is dispensable in Leishmania major. Scientific Reports. 9, 7602(2019).
- Keyel, P. A., Heid, M. E., Watkins, S. C., Salter, R. D. Visualization of bacterial toxin induced responses using live cell fluorescence microscopy. Journal of Visualized Experiments. (68), 4227(2012).
- Romero, M., et al. Intrinsic repair protects cells from pore-forming toxins by microvesicle shedding. Cell Death & Differentiation. 24 (5), 798-808 (2017).
- Keyel, P. A., et al. Streptolysin O clearance through sequestration into blebs that bud passively from the plasma membrane. Journal of Cell Science. 124, 2414-2423 (2011).
- Dong, Z., Patel, Y., Saikumar, P., Weinberg, J. M., Venkatachalam, M. A. Development of porous defects in plasma membranes of adenosine triphosphate-depleted Madin-Darby canine kidney cells and its inhibition by glycine. Laboratory Investigations. 78 (6), 657-668 (1998).
- Loomis, W. P., den Hartigh, A. B., Cookson, B. T., Fink, S. L. Diverse small molecules prevent macrophage lysis during pyroptosis. Cell Death & Disease. 10 (4), 326(2019).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon