Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Entschlüsselung des molekularen Mechanismus und der Funktion von porenbildenden Toxinen mit Leishmania major
In diesem Artikel
Zusammenfassung
Hier wird ein Protokoll vorgestellt, das Leishmania major promastigotes verwendet, um die Bindung, Zytotoxizität und Signalisierung zu bestimmen, die durch porenbildende Toxine induziert werden. Ein Proof-of-Concept mit Streptolysin O wird bereitgestellt. Andere Toxine können auch verwendet werden, um die in L. major verfügbaren genetischen Mutanten zu nutzen, um neue Mechanismen der Toxinresistenz zu definieren.
Zusammenfassung
Das Verständnis der Funktion und des Mechanismus von porenbildenden Toxinen (PFTs) ist eine Herausforderung, da Zellen den durch PFTs verursachten Membranschäden widerstehen. Während biophysikalische Ansätze helfen, die Porenbildung zu verstehen, stützen sie sich oft auf reduktionistische Ansätze, denen das vollständige Komplement von Membranlipiden und Proteinen fehlt. Kultivierte menschliche Zellen bieten ein alternatives System, aber ihre Komplexität und Redundanzen in Reparaturmechanismen erschweren die Identifizierung spezifischer Mechanismen. Im Gegensatz dazu bietet der für die kutane Leishmaniose verantwortliche humane Protozoenerreger Leishmania major eine optimale Balance zwischen Komplexität und physiologischer Relevanz. L. major ist genetisch beherrschbar und kann in vitro zu hoher Dichte kultiviert werden, und jeder Einfluss von Störungen auf Infektionen kann in etablierten Mausmodellen gemessen werden. Darüber hinaus synthetisiert L. major Lipide, die sich von ihren Gegenstücken bei Säugetieren unterscheiden, was die Membrandynamik verändern könnte. Diese Veränderungen in der Membrandynamik können mit PFTs aus der am besten charakterisierten Toxinfamilie, den cholesterinabhängigen Cytolysinen (CDCs), untersucht werden. CDCs binden an Ergosterol in der Leishmania-Membran und können L. major promastigotes abtöten, was darauf hindeutet, dass L. major ein geeignetes Modellsystem zur Bestimmung der zellulären und molekularen Mechanismen der PFT-Funktion ist . Diese Arbeit beschreibt Methoden zum Testen der PFT-Funktion in L. major Promastigoten, einschließlich Parasitenkultur, genetischen Werkzeugen zur Beurteilung der Lipidempfindlichkeit, Membranbindungsassays und Zelltodtests. Diese Assays werden die schnelle Verwendung von L. major als leistungsfähiges Modellsystem zum Verständnis der PFT-Funktion über eine Reihe von evolutionär unterschiedlichen Organismen und Gemeinsamkeiten in der Lipidorganisation ermöglichen.
Einleitung
Porenbildende Toxine (PFTs) sind die größte Familie bakterieller Toxine1, aber die Mechanismen, mit denen sie Zellen perforieren und zerstören, sind kaum verstanden. Die am besten untersuchte Familie porenbildender Toxine ist die der cholesterinabhängigen Cytolysine (CDCs). CDCs werden hauptsächlich von grampositiven Bakterien synthetisiert, einschließlich des Erregers der nekrotisierenden Fasziitis, Streptococcus pyogenes2. S. pyogenes sezerniert das CDC-Streptolysin O (SLO), das als Monomere an Sterole in der Plasmamembran von Wirtszellen bindet, oligomerisiert und ~20-30 nm Poren in die Membraneinfügt 1. Die Rolle, die Lipide in diesem Prozess spielen, ist nach wie vor schlecht bestimmt.
Ein Ansatz zur Untersuchung von Lipid-CDC-Wechselwirkungen ist die Verwendung chemisch definierter Liposomen. Während definierte Liposomen Informationen über die notwendigen Schwellenwerte von Lipiden liefern, um die Toxinbindung und Porenbildung aufrechtzuerhalten3,4, rekapitulieren sie die zellulären Funktionen nicht vollständig. Zum Beispiel fehlt rekonstituierten Liposomen die Lipidasymmetrie von Säugetierwirten und Lipidmodifikationen als Reaktion auf Toxine5. Eine Alternative zu Liposomen ist die Verwendung von Säugetierzelllinien. Während diese Zelllinien physiologisch relevanter sind, gibt es ein hohes Maß an Redundanz in den Toxinsensor- und Resistenzmechanismen2. Infolgedessen bleiben die Reparaturwege, die verwendet werden, um CDCs zu widerstehen, schlecht bestimmt. Insbesondere ist der Ca2+ Zustrom der primäre Aktivator der Membranreparatur1. Stromabwärts des Ca2+-Zustroms sind mehrere Signalwege angelegt, darunter ein Ceramid-abhängiger Reparaturweg 6,7 und ein MEK-abhängiger Reparaturweg6. Diese Signalwege interagieren mit anderen Proteineffektoren, einschließlich des für den Transport erforderlichen endosomalen Sortierkomplexes (ESCRT)8 und der Annexine 6,9,10. Die Analyse dieser Signalwege in Säugetierzellen ist aufgrund der Redundanz, die die Dateninterpretation durcheinander bringt, eine Herausforderung.
Eine Möglichkeit, Komplexität mit Einfachheit für die Analyse von Reparaturwegen in Einklang zu bringen, ist die Verwendung einfacherer Organismen wie Protozoenpathogene der Gattung Leishmania. Leishmania sp. verursachen Leishmaniose bei Menschen und anderen Tieren. Die Leishmaniose reicht von der kutanen Leishmaniose (selbstlimitierende Hautläsionen) bis zur tödlichen viszeralen Leishmaniose (Hepatosplenomegalie), abhängig von der Spezies und anderen Faktoren11. Leishmania major, der Erreger der kutanen Leishmaniose, wird über einen Sandmückenvektor auf den Menschen übertragen und wird verwendet, um die Leishmanienfunktion und -infektion zu verstehen12. Darüber hinaus sind Leishmania sp. digenic12. Sie existieren als intrazelluläre Makrophagenparasiten von Säugetieren, die als Amastigotes bezeichnet werden, und als frei schwimmende, begeißelte Promastigoten in der Sandfliege12. L. major Promastigoten können in serumergänzten Medien wie M199 zu hoher Dichte kultiviert werden13. Promastigoten sind auch genetisch beherrschbar; Es gibt viele Gen-Knockouts, einschließlich solcher, die auf Lipidbiosynthesewege abzielen13. Diese Knockouts können hinsichtlich des Wachstums und der Unterschiede in der Infektiosität und Läsionsentwicklung durch Infektion von Balb/c-Mäusen bewertet werden13.
Zusätzlich zu der relativen Leichtigkeit der Leishmania-Kultur und der Bandbreite der Lipidbiosynthese-Knockouts hat der Parasit ein einfacheres Genom als Säugetiere. Die am besten charakterisierte Art von Leishmania ist L. major, die viele genetische Werkzeuge hat, wie Mutanten mit defektem Fettstoffwechsel14. Bemerkenswerterweise fehlen viele Reparaturproteine. Für L. major wurden bisher keine Homologe für wichtige Reparaturproteine von Säugetieren wie Annexine identifiziert. Dies ermöglicht die Charakterisierung evolutionär konservierter Reparaturwege ohne die Komplexität von Säugetiersystemen. Reparaturwege wurden in Leishmanien bisher jedoch nicht charakterisiert. Gleichzeitig sind wichtige Signalwege, die an der Reparatur beteiligt sind, wie der MEK-Signalweg6, in Leishmania sp.15,16 erhalten, obwohl Homologe validiert werden müssen. Der Mitogen-aktivierte Proteinkinase-Signalweg (MAPK) ist in L. mexicana gut untersucht, wo er zum intrazellulären Überleben und zur Thermostabilität in Säugetierzellen beiträgt und die Metazyklogenese steuert16. In Leishmania sp. wurden 10 der 15 MAPKs17 charakterisiert. Es wird vorhergesagt, dass LmMAPK9 und LmMAPK13 dem ERK1/2 von Säugetieren am ähnlichsten sind, basierend auf der Identität in der konservierten Phosphorylierungslippensequenz. Die Phosphorylierungslippensequenz ist TEY sowohl für Säugetiere ERK1/2 als auch für LmMAPK9 und LmMAPK13. Acht der Leishmania MAPKs haben jedoch ein TDY-Phosphorylierungsmotiv15. Mindestens zwei Homologe von MEK wurden in Leishmania sp., LmxMKK18 und MEKK-related kinase (MRK1)19 identifiziert. Dies deutet darauf hin, dass die in Leishmanien identifizierten Erkenntnisse auf Säugetiersysteme übertragen werden könnten. Wo sie nicht in Säugetiersysteme übersetzt werden, stellen sie therapeutische Ziele für die Behandlung von Leishmaniose dar.
Um L. major Promastigoten zur Untersuchung der Membranreparatur und der Wechselwirkungen mit Toxinen zu verwenden, sind Techniken mit mittlerem Durchsatz erforderlich. Während hochauflösende Lebendzellbildgebung die Visualisierung markierter Proteine und Membranen in Echtzeit ermöglicht, ist sie ein geringer Durchsatz und misst möglicherweise nicht das Überleben der Zellen. Medium-Throughput-Rentabilitätstests umfassen die mittels Durchflusszytometrie gemessene Farbstoffaufnahme, die Messung der mitochondrialen Aktivität oder die Freisetzung zellulärer Proteine wie Laktatdehydrogenase (LDH). In Säugetierzellen messen LDH-Assays den Zelltod nicht quantitativ20. Darüber hinaus erlauben populationsbasierte Assays wie LDH-Freisetzung oder mitochondriale Aktivität keine robuste Einzelzell- oder multiparametrische Analyse20. Im Gegensatz dazu ermöglichen durchflusszytometrische Assays eine multiparametrische Einzelzellanalyse20. Diese Assays wurden jedoch nicht angewendet, um die Toxinbiologie oder Reaktionen auf Toxine in L. major promastigotes zu verstehen.
In dieser Studie wird SLO als Werkzeug verwendet, um die Plasmamembranstörung der Sphingolipid-Nullmutante von L. major in zwei verschiedenen Puffern zu verstehen - dem M199-Medium, das routinemäßig zur Kultivierung von L. major-Promastigoten verwendet wird, und dem einfacheren Tyrode-Puffer. Ein Durchflusszytometrie-Assay mit mittlerem Durchsatz wird beschrieben und verwendet, um Toxin-Dosis-Wirkungs-Kurven zu erzeugen. Die Daten des durchflusszytometrischen Assays werden zu einer logistischen Kurve modelliert, um die LC50-Werte zu bestimmen. Mit diesen Informationen kann eine sublytische Dosis von SLO bestimmt werden, so dass MAPK-Antikörper mittels Western Blotting validiert werden können.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Für die Verwendung und Handhabung des RG2-Erregers Leishmania major und der rekombinanten DNA wurden alle geeigneten Richtlinien und mikrobiologischen, Sicherheits- und Zellkulturpraktiken angewendet. Alle Experimente mit lebendem L. major wurden in einer Biosicherheitswerkbank in einem BSL-2-zertifizierten Labor durchgeführt. Die Arbeit wurde vom Texas Tech University Institutional Biosafety Committee überwacht.
HINWEIS: Aus Sicherheitsgründen sind lebende L. major Promastigoten Risikoerreger der Gruppe 2. Handhabung mit geeigneten Eindämmungsmaßnahmen, Vorsichtsmaßnahmen und Aufsicht durch das Institutional Biosafety Committee (IBC). Umgang mit toxischen Substanzen und Chemikalien in Übereinstimmung mit institutionellen Verfahren für toxische Substanzen. Wenn rekombinante Toxine verwendet werden, kann eine IBC-Zulassung und -Aufsicht für rekombinante DNA-Arbeiten erforderlich sein.
1. Kultivierung und Zubereitung von L. major promastigotes
- Gewinnung oder Herstellung und Validierung von L. major genetischen Mutanten, wie zuvor beschrieben, entweder unter Verwendung homologer Rekombination oder CRISPR-basierter Methoden13,21. Verwenden Sie Knockouts, die mit dem Gen ergänzt werden, das wieder auf einem Plasmid hinzugefügt wird, um die Spezifität des Knockouts sicherzustellen.
- Kultur Wildtyp L. major und spt2- Promastigoten bei 27 °C im kompletten M199-Medium. Kultur der episomalen Addback-Zellen (spt2-/+SPT2) in vollständigem M199 plus 10 μg/ml G418 (siehe Tabelle 1 und Materialtabelle).
HINWEIS: Der gesamte Versuchsaufbau mit Experimenten mit L. major Zellen muss in einer BSL2-zertifizierten Biosicherheitswerkbank durchgeführt werden. - Die Promastigoten werden in vollständigem M199-Medium22 kultiviert, bis sie die logarithmische Phase (2-8 x 106 Zellen/ml) erreichen, wie durch L. major growth curve Assays bestimmt, die zuvor23 durchgeführt wurden. Planen Sie 1 x 10 5 Zellen für jede Vertiefung auf Zytotoxizität, plus 5 x 105 Zellen für die Färbekontrolle. Für Western Blot planen Sie 2 x 107 Zellen pro Vertiefung ein.
HINWEIS: Führen Sie einen Zytotoxizitätstest mit zwei technischen Replikaten durch. - Um die richtige Zelldichte zu überprüfen, mischen Sie ein Aliquot (10-40 μL) Promastigoten mit einem gleichen Volumen Fixiermittel (3,7% Paraformaldehyd in 1x PBS). Laden Sie 10 μL der fixierten Probe auf jede Seite des Hämazytometers.
ACHTUNG: Formaldehyd ist eine giftige Chemikalie. Handhabung in Übereinstimmung mit den institutionellen Richtlinien für gefährliche Chemikalien. - Führen Sie die Zellzählung mit einem Mikroskop bei 20-facher Vergrößerung durch. Zählen Sie alle Zellen in den 25 kleinen Quadraten in der Mitte des Hämazytometers. Wiederholen Sie dies für die Quadrate auf beiden Seiten und durchschnittlich die Anzahl.
HINWEIS: Wenn die Abweichung zwischen den Zählungen >10 beträgt, zählen Sie erneut und durchschnittlich. Wenn die durchschnittliche Anzahl <10 oder >100 beträgt, ändern Sie die Verdünnung und Neuzählung, und berechnen Sie dann die Kulturdichte mit der folgenden Formel: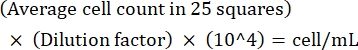 (Gl. 1)
(Gl. 1)
Wenn es beispielsweise durchschnittlich 250 Leishmanien in 25 Quadraten gibt, beträgt die Kulturdichte 5 x 106 Zellen / ml. - Nach dem Zählen 5 x 106 Zellen in ein 15 ml konisches Röhrchen überführen und bei 1.500 x g für 8 min bei Raumtemperatur zentrifugieren, um die Zellen zu pelletieren.
- Verwerfen Sie den Überstand mit einer 10-ml-Pipette und wirbeln Sie das Zellpellet kurz. Fügen Sie 5 ml 1x PBS in die gleiche Tube und waschen Sie die Zellen, indem Sie 3-6x vorsichtig invertieren. Zentrifugieren bei 1.500 x g für 8 min bei Raumtemperatur, um die Zellen zu pelletieren.
- Verwerfen Sie den Überstand mit einer 10-ml-Pipette und resuspendieren Sie das 5 x 10 6-Zell-Pellet in 5 ml Medium (z. B. M199 oder 1x Tyrode-Puffer), das für die Experimente mit einer 5-ml-Pipette verwendet wurde, um eine Endkonzentration von 1 x 106 Zellen / ml zu erhalten.
2. Zytotoxizitätstest
- Experimentelle Vorbereitung
- Reinigen Sie das Toxin wie zuvor beschrieben24 oder kaufen Sie das Toxin von einem Händler. Aliquot in Einwegaliquots umwandeln und −80 °C bis zu 1 Jahr lagern. Vermeiden Sie mehrere Gefrier-Tau-Zyklen.
- Bestimmen Sie die hämolytische Aktivität für jedes Toxin unter Verwendung menschlicher roter Blutkörperchen (siehe Materialtabelle)24.
HINWEIS: Hämolytische Aktivität wird verwendet, weil sie Unterschiede in der Aktivität aufgrund von Reinigung, Mutationen usw. kontrolliert. Die Spezieswahl der Erythrozyten kann die hämolytische Aktivität verändern (z. B. erfordert Intermedilysin menschliche rote Blutkörperchen). - Planen Sie zwei technische Replikate für jede Bedingung, sieben Verdünnungen für die Dosis-Wirkungs-Kurve und eine No-Toxin-Kontrolle.
HINWEIS: Bei Wildtyp-Promastigoten (WT), spt2- und spt2-/+SPT2-Promastigoten können zwei Behandlungen in einer 96-Well-Platte mit V-Boden getestet werden. So konnte beispielsweise die Empfindlichkeit gegenüber Medien verglichen werden (Abbildung 1). Anstelle einer V-Bodenplatte können 1,2-ml-Mikrotiterröhren (siehe Materialtabelle) verwendet werden. Aufgrund der Erfassungszeiten auf dem Zytometer wird nicht empfohlen, mehr als eine Platte gleichzeitig zu laufen. - Bestimmen Sie, welcher Assay-Puffer basierend auf den erforderlichen Testbedingungen und dem Zweck des Experiments verwendet werden soll.
HINWEIS: In diesem Beispiel werden zwei Assay-Puffer verglichen: M199 und Tyrodes Puffer, ergänzt mit dem Lebensfähigkeitsfarbstoff Propidiumiodid (PI). Cholesterin im Serum stört die CDC-Aktivität24. - Berechnen Sie die benötigte Toxinmenge basierend auf den Bedingungen und der Anzahl der behandelten Genotypen. Stellen Sie sicher, dass die 50%ige spezifische Lyse auf halber Höhe der Verdünnungskurve stattfindet.
HINWEIS: Für CDCs ergibt eine zweifache serielle Verdünnung einen guten Bereich für die spätere logistische Modellierung. Für spt2-Promastigoten ist 4.000 HU/ml SLO die empfohlene Ausgangsverdünnung. Wenn inaktive Toxine verwendet werden, kann stattdessen eine Masse verwendet werden, die der höchsten Dosis entspricht. - Planen Sie ein Endvolumen von 200 μL pro Vertiefung und fügen Sie ein kleines Volumen (50-100 μL) hinzu, um Pipettenfehler zu berücksichtigen.
HINWEIS: Bei drei Genotypen, die jeweils doppelt durchgeführt werden, gibt es sechs Proben für die serielle Verdünnung. - Bestimmen Sie die Gesamtmenge des benötigten Toxins mit der folgenden Formel:
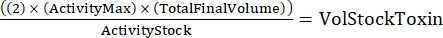 (Gleich 2)
(Gleich 2)
wobei ActivityMax die höchste verwendete Konzentration (HU/ml) ist; TotalFinalVolume ist das Gesamtvolumen (für sechs Proben, 200 x 6 + 100 = 1.300 μL); ActivityStock ist die Aktivität des Toxinvorrats (HU/ml); und VolStockToxin ist die Menge des benötigten Toxinbestands. - Bereiten Sie einen ausreichenden Assay-Puffer vor, der für das Experiment benötigt wird. Ergänzen Sie das Basalmedium mit einem Lebensfähigkeitsfarbstoff und Ca 2+ oder EGTA, die zur Kontrolle des Ca2+ -Spiegels benötigt werden. Wirbel zum Mischen.
HINWEIS: Fügen Sie beispielsweise für jeden 10-ml-Tyrode-Puffer 50 μL 2 mg/ml PI und 200 μL 100 mM CaCl 2 hinzu, was Endkonzentrationen von 10 μg/ml bzw.2 mM ergibt. - Stellen Sie sicher, dass der Lebensfähigkeitsfarbstoff PI nicht mit anderen verwendeten Sonden kollidiert, z. B. für Fluoreszenzbindungstests mit Cy5 oder AlexaFluor647-konjugierten Toxinen20,25.
- Planen Sie Toxinverdünnungen, um eine 2x-Lösung von Toxin herzustellen. 1,5 ml Zentrifugenröhrchen mit Assaypuffer füllen und auf Eis kühlen. Fügen Sie das Toxin nur unmittelbar vor Beginn des Assays hinzu.
HINWEIS: In diesem Beispiel würden 1,3 ml Assay-Puffer für die obere Verdünnung und 650 μL für serielle Verdünnungen hinzugefügt, um die 2x-Toxinlösung herzustellen.
- Experiment
- 0,5 ml verarbeitete Promastigoten aus Schritt 1.8 in einem separaten Röhrchen als "Unstained control" reservieren.
HINWEIS: Diese Probe wird verwendet, um die Ansteuerung auf dem Durchflusszytometer einzurichten. - 2 mg/ml PI zu einer Endkonzentration von 10 μg/ml zu den verbleibenden Promastigoten geben. Wirbel für 3 s.
HINWEIS: Nach der Zugabe von PI zu den verarbeiteten Promastigoten können diese Zellen nur für einen Zeitraum von 2,5 h verwendet werden. Nach 2,5 h beginnen die Zellen zu sterben und die Ergebnisse werden fehlerhaft. - Fügen Sie 1 x 105 (in 100 μL/Well) verarbeitete Promastigoten zu jeder Vertiefung einer V-Bodenplatte mit 96 Vertiefungen oder 1,2-ml-Mikrotiterröhren hinzu (Abbildung 1). Stellen Sie die Platte oder das Rohrgestell in einem Winkel von etwa 45° vor der Betrachtung auf Eis. Führen Sie die Arbeit in einer Biosicherheitswerkbank durch, wenn Sie Leishmania-Promastigoten handhaben.
- Fügen Sie 100 μL Assay-Puffer mit PI zu jeder No-Toxin-Kontrolle hinzu (letzte Reihe). Überprüfen Sie, ob das Steuerelement korrekt hinzugefügt wurde, indem Sie Röhren mit einem Gesamtvolumen von 200 μL, die dunkler erscheinen, visuell identifizieren.
- Entfernen Sie die Toxinaliquots von −80 °C, tauen Sie auf Eis auf und lagern Sie nach Bedarf. Das in Schritt 2.1.5 berechnete Toxinvolumen wird der höchsten Verdünnung (hergestellt in Schritt 2.1.10) zugegeben. Dann verdünnen Sie das Toxin seriell (Abbildung 1). Pipettieren Sie mindestens 8x nach oben und unten, um das Mischen zu gewährleisten.
HINWEIS: Führen Sie die Leistung auf Eis durch, da CDCs bei Raumtemperatur schnell inaktiviert werden. - Ausgehend von der niedrigsten Toxinkonzentration fügen Sie schnell 100 μL Toxin in die richtige Reihe (Abbildung 1 und Abbildung 2) und fahren Sie fort, bis das gesamte Toxin in die Zellen gegeben wurde.
- Verschließen Sie die Platte mit Dichtband. Bei 37 °C 30 min inkubieren. Nach der Inkubationszeit verpacken und transportieren Sie die Platte zum Durchflusszytometer.
- 0,5 ml verarbeitete Promastigoten aus Schritt 1.8 in einem separaten Röhrchen als "Unstained control" reservieren.
- Datenerfassung
- Richten Sie das Durchflusszytometer (siehe Materialtabelle) und die Erfassungssoftware gemäß den Anweisungen des Herstellers und den Richtlinien pro Einrichtung ein. Führen Sie das Durchflusszytometrieverfahren nicht ohne vorherige Schulung auf dem Zytometer durch.
HINWEIS: In diesem Beispiel wurde ein 4-Laser-Attune NxT verwendet. PI wurde auf dem YL-1-Kanal gesammelt (angeregt durch einen 561-nm-Laser, geleitet durch einen 577 LP, reflektiert von 600 DLP und gefiltert durch 585/16-Bandpass), obwohl das breite Spektrum von PI die Erfassung auf anderen Kanälen ermöglicht. - Stellen Sie unter Verwendung der ungefärbten L. major-Promastigotenprobe die Tore für die Vorwärts- und Seitenstreuung und die anfänglichen Fluoreszenzparameter basierend auf den ausgewählten Farbstoffen ein.
- Fügen Sie bei Bedarf einen zusätzlichen Parameter für Punktdiagramme hinzu, um die Autofluoreszenz zu überprüfen (z. B. "keine Färbung") (Abbildung 3).
HINWEIS: In diesem Beispiel wurde der BL-1-Kanal (angeregt durch einen 488-nm-Laser, durch 495 DLP und 503 LP geleitet, von 555 DLP reflektiert und über 530/30-Bandpass gefiltert) verwendet. - Stellen Sie mit einzeln gefärbten Kontrollen die Tore für den Lebensfähigkeitsfarbstoff (PI in dieser Studie) und alle fluoreszenzmarkierten Toxine ein. Überwachen Sie die Vorwärtsstreuung im Vergleich zur Zeit auf Mikroverstopfungen.
HINWEIS: PI färbt alle Zellen über ungefärbten Kontrollen schwach an. Tote Zellen werden leicht trennbar sein, mit vorübergehend permeabilisierten Zellen zwischen den Populationen. - Erfassen Sie >10.000 gesteuerte Ereignisse für jede Probe auf dem Zytometer.
HINWEIS: Es wird empfohlen, von der empfindlichsten zur am wenigsten empfindlichen zu lesen, aber die Reihenfolge der Erfassung kann umgekehrt werden, um die Auswirkungen der Lesereihenfolge auf die Probenergebnisse zu bestimmen. - Speichern Sie die Daten und exportieren Sie sie nach Bedarf für die Analyse.
- Richten Sie das Durchflusszytometer (siehe Materialtabelle) und die Erfassungssoftware gemäß den Anweisungen des Herstellers und den Richtlinien pro Einrichtung ein. Führen Sie das Durchflusszytometrieverfahren nicht ohne vorherige Schulung auf dem Zytometer durch.
- Datenanalyse
Hinweis: In dieser Studie wurde Excel mit Solver-Plug-In (siehe Tabelle der Materialien) wurde für die Datenanalyse verwendet (siehe Ergänzende Datei 1).- Gate insgesamt, einzellige L. major Promastigoten durch Gating auf Vorwärts- und Seitenstreuung und Zeit nach Bedarf (Abbildung 3). Verwenden Sie Höhe oder Fläche wie für das Durchflusszytometer empfohlen.
HINWEIS: Für das hier verwendete Durchflusszytometer ist die Höhe anstelle der Fläche der empfohlene Parameter. - Identifizieren und markieren Sie tote Zellen als "PI high". Gate-Zwischenzellen als "PI low". PI-hohe Zellen sind tote Zellen, während PI-niedrige Zellen vorübergehend permeabilisiert sind26.
HINWEIS: PI-hohe Zellen zeigen typischerweise eine Verschiebung von 2-3 log von negativen Zellen. - Exportieren Sie die Daten nach Excel. Rufen Sie den Probennamen/die Stichproben-ID und %PI high ab, um die Tötung zu bestimmen.
HINWEIS: Wenn fluoreszierende Toxine verwendet wurden, wird die mediane Fluoreszenzintensität (MFI) von lebenden, vorübergehend permeabilisierten und negativen Populationen benötigt. %PI niedrig kann zur transienten Permeabilisierung exportiert werden. - Bestimmen Sie den durchschnittlichen %PI hoch für jede Bedingung zwischen den beiden technischen Replikaten.
HINWEIS: Wenn ein durchschnittlicher MFI für fluoreszierende Toxine benötigt wird, berechnen Sie dies ebenfalls. - Berechnen Sie %Specific Lysis aus dem %PI-Hoch mit der folgenden Formel24,25:
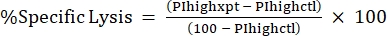 (Gleichung 3)
(Gleichung 3)
wobei PIhighxpt der %PI hoch für die Versuchsbedingung ist; und PIhighctl ist der %PI hoch für die No-Toxin-Kontrolle. - Darstellung der %spezifischen Lyse gegen die Toxinkonzentration für die Dosis-Wirkungs-Kurve (Abbildung 4).
- Organisieren Sie die Dosis-Wirkungs-Kurve in Excel für die logistische Modellierung. Fügen Sie die Toxinkonzentration und die durchschnittliche %spezifische Lyse zusammen mit experimentellen Details und/oder rohen %PI-Hochberechnungen hinzu (Tabelle 2).
- Stellen Sie sicher, dass das Solver-Add-In aktiviert ist.
Hinweis: Um den Solver in der Desktopversion von Excel zu aktivieren, wechseln Sie zu Datei- > Optionen > Add-Ins, und aktivieren Sie das Kontrollkästchen Solver . Starten Sie Excel neu. - Beschriften Sie vier weitere Spalten als "modelliert", "Residuen", "Parameter" und "Parameterwerte". Überprüfen Sie, ob die ersten Spalten den experimentellen Parametern, der Toxinkonzentration und der %spezifischen Lyse entsprechen (Tabelle 2).
- Fügen Sie die folgenden Parameter in die Spalte "Parameter" ein: L, k, c, SUM und LC50. Initialisieren Sie die Parameter L, k und c, indem Sie die folgenden Werte in die Spalte "Parameterwerte" eingeben: 100, 0,05, 1.000.
- Erstellen Sie in der Spalte "modelliert" das Logistikmodell mit der folgenden Formel:
 (Gleich 4)
(Gleich 4)
Legen Sie L, k und c auf die Zellen fest, die diese Parameter in der Spalte "Parameterwerte" enthalten.
Setzen Sie x auf die Zelle, die die Toxinkonzentration enthält.
Hinweis: Für Tabelle 2, Zelle G4, lautet die Formel wie folgt: =$J$3/(1+EXP(-$J$4*(D4-$J$5))) - Wenden Sie diese Gleichung auf alle %Specific Lysis-Werte mit Ausnahme der No-Toxin-Kontrolle an.
- Berechnen Sie in der Spalte "Residuen" das Quadrat der Differenz zwischen der modellierten Zahl und der tatsächlichen spezifischen Lyse mit der folgenden Gleichung:
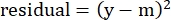 (Gleichung 5)
(Gleichung 5)
wobei y die experimentelle %spezifische Lyse ist; und m ist der entsprechende Wert in der Spalte "modelliert", der in den Schritten 2.4.10-2.4.11 berechnet wurde. - Summieren Sie in der Spalte "Parameterwerte" neben "SUMME" alle Werte in der Spalte Residuen.
- Initialisieren Sie in der Spalte "Parameterwerte" neben "LC50" die Gleichung, um den LC50 aus den ermittelten Werten zu berechnen. Dies löst Gleichung 4 für x , wenn m = 50 ist.
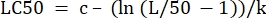 (Gleich 6)
(Gleich 6)
Für Tabelle 2 lautet die Excel-Formel wie folgt: =J5-(LN(J3/(50)-1))/J4 - Öffnen Sie den Solver auf der Registerkarte Daten. Wählen Sie als Ziel festlegen die Zelle aus, die die Summe der berechneten Residuen enthält. Stellen Sie es auf Min.
- Ändern Sie die variablen Zellen für die Parameterwerte L, k und c.
HINWEIS: Der Gleichungslöser verhält sich möglicherweise besser, wenn "Make Unconstrained Variables Non-negative" aktiviert ist. - Für negative k-Werte modifizieren Sie Gleichung 4 und Gleichung 6, indem Sie −1 von k ausschließen, um k in positiv zu ändern. Verwenden Sie die GRG Nichtlineare Lösungsmethode. Klicken Sie auf Lösen.
- Überprüfen Sie die Kurve und ob der LC50 automatisch mit Gleichung 6 berechnet wird (Tabelle 2). Überprüfen Sie die Passung, indem Sie sowohl die %spezifische Lyse als auch die Modellierung gegen die Toxinkonzentration grafisch darstellen.
HINWEIS: Es kann auch überprüft werden, indem dieR2 für die Kurve berechnet wird.
- Gate insgesamt, einzellige L. major Promastigoten durch Gating auf Vorwärts- und Seitenstreuung und Zeit nach Bedarf (Abbildung 3). Verwenden Sie Höhe oder Fläche wie für das Durchflusszytometer empfohlen.
3. Proteinanalyse von toxin-challenged L. major promastigotes
- Bereiten Sie L. major promastigotes wie in Abschnitt 1 beschrieben vor.
- Resuspendieren Sie 2 x 107 WT, spt2- und spt2-/+SPT2 L. major Promastigoten in 2 mL des gewünschten Assaypuffers mit einer 5 mL Pipette (z. B. serumfreies M199). Toxin auf eine endgültige sublytische Konzentration geben und bei 37 °C für 30 min inkubieren.
HINWEIS: Zum Beispiel beträgt eine sublytische Dosis von SLO für spt2-Promastigoten 500 HU / ml. - Schließen Sie andere Genotypen und No-Toxin-Kontrollen ein.
- Zentrifugieren Sie die Promastigoten bei 1.500 x g für 10 min, um die Zellen zu pelletieren. Verwerfen Sie den Überstand mit einer 10-ml-Pipette. Führen Sie das geschlossene Röhrchen mit dem Zellpellet dreimal schnell über eine unregelmäßige Oberfläche, z. B. den Grill der Biosicherheitswerkbank, um das Zellpellet aufzubrechen.
HINWEIS: Das Zellpellet ist für das bloße Auge fast unsichtbar. - 1x SDS-PAGE Probenpuffer mit 2-Mercaptoethanol unmittelbar vor Gebrauch rekonstituieren und 10 min vor Zugabe zum Zellpellet auf 95 °C erhitzen. Resuspendieren Sie das Zellpellet in heißem 1x SDS-PAGE Probenpuffer und mischen Sie gut, indem Sie auf und ab pipettieren. Das resuspendierte Zellpellet in 1x Probenpuffer bei 95 °C für 10 min erhitzen.
HINWEIS: Nach der Solubilisierung im Probenpuffer lagern Sie die Proben bei Bedarf langfristig bei −20 °C. - Bereiten Sie das auflösende Gel vor. Entgasen Sie alle Komponenten außer Ammoniumpersulfat (APS) und TEMED für 15 min.
- Fügen Sie APS und TEMED unmittelbar vor dem Gießen des Gels hinzu. Überlagern Sie das auflösende Gel vorsichtig mit Wasser. Lassen Sie das auflösende Gel polymerisieren, ~30-45 min.
- Dekantieren Sie das Wasser und bereiten Sie das Stapelgel vor. Fügen Sie das Stapelgel hinzu und achten Sie darauf, Blasen zu vermeiden. Führen Sie einen Kamm mit der entsprechenden Anzahl von Vertiefungen ein und lassen Sie ihn 5 min polymerisieren. Überwachen Sie die Polymerisation mit einem übrig gebliebenen Stapelgel.
- Montieren Sie das Gel für den Betrieb von SDS-PAGE und fügen Sie der Kammer einen Reservoirpuffer hinzu.
- Laden Sie 10 μL jeder Probe pro Vertiefung oder 8 μL der Proteinleiter. Laufen Sie bei 180 V, bis die Proben in das auflösende Gel eintreten, und reduzieren Sie dann die Spannung auf ~ 160 V und laufen Sie, bis die Farbstofffront ~ 0,5 cm vom Rand der Platte entfernt ist.
HINWEIS: Die Zeit, die benötigt wird, bis die Proben den Boden erreichen, kann zwischen 1-1,5 h variieren. Die Zeit kann durch Reduzieren der Spannung verlängert werden. Reduzieren Sie die Spannung niemals auf Null. Höhere Spannungen können das Gel "lächelnd" erhöhen und die Platten reißen. - Übertragen Sie das Gel entweder auf die Coomassie-Färbung (zur Proteinfärbung) oder auf 1x Transferpuffer (für Western Blotting). Für die Coomassie-Färbung über Nacht färben und dann entfärben, abbilden und trocknen.
- Für Western Blot bereiten Sie das Transfersystem gemäß den Anweisungen des Herstellers vor.
- Verwenden Sie für einen Nasstransfer kalte 1x Transferpuffer und vornasse Pads, Filterpapier und Nitrozellulose. Für das hier verwendete Bio-rad Protean III System (siehe Materialtabelle) kann das Filterpapier auf 10 cm x 7,5 cm zugeschnitten werden. Schneiden Sie die Nitrocellulose auf 9 cm x 6,75 cm.
HINWEIS: Nitrocellulose ist leicht entzündlich. Vermeiden Sie offene Flammen und andere potenzielle Zündquellen. - Legen Sie die Transferkassette mit Pads, Filterpapier und Nitrozellulose aus. Luftblasen ausrollen. Fügen Sie das Gel vorsichtig hinzu.
- Fügen Sie das Filterpapier hinzu und rollen Sie Luftblasen aus. Fügen Sie das Pad hinzu, schließen Sie die Kassette und legen Sie es in der richtigen Ausrichtung in die Halterung ein (stellen Sie sicher, dass Nitrozellulose dem roten Anschluss zugewandt ist). Fügen Sie einen Rührbalken und einen Eisbeutel zur Seite hinzu und füllen Sie das Reservoir mit kaltem 1x Transferpuffer auf. Transfer bei 110 V für 90 min.
HINWEIS: Die während der Übertragung erzeugte Wärme kann sich nachteilig auf die Übertragung auswirken. Um eine gute Übertragung zu gewährleisten, verwenden Sie immer einen kalten 1x Transferpuffer. - Entfernen Sie die Nitrocellulose und färben Sie sie mit Ponceau-Lösung für ~5 min. Mit Reinstwasser abspülen. Markieren Sie die Proteinleiter mit Bleistift und schneiden Sie den Blot nach Bedarf ab. Defärben Sie den Blot mit dem übrig gebliebenen Transferpuffer.
HINWEIS: Ponceau kann viele Male wiederverwendet werden. - Blockieren Sie die Nitrocellulose in 25 mL 5% BSA in 1x TBST bei 4 °C, mit Schütteln, über Nacht. Dann verwerfen Sie die blockierende Lösung und fügen Sie einen primären Antikörper (1:1.000) in 1% BSA in 1x TBST hinzu. Über Nacht bei 4 °C schütteln.
HINWEIS: Der primäre Antikörper kann bei −20 °C gespeichert und mehrmals wiederverwendet werden. - Waschen Sie die Nitrocellulose 3x für je 10 min in 1x TBST unter Schütteln. Verwerfen Sie die Wäsche und fügen Sie 10 ml HRP-konjugierten sekundären Antikörper (1:10.000) in 1% BSA in 1x TBST hinzu. Bei Raumtemperatur 1 h schütteln. Waschen Sie die Nitrocellulose 3x für je 10 min in 1x TBST unter Schütteln.
- Bereiten Sie das ECL-Reagenz unmittelbar vor der Bildgebung der Nitrocellulose vor. Unmittelbar vor der Bildgebung dekantieren Sie die TBST und fügen Sie das ECL-Reagenz zur Nitrocellulose hinzu. 1 min schütteln. Stellen Sie das Gel vor.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Erhöhte Promastigotenempfindlichkeit gegenüber SLO im Tyrode-Puffer im Vergleich zu M199
Die SLO-Sensitivität von L. major Promastigoten wurde zwischen verschiedenen Assay-Puffern verglichen. Wildtyp-, spt2- und spt2-/+SPT2-Promastigoten wurden vor der Analyse auf einem Durchflusszytometer mit SLO in serumfreiem M199 oder Tyrode-Puffer mit 2 mM CaCl2 für 30 min herausgefordert. Geeignete Parasiten für die Analyse waren einzelne Zellen, ...
Access restricted. Please log in or start a trial to view this content.
Diskussion
In dieser Studie wurden Methoden zur Untersuchung der molekularen Mechanismen und Funktionen von PFTs beschrieben, wobei der menschliche Erreger Leishmania major als Modellsystem verwendet wurde. Es wurde ein auf Durchflusszytometrie basierender Zytotoxizitätstest mit mittlerem Durchsatz zur Messung der Einzelzelllebensfähigkeit entwickelt. Die Lebensfähigkeit ist auf Bevölkerungsebene quantitativ, da LC50-Werte aus der Dosis-Wirkungs-Kurve mittels logistischer Modellierung berechnet werden könne...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Diese Arbeit wurde vom National Institute of Allergy And Infectious Diseases der National Institutes of Health unterstützt, das PAK und KZ (co-I) R01AI139198 an KZ (co-I) gewährt. CH dankt dem Departement für Biowissenschaften für die Lehrassistenz, die während der Zeit dieser Studie zur Verfügung gestellt wurde.
Die Förderorganisationen spielten keine Rolle bei der Konzeption der Studie; bei der Erhebung, Analyse oder Interpretation von Daten; beim Schreiben des Manuskripts; noch in der Entscheidung, die Ergebnisse zu veröffentlichen. Der Inhalt liegt allein in der Verantwortung der Autoren und gibt nicht notwendigerweise die offizielle Meinung der Fördergeber wieder. Die Autoren erklären, dass sie keine konkurrierenden Interessenkonflikte haben.
Danksagungen
Die Autoren danken den Mitgliedern der Keyel- und Zhang-Labore für ihre kritische Durchsicht des Manuskripts. Die Autoren danken dem College of Arts and Sciences Microscopy für die Nutzung der Einrichtungen.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.2 mL microtiter (Marsh) tubes | Fisher | 02-681-376 | Cytotoxicity assay |
| 1.5 mL microcentrifuge tube | Fisher | 05-408-129 | Toxin dilutions |
| 15 mL centrifuge tube | Avantor VWR (Radnor, PA) | 89039-666 | To hold cells and media |
| 1x Phosphate buffered saline (PBS) | Fisher | BP399 | For cell processing |
| 3% H2O2 | Walmart (Fayetteville, AR) | N/A | For ECL |
| 5x M199 | Cell-gro | 11150067 | Basal growth media for L. major promastigotes |
| Biosafety cabinet | Baker | To culture cells in sterile conditions | |
| Bovine serum albumin (BSA) | Fisher | BP1605-100 | Fraction V acceptable purity |
| CaCl2 | Fisher | BP510-100 | Stock concentration 100 mM |
| Centrifuge | Thermo Fisher | Heraeus Megafuge 40R | To pellet the cells from culture |
| Cy5 Mono-reactive dye pack | Cytiva (Marlborough, MA) | PA25031 | Fluorophore label for toxins |
| Digital dry bath | Benchmark | BSH1002 | To denature protein samples |
| EGTA | Amresco | 0732-100G | Stock concentration 0.5 M |
| Excel | Microsoft (Redmond, VA) | Data analysis software | |
| Flow cytometer (4-laser Attune NxT) | Fisher | Cytometer for data acquisition | |
| FlowJo | BD (Ashland, OR) | Software | |
| Formaldehyde | Fisher | BP531-500 | Fixative for counting cells |
| G418 | Fisher | BP673-1 | Selection agent for cells |
| Hellmanex III | Sigma | Z805939 | Dilute 1:4 for cleaning cytometer |
| Hemacytometer | Fisher | 0267151B | For counting cells |
| Human red blood cells | Zen-bio (Durham, NC) | SER-10MLRBC | To validate toxin activity |
| Ice bucket | |||
| Light microscope | Nikon | Eclipse 55i | To visualize cells |
| Nitrocellulose | Fisher | 88018 | For probing proteins via antibodies |
| Pipettors and tips | Avantor VWR | To dispense reagents | |
| Power supply | Bio-Rad | To run SDS-PAGE and transfers | |
| Propidium iodide | Biotium | 40016 | Stock concentration 2 mg/mL in water |
| Protein ladder | Bio-Rad | 161-0373 | To determine molecular weight of proteins |
| SDS-PAGE Running Apparatus (Mini Protean III) | Bio-Rad | 165-3302 | To separate proteins based on their size |
| Sealing tape | R&D | DY992 | To seal plates with cells |
| Streptolysin O C530A plasmid insert | Cloned into pBAD-gIII vector (Reference: 7) | ||
| Streptolysin O C530A toxin | Lab purified | Specific activity 4.34 x 105 HU/mg | |
| Swinging bucket rotor | Thermo Fisher | 75003607 | To centrifuge cells |
| V-bottom plate | Greiner Bio-one | 651206 | For cytotoxicity assay |
| Vortex | Benchmark | BV1000 | To mix cells |
| Western blot imaging system (Chemi-doc) | Bio-Rad | To visualize proteins by western blot | |
| Western Blot Transfer Apparatus (Mini Protean III) | Bio-Rad | 170-3930 | Transfer proteins to nitrocellulose |
| Whatman Filter paper | GE Healthcare Life Sciences | 3030-700 | Used in transfer of proteins to nitrocellulose |
| Antibody | |||
| Anti-ERK antibody | Cell Signaling Technologies | Cat# 9102S | Rabbit (1:1000 dilution) |
| Anti-lipophosphoglycan (LPG) antibody | CreativeBioLabs | Cat# WIC79.3 | Mouse (1: 1000) |
| Anti-MEK antibody | Cell Signaling Technologies | Cat# 9122L | Rabbit (1:1000) |
| Anti-mouse IgG, HRP conjugate | Jackson Immunoresearch | Cat#715-035-151 | Donkey (1:10000) |
| Anti-phosphoERK antibody | Cell Signaling Technologies | Cat# 9101S | Rabbit (1:1000) |
| Anti-pMEK antibody | Cell Signaling Technologies | Cat# 9121S | Rabbit (1:1000) |
| Anti-rabbit IgG, HRP conjugate | Jackson Immunoresearch | Cat#711-035-152 | Donkey (1:10000) |
| Anti-tubulin antibody | Sigma | Cat# T5168 | Mouse (1: 2000) |
| Leishmania major Genotypes | Reference: 13 | ||
| Episomal addback (spt2-/+SPT2) | Δspt2::HYG/Δspt2:PAC/+pXG-SPT2 | ||
| Serine palmitoyltransferase subunit 2 knockout (spt2-) | Δspt2::HYG/Δspt2::PAC | ||
| Wild type (WT) | LV39 clone 5 (Rho/SU/59/P) |
Referenzen
- Thapa, R., Ray, S., Keyel, P. A. Interaction of macrophages and cholesterol-dependent cytolysins: The impact on immune response and cellular survival. Toxins. 12 (9), 531(2020).
- Limbago, B., Penumalli, V., Weinrick, B., Scott, J. R. Role of streptolysin O in a mouse model of invasive group A streptococcal disease. Infection & Immunity. 68 (11), 6384-6390 (2000).
- Farrand, A. J., et al. The cholesterol-dependent cytolysin membrane-binding interface discriminates lipid environments of cholesterol to support beta-barrel pore insertion. Journal of Biological Chemistry. 290 (29), 17733-17744 (2015).
- Soltani, C. E., Hotze, E. M., Johnson, A. E., Tweten, R. K. Structural elements of the cholesterol-dependent cytolysins that are responsible for their cholesterol-sensitive membrane interactions. Proceedings of the National Academy of Sciences of the United States of America. 104 (51), 20226-20231 (2007).
- Schoenauer, R., et al. Down-regulation of acid sphingomyelinase and neutral sphingomyelinase-2 inversely determines the cellular resistance to plasmalemmal injury by pore-forming toxins. FASEB Journal. 33 (1), 275-285 (2019).
- Ray, S., Roth, R., Keyel, P. A. Membrane repair triggered by cholesterol-dependent cytolysins is activated by mixed lineage kinases and MEK. Science Advances. 8 (11), (2022).
- Babiychuk, E. B., Monastyrskaya, K., Draeger, A. Fluorescent annexin A1 reveals dynamics of ceramide platforms in living cells. Traffic. 9 (10), 1757-1775 (2008).
- Jimenez, A. J., et al. ESCRT machinery is required for plasma membrane repair. Science. 343 (6174), 1247136(2014).
- Demonbreun, A. R., et al. An actin-dependent annexin complex mediates plasma membrane repair in muscle. Journal of Cell Biology. 213 (6), 705-718 (2016).
- Wolfmeier, H., et al. Ca(2)(+)-dependent repair of pneumolysin pores: A new paradigm for host cellular defense against bacterial pore-forming toxins. Biochimica et Biophysica Acta. 1853 (2), 2045-2054 (2015).
- Bravo, F., Sanchez, M. R. New and re-emerging cutaneous infectious diseases in Latin America and other geographic areas. Dermatologic Clinics. 21 (4), 655-668 (2003).
- Manfredi, M., Iuliano, S. Cutaneous leishmaniasis with long duration and bleeding ulcer. Clinical Microbiology Open Access. 05, 2-6 (2016).
- Zhang, K., et al. Sphingolipids are essential for differentiation but not growth in Leishmania. EMBO Journal. 22 (22), 6016-6026 (2003).
- Zhang, K. Balancing de novo synthesis and salvage of lipids by Leishmania amastigotes. Current Opinions in Microbiology. 63, 98-103 (2021).
- Kaur, P., Goyal, N. Pathogenic role of mitogen activated protein kinases in protozoan parasites. Biochimie. 193, 78-89 (2022).
- Wiese, M. Leishmania MAP kinases--Familiar proteins in an unusual context. International Journal of Parasitology. 37 (10), 1053-1062 (2007).
- Brumlik, M. J., Pandeswara, S., Ludwig, S. M., Murthy, K., Curiel, T. J. Parasite mitogen-activated protein kinases as drug discovery targets to treat human protozoan pathogens. Journal of Signal Transduction. 2011, 971968(2011).
- Wiese, M., Kuhn, D., Grunfelder, C. G. Protein kinase involved in flagellar-length control. Eukaryotic Cell. 2 (4), 769-777 (2003).
- Agron, P. G., Reed, S. L., Engel, J. N. An essential, putative MEK kinase of Leishmania major. Molecular Biochemistry of Parasitology. 142 (1), 121-125 (2005).
- Ray, S., Thapa, R., Keyel, P. A. Multiple parameters beyond lipid binding affinity drive cytotoxicity of cholesterol-dependent cytolysins. Toxins. 11 (1), (2018).
- Beneke, T., et al. A CRISPR Cas9 high-throughput genome editing toolkit for kinetoplastids. Royal Society Open Science. 4 (5), 170095(2017).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular and Cellular Biology. 10 (3), 1084-1094 (1990).
- Moitra, S., Pawlowic, M. C., Hsu, F. F., Zhang, K. Phosphatidylcholine synthesis through cholinephosphate cytidylyltransferase is dispensable in Leishmania major. Scientific Reports. 9, 7602(2019).
- Keyel, P. A., Heid, M. E., Watkins, S. C., Salter, R. D. Visualization of bacterial toxin induced responses using live cell fluorescence microscopy. Journal of Visualized Experiments. (68), 4227(2012).
- Romero, M., et al. Intrinsic repair protects cells from pore-forming toxins by microvesicle shedding. Cell Death & Differentiation. 24 (5), 798-808 (2017).
- Keyel, P. A., et al. Streptolysin O clearance through sequestration into blebs that bud passively from the plasma membrane. Journal of Cell Science. 124, 2414-2423 (2011).
- Dong, Z., Patel, Y., Saikumar, P., Weinberg, J. M., Venkatachalam, M. A. Development of porous defects in plasma membranes of adenosine triphosphate-depleted Madin-Darby canine kidney cells and its inhibition by glycine. Laboratory Investigations. 78 (6), 657-668 (1998).
- Loomis, W. P., den Hartigh, A. B., Cookson, B. T., Fink, S. L. Diverse small molecules prevent macrophage lysis during pyroptosis. Cell Death & Disease. 10 (4), 326(2019).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten