Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Déchiffrer le mécanisme moléculaire et la fonction des toxines formant des pores à l’aide de Leishmania major
Dans cet article
Résumé
Un protocole utilisant des promastigotes majeurs de Leishmania est présenté ici pour déterminer la liaison, la cytotoxicité et la signalisation induites par les toxines formant des pores. Une preuve de concept avec la streptolysine O est fournie. D’autres toxines peuvent également être utilisées pour tirer parti des mutants génétiques disponibles chez L. major afin de définir de nouveaux mécanismes de résistance aux toxines.
Résumé
Comprendre la fonction et le mécanisme des toxines formant des pores (PFT) est difficile parce que les cellules résistent aux dommages membranaires causés par les PFT. Bien que les approches biophysiques aident à comprendre la formation des pores, elles reposent souvent sur des approches réductionnistes dépourvues du complément complet de lipides et de protéines membranaires. Les cellules humaines cultivées fournissent un système alternatif, mais leur complexité et les redondances dans les mécanismes de réparation rendent difficile l’identification de mécanismes spécifiques. En revanche, le protozoaire humain pathogène responsable de la leishmaniose cutanée, Leishmania major, offre un équilibre optimal entre complexité et pertinence physiologique. L. major est génétiquement traitable et peut être cultivé à haute densité in vitro, et tout impact des perturbations sur l’infection peut être mesuré dans des modèles murins établis. De plus, L. major synthétise des lipides distincts de leurs homologues mammifères, ce qui pourrait modifier la dynamique membranaire. Ces altérations de la dynamique membranaire peuvent être sondées avec des PFT de la famille de toxines la mieux caractérisée, les cytolysines dépendantes du cholestérol (CDC). Les CDC se lient à l’ergostérol dans la membrane de Leishmania et peuvent tuer les promastigotes de L. major, ce qui indique que L. major est un système modèle approprié pour déterminer les mécanismes cellulaires et moléculaires de la fonction PFT. Ce travail décrit les méthodes de test de la fonction PFT chez les promastigotes de L. major, y compris la culture de parasites, les outils génétiques pour évaluer la sensibilité aux lipides, les tests de liaison membranaire et les tests de mort cellulaire. Ces essais permettront l’utilisation rapide de L. major en tant que système modèle puissant pour comprendre la fonction PFT à travers une gamme d’organismes évolutifs divers et de points communs dans l’organisation des lipides.
Introduction
Les toxines formant des pores (PFT) constituent la plus grande famille de toxines bactériennes1, mais les mécanismes par lesquels elles perforent et détruisent les cellules sont mal compris. La famille de toxines formant des pores la mieux étudiée est celle des cytolysines dépendantes du cholestérol (CDC). Les CDC sont principalement synthétisés par des bactéries à Gram positif, y compris l’agent causal de la fasciite nécrosante, Streptococcus pyogenes2. S. pyogenes sécrète la streptolysine O (SLO) du CDC, qui se lie aux stérols dans la membrane plasmique des cellules hôtes sous forme de monomères, oligomérise et insère ~20-30 nm pores dans la membrane1. Le rôle que jouent les lipides dans ce processus reste mal déterminé.
Une approche pour étudier les interactions lipides-CDC est l’utilisation de liposomes chimiquement définis. Bien que les liposomes définis fournissent des informations sur les seuils de lipides nécessaires pour soutenir la liaison aux toxines et la formation de pores3,4, ils ne récapitulent pas complètement les fonctions cellulaires. Par exemple, les liposomes reconstitués n’ont pas l’asymétrie lipidique des hôtes mammifères et les modifications lipidiques en réponse aux toxines5. Une alternative aux liposomes consiste à utiliser des lignées cellulaires de mammifères. Bien que ces lignées cellulaires soient plus pertinentes sur le plan physiologique, il existe un degré élevé de redondance dans les mécanismes de détection des toxines et de résistance2. En conséquence, les voies de réparation utilisées pour résister aux CDC restent mal déterminées. Notamment, l’afflux de Ca2+ est le principal activateur de la réparation membranaire1. En aval de l’afflux de Ca2+, de multiples voies sont engagées, y compris une réparation dépendante des céramides 6,7 et une voie de réparation dépendante de MEK6. Ces voies interagissent avec d’autres effecteurs protéiques, y compris le complexe de tri endosomal nécessaire au transport (ESCRT)8 et les annexines 6,9,10. La dissection de ces voies dans les cellules de mammifères est difficile en raison de la redondance, qui brouille l’interprétation des données.
Une façon d’équilibrer la complexité et la simplicité pour disséquer les voies de réparation est l’utilisation d’organismes plus simples, tels que les pathogènes protozoaires du genre Leishmania. Leishmania sp. cause la leishmaniose chez les humains et d’autres animaux. La leishmaniose va de la leishmaniose cutanée (lésions cutanées spontanément résolutives) à la leishmaniose viscérale mortelle (hépatosplénomégalie), selon l’espèce et d’autres facteurs11. Leishmania major, l’agent causal de la leishmaniose cutanée, est transmis à l’homme via un vecteur phlébotome et est utilisé pour comprendre la fonction et l’infection de Leishmania 12. De plus, Leishmania sp. sont digéniques12. Ils existent sous forme de parasites macrophages mammifères intracellulaires appelés amastigotes et de promastigotes flagellés nageant librement chez le phlébotome12. Les promastigotes de L. major peuvent être cultivés dans des milieux supplémentés en sérum tels que M199 à haute densité13. Les promastigotes sont également génétiquement traitables; Il existe de nombreux gènes knockouts, y compris ceux ciblant les voies de biosynthèse des lipides13. Ces knockouts peuvent être évalués pour la croissance et les différences dans l’infectiosité et le développement de lésions via l’infection de souris Balb / c13.
En plus de la facilité relative de la culture de Leishmania et de la gamme des knockouts de biosynthèse lipidique, le parasite a un génome plus simple que les mammifères. L’espèce de Leishmania la mieux caractérisée est L. major, qui possède de nombreux outils génétiques existants, tels que des mutants avec un métabolisme lipidique défectueux14. Notamment, de nombreuses protéines de réparation sont absentes. L. major n’a pas d’homologues identifiés à ce jour pour les protéines de réparation des mammifères clés telles que les annexines. Cela permet de caractériser les voies de réparation conservées au cours de l’évolution sans la complexité des systèmes de mammifères. Cependant, les voies de réparation n’ont pas été caractérisées à Leishmania à ce jour. Dans le même temps, les principales voies de signalisation impliquées dans la réparation, telles que la voie MEK6, sont conservées dans Leishmania sp.15,16, bien que les homologues doivent être validés. La voie de la protéine kinase activée par les mitogènes (MAPK) est bien étudiée chez L. mexicana, où elle contribue à la survie intracellulaire et à la thermostabilité dans les cellules de mammifères et contrôle la métacyclogenèse16. Chez Leishmania sp., 10 des 15 MAPK ont été caractérisés17. LmMAPK9 et LmMAPK13 devraient être les plus semblables aux mammifères ERK1/2 d’après l’identité dans la séquence labiale de phosphorylation conservée. La séquence de phosphorylation des lèvres est TEY pour les mammifères ERK1/2 et LmMAPK9 et LmMAPK13. Cependant, huit des MAPK de Leishmania ont un motif de phosphorylation TDY15. Au moins deux homologues de MEK ont été identifiés dans Leishmania sp., LmxMKK18 et MEKK-related kinase (MRK1)19. Cela suggère que les idées identifiées dans Leishmania pourraient se traduire dans les systèmes de mammifères. Lorsqu’ils ne se traduisent pas dans les systèmes mammifères, ils représentent des cibles thérapeutiques pour le traitement de la leishmaniose.
Afin d’utiliser les promastigotes de L. major pour étudier la réparation membranaire et les interactions avec les toxines, des techniques à débit moyen sont nécessaires. Bien que l’imagerie de cellules vivantes à haute résolution permette de visualiser les protéines et les membranes marquées en temps réel, elle est à faible débit et peut ne pas mesurer la survie cellulaire. Les tests de viabilité à débit moyen comprennent l’absorption de colorants mesurée par cytométrie en flux, la mesure de l’activité mitochondriale ou la libération de protéines cellulaires comme la lactate déshydrogénase (LDH). Dans les cellules de mammifères, les tests LDH ne mesurent pas quantitativement la mort cellulaire20. De plus, les tests basés sur la population comme la libération de LDH ou l’activité mitochondriale ne permettent pas une analyse monocellulaire ou multiparamétriquerobuste 20. En revanche, les tests basés sur la cytométrie en flux permettent une analyse multiparamétrique d’une seule cellule20. Cependant, ces tests n’ont pas été appliqués à la compréhension de la biologie des toxines ou des réponses aux toxines chez L. major promastigotes.
Dans cette étude, le SLO est utilisé comme outil pour comprendre la perturbation de la membrane plasmique du mutant nul sphingolipide de L. major dans deux tampons différents - le milieu M199 couramment utilisé pour la culture de promastigotes de L. major et le tampon de Tyrode plus simple . Un essai de cytométrie en flux à débit moyen est décrit et utilisé pour générer des courbes dose-réponse de toxines. Les données du test de cytométrie en flux sont modélisées sur une courbe logistique pour déterminer les valeurs CL50 . Avec cette information, une dose sublytique de SLO peut être déterminée afin que les anticorps MAPK puissent être validés par transfert Western.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les lignes directrices appropriées et les pratiques normalisées en matière de microbiologie, d’innocuité et de culture cellulaire ont été utilisées pour l’utilisation et la manipulation de l’agent pathogène du GR2 Leishmania major et de l’ADN recombinant. Toutes les expériences avec L. major vivant ont été réalisées dans une enceinte de biosécurité dans un laboratoire certifié BSL-2. Le travail a été supervisé par le comité institutionnel de biosécurité de l’Université Texas Tech.
REMARQUE : Du point de vue de l’innocuité, les promastigotes L. major vivants sont des agents pathogènes du groupe de risque 2 . Manipuler en utilisant le confinement, les précautions et la surveillance appropriés du Comité institutionnel de biosécurité (CIB). Manipuler les substances toxiques et les produits chimiques conformément aux procédures institutionnelles pour les substances toxiques. Si des toxines recombinantes sont utilisées, l’approbation et la surveillance du GRV peuvent être nécessaires pour les travaux sur l’ADN recombinant.
1. Culture et préparation des promastigotes de L. major
- Obtenir, ou fabriquer et valider, des mutants génétiques majeurs de L. comme décrit précédemment en utilisant soit la recombinaison homologue, soit des méthodes basées sur CRISPR13,21. Utilisez des knockouts complétés par le gène ajouté sur un plasmide pour assurer la spécificité du knockout.
- Culture sauvage de type L. major et promastigotes spt2- à 27 °C en milieu M199 complet. Culture des cellules d’addition épisomiques (spt2-/+SPT2) en M199 complet plus 10 μg/mL de G418 (voir le tableau 1 et le tableau des matériaux).
NOTE: L’ensemble de la configuration expérimentale impliquant des expériences avec des cellules L. major doit être effectué dans une enceinte de biosécurité certifiée BSL2. - Cultiver les promastigotes dans un milieu M199 complet22 jusqu’à ce qu’ils atteignent la phase logarithmique (2-8 x 106 cellules/mL), tel que déterminé par les tests de la courbe de croissance de L. major effectués précédemment23. Prévoyez 1 x 10 5 cellules pour chaque puits pour la cytotoxicité, plus 5 x 105 cellules pour le contrôle de la coloration. Pour le transfert Western, prévoyez 2 x 107 cellules par puits.
NOTE: Effectuer un test de cytotoxicité avec deux répétitions techniques. - Pour vérifier la densité cellulaire appropriée, mélanger une partie aliquote (10-40 μL) de promastigotes avec un volume égal de fixateur (3,7% de paraformaldéhyde dans 1x PBS). Charger 10 μL de l’échantillon fixe de chaque côté de l’hémocytomètre.
ATTENTION : Le formaldéhyde est un produit chimique toxique. Manipuler conformément aux politiques institutionnelles pour les produits chimiques dangereux. - Effectuez le comptage des cellules à l’aide d’un microscope à un grossissement de 20x. Comptez toutes les cellules des 25 petits carrés au centre de l’hémocytomètre. Répétez l’opération pour les carrés des deux côtés et faites la moyenne des comptes.
REMARQUE : Si la variation entre les comptes est de >10, recomptage et moyenne. Si le nombre moyen est de <10 ou >100, modifier la dilution et recompter, puis calculer la densité de culture à l’aide de la formule suivante :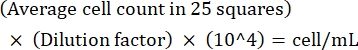 (Éq 1)
(Éq 1)
Par exemple, s’il y a en moyenne 250 Leishmania dans 25 carrés, la densité de culture est de 5 x 106 cellules/mL. - Après le comptage, transférer 5 x 106 cellules dans un tube conique de 15 ml et centrifuger à 1 500 x g pendant 8 minutes à température ambiante pour enduire les cellules.
- Jeter le surnageant à l’aide d’une pipette de 10 ml et vorter brièvement la pastille cellulaire. Ajouter 5 mL de 1x PBS dans le même tube et laver les cellules en retournant doucement 3-6x. Centrifuger à 1 500 x g pendant 8 min à température ambiante pour granuler les cellules.
- Jeter le surnageant à l’aide d’une pipette de 10 mL et remettre en suspension la pastille de 5 x 10 6 cellules dans 5 mL de milieu (p. ex. M199 ou 1x tampon de Tyrode) utilisée pour les expériences avec une pipette de 5 mL pour obtenir une concentration finale de 1 x 106 cellules/mL.
2. Essai de cytotoxicité
- Préparation expérimentale
- Purifiez la toxine comme décrit précédemment24, ou achetez la toxine auprès d’un vendeur. Aliquote dans des aliquotes à usage unique et conserver −80 °C jusqu’à 1 an. Évitez les cycles multiples de gel-dégel.
- Déterminer l’activité hémolytique de chaque toxine à l’aide de globules rouges humains (voir le tableau des matières)24.
REMARQUE: L’activité hémolytique est utilisée car elle contrôle les différences d’activité dues à la purification, aux mutations, etc. Le choix des érythrocytes par espèce peut modifier l’activité hémolytique (p. ex., l’intermédilysine nécessite des globules rouges humains). - Prévoir deux répétitions techniques pour chaque condition, sept dilutions pour la courbe dose-réponse et un contrôle sans toxine.
REMARQUE: Avec les promastigotes de type sauvage (WT), spt2 et spt2-/+SPT2, deux traitements peuvent être testés dans une plaque à fond en V à 96 puits. Par exemple, la sensibilité aux milieux pourrait être comparée (figure 1). Au lieu d’une plaque à fond en V, des tubes de microtitrage de 1,2 mL (voir le tableau des matériaux) peuvent être utilisés. En raison des temps d’acquisition sur le cytomètre, il n’est pas recommandé d’exécuter plus d’une plaque à la fois. - Déterminez quel tampon de dosage utiliser en fonction des conditions de test nécessaires et du but de l’expérience.
REMARQUE : Dans cet exemple, deux tampons de dosage sont comparés : M199 et le tampon de Tyrode complété par le colorant de viabilité iodure de propidium (PI). Le cholestérol dans le sérum interférera avec l’activité CDC24. - Calculer la quantité de toxine nécessaire en fonction des conditions et du nombre de génotypes traités. S’assurer que la lyse spécifique à 50 % se produit à mi-chemin de la courbe de dilution.
NOTE: Pour les CDC, une dilution en série à deux volets donnera une bonne plage pour une modélisation logistique ultérieure. Pour les promastigotes spt2, 4 000 HU/mL SLO est la dilution initiale recommandée. Lorsque des toxines inactives sont utilisées, une masse équivalente à la dose la plus élevée peut être utilisée à la place. - Planifiez un volume final de 200 μL par puits et ajoutez un petit volume supplémentaire (50-100 μL) pour tenir compte de l’erreur de pipette.
NOTE: Avec trois génotypes, chacun fait en double, il y aura six échantillons pour la dilution en série. - Déterminez la quantité totale de toxine nécessaire à l’aide de la formule suivante:
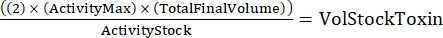 (Éq. 2)
(Éq. 2)
où ActivityMax est la concentration la plus élevée utilisée (HU/mL); TotalFinalVolume est le volume total (pour six échantillons, 200 x 6 + 100 = 1 300 μL); ActivityStock est l’activité du stock de toxines (HU/mL); et VolStockToxin est le volume de toxine nécessaire pour le stock. - Préparez suffisamment de tampon d’essai nécessaire pour l’expérience. Compléter le milieu basal avec un colorant de viabilité et tout Ca 2+ ou EGTA nécessaire pour contrôler les niveaux de Ca2+. Vortex à mélanger.
REMARQUE : Par exemple, pour chaque tampon de Tyrode de 10 mL, ajouter 50 μL de 2 mg/mL PI et 200 μL de CaCl 2 de 100 mM, ce qui donne des concentrations finales de 10 μg/mL et2 mM, respectivement. - S’assurer que le colorant de viabilité PI n’entre pas en conflit avec d’autres sondes utilisées, comme pour les tests de liaison fluorescente utilisant des toxines conjuguées Cy5 ou AlexaFluor64720,25.
- Planifiez les dilutions de toxines pour faire une solution 2x de toxine. Ajouter le tampon d’essai dans des tubes centrifugeux de 1,5 mL et refroidir sur de la glace. N’ajoutez la toxine qu’immédiatement avant de commencer l’essai.
NOTA : Dans cet exemple, 1,3 mL de tampon d’essai serait ajouté pour la dilution par le haut, et 650 μL pour les dilutions en série afin de préparer la solution de toxine 2x.
- Expérience
- Réserver 0,5 mL de promastigotes traités de l’étape 1,8 dans un tube séparé comme « témoin non coloré ».
REMARQUE: Cet échantillon sera utilisé pour configurer le contrôle sur le cytomètre de flux. - Ajouter 2 mg/mL d’IP à une concentration finale de 10 μg/mL aux promastigotes restants. Vortex pendant 3 s.
REMARQUE: Après l’ajout de PI aux promastigotes traités, ces cellules ne peuvent être utilisées que pendant une période de 2,5 h. Après 2,5 heures, les cellules commencent à mourir et les résultats deviennent erronés. - Ajouter 1 x 105 (dans 100 μL/puits) de promastigotes traités à chaque puits d’une plaque de 96 puits à fond en V ou de tubes de microtitrage de 1,2 mL (figure 1). Placez la plaque ou le support à tubes sur de la glace à un angle d’environ 45° par rapport à la vue. Effectuer le travail dans une enceinte de biosécurité lors de la manipulation de Leishmania promastigotes.
- Ajouter 100 μL de tampon de dosage avec PI à chaque contrôle sans toxine (dernière rangée). Vérifiez que le contrôle a été correctement ajouté en identifiant visuellement les tubes d’un volume total de 200 μL qui apparaissent de couleur plus foncée.
- Retirer les aliquotes de toxines à −80 °C, décongeler sur la glace et accumuler au besoin. Ajouter le volume de toxine calculé à l’étape 2.1.5 à la dilution la plus élevée (préparée à l’étape 2.1.10). Ensuite, diluez la toxine en série (Figure 1). Pipeter de haut en bas au moins 8x pour assurer le mélange.
REMARQUE: Effectuer sur la glace parce que les CDC se désactivent rapidement à température ambiante. - En partant de la concentration de toxine la plus faible, ajoutez rapidement 100 μL de toxine à la bonne rangée (Figure 1 et Figure 2) et continuez jusqu’à ce que toute la toxine ait été ajoutée aux cellules.
- Scellez la plaque avec du ruban adhésif. Incuber à 37 °C pendant 30 min. Après la période d’incubation, emballer et transporter la plaque jusqu’au cytomètre en flux.
- Réserver 0,5 mL de promastigotes traités de l’étape 1,8 dans un tube séparé comme « témoin non coloré ».
- Acquisition de données
- Configurez le cytomètre en flux (voir le tableau des matériaux) et le logiciel d’acquisition conformément aux instructions du fabricant et à la politique de l’installation. Ne pas effectuer la procédure de cytométrie en flux sans formation préalable sur le cytomètre.
REMARQUE: Dans cet exemple, un Attune NxT à 4 lasers a été utilisé. PI a été collecté sur le canal YL-1 (excité par un laser 561 nm, passé à travers un 577 LP, réfléchi par 600 DLP et filtré par 585/16 bande passante), bien que le large spectre de PI permette la collecte sur d’autres canaux. - À l’aide de l’échantillon non coloré de L. major promastigote, régler les portes pour la diffusion avant et latérale et les paramètres fluorescents initiaux en fonction des colorants choisis.
- Inclure un paramètre supplémentaire si désiré pour les diagrammes à points afin de vérifier l’autofluorescence (par exemple, « pas de coloration ») (Figure 3).
REMARQUE: Dans cet exemple, le canal BL-1 (excité par un laser de 488 nm, passé à travers 495 DLP et 503 LP, réfléchi à partir de 555 DLP et filtré via 530/30 bande passante) a été utilisé. - À l’aide de témoins à coloration unique, régler les barrières pour le colorant de viabilité (PI dans cette étude) et toutes les toxines marquées par fluorescence. Surveillez la diffusion vers l’avant en fonction du temps pour les micro-sabots.
REMARQUE: PI colorera faiblement toutes les cellules au-dessus des contrôles non colorés. Les cellules mortes seront facilement séparables, avec des cellules perméabilisées transitoirement entre les populations. - Acquérir > 10 000 événements fermés pour chaque échantillon sur le cytomètre.
REMARQUE : Il est recommandé de lire du plus sensible au moins sensible, mais l’ordre d’acquisition peut être inversé pour déterminer l’impact de l’ordre de lecture sur les résultats de l’échantillon. - Enregistrez les données et exportez-les selon vos besoins pour l’analyse.
- Configurez le cytomètre en flux (voir le tableau des matériaux) et le logiciel d’acquisition conformément aux instructions du fabricant et à la politique de l’installation. Ne pas effectuer la procédure de cytométrie en flux sans formation préalable sur le cytomètre.
- Analyse des données
REMARQUE: Dans cette étude, Excel avec le plug-in Solver (voir Tableau des matériaux) a été utilisé pour l’analyse des données (voir Fichier supplémentaire 1).- Promastigotes L . major à une seule cellule par déclenchement sur la diffusion avant et latérale et le temps au besoin (figure 3). Utilisez la hauteur ou la surface recommandée pour le cytomètre en flux.
REMARQUE: Pour le cytomètre de flux utilisé ici, la hauteur est le paramètre recommandé au lieu de la surface. - Identifiez et portez les cellules mortes comme « PI élevé ». Grille des cellules intermédiaires comme « PI faible ». Les cellules PI élevées sont des cellules mortes, tandis que les cellules PI basses sont perméabiliséestransitoirement 26.
REMARQUE: Les cellules PI élevées montrent généralement un décalage logarithmique de 2-3 par rapport aux cellules négatives. - Exportez les données vers Excel. Obtenez le nom/ID de l’échantillon et %PI élevé pour déterminer la mise à mort.
REMARQUE : Si des toxines fluorescentes étaient utilisées, l’intensité fluorescente médiane (IFM) des populations vivantes, perméabilisées transitoirement et négatives serait nécessaire. %PI faible peut être exporté pour la perméabilisation transitoire. - Déterminez le pourcentage d’IP moyen élevé pour chaque condition entre les deux répétitions techniques.
REMARQUE: Si une IMF moyenne est nécessaire pour les toxines fluorescentes, calculez-la également. - Calculez %Specific Lysis à partir du %PI élevé en utilisant la formule suivante24,25:
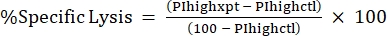 (Éq 3)
(Éq 3)
où PIhighxpt est le %PI élevé pour la condition expérimentale; et PIhighctl est le %PI élevé pour le contrôle sans toxine. - Tracer le pourcentage de lyse spécifique par rapport à la concentration de toxines pour la courbe dose-réponse (figure 4).
- Organiser la courbe dose-réponse dans Excel pour la modélisation logistique. Inclure la concentration de toxines et la moyenne en % de lyse spécifique ainsi que les détails expérimentaux et/ou les calculs bruts du %IP élevé (tableau 2).
- Vérifiez que le complément Solveur est activé.
Remarque : Pour activer le solveur dans la version de bureau d’Excel, accédez à Options de > de fichiers > compléments et cochez la case Solver . Redémarrez Excel. - Étiquetez quatre autres colonnes comme « modélisées », « résidus », « paramètres » et « valeurs des paramètres ». Vérifiez que les premières colonnes correspondent aux paramètres expérimentaux, à la concentration en toxines et au % de lyse spécifique (tableau 2).
- Ajoutez les paramètres suivants dans la colonne « paramètres » : L, k, c, SUM et LC50. Initialisez les paramètres L, k et c en entrant les valeurs suivantes dans la colonne « valeurs des paramètres » : 100, 0,05, 1 000.
- Dans la colonne « modélisé », créez le modèle logistique à l’aide de la formule suivante :
 (Éq. 4)
(Éq. 4)
Définissez L, k et c sur les cellules contenant ces paramètres dans la colonne « valeurs des paramètres ».
Définir x sur la cellule contenant la concentration de toxine.
REMARQUE : Pour le tableau 2, cellule G4, la formule est la suivante : =$J$3/(1+EXP(-$J$4*(D4-$J$5))) - Appliquez cette équation à toutes les valeurs de lyse spécifique à l’exception du contrôle sans toxine.
- Dans la colonne « résidus », calculez le carré de la différence entre le nombre modélisé et la lyse spécifique réelle à l’aide de l’équation suivante :
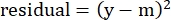 (Éq. 5)
(Éq. 5)
où y est la %Lyse spécifique expérimentale; et m est la valeur correspondante dans la colonne « modélisée » calculée aux étapes 2.4.10-2.4.11. - Dans la colonne « valeurs des paramètres » à côté de « SOMME », additionnez toutes les valeurs de la colonne des résidus.
- Dans la colonne « valeurs des paramètres » à côté de « CL50 », initialisez l’équation pour calculer la CL50 à partir des valeurs déterminées. Il s’agit de résoudre Eq 4 pour x lorsque m = 50.
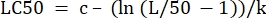 (Éq 6)
(Éq 6)
Pour le tableau 2, la formule Excel est la suivante : =J5-(LN(J3/(50)-1))/J4 - Ouvrez le solveur à partir de l’onglet Données. Sélectionnez l’objectif défini comme étant la cellule contenant la somme des résidus calculés. Réglez-le sur Min.
- Modifiez les cellules variables pour les valeurs de paramètre L, k et c.
Remarque : Le solveur peut se comporter mieux si l’option « Rendre les variables non contraintes non négatives » est laissée cochée. - Pour les valeurs k négatives, modifiez Eq 4 et Eq 6 en factorisant −1 de k pour changer k en positif. Utilisez la méthode de résolution non linéaire GRG. Cliquez sur Résoudre.
- Vérifiez la courbe et assurez-vous que la LC50 est automatiquement calculée à l’aide de l’éq 6 (tableau 2). Vérifiez l’ajustement en traçant graphiquement à la fois le pourcentage de lyse spécifique et la modélisation par rapport à la concentration de toxines.
NOTE: Il peut également être vérifié en calculant le R2 pour la courbe.
- Promastigotes L . major à une seule cellule par déclenchement sur la diffusion avant et latérale et le temps au besoin (figure 3). Utilisez la hauteur ou la surface recommandée pour le cytomètre en flux.
3. Analyse protéique des promastigotes de L. major défiés par les toxines
- Préparer des promastigotes de L. major comme décrit à la rubrique 1.
- Resuspendre 2 promastigotes majeurs de L. 107 WT, spt2 et spt2-/+SPT2 L. dans 2 mL du tampon de dosage désiré à l’aide d’une pipette de 5 mL (p. ex. M199 sans sérum). Ajouter la toxine à une concentration sublytique finale et incuber à 37 °C pendant 30 min.
REMARQUE: Par exemple, une dose sublytique de SLO pour les promastigotes spt2- est de 500 HU / mL. - Inclure d’autres génotypes et des contrôles sans toxines.
- Centrifuger les promastigotes à 1 500 x g pendant 10 min pour granuler les cellules. Jeter le surnageant à l’aide d’une pipette de 10 mL. Faites passer le tube fermé contenant la pastille de cellule rapidement trois fois sur une surface irrégulière, telle que la grille de l’armoire de biosécurité, pour briser la pastille de cellule.
REMARQUE: La pastille de cellule est presque invisible à l’œil nu. - Reconstituer 1x tampon d’échantillon SDS-PAGE avec du 2-mercaptoéthanol immédiatement avant utilisation et chauffer à 95 °C pendant 10 minutes avant l’ajout à la pastille de cellule. Remettez en suspension la pastille de cellule dans un tampon d’échantillon SDS-PAGE 1x chaud et mélangez bien en pipetant de haut en bas. Chauffer la pastille de cellule remise en suspension dans 1x tampon d’échantillon à 95 °C pendant 10 min.
NOTA: Après solubilisation dans un tampon d’échantillonnage, conserver les échantillons à long terme à −20 °C si nécessaire. - Préparez le gel de résolution. Dégazer tous les composants sauf le persulfate d’ammonium (APS) et TEMED pendant 15 min.
- Ajouter APS et TEMED immédiatement avant de couler le gel. Recouvrir soigneusement le gel de résolution avec de l’eau. Laisser le gel de résolution polymériser, ~30-45 min.
- Décanter l’eau et préparer le gel empilable. Ajouter le gel empilable en prenant soin d’éviter les bulles. Insérez un peigne avec le nombre de puits approprié et laissez polymériser pendant 5 min. Surveillez la polymérisation à l’aide de tout gel empilable restant.
- Assemblez le gel pour faire fonctionner SDS-PAGE et ajoutez un tampon de réservoir à la chambre.
- Charge de 10 μL de chaque échantillon par puits, ou 8 μL de l’échelle des protéines. Exécuter à 180 V jusqu’à ce que les échantillons pénètrent dans le gel de résolution, puis réduire la tension à ~160 V et fonctionner jusqu’à ce que le front du colorant soit à ~0,5 cm du bord de la plaque.
NOTE: Le temps nécessaire pour que les échantillons atteignent le fond peut varier entre 1 et 1,5 h. Le temps peut être allongé en réduisant la tension. Ne réduisez jamais la tension à zéro. Des tensions plus élevées peuvent augmenter le gel « souriant » et fissurer les plaques. - Transférer le gel soit sur la coloration de Coomassie (pour la coloration des protéines), soit sur un tampon de transfert 1x (pour le Western blotting). Pour la coloration de Coomassie, colorer pendant la nuit, puis décolorer, imager et sécher.
- Pour le transfert Western, préparer le système de transfert conformément aux instructions du fabricant.
- Pour un transfert humide, utilisez un tampon de transfert froid 1x et des tampons pré-humides, du papier filtre et de la nitrocellulose. Pour le système Bio-rad Protean III (voir tableau des matériaux) utilisé ici, le papier filtre peut être coupé à 10 cm x 7,5 cm. Couper la nitrocellulose à 9 cm x 6,75 cm.
REMARQUE : La nitrocellulose est facilement inflammable. Évitez les flammes nues et les autres sources potentielles d’inflammation. - Disposez la cassette de transfert avec des tampons, du papier filtre et de la nitrocellulose. Déroulez des bulles d’air. Ajouter le gel avec précaution.
- Ajouter le papier filtre et étaler les bulles d’air. Ajouter le tampon, fermer la cassette et l’insérer dans le support dans la bonne orientation (assurez-vous que la nitrocellulose fait face à la borne rouge). Ajouter une barre d’agitation et un sac de glace sur le côté, et compléter le réservoir avec un tampon de transfert froid 1x. Transfert à 110 V pendant 90 min.
REMARQUE: La chaleur générée pendant le transfert peut nuire au transfert. Pour assurer un bon transfert, utilisez toujours un tampon de transfert froid 1x. - Enlever la nitrocellulose et colorer avec la solution de Ponceau pendant ~5 min. Rincer à l’eau ultrapure. Marquez l’échelle de protéines au crayon et coupez la tache au besoin. Détachez le transfert à l’aide du tampon de transfert restant.
REMARQUE : Ponceau peut être réutilisé plusieurs fois. - Bloquer la nitrocellulose dans 25 mL de BSA à 5 % dans 1x TBST à 4 °C, en agitant pendant la nuit. Ensuite, jetez la solution bloquante et ajoutez l’anticorps primaire (1:1 000) dans 1% BSA dans 1x TBST. Agiter à 4 °C pendant la nuit.
REMARQUE : L’anticorps primaire peut être conservé à -20 °C et réutilisé plusieurs fois. - Laver la nitrocellulose 3x pendant 10 min chacune dans 1x TBST en agitant le tas de la nitrocellulose. Jeter le lavage et ajouter 10 mL d’anticorps secondaire conjugué HRP (1:10 000) dans 1% BSA dans 1x TBST. Agiter à température ambiante pendant 1 h. Laver la nitrocellulose 3x pendant 10 min chacune dans 1x TBST en agitant le tas de la nitrocellulose.
- Préparer le réactif ECL immédiatement avant d’imager la nitrocellulose. Immédiatement avant l’imagerie, décanter le TBST et ajouter le réactif ECL à la nitrocellulose. Agiter pendant 1 min. Imaginez le gel.
Access restricted. Please log in or start a trial to view this content.
Résultats
Augmentation de la sensibilité promastigote au SLO dans le tampon de Tyrode par rapport à M199
La sensibilité SLO des promastigotes de L. major a été comparée entre différents tampons d’essai. Les promastigotes de type sauvage, spt2 et spt2-/+SPT2 ont été testés avec SLO dans du M199 sans sérum ou du tampon de Tyrode complété par 2 mM CaCl2 pendant 30 minutes avant l’analyse sur un cytomètre en flux. Les parasites approprié...
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans cette étude, des méthodes pour étudier les mécanismes moléculaires et les fonctions des PFT ont été décrites, en utilisant l’agent pathogène humain Leishmania major comme système modèle. Un test de cytotoxicité basé sur la cytométrie en flux à moyen débit pour mesurer la viabilité d’une cellule unique a été mis au point. La viabilité est quantitative au niveau de la population parce que les valeurs de CL50 peuvent être calculées à partir de la courbe dose-réponse à l?...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Ce travail a été soutenu par l’Institut national des allergies et des maladies infectieuses des National Institutes of Health subvention R21AI156225 à PAK et KZ (co-I) et R01AI139198 à KZ (co-I). CH tient à remercier le Département des sciences biologiques pour l’assistanat d’enseignement fourni pendant la durée de cette étude.
Les organismes de financement n’ont joué aucun rôle dans la conception de l’étude; dans la collecte, l’analyse ou l’interprétation des données; dans la rédaction du manuscrit; ni dans la décision de publier les résultats. Le contenu relève de la seule responsabilité des auteurs et ne représente pas nécessairement les opinions officielles des organismes de financement. Les auteurs déclarent qu’ils n’ont pas de conflits d’intérêts concurrents.
Remerciements
Les auteurs tiennent à remercier les membres des laboratoires Keyel et Zhang pour leur examen critique du manuscrit. Les auteurs remercient le College of Arts and Sciences Microscopy pour l’utilisation des installations.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 1.2 mL microtiter (Marsh) tubes | Fisher | 02-681-376 | Cytotoxicity assay |
| 1.5 mL microcentrifuge tube | Fisher | 05-408-129 | Toxin dilutions |
| 15 mL centrifuge tube | Avantor VWR (Radnor, PA) | 89039-666 | To hold cells and media |
| 1x Phosphate buffered saline (PBS) | Fisher | BP399 | For cell processing |
| 3% H2O2 | Walmart (Fayetteville, AR) | N/A | For ECL |
| 5x M199 | Cell-gro | 11150067 | Basal growth media for L. major promastigotes |
| Biosafety cabinet | Baker | To culture cells in sterile conditions | |
| Bovine serum albumin (BSA) | Fisher | BP1605-100 | Fraction V acceptable purity |
| CaCl2 | Fisher | BP510-100 | Stock concentration 100 mM |
| Centrifuge | Thermo Fisher | Heraeus Megafuge 40R | To pellet the cells from culture |
| Cy5 Mono-reactive dye pack | Cytiva (Marlborough, MA) | PA25031 | Fluorophore label for toxins |
| Digital dry bath | Benchmark | BSH1002 | To denature protein samples |
| EGTA | Amresco | 0732-100G | Stock concentration 0.5 M |
| Excel | Microsoft (Redmond, VA) | Data analysis software | |
| Flow cytometer (4-laser Attune NxT) | Fisher | Cytometer for data acquisition | |
| FlowJo | BD (Ashland, OR) | Software | |
| Formaldehyde | Fisher | BP531-500 | Fixative for counting cells |
| G418 | Fisher | BP673-1 | Selection agent for cells |
| Hellmanex III | Sigma | Z805939 | Dilute 1:4 for cleaning cytometer |
| Hemacytometer | Fisher | 0267151B | For counting cells |
| Human red blood cells | Zen-bio (Durham, NC) | SER-10MLRBC | To validate toxin activity |
| Ice bucket | |||
| Light microscope | Nikon | Eclipse 55i | To visualize cells |
| Nitrocellulose | Fisher | 88018 | For probing proteins via antibodies |
| Pipettors and tips | Avantor VWR | To dispense reagents | |
| Power supply | Bio-Rad | To run SDS-PAGE and transfers | |
| Propidium iodide | Biotium | 40016 | Stock concentration 2 mg/mL in water |
| Protein ladder | Bio-Rad | 161-0373 | To determine molecular weight of proteins |
| SDS-PAGE Running Apparatus (Mini Protean III) | Bio-Rad | 165-3302 | To separate proteins based on their size |
| Sealing tape | R&D | DY992 | To seal plates with cells |
| Streptolysin O C530A plasmid insert | Cloned into pBAD-gIII vector (Reference: 7) | ||
| Streptolysin O C530A toxin | Lab purified | Specific activity 4.34 x 105 HU/mg | |
| Swinging bucket rotor | Thermo Fisher | 75003607 | To centrifuge cells |
| V-bottom plate | Greiner Bio-one | 651206 | For cytotoxicity assay |
| Vortex | Benchmark | BV1000 | To mix cells |
| Western blot imaging system (Chemi-doc) | Bio-Rad | To visualize proteins by western blot | |
| Western Blot Transfer Apparatus (Mini Protean III) | Bio-Rad | 170-3930 | Transfer proteins to nitrocellulose |
| Whatman Filter paper | GE Healthcare Life Sciences | 3030-700 | Used in transfer of proteins to nitrocellulose |
| Antibody | |||
| Anti-ERK antibody | Cell Signaling Technologies | Cat# 9102S | Rabbit (1:1000 dilution) |
| Anti-lipophosphoglycan (LPG) antibody | CreativeBioLabs | Cat# WIC79.3 | Mouse (1: 1000) |
| Anti-MEK antibody | Cell Signaling Technologies | Cat# 9122L | Rabbit (1:1000) |
| Anti-mouse IgG, HRP conjugate | Jackson Immunoresearch | Cat#715-035-151 | Donkey (1:10000) |
| Anti-phosphoERK antibody | Cell Signaling Technologies | Cat# 9101S | Rabbit (1:1000) |
| Anti-pMEK antibody | Cell Signaling Technologies | Cat# 9121S | Rabbit (1:1000) |
| Anti-rabbit IgG, HRP conjugate | Jackson Immunoresearch | Cat#711-035-152 | Donkey (1:10000) |
| Anti-tubulin antibody | Sigma | Cat# T5168 | Mouse (1: 2000) |
| Leishmania major Genotypes | Reference: 13 | ||
| Episomal addback (spt2-/+SPT2) | Δspt2::HYG/Δspt2:PAC/+pXG-SPT2 | ||
| Serine palmitoyltransferase subunit 2 knockout (spt2-) | Δspt2::HYG/Δspt2::PAC | ||
| Wild type (WT) | LV39 clone 5 (Rho/SU/59/P) |
Références
- Thapa, R., Ray, S., Keyel, P. A. Interaction of macrophages and cholesterol-dependent cytolysins: The impact on immune response and cellular survival. Toxins. 12 (9), 531(2020).
- Limbago, B., Penumalli, V., Weinrick, B., Scott, J. R. Role of streptolysin O in a mouse model of invasive group A streptococcal disease. Infection & Immunity. 68 (11), 6384-6390 (2000).
- Farrand, A. J., et al. The cholesterol-dependent cytolysin membrane-binding interface discriminates lipid environments of cholesterol to support beta-barrel pore insertion. Journal of Biological Chemistry. 290 (29), 17733-17744 (2015).
- Soltani, C. E., Hotze, E. M., Johnson, A. E., Tweten, R. K. Structural elements of the cholesterol-dependent cytolysins that are responsible for their cholesterol-sensitive membrane interactions. Proceedings of the National Academy of Sciences of the United States of America. 104 (51), 20226-20231 (2007).
- Schoenauer, R., et al. Down-regulation of acid sphingomyelinase and neutral sphingomyelinase-2 inversely determines the cellular resistance to plasmalemmal injury by pore-forming toxins. FASEB Journal. 33 (1), 275-285 (2019).
- Ray, S., Roth, R., Keyel, P. A. Membrane repair triggered by cholesterol-dependent cytolysins is activated by mixed lineage kinases and MEK. Science Advances. 8 (11), (2022).
- Babiychuk, E. B., Monastyrskaya, K., Draeger, A. Fluorescent annexin A1 reveals dynamics of ceramide platforms in living cells. Traffic. 9 (10), 1757-1775 (2008).
- Jimenez, A. J., et al. ESCRT machinery is required for plasma membrane repair. Science. 343 (6174), 1247136(2014).
- Demonbreun, A. R., et al. An actin-dependent annexin complex mediates plasma membrane repair in muscle. Journal of Cell Biology. 213 (6), 705-718 (2016).
- Wolfmeier, H., et al. Ca(2)(+)-dependent repair of pneumolysin pores: A new paradigm for host cellular defense against bacterial pore-forming toxins. Biochimica et Biophysica Acta. 1853 (2), 2045-2054 (2015).
- Bravo, F., Sanchez, M. R. New and re-emerging cutaneous infectious diseases in Latin America and other geographic areas. Dermatologic Clinics. 21 (4), 655-668 (2003).
- Manfredi, M., Iuliano, S. Cutaneous leishmaniasis with long duration and bleeding ulcer. Clinical Microbiology Open Access. 05, 2-6 (2016).
- Zhang, K., et al. Sphingolipids are essential for differentiation but not growth in Leishmania. EMBO Journal. 22 (22), 6016-6026 (2003).
- Zhang, K. Balancing de novo synthesis and salvage of lipids by Leishmania amastigotes. Current Opinions in Microbiology. 63, 98-103 (2021).
- Kaur, P., Goyal, N. Pathogenic role of mitogen activated protein kinases in protozoan parasites. Biochimie. 193, 78-89 (2022).
- Wiese, M. Leishmania MAP kinases--Familiar proteins in an unusual context. International Journal of Parasitology. 37 (10), 1053-1062 (2007).
- Brumlik, M. J., Pandeswara, S., Ludwig, S. M., Murthy, K., Curiel, T. J. Parasite mitogen-activated protein kinases as drug discovery targets to treat human protozoan pathogens. Journal of Signal Transduction. 2011, 971968(2011).
- Wiese, M., Kuhn, D., Grunfelder, C. G. Protein kinase involved in flagellar-length control. Eukaryotic Cell. 2 (4), 769-777 (2003).
- Agron, P. G., Reed, S. L., Engel, J. N. An essential, putative MEK kinase of Leishmania major. Molecular Biochemistry of Parasitology. 142 (1), 121-125 (2005).
- Ray, S., Thapa, R., Keyel, P. A. Multiple parameters beyond lipid binding affinity drive cytotoxicity of cholesterol-dependent cytolysins. Toxins. 11 (1), (2018).
- Beneke, T., et al. A CRISPR Cas9 high-throughput genome editing toolkit for kinetoplastids. Royal Society Open Science. 4 (5), 170095(2017).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular and Cellular Biology. 10 (3), 1084-1094 (1990).
- Moitra, S., Pawlowic, M. C., Hsu, F. F., Zhang, K. Phosphatidylcholine synthesis through cholinephosphate cytidylyltransferase is dispensable in Leishmania major. Scientific Reports. 9, 7602(2019).
- Keyel, P. A., Heid, M. E., Watkins, S. C., Salter, R. D. Visualization of bacterial toxin induced responses using live cell fluorescence microscopy. Journal of Visualized Experiments. (68), 4227(2012).
- Romero, M., et al. Intrinsic repair protects cells from pore-forming toxins by microvesicle shedding. Cell Death & Differentiation. 24 (5), 798-808 (2017).
- Keyel, P. A., et al. Streptolysin O clearance through sequestration into blebs that bud passively from the plasma membrane. Journal of Cell Science. 124, 2414-2423 (2011).
- Dong, Z., Patel, Y., Saikumar, P., Weinberg, J. M., Venkatachalam, M. A. Development of porous defects in plasma membranes of adenosine triphosphate-depleted Madin-Darby canine kidney cells and its inhibition by glycine. Laboratory Investigations. 78 (6), 657-668 (1998).
- Loomis, W. P., den Hartigh, A. B., Cookson, B. T., Fink, S. L. Diverse small molecules prevent macrophage lysis during pyroptosis. Cell Death & Disease. 10 (4), 326(2019).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon