Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación de los niveles de autofagia en dos modelos diferentes de células pancreáticas utilizando inmunofluorescencia LC3
En este artículo
Resumen
El objetivo de este protocolo es determinar los niveles autofágicos en el cáncer de páncreas y las células acinares pancreáticas a través de la inmunofluorescencia LC3 y la cuantificación de puntos LC3.
Resumen
La autofagia es un proceso catabólico especializado que degrada selectivamente los componentes citoplasmáticos, incluidas las proteínas y los orgánulos dañados. La autofagia permite a las células responder fisiológicamente a los estímulos de estrés y, por lo tanto, mantener la homeostasis celular. Las células cancerosas pueden modular sus niveles de autofagia para adaptarse a condiciones adversas como hipoxia, deficiencia de nutrientes o daño causado por la quimioterapia. El adenocarcinoma pancreático ductal es uno de los tipos de cáncer más mortales. Las células de cáncer de páncreas tienen una alta actividad de autofagia debido a la regulación transcripcional positiva y la activación postraduccional de las proteínas de autofagia.
Aquí, la línea celular PANC-1 se utilizó como modelo de células de cáncer de páncreas humano, y la línea celular acinar pancreática AR42J se utilizó como modelo fisiológico de células de mamíferos altamente diferenciadas. Este estudio utilizó la inmunofluorescencia de la cadena ligera 3 de la proteína asociada a microtúbulos (CL3) como un indicador del estado de activación de la autofagia. La CL3 es una proteína de autofagia que, en condiciones basales, muestra un patrón difuso de distribución en el citoplasma (conocido como LC3-I en esta condición). La inducción de autofagia desencadena la conjugación de CL3 a fosfatidiletanolamina en la superficie de los autofagosomas recién formados para formar LC3-II, una proteína unida a la membrana que ayuda en la formación y expansión de autofagosomas. Para cuantificar el número de estructuras autofágicas etiquetadas, se utilizó el software de código abierto FIJI con la ayuda de la herramienta "3D Objects Counter".
La medida de los niveles autofágicos tanto en condiciones fisiológicas como en células cancerosas nos permite estudiar la modulación de la autofagia en diversas condiciones como la hipoxia, el tratamiento de quimioterapia o el derribo de ciertas proteínas.
Introducción
La macroautofagia (comúnmente conocida como autofagia) es un proceso catabólico especializado que degrada selectivamente los componentes citoplasmáticos, incluidas las proteínas y los orgánulos dañados 1,2. La autofagia permite a las células responder fisiológicamente a los estímulos de estrés y, así, mantener la homeostasis celular3. Durante la autofagia, se forma una vesícula de doble membrana: el autofagosoma. El autofagosoma contiene las moléculas de carga y las conduce al lisosoma para su degradación 1,4.
Los autofagosomas están decorados por la proteína autofágica de la cadena ligera 3 (CL3)5. Cuando no se induce la autofagia, la CL3 se difunde en el citoplasma y el núcleo en la conformación LC3-I. Por otro lado, cuando se induce la autofagia, la CL3 se conjuga con una fosfatidiletanolamina en la membrana de las estructuras autofágicas6. Esta nueva conformación LC3 se conoce como LC3-II1. El cambio de conformación de LC3 provoca cambios en su localización celular y su migración de electroforesis en gel de dodecil sulfato de sodio-poliacrilamida (SDS-PAGE), que puede ser detectada por técnicas como la inmunofluorescencia y el western blot 5,7. De esta manera, la conjugación de CL3 es un evento clave en el proceso autofágico que se puede utilizar para medir la actividad autofágica.
La célula acinar pancreática es una célula altamente diferenciada que, en condiciones saludables, tiene una baja tasa de autofagia. Sin embargo, en diferentes condiciones fisiológicas o bajo estimulación farmacológica, pueden activar la autofagia. Por lo tanto, la determinación de los niveles autofágicos en esta línea celular es útil para estudiar los posibles efectos directos o indirectos de diferentes agentes farmacológicos o biológicos sobre la autofagia 8,9.
El adenocarcinoma pancreático ductal es uno de los tipos de cáncer más mortales, dado su diagnóstico tardío y su alta resistencia a la quimioterapia10. Las células de cáncer de páncreas tienen una alta actividad de autofagia debido a la regulación transcripcional positiva y la activación postraduccional de proteínas relacionadas con la autofagia11. Las células de cáncer de páncreas pueden ajustar sus niveles de autofagia en respuesta a condiciones desfavorables como hipoxia, privación de nutrientes o daño inducido por la quimioterapia11. Por lo tanto, analizar los niveles de autofagia en las células de cáncer de páncreas puede ayudar a comprender cómo se adaptan a diferentes entornos y evaluar la efectividad de los tratamientos moduladores de la autofagia.
Este estudio muestra un método para realizar la inmunofluorescencia de LC3 en dos modelos celulares pancreáticos distintos. El primer modelo, las células PANC-1, sirvió como modelo para el adenocarcinoma ductal pancreático. Estas células fueron tratadas con gemcitabina, un agente quimioterápico que previamente ha demostrado inducir autofagia, específicamente en células de cáncer de páncreas portadoras del gen oncogénico del virus del sarcoma de rata Kirsten (KRAS)12,13. El segundo modelo, las células AR42J, sirvió como un modelo más fisiológico de las células pancreáticas exocrinas. Estas células fueron diferenciadas con dexametasona para volverse más similares a las células pancreáticas acinares14. En estas células, la autofagia fue inducida farmacológicamente mediante el uso de PP242, que es un potente inhibidor de mTOR15. En este estudio, demostramos la aplicabilidad del protocolo descrito con dos modelos pancreáticos diferentes y su capacidad para discriminar entre estados de baja y alta autofagia.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparación celular
- Remoje los cubreobjetos redondos de 12 mm en etanol absoluto y colóquelos verticalmente en los pocillos de una placa de 24 pocillos.
- Retire la cubierta y exponga la placa de pocillos múltiples a la radiación ultravioleta durante 15 minutos.
- Coloque los cubreobjetos horizontalmente y lávelos con el medio de águila modificado (DMEM) de Dulbecco.
- Sembrar un número bajo de células pancreáticas. La cantidad debe ajustarse para obtener una confluencia del 50%-75% el día de la fijación16.
NOTA: Se recomienda sembrar 2.5 × 10 4 PANC-1 o 4 × 104 células AR42J por pocillo para fijar las células después de 3 días. - Cultivar las células en DMEM que contiene 10% de suero bovino fetal, 100 U/ml de penicilina y 100 μg/ml de estreptomicina en una incubadora a 37 °C bajo una atmósfera humidificada con 5% de dióxido de carbono (CO2).
NOTA: Para las células PANC-1, se recomienda incubar las células durante 2 días entre la siembra celular y los siguientes pasos. Después de este tiempo, las células pueden ser transfectadas, tratadas o fijadas. Este protocolo ejemplifica el tratamiento con gemcitabina en células PANC-1 no transfectadas y la diferenciación y el tratamiento PP242 para células AR42J no transfectadas.
2. Tratamiento de las células
- Tratamiento con gemcitabina para las células PANC-1
- Preparar una solución de gemcitabina de 1 μg/μL en DMEM 2 días después de la siembra. Tratar cada pocillo con 2,6 μL de la solución de gemcitabina de 1 μg/μL para lograr una dilución final de 20 μM.
- Incubar las células durante 24 h en la incubadora.
- Diferenciación AR42J y tratamiento PP242
- Preparar una solución de 4 μg/ml de dexametasona en DMEM.
- Tratar cada pocillo con 4,9 μL de solución de dexametasona de 4 μg/ml para obtener una dilución final de 100 nM.
- Incubar las células durante 48 h en la incubadora.
- Retirar el medio, y tratar cada pocillo con 0,5 μL de 1 mM PP242 para obtener una dilución final de 1 μM.
- Incubar las células durante 2 h en la incubadora.
3. Fijación y permeabilización de las células
- Prepare una placa de 24 pocillos con metanol frío y una placa de 6 pocillos con solución salina tamponada con fosfato frío (PBS; 137 mM NaCl, 2,7 mM KCl, 8 mM Na 2 HPO 4,2 mM KH2PO4). Manténgalos en hielo.
- Tome cada cubreobjetos con pinzas, lávelos dos veces en PBS e incube durante 6 minutos en metanol.
4. Bloqueo de las células
- Lave cada cubreobjetos dos veces en PBS e incube durante 1 h en suero bovino fetal al 10% en PBS (solución de bloqueo).
Nota : en este paso, el protocolo podría estar en pausa. Los cubreobjetos se pueden almacenar durante la noche en la nevera en la solución de bloqueo, y el protocolo se puede continuar al día siguiente.
5. Incubación de los cubobjetos con el anticuerpo primario
- Prepare una solución 1:1.000 de anti-LC3 en la solución de bloqueo y manténgala en hielo.
- Coloque un trozo de película de sellado de laboratorio sobre la tapa de varios pocillos.
- Coloque una gota (25 μL) por cubreobjetos de solución anti-LC3 sobre la película de sellado.
- Tome cada cubreobjetos con pinzas y colóquelo sobre la gota de anticuerpos primarios, teniendo cuidado de que el lado celular esté en contacto con la solución.
- Prepare una cámara húmeda colocando un pedazo de papel húmedo en una caja de plástico de fondo plano.
- Coloque la placa de pocillos múltiples en la cámara de humedad, cúbrala con papel de aluminio e incube durante la noche en el refrigerador.
6. Incubación de los cubobjetos con el anticuerpo secundario
- Retire la placa de pocillos múltiples de la cámara de humedad y vuelva a colocar los cubreobjetos en la placa de pocillos múltiples.
- Realice tres lavados con PBS.
- Prepare una solución de anticonejo marcado con fluorescencia con una dilución de 1:800 en la solución de bloqueo y manténgala en hielo protegido de la luz.
- Coloque una pieza de película de sellado sobre la tapa de varios pocillos.
- Coloque una gota (25 μL) por cubreobjetos de solución anticonejo sobre la película de sellado.
- Tome cada cubreobjetos con pinzas y colóquelo sobre la gota de anticuerpos primarios, teniendo cuidado de que el lado celular esté en contacto con la solución.
- Incubar la placa multipocillo en la cámara de humedad durante 2 h a temperatura ambiente (RT) protegida de la luz.
7. Teñir las células con 4′,6-diamidino-2-fenilindol (DAPI)
- Retire la placa de pocillos múltiples de la cámara de humedad y vuelva a colocar los cubreobjetos en la placa de pocillos múltiples.
- Realice tres lavados con PBS.
- Prepare una solución de 300 nM de DAPI en PBS (protegido de la luz).
- Incubar cada cubador con la solución DAPI durante 10 minutos.
- Realice tres lavados con PBS. Mantenga la placa de pocillos múltiples protegida de la luz.
8. Montaje
- Prepara dos vasos de precipitados con agua y un pedazo de papel.
- Colocar una gota (10 μL) por cubreobjetos de una solución de aducto de polivinílico alcohol-Bis(trimetilaluminio)-1,4-diazabiciclo[2.2.2]octano (PVA-DABCO) sobre un portaobjetos.
NOTA: PVA-DABCO se prepara combinando 0,25 M DABCO, 10% p/v PVA, 20% de glicerol y 50% de Tris HCl (1,5 M, pH 8,8) en agua ultrapura. - Tome cada cubreobjetos con pinzas, lávelos en cada vaso de precipitados de agua, séquelo en el papel y colóquelo sobre la gota de PVA-DABCO (con las células en contacto con la solución).
- Dejar secar toda la noche, protegido de la luz.
9. Visualización por microscopía confocal y captura de imágenes
- Visualice los cubreobjetos en un microscopio confocal invertido utilizando un objetivo de alrededor de 63x17.
- Capture imágenes representativas de las celdas etiquetadas.
10. Cuantificación de los puntos LC3
- Arrastre y suelte cada archivo de imagen que contenga los canales capturados, como ".czi", en la pantalla ImageJ (FIJI) para abrirlo. Haga clic en Aceptar en el cuadro de diálogo y cierre la ventana Consola .
- En la pestaña Imagen , seleccione Color > Canales divididos.
- Cierre las imágenes correspondientes a los canales que no sean la imagen LC3.
- En la pestaña Imagen , seleccione Ajustar > balance de color
- Mueva el control deslizante Máximo hacia la izquierda hasta que la imagen esté saturada para visualizar los contornos de las celdas.
- Dibuje el contorno de la celda con la herramienta Selección a mano alzada .
- Haga clic en el botón Restablecer para restablecer el ajuste de color.
- En la ficha Editar , seleccione Cortar para cortar el elemento seleccionado.
- Cierre la imagen sin guardarla.
- En la pestaña Editar , seleccione Pegar.
- En el menú Analizar , elija la herramienta Contador de objetos 3D.
- Establezca el umbral. En el ejemplo proporcionado en este estudio, el umbral se establece en 2.000.
- Establezca el filtro de tamaño. En este estudio, se establece entre 50 y 500.
- Asegúrese de que las casillas Objetos y Resumen estén marcadas.
- Haga clic en Aceptar. El número de puntos se describirá como Objetos detectados en el Resumen.
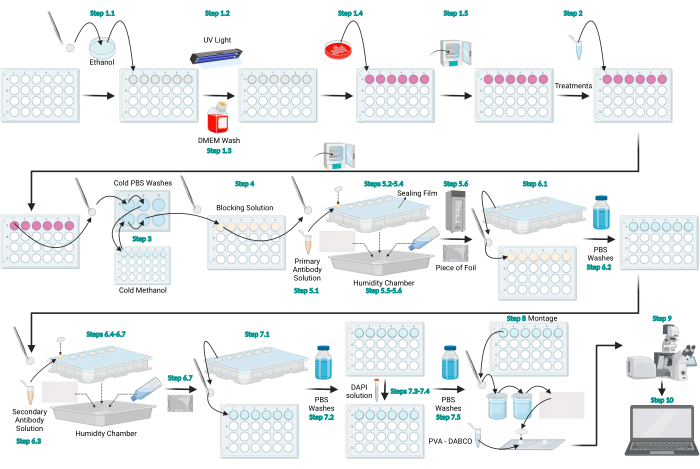
Figura 1: Diagrama esquemático del protocolo de inmunofluorescencia LC3. Diagrama esquemático que representa el protocolo general proporcionado para la inmunofluorescencia LC3. Figura creada con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Este protocolo realiza inmunofluorescencia de CL3 en líneas celulares pancreáticas para determinar los niveles de autofagia en diferentes condiciones. El resultado de este experimento fue la obtención de imágenes celulares de los canales rojo y azul, correspondientes a LC3 y DAPI. Las imágenes de LC3 indican la distribución celular de esta proteína, mientras que la DAPI muestra la localización nuclear. La Figura 2A muestra una imagen representativa de la inmunofluorescencia de LC3 y ...
Access restricted. Please log in or start a trial to view this content.
Discusión
El método descrito en este protocolo permite visualizar la distribución endógena de CL3 en la célula y cuantificar los niveles autofágicos en diferentes condiciones. Otro método similar utilizado para analizar la distribución de CL3 y determinar la activación de la autofagia implica la transfección de LC3 marcada con fluorescencia (como RFP-LC3)19. La transfección RFP-LC3 tiene las ventajas de no necesitar fijación (lo que permite aplicar este método en imágenes de células vivas
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
No se declararon conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por becas de la Universidad de Buenos Aires (UBACyT 2018-2020 20020170100082BA), el Consejo Nacional de Investigación Científica y Tecnológica (CONICET) (PIP 2021-2023 GI− 11220200101549CO; y PUE 22920170100033) y la Agencia Nacional de Promoción Científica y Tecnológica (PICT 2019-01664).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 10x Phosphate-Buffered Saline (PBS) | Corning | 46-013-CM | |
| 12 mm round coverslips | HDA | CBR_OBJ_6467 | |
| 24 Well- Cell Culture Plate | Sorfa | 220300 | |
| Absolute ethanol | Biopack | 2207.10.00 | |
| Alexa Fluor 594 Donkey anti-rabbit IgG (H+L) | Invitrogen | R37119 | |
| Confocal Laser Scanning Microscope | Zeiss | LSM 800 | |
| Dexamethasone | Sigma Aldrich | D4902 | |
| DMEN | Sartorius | 01-052-1A | |
| Fetal Bovine Serum | NATOCOR | Lintc-634 | |
| Gemcitabina | Eli Lilly | VL7502 | |
| LC3B (D11) XP Rabbit mAb | Cell Signaling Technology | 3868S | |
| Methanol | Anedra | 6197 | |
| Parafilm "M" (Laboratory Sealing Film) | Bemis/Curwood | PM-996 | |
| Pen-Strep Solution | Sartorius | 03-031-1B | |
| PP242 | Santa Cruz Biotechnology | SC-301606 | |
| Trypsin EDTA | Gibco | 11570626 |
Referencias
- Grasso, D., Renna, F. J., Vaccaro, M. I. Initial steps in mammalian autophagosome biogenesis. Frontiers in Cell and Developmental Biology. 6, 146(2018).
- Galluzzi, L., et al. Molecular definitions of autophagy and related processes. EMBO Journal. 36 (13), 1811-1836 (2017).
- Kitada, M., Koya, D. Autophagy in metabolic disease and ageing. Nature Reviews Endocrinology. 17 (11), 647-661 (2021).
- Ktistakis, N. T., Tooze, S. A. Digesting the expanding mechanisms of autophagy. Trends in Cell Biology. 26 (8), 624-635 (2016).
- Tanida, I., Ueno, T., Kominami, E. LC3 and autophagy. Methods in Molecular Biology. 445, 77-88 (2008).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO Journal. 19 (21), 5720-5728 (2000).
- Mizushima, N., Yoshimori, T. How to interpret LC3 immunoblotting. Autophagy. 3 (6), 542-545 (2007).
- Vanasco, V., et al. Mitochondrial dynamics and VMP1-related selective mitophagy in experimental acute pancreatitis. Frontiers in Cell and Developmental Biology. 9, 640094(2021).
- Williams, J. A. Regulatory mechanisms in pancreas and salivary acini. Annual Review of Physiology. 46, 361-375 (1984).
- Mizrahi, J. D., Surana, R., Valle, J. W., Shroff, R. T. Pancreatic cancer. Lancet. 395 (10242), 2008-2020 (2020).
- Li, J., et al. Regulation and function of autophagy in pancreatic cancer. Autophagy. 17 (11), 3275-3296 (2021).
- Ropolo, A., et al. A novel E2F1-EP300-VMP1 pathway mediates gemcitabine-induced autophagy in pancreatic cancer cells carrying oncogenic KRAS. Frontiers in Endocrinology. 11, 411(2020).
- Pardo, R., et al. Gemcitabine induces the VMP1-mediated autophagy pathway to promote apoptotic death in human pancreatic cancer cells. Pancreatology. 10 (1), 19-26 (2010).
- Logsdon, C. D., Moessner, J., Williams, J. A., Goldfine, I. D. Glucocorticoids increase amylase mRNA levels, secretory organelles, and secretion in pancreatic acinar AR42J cells. Journal of Cell Biology. 100 (4), 1200-1208 (1985).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (4th edition). Autophagy. 17 (1), 1(2021).
- Segeritz, C. -P., Vallier, L. Chapter 9 - Cell culture: Growing cells as model systems in vitro. Basic Science Methods for Clinical Researchers. Jalali, M., Saldanha, F. Y. L., Jalali, M. , Academic Press. Cambridge, MA. 151-172 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Current Protocols in Cytometry. 92 (1), 68(2020).
- Grasso, D., et al. a novel selective autophagy pathway mediated by VMP1-USP9x-p62, prevents pancreatic cell death. Journal of Biological Chemistry. 286 (10), 8308-8324 (2011).
- Ropolo, A., et al. The pancreatitis-induced vacuole membrane protein 1 triggers autophagy in mammalian cells. Journal of Biological Chemistry. 282 (51), 37124-37133 (2007).
- Karanasios, E., Stapleton, E., Walker, S. A., Manifava, M., Ktistakis, N. T. Live cell imaging of early autophagy events: Omegasomes and beyond. Journal of Visualized Experiments. (77), e50484(2013).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Zhang, Z., Singh, R., Aschner, M. Methods for the detection of autophagy in mammalian cells. Current Protocols in Toxicology. 69, 1-26 (2016).
- Yoshii, S. R., Mizushima, N. Monitoring and measuring autophagy. International Journal of Molecular Sciences. 18 (9), 1865(2017).
- Betriu, N., Andreeva, A., Semino, C. E. Erlotinib promotes ligand-induced EGFR degradation in 3D but not 2D cultures of pancreatic ductal adenocarcinoma cells. Cancers. 13 (18), 4504(2021).
- Wang, W., Dong, L., Zhao, B., Lu, J., Zhao, Y. E-cadherin is downregulated by microenvironmental changes in pancreatic cancer and induces EMT. Oncology Reports. 40 (3), 1641-1649 (2018).
- Kim, S. K., et al. Phenotypic heterogeneity and plasticity of cancer cell migration in a pancreatic tumor three-dimensional culture model. Cancers. 12 (5), 1305(2020).
- Meng, Y., et al. Cytoplasmic EpCAM over-expression is associated with favorable clinical outcomes in pancreatic cancer patients with Hepatitis B virus negative infection. International Journal of Clinical and Experimental Medicine. 8 (12), 22204-22216 (2015).
- Brock, R., Hamelers, I. H., Jovin, T. M. Comparison of fixation protocols for adherent cultured cells applied to a GFP fusion protein of the epidermal growth factor receptor. Cytometry. 35 (4), 353-362 (1999).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados