Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluation des niveaux d’autophagie dans deux modèles de cellules pancréatiques différents à l’aide de l’immunofluorescence LC3
Dans cet article
Résumé
L’objectif de ce protocole est de déterminer les niveaux autophagiques dans le cancer du pancréas et les cellules acineuses pancréatiques par immunofluorescence CL3 et quantification des points CL3.
Résumé
L’autophagie est un processus catabolique spécialisé qui dégrade sélectivement les composants cytoplasmiques, y compris les protéines et les organites endommagés. L’autophagie permet aux cellules de répondre physiologiquement aux stimuli de stress et, ainsi, de maintenir l’homéostasie cellulaire. Les cellules cancéreuses peuvent moduler leurs niveaux d’autophagie pour s’adapter à des conditions défavorables telles que l’hypoxie, une carence en nutriments ou des dommages causés par la chimiothérapie. L’adénocarcinome pancréatique canalaire est l’un des types de cancer les plus mortels. Les cellules cancéreuses du pancréas ont une activité d’autophagie élevée en raison de la régulation transcriptionnelle à la hausse et de l’activation post-traductionnelle des protéines d’autophagie.
Ici, la lignée cellulaire PANC-1 a été utilisée comme modèle de cellules cancéreuses humaines pancréatiques, et la lignée cellulaire acineuse pancréatique AR42J a été utilisée comme modèle physiologique de cellules de mammifères hautement différenciées. Cette étude a utilisé l’immunofluorescence de la chaîne légère 3 (CL3) de la protéine associée aux microtubules comme indicateur de l’état de l’activation de l’autophagie. LC3 est une protéine d’autophagie qui, dans des conditions basales, présente un schéma diffus de distribution dans le cytoplasme (connu sous le nom de LC3-I dans cette condition). L’induction de l’autophagie déclenche la conjugaison de la CL3 à la phosphatidyléthanolamine à la surface des autophagosomes nouvellement formés pour former la LC3-II, une protéine liée à la membrane qui aide à la formation et à l’expansion des autophagosomes. Pour quantifier le nombre de structures autophagiques étiquetées, le logiciel open source FIJI a été utilisé à l’aide de l’outil « 3D Objects Counter ».
La mesure des niveaux autophagiques à la fois dans les conditions physiologiques et dans les cellules cancéreuses nous permet d’étudier la modulation de l’autophagie dans diverses conditions telles que l’hypoxie, le traitement de chimiothérapie ou l’élimination de certaines protéines.
Introduction
La macroautophagie (communément appelée autophagie) est un processus catabolique spécialisé qui dégrade sélectivement les composants cytoplasmiques, y compris les protéines et les organites endommagés 1,2. L’autophagie permet aux cellules de répondre physiologiquement aux stimuli de stress et, ainsi, de maintenir l’homéostasie cellulaire3. Au cours de l’autophagie, une vésicule à double membrane se forme : l’autophagosome. L’autophagosome contient les molécules de cargaison et les conduit au lysosome pour la dégradation 1,4.
Les autophagosomes sont décorés par la chaîne légère 3 (CL3)5 de la protéine autophagique associée aux microtubules. Lorsque l’autophagie n’est pas induite, la CL3 est diffusée dans le cytoplasme et le noyau dans la conformation LC3-I. En revanche, lorsque l’autophagie est induite, la CL3 est conjuguée à une phosphatidyléthanolamine dans la membrane des structures autophagiques6. Cette nouvelle conformation LC3 est connue sous le nom de LC3-II1. Le décalage de conformation de la CL3 provoque des changements dans sa localisation cellulaire et sa migration par électrophorèse sur gel de sulfate de sodium dodécyle-polyacrylamide (SDS-PAGE), qui peuvent être détectées par des techniques telles que l’immunofluorescence et le Western blot 5,7. De cette façon, la conjugaison de la CL3 est un événement clé dans le processus autophagique qui peut être utilisé pour mesurer l’activité autophagique.
La cellule acineuse pancréatique est une cellule hautement différenciée qui, dans des conditions saines, a un faible taux d’autophagie. Cependant, dans différentes conditions physiologiques ou sous stimulation pharmacologique, ils peuvent activer l’autophagie. Par conséquent, la détermination des niveaux autophagiques dans cette lignée cellulaire est utile pour étudier les effets directs ou indirects potentiels de différents agents pharmacologiques ou biologiques sur l’autophagie 8,9.
L’adénocarcinome pancréatique canalaire est l’un des types de cancer les plus mortels, compte tenu de son diagnostic tardif et de sa résistance élevée à la chimiothérapie10. Les cellules cancéreuses du pancréas ont une activité d’autophagie élevée en raison de la régulation transcriptionnelle positive et de l’activation post-traductionnelle des protéines liées à l’autophagie11. Les cellules cancéreuses du pancréas peuvent ajuster leurs niveaux d’autophagie en réponse à des conditions défavorables telles que l’hypoxie, la privation de nutriments ou les dommages induits par la chimiothérapie11. Par conséquent, l’analyse des niveaux d’autophagie dans les cellules cancéreuses du pancréas peut aider à comprendre comment elles s’adaptent à divers environnements et à évaluer l’efficacité des traitements modulateurs de l’autophagie.
Cette étude montre une méthode pour effectuer l’immunofluorescence LC3 dans deux modèles cellulaires pancréatiques distincts. Le premier modèle, les cellules PANC-1, a servi de modèle pour l’adénocarcinome canalaire pancréatique. Ces cellules ont été traitées avec de la gemcitabine, un agent de chimiothérapie dont il a déjà été démontré qu’il induisait l’autophagie, en particulier dans les cellules cancéreuses du pancréas porteuses du gène oncogène du virus du sarcome du rat de Kirsten (KRAS)12,13. Le deuxième modèle, les cellules AR42J, a servi de modèle plus physiologique des cellules pancréatiques exocrines. Ces cellules ont été différenciées avec la dexaméthasone pour devenir plus semblables aux cellules pancréatiques acineuses14. Dans ces cellules, l’autophagie a été induite pharmacologiquement par l’utilisation de PP242, qui est un puissant inhibiteur de mTOR15. Dans cette étude, nous démontrons l’applicabilité du protocole décrit avec deux modèles pancréatiques différents et sa capacité à discriminer entre les états d’autophagie basse et élevée.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparation cellulaire
- Trempez des lamelles de couverture rondes de 12 mm dans de l’éthanol absolu et placez-les verticalement dans les puits d’une plaque de 24 puits.
- Retirez le couvercle et exposez la plaque multipuits aux rayons ultraviolets pendant 15 min.
- Positionnez les lamelles horizontalement et lavez-les avec le milieu Eagle modifié (DMEM) de Dulbecco.
- Ensemencer un faible nombre de passages de cellules pancréatiques. Le montant doit être ajusté pour obtenir une confluence de 50% à 75% le jour de la fixation16.
NOTE: Il est recommandé d’ensemencer 2,5 × 10 4 PANC-1 ou 4 × 104 cellules AR42J par puits pour fixer les cellules après 3 jours. - Culture des cellules dans du DMEM contenant 10 % de sérum bovin fœtal, 100 U/mL de pénicilline et 100 μg/mL de streptomycine dans un incubateur à 37 °C sous atmosphère humidifiée contenant 5 % de dioxyde de carbone (CO2).
REMARQUE: Pour les cellules PANC-1, il est recommandé d’incuber les cellules pendant 2 jours entre l’ensemencement cellulaire et les étapes suivantes. Passé ce délai, les cellules peuvent être transfectées, traitées ou fixées. Ce protocole illustre le traitement à la gemcitabine dans les cellules PANC-1 non transfectées et le traitement de différenciation et de PP242 pour les cellules AR42J non transfectées.
2. Traitement des cellules
- Traitement à la gemcitabine pour les cellules PANC-1
- Préparer une solution de 1 μg/μL de gemcitabine dans du DMEM 2 jours après l’ensemencement. Traiter chaque puits avec 2,6 μL de la solution de gemcitabine à 1 μg/μL pour obtenir une dilution finale de 20 μM.
- Incuber les cellules pendant 24 h dans l’incubateur.
- Différenciation AR42J et traitement PP242
- Préparer une solution de 4 μg/mL de dexaméthasone dans du DMEM.
- Traiter chaque puits avec 4,9 μL de solution de dexaméthasone à 4 μg/mL pour obtenir une dilution finale de 100 nM.
- Incuber les cellules pendant 48 h dans l’incubateur.
- Retirer le milieu et traiter chaque puits avec 0,5 μL de 1 mM PP242 pour obtenir une dilution finale de 1 μM.
- Incuber les cellules pendant 2 h dans l’incubateur.
3. Fixation et perméabilisation des cellules
- Préparer une plaque de 24 puits avec du méthanol froid et une plaque de 6 puits avec une solution saline tamponnée au phosphate froid (PBS; 137 mM NaCl, 2,7 mM KCl, 8 mM Na 2 HPO 4, 2 mMKH2PO4). Entretenez-les sur la glace.
- Prenez chaque capiton avec une pince à épiler, lavez-le deux fois dans du PBS et incuber pendant 6 minutes dans du méthanol.
4. Blocage des cellules
- Laver chaque lamelle deux fois dans du PBS et incuber pendant 1 h dans du sérum fœtal bovin à 10% dans du PBS (solution bloquante).
Remarque : Dans cette étape, le protocole peut être suspendu. Les lamelles de couverture peuvent être conservées pendant la nuit au réfrigérateur dans la solution de blocage, et le protocole peut être poursuivi le lendemain.
5. Incubation des lamelles de couverture avec l’anticorps primaire
- Préparez une solution 1:1 000 d’anti-CL3 dans la solution bloquante et maintenez-la sur de la glace.
- Placez un morceau de film d’étanchéité de laboratoire sur le couvercle multipuits.
- Déposer une goutte (25 μL) par capseau de solution anti-LC3 sur le film d’étanchéité.
- Prenez chaque capseau avec une pince à épiler et placez-le sur la goutte d’anticorps primaire, en veillant à ce que le côté cellulaire soit en contact avec la solution.
- Préparez une chambre humide en plaçant un morceau de papier humide dans une boîte en plastique à fond plat.
- Placez l’assiette multipuits dans la chambre d’humidité, recouvrez-la de papier d’aluminium et incuber toute la nuit au réfrigérateur.
6. Incubation des lamelles de couverture avec l’anticorps secondaire
- Retirez la plaque multipuits de la chambre d’humidité et replacez les lamelles de couverture dans la plaque multipuits.
- Effectuez trois lavages avec PBS.
- Préparer une solution d’anti-lapin marquée par fluorescence avec une dilution de 1:800 dans la solution de blocage et la maintenir sur de la glace à l’abri de la lumière.
- Placez un morceau de film d’étanchéité sur le couvercle multipuits.
- Déposer une goutte (25 μL) par capseau de solution anti-lapin sur le film d’étanchéité.
- Prenez chaque capseau avec une pince à épiler et placez-le sur la goutte d’anticorps primaire, en veillant à ce que le côté cellulaire soit en contact avec la solution.
- Incuber la plaque multipuits dans la chambre d’humidité pendant 2 h à température ambiante (RT) à l’abri de la lumière.
7. Coloration des cellules avec 4′,6-diamidino-2-phénylindole (DAPI)
- Retirez la plaque multipuits de la chambre d’humidité et replacez les lamelles de couverture dans la plaque multipuits.
- Effectuez trois lavages avec PBS.
- Préparer une solution 300 nM de DAPI en PBS (protégé de la lumière).
- Incuber chaque capuchon avec la solution DAPI pendant 10 min.
- Effectuez trois lavages avec PBS. Entretenez la plaque multipuits à l’abri de la lumière.
8. Montage
- Préparez deux béchers avec de l’eau et un morceau de papier.
- Déposer une goutte (10 μL) par lamelle de couverture d’une solution d’adduit à l’alcool polyvinylique-bis(triméthylaluminium)-1,4-diazabicyclo[2.2.2]octane (PVA-DABCO) sur une lame.
REMARQUE : PVA-DABCO est préparé en combinant 0,25 M de DABCO, 10 % P/V PVA, 20 % de glycérol et 50 % de Tris HCl (1,5 M, pH 8,8) dans de l’eau ultrapure. - Prenez chaque capuchon avec une pince à épiler, lavez-le dans chaque bécher d’eau, séchez-le dans le papier et placez-le sur la goutte PVA-DABCO (avec les cellules en contact avec la solution).
- Laissez-le sécher toute la nuit, à l’abri de la lumière.
9. Visualisation par microscopie confocale et capture d’images
- Visualisez les lames de couverture dans un microscope confocale inversé à l’aide d’un objectif d’environ 63x17.
- Capturez des images représentatives des cellules étiquetées.
10. Quantifier les points CL3
- Faites glisser et déposez chaque fichier image contenant les canaux capturés, tels que « .czi », dans l’écran ImageJ (FIJI) pour l’ouvrir. Cliquez sur OK dans la boîte de dialogue et fermez la fenêtre de la console .
- Dans l’onglet Image , sélectionnez Couleur > Fractionner les couches.
- Fermez les images correspondant aux canaux autres que l’image LC3.
- Dans l’onglet Image , sélectionnez Ajuster > balance des couleurs
- Déplacez le curseur Maximum vers la gauche jusqu’à ce que l’image soit saturée pour visualiser les contours de la cellule.
- Dessinez le contour de la cellule à l’aide de l’outil Sélection à main levée .
- Cliquez sur le bouton Réinitialiser pour réinitialiser le réglage des couleurs.
- Dans l’onglet Modifier , sélectionnez Couper pour couper l’élément sélectionné.
- Fermez l’image sans l’enregistrer.
- Dans l’onglet Modifier , sélectionnez Coller.
- Dans le menu Analyser , choisissez l’outil Compteur d’objets 3D.
- Définissez le seuil. Dans l’exemple fourni dans cette étude, le seuil est fixé à 2 000.
- Définissez le filtre de taille. Dans cette étude, il est fixé entre 50 et 500.
- Assurez-vous que les cases Objets et Résumé sont cochées.
- Cliquez sur Ok. Le nombre de points sera décrit comme Objets détectés dans le résumé.
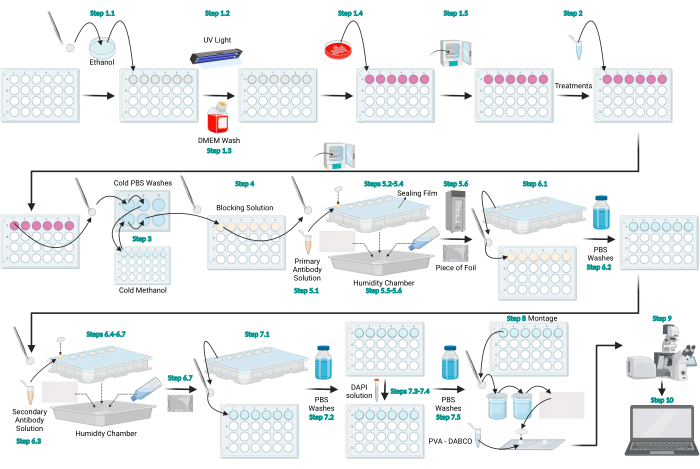
Figure 1 : Schéma du protocole d’immunofluorescence CL3. Diagramme schématique qui représente le protocole général fourni pour l’immunofluorescence CL3. Figure créée avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Ce protocole effectue une immunofluorescence de CL3 dans des lignées cellulaires pancréatiques pour déterminer les niveaux d’autophagie dans différentes conditions. Le résultat de cette expérience a été l’obtention d’images cellulaires à partir des canaux rouge et bleu, correspondant à la CL3 et au DAPI. Les images CL3 indiquent la distribution cellulaire de cette protéine, tandis que le DAPI montre la localisation nucléaire. La figure 2A montre une image représentative de...
Access restricted. Please log in or start a trial to view this content.
Discussion
La méthode décrite dans ce protocole permet de visualiser la distribution endogène de la CL3 dans la cellule et de quantifier les niveaux autophagiques dans différentes conditions. Une autre méthode similaire utilisée pour analyser la distribution de la CL3 et déterminer l’activation de l’autophagie implique la transfection de CL3 marquée par fluorescence (telle que RFP-LC3)19. La transfection RFP-LC3 présente l’avantage de ne pas avoir besoin de fixation (ce qui permet d’applique...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Aucun conflit d’intérêts n’a été déclaré.
Remerciements
Ce travail a été soutenu par des subventions de l’Université de Buenos Aires (UBACyT 2018-2020 20020170100082BA), du Conseil national pour la recherche scientifique et la technologie (CONICET) (PIP 2021-2023 GI− 11220200101549CO; et PUE 22920170100033) et de l’Agence nationale pour la promotion scientifique et technologique (PICT 2019-01664).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 10x Phosphate-Buffered Saline (PBS) | Corning | 46-013-CM | |
| 12 mm round coverslips | HDA | CBR_OBJ_6467 | |
| 24 Well- Cell Culture Plate | Sorfa | 220300 | |
| Absolute ethanol | Biopack | 2207.10.00 | |
| Alexa Fluor 594 Donkey anti-rabbit IgG (H+L) | Invitrogen | R37119 | |
| Confocal Laser Scanning Microscope | Zeiss | LSM 800 | |
| Dexamethasone | Sigma Aldrich | D4902 | |
| DMEN | Sartorius | 01-052-1A | |
| Fetal Bovine Serum | NATOCOR | Lintc-634 | |
| Gemcitabina | Eli Lilly | VL7502 | |
| LC3B (D11) XP Rabbit mAb | Cell Signaling Technology | 3868S | |
| Methanol | Anedra | 6197 | |
| Parafilm "M" (Laboratory Sealing Film) | Bemis/Curwood | PM-996 | |
| Pen-Strep Solution | Sartorius | 03-031-1B | |
| PP242 | Santa Cruz Biotechnology | SC-301606 | |
| Trypsin EDTA | Gibco | 11570626 |
Références
- Grasso, D., Renna, F. J., Vaccaro, M. I. Initial steps in mammalian autophagosome biogenesis. Frontiers in Cell and Developmental Biology. 6, 146(2018).
- Galluzzi, L., et al. Molecular definitions of autophagy and related processes. EMBO Journal. 36 (13), 1811-1836 (2017).
- Kitada, M., Koya, D. Autophagy in metabolic disease and ageing. Nature Reviews Endocrinology. 17 (11), 647-661 (2021).
- Ktistakis, N. T., Tooze, S. A. Digesting the expanding mechanisms of autophagy. Trends in Cell Biology. 26 (8), 624-635 (2016).
- Tanida, I., Ueno, T., Kominami, E. LC3 and autophagy. Methods in Molecular Biology. 445, 77-88 (2008).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO Journal. 19 (21), 5720-5728 (2000).
- Mizushima, N., Yoshimori, T. How to interpret LC3 immunoblotting. Autophagy. 3 (6), 542-545 (2007).
- Vanasco, V., et al. Mitochondrial dynamics and VMP1-related selective mitophagy in experimental acute pancreatitis. Frontiers in Cell and Developmental Biology. 9, 640094(2021).
- Williams, J. A. Regulatory mechanisms in pancreas and salivary acini. Annual Review of Physiology. 46, 361-375 (1984).
- Mizrahi, J. D., Surana, R., Valle, J. W., Shroff, R. T. Pancreatic cancer. Lancet. 395 (10242), 2008-2020 (2020).
- Li, J., et al. Regulation and function of autophagy in pancreatic cancer. Autophagy. 17 (11), 3275-3296 (2021).
- Ropolo, A., et al. A novel E2F1-EP300-VMP1 pathway mediates gemcitabine-induced autophagy in pancreatic cancer cells carrying oncogenic KRAS. Frontiers in Endocrinology. 11, 411(2020).
- Pardo, R., et al. Gemcitabine induces the VMP1-mediated autophagy pathway to promote apoptotic death in human pancreatic cancer cells. Pancreatology. 10 (1), 19-26 (2010).
- Logsdon, C. D., Moessner, J., Williams, J. A., Goldfine, I. D. Glucocorticoids increase amylase mRNA levels, secretory organelles, and secretion in pancreatic acinar AR42J cells. Journal of Cell Biology. 100 (4), 1200-1208 (1985).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (4th edition). Autophagy. 17 (1), 1(2021).
- Segeritz, C. -P., Vallier, L. Chapter 9 - Cell culture: Growing cells as model systems in vitro. Basic Science Methods for Clinical Researchers. Jalali, M., Saldanha, F. Y. L., Jalali, M. , Academic Press. Cambridge, MA. 151-172 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Current Protocols in Cytometry. 92 (1), 68(2020).
- Grasso, D., et al. a novel selective autophagy pathway mediated by VMP1-USP9x-p62, prevents pancreatic cell death. Journal of Biological Chemistry. 286 (10), 8308-8324 (2011).
- Ropolo, A., et al. The pancreatitis-induced vacuole membrane protein 1 triggers autophagy in mammalian cells. Journal of Biological Chemistry. 282 (51), 37124-37133 (2007).
- Karanasios, E., Stapleton, E., Walker, S. A., Manifava, M., Ktistakis, N. T. Live cell imaging of early autophagy events: Omegasomes and beyond. Journal of Visualized Experiments. (77), e50484(2013).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Zhang, Z., Singh, R., Aschner, M. Methods for the detection of autophagy in mammalian cells. Current Protocols in Toxicology. 69, 1-26 (2016).
- Yoshii, S. R., Mizushima, N. Monitoring and measuring autophagy. International Journal of Molecular Sciences. 18 (9), 1865(2017).
- Betriu, N., Andreeva, A., Semino, C. E. Erlotinib promotes ligand-induced EGFR degradation in 3D but not 2D cultures of pancreatic ductal adenocarcinoma cells. Cancers. 13 (18), 4504(2021).
- Wang, W., Dong, L., Zhao, B., Lu, J., Zhao, Y. E-cadherin is downregulated by microenvironmental changes in pancreatic cancer and induces EMT. Oncology Reports. 40 (3), 1641-1649 (2018).
- Kim, S. K., et al. Phenotypic heterogeneity and plasticity of cancer cell migration in a pancreatic tumor three-dimensional culture model. Cancers. 12 (5), 1305(2020).
- Meng, Y., et al. Cytoplasmic EpCAM over-expression is associated with favorable clinical outcomes in pancreatic cancer patients with Hepatitis B virus negative infection. International Journal of Clinical and Experimental Medicine. 8 (12), 22204-22216 (2015).
- Brock, R., Hamelers, I. H., Jovin, T. M. Comparison of fixation protocols for adherent cultured cells applied to a GFP fusion protein of the epidermal growth factor receptor. Cytometry. 35 (4), 353-362 (1999).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon