このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
LC3免疫蛍光を用いた2つの異なる膵臓細胞モデルにおけるオートファジーレベルの評価(英語)
要約
このプロトコルの目的は、LC3免疫蛍光およびLC3ドット定量を通じて、膵臓癌および膵臓腺房細胞のオートファジーレベルを決定することです。
要約
オートファジーは、タンパク質や損傷した細胞小器官などの細胞質成分を選択的に分解する特殊な異化プロセスです。オートファジーは、細胞がストレス刺激に生理学的に応答することを可能にし、したがって、細胞の恒常性を維持する。がん細胞は、オートファジーレベルを調節して、低酸素症、栄養不足、化学療法による損傷などの悪条件に適応する可能性があります。乳管膵臓腺癌は、最も致命的な種類の癌の1つです。膵臓がん細胞は、オートファジータンパク質の転写アップレギュレーションと翻訳後活性化により、高いオートファジー活性を示します。
ここでは、膵臓ヒト癌細胞のモデルとしてPANC-1細胞株を用い、高分化哺乳類細胞の生理モデルとしてAR42J膵腺房細胞株を用いた。この研究では、オートファジー活性化の状態の指標として、微小管関連タンパク質軽鎖3(LC3)の免疫蛍光を使用しました。LC3はオートファジータンパク質であり、基底条件下では細胞質内の分布の拡散パターンを示します(この状態ではLC3-Iとして知られています)。オートファジー誘導は、新しく形成されたオートファゴソームの表面上のLC3とホスファチジルエタノールアミンの結合を引き起こし、オートファゴソームの形成と増殖を助ける膜結合タンパク質であるLC3-IIを形成します。標識されたオートファジー構造の数を定量化するために、オープンソースソフトウェアFIJIが「3Dオブジェクトカウンター」ツールの助けを借りて利用されました。
生理学的条件と癌細胞の両方におけるオートファジーレベルの測定により、低酸素症、化学療法治療、または特定のタンパク質のノックダウンなどの多様な条件下でのオートファジーの調節を研究することができます。
概要
マクロオートファジー(一般にオートファジーと呼ばれる)は、タンパク質や損傷した細胞小器官などの細胞質成分を選択的に分解する特殊な異化プロセスです1,2。オートファジーは、細胞がストレス刺激に生理学的に応答することを可能にし、したがって、細胞の恒常性を維持する3。オートファジーの間に、二重膜小胞、すなわちオートファゴソームが形成される。オートファゴソームは貨物分子を含み、分解のためにそれらをリソソームに駆動します1,4。
オートファゴソームは、オートファジータンパク質微小管関連タンパク質軽鎖3(LC3)5によって装飾されています。オートファジーが誘導されない場合、LC3は細胞質に拡散し、核はLC3-I立体構造に拡散します。一方、オートファジーが誘導されると、LC3はオートファジー構造6の膜においてホスファチジルエタノールアミンと共役する。この新しいLC3立体配座はLC3-II1として知られています。LC3コンフォメーションシフトは、その細胞局在とドデシル硫酸ナトリウム-ポリアクリルアミドゲル電気泳動(SDS-PAGE)の移行に変化を引き起こし、免疫蛍光法やウェスタンブロットなどの手法で検出できます5,7。このように、LC3コンジュゲーションは、オートファジー活動の測定に使用できるオートファジープロセスの重要なイベントです。
膵臓腺房細胞は高度に分化した細胞であり、健康な条件下ではオートファジーの発生率が低い。しかしながら、異なる生理学的条件において、または薬理学的刺激下で、それらはオートファジーを活性化することができる。したがって、この細胞株におけるオートファジーレベルの決定は、オートファジーに対する異なる薬理学的または生物学的薬剤の潜在的な直接的または間接的な効果を研究するのに有用である8,9。
乳管膵臓腺癌は、診断が遅く、化学療法耐性が高いことを考えると、最も致命的な種類の癌の10つです。膵臓がん細胞は、オートファジー関連タンパク質の転写アップレギュレーションと翻訳後活性化により、高いオートファジー活性を示します11。膵臓がん細胞は、低酸素症、栄養不足、化学療法による損傷などの好ましくない条件に応じてオートファジーレベルを調整する可能性があります11。したがって、膵臓がん細胞のオートファジーレベルを分析することは、それらがさまざまな環境にどのように適応するかを理解し、オートファジー調節治療の有効性を評価するのに役立ちます。
この研究は、2つの異なる膵臓細胞モデルでLC3免疫蛍光を実行する方法を示しています。最初のモデルであるPANC-1細胞は、膵管腺癌のモデルとして機能しました。これらの細胞は、発癌性キルステンラット肉腫ウイルス遺伝子(KRAS)12,13を有する膵臓癌細胞において、オートファジーを誘導することが以前に示されている化学療法剤であるゲムシタビンで処理されました。第2のモデルであるAR42J細胞は、膵臓外分泌細胞のより生理学的なモデルとして役立った。これらの細胞をデキサメタゾンで分化させて、腺房膵臓細胞14により類似したものとなった。これらの細胞では、強力なmTOR阻害剤であるPP242を使用することでオートファジーが薬理学的に誘導されました15。本研究では、2つの異なる膵臓モデルで記述されたプロトコルの適用性と、低オートファジーと高オートファジーの状態を区別する能力を実証します。
プロトコル
1.細胞調製
- 12 mmの丸いカバーガラスを無水エタノールに浸し、24ウェルプレートのウェルに垂直に置きます。
- カバーを取り外し、マルチウェルプレートを紫外線に15分間さらします。
- カバーガラスを水平に置き、ダルベッコのモディファイドイーグルミディアム(DMEM)で洗います。
- 低継代数の膵臓細胞を播種する。固定日に50%〜75%のコンフルエンシーが得られるように量を調整する必要があります16。
注:ウェルあたり2.5 ×10 4 PANC-1または4 ×10 4 AR42J細胞を播種して、3日後に細胞を固定することをお勧めします。 - 10%ウシ胎児血清、100 U/mLペニシリン、および100 μg/mLストレプトマイシンを含むDMEMで、5%二酸化炭素(CO2)を含む加湿雰囲気下、37°Cのインキュベーター内で細胞を培養します。
注:PANC-1細胞の場合、細胞の播種と次の手順の間に2日間細胞をインキュベートすることをお勧めします。この時間の後、細胞をトランスフェクト、処理、または固定することができます。このプロトコルは、非トランスフェクションPANC-1細胞におけるゲムシタビンによる治療、ならびに非トランスフェクションAR42J細胞に対する分化およびPP242治療を例示する。
2.細胞を処理する
- PANC-1細胞に対するゲムシタビン治療
- 播種後2日目に1 μg/μLゲムシタビンをDMEM溶液で調製します。各ウェルを2.6 μLの1 μg/μLゲムシタビン溶液で処理して、20 μMの最終希釈を達成します。
- インキュベーター内で細胞を24時間インキュベートします。
- AR42J分化とPP242治療
- 4 μg/mL デキサメタゾンの DMEM 溶液を準備します。
- 各ウェルを4.9 μLの4 μg/mLデキサメタゾン溶液で処理して、100 nMの最終希釈液を得ます。
- インキュベーター内で細胞を48時間インキュベートします。
- 培地を取り出し、各ウェルを0.5 μLの1 mM PP242で処理して、1 μMの最終希釈液を得ました。
- インキュベーター内で細胞を2時間インキュベートします。
3.細胞の固定と透過処理
- 冷メタノールを含む24ウェルプレートと、冷リン酸緩衝生理食塩水(PBS;137 mM NaCl、2.7 mM KCl、8 mM Na 2 HPO 4、2 mM KH2PO4)を含む6ウェルプレートを準備します。氷の上でそれらを維持してください。
- 各カバーガラスをピンセットで取り、PBSで2回洗浄し、メタノール中で6分間インキュベートします。
4.セルをブロックする
- 各カバーガラスをPBSで2回洗浄し、PBS(ブロッキング溶液)中の10%ウシ胎児血清中で1時間インキュベートします。
注 : この手順では、プロトコルが一時停止される場合があります。カバーガラスはブロッキング溶液中の冷蔵庫に一晩保管することができ、プロトコルは翌日も続けることができます。
5. カバーガラスと一次抗体のインキュベーション
- ブロッキング溶液に抗LC3の1:1,000溶液を調製し、氷上で維持します。
- マルチウェルの蓋の上に実験室のシーリングフィルムを置きます。
- シーリングフィルムの上に、抗LC3溶液のカバーガラスごとに1滴(25μL)を置きます。
- 各カバーガラスをピンセットで取り、細胞側が溶液に接触するように注意しながら、一次抗体ドロップの上に置きます。
- 湿った紙を平底のプラスチックの箱に入れて、湿った部屋を準備します。
- マルチウェルプレートを湿度チャンバーに入れ、ホイルで覆い、冷蔵庫で一晩インキュベートします。
6. カバーガラスと二次抗体のインキュベーション
- マルチウェルプレートを湿度チャンバーから取り外し、カバーガラスをマルチウェルプレートに戻します。
- PBSで3回洗浄します。
- ブロッキング溶液中で1:800に希釈した蛍光標識抗ウサギの溶液を調製し、光から保護された氷上で維持する。
- マルチウェル蓋の上にシーリングフィルム片を置きます。
- 抗ウサギ溶液のカバーガラスごとに一滴(25μL)をシーリングフィルムの上に置きます。
- 各カバーガラスをピンセットで取り、細胞側が溶液に接触するように注意しながら、一次抗体ドロップの上に置きます。
- マルチウェルプレートを湿度チャンバー内で、光から保護された室温(RT)で2時間インキュベートします。
7. 4′,6-ジアミジノ-2-フェニルインドール(DAPI)による細胞の染色
- マルチウェルプレートを湿度チャンバーから取り外し、カバーガラスをマルチウェルプレートに戻します。
- PBSで3回洗浄します。
- PBS(光から保護されている)中のDAPIの300 nM溶液を調製します。
- 各カバーガラスをDAPI溶液で10分間インキュベートします。
- PBSで3回洗浄します。マルチウェルプレートを光から保護してください。
8.モンタージュ
- 水と一枚の紙で2つのビーカーを準備します。
- ポリビニルアルコール-ビス(トリメチルアルミニウム)-1,4-ジアザビシクロ[2.2.2]オクタン付加物(PVA-DABCO)溶液のカバーガラス1枚につき1滴(10 μL)をスライドに置きます。
注:PVA-DABCOは、0.25 M DABCO、10% W/V PVA、20% グリセロール、および 50% トリス塩酸塩 (1.5 M、pH 8.8) を超純水中で組み合わせて調製されます。 - ピンセットで各カバーガラスを取り、各ウォータービーカーで洗い、紙で乾かし、PVA-DABCOドロップの上に置きます(セルを溶液と接触させます)。
- 光から保護して一晩乾かします。
9. 共焦点顕微鏡観察と画像キャプチャ
- 倒立共焦点顕微鏡でカバーガラスを約63x17の対物レンズで可視化します。
- 標識された細胞の代表的な画像をキャプチャします。
10. LC3ドットの定量化
- キャプチャしたチャンネルを含む各画像ファイル(「.czi」など)をImageJ(FIJI)画面にドラッグ&ドロップして開きます。ダイアログ ボックスで [OK ] をクリックし、[ コンソール ] ウィンドウを閉じます。
- 「画像」タブで、「カラー>分割チャンネル」を選択します。
- LC3画像以外のチャンネルに対応する画像を閉じます。
- [画像]タブから、[>カラーバランスの調整]を選択します
- 画像が飽和するまで [ 最大 ] スライダーを左に動かして、セルの輪郭を視覚化します。
- フリーハンド選択ツールでセルのアウトラインを描画します。
- [リセット]ボタンをクリックして、色調整をリセットします。
- [編集] タブで、[切り取り] を選択して、選択した項目を切り取ります。
- 画像を保存せずに閉じます。
- [ 編集 ] タブで、[ 貼り付け] を選択します。
- [ 分析] メニューで、ツール[ 3Dオブジェクトカウンター]を選択します。
- しきい値を設定します。この調査で提供される例では、しきい値は 2,000 に設定されています。
- サイズ フィルターを設定します。この研究では、50から500の間に設定されています。
- [オブジェクト] ボックスと [概要] ボックスにマークが付いていることを確認します。
- OKをクリックします。ドットの数は、概要で検出されたオブジェクトとして説明されます。
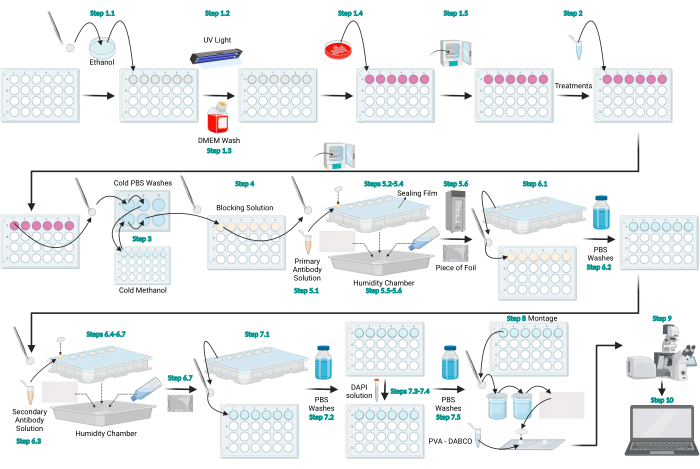
図1:LC3免疫蛍光プロトコルの概略図。LC3免疫蛍光法のために提供される一般的なプロトコールを表す概略図。BioRender.com で作成された図。この図の拡大版を表示するには、ここをクリックしてください。
結果
このプロトコルは、膵臓細胞株におけるLC3の免疫蛍光を行い、異なる条件下でのオートファジーレベルを決定する。この実験の結果は、LC3とDAPIに対応する赤と青のチャネルからの細胞画像の観察でした。LC3画像はこのタンパク質の細胞分布を示し、DAPIは核局在を示しています。 図2A は、基礎またはゲムシタビン処理条件下でのPANC-1細胞におけるLC3およびそのDAPI染色と...
ディスカッション
このプロトコルに記載されている方法は、細胞内の内因性LC3分布を視覚化し、異なる条件下でオートファジーレベルを定量化することを可能にする。LC3の分布を分析し、オートファジーの活性化を決定するために使用される別の同様の方法には、蛍光標識されたLC3トランスフェクション(RFP-LC3など)が含まれます19。RFP-LC3トランスフェクションには、固定が不要(この方法をラ...
開示事項
利益相反は宣言されていません。
謝辞
この研究は、ブエノスアイレス大学(UBACyT 2018-2020 20020170100082BA)、国立科学研究技術評議会(CONICET)(PIP 2021-2023 GI− 11220200101549CO;およびPUE 22920170100033)、および国立科学技術振興庁(PICT 2019-01664)からの助成金によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 10x Phosphate-Buffered Saline (PBS) | Corning | 46-013-CM | |
| 12 mm round coverslips | HDA | CBR_OBJ_6467 | |
| 24 Well- Cell Culture Plate | Sorfa | 220300 | |
| Absolute ethanol | Biopack | 2207.10.00 | |
| Alexa Fluor 594 Donkey anti-rabbit IgG (H+L) | Invitrogen | R37119 | |
| Confocal Laser Scanning Microscope | Zeiss | LSM 800 | |
| DAPI (4',6-diamidino-2-phenylindole, dihydrochloride) | Invitrogen | 62248 | |
| Dexamethasone | Sigma Aldrich | D4902 | |
| DMEN | Sartorius | 01-052-1A | |
| Fetal Bovine Serum | NATOCOR | Lintc-634 | |
| Gemcitabina | Eli Lilly | VL7502 | |
| LC3B (D11) XP Rabbit mAb | Cell Signaling Technology | 3868S | |
| Methanol | Anedra | 6197 | |
| Parafilm "M" (Laboratory Sealing Film) | Bemis/Curwood | PM-996 | |
| Pen-Strep Solution | Sartorius | 03-031-1B | |
| PP242 | Santa Cruz Biotechnology | SC-301606 | |
| Trypsin EDTA | Gibco | 11570626 |
参考文献
- Grasso, D., Renna, F. J., Vaccaro, M. I. Initial steps in mammalian autophagosome biogenesis. Frontiers in Cell and Developmental Biology. 6, 146 (2018).
- Galluzzi, L., et al. Molecular definitions of autophagy and related processes. EMBO Journal. 36 (13), 1811-1836 (2017).
- Kitada, M., Koya, D. Autophagy in metabolic disease and ageing. Nature Reviews Endocrinology. 17 (11), 647-661 (2021).
- Ktistakis, N. T., Tooze, S. A. Digesting the expanding mechanisms of autophagy. Trends in Cell Biology. 26 (8), 624-635 (2016).
- Tanida, I., Ueno, T., Kominami, E. LC3 and autophagy. Methods in Molecular Biology. 445, 77-88 (2008).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO Journal. 19 (21), 5720-5728 (2000).
- Mizushima, N., Yoshimori, T. How to interpret LC3 immunoblotting. Autophagy. 3 (6), 542-545 (2007).
- Vanasco, V., et al. Mitochondrial dynamics and VMP1-related selective mitophagy in experimental acute pancreatitis. Frontiers in Cell and Developmental Biology. 9, 640094 (2021).
- Williams, J. A. Regulatory mechanisms in pancreas and salivary acini. Annual Review of Physiology. 46, 361-375 (1984).
- Mizrahi, J. D., Surana, R., Valle, J. W., Shroff, R. T. Pancreatic cancer. Lancet. 395 (10242), 2008-2020 (2020).
- Li, J., et al. Regulation and function of autophagy in pancreatic cancer. Autophagy. 17 (11), 3275-3296 (2021).
- Ropolo, A., et al. A novel E2F1-EP300-VMP1 pathway mediates gemcitabine-induced autophagy in pancreatic cancer cells carrying oncogenic KRAS. Frontiers in Endocrinology. 11, 411 (2020).
- Pardo, R., et al. Gemcitabine induces the VMP1-mediated autophagy pathway to promote apoptotic death in human pancreatic cancer cells. Pancreatology. 10 (1), 19-26 (2010).
- Logsdon, C. D., Moessner, J., Williams, J. A., Goldfine, I. D. Glucocorticoids increase amylase mRNA levels, secretory organelles, and secretion in pancreatic acinar AR42J cells. Journal of Cell Biology. 100 (4), 1200-1208 (1985).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (4th edition). Autophagy. 17 (1), 1 (2021).
- Segeritz, C. -. P., Vallier, L., Jalali, M., Saldanha, F. Y. L., Jalali, M. Chapter 9 - Cell culture: Growing cells as model systems in vitro. Basic Science Methods for Clinical Researchers. , 151-172 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Current Protocols in Cytometry. 92 (1), 68 (2020).
- Grasso, D., et al. a novel selective autophagy pathway mediated by VMP1-USP9x-p62, prevents pancreatic cell death. Journal of Biological Chemistry. 286 (10), 8308-8324 (2011).
- Ropolo, A., et al. The pancreatitis-induced vacuole membrane protein 1 triggers autophagy in mammalian cells. Journal of Biological Chemistry. 282 (51), 37124-37133 (2007).
- Karanasios, E., Stapleton, E., Walker, S. A., Manifava, M., Ktistakis, N. T. Live cell imaging of early autophagy events: Omegasomes and beyond. Journal of Visualized Experiments. (77), e50484 (2013).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Zhang, Z., Singh, R., Aschner, M. Methods for the detection of autophagy in mammalian cells. Current Protocols in Toxicology. 69, 1-26 (2016).
- Yoshii, S. R., Mizushima, N. Monitoring and measuring autophagy. International Journal of Molecular Sciences. 18 (9), 1865 (2017).
- Betriu, N., Andreeva, A., Semino, C. E. Erlotinib promotes ligand-induced EGFR degradation in 3D but not 2D cultures of pancreatic ductal adenocarcinoma cells. Cancers. 13 (18), 4504 (2021).
- Wang, W., Dong, L., Zhao, B., Lu, J., Zhao, Y. E-cadherin is downregulated by microenvironmental changes in pancreatic cancer and induces EMT. Oncology Reports. 40 (3), 1641-1649 (2018).
- Kim, S. K., et al. Phenotypic heterogeneity and plasticity of cancer cell migration in a pancreatic tumor three-dimensional culture model. Cancers. 12 (5), 1305 (2020).
- Meng, Y., et al. Cytoplasmic EpCAM over-expression is associated with favorable clinical outcomes in pancreatic cancer patients with Hepatitis B virus negative infection. International Journal of Clinical and Experimental Medicine. 8 (12), 22204-22216 (2015).
- Brock, R., Hamelers, I. H., Jovin, T. M. Comparison of fixation protocols for adherent cultured cells applied to a GFP fusion protein of the epidermal growth factor receptor. Cytometry. 35 (4), 353-362 (1999).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved