Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Evaluierung der Autophagie in zwei verschiedenen Pankreaszellmodellen mittels LC3-Immunfluoreszenz
In diesem Artikel
Zusammenfassung
Das Ziel dieses Protokolls ist es, die autophagischen Konzentrationen bei Bauchspeicheldrüsenkrebs und Pankreasazinuszellen durch LC3-Immunfluoreszenz und LC3-Punktquantifizierung zu bestimmen.
Zusammenfassung
Autophagie ist ein spezialisierter kataboler Prozess, bei dem zytoplasmatische Komponenten, einschließlich Proteine und beschädigte Organellen, selektiv abgebaut werden. Autophagie ermöglicht es Zellen, physiologisch auf Stressreize zu reagieren und so die zelluläre Homöostase aufrechtzuerhalten. Krebszellen könnten ihre Autophagie modulieren, um sich an widrige Bedingungen wie Hypoxie, Nährstoffmangel oder Schäden durch Chemotherapie anzupassen. Das duktale Pankreas-Adenokarzinom ist eine der tödlichsten Krebsarten. Pankreaskarzinomzellen weisen aufgrund der transkriptionellen Hochregulation und posttranslationalen Aktivierung von Autophagieproteinen eine hohe Autophagieaktivität auf.
Hier wurde die PANC-1-Zelllinie als Modell für menschliche Krebszellen der Bauchspeicheldrüse und die AR42J-Pankreas-Azinuszelllinie als physiologisches Modell für hochdifferenzierte Säugetierzellen verwendet. In dieser Studie wurde die Immunfluoreszenz der Mikrotubuli-assoziierten Protein-Leichtkette 3 (LC3) als Indikator für den Status der Autophagie-Aktivierung verwendet. LC3 ist ein Autophagieprotein, das unter basalen Bedingungen ein diffuses Verteilungsmuster im Zytoplasma aufweist (in diesem Zustand als LC3-I bekannt). Die Autophagie-Induktion löst die Konjugation von LC3 mit Phosphatidylethanolamin auf der Oberfläche neu gebildeter Autophagosomen aus, um LC3-II zu bilden, ein membrangebundenes Protein, das die Bildung und Expansion von Autophagosomen unterstützt. Um die Anzahl der markierten autophagischen Strukturen zu quantifizieren, wurde die Open-Source-Software FIJI mit Hilfe des Tools "3D Objects Counter" eingesetzt.
Die Messung der autophagischen Konzentrationen sowohl unter physiologischen Bedingungen als auch in Krebszellen ermöglicht es uns, die Modulation der Autophagie unter verschiedenen Bedingungen wie Hypoxie, Chemotherapie oder dem Knockdown bestimmter Proteine zu untersuchen.
Einleitung
Makroautophagie (allgemein als Autophagie bezeichnet) ist ein spezialisierter kataboler Prozess, bei dem zytoplasmatische Komponenten, einschließlich Proteine und beschädigte Organellen, selektiv abgebaut werden 1,2. Autophagie ermöglicht es Zellen, physiologisch auf Stressreize zu reagieren und so die zelluläre Homöostase aufrechtzuerhalten3. Bei der Autophagie entsteht ein Doppelmembranvesikel: das Autophagosom. Das Autophagosom enthält die Frachtmoleküle und treibt sie zum Abbau in das Lysosom 1,4.
Autophagosomen werden durch das autophagische Protein Mikrotubuli-assoziiertes Protein Light Chain 3 (LC3)5 dekoriert. Wenn keine Autophagie induziert wird, diffundiert LC3 im Zytoplasma und Zellkern in der LC3-I-Konformation. Wenn andererseits Autophagie induziert wird, wird LC3 mit einem Phosphatidylethanolamin in der Membran der autophagischen Strukturenkonjugiert 6. Diese neue LC3-Konformation wird als LC3-II1 bezeichnet. Die Konformationsverschiebung von LC3 führt zu Veränderungen in der zellulären Lokalisation und der Migration der Dodecylnatriumsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE), die durch Techniken wie Immunfluoreszenz und Western Blot 5,7 nachgewiesen werden können. Auf diese Weise ist die LC3-Konjugation ein Schlüsselereignis im autophagischen Prozess, das zur Messung der autophagischen Aktivität verwendet werden kann.
Die Azinuszelle der Bauchspeicheldrüse ist eine hochdifferenzierte Zelle, die unter gesunden Bedingungen eine geringe Autophagierate aufweist. Unter verschiedenen physiologischen Bedingungen oder unter pharmakologischer Stimulation können sie jedoch die Autophagie aktivieren. Daher ist die Bestimmung der autophagischen Konzentrationen in dieser Zelllinie nützlich, um die möglichen direkten oder indirekten Auswirkungen verschiedener pharmakologischer oder biologischer Wirkstoffe auf die Autophagie zu untersuchen 8,9.
Das duktale Pankreas-Adenokarzinom ist aufgrund seiner späten Diagnose und seiner hohen Chemotherapieresistenz eine der tödlichsten Krebsarten10. Pankreaskarzinomzellen weisen aufgrund der transkriptionellen Hochregulation und posttranslationalen Aktivierung von Autophagie-verwandten Proteinen eine hohe Autophagieaktivität auf11. Bauchspeicheldrüsenkrebszellen können ihre Autophagie als Reaktion auf ungünstige Bedingungen wie Hypoxie, Nährstoffmangel oder Chemotherapie-induzierte Schäden anpassen11. Daher kann die Analyse des Autophagieniveaus in Bauchspeicheldrüsenkrebszellen helfen zu verstehen, wie sie sich an unterschiedliche Umgebungen anpassen, und die Wirksamkeit von autophagiemodulierenden Behandlungen bewerten.
Diese Studie zeigt eine Methode zur Durchführung von LC3-Immunfluoreszenz in zwei verschiedenen zellulären Modellen der Bauchspeicheldrüse. Das erste Modell, PANC-1-Zellen, diente als Modell für das duktale Adenokarzinom der Bauchspeicheldrüse. Diese Zellen wurden mit Gemcitabin behandelt, einem Chemotherapeutikum, von dem bereits gezeigt wurde, dass es Autophagie induziert, insbesondere in Bauchspeicheldrüsenkrebszellen, die das onkogene Kirsten-Rattensarkomvirus-Gen (KRAS) tragen12,13. Das zweite Modell, AR42J-Zellen, diente als physiologischeres Modell exokriner Pankreaszellen. Diese Zellen wurden mit Dexamethason differenziert, um den azinären Pankreaszellen ähnlicher zu werden14. In diesen Zellen wurde die Autophagie pharmakologisch durch die Verwendung von PP242 induziert, das ein potenter mTOR-Inhibitor ist15. In dieser Studie demonstrieren wir die Anwendbarkeit des beschriebenen Protokolls mit zwei verschiedenen Pankreasmodellen und seine Fähigkeit, zwischen Zuständen niedriger und hoher Autophagie zu unterscheiden.
Protokoll
1. Zellvorbereitung
- Tränken Sie 12 mm runde Deckgläser in absolutem Ethanol und legen Sie sie senkrecht in die Vertiefungen einer 24-Well-Platte.
- Entfernen Sie die Abdeckung und setzen Sie die Multi-Well-Platte 15 Minuten lang ultravioletter Strahlung aus.
- Positionieren Sie die Deckgläser horizontal und waschen Sie sie mit Dulbeccos Modified Eagle Medium (DMEM).
- Säen Sie eine geringe Anzahl von Pankreaszellen. Die Menge sollte so angepasst werden, dass am Tag der Fixierung eine Konfluenz von 50%-75% erreicht wird16.
HINWEIS: Es wird empfohlen, 2,5 × 10 4 PANC-1 oder 4 × 104 AR42J-Zellen pro Vertiefung zu säen, um die Zellen nach 3 Tagen zu fixieren. - Kultur der Zellen in DMEM mit 10 % fötalem Kälberserum, 100 U/ml Penicillin und 100 μg/ml Streptomycin in einem Inkubator bei 37 °C unter befeuchteter Atmosphäre mit 5 % Kohlendioxid (CO2).
HINWEIS: Für PANC-1-Zellen wird empfohlen, die Zellen zwischen der Zellaussaat und den folgenden Schritten 2 Tage lang zu inkubieren. Nach dieser Zeit können die Zellen transfiziert, behandelt oder fixiert werden. Dieses Protokoll veranschaulicht beispielhaft die Behandlung mit Gemcitabin in nicht-transfizierten PANC-1-Zellen und die Differenzierung und PP242-Behandlung bei nicht-transfizierten AR42J-Zellen.
2. Behandlung der Zellen
- Gemcitabin-Behandlung für PANC-1-Zellen
- Bereiten Sie 2 Tage nach der Aussaat eine Lösung von 1 μg/μl Gemcitabin in DMEM vor. Behandeln Sie jede Vertiefung mit 2,6 μl der 1 μg/μl Gemcitabinlösung, um eine endgültige Verdünnung von 20 μM zu erreichen.
- Inkubieren Sie die Zellen für 24 Stunden im Inkubator.
- AR42J-Differenzierung und PP242-Behandlung
- Bereiten Sie eine Lösung von 4 μg/ml Dexamethason in DMEM vor.
- Behandeln Sie jede Vertiefung mit 4,9 μl 4 μg/ml Dexamethasonlösung, um eine endgültige Verdünnung von 100 nM zu erhalten.
- Inkubieren Sie die Zellen für 48 Stunden im Inkubator.
- Entfernen Sie das Medium und behandeln Sie jede Vertiefung mit 0,5 μl 1 mM PP242, um eine endgültige Verdünnung von 1 μM zu erhalten.
- Inkubieren Sie die Zellen für 2 Stunden im Inkubator.
3. Fixierung und Permeabilisierung der Zellen
- Bereiten Sie eine 24-Well-Platte mit kaltem Methanol und eine 6-Well-Platte mit kaltphosphatgepufferter Kochsalzlösung (PBS; 137 mM NaCl, 2,7 mM KCl, 8 mM Na2HPO4, 2 mMKH2PO4) vor. Halte sie auf Eis.
- Nehmen Sie jedes Deckglas mit einer Pinzette, waschen Sie es zweimal in PBS und inkubieren Sie es 6 Minuten lang in Methanol.
4. Blockieren der Zellen
- Waschen Sie jedes Deckglas zweimal in PBS und inkubieren Sie es 1 h lang in 10%igem fötalem Kälberserum in PBS (Blockierlösung).
HINWEIS: In diesem Schritt kann das Protokoll angehalten werden. Die Deckgläser können über Nacht im Kühlschrank in der Blockierlösung aufbewahrt werden, und das Protokoll kann am nächsten Tag fortgesetzt werden.
5. Inkubation der Deckgläser mit dem Primärantikörper
- Bereiten Sie eine 1:1.000-Lösung von Anti-LC3 in der Blockierlösung vor und halten Sie sie auf Eis.
- Legen Sie ein Stück Laborversiegelungsfolie über den Multiwell-Deckel.
- Geben Sie einen Tropfen (25 μl) pro Deckglas der Anti-LC3-Lösung auf die Versiegelungsfolie.
- Nehmen Sie jedes Deckglas mit einer Pinzette und legen Sie es über den primären Antikörpertropfen, wobei Sie darauf achten sollten, dass die Zellseite mit der Lösung in Kontakt kommt.
- Bereiten Sie eine feuchte Kammer vor, indem Sie ein feuchtes Stück Papier in eine Plastikbox mit flachem Boden legen.
- Legen Sie die Multiwell-Platte in die Feuchtigkeitskammer, decken Sie sie mit Folie ab und inkubieren Sie sie über Nacht im Kühlschrank.
6. Inkubation der Deckgläser mit dem Sekundärantikörper
- Nehmen Sie die Multi-Well-Platte aus der Feuchtigkeitskammer und legen Sie die Deckgläser wieder in die Multi-Well-Platte.
- Führen Sie drei Wäschen mit PBS durch.
- Bereiten Sie eine Lösung aus fluoreszenzmarkiertem Anti-Kaninchen mit einer Verdünnung von 1:800 in der Blockierlösung vor und bewahren Sie sie lichtgeschützt auf Eis auf.
- Legen Sie ein Stück Siegelfolie über den Multiwell-Deckel.
- Geben Sie einen Tropfen (25 μl) pro Deckglas der Anti-Kaninchen-Lösung auf die Versiegelungsfolie.
- Nehmen Sie jedes Deckglas mit einer Pinzette und legen Sie es über den primären Antikörpertropfen, wobei Sie darauf achten sollten, dass die Zellseite mit der Lösung in Kontakt kommt.
- Inkubieren Sie die Multiwell-Platte in der Feuchtekammer für 2 h bei Raumtemperatur (RT) lichtgeschützt.
7. Färbung der Zellen mit 4′,6-Diamidino-2-phenylindol (DAPI)
- Nehmen Sie die Multi-Well-Platte aus der Feuchtigkeitskammer und legen Sie die Deckgläser wieder in die Multi-Well-Platte.
- Führen Sie drei Wäschen mit PBS durch.
- Bereiten Sie eine 300-nM-DAPI-Lösung in PBS (lichtgeschützt) vor.
- Inkubieren Sie jedes Deckglas 10 Minuten lang mit der DAPI-Lösung.
- Führen Sie drei Wäschen mit PBS durch. Bewahren Sie die Multi-Well-Platte vor Licht geschützt auf.
8. Montage
- Bereiten Sie zwei Becher mit Wasser und einem Blatt Papier vor.
- Geben Sie einen Tropfen (10 μl) pro Deckglas einer Polyvinylalkohol-Bis(trimethylalumin)-1,4-diazabicyclo[2.2.2]octan-Addukt-Lösung (PVA-DABCO) auf einen Objektträger.
HINWEIS: PVA-DABCO wird durch die Kombination von 0,25 M DABCO, 10 % W/V PVA, 20 % Glycerin und 50 % Tris HCl (1,5 M, pH 8,8) in Reinstwasser hergestellt. - Nehmen Sie jedes Deckglas mit einer Pinzette, waschen Sie es in jedem Wasserbecher, trocknen Sie es im Papier ab und legen Sie es über den PVA-DABCO-Tropfen (wobei die Zellen mit der Lösung in Kontakt kommen).
- Über Nacht lichtgeschützt trocknen lassen.
9. Konfokale Mikroskopiebetrachtung und Bildaufnahme
- Visualisieren Sie die Deckgläser in einem inversen konfokalen Mikroskop mit einem Objektiv von etwa 63x17.
- Nehmen Sie repräsentative Bilder der markierten Zellen auf.
10. Quantifizierung der LC3-Punkte
- Ziehen Sie jede Bilddatei, die die erfassten Kanäle enthält, z. B. ".czi", per Drag & Drop in den ImageJ (FIJI)-Bildschirm, um sie zu öffnen. Klicken Sie im Dialogfeld auf OK und schließen Sie das Konsolenfenster .
- Wählen Sie auf der Registerkarte " Bild " die Option " Farbe" > "Kanäle teilen" aus.
- Schließen Sie die Bilder, die den anderen Kanälen als dem LC3-Bild entsprechen.
- Wählen Sie auf der Registerkarte Bild die Option > Farbbalance anpassen aus
- Verschieben Sie den Schieberegler "Maximum " nach links, bis das Bild gesättigt ist, um die Zellenkonturen zu visualisieren.
- Zeichnen Sie den Zellenumriss mit dem Freihandauswahl-Werkzeug .
- Klicken Sie auf die Schaltfläche Zurücksetzen , um die Farbanpassung zurückzusetzen.
- Wählen Sie auf der Registerkarte " Bearbeiten " die Option "Ausschneiden " aus, um das ausgewählte Element auszuschneiden.
- Schließen Sie das Bild, ohne es zu speichern.
- Wählen Sie auf der Registerkarte " Bearbeiten " die Option " Einfügen" aus.
- Wählen Sie im Menü " Analysieren " das Werkzeug " Zähler für 3D-Objekte" aus.
- Legen Sie den Schwellenwert fest. Im Beispiel dieser Studie ist der Schwellenwert auf 2.000 festgelegt.
- Legen Sie den Größenfilter fest. In dieser Studie liegt sie zwischen 50 und 500.
- Achten Sie darauf, dass die Kästchen Objekte und Zusammenfassung markiert sind.
- Klicken Sie auf OK. Die Anzahl der Punkte wird in der Zusammenfassung als erkannte Objekte beschrieben.
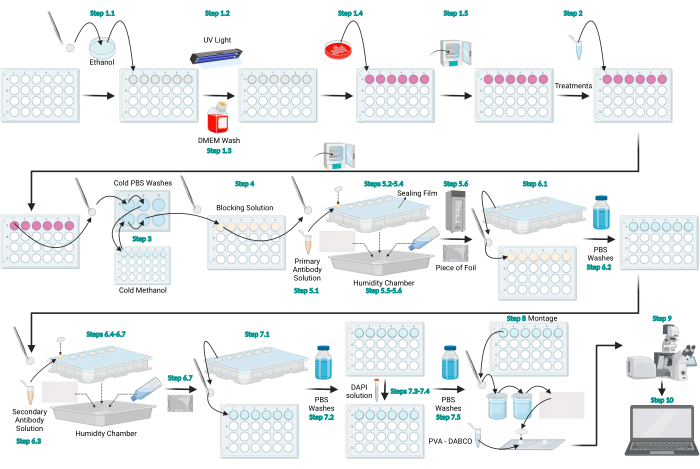
Abbildung 1: Schematische Darstellung des LC3-Immunfluoreszenzprotokolls. Schematische Darstellung, die das allgemeine Protokoll für die LC3-Immunfluoreszenz darstellt. Mit BioRender.com erstellte Figur. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergebnisse
Dieses Protokoll führt die Immunfluoreszenz von LC3 in Pankreaszelllinien durch, um die Autophagieniveaus unter verschiedenen Bedingungen zu bestimmen. Das Ergebnis dieses Experiments war die Gewinnung zellulärer Bilder aus dem roten und blauen Kanal, die LC3 und DAPI entsprechen. Die LC3-Bilder zeigen die zelluläre Verteilung dieses Proteins, während die DAPI die Kernlokalisation zeigt. Abbildung 2A zeigt ein repräsentatives Bild der Immunfluoreszenz von LC3 und seiner Verschmelzung mi...
Diskussion
Die in diesem Protokoll beschriebene Methode ermöglicht es, die endogene LC3-Verteilung in der Zelle zu visualisieren und die autophagischen Konzentrationen unter verschiedenen Bedingungen zu quantifizieren. Eine weitere ähnliche Methode zur Analyse der LC3-Verteilung und zur Bestimmung der Autophagieaktivierung ist die fluoreszenzmarkierte LC3-Transfektion (z. B. RFP-LC3)19. Die RFP-LC3-Transfektion hat den Vorteil, dass keine Fixierung erforderlich ist (was die Anwendung dieser Methode in der ...
Offenlegungen
Es wurden keine Interessenkonflikte erklärt.
Danksagungen
Diese Arbeit wurde durch Zuschüsse der Universität Buenos Aires (UBACyT 2018-2020 20020170100082BA), des Nationalen Rates für wissenschaftliche Forschung und Technologie (CONICET) (PIP 2021-2023 GI− 11220200101549CO; und PUE 22920170100033) und der Nationalen Agentur für wissenschaftliche und technologische Förderung (PICT 2019-01664) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 10x Phosphate-Buffered Saline (PBS) | Corning | 46-013-CM | |
| 12 mm round coverslips | HDA | CBR_OBJ_6467 | |
| 24 Well- Cell Culture Plate | Sorfa | 220300 | |
| Absolute ethanol | Biopack | 2207.10.00 | |
| Alexa Fluor 594 Donkey anti-rabbit IgG (H+L) | Invitrogen | R37119 | |
| Confocal Laser Scanning Microscope | Zeiss | LSM 800 | |
| DAPI (4',6-diamidino-2-phenylindole, dihydrochloride) | Invitrogen | 62248 | |
| Dexamethasone | Sigma Aldrich | D4902 | |
| DMEN | Sartorius | 01-052-1A | |
| Fetal Bovine Serum | NATOCOR | Lintc-634 | |
| Gemcitabina | Eli Lilly | VL7502 | |
| LC3B (D11) XP Rabbit mAb | Cell Signaling Technology | 3868S | |
| Methanol | Anedra | 6197 | |
| Parafilm "M" (Laboratory Sealing Film) | Bemis/Curwood | PM-996 | |
| Pen-Strep Solution | Sartorius | 03-031-1B | |
| PP242 | Santa Cruz Biotechnology | SC-301606 | |
| Trypsin EDTA | Gibco | 11570626 |
Referenzen
- Grasso, D., Renna, F. J., Vaccaro, M. I. Initial steps in mammalian autophagosome biogenesis. Frontiers in Cell and Developmental Biology. 6, 146 (2018).
- Galluzzi, L., et al. Molecular definitions of autophagy and related processes. EMBO Journal. 36 (13), 1811-1836 (2017).
- Kitada, M., Koya, D. Autophagy in metabolic disease and ageing. Nature Reviews Endocrinology. 17 (11), 647-661 (2021).
- Ktistakis, N. T., Tooze, S. A. Digesting the expanding mechanisms of autophagy. Trends in Cell Biology. 26 (8), 624-635 (2016).
- Tanida, I., Ueno, T., Kominami, E. LC3 and autophagy. Methods in Molecular Biology. 445, 77-88 (2008).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO Journal. 19 (21), 5720-5728 (2000).
- Mizushima, N., Yoshimori, T. How to interpret LC3 immunoblotting. Autophagy. 3 (6), 542-545 (2007).
- Vanasco, V., et al. Mitochondrial dynamics and VMP1-related selective mitophagy in experimental acute pancreatitis. Frontiers in Cell and Developmental Biology. 9, 640094 (2021).
- Williams, J. A. Regulatory mechanisms in pancreas and salivary acini. Annual Review of Physiology. 46, 361-375 (1984).
- Mizrahi, J. D., Surana, R., Valle, J. W., Shroff, R. T. Pancreatic cancer. Lancet. 395 (10242), 2008-2020 (2020).
- Li, J., et al. Regulation and function of autophagy in pancreatic cancer. Autophagy. 17 (11), 3275-3296 (2021).
- Ropolo, A., et al. A novel E2F1-EP300-VMP1 pathway mediates gemcitabine-induced autophagy in pancreatic cancer cells carrying oncogenic KRAS. Frontiers in Endocrinology. 11, 411 (2020).
- Pardo, R., et al. Gemcitabine induces the VMP1-mediated autophagy pathway to promote apoptotic death in human pancreatic cancer cells. Pancreatology. 10 (1), 19-26 (2010).
- Logsdon, C. D., Moessner, J., Williams, J. A., Goldfine, I. D. Glucocorticoids increase amylase mRNA levels, secretory organelles, and secretion in pancreatic acinar AR42J cells. Journal of Cell Biology. 100 (4), 1200-1208 (1985).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (4th edition). Autophagy. 17 (1), 1 (2021).
- Segeritz, C. -. P., Vallier, L., Jalali, M., Saldanha, F. Y. L., Jalali, M. Chapter 9 - Cell culture: Growing cells as model systems in vitro. Basic Science Methods for Clinical Researchers. , 151-172 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Current Protocols in Cytometry. 92 (1), 68 (2020).
- Grasso, D., et al. a novel selective autophagy pathway mediated by VMP1-USP9x-p62, prevents pancreatic cell death. Journal of Biological Chemistry. 286 (10), 8308-8324 (2011).
- Ropolo, A., et al. The pancreatitis-induced vacuole membrane protein 1 triggers autophagy in mammalian cells. Journal of Biological Chemistry. 282 (51), 37124-37133 (2007).
- Karanasios, E., Stapleton, E., Walker, S. A., Manifava, M., Ktistakis, N. T. Live cell imaging of early autophagy events: Omegasomes and beyond. Journal of Visualized Experiments. (77), e50484 (2013).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Zhang, Z., Singh, R., Aschner, M. Methods for the detection of autophagy in mammalian cells. Current Protocols in Toxicology. 69, 1-26 (2016).
- Yoshii, S. R., Mizushima, N. Monitoring and measuring autophagy. International Journal of Molecular Sciences. 18 (9), 1865 (2017).
- Betriu, N., Andreeva, A., Semino, C. E. Erlotinib promotes ligand-induced EGFR degradation in 3D but not 2D cultures of pancreatic ductal adenocarcinoma cells. Cancers. 13 (18), 4504 (2021).
- Wang, W., Dong, L., Zhao, B., Lu, J., Zhao, Y. E-cadherin is downregulated by microenvironmental changes in pancreatic cancer and induces EMT. Oncology Reports. 40 (3), 1641-1649 (2018).
- Kim, S. K., et al. Phenotypic heterogeneity and plasticity of cancer cell migration in a pancreatic tumor three-dimensional culture model. Cancers. 12 (5), 1305 (2020).
- Meng, Y., et al. Cytoplasmic EpCAM over-expression is associated with favorable clinical outcomes in pancreatic cancer patients with Hepatitis B virus negative infection. International Journal of Clinical and Experimental Medicine. 8 (12), 22204-22216 (2015).
- Brock, R., Hamelers, I. H., Jovin, T. M. Comparison of fixation protocols for adherent cultured cells applied to a GFP fusion protein of the epidermal growth factor receptor. Cytometry. 35 (4), 353-362 (1999).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten