Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes de calcio en el colículo superior de ratón
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo detalla el procedimiento para obtener imágenes de las respuestas de calcio en el colículo superior (SC) de ratones despiertos, incluida la obtención de imágenes de la actividad de una sola neurona con microscopía de dos fotones mientras se deja la corteza intacta en ratones de tipo salvaje, y la obtención de imágenes de todo el SC con microscopía de campo amplio en ratones mutantes de corteza parcial.
Resumen
El colículo superior (SC), una estructura evolutivamente conservada del mesencéfalo en todos los vertebrados, es el centro visual más sofisticado antes de la aparición de la corteza cerebral. Recibe entradas directas de ~ 30 tipos de células ganglionares de la retina (RGC), cada una de las cuales codifica una característica visual específica. Sigue siendo difícil de entender si el SC simplemente hereda las características de la retina o si se produce un procesamiento adicional y potencialmente de novo en el SC. Para revelar la codificación neuronal de la información visual en el SC, proporcionamos aquí un protocolo detallado para registrar ópticamente las respuestas visuales con dos métodos complementarios en ratones despiertos. Un método utiliza la microscopía de dos fotones para obtener imágenes de la actividad del calcio con una resolución de una sola célula sin ablacionar la corteza superpuesta, mientras que el otro utiliza la microscopía de campo amplio para obtener imágenes de todo el SC de un ratón mutante cuya corteza está en gran parte subdesarrollada. Este protocolo detalla estos dos métodos, incluida la preparación de animales, la inyección viral, la implantación de la placa de cabeza, la implantación del tapón, la adquisición de datos y el análisis de datos. Los resultados representativos muestran que las imágenes de calcio de dos fotones revelan respuestas neuronales evocadas visualmente a una resolución de una sola célula, y las imágenes de calcio de campo amplio revelan actividad neuronal en todo el SC. Al combinar estos dos métodos, se puede revelar la codificación neuronal en el SC a diferentes escalas, y dicha combinación también se puede aplicar a otras regiones del cerebro.
Introducción
El colículo superior (SC) es un centro visual importante en todos los vertebrados. En los mamíferos, recibe información directa de la retina y la corteza visual1. Si bien el registro óptico se ha aplicado ampliamente a la corteza 2,3,4,5, su aplicación en el SC se ve obstaculizada por accesos ópticos deficientes 6,7,8,9,10,11,12,13,14,15,16,17 ,18,19. El objetivo de este protocolo es proporcionar detalles sobre dos métodos complementarios para el registro óptico de la actividad neuronal en el SC.
El SC se encuentra debajo de la corteza y el seno transverso, lo que limita el acceso óptico a las neuronas coliares. Un enfoque para superar esta limitación es aspirar la corteza suprayacente y exponer el SCanterolateral 7,9,10,13,14,19. Sin embargo, debido a que el SC recibe entradas corticales, tal operación podría afectar la forma en que las neuronas SC responden a los estímulos visuales. Para superar esta limitación, detallamos aquí un protocolo alternativo para obtener imágenes de la capa superficial del SC posterior-medial con un tapón de silicio, dejando intacta la corteza 8,11. Específicamente, para lograr la resolución de una sola célula, aplicamos microscopía de dos fotones para obtener imágenes de las respuestas de calcio en el SC posterior-medial de ratones de tipo salvaje. Además, para lograr una amplia cobertura, aplicamos microscopía de campo amplio para obtener imágenes de todo el SC de un ratón mutante cuya corteza posterior no se ha desarrollado20.
Los dos métodos descritos en este protocolo son complementarios entre sí. Las imágenes de calcio de dos fotones sin ablar la corteza son apropiadas para registrar la actividad neuronal a una resolución de una sola célula con entradas corticales intactas. Las imágenes de calcio de campo amplio son apropiadas para registrar la actividad neuronal en todo el SC al tiempo que sacrifican la resolución espacial.
Protocolo
Todos los procedimientos experimentales se realizaron de acuerdo con las directrices de bienestar animal y fueron aprobados por la IACUC en el Instituto Chino de Investigación del Cerebro, Beijing.
NOTA: El cronograma para este protocolo es el siguiente: 1) hacer la ventosa; 2) inyectar el virus; 3) implantar la placa de la cabeza; 4) después de 3 semanas, implantar el tapón; 5) Después de una recuperación de ~ 3 días y habituación en la cinta de correr, realice imágenes de dos fotones / campo amplio.
1. Preparación de una ventosa (Figura 1A)
- Depositar una gota de solución salina tamponada con fosfato (PBS, 1x) en una placa acrílica y tocarla con una aguja plana de 21 G para rellenar la punta por capilaridad.
- Cubra la punta de la aguja con un adhesivo de silicona translúcido y colóquela durante aproximadamente ~ 10 minutos.
- Corta el adhesivo de silicona con unas tijeras hasta formar un disco de ~2 mm de diámetro.
2. Preparación de los tapones (Figura 1B)
- Prepara una cuña de plástico de 0,75 mm de grosor y recorta un cuadrado de 1 cm x 1 cm en el centro. Prepara dos bloques acrílicos. Limpie la cuña y los bloques acrílicos con alcohol y límpielos con papel para lentes.
- Coloque la cuña en un bloque acrílico, deposite el adhesivo de silicona en el centro para llenar ~ 90% de la abertura (evite las burbujas), agregue otro bloque acrílico y ~ 1 kg de fuerza, y espere 20 min.
- Bajo un microscopio quirúrgico, corte el adhesivo de silicona con un bisturí a prismas triangulares de 1 mm de alto por 1,5 mm de ancho sobre una hoja de papel con triángulos impresos en ella.
- Retire el polvo de los prismas triangulares con cinta adhesiva de plástico y colóquelo en un cubreobjetos de vidrio de 5 mm de diámetro y 0,15 mm de grosor.
- Coloque el cubreobjetos con los prismas triangulares debajo de un tratador de corona, encienda el tratante de corona y acérquelo al cubreobjetos hasta que aparezca un rayo. Manténgalo en esa posición durante unos minutos hasta que estén unidos entre sí.
NOTA: Este paso puede ser diferente para otros tratantes de corona. - Coloque los tapones en una placa de Petri y colóquela en una incubadora a 70 °C durante la noche.
3. Preparación de los animales e inyección de la construcción viral
- Utilice ratones C57BL/6J (tipo salvaje) y Emx1-Cre:Pals1flox/wt (corteza parcial)20 de ambos sexos a las 6 semanas de edad (9 semanas en el momento de la obtención de imágenes).
- Esterilizar todos los materiales e instrumentos utilizados para el procedimiento quirúrgico.
- Anestesiar al ratón con isoflurano al 5% y luego mantener el isoflurano al 1%-2% con un caudal de 1 L/min durante la cirugía. Confirme el nivel de anestesia por la falta de reflejo de pellizco de los dedos de los pies.
- Fije la cabeza del mouse en un marco estereotáxico con barras en las orejas. A lo largo de la cirugía, coloque una pomada oftálmica en los ojos del ratón para evitar que se seque y mantenga su temperatura corporal a 37 °C con una almohadilla térmica termostática.
- Afeitar el pelaje y esterilizar la superficie de la piel con yodóforo y etanol al 75%. Inyecte lidocaína (5 mg/kg, inyección subcutánea [SQ]), tilidina (2 mg/kg, SQ) y meloxicam (2 mg/kg, SQ).
NOTA: El procedimiento quirúrgico para la esterilización, la analgesia y la anestesia local puede diferir de una institución a otra. - Retire la piel sobre el SC con unas tijeras quirúrgicas para exponer el cráneo. Limpie el cráneo con un hisopo de algodón.
- Ajuste el marco estereotáxico hasta que la superficie del cráneo esté plana. Taladre un orificio de 0,5 mm lateral y 0,42 mm anterior a la lambda hasta que la duramadre quede expuesta.
- Inyectar virus adenoasociados (AAV) que expresen GCaMP6m (rAAV2/9-hsyn-GCaMP6m; 1 x10 13 GC/mL disuelto en 1x PBS) a profundidades de 1 mm y 1,6 mm de la lambda con una micropipeta de vidrio biselado (la punta está biselada a 25° con un diámetro de 40-50 μm).
- En cada profundidad, inyecte 100 nL a una velocidad de 50 nL/min y espere 5 min después de cada inyección. Después de la inyección, retire lentamente el inyector.
4. Implantación de la placa de cabeza
- Limpie los músculos y tejidos sobre el cráneo y rasque el cráneo con una cuchilla. Si se produce sangrado, deténgalo con espuma de gel y limpie la sangre. Fije la placa de la cabeza (Figura 1C) al cráneo con un cemento de resina adhesivo dental autopolimerizable. Específicamente, mezcle 3/4 de cucharada de polímero, tres gotas de monómero y una gota de catalizador en un recipiente de cerámica con hielo. Aplica la mezcla en la placa de la cabeza y el cráneo. Únalos y espere 5 minutos para que el adhesivo se asiente.
- Inyecte antibióticos (ceftiofur sódico, 5 mg/kg, SQ) para prevenir infecciones. Retire el mouse del marco estereotáxico y colóquelo sobre una almohadilla térmica para recuperarlo. Devuélvalo a la jaula de la casa después de recuperarse de la anestesia. Para el manejo del dolor, proporcione agua potable que contenga ibuprofeno (0,5 ml/50 ml) al ratón durante 3 días después de la cirugía.
5. Implantación del tapón (Figura 2)
- Implante el tapón 3 semanas después de la inyección viral. Anestesiar y fijar el ratón en un marco estereotáxico, como se describe en los pasos 3.2-3.5. Prepare espuma de gel empapada en solución salina para detener posibles hemorragias.
- Taladre un óvalo de 3 mm x 2 mm con un microtaladro centrado a 0,5 mm posterior a la lambda. Adelgaza el límite del óvalo hasta que se agriete. Adelgace el cráneo frente a la ventana de grabación ovalada para hacer que el cubreobjetos esté cerca del SC.
- Retire los colgajos óseos con unas pinzas finas. Haga un corte en la duramadre posterior al seno transverso y retire el trozo de duramadre. Si es necesario, agregue líquido cefalorraquídeo artificial (ACSF, por sus siglas en inglés) al cerebro con una jeringa de 3 ml para evitar que se seque. Seque el cráneo y la ventana de grabación con espuma de gel e hisopos de algodón.
- Deposite una gota de adhesivo de silicona en la ventana de grabación. Utilice la ventosa para sujetar el tapón con presión negativa. Use un micromanipulador motorizado para mover la ventosa y baje el tapón en el adhesivo de silicona hasta que el cubreobjetos toque el cráneo. Luego, mueva el tapón hacia adelante ~ 1 mm para alejar el seno transverso y exponer el SC. Este paso debe realizarse en 1-2 minutos antes de que el adhesivo de silicona se vuelva pegajoso.
- Limpie la placa de la cabeza y aplique cianoacrilato de butilo y cemento de resina alrededor del límite del tapón para fijarlo a la placa de la cabeza.
NOTA: Proporcione cuidados postoperatorios, como se describe en el paso 4.2.
6. Imágenes de dos fotones (Figura 3)
- Fije la cabeza del animal en una cinta de correr giratoria con su placa para la cabeza. Coloque la cinta de correr bajo un microscopio de dos fotones y ajuste la altura a una posición adecuada. Coloque un cono de aluminio negro entre el objetivo y la placa frontal para evitar la contaminación lumínica del monitor para la estimulación visual. Habitúa al ratón durante ~15 min.
- Realice imágenes de dos fotones en el microscopio con un aumento de 16x, una apertura numérica (NA) de 0,8 y un objetivo de distancia de trabajo (WD) de 3 mm. Utilice un láser Ti:zafiro con técnica de bloqueo de modo controlado por dos escáneres galvo para excitar GCaMP6m a 920 nm. Ajuste la potencia del láser en el plano de muestra entre 20 y 80 mW.
- Escanee un campo de visión de 600 μm x 600 μm a 4,8 Hz con una resolución espacial de 2,4 μ m/píxel y actividad neuronal de la imagen a una profundidad de hasta 350 μ m.
NOTA: La frecuencia de muestreo y la resolución espacial son para escáneres galvo y deben ajustarse para escáneres resonantes. - Recoge la fluorescencia emitida con un espejo dicroico y detecta mediante un tubo fotomultiplicador (PMT) después de pasarla por un filtro de paso de banda.
- Registre la locomoción del ratón en la cinta de correr con un codificador rotatorio. Utilice una cámara con una lente de 50 mm para registrar el tamaño y la posición de su pupila. Sincronice estas grabaciones y adquisiciones de imágenes con la estimulación visual mediante la grabación de los disparadores enviados por la computadora de estimulación.
7. Análisis de las respuestas de calcio medidas mediante imágenes de dos fotones
- Corrija el movimiento del cerebro durante la obtención de imágenes con SIMA21 o NoRMCorre22.
- Dibuje manualmente una región de interés (ROI) con la herramienta Varita mágica de celda (ImageJ) y ajústela con una elipse en MATLAB. Extraiga los rastros de fluorescencia de cada ROI después de eliminar la contaminación por neuropil:
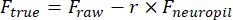
F true y F rawson las amplitudes de fluorescencia corregida y bruta del ROI, respectivamente, Fneuropiles la amplitud de fluorescencia del neuropilo circundante y r es el factor de contaminación del neuropilo desenfocado (0,7 para nuestra configuración). La r se estima midiendo las señales en un vaso sanguíneo para el cual Fverdadero = 0, como se describió anteriormente23,24. - Elimine las fluctuaciones lentas de la línea de base restando el valor del octavo percentil de una ventana de 15 s centrada en cada fotograma25.
- Defina las respuestas visuales de una neurona como el cambio relativo de la amplitud de fluorescencia en su ROI durante el período de estímulo:
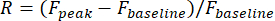
Elpico F es la amplitud máxima de la traza de fluorescencia durante la estimulación visual, y la línea debaseFes la amplitud media durante un período de 0,5 s antes de la estimulación.
8. Imágenes de campo amplio y análisis de datos (Figura 4)
- Construya un macroscopio de campo amplio con una lente tándem4 (Figura 4A). El macroscopio de lente tándem son dos lentes de cámara (50 mm y 105 mm) conectadas a través de un adaptador, y el aumento es de ~2x.
- Prepare ratones mutantes de corteza parcial como se describe en los pasos 3.2-3.5. Inyectar 100 nL de AAV que exprese GCaMP6m a una profundidad de 200 μm en el centro de cada lado del SC.
- Después de ~3 semanas, implante un cubreobjetos de 5 mm de diámetro sobre el SC. Realice una craneotomía ovalada de 4 mm x 3 mm centrada en el SC y adelgace el hueso alrededor de él. A continuación, presione el cubreobjetos directamente sobre el SC y fíjelo con cemento de resina.
- Excite GCaMP6m con un diodo emisor de luz azul (LED) con un filtro de excitación (469 nm ± 35 nm). Adquiera imágenes con una cámara de semiconductor de óxido metálico complementario (CMOS) después de pasar un filtro de emisión (525 nm ± 39 nm) a 10 Hz. La cámara cubre un área de 5,36 mm x 2,85 mm con una resolución espacial de 2,63 μm/píxel.
- Convoluciona cada imagen adquirida con un filtro de caja normalizado y reduce la resolución a 1/4 del tamaño original. Para cada píxel de la imagen, defina la respuesta, como se describe en el paso 7.4.
NOTA: Proporcione cuidados postoperatorios, como se describe en el paso 4.2.
Resultados
Las figuras 1A, B muestran cómo hacer la ventosa y los tapones, respectivamente. La figura 2 muestra cómo implantar el tapón con éxito. Después de implantar el tapón, el SC posterior-medial queda expuesto, como se muestra en la Figura 2D. La Figura 3 muestra las respuestas de calcio de las neuronas SC de un ejemplo de ratón de tipo salvaje fotografiado mediante microscopía de do...
Discusión
Pasos críticos en el protocolo
El paso más crítico es la craneotomía en los pasos 5.2 y 5.3. En primer lugar, el hueso a 0,5 mm posterior a la lambda es grueso y tiene vasos sanguíneos en su interior, lo que puede causar sangrado durante el proceso de perforación. Se debe preparar una espuma de gel adecuada para detener el sangrado. En segundo lugar, existe una buena probabilidad de angiorrexis cuando se extirpa el hueso justo por encima del seno transverso. Para la resolución de problemas, un ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo cuenta con el apoyo de la Fundación Nacional de Ciencias Naturales de China (32271060). Y.-t.L. diseñó la investigación, realizó el experimento, analizó los datos y escribió el manuscrito. Z.L. y R.W. realizaron el experimento.
Materiales
| Name | Company | Catalog Number | Comments |
| 16x objective | Nikon | ||
| 50-mm lens | Computar | M5018-MP2 | |
| 5-mm coverslip | Warner instruments | CS-5R | |
| bandpass filter | Chroma Technology | HQ575/250 m-2p | |
| butyl cyanoacrylate | Vetbond, World Precision Instruments | ||
| camera for monitoring pupil | FLIR | BFS-U3-04S2M-CS | |
| camera for widefield imaging | Basler | acA2000-165µm | |
| corona treater | Electro-Technic Products | BD-20AC | |
| dichroic | Chroma Technology | T600/200dcrb | |
| galvanometers | Cambridge Technology | ||
| glass bead sterilizer | RWD | RS1502 | |
| microdrill | RWD | 78001 | |
| micromanipulator | Sutter Instruments | QUAD | |
| photomultiplier tube | Hamamatsu | R3896 | |
| rotory encoder | USdigital | MA3-A10-125-N | |
| self-curing dental adhesive resin cement | SuperBond C&B, Sun Medical Co, Ltd. Moriyama, Japan | ||
| thermostatic heating pad | RWD | 69020 | |
| Ti:Sapphire laser | Spectra-Physics | Mai Tai HP DeepSee | |
| translucent silicone adhesive | Kwik-Sil, World Precision Instruments | ||
| treadmill | Xinglin Biology | ||
| Virus Strains | |||
| rAAV2/9-hsyn-Gcamp6m | Vector Core at Chinese Institute for Brain Research, Beijing | ||
| Animals | |||
| C57BL/6J wild type | Laboratory Animal Resource Center at Chinese Institute for Brain Research, Beijing | ||
| Emx1-Cre | The Jackson Laboratory | 5628 | |
| Pals1flox/wt | Christopher A. Walsh Lab | ||
| Software | |||
| ImageJ | NIH Image | ||
| Labview | National Instruments | ||
| MATLAB | Mathworks |
Referencias
- May, P. J. The mammalian superior colliculus: laminar structure and connections. Progress in Brain Research. 151, 321-378 (2006).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. 248 (4951), 73-76 (1990).
- Ohki, K., Chung, S., Ch'ng, Y. H., Kara, P., Reid, R. C. Functional imaging with cellular resolution reveals precise micro-architecture in visual cortex. Nature. 433 (7026), 597-603 (2005).
- Ratzlaff, E. H., Grinvald, A. A tandem-lens epifluorescence macroscope: Hundred-fold brightness advantage for wide-field imaging. Journal of Neuroscience Methods. 36 (2-3), 127-137 (1991).
- de Vries, S. E. J., et al. A large-scale standardized physiological survey reveals functional organization of the mouse visual cortex. Nature Neuroscience. 23 (1), 138-151 (2020).
- Mrsic-Flogel, T. D., et al. Altered map of visual space in the superior colliculus of mice lacking early retinal waves. The Journal of Neuroscience. 25 (29), 6921-6928 (2005).
- Cang, J., Wang, L., Stryker, M. P., Feldheim, D. A. Roles of ephrin-as and structured activity in the development of functional maps in the superior colliculus. The Journal of Neuroscience. 28 (43), 11015-11023 (2008).
- Feinberg, E. H., Meister, M. Orientation columns in the mouse superior colliculus. Nature. 519 (7542), 229-232 (2015).
- Ahmadlou, M., Heimel, J. A. Preference for concentric orientations in the mouse superior colliculus. Nature Communications. 6, 6773 (2015).
- de Malmazet, D., Kühn, N. K., Farrow, K. Retinotopic separation of nasal and temporal motion selectivity in the mouse superior colliculus. Current Biology. 28 (18), 2961-2969 (2018).
- Li, Y. T., Turan, Z., Meister, M. Functional architecture of motion direction in the mouse superior colliculus. Current Biology. 30 (17), 3304-3315 (2020).
- Gribizis, A., et al. Visual cortex gains independence from peripheral drive before eye opening. Neuron. 104 (4), 711-723 (2019).
- Inayat, S., et al. Neurons in the most superficial lamina of the mouse superior colliculus are highly selective for stimulus direction. The Journal of Neuroscience. 35 (20), 7992-8003 (2015).
- Barchini, J., Shi, X., Chen, H., Cang, J. Bidirectional encoding of motion contrast in the mouse superior colliculus. eLife. 7, 35261 (2018).
- Savier, E. L., Chen, H., Cang, J. Effects of locomotion on visual responses in the mouse superior colliculus. The Journal of Neuroscience. 39 (47), 9360-9368 (2019).
- Schröder, S., et al. Arousal modulates retinal output. Neuron. 107 (3), 487-495 (2020).
- Ge, X., et al. Retinal waves prime visual motion detection by simulating future optic flow. Science. 373 (6553), (2021).
- Chen, H., Savier, E. L., DePiero, V. J., Cang, J. Lack of evidence for stereotypical direction columns in the mouse superior colliculus. The Journal of Neuroscience. 41 (3), 461-473 (2021).
- Kasai, M., Isa, T. Effects of light isoflurane anesthesia on organization of direction and orientation selectivity in the superficial layer of the mouse superior colliculus. The Journal of Neuroscience. 42 (4), 619-630 (2022).
- Kim, S., et al. The apical complex couples cell fate and cell survival to cerebral cortical development. Neuron. 66 (1), 69-84 (2010).
- Kaifosh, P., Zaremba, J. D., Danielson, N. B., Losonczy, A. S. I. M. A. Python software for analysis of dynamic fluorescence imaging data. Frontiers in Neuroinformatics. 8, 80 (2014).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Kerlin, A. M., Andermann, M. L., Berezovskii, V. K., Reid, R. C. Broadly tuned response properties of diverse inhibitory neuron subtypes in mouse visual cortex. Neuron. 67 (5), 858-871 (2010).
- Göbel, W., Helmchen, F. In vivo calcium imaging of neural network function. Physiology. 22 (6), 358-365 (2007).
- Dombeck, D. A., Khabbaz, A. N., Collman, F., Adelman, T. L., Tank, D. W. Imaging large-scale neural activity with cellular resolution in awake, mobile mice. Neuron. 56 (1), 43-57 (2007).
- Evans, D. A., et al. A synaptic threshold mechanism for computing escape decisions. Nature. 558 (7711), 590-594 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados