Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Imagerie calcique dans le colliculus supérieur de souris
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole détaille la procédure d’imagerie des réponses calciques dans le colliculus supérieur (SC) des souris éveillées, y compris l’imagerie de l’activité d’un seul neurone avec la microscopie à deux photons tout en laissant le cortex intact chez les souris de type sauvage, et l’imagerie de l’ensemble du SC avec la microscopie à grand champ chez les souris mutantes à cortex partiel.
Résumé
Le colliculus supérieur (SC), une structure du mésencéphale conservée au cours de l’évolution chez tous les vertébrés, est le centre visuel le plus sophistiqué avant l’émergence du cortex cérébral. Il reçoit des entrées directes de ~30 types de cellules ganglionnaires rétiniennes (RGC), chacune codant pour une caractéristique visuelle spécifique. Il reste difficile de savoir si le CS hérite simplement des caractéristiques rétiniennes ou si un traitement supplémentaire et potentiellement de novo se produit dans le CS. Pour révéler le codage neuronal de l’information visuelle dans le SC, nous fournissons ici un protocole détaillé pour enregistrer optiquement les réponses visuelles avec deux méthodes complémentaires chez les souris éveillées. Une méthode utilise la microscopie à deux photons pour imager l’activité du calcium à une seule résolution cellulaire sans ablation du cortex superposé, tandis que l’autre utilise la microscopie à grand champ pour imager l’ensemble du SC d’une souris mutante dont le cortex est en grande partie sous-développé. Ce protocole détaille ces deux méthodes, y compris la préparation des animaux, l’injection virale, l’implantation de la plaque de tête, l’implantation du bouchon, l’acquisition de données et l’analyse des données. Les résultats représentatifs montrent que l’imagerie calcique à deux photons révèle des réponses neuronales évoquées visuellement à une résolution unicellulaire, et que l’imagerie calcique à grand champ révèle une activité neuronale dans l’ensemble du SC. En combinant ces deux méthodes, on peut révéler le codage neuronal dans le CS à différentes échelles, et une telle combinaison peut également être appliquée à d’autres régions du cerveau.
Introduction
Le colliculus supérieur (SC) est un centre visuel important chez tous les vertébrés. Chez les mammifères, il reçoit des entrées directes de la rétine et du cortex visuel1. Alors que l’enregistrement optique a été largement appliqué au cortex 2,3,4,5, son application dans le SC est entravée par de mauvais accès optiques 6,7,8,9,10,11,12,13,14,15,16,17 ,18,19. L’objectif de ce protocole est de fournir des détails sur deux méthodes complémentaires d’enregistrement optique de l’activité neuronale dans le CS.
Le SC est situé sous le cortex et le sinus transverse, ce qui limite l’accès optique aux neurones colliculaires. Une approche pour surmonter cette limitation est d’aspirer le cortex superposé et d’exposer le SC antéro-latéral 7,9,10,13,14,19. Cependant, comme le SC reçoit des entrées corticales, une telle opération peut affecter la façon dont les neurones du SC répondent aux stimuli visuels. Pour pallier cette limitation, nous détaillons ici un protocole alternatif permettant d’imager la couche superficielle du SC postérieur-médial avec un bouchon en silicone, tout en laissant le cortex intact 8,11. Plus précisément, pour obtenir une résolution unicellulaire, nous avons appliqué la microscopie à deux photons pour imager les réponses calciques dans le SC postérieur-médial de souris de type sauvage. De plus, pour obtenir une large couverture, nous avons appliqué la microscopie à grand champ pour imager l’ensemble du SC d’une souris mutante dont le cortex postérieur ne s’est pas développé20.
Les deux méthodes décrites dans ce protocole sont complémentaires l’une de l’autre. L’imagerie calcique à deux photons sans ablation du cortex est appropriée pour enregistrer l’activité neuronale à une résolution de cellule unique avec des entrées corticales intactes. L’imagerie calcique à grand champ est appropriée pour enregistrer l’activité neuronale dans l’ensemble du SC tout en sacrifiant la résolution spatiale.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les procédures expérimentales ont été réalisées conformément aux directives sur le bien-être animal et approuvées par l’IACUC de l’Institut chinois de recherche sur le cerveau de Beijing.
REMARQUE : L’échéancier de ce protocole est le suivant : 1) faire la ventouse ; 2) injecter le virus ; 3) implanter la plaque de tête ; 4) après 3 semaines, implantez le bouchon ; 5) Après une récupération et une accoutumance de ~3 jours sur le tapis roulant, effectuez une imagerie à deux photons/grand champ.
1. Préparation d’une ventouse (Figure 1A)
- Déposez une goutte de sérum physiologique tamponné au phosphate (PBS, 1x) dans un plat en acrylique et touchez-le avec une aiguille plate de 21 G pour remplir l’embout par capillarité.
- Couvrez la pointe de l’aiguille d’un adhésif en silicone translucide et laissez reposer environ ~10 min.
- Coupez l’adhésif silicone avec des ciseaux en un disque d’un diamètre de ~2 mm.
2. Préparation des bouchons (Figure 1B)
- Préparez une cale en plastique de 0,75 mm d’épaisseur et découpez un carré de 1 cm x 1 cm au centre. Préparez deux blocs d’acrylique. Nettoyez la cale et les blocs acryliques avec de l’alcool et essuyez-les avec du papier pour lentilles.
- Placez la cale sur un bloc acrylique, déposez l’adhésif silicone au centre pour remplir ~90% de l’ouverture (évitez les bulles), ajoutez un autre bloc acrylique et ~1 kg de force, et attendez 20 min.
- À l’aide d’un microscope chirurgical, découpez l’adhésif silicone à l’aide d’un scalpel à des prismes triangulaires de 1 mm de haut sur 1,5 mm de large au-dessus d’une feuille de papier sur laquelle sont imprimés des triangles.
- Enlevez la poussière des prismes triangulaires avec du ruban adhésif en plastique et placez-le sur une lamelle en verre de 5 mm de diamètre et 0,15 mm d’épaisseur.
- Placez la lamelle avec les prismes triangulaires sous un traitement corona, allumez le traitement corona et rapprochez-le de la lamelle jusqu’à ce que l’éclaircissement apparaisse. Maintenez-le dans cette position pendant quelques minutes jusqu’à ce qu’ils soient attachés l’un à l’autre.
REMARQUE : Cette étape peut être différente pour d’autres personnes traitant le coronavirus. - Mettez les bouchons dans une boîte de Pétri et placez-les dans un incubateur à 70 °C pendant la nuit.
3. Préparation d’animaux et injection de construction virale
- Utiliser C57BL/6J (type sauvage) et Emx1-Cre :Pals1flox/wt (cortex partiel)20 souris des deux sexes à l’âge de 6 semaines (9 semaines au moment de l’imagerie).
- Stériliser tout le matériel et les instruments utilisés pour l’intervention chirurgicale.
- Anesthésier la souris avec de l’isoflurane à 5 %, puis maintenir l’isoflurane à 1 % à 2 % avec un débit de 1 L/min pendant la chirurgie. Confirmer le niveau d’anesthésie par l’absence de réflexe de pincement des orteils.
- Fixez la tête de la souris sur un cadre stéréotaxique avec des barres d’oreille. Tout au long de l’opération, mettez une pommade ophtalmique sur les yeux de la souris pour éviter qu’ils ne se dessèchent et maintenez sa température corporelle à 37 °C avec un coussin chauffant thermostatique.
- Rasez la fourrure et stérilisez la surface de la peau avec de l’iodophor et de l’éthanol à 75 %. Injecter de la lidocaïne (5 mg/kg, injection sous-cutanée [SQ]), de la tilidine (2 mg/kg, SQ) et du méloxicam (2 mg/kg, SQ).
REMARQUE : La procédure chirurgicale pour la stérilisation, l’analgésie et l’anesthésie locale peut différer d’un établissement à l’autre. - Retirez la peau au-dessus du SC avec des ciseaux chirurgicaux pour exposer le crâne. Nettoyez le crâne avec un coton-tige.
- Ajustez le cadre stéréotaxique jusqu’à ce que la surface du crâne soit plane. Percez un trou de 0,5 mm latéralement et de 0,42 mm en avant de la lambda jusqu’à ce que la dure-mère soit exposée.
- Injecter le virus adéno-associé (AAV) exprimant GCaMP6m (rAAV2/9-hsyn-GCaMP6m ; 1 x10 13 GC/mL dissous dans 1x PBS) à des profondeurs de 1 mm et 1,6 mm à partir du lambda à l’aide d’une micropipette en verre biseauté (la pointe est biseautée à 25° avec un diamètre de 40-50 μm).
- À chaque profondeur, injecter 100 nL à une vitesse de 50 nL/min et attendre 5 min après chaque injection. Après l’injection, retirez lentement l’injecteur.
4. Implantation de la plaque de tête
- Nettoyez les muscles et les tissus au-dessus du crâne et grattez le crâne avec une lame. En cas de saignement, arrêtez-le avec de la mousse de gel et nettoyez le sang. Fixez la plaque de tête (Figure 1C) au crâne à l’aide d’un ciment à base de résine adhésive dentaire autopolymérisable. Plus précisément, mélangez 3/4 cuillère de polymère, trois gouttes de monomère et une goutte de catalyseur dans un bol en céramique sur de la glace. Appliquez le mélange sur la plaque de tête et le crâne. Fixez-les ensemble et attendez 5 minutes que l’adhésif prenne.
- Injecter des antibiotiques (ceftiofur sodique, 5 mg/kg, SQ) pour prévenir les infections. Retirez la souris du cadre stéréotaxique et placez-la sur un coussin chauffant pour la récupérer. Remettez-le dans la cage d’accueil après avoir récupéré de l’anesthésie. Pour la gestion de la douleur, fournissez de l’eau potable contenant de l’ibuprofène (0,5 mL/50 mL) à la souris pendant 3 jours après la chirurgie.
5. Implantation du bouchon (Figure 2)
- Implantez le bouchon 3 semaines après l’injection virale. Anesthésiez et fixez la souris sur un cadre stéréotaxique, comme décrit aux étapes 3.2 à 3.5. Préparez du gel mousse imbibé de solution saline pour arrêter les saignements potentiels.
- Percer un ovale de 3 mm x 2 mm à l’aide d’un microforet centré à 0,5 mm en arrière du lambda. Amincissez la limite de l’ovale jusqu’à ce qu’elle se fissure. Amincissez le crâne devant la fenêtre d’enregistrement ovale pour que la lamelle soit proche du SC.
- Retirez les lambeaux osseux à l’aide d’une pince fine. Faites une incision sur la dure-mère postérieure au sinus transverse et retirez le morceau de dure-mère. Si nécessaire, ajoutez du liquide céphalorachidien artificiel (ACSF) au cerveau à l’aide d’une seringue de 3 ml pour éviter qu’il ne se dessèche. Séchez le crâne et la fenêtre d’enregistrement avec de la mousse de gel et des cotons-tiges.
- Déposez une goutte d’adhésif silicone dans la fenêtre d’enregistrement. Utilisez la ventouse pour maintenir le bouchon avec une pression négative. Utilisez un micromanipulateur motorisé pour déplacer la ventouse et abaissez le bouchon dans l’adhésif silicone jusqu’à ce que la lamelle touche le crâne. Ensuite, déplacez le bouchon vers l’avant ~1 mm pour repousser le sinus transverse et exposer le SC. Cette étape doit être effectuée en 1 à 2 minutes avant que l’adhésif en silicone ne devienne collant.
- Nettoyez la plaque de tête et appliquez du cyanoacrylate de butyle et du ciment résine autour de la limite du bouchon pour le fixer à la plaque de tête.
REMARQUE : Fournir des soins postopératoires, comme décrit à l’étape 4.2.
6. Imagerie à deux photons (Figure 3)
- Fixez la tête de l’animal sur un tapis roulant rotatif avec sa plaque de tête. Placez le tapis roulant sous un microscope à deux photons et ajustez la hauteur dans une position appropriée. Placez un cône en aluminium noir entre l’objectif et la plaque de tête pour éviter la contamination lumineuse du moniteur pour la stimulation visuelle. Habituez la souris pendant ~15 min.
- Effectuez une imagerie à deux photons au microscope avec un grossissement de 16x, une ouverture numérique (NA) de 0,8 et un objectif de distance de travail (WD) de 3 mm. Utilisez un laser Ti :saphir avec une technique de verrouillage de mode contrôlée par deux scanners galvo pour exciter GCaMP6m à 920 nm. Ajustez la puissance du laser sur le plan de l’échantillon entre 20 et 80 mW.
- Numérisez un champ de vision de 600 μm x 600 μm à 4,8 Hz avec une résolution spatiale de 2,4 μ m/pixel et imagez l’activité neuronale à une profondeur allant jusqu’à 350 μ m.
REMARQUE : La fréquence d’échantillonnage et la résolution spatiale sont pour les scanners galvo et doivent être ajustées pour les scanners résonants. - Collectez la fluorescence émise à l’aide d’un miroir dichroïque et détectez-la à l’aide d’un tube photomultiplicateur (PMT) après l’avoir passée à travers un filtre passe-bande.
- Enregistrez la locomotion de la souris sur le tapis roulant à l’aide d’un encodeur rotatif. Utilisez un appareil photo avec un objectif de 50 mm pour enregistrer la taille et la position de sa pupille. Synchronisez ces enregistrements et acquisitions d’images avec la stimulation visuelle en enregistrant les déclencheurs envoyés par l’ordinateur de stimulation.
7. Analyse des réponses calciques mesurées par imagerie à deux photons
- Corrigez les mouvements du cerveau pendant l’imagerie à l’aide de SIMA21 ou NoRMCorre22.
- Dessinez manuellement une région d’intérêt (ROI) à l’aide de l’outil Cell Magic Wand (ImageJ) et ajustez-la à l’aide d’une ellipse dans MATLAB. Extraire les traces de fluorescence de chaque ROI après avoir éliminé la contamination par les neuropils :
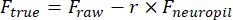
F true et F rawsont les amplitudes de fluorescence corrigées et brutes du ROI, respectivement, F neuropil est l’amplitude de fluorescence du neuropil environnant et r est le facteur de contamination du neuropilflou (0,7 pour notre configuration). Le r est estimé en mesurant les signaux sur un vaisseau sanguin pour lequel Fvrai = 0, comme décrit précédemment23,24. - Supprimez les fluctuations lentes de la ligne de base en soustrayant la valeur du huitième centile d’une fenêtre de 15 s centrée sur chaque image25.
- Définissez les réponses visuelles d’un neurone comme la variation relative de l’amplitude de fluorescence dans son ROI pendant la période de stimulus :
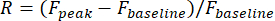
Lepic F est l’amplitude maximale de la trace de fluorescence pendant la stimulation visuelle, et la ligne de baseFest l’amplitude moyenne pendant une période de 0,5 s avant la stimulation.
8. Imagerie à grand champ et analyse de données (Figure 4)
- Construire un macroscope à grand champ avec une lentille tandem4 (Figure 4A). Le macroscope à objectif tandem est constitué de deux objectifs de caméra (50 mm et 105 mm) connectés via un adaptateur, et le grossissement est de ~2x.
- Préparez les souris mutantes du cortex partiel comme décrit aux étapes 3.2 à 3.5. Injecter 100 nL d’AAV exprimant GCaMP6m à une profondeur de 200 μm au centre de chaque côté du CS.
- Après ~3 semaines, implantez une lamelle de 5 mm de diamètre sur le CS. Effectuez une craniotomie ovale de 4 mm x 3 mm centrée au niveau du SC et amincissez l’os qui l’entoure. Ensuite, appuyez la lamelle directement sur le SC et fixez-la avec de la ciment résine.
- Excitez GCaMP6m avec une diode électroluminescente bleue (LED) avec un filtre d’excitation (469 nm ± 35 nm). Acquérez des images à l’aide d’une caméra CMOS (Complementary Metal Oxide Semiconductor) après avoir passé un filtre d’émission (525 nm ± 39 nm) à 10 Hz. La caméra couvre une surface de 5,36 mm x 2,85 mm avec une résolution spatiale de 2,63 μm/pixel.
- Convoluez chaque image acquise à l’aide d’un filtre de boîte normalisé et sous-échantillonnez-la à 1/4 de la taille d’origine. Pour chaque pixel de l’image, définissez la réponse, comme décrit à l’étape 7.4.
REMARQUE : Fournir des soins postopératoires, comme décrit à l’étape 4.2.
Access restricted. Please log in or start a trial to view this content.
Résultats
Les figures 1A et B montrent comment fabriquer respectivement la ventouse et les bouchons. La figure 2 montre comment implanter le bouchon avec succès. Après l’implantation du bouchon, le CS postérieur-médial est exposé, comme le montre la figure 2D. La figure 3 montre les réponses calciques des neurones SC d’un exemple de souris de type sauvage imagé à l’aide de la micros...
Access restricted. Please log in or start a trial to view this content.
Discussion
Étapes critiques du protocole
L’étape la plus critique est la craniotomie aux étapes 5.2 et 5.3. Tout d’abord, l’os à 0,5 mm en arrière de la lambda est épais et contient des vaisseaux sanguins, ce qui peut provoquer des saignements pendant le processus de forage. Une mousse de gel adéquate doit être préparée pour arrêter le saignement. Deuxièmement, il y a un bon risque d’angiorrhexie lors de l’ablation de l’os juste au-dessus du sinus transverse. Pour le dépannage, une autre...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ces travaux sont soutenus par la Fondation nationale des sciences naturelles de Chine (32271060). Y.-t.L. a conçu la recherche, réalisé l’expérience, analysé les données et rédigé le manuscrit. Z.L. et R.W. ont réalisé l’expérience.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 16x objective | Nikon | ||
| 50-mm lens | Computar | M5018-MP2 | |
| 5-mm coverslip | Warner instruments | CS-5R | |
| bandpass filter | Chroma Technology | HQ575/250 m-2p | |
| butyl cyanoacrylate | Vetbond, World Precision Instruments | ||
| camera for monitoring pupil | FLIR | BFS-U3-04S2M-CS | |
| camera for widefield imaging | Basler | acA2000-165µm | |
| corona treater | Electro-Technic Products | BD-20AC | |
| dichroic | Chroma Technology | T600/200dcrb | |
| galvanometers | Cambridge Technology | ||
| glass bead sterilizer | RWD | RS1502 | |
| microdrill | RWD | 78001 | |
| micromanipulator | Sutter Instruments | QUAD | |
| photomultiplier tube | Hamamatsu | R3896 | |
| rotory encoder | USdigital | MA3-A10-125-N | |
| self-curing dental adhesive resin cement | SuperBond C&B, Sun Medical Co, Ltd. Moriyama, Japan | ||
| thermostatic heating pad | RWD | 69020 | |
| Ti:Sapphire laser | Spectra-Physics | Mai Tai HP DeepSee | |
| translucent silicone adhesive | Kwik-Sil, World Precision Instruments | ||
| treadmill | Xinglin Biology | ||
| Virus Strains | |||
| rAAV2/9-hsyn-Gcamp6m | Vector Core at Chinese Institute for Brain Research, Beijing | ||
| Animals | |||
| C57BL/6J wild type | Laboratory Animal Resource Center at Chinese Institute for Brain Research, Beijing | ||
| Emx1-Cre | The Jackson Laboratory | 5628 | |
| Pals1flox/wt | Christopher A. Walsh Lab | ||
| Software | |||
| ImageJ | NIH Image | ||
| Labview | National Instruments | ||
| MATLAB | Mathworks |
Références
- May, P. J. The mammalian superior colliculus: laminar structure and connections. Progress in Brain Research. 151, 321-378 (2006).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. 248 (4951), 73-76 (1990).
- Ohki, K., Chung, S., Ch'ng, Y. H., Kara, P., Reid, R. C. Functional imaging with cellular resolution reveals precise micro-architecture in visual cortex. Nature. 433 (7026), 597-603 (2005).
- Ratzlaff, E. H., Grinvald, A. A tandem-lens epifluorescence macroscope: Hundred-fold brightness advantage for wide-field imaging. Journal of Neuroscience Methods. 36 (2-3), 127-137 (1991).
- de Vries, S. E. J., et al. A large-scale standardized physiological survey reveals functional organization of the mouse visual cortex. Nature Neuroscience. 23 (1), 138-151 (2020).
- Mrsic-Flogel, T. D., et al. Altered map of visual space in the superior colliculus of mice lacking early retinal waves. The Journal of Neuroscience. 25 (29), 6921-6928 (2005).
- Cang, J., Wang, L., Stryker, M. P., Feldheim, D. A. Roles of ephrin-as and structured activity in the development of functional maps in the superior colliculus. The Journal of Neuroscience. 28 (43), 11015-11023 (2008).
- Feinberg, E. H., Meister, M. Orientation columns in the mouse superior colliculus. Nature. 519 (7542), 229-232 (2015).
- Ahmadlou, M., Heimel, J. A. Preference for concentric orientations in the mouse superior colliculus. Nature Communications. 6, 6773(2015).
- de Malmazet, D., Kühn, N. K., Farrow, K. Retinotopic separation of nasal and temporal motion selectivity in the mouse superior colliculus. Current Biology. 28 (18), 2961-2969 (2018).
- Li, Y. T., Turan, Z., Meister, M. Functional architecture of motion direction in the mouse superior colliculus. Current Biology. 30 (17), 3304-3315 (2020).
- Gribizis, A., et al. Visual cortex gains independence from peripheral drive before eye opening. Neuron. 104 (4), 711-723 (2019).
- Inayat, S., et al. Neurons in the most superficial lamina of the mouse superior colliculus are highly selective for stimulus direction. The Journal of Neuroscience. 35 (20), 7992-8003 (2015).
- Barchini, J., Shi, X., Chen, H., Cang, J. Bidirectional encoding of motion contrast in the mouse superior colliculus. eLife. 7, 35261(2018).
- Savier, E. L., Chen, H., Cang, J. Effects of locomotion on visual responses in the mouse superior colliculus. The Journal of Neuroscience. 39 (47), 9360-9368 (2019).
- Schröder, S., et al. Arousal modulates retinal output. Neuron. 107 (3), 487-495 (2020).
- Ge, X., et al. Retinal waves prime visual motion detection by simulating future optic flow. Science. 373 (6553), (2021).
- Chen, H., Savier, E. L., DePiero, V. J., Cang, J. Lack of evidence for stereotypical direction columns in the mouse superior colliculus. The Journal of Neuroscience. 41 (3), 461-473 (2021).
- Kasai, M., Isa, T. Effects of light isoflurane anesthesia on organization of direction and orientation selectivity in the superficial layer of the mouse superior colliculus. The Journal of Neuroscience. 42 (4), 619-630 (2022).
- Kim, S., et al. The apical complex couples cell fate and cell survival to cerebral cortical development. Neuron. 66 (1), 69-84 (2010).
- Kaifosh, P., Zaremba, J. D., Danielson, N. B., Losonczy, A. S. I. M. A. Python software for analysis of dynamic fluorescence imaging data. Frontiers in Neuroinformatics. 8, 80(2014).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Kerlin, A. M., Andermann, M. L., Berezovskii, V. K., Reid, R. C. Broadly tuned response properties of diverse inhibitory neuron subtypes in mouse visual cortex. Neuron. 67 (5), 858-871 (2010).
- Göbel, W., Helmchen, F. In vivo calcium imaging of neural network function. Physiology. 22 (6), 358-365 (2007).
- Dombeck, D. A., Khabbaz, A. N., Collman, F., Adelman, T. L., Tank, D. W. Imaging large-scale neural activity with cellular resolution in awake, mobile mice. Neuron. 56 (1), 43-57 (2007).
- Evans, D. A., et al. A synaptic threshold mechanism for computing escape decisions. Nature. 558 (7711), 590-594 (2018).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon