Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Methods Article
Kalzium-Bildgebung beim Colliculus superior der Maus
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt das Verfahren zur Bildgebung von Kalziumantworten im Colliculus superior (SC) von wachen Mäusen, einschließlich der Abbildung der Aktivität einzelner Neuronen mit Zwei-Photonen-Mikroskopie, während der Kortex bei Wildtyp-Mäusen intakt bleibt, und der Abbildung des gesamten SC mit Weitfeldmikroskopie bei Mäusen mit Teilkortex-Mutante.
Zusammenfassung
Der Colliculus superior (SC), eine evolutionär konservierte Mittelhirnstruktur bei allen Wirbeltieren, ist das am weitesten entwickelte Sehzentrum vor der Entstehung der Großhirnrinde. Es empfängt direkte Eingaben von ~30 Arten von retinalen Ganglienzellen (RGCs), von denen jede ein bestimmtes visuelles Merkmal kodiert. Es bleibt unklar, ob der SC einfach nur Netzhautmerkmale erbt oder ob eine zusätzliche und möglicherweise de novo Verarbeitung im SC stattfindet. Um die neuronale Kodierung visueller Informationen im SC aufzudecken, stellen wir hier ein detailliertes Protokoll zur optischen Aufzeichnung visueller Reaktionen mit zwei komplementären Methoden in wachen Mäusen zur Verfügung. Eine Methode verwendet Zwei-Photonen-Mikroskopie, um die Kalziumaktivität mit Einzelzellauflösung abzubilden, ohne den überlagernden Kortex abzutragen, während die andere Weitfeldmikroskopie verwendet, um die gesamte SC einer mutierten Maus abzubilden, deren Kortex weitgehend unentwickelt ist. In diesem Protokoll werden diese beiden Methoden detailliert beschrieben, einschließlich der Tierpräparation, der Virusinjektion, der Kopfplattenimplantation, der Plug-Implantation, der Datenerfassung und der Datenanalyse. Die repräsentativen Ergebnisse zeigen, dass die Zwei-Photonen-Kalzium-Bildgebung visuell evozierte neuronale Reaktionen bei Einzelzellauflösung zeigt, und die Weitfeld-Kalzium-Bildgebung zeigt neuronale Aktivität über das gesamte SC. Durch die Kombination dieser beiden Methoden kann man die neuronale Kodierung im SC auf verschiedenen Skalen aufdecken, und eine solche Kombination kann auch auf andere Hirnregionen angewendet werden.
Einleitung
Der Colliculus superior (SC) ist ein wichtiges Sehzentrum bei allen Wirbeltieren. Bei Säugetieren erhält es direkte Eingaben von der Netzhaut und dem visuellen Kortex1. Während die optische Aufzeichnung im Kortex 2,3,4,5 weit verbreitet ist, wird ihre Anwendung im SC durch schlechte optische Zugänge behindert 6,7,8,9,10,11,12,13,14,15,16,17 ,18,19. Das Ziel dieses Protokolls ist es, Details über zwei komplementäre Methoden zur optischen Aufzeichnung der neuronalen Aktivität im SC bereitzustellen.
Der SC befindet sich unter dem Kortex und dem Sinus transversal, was den optischen Zugang zu den kollikulären Neuronen einschränkt. Ein Ansatz, um diese Einschränkung zu überwinden, besteht darin, den überlagernden Kortex zu aspirieren und den anterior-lateralen SCfreizulegen 7,9,10,13,14,19. Da der SC jedoch kortikale Inputs empfängt, kann eine solche Operation beeinflussen, wie die SC-Neuronen auf visuelle Reize reagieren. Um diese Einschränkung zu überwinden, beschreiben wir hier ein alternatives Protokoll, um die oberflächliche Schicht des posterior-medialen SC mit einem Silikonpfropfen abzubilden, während der Kortex intakt bleibt 8,11. Um eine Einzelzellauflösung zu erreichen, haben wir die Zwei-Photonen-Mikroskopie angewendet, um die Kalziumantwort im posterior-medialen SC von Wildtyp-Mäusen abzubilden. Um eine breite Abdeckung zu erreichen, haben wir außerdem Weitfeldmikroskopie angewendet, um das gesamte SC einer mutierten Maus abzubilden, deren hinterer Kortex sich nicht entwickelt hat20.
Die beiden in diesem Protokoll beschriebenen Methoden ergänzen einander. Die Zwei-Photonen-Kalzium-Bildgebung ohne Ablation des Kortex eignet sich für die Aufzeichnung neuronaler Aktivität mit Einzelzellauflösung und intaktem kortikalem Input. Die Weitfeld-Kalzium-Bildgebung eignet sich für die Aufzeichnung der neuronalen Aktivität im gesamten SC unter Einbußen bei räumlicher Auflösung.
Protokoll
Alle experimentellen Verfahren wurden in Übereinstimmung mit den Tierschutzrichtlinien durchgeführt und von der IACUC am Chinesischen Institut für Hirnforschung, Peking, genehmigt.
HINWEIS: Der Zeitplan für dieses Protokoll ist wie folgt: 1) Herstellung des Saugnapfes; 2) das Virus injizieren; 3) Implantieren Sie die Kopfplatte; 4) Implantieren Sie nach 3 Wochen den Pfropfen; 5) Führen Sie nach einer ~3-tägigen Erholung und Gewöhnung auf dem Laufband eine Zwei-Photonen-/Weitfeld-Bildgebung durch.
1. Vorbereitung eines Saugnapfes (Abbildung 1A)
- Geben Sie einen Tropfen phosphatgepufferte Kochsalzlösung (PBS, 1x) in eine Acrylschale und berühren Sie sie mit einer 21 G flachen Nadel, um die Spitze durch Kapillarität zu füllen.
- Decken Sie die Nadelspitze mit einem durchscheinenden Silikonkleber ab und lassen Sie sie ca. ~10 Minuten einwirken.
- Schneide den Silikonkleber mit einer Schere zu einer Scheibe mit ~2 mm Durchmesser zu.
2. Vorbereitung der Stopfen (Abbildung 1B)
- Bereiten Sie eine 0,75 mm dicke Kunststoffscheibe vor und schneiden Sie in der Mitte ein 1 cm x 1 cm großes Quadrat aus. Bereite zwei Acrylblöcke vor. Reinigen Sie den Shimstock und die Acrylblöcke mit Alkohol und wischen Sie sie mit Linsenpapier ab.
- Legen Sie die Unterlegscheibe auf einen Acrylblock, legen Sie den Silikonkleber in die Mitte, um ~90% der Öffnung zu füllen (vermeiden Sie Blasen), fügen Sie einen weiteren Acrylblock und ~1 kg Kraft hinzu und warten Sie 20 Minuten.
- Schneiden Sie unter dem Operationsmikroskop den Silikonkleber mit einem Skalpell auf 1 mm hohe und 1,5 mm breite dreieckige Prismen über einem Blatt Papier mit aufgedruckten Dreiecken zu.
- Entfernen Sie den Staub von den dreieckigen Prismen mit Kunststoffklebeband und legen Sie es auf ein Deckglas mit einem Durchmesser von 5 mm und einer Dicke von 0,15 mm.
- Legen Sie das Deckglas mit den dreieckigen Prismen unter einen Korona-Behandler, schalten Sie den Corona-Behandler ein und bringen Sie es näher an das Deckglas, bis eine Aufhellung auftritt. Halten Sie es einige Minuten lang in dieser Position, bis sie aneinander befestigt sind.
HINWEIS: Dieser Schritt kann bei anderen Corona-Behandlern anders sein. - Legen Sie die Plugs in eine Petrischale und legen Sie sie über Nacht bei 70 °C in einen Inkubator.
3. Vorbereitung der Tiere und Injektion des viralen Konstrukts
- Verwenden Sie C57BL/6J (Wildtyp) und Emx1-Cre:Pals1flox/wt (partieller Kortex)20 Mäuse beiderlei Geschlechts im Alter von 6 Wochen (9 Wochen zum Zeitpunkt der Bildgebung).
- Sterilisieren Sie alle Materialien und Instrumente, die für den chirurgischen Eingriff verwendet werden.
- Betäuben Sie die Maus mit 5% Isofluran und halten Sie dann das Isofluran bei 1%-2% mit einer Flussrate von 1 l/min während der Operation. Bestätigen Sie den Grad der Anästhesie durch das Fehlen eines Zehenkneifreflexes.
- Befestigen Sie die Maus mit dem Kopf auf einem stereotaktischen Rahmen mit Ohrbügeln. Tragen Sie während der gesamten Operation Augensalbe auf die Augen der Maus auf, um ein Austrocknen zu verhindern, und halten Sie die Körpertemperatur mit einem thermostatischen Heizkissen bei 37 °C.
- Rasieren Sie das Fell und sterilisieren Sie die Hautoberfläche mit Jodophor und 75%igem Ethanol. Injizieren Sie Lidocain (5 mg/kg, subkutane Injektion [SQ]), Tilidin (2 mg/kg, SQ) und Meloxicam (2 mg/kg, SQ).
HINWEIS: Das chirurgische Verfahren für Sterilisation, Analgesie und Lokalanästhesie kann je nach Einrichtung unterschiedlich sein. - Entfernen Sie die Haut über dem SC mit einer chirurgischen Schere, um den Schädel freizulegen. Reinigen Sie den Schädel mit einem Wattestäbchen.
- Passen Sie den stereotaktischen Rahmen an, bis die Schädeloberfläche flach ist. Bohren Sie ein Loch 0,5 mm seitlich und 0,42 mm vor dem Lambda, bis die Dura freiliegt.
- Adeno-assoziiertes Virus (AAV), das GCaMP6m exprimiert (rAAV2/9-hsyn-GCaMP6m; 1 x 10 13 GC/ml gelöst in1x PBS) in einer Tiefe von 1 mm und 1,6 mm aus dem Lambda mit einer abgeschrägten Glasmikropipette injizieren (Spitze ist auf 25° abgeschrägt und hat einen Durchmesser von 40-50 μm).
- In jeder Tiefe 100 nL mit einer Geschwindigkeit von 50 nL/min injizieren und nach jeder Injektion 5 min warten. Nach der Injektion den Injektor langsam herausziehen.
4. Implantation der Kopfplatte
- Reinigen Sie die Muskeln und das Gewebe über dem Schädel und kratzen Sie den Schädel mit einer Klinge ab. Wenn Blutungen auftreten, stoppen Sie sie mit Gelschaum und reinigen Sie das Blut. Befestigen Sie die Kopfplatte (Abbildung 1C) mit einem selbsthärtenden Zahnadhäsivharzzement am Schädel. Mischen Sie einen 3/4 Löffel Polymer, drei Tropfen Monomer und einen Tropfen Katalysator in einer Keramikschüssel auf Eis. Tragen Sie die Mischung auf die Kopfplatte und den Schädel auf. Befestigen Sie sie zusammen und warten Sie 5 Minuten, bis der Kleber ausgehärtet ist.
- Injizieren Sie Antibiotika (Ceftiofur-Natrium, 5 mg/kg, SQ), um Infektionen zu verhindern. Nehmen Sie die Maus aus dem stereotaktischen Rahmen und legen Sie sie zur Erholung auf ein Heizkissen. Bringen Sie es nach der Genesung von der Narkose in den häuslichen Käfig zurück. Zur Schmerzbehandlung der Maus 3 Tage nach der Operation Ibuprofen-haltiges Trinkwasser (0,5 ml/50 ml) zur Verfügung stellen.
5. Implantation des Steckers (Abbildung 2)
- Implantieren Sie den Pfropfen 3 Wochen nach der Virusinjektion. Betäuben und fixieren Sie die Maus auf einem stereotaktischen Rahmen, wie in den Schritten 3.2-3.5 beschrieben. Bereiten Sie in Kochsalzlösung getränkten Gelschaum vor, um mögliche Blutungen zu stoppen.
- Bohren Sie ein 3 mm x 2 mm großes Oval mit einem Mikrobohrer, der 0,5 mm hinter dem Lambda zentriert ist. Dünne den Rand des Ovals aus, bis er reißt. Verdünnen Sie den Schädel vor dem ovalen Aufnahmefenster, um das Deckglas dicht an der SC zu machen.
- Entfernen Sie Knochenlappen mit einer feinen Pinzette. Machen Sie einen Schnitt an der Dura posterior des Sinus transversum und entfernen Sie das Stück Dura. Falls erforderlich, geben Sie dem Gehirn künstliche Zerebrospinalflüssigkeit (ACSF) mit einer 3-ml-Spritze zu, um ein Austrocknen zu verhindern. Trocknen Sie den Schädel und das Aufnahmefenster mit Gelschaum und Wattestäbchen.
- Geben Sie einen Tropfen Silikonkleber in das Aufnahmefenster. Verwenden Sie den Saugnapf, um den Stecker mit Unterdruck zu halten. Verwenden Sie einen motorisierten Mikromanipulator, um den Saugnapf zu bewegen, und senken Sie den Stecker in den Silikonkleber, bis das Deckglas den Schädel berührt. Bewegen Sie dann den Plug ~1 mm nach vorne, um den Sinus transversum wegzuschieben und den SC freizulegen. Dieser Schritt sollte in 1-2 Minuten erledigt sein, bevor der Silikonkleber klebrig wird.
- Reinigen Sie die Kopfplatte und tragen Sie Butylcyanacrylat und Harzzement um die Begrenzung des Steckers auf, um ihn an der Kopfplatte zu befestigen.
HINWEIS: Führen Sie eine postoperative Versorgung durch, wie in Schritt 4.2 beschrieben.
6. Zwei-Photonen-Bildgebung (Abbildung 3)
- Fixieren Sie das Tier mit der Kopfplatte auf einem rotierenden Laufband. Stellen Sie das Laufband unter ein Zwei-Photonen-Mikroskop und stellen Sie die Höhe auf eine geeignete Position ein. Platzieren Sie einen schwarzen Aluminiumkegel zwischen dem Objektiv und der Kopfplatte, um eine Lichtverschmutzung durch den Monitor für die visuelle Stimulation zu verhindern. Gewöhnen Sie die Maus für ~15 Minuten.
- Führen Sie Zwei-Photonen-Bildgebung auf dem Mikroskop mit einer 16-fachen Vergrößerung, einer numerischen Apertur (NA) von 0,8 und einem Objektiv mit einem Arbeitsabstand von 3 mm durch. Verwenden Sie einen Ti:Saphir-Laser mit Modenkopplungstechnik, der von zwei Galvo-Scannern gesteuert wird, um GCaMP6m bei 920 nm anzuregen. Stellen Sie die Laserleistung an der Probenebene zwischen 20 und 80 mW ein.
- Scannen Sie ein Sichtfeld von 600 μm x 600 μm bei 4,8 Hz mit einer räumlichen Auflösung von 2,4 μ m/Pixel und bilden Sie die neuronale Aktivität über eine Tiefe von bis zu 350 μ m ab.
HINWEIS: Die Abtastrate und die räumliche Auflösung gelten für Galvo-Scanner und sollten für resonante Scanner angepasst werden. - Sammeln Sie die emittierte Fluoreszenz mit einem dichroitischen Spiegel und detektieren Sie sie mit einer Photomultiplier-Röhre (PMT), nachdem Sie sie durch einen Bandpassfilter geleitet haben.
- Zeichnen Sie die Fortbewegung der Maus auf dem Laufband mit einem Drehgeber auf. Verwenden Sie eine Kamera mit einem 50-mm-Objektiv, um die Pupillengröße und -position aufzuzeichnen. Synchronisieren Sie diese Aufzeichnungen und Bildaufnahmen mit der visuellen Stimulation, indem Sie die vom Stimulationscomputer gesendeten Trigger aufzeichnen.
7. Analyse der Kalziumreaktionen, gemessen durch Zwei-Photonen-Bildgebung
- Korrigieren Sie die Gehirnbewegung während der Bildgebung mit SIMA21 oder NoRMCorre22.
- Zeichnen Sie manuell eine Region of Interest (ROI) mit dem Cell Magic Wand (ImageJ) und passen Sie sie mit einer Ellipse in MATLAB an. Extrahieren Sie die Fluoreszenzspuren jedes ROI nach der Entfernung der Neuropil-Kontamination:
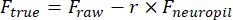
Ftrue und Frawsind die korrigierten bzw. rohen Fluoreszenzamplituden des ROI, F Neuropil ist die Fluoreszenzamplitude des umgebenden Neuropils und r ist der unscharfe Neuropil-Kontaminationsfaktor(0,7 für unser Setup). Das r wird geschätzt, indem die Signale an einem Blutgefäß gemessen werden, für das Fwahr = 0 ist, wie zuvor beschrieben23,24. - Entfernen Sie langsame Grundlinienschwankungen, indem Sie den achten Perzentilwert von einem 15-Sekunden-Fenster subtrahieren, das auf jedem Framezentriert ist 25.
- Definieren Sie die visuellen Reaktionen eines Neurons als die relative Änderung der Fluoreszenzamplitude in seinem ROI während der Stimulusperiode:
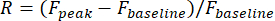
F-Peak ist die Peak-Amplitude der Fluoreszenzspur während der visuellen Stimulation, und F-Baselineist die mittlere Amplitude während eines Zeitraums von 0,5 s vor der Stimulation.
8. Weitfeld-Bildgebung und Datenanalyse (Abbildung 4)
- Bauen Sie ein Weitfeldmakroskop mit einem Tandemobjektiv4 (Abbildung 4A). Das Tandem-Makroskop besteht aus zwei Kameraobjektiven (50 mm und 105 mm), die über einen Adapter miteinander verbunden sind, und die Vergrößerung beträgt ~2x.
- Bereiten Sie Mäuse mit Teilrindenmutanten vor, wie in den Schritten 3.2-3.5 beschrieben. 100 nL AAV, das GCaMP6m exprimiert, werden in einer Tiefe von 200 μm in die Mitte jeder Seite des SC injiziert.
- Nach ~3 Wochen implantieren Sie ein Deckglas mit 5 mm Durchmesser über den SC. Führen Sie eine ovale Kraniotomie von 4 mm x 3 mm mittig am SC durch und verdünnen Sie den Knochen um ihn herum. Drücken Sie dann das Deckglas direkt auf den SC und fixieren Sie es mit Kunstzement.
- GCaMP6m mit einer blauen Leuchtdiode (LED) mit Anregungsfilter (469 nm ± 35 nm) anregen. Erfassen Sie Bilder mit einer CMOS-Kamera (Complementary Metal Oxide Semiconductor), nachdem Sie einen Emissionsfilter (525 nm ± 39 nm) bei 10 Hz passiert haben. Die Kamera deckt einen Bereich von 5,36 mm x 2,85 mm bei einer räumlichen Auflösung von 2,63 μm/Pixel ab.
- Wickeln Sie jedes aufgenommene Bild mit einem normalisierten Box-Filter und reduzieren Sie es auf 1/4 der Originalgröße. Definieren Sie für jedes Pixel im Bild die Antwort, wie in Schritt 7.4 beschrieben.
HINWEIS: Führen Sie eine postoperative Versorgung durch, wie in Schritt 4.2 beschrieben.
Ergebnisse
Die Abbildungen 1A,B zeigen, wie der Saugnapf bzw. die Stopfen hergestellt werden. Abbildung 2 zeigt, wie der Plug erfolgreich implantiert wird. Nach der Implantation des Plugs wird der posterior-mediale SC freigelegt, wie in Abbildung 2D dargestellt. Abbildung 3 zeigt die Kalziumantwort von SC-Neuronen an einer Wildtyp-Maus, die mit Zwei-Photonen-Mikroskopie aufgenommen wurde. Das drei...
Diskussion
Kritische Schritte im Protokoll
Der kritischste Schritt ist die Kraniotomie in den Schritten 5.2 und 5.3. Erstens ist der Knochen 0,5 mm hinter dem Lambda dick und enthält Blutgefäße im Inneren, die während des Bohrvorgangs zu Blutungen führen können. Es sollte ein geeigneter Gelschaum vorbereitet werden, um die Blutung zu stoppen. Zweitens besteht eine gute Wahrscheinlichkeit für eine Angiorrhexis, wenn der Knochen direkt über der Sinus transversum entfernt wird. Zur Fehlerbehebung besteht ei...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wird von der National Natural Science Foundation of China (32271060) unterstützt. Y.-t.L. entwarf die Forschung, führte das Experiment durch, analysierte die Daten und schrieb das Manuskript. Z.L. und R.W. führten das Experiment durch.
Materialien
| Name | Company | Catalog Number | Comments |
| 16x objective | Nikon | ||
| 50-mm lens | Computar | M5018-MP2 | |
| 5-mm coverslip | Warner instruments | CS-5R | |
| bandpass filter | Chroma Technology | HQ575/250 m-2p | |
| butyl cyanoacrylate | Vetbond, World Precision Instruments | ||
| camera for monitoring pupil | FLIR | BFS-U3-04S2M-CS | |
| camera for widefield imaging | Basler | acA2000-165µm | |
| corona treater | Electro-Technic Products | BD-20AC | |
| dichroic | Chroma Technology | T600/200dcrb | |
| galvanometers | Cambridge Technology | ||
| glass bead sterilizer | RWD | RS1502 | |
| microdrill | RWD | 78001 | |
| micromanipulator | Sutter Instruments | QUAD | |
| photomultiplier tube | Hamamatsu | R3896 | |
| rotory encoder | USdigital | MA3-A10-125-N | |
| self-curing dental adhesive resin cement | SuperBond C&B, Sun Medical Co, Ltd. Moriyama, Japan | ||
| thermostatic heating pad | RWD | 69020 | |
| Ti:Sapphire laser | Spectra-Physics | Mai Tai HP DeepSee | |
| translucent silicone adhesive | Kwik-Sil, World Precision Instruments | ||
| treadmill | Xinglin Biology | ||
| Virus Strains | |||
| rAAV2/9-hsyn-Gcamp6m | Vector Core at Chinese Institute for Brain Research, Beijing | ||
| Animals | |||
| C57BL/6J wild type | Laboratory Animal Resource Center at Chinese Institute for Brain Research, Beijing | ||
| Emx1-Cre | The Jackson Laboratory | 5628 | |
| Pals1flox/wt | Christopher A. Walsh Lab | ||
| Software | |||
| ImageJ | NIH Image | ||
| Labview | National Instruments | ||
| MATLAB | Mathworks |
Referenzen
- May, P. J. The mammalian superior colliculus: laminar structure and connections. Progress in Brain Research. 151, 321-378 (2006).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. 248 (4951), 73-76 (1990).
- Ohki, K., Chung, S., Ch'ng, Y. H., Kara, P., Reid, R. C. Functional imaging with cellular resolution reveals precise micro-architecture in visual cortex. Nature. 433 (7026), 597-603 (2005).
- Ratzlaff, E. H., Grinvald, A. A tandem-lens epifluorescence macroscope: Hundred-fold brightness advantage for wide-field imaging. Journal of Neuroscience Methods. 36 (2-3), 127-137 (1991).
- de Vries, S. E. J., et al. A large-scale standardized physiological survey reveals functional organization of the mouse visual cortex. Nature Neuroscience. 23 (1), 138-151 (2020).
- Mrsic-Flogel, T. D., et al. Altered map of visual space in the superior colliculus of mice lacking early retinal waves. The Journal of Neuroscience. 25 (29), 6921-6928 (2005).
- Cang, J., Wang, L., Stryker, M. P., Feldheim, D. A. Roles of ephrin-as and structured activity in the development of functional maps in the superior colliculus. The Journal of Neuroscience. 28 (43), 11015-11023 (2008).
- Feinberg, E. H., Meister, M. Orientation columns in the mouse superior colliculus. Nature. 519 (7542), 229-232 (2015).
- Ahmadlou, M., Heimel, J. A. Preference for concentric orientations in the mouse superior colliculus. Nature Communications. 6, 6773 (2015).
- de Malmazet, D., Kühn, N. K., Farrow, K. Retinotopic separation of nasal and temporal motion selectivity in the mouse superior colliculus. Current Biology. 28 (18), 2961-2969 (2018).
- Li, Y. T., Turan, Z., Meister, M. Functional architecture of motion direction in the mouse superior colliculus. Current Biology. 30 (17), 3304-3315 (2020).
- Gribizis, A., et al. Visual cortex gains independence from peripheral drive before eye opening. Neuron. 104 (4), 711-723 (2019).
- Inayat, S., et al. Neurons in the most superficial lamina of the mouse superior colliculus are highly selective for stimulus direction. The Journal of Neuroscience. 35 (20), 7992-8003 (2015).
- Barchini, J., Shi, X., Chen, H., Cang, J. Bidirectional encoding of motion contrast in the mouse superior colliculus. eLife. 7, 35261 (2018).
- Savier, E. L., Chen, H., Cang, J. Effects of locomotion on visual responses in the mouse superior colliculus. The Journal of Neuroscience. 39 (47), 9360-9368 (2019).
- Schröder, S., et al. Arousal modulates retinal output. Neuron. 107 (3), 487-495 (2020).
- Ge, X., et al. Retinal waves prime visual motion detection by simulating future optic flow. Science. 373 (6553), (2021).
- Chen, H., Savier, E. L., DePiero, V. J., Cang, J. Lack of evidence for stereotypical direction columns in the mouse superior colliculus. The Journal of Neuroscience. 41 (3), 461-473 (2021).
- Kasai, M., Isa, T. Effects of light isoflurane anesthesia on organization of direction and orientation selectivity in the superficial layer of the mouse superior colliculus. The Journal of Neuroscience. 42 (4), 619-630 (2022).
- Kim, S., et al. The apical complex couples cell fate and cell survival to cerebral cortical development. Neuron. 66 (1), 69-84 (2010).
- Kaifosh, P., Zaremba, J. D., Danielson, N. B., Losonczy, A. S. I. M. A. Python software for analysis of dynamic fluorescence imaging data. Frontiers in Neuroinformatics. 8, 80 (2014).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Kerlin, A. M., Andermann, M. L., Berezovskii, V. K., Reid, R. C. Broadly tuned response properties of diverse inhibitory neuron subtypes in mouse visual cortex. Neuron. 67 (5), 858-871 (2010).
- Göbel, W., Helmchen, F. In vivo calcium imaging of neural network function. Physiology. 22 (6), 358-365 (2007).
- Dombeck, D. A., Khabbaz, A. N., Collman, F., Adelman, T. L., Tank, D. W. Imaging large-scale neural activity with cellular resolution in awake, mobile mice. Neuron. 56 (1), 43-57 (2007).
- Evans, D. A., et al. A synaptic threshold mechanism for computing escape decisions. Nature. 558 (7711), 590-594 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten