Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis automatizado optimizado de la modulación de la homeostasis de las mitocondrias neuronales vivas mediante receptores de ácido retinoico específicos de isoformas
En este artículo
Resumen
La red mitocondrial es extremadamente compleja, lo que hace que sea muy difícil de analizar. Una novedosa herramienta de MATLAB analiza mitocondrias con imágenes confocales en vivo en imágenes timelapse, pero da como resultado un gran volumen de salida que requiere atención manual individual. Para solucionar este problema, se desarrolló una optimización de rutina que permite un análisis rápido de los archivos.
Resumen
La compleja red mitocondrial hace que sea muy difícil segmentar, seguir y analizar las células vivas. Las herramientas de MATLAB permiten el análisis de mitocondrias en archivos timelapse, simplificando y agilizando considerablemente el proceso de procesamiento de imágenes. No obstante, las herramientas existentes producen un gran volumen de salida, lo que requiere atención manual individual, y las configuraciones experimentales básicas tienen una salida de miles de archivos, cada uno de los cuales requiere un manejo extenso y lento.
Para abordar estos problemas, se desarrolló una optimización rutinaria, tanto en código de MATLAB como en forma de scripts en vivo, lo que permite un análisis rápido de archivos y reduce significativamente la lectura de documentos y el procesamiento de datos. Con una velocidad de 100 archivos/min, la optimización permite un análisis rápido en general. La optimización logra la salida de resultados promediando los datos específicos del marco para las mitocondrias individuales a lo largo de los marcos de tiempo, analizando los datos de una manera definida, consistente con la salida de las herramientas existentes. Se realizaron imágenes confocales en vivo utilizando el colorante tetrametilrodamina metil éster, y la optimización de rutina se validó mediante el tratamiento de células neuronales con agonistas del receptor de ácido retinoico (RAR), cuyos efectos sobre las mitocondrias neuronales están establecidos en la literatura. Los resultados fueron consistentes con la literatura y permitieron una mayor caracterización del comportamiento de la red mitocondrial en respuesta a la modulación de RAR específica de isoformas.
Esta nueva metodología permitió una caracterización rápida y validada de la red de mitocondrias de toda la neurona, pero también permite la diferenciación entre las mitocondrias axónicas y las del cuerpo celular, una característica esencial para aplicar en el campo de la neurociencia. Además, este protocolo se puede aplicar a experimentos que utilizan tratamientos de acción rápida, permitiendo la obtención de imágenes de las mismas células antes y después de los tratamientos, trascendiendo el campo de la neurociencia.
Introducción
Las mitocondrias celulares se encuentran en el centro de todos los estados fisiológicos, y una comprensión profunda de su homeostasis (mitástasis) y comportamiento es primordial para ayudar a identificar el tratamiento farmacológico para una amplia gama de enfermedades, incluyendo el cáncer y la enfermedad de Alzheimer 1,2.
Las mitocondrias desempeñan un papel celular crucial en la homeostasis energética, la generación de ATP, la amortiguación del calcio y la regulación de ROS, y la mitostasis es esencial para mantener la homeostasis de las proteínas, ya que las chaperonas moleculares dependen de la energía3. Estos requieren una modulación y adaptación de red constante y dinámica para satisfacer eficientemente las necesidades celulares, y el transporte de mitocondrias está regulado por diferentes vías de señalización; Trabajos previos han descrito una de estas vías, la de los receptores de ácido retinoico (RARs)4,5. El ácido retinoico (AR) promueve el crecimiento axonal y de neuritas a través de la activación de RAR. En las neuronas corticales primarias de ratón, la activación de RAR-β estimula el crecimiento, la velocidad y la movilidad mitocondrial en la neurita6.
Teniendo en cuenta la adaptabilidad y la dinámica de la red mitocondrial, la posibilidad de evaluar la mitostasis en "tiempo real" es esencial no solo para investigar la homeostasis energética, sino también para la proteostasis, la salud celular, la proliferación o la señalización. Un método comúnmente utilizado para evaluar la mitostasis se basa en la microscopía confocal después de resaltar las mitocondrias utilizando un colorante o marcador fluorescente, así como una configuración de microscopía específica que permite la regulación de la temperatura y/o elCO2 7. Este tipo de configuración experimental implica que se realice una réplica experimental a la vez. Además de la repetición experimental de diferentes tratamientos, se debe considerar que la mayoría de los experimentos deben tener sus réplicas técnicas (donde se visualiza más de una posición por placa), con una serie de planos focales (pilas z) que se registran en una serie de puntos de tiempo. Así, un diseño experimental con tres repeticiones de un control y dos tratamientos, con cinco posiciones de imagen por placa, y 15 puntos de tiempo, da como resultado 225 pilas a procesar. Clásicamente, los videos de mitocondrias vivas se analizaban mediante el trazado de quimógrafos, que se analizaban individualmente8, en un proceso que consumía mucho tiempo y que requería una amplia entrada manual, incluso cuando se dependía de herramientas informáticas.
Recientemente se ha descrito un algoritmo9 que permite la segmentación y el seguimiento automatizados de las mitocondrias en archivos time-lapse 2D y 3D de células vivas. Existen otras técnicas de cuantificación y todas tienen sus limitaciones10. Mitometer, una aplicación automatizada de código abierto, es particularmente adecuada para el análisis de lapso de tiempo y dinámica mitocondrial, que requiere poca participación del usuario. Esta aplicación tiene una serie de ventajas frente a otras herramientas existentes basadas en MATLAB, a saber, permite el procesamiento automático de pilas TIF individuales, utilizando hasta 13 parámetros diferentes, especialmente interesante para las neurociencias, ya que diferencia entre mitocondrias perinucleares y telenucleares.
Sin embargo, para un experimento como el descrito anteriormente, estos 13 parámetros aplicados a 225 pilas dan como resultado 2.925 archivos de salida individuales. Estos requieren cuatro entradas individuales de computadora, que suman más de 10,000 entradas manuales que se requieren para descargar todos los archivos de salida. En el caso de los diseños experimentales de gran envergadura, esto da lugar a un análisis de cada archivo e integración de datos que requiere mucho tiempo. En este trabajo presentamos una optimización rutinaria que permite un análisis rápido de archivos, reduciendo en gran medida la lectura de documentos y el procesamiento de datos, analizando los datos de una manera definida, consistente con la salida de las herramientas existentes.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: Este protocolo tiene dos pasos principales: un paso de laboratorio húmedo, que involucra cultivo celular y microscopía confocal viva para obtener imágenes de mitocondrias vivas (Figura 1) y un paso in silico para analizar las imágenes obtenidas (Figura 2). Para el análisis automatizado de datos de mitocondrias en vivo en 3D, se utilizó la aplicación Mitometer de MATLAB proporcionada por Lefebvre et al.9. La optimización de la rutina está escrita en MATLAB. El software, las versiones actualizadas y el procesamiento de macros de ImageJ están disponibles gratuitamente en línea a través de GitHub, en https://github.com/JoseJoaoMV/Routine_Optimization_Mitometer_APP_MATLAB.
1. Microscopía en vivo
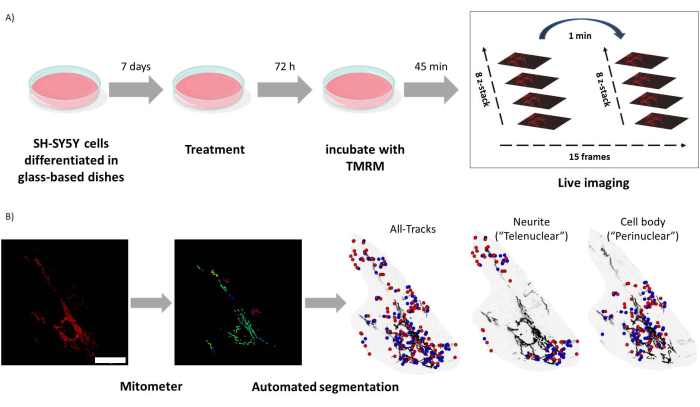
Figura 1: Protocolo experimental. Las células SH-SY5Y se diferenciaron y trataron con retinoides. (A) La TMRM se utilizó para obtener imágenes en vivo de mitocondrias sanas en células tratadas utilizando un microscopio confocal, capturando una pila Z de lapso de tiempo de cinco campos visuales. (B) Aplicación de mitómetros MATLAB segmenta y analiza automáticamente las imágenes de mitocondrias. Además de analizar, este software discrimina automáticamente las mitocondrias en función de la proximidad nuclear. Los puntos azules son las posiciones iniciales de las mitocondrias; Los puntos rojos son las posiciones finales. Barra de escala = 30 μm. Abreviatura: TMRM = tetrametilrodamina, éster metílico. Haga clic aquí para ver una versión más grande de esta figura.
- Cultivo celular
- Incubar células SH-SY5Y a 37 °C en una atmósfera humidificada de 5% de CO2 y 95% de aire, cultivadas en partes iguales de medio MEM (Minimal Essential Medium) y F12 (Ham's F12 Nutrient Mix), suplementado con 10% de suero fetal bovino (FBS).
- Placa de células SH-SY5Y en placas de células con fondo de vidrio a una densidad de 15 × 104 células/ml.
- Diferenciar las células con 5 días de tratamiento con 10 μM de ácido transretinoico all-trans en medio de cultivo que contenga FBS al 1%, seguido de 2 días de tratamiento con 10 ƞg/mL de factor neurotrófico derivado del cerebro (BDNF).
- Tratamiento celular
- Lavar las células con solución salina estéril tamponada con fosfato (PBS) y tratar durante 72 h con agonistas de isoformas RAR 10-7 M, en partes iguales de medio MEM (medio esencial mínimo) y medio F12 (mezcla de nutrientes F12 de Ham), suplementado con FBS al 1%.
NOTA: El agonista RARα utilizado fue AM580; El agonista de RARβ utilizado fue CD2314; Ch55 se utilizó como agonista como RARα y β co-agonista; enSe utilizó la AR como control positivo; El BMS493 se utilizó como pan-antagonista del RAR.
- Lavar las células con solución salina estéril tamponada con fosfato (PBS) y tratar durante 72 h con agonistas de isoformas RAR 10-7 M, en partes iguales de medio MEM (medio esencial mínimo) y medio F12 (mezcla de nutrientes F12 de Ham), suplementado con FBS al 1%.
- Imágenes confocales en vivo
- Reemplace el medio de cultivo con un medio de cultivo fresco que contenga FBS al 1% con 20 nM de tetrametilrodamina, éster metílico (TMRM) durante 45 min.
NOTA: La TMRM es un colorante fluorescente permeable a las células, secuestrado por mitocondrias activas, y este período de incubación permite que la TMRM alcance un equilibrio y sea absorbida por las mitocondrias polarizadas11. La obtención de imágenes debe iniciarse antes de que se establezca el equilibrio, ya que la intensidad de la señal TMRM podría aumentar artificialmente durante la obtención de imágenes. - Coloque las células en una incubadora conectada a un microscopio confocal de barrido láser a 37 °C.
- Capture imágenes utilizando un objetivo apocromático de inmersión en aceite de 63x, con un tamaño de imagen de 512 x 512 píxeles obtenido con una apertura estenopeica de 1 unidad aireada, capturando una serie temporal de 15 fotogramas de cinco campos visuales diferentes en cada placa celular y una pila z de 8 planos z equidistantes. El archivo .lsm resultante es una pila de tiempo, posición y z.
NOTA: Los ajustes de ganancia, contraste y brillo deben optimizarse inicialmente utilizando la potencia láser mínima necesaria para utilizar todo el rango dinámico del detector y mantenerse constantes durante todo el estudio, asegurando que todas las imágenes se realicen en las mismas condiciones. En esta combinación de hardware y software se pueden registrar hasta nueve posiciones diferentes y el microscopio alterna automáticamente entre las posiciones de obtención de imágenes.
- Reemplace el medio de cultivo con un medio de cultivo fresco que contenga FBS al 1% con 20 nM de tetrametilrodamina, éster metílico (TMRM) durante 45 min.
2. Análisis de imágenes
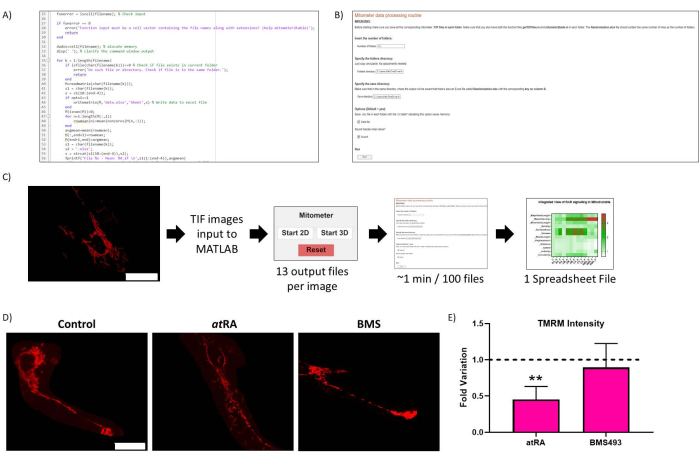
Figura 2: Optimización de rutinas. (A) Código representativo de la optimización de rutinas. (B) Optimización rutinaria Live-Script. (C) Flujo de trabajo de optimización rutinaria. (D) Validación de resultados de optimización de rutina: imagen representativa de mitocondrias en células no tratadas (panel izquierdo), tratadas con AT (10-7 M, 72 h, panel central) y tratadas con elantagonista RAR BMS493 (10-7 M, 72 h, panel derecho), fotografiadas después de la incubación con TMRM (20 nM, 45 min de incubación). Barra de escala = 30 μm. (E) TMRM Intensidad en las mitocondrias del cuerpo celular. Disminución significativa con el tratamiento con ácido transretinoico (enAR, 10-7 M, 72 h), en comparación con el control (p=0,0062), no observada cuando se trató con antagonista de RAR (BMS493, 10-7 M, 72 horas). Se cuantificaron cinco células de cada una de las tres repeticiones por condición. Haga clic aquí para ver una versión más grande de esta figura.
- Tratamiento de imágenes
- Abra archivos en ImageJ 2.1.0 y separe las pilas de posiciones por campo visual: Abra ImageJ y haga clic en Barra de menús | Imagen | Duplicar | Segmentos de entrada/número de fotograma | De acuerdo.
NOTA: Para reducir las entradas repetitivas en el software, se desarrolló una macro ImageJ para facilitar la duplicación y el almacenamiento de imágenes del campo visual.
- Abra archivos en ImageJ 2.1.0 y separe las pilas de posiciones por campo visual: Abra ImageJ y haga clic en Barra de menús | Imagen | Duplicar | Segmentos de entrada/número de fotograma | De acuerdo.
- Protocolo de macros
- Dibuje una región de interés (ROI) alrededor de la celda con la herramienta de selección libre.
- Ejecute la macro abriendo ImageJ y navegando a la barra de menús | Plugins | Macros | Ejecute la macro Borrar el fondo y guardar imágenes.
NOTA: Las imágenes se pueden aleatorizar en este proceso, almacenando la clave de la solución de acuerdo con los requisitos de optimización. - Determinar el tamaño de píxel y la profundidad del vóxel: Abra ImageJ | la imagen y navegue hasta la barra de menús | Imagen | Propiedades.
- Protocolo directo alternativo
NOTA: Esta alternativa no utiliza macros para procesar imágenes- Encuentre el tamaño de píxel y la profundidad del vóxel: Abrir ImageJ | Abra Imagen y navegue hasta Barra de menú | Imagen | Propiedades.
- Dibuje un ROI alrededor de la celda con la herramienta de selección libre, asegurándose de que abarque toda la celda a lo largo de los 15 fotogramas.
- Vaya a la barra de menús | Editar | Despeje el exterior.
- Vaya a la barra de menús | Archivo | Guardar como | Seleccione Tiff.
- Seleccione Guardar carpeta y haga clic en Guardar.
NOTA: Las imágenes se pueden cegar/aleatorizar manualmente en este punto antes de continuar con el análisis.
- Análisis automatizado de imágenes
- Prepare las carpetas para los archivos de análisis.
- Cree tres carpetas principales, tituladas "Todas las pistas", "Pistas perinucleares" y "Pistas telenucleares".
NOTA: Estos coinciden con las principales opciones de pistas automatizadas. - En cada carpeta principal, cree una subcarpeta para cada imagen que se va a procesar, identificada numéricamente desde 1 en adelante.
- Agregue una copia de los dos archivos complementarios de optimización de rutina (mitometer2table.m y getTXTfiles.m) a cada carpeta de imágenes.
NOTA: Estos archivos ayudan en el análisis de datos y en la disposición final del formato. El número de carpetas debe coincidir con el número de elementos de la hoja de cálculo aleatoria (.xlsx). Después de crear todas las subcarpetas numeradas con archivos complementarios para un conjunto de datos, se pueden copiar por lotes y pegar en los conjuntos de datos restantes.
- Cree tres carpetas principales, tituladas "Todas las pistas", "Pistas perinucleares" y "Pistas telenucleares".
- Prepare las carpetas para los archivos de análisis.
- Utilice la aplicación Mitometer de MATLAB para analizar imágenes.
NOTA: Este protocolo se ejecutó en un mitómetro, instalado en MATLAB R2022a. Cargue imágenes en lotes de 30 para optimizar el tiempo de ejecución y el equilibrio de salida. El tamaño máximo del archivo MAT lo impone el sistema de archivos nativo: de forma predeterminada, las operaciones de "guardar" pueden crear un archivo < 231 bytes (~2 GB); En su lugar, se puede utilizar la versión 7.3 de MAT-Files, ya que permite tamaños variables máximos superiores a 2 GB.- Identifique/modifique la versión predeterminada del archivo MAT: en la pestaña Inicio de la sección Entorno, haga clic en Preferencias y seleccione MATLAB | Generalidades | Archivos MAT.
- Utilice la herramienta de MATLAB Mitómetro para analizar: Abra MATLAB y vaya a la barra de menús | APLICACIONES | Mitómetro abierto | Seleccione Iniciar 3D | Datos de entrada (Tamaño de píxel (μm): 0,1395089/Tiempo entre fotogramas (s): 2/Número de planos z: 8/Distancia axial entre planos z (μm): 0,418809 | Seleccione las imágenes que desea introducir.
- Vaya al menú lateral del mitómetro, seleccione Imagen, haga clic en Seleccionar resaltado, navegue hasta la barra de menú del mitómetro | Seleccionar pistas | Vistas de pista | seleccione Todas las pistas, Telenuclear o Perinuclear | Barra de menú de mitómetro | Seleccionar análisis | Elija un elemento (es decir, Longitud) | seleccione Guardar en ".txt".
NOTA: Se puede descargar más de un parámetro a la vez, si se selecciona concomitantemente. - Extraiga los archivos de resultados en las carpetas creadas (2.2.1).
- Optimización rutinaria y análisis de datos
- Prepare/adapte el archivo "Randomization.xlsx" que contiene la clave para la codificación de imágenes.
- Inserte una lista de enteros consecutivos, de 1 en adelante, en la columna A.
NOTA: Es recomendable mantener una carpeta duplicada con las imágenes nombradas originalmente. - Coloque las variables analíticas en la columna B, compuesta por caracteres alfanuméricos.
NOTA: El número de líneas del documento debe ser coherente con el número de carpetas presentes en el conjunto de datos principal. Copie y pegue este archivo "Randomization.xlsx" en los otros dos conjuntos de datos principales.
- Inserte una lista de enteros consecutivos, de 1 en adelante, en la columna A.
- Análisis de datos optimizado
- Haga doble clic en "ejecutable.mlx", ingrese el número de carpetas, especifique el directorio de carpetas (copie el directorio de la parte superior de la carpeta) | el directorio de guardado (copie el directorio de la parte superior de la carpeta) | el nombre de la hoja de cálculo en el archivo de salida y haga clic en Ejecutar.
- Realice análisis estadísticos según sea necesario.
NOTA: Se puede seleccionar la creación opcional de un archivo .xlsx en cada carpeta con los datos .txt y la alerta sonora al final del análisis. Live Script genera un único archivo de hoja de cálculo en formato de tabla. En este resultado, las columnas representan los parámetros analizados (por ejemplo, "Longitud del eje principal"; "Intensidad") y las líneas representan campos visuales de variables analíticas (por ejemplo, "Control").
- Prepare/adapte el archivo "Randomization.xlsx" que contiene la clave para la codificación de imágenes.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para mejorar y acelerar el análisis de los archivos de salida en formato .txt, se codificó una optimización de rutina que lee datos consistentes con los archivos de salida de Mitómetro .txt, con columnas que representan un marco y líneas que representan mitocondrias identificadas. La optimización de rutina produce datos en un solo valor por parámetro promediando los fotogramas de cada mitocondria identificada y, a continuación, promediando los resultados de todas las mitocondrias por campo visual. La rutina desar...
Access restricted. Please log in or start a trial to view this content.
Discusión
Las imágenes de células vivas producen archivos de gran tamaño que requieren un procesamiento informático serio, pero incluso las herramientas más recientes requieren una amplia entrada manual para procesarlas. Esta optimización rutinaria se centra en simplificar el proceso de análisis de mitocondrias en el mitómetro, ya que esta herramienta presenta un muy buen equilibrio entre la entrada del usuario y la salida de datos. Previamente se ha revisado una comparación exhaustiva entre diferentes herramientas para e...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
La adquisición de imágenes se realizó en la instalación LiM de iBiMED, un nodo de PPBI (Plataforma Portuguesa de BioImagen): POCI-01-0145-FEDER-022122. Este trabajo contó con el apoyo de la FCT (EXPL/BTM-SAL/0902/2021), LCF (CI21-00276), una beca a DT de la Fundação para a Ciência e Tecnologia del Ministério da Educação e Ciência (2020.02006.CEECIND), una beca de ATG-Asociación de Antiguos Alumnos de Gabba a VP y del Instituto de Biomedicina-iBiMED, Universidad de Aveiro.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| AM580 | Sigma-Aldrich | A8843 | |
| BDNF | Thermo-Fisher | RP8642 | |
| BMS493 | Tocris Bioscience | 3409 | |
| CD2314 | Tocris Bioscience | 3824 | |
| Ch55 | Tocris Bioscience | 2020 | |
| Foetal Bovine Serum | Thermo-Fisher | 10270106 | |
| GraphPad Prism v4.0 | GraphPad Software, La Jolla | n/a | |
| Ham’s F12 Nutrient Mix | Thermo-Fisher | 21765029 | |
| MATLAB R2022a | MathWorks | n/a | |
| Minimal Essential Medium | Thermo-Fisher | 31095 | |
| Nunc Glass Bottom Dishes | Thermo-Fisher | 150680 | |
| Phosphate Buffer Saline Solution | Thermo-Fisher | 28372 | |
| Retinoic acid | Sigma-Aldrich | R2625 | |
| TMRM | Thermo-Fisher | T668 | |
| Zeiss LSM 510 | Carl Zeiss | n/a | Equiped with live-cell imaging culture chamber and 63x oil immersion objective |
Referencias
- Trigo, D., Avelar, C., Fernandes, M., Sa, J., da Cruz, E. S. O. Mitochondria, energy, and metabolism in neuronal health and disease. FEBS Letters. 596 (9), 1095-1110 (2022).
- Zong, W. X., Rabinowitz, J. D., White, E. Mitochondria and cancer. Molecular Cell. 61 (5), 667-676 (2016).
- Clare, D. K., Saibil, H. R. ATP-driven molecular chaperone machines. Biopolymers. 99 (11), 846-859 (2013).
- Tourniaire, F., et al. All-trans retinoic acid induces oxidative phosphorylation and mitochondria biogenesis in adipocytes. Journal of Lipid Research. 56 (6), 1100-1109 (2015).
- Psarra, A. M., Sekeris, C. E. Nuclear receptors and other nuclear transcription factors in mitochondria: regulatory molecules in a new environment. Biochimica et Biophysica Acta. 1783 (1), 1-11 (2008).
- Trigo, D., Goncalves, M. B., Corcoran, J. P. T. The regulation of mitochondrial dynamics in neurite outgrowth by retinoic acid receptor beta signaling. FASEB Journal. 33 (6), 7225-7235 (2019).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Current Protocols in Cell Biology. Chapter 4 (Unit 4), 1-21 (2010).
- Sajic, M., et al. Impulse conduction increases mitochondrial transport in adult mammalian peripheral nerves in vivo. PLoS Biology. 11 (12), e1001754(2013).
- Lefebvre, A., Ma, D., Kessenbrock, K., Lawson, D. A., Digman, M. A. Automated segmentation and tracking of mitochondria in live-cell time-lapse images. Nature Methods. 18 (9), 1091-1102 (2021).
- Chu, C. -H., Tseng, W. -W., Hsu, C. -M., Wei, A. -C. Image analysis of the mitochondrial network morphology with applications in cancer research. Frontiers in Physics. 10, 855775(2022).
- Creed, S., McKenzie, M. Measurement of mitochondrial membrane potential with the fluorescent dye tetramethylrhodamine methyl ester (TMRM). Methods in Molecular Biology. 1928, 69-76 (2019).
- Kovalevich, J., Langford, D. Considerations for the use of SH-SY5Y neuroblastoma cells in neurobiology. Methods in Molecular Biology. 1078, 9-21 (2013).
- Sahin, M., Oncu, G., Yilmaz, M. A., Ozkan, D., Saybasili, H. Transformation of SH-SY5Y cell line into neuron-like cells: Investigation of electrophysiological and biomechanical changes. Neuroscience Letters. 745, 135628(2021).
- Trigo, D., et al. Mitochondria dysfunction and impaired response to oxidative stress promotes proteostasis disruption in aged human cells. Mitochondrion. 69, 1-9 (2022).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados