JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
동형 특이적 레티노산 수용체에 의한 살아있는 신경 미토콘드리아 항상성 조절의 최적화된 자동 분석
요약
미토콘드리아 네트워크는 매우 복잡하여 분석하기가 매우 어렵습니다. 새로운 MATLAB 툴은 타임랩스 이미지에서 실시간 컨포칼 이미징 미토콘드리아를 분석하지만, 출력 볼륨이 커서 개별 수동 주의가 필요합니다. 이 문제를 해결하기 위해 신속한 파일 분석이 가능한 루틴 최적화가 개발되었습니다.
초록
복잡한 미토콘드리아 네트워크는 살아있는 세포를 분할, 추적 및 분석하는 것을 매우 어렵게 만듭니다. MATLAB 툴을 사용하면 타임랩스 파일의 미토콘드리아를 분석할 수 있어 이미지 처리 프로세스가 상당히 간소화되고 속도가 빨라집니다. 그럼에도 불구하고 기존 도구는 많은 양의 출력을 생성하므로 개별적인 수동 주의가 필요하며, 기본 실험 설정에는 수천 개의 파일이 출력되어 각각 광범위하고 시간이 많이 소요되는 처리가 필요합니다.
이러한 문제를 해결하기 위해 MATLAB 코드 및 라이브 스크립트 형식 모두에서 루틴 최적화가 개발되어 신속한 파일 분석이 가능하고 문서 읽기 및 데이터 처리가 크게 줄었습니다. 100개 파일/분의 속도로 최적화를 통해 전반적으로 빠른 분석이 가능합니다. 최적화는 시간 프레임 전반에 걸쳐 개별 미토콘드리아에 대한 프레임별 데이터의 평균을 구하고 기존 도구의 출력과 일치하는 정의된 방식으로 데이터를 분석하여 결과 출력을 달성합니다. 염료 테트라메틸로다민 메틸 에스테르를 사용하여 실시간 공초점 이미징을 수행했으며, 신경 세포 미토콘드리아에 대한 효과가 문헌에 확립된 레티노산 수용체(RAR) 작용제로 신경 세포를 처리하여 일상적인 최적화를 검증했습니다. 결과는 문헌과 일치했으며 동형 특이적 RAR 변조에 대한 반응으로 미토콘드리아 네트워크 행동의 추가 특성화를 허용했습니다.
이 새로운 방법론은 전체 뉴런 미토콘드리아 네트워크의 빠르고 검증된 특성 분석을 가능하게 할 뿐만 아니라 신경과학 분야에 적용하기 위한 필수 기능인 축삭돌기와 세포체 미토콘드리아를 구별할 수 있게 해줍니다. 또한, 이 프로토콜은 속효성 치료법을 사용하는 실험에 적용할 수 있어 치료 전후에 동일한 세포를 이미징할 수 있어 신경 과학 분야를 초월합니다.
서문
세포 미토콘드리아는 모든 생리적 상태의 중심에 있으며, 세포 미토콘드리아의 항상성(유사분열)과 행동에 대한 철저한 이해는 암 및 알츠하이머병을 포함한 광범위한 질병에 대한 약리학적 치료법을 식별하는 데 가장 중요합니다 1,2.
미토콘드리아는 에너지 항상성, ATP 생성, 칼슘 완충 및 ROS 조절에 중요한 세포 역할을 하며, 분자 샤페론은 에너지 의존적이기 때문에 단백질 항상성을 유지하는 데 유사분열은 필수적입니다3. 이를 위해서는 세포의 요구를 효율적으로 충족하기 위해 지속적이고 역동적인 네트워크 변조 및 적응이 필요하며, 미토콘드리아 수송은 다양한 신호 경로에 의해 조절됩니다. 이전 연구에서는 이러한 경로 중 하나인 레티노산 수용체(RAR)4,5에 대해 설명했습니다. 레티노산(RA)은 RAR 활성화를 통해 축삭돌기 및 신경돌기 성장을 촉진합니다. 생쥐의 일차 피질 뉴런에서 RAR-β의 활성화는 신경돌기의 미토콘드리아 성장, 속도 및 이동성을 촉진한다6.
미토콘드리아 네트워크 적응성과 역동성을 고려할 때, "실시간"으로 유사분열을 평가할 수 있는 가능성은 에너지 항상성을 조사하는 것뿐만 아니라 단백질 항상성, 세포 건강, 증식 또는 신호 전달을 조사하는 데에도 필수적입니다. 유사분열을 평가하기 위해 일반적으로 사용되는 방법은 형광 염료 또는 마커를 사용하여 미토콘드리아를 강조 표시한 후 컨포칼 현미경 검사와 온도 및/또는CO2 조절을 허용하는 특정 현미경 설정에 의존합니다7. 이러한 유형의 실험 설정에서는 한 번에 하나의 실험 반복실험을 수행해야 합니다. 다양한 처리의 실험적 반복 외에도 대부분의 실험에는 일련의 시점에 일련의 초점면(z-스택)이 기록되는 일련의 초점면(z-스택)과 함께 기술적 복제(플레이트당 두 개 이상의 위치가 이미지화됨)가 있어야 한다는 점을 고려해야 합니다. 따라서 1개의 대조군과 2개의 처리를 3회 반복하고 플레이트당 5개의 이미징 위치와 15개의 시점을 사용하는 실험 설계에서는 225개의 스택을 처리해야 합니다. 전통적으로 살아있는 미토콘드리아의 비디오는 카이모그래프를 플로팅하여 분석했으며, 이 그래프는 컴퓨터 도구에 의존하는 경우에도 광범위한 수동 입력이 필요한 시간 소모적인 프로세스에서 개별적으로 분석되었습니다8.
최근 라이브 셀 2D 및 3D 타임랩스 파일에서 미토콘드리아의 자동 분할 및 추적을 가능하게 하는 알고리즘이 설명되었습니다(9). 다른 정량화 기법을 사용할 수 있으며, 모두 한계가 있습니다10. 자동화된 오픈 소스 애플리케이션인 Mitometer는 시간 경과 및 미토콘드리아 역학 분석에 특히 적합하며 사용자 입력이 적습니다. 이 응용 프로그램은 기존의 다른 MATLAB 기반 도구에 비해 여러 가지 장점이 있는데, 최대 13개의 서로 다른 파라미터를 사용하여 개별 TIF 스택을 자동으로 처리할 수 있다는 점으로, 특히 신경 과학에서 흥미로운 점은 핵 주위 미토콘드리아와 원격 핵 미토콘드리아를 구분하기 때문입니다.
그러나 위에서 설명한 것과 같은 실험의 경우 이러한 13개의 매개 변수를 225개의 스택에 적용하면 2,925개의 개별 출력 파일이 생성됩니다. 이를 위해서는 4개의 개별 컴퓨터 입력이 필요하며, 모든 출력 파일을 다운로드하는 데 최대 10,000개 이상의 수동 입력이 필요합니다. 대규모 실험 설계의 경우 이로 인해 각 파일 및 데이터 통합에 대한 분석에 불필요하게 시간이 많이 소요됩니다. 여기에서는 신속한 파일 분석을 가능하게 하는 일상적인 최적화를 제시하여 문서 읽기 및 데이터 처리를 크게 줄이고 기존 도구의 출력과 일치하는 정의된 방식으로 데이터를 분석합니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
참고: 이 프로토콜에는 살아있는 미토콘드리아의 이미지를 얻기 위해 세포 배양 및 살아있는 공초점 현미경을 사용하는 습식 실험실 단계(그림 1)와 얻은 이미지를 분석하기 위한 인실리코 단계(그림 2)의 두 가지 주요 단계가 있습니다. 3D 실시간 이미징 미토콘드리아의 자동화된 데이터 분석을 위해 Lefebvre et al.9에서 제공한 MATLAB 응용 프로그램 Mitometer를 사용했습니다. 루틴 최적화는 MATLAB으로 작성되었습니다. 소프트웨어, 업데이트된 버전 및 처리 ImageJ 매크로는 GitHub(https://github.com/JoseJoaoMV/Routine_Optimization_Mitometer_APP_MATLAB)를 통해 온라인으로 무료로 사용할 수 있습니다.
1. 살아있는 현미경 검사
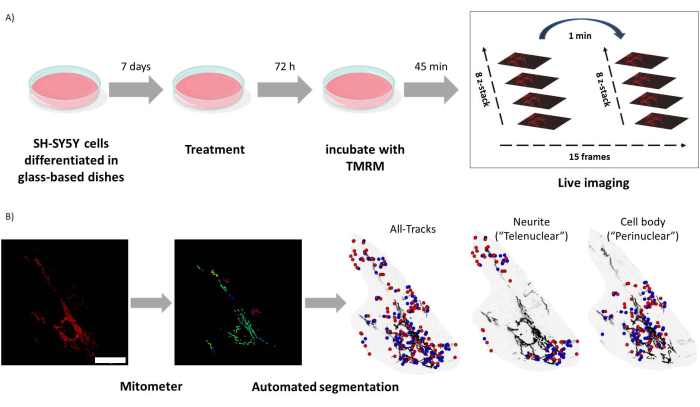
그림 1: 실험적 프로토콜. SH-SY5Y 세포를 분화하고 레티노이드로 처리했습니다. (A) TMRM은 컨포칼 현미경을 사용하여 처리된 세포의 건강한 미토콘드리아를 생생하게 이미지화하는 데 사용되었으며, 5개의 시야로 구성된 타임 랩스 z-스택을 캡처했습니다. (B) Mitometer 응용 프로그램 MATLAB은 미토콘드리아 영상을 자동으로 분할하고 분석합니다. 분석 외에도 이 소프트웨어는 핵 근접성에 따라 미토콘드리아를 자동으로 구별합니다. 파란색 점은 미토콘드리아의 초기 위치입니다. 빨간색 점은 최종 위치입니다. 눈금 막대 = 30μm. 약어: TMRM = 테트라메틸로다민, 메틸 에스테르. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 세포 배양
- 37°C에서 5%CO2 및 95% 공기의 가습된 분위기에서 MEM(Minimal Essential Medium) 및 F12(Ham's F12 Nutrient Mix) 배지를 동일한 비율로 배양하고 10% 소 태아 혈청(FBS)을 보충하여 SH-SY5Y 세포를 배양합니다.
- 유리 바닥 셀 접시에 SH-SY5Y 세포를 15 × 104 cells/mL의 밀도로 플레이트합니다.
- 1% FBS 함유 배양 배지에서 10μM all-trans 레티노산으로 5일 동안 처리한 후 10ƞg/mL 뇌 유래 신경 영양 인자(BDNF)로 2일 동안 처리하여 세포를 분화합니다.
- 세포 처리
- 멸균 인산염 완충 식염수(PBS)로 세포를 세척하고 MEM(최소 필수 배지) 및 F12(Ham's F12 영양 믹스) 배지를 동일한 비율로 10-7 M RAR 동형 작용제로 72시간 동안 처리하고 1% FBS를 보충합니다.
참고: 사용된 RARα 작용제는 AM580이었습니다. 사용된 RARβ 작용제는 CD2314였다. Ch55는 RARα 및 β 공동 작용제로서 작용제로 사용되었습니다. 에RA는 양성 대조군으로 사용되었습니다. BMS493은 RAR 팬 길항제로 사용되었습니다.
- 멸균 인산염 완충 식염수(PBS)로 세포를 세척하고 MEM(최소 필수 배지) 및 F12(Ham's F12 영양 믹스) 배지를 동일한 비율로 10-7 M RAR 동형 작용제로 72시간 동안 처리하고 1% FBS를 보충합니다.
- 실시간 공초점 이미징
- 배양 배지를 20nM 테트라메틸로다민, 메틸 에스테르(TMRM)가 포함된 신선한 1% FBS 함유 배양 배지로 45분 동안 교체합니다.
참고: TMRM은 활성 미토콘드리아에 의해 격리된 세포 투과성 형광 염료이며, 이 잠복기를 통해 TMRM은 평형에 도달하고 편광 미토콘드리아에 의해 흡수될 수 있습니다11. 이미징 중에 TMRM 신호 강도가 인위적으로 증가할 수 있으므로 평형이 설정되기 전에 이미징을 시작해야 합니다. - 37°C에서 레이저 스캐닝 컨포칼 현미경에 부착된 인큐베이터에 세포를 넣습니다.
- 63x 오일 이멀젼 아포크로맷 대물렌즈를 사용하여 1 airy 단위의 핀홀 조리개로 얻은 512 x 512 픽셀의 이미지 크기로 각 셀 플레이트의 5가지 서로 다른 시야에서 15개 프레임의 시계열과 8개의 등거리 z 평면으로 구성된 z 스택을 캡처합니다. 결과 .lsm 파일은 시간, 위치 및 z 스택입니다.
알림: 게인, 대비 및 밝기 설정은 처음에 검출기의 전체 동적 범위를 사용하는 데 필요한 최소 레이저 출력을 사용하여 최적화해야 하며 모든 이미징이 동일한 조건에서 수행되도록 연구 전반에 걸쳐 일정하게 유지되어야 합니다. 이 하드웨어-소프트웨어 조합에서 최대 9개의 서로 다른 위치를 기록할 수 있으며 현미경은 이미징 위치 사이를 자동으로 순환합니다.
- 배양 배지를 20nM 테트라메틸로다민, 메틸 에스테르(TMRM)가 포함된 신선한 1% FBS 함유 배양 배지로 45분 동안 교체합니다.
2. 이미지 분석
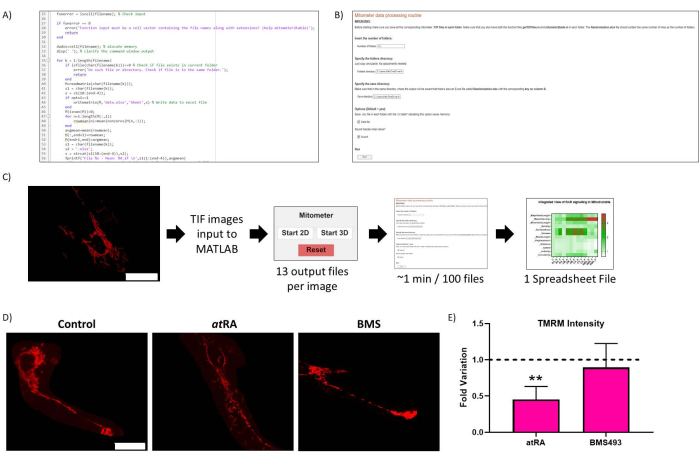
그림 2: 루틴 최적화. (A) 루틴 최적화의 대표 코드. (B) 루틴 최적화 라이브 스크립트. (C) 일상적인 최적화 작업 흐름. (D) 일상적인 최적화 결과 검증: 처리되지 않은 세포(왼쪽 패널)에서 미토콘드리아의 대표 이미지, RA(10-7M, 72시간, 중간 패널)에서처리하고 RAR 길항제 BMS493(10-7M, 72시간, 오른쪽 패널)으로 처리, TMRM(20nM, 45분 배양)으로 배양 후 이미지화. 척도 막대 = 30 μm. (E) TMRM 세포체 미토콘드리아의 강도. 대조군(p=0,0062)과 비교하여 all-trans 레티노산(RA에서, 10-7M, 72시간)으로 유의한 감소를 보였으며, RAR 길항제(BMS493, 10-7 M, 72시간)로 치료했을 때는 관찰되지 않았습니다. 조건당 3회 반복 각각에서 5개의 세포를 정량화했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 이미지 처리
- ImageJ 2.1.0에서 파일을 열고 시야각별로 위치 스택을 구분합니다. ImageJ를 열고 메뉴 모음 | 이미지 | 복제 | 입력 슬라이스/프레임 번호 | 확인.
알림: 소프트웨어에 대한 반복적인 입력을 줄이기 위해 ImageJ 매크로가 개발되어 시야 이미지의 복제 및 저장이 용이합니다.
- ImageJ 2.1.0에서 파일을 열고 시야각별로 위치 스택을 구분합니다. ImageJ를 열고 메뉴 모음 | 이미지 | 복제 | 입력 슬라이스/프레임 번호 | 확인.
- 매크로 프로토콜
- 자유 선택 도구를 사용하여 셀 주위에 관심 영역(ROI)을 그립니다.
- ImageJ를 열고 메뉴 모음 | 플러그인 | 매크로 | 배경 지우기 및 이미지 저장 매크로를 실행합니다.
참고: 이 프로세스에서 이미지를 무작위화하여 최적화 요구 사항에 따라 솔루션 키를 저장할 수 있습니다. - 픽셀 크기 및 복셀 심도 결정: ImageJ | 이미지를 열고 메뉴 모음 | 이미지 | 속성.
- 대체 직접 프로토콜
참고: 이 대안은 매크로를 사용하여 이미지를 처리하지 않습니다- 픽셀 크기 및 복셀 심도 찾기: ImageJ 열기 | Image(이미지)를 열고 Menu Bar(메뉴 모음) | 이미지 | 속성.
- 자유 선택 도구를 사용하여 셀 주위에 ROI를 그려 15개 프레임 전체에 걸쳐 전체 셀을 포함하도록 합니다.
- Menu Bar(메뉴 모음) | 편집 | 외부를 비우십시오.
- Menu Bar(메뉴 모음) | 파일 | 다른 이름으로 저장 | Tiff를 선택합니다.
- Saving Folder(폴더 저장)를 선택하고 Save(저장)를 클릭합니다.
참고: 분석을 계속하기 전에 이 시점에서 이미지를 수동으로 블라인딩/무작위화할 수 있습니다.
- 자동화된 이미지 분석
- 분석 파일에 대한 폴더를 준비합니다.
- "All tracks", "Perinuclear tracks" 및 "Telenuclear tracks"라는 제목의 세 가지 기본 폴더를 만듭니다.
알림: 이는 기본 자동 트랙 옵션과 일치합니다. - 각 기본 폴더에서 처리할 각 이미지에 대한 하위 폴더를 1부터 숫자로 식별합니다.
- 루틴 최적화 보조 파일 2개(mitometer2table.m 및 getTXTfiles.m)의 복사본을 각 영상 폴더에 추가합니다.
참고: 이 파일은 데이터 분석 및 최종 형식 정렬에 도움이 됩니다. 폴더 수는 무작위 스프레드시트(.xlsx)의 요소 수와 일치해야 합니다. 하나의 데이터 세트에 대한 보조 파일이 있는 번호가 매겨진 모든 하위 폴더를 만든 후 일괄 복사하여 나머지 데이터 세트에 붙여넣을 수 있습니다.
- "All tracks", "Perinuclear tracks" 및 "Telenuclear tracks"라는 제목의 세 가지 기본 폴더를 만듭니다.
- 분석 파일에 대한 폴더를 준비합니다.
- MATLAB 응용 프로그램 Mitometer를 사용하여 영상을 분석합니다.
참고: 이 프로토콜은 MATLAB R2022a에 설치된 mitometer에서 실행되었습니다. 최적의 시간 실행 및 출력 균형을 위해 30개의 배치로 이미지를 로드합니다. MAT 파일의 최대 크기는 네이티브 파일 시스템에 의해 결정되며, 기본적으로 "저장" 작업은 231 바이트(~2GB)< 파일을 만들 수 있습니다. 저장 형식 MAT 파일 버전 7.3을 대신 사용할 수 있는데, 이 경우 최대 변수 크기가 2GB보다 큽니다.- 디폴트 MAT 파일 버전 식별/변경: 홈(Home ) 탭의 환경( Environment ) 섹션에서 기본 설정(Preferences )을 클릭하고 MATLAB | 일반 | MAT 파일.
- MATLAB 툴 Mitometer를 사용하여 분석: MATLAB을 열고 메뉴 모음 | 앱 | 개방 Mitometer | 3D 시작 | 입력 데이터 (픽셀 크기(μm): 0.1395089/프레임 간격: 2/Z 평면 수: 8/Z 평면 사이의 축 방향 거리(μm): 0.418809 | 입력할 이미지를 선택합니다.
- Mitometer Side Menu(마이토미터 사이드 메뉴)로 이동하여 Image(이미지)를 선택하고 Select Highlighted(강조 표시된 선택)를 클릭한 다음 Mitometer Menu Bar(마이토미터 메뉴 표시줄)로 이동합니다 . 트랙 선택 | 조회수 추적 | All-Tracks, Telenuclear 또는 Perinuclear | mitometer 메뉴 모음 | 분석 | 요소 선택 (예: 길이) | ".txt"에 저장을 선택합니다.
알림: 동시에 선택한 경우 한 번에 두 개 이상의 매개변수를 다운로드할 수 있습니다. - 생성된 폴더에 결과 파일을 추출합니다(2.2.1).
- 일상적인 최적화 및 데이터 분석
- 이미지 인코딩을 위한 키가 포함된 "Randomization.xlsx" 파일을 준비/조정합니다.
- 1부터 연속된 정수 목록을 열 A에 삽입합니다.
참고: 원래 이름이 지정된 이미지와 함께 복제된 폴더를 유지하는 것이 좋습니다. - 영숫자로 구성된 B 열에 분석 변수를 배치합니다.
참고: 문서의 줄 수는 기본 데이터 세트에 있는 폴더 수와 일치해야 합니다. 이 "Randomization.xlsx" 파일을 복사하여 다른 두 개의 기본 데이터 세트에 붙여넣습니다.
- 1부터 연속된 정수 목록을 열 A에 삽입합니다.
- 최적화된 데이터 분석
- "executable.mlx"를 두 번 클릭하고, 폴더 수를 입력하고, 폴더 디렉토리를 지정합니다 (폴더 상단에서 디렉토리 복사) | 저장 디렉토리 (폴더 상단에서 디렉토리 복사) | 출력 파일의 스프레드 시트 이름을 지정 하고 실행을 클릭하십시오.
- 필요에 따라 통계 분석을 수행합니다.
참고: 각 폴더에 .txt 데이터와 분석 종료에 대한 경고음이 있는 .xlsx 파일을 선택적으로 생성하도록 선택할 수 있습니다. 라이브 스크립트는 단일 스프레드시트 파일을 테이블 형식으로 출력합니다. 이 출력에서 열은 분석된 매개변수(예 : "Major Axis Length"; "강도") 및 선은 분석 변수의 시각적 필드(예: "제어")를 나타냅니다.
- 이미지 인코딩을 위한 키가 포함된 "Randomization.xlsx" 파일을 준비/조정합니다.
Access restricted. Please log in or start a trial to view this content.
결과
.txt 형식의 출력 파일 분석을 강화하고 가속화하기 위해 Mitometer .txt 출력 파일과 일치하는 데이터를 읽는 루틴 최적화가 코딩되었으며, 열은 프레임을 나타내고 선은 식별된 미토콘드리아를 나타냅니다. 루틴 최적화는 식별된 각 미토콘드리아의 프레임을 평균화한 다음 시야당 모든 미토콘드리아의 결과를 평균화하여 매개변수당 단일 값으로 데이터를 생성합니다. 개발된 루틴은 1부터 번호가 ?...
Access restricted. Please log in or start a trial to view this content.
토론
라이브 셀 이미징은 고도의 컴퓨팅 처리가 필요한 대용량 파일을 생성하지만, 최신 도구조차도 처리하려면 광범위한 수동 입력이 필요합니다. 이 루틴 최적화는 Mitometer에서 미토콘드리아 분석 프로세스를 단순화하는 데 중점을 두는데, 이 도구는 사용자 입력과 데이터 출력 간에 매우 좋은 균형을 제공하기 때문입니다. 미토콘드리아 이미지 분석을 위한 다양한 도구 간의 포괄적인 비교는 이전?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 선언할 이해 상충이 없습니다.
감사의 말
이미지 획득은 PPBI(Portuguese Platform of BioImaging): POCI-01-0145-FEDER-022122의 노드인 iBiMED의 LiM 시설에서 수행되었습니다. 이 연구는 FCT (EXPL/BTM-SAL/0902/2021) LCF(CI21-00276), Fundação para a Ciência e Tecnologia of the Ministério da Educação e Ciência (2020.02006.CEECIND)의 DT 보조금, ATG-The Gabba Alumni Association의 VP 보조금 및 Institute for Biomedicine-iBiMED의 Aveiro 대학의 지원을 받았습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| AM580 | Sigma-Aldrich | A8843 | |
| BDNF | Thermo-Fisher | RP8642 | |
| BMS493 | Tocris Bioscience | 3409 | |
| CD2314 | Tocris Bioscience | 3824 | |
| Ch55 | Tocris Bioscience | 2020 | |
| Foetal Bovine Serum | Thermo-Fisher | 10270106 | |
| GraphPad Prism v4.0 | GraphPad Software, La Jolla | n/a | |
| Ham’s F12 Nutrient Mix | Thermo-Fisher | 21765029 | |
| MATLAB R2022a | MathWorks | n/a | |
| Minimal Essential Medium | Thermo-Fisher | 31095 | |
| Nunc Glass Bottom Dishes | Thermo-Fisher | 150680 | |
| Phosphate Buffer Saline Solution | Thermo-Fisher | 28372 | |
| Retinoic acid | Sigma-Aldrich | R2625 | |
| TMRM | Thermo-Fisher | T668 | |
| Zeiss LSM 510 | Carl Zeiss | n/a | Equiped with live-cell imaging culture chamber and 63x oil immersion objective |
참고문헌
- Trigo, D., Avelar, C., Fernandes, M., Sa, J., da Cruz, E. S. O. Mitochondria, energy, and metabolism in neuronal health and disease. FEBS Letters. 596 (9), 1095-1110 (2022).
- Zong, W. X., Rabinowitz, J. D., White, E. Mitochondria and cancer. Molecular Cell. 61 (5), 667-676 (2016).
- Clare, D. K., Saibil, H. R. ATP-driven molecular chaperone machines. Biopolymers. 99 (11), 846-859 (2013).
- Tourniaire, F., et al. All-trans retinoic acid induces oxidative phosphorylation and mitochondria biogenesis in adipocytes. Journal of Lipid Research. 56 (6), 1100-1109 (2015).
- Psarra, A. M., Sekeris, C. E. Nuclear receptors and other nuclear transcription factors in mitochondria: regulatory molecules in a new environment. Biochimica et Biophysica Acta. 1783 (1), 1-11 (2008).
- Trigo, D., Goncalves, M. B., Corcoran, J. P. T. The regulation of mitochondrial dynamics in neurite outgrowth by retinoic acid receptor beta signaling. FASEB Journal. 33 (6), 7225-7235 (2019).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Current Protocols in Cell Biology. Chapter 4 (Unit 4), 1-21 (2010).
- Sajic, M., et al. Impulse conduction increases mitochondrial transport in adult mammalian peripheral nerves in vivo. PLoS Biology. 11 (12), e1001754(2013).
- Lefebvre, A., Ma, D., Kessenbrock, K., Lawson, D. A., Digman, M. A. Automated segmentation and tracking of mitochondria in live-cell time-lapse images. Nature Methods. 18 (9), 1091-1102 (2021).
- Chu, C. -H., Tseng, W. -W., Hsu, C. -M., Wei, A. -C. Image analysis of the mitochondrial network morphology with applications in cancer research. Frontiers in Physics. 10, 855775(2022).
- Creed, S., McKenzie, M. Measurement of mitochondrial membrane potential with the fluorescent dye tetramethylrhodamine methyl ester (TMRM). Methods in Molecular Biology. 1928, 69-76 (2019).
- Kovalevich, J., Langford, D. Considerations for the use of SH-SY5Y neuroblastoma cells in neurobiology. Methods in Molecular Biology. 1078, 9-21 (2013).
- Sahin, M., Oncu, G., Yilmaz, M. A., Ozkan, D., Saybasili, H. Transformation of SH-SY5Y cell line into neuron-like cells: Investigation of electrophysiological and biomechanical changes. Neuroscience Letters. 745, 135628(2021).
- Trigo, D., et al. Mitochondria dysfunction and impaired response to oxidative stress promotes proteostasis disruption in aged human cells. Mitochondrion. 69, 1-9 (2022).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유