È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi automatizzata ottimizzata della modulazione dell'omeostasi dei mitocondri neuronali vivi da parte dei recettori dell'acido retinoico isoform-specifici
In questo articolo
Riepilogo
La rete mitocondriale è estremamente complessa, il che la rende molto difficile da analizzare. Un nuovo strumento MATLAB analizza i mitocondri confocali in tempo reale nelle immagini timelapse, ma produce un grande volume di output che richiede un'attenzione manuale individuale. Per risolvere questo problema, è stata sviluppata un'ottimizzazione di routine, che consente una rapida analisi dei file.
Abstract
La complessa rete mitocondriale rende molto difficile segmentare, seguire e analizzare le cellule vive. Gli strumenti MATLAB consentono l'analisi dei mitocondri in file timelapse, semplificando e velocizzando notevolmente il processo di elaborazione delle immagini. Ciononostante, gli strumenti esistenti producono un grande volume di output, che richiede un'attenzione manuale individuale, e le configurazioni sperimentali di base hanno un output di migliaia di file, ognuno dei quali richiede una gestione estesa e dispendiosa in termini di tempo.
Per risolvere questi problemi, è stata sviluppata un'ottimizzazione di routine, sia in codice MATLAB che in forma live-script, che consente una rapida analisi dei file e riduce significativamente la lettura e l'elaborazione dei dati. Con una velocità di 100 file/min, l'ottimizzazione consente un'analisi complessivamente rapida. L'ottimizzazione raggiunge l'output dei risultati calcolando la media dei dati specifici del frame per i singoli mitocondri in intervalli di tempo, analizzando i dati in modo definito, coerente con quelli degli strumenti esistenti. L'imaging confocale dal vivo è stato eseguito utilizzando il colorante tetrametilrodamina estere metilico e l'ottimizzazione di routine è stata convalidata trattando le cellule neuronali con agonisti del recettore dell'acido retinoico (RAR), i cui effetti sui mitocondri neuronali sono stabiliti in letteratura. I risultati sono stati coerenti con la letteratura e hanno permesso un'ulteriore caratterizzazione del comportamento della rete mitocondriale in risposta alla modulazione RAR isoforma-specifica.
Questa nuova metodologia ha permesso una caratterizzazione rapida e validata della rete mitocondriale dell'intero neurone, ma consente anche la differenziazione tra mitocondri assone e corpo cellulare, una caratteristica essenziale da applicare nel campo delle neuroscienze. Inoltre, questo protocollo può essere applicato a esperimenti che utilizzano trattamenti ad azione rapida, consentendo l'imaging delle stesse cellule prima e dopo i trattamenti, trascendendo il campo delle neuroscienze.
Introduzione
I mitocondri cellulari sono al centro di tutti gli stati fisiologici e una comprensione approfondita della loro omeostasi (mitostasi) e del loro comportamento è fondamentale per aiutare a identificare il trattamento farmacologico per una vasta gamma di malattie, tra cui il cancro e il morbo di Alzheimer 1,2.
I mitocondri svolgono ruoli cellulari cruciali nell'omeostasi energetica, nella generazione di ATP, nel tamponamento del calcio e nella regolazione dei ROS, e la mitostasi è essenziale per mantenere l'omeostasi proteica poiché gli chaperoni molecolari sono dipendenti dall'energia3. Questi richiedono una modulazione e un adattamento di rete costanti e dinamici per soddisfare in modo efficiente le esigenze cellulari e il trasporto dei mitocondri è regolato da diverse vie di segnalazione; Lavori precedenti hanno descritto uno di questi percorsi, quello dei recettori dell'acido retinoico (RARs)4,5. L'acido retinoico (RA) promuove la crescita assonale e neuritica attraverso l'attivazione di RAR. Nei neuroni corticali primari del topo, l'attivazione di RAR-β incoraggia la crescita mitocondriale, la velocità e la mobilità nel neurite6.
Considerando l'adattabilità e la dinamica della rete mitocondriale, la possibilità di valutare la mitostasi in "tempo reale" è essenziale non solo per studiare l'omeostasi energetica, ma anche per la proteostasi, la salute cellulare, la proliferazione o la segnalazione. Un metodo comunemente usato per valutare la mitostasi si basa sulla microscopia confocale dopo aver evidenziato i mitocondri utilizzando un colorante o un marcatore fluorescente, nonché una configurazione di microscopia specifica che consente la regolazione della temperatura e/o della CO2 7. Questo tipo di configurazione sperimentale prevede l'esecuzione di una replica sperimentale alla volta. Oltre alla ripetizione sperimentale di diversi trattamenti, va considerato che la maggior parte degli esperimenti dovrebbe avere le proprie repliche tecniche (in cui viene ripresa più di una posizione per lastra), con una serie di piani focali (z-stack) registrati in una serie di punti temporali. Pertanto, un disegno sperimentale con tre ripetizioni di un controllo e due trattamenti, con cinque posizioni di imaging per piastra e 15 punti temporali, si traduce in 225 pile da elaborare. Classicamente, i video dei mitocondri vivi sono stati analizzati tracciando i kymografi, che sarebbero stati analizzati individualmente8, in un processo che richiedeva molto tempo e che richiedeva un ampio input manuale, anche quando ci si affidava a strumenti informatici.
Recentemente è stato descritto un algoritmo9 che consente la segmentazione e il tracciamento automatizzati dei mitocondri in file time-lapse 2D e 3D di cellule vive. Sono disponibili altre tecniche di quantificazione, e tutte hanno i loro limiti10. Mitometer, un'applicazione open source automatizzata, è particolarmente adatta per l'analisi del time lapse e della dinamica dei mitocondri, richiedendo un basso input da parte dell'utente. Questa applicazione presenta una serie di vantaggi rispetto ad altri strumenti esistenti basati su MATLAB, ovvero la possibilità di elaborare automaticamente i singoli stack TIF, utilizzando fino a 13 parametri diversi, particolarmente interessante per le neuroscienze, in quanto differenzia tra mitocondri peri- e tele-nucleari.
Tuttavia, per un esperimento come quello sopra descritto, questi 13 parametri applicati a 225 stack danno come risultato 2.925 singoli file di output. Questi richiedono quattro input individuali del computer, che sommano oltre 10.000 input manuali necessari per scaricare tutti i file di output. Per i progetti sperimentali di grandi dimensioni, ciò si traduce in un'analisi inutilmente estremamente dispendiosa in termini di tempo di ogni file e integrazione dei dati. Qui presentiamo un'ottimizzazione di routine che consente una rapida analisi dei file, riducendo notevolmente la lettura dei documenti e l'elaborazione dei dati, analizzando i dati in modo definito, coerente con l'output degli strumenti esistenti.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Questo protocollo prevede due fasi principali: una fase di laboratorio umido, che coinvolge la coltura cellulare e la microscopia confocale dal vivo per ottenere immagini di mitocondri vivi (Figura 1) e una fase in silico per analizzare le immagini ottenute (Figura 2). Per l'analisi automatizzata dei dati dei mitocondri 3D con immagini dal vivo, è stata utilizzata l'applicazione MATLAB Mitometer fornita da Lefebvre et al.9. L'ottimizzazione di routine è scritta in MATLAB. Il software, le versioni aggiornate e le macro ImageJ per l'elaborazione sono disponibili gratuitamente online tramite GitHub, all'indirizzo https://github.com/JoseJoaoMV/Routine_Optimization_Mitometer_APP_MATLAB.
1. Microscopia dal vivo
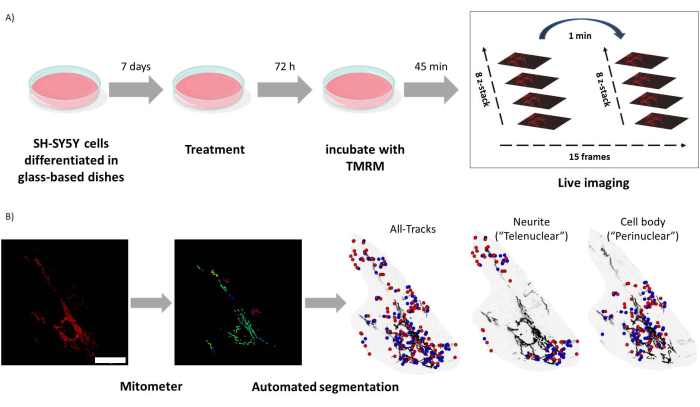
Figura 1: Protocollo sperimentale. Le cellule SH-SY5Y sono state differenziate e trattate con retinoidi. (A) Il TMRM è stato utilizzato per visualizzare in tempo reale i mitocondri sani nelle cellule trattate utilizzando un microscopio confocale, catturando una pila z time lapse di cinque campi visivi. (B) L'applicazione del mitometro MATLAB segmenta e analizza automaticamente le immagini dei mitocondri. Oltre ad analizzare, questo software discrimina automaticamente i mitocondri in base alla vicinanza nucleare. I punti blu sono le posizioni iniziali mitocondriali; I punti rossi sono le posizioni finali. Barra della scala = 30 μm. Abbreviazione: TMRM = tetrametilrodammina, estere metilico. Fare clic qui per visualizzare una versione più grande di questa figura.
- Colture cellulari
- Incubare le cellule SH-SY5Y a 37 °C in un'atmosfera umidificata con il 5% di CO2 e il 95% di aria, coltivate in parti uguali di MEM (Minimal Essential Medium) e F12 (Ham's F12 Nutrient Mix) Medium, integrate con il 10% di siero fetale bovino (FBS).
- Piastre di cellule SH-SY5Y in piastre con fondo di vetro a una densità di 15 × 104 cellule/mL.
- Differenziare le cellule con 5 giorni di trattamento con 10 μM di acido all-trans retinoico in terreno di coltura contenente FBS all'1%, seguito da 2 giorni di trattamento con fattore neurotrofico derivato dal cervello (BDNF) 10 ƞg/mL.
- Trattamento cellulare
- Lavare le cellule con soluzione fisiologica sterile tamponata con fosfato (PBS) e trattare per 72 ore con agonisti dell'isoforma RAR 10-7 M, in parti uguali di terreno MEM (Minimal Essential Medium) e F12 (Ham's F12 Nutrient Mix), integrati con FBS all'1%.
NOTA: L'agonista RARα utilizzato è stato AM580; L'agonista RARβ utilizzato è stato CD2314; Ch55 è stato usato come agonista come RARα e β co-agonista; aL'artrite reumatoide è stata utilizzata come controllo positivo; BMS493 è stato utilizzato come pan-antagonista RAR.
- Lavare le cellule con soluzione fisiologica sterile tamponata con fosfato (PBS) e trattare per 72 ore con agonisti dell'isoforma RAR 10-7 M, in parti uguali di terreno MEM (Minimal Essential Medium) e F12 (Ham's F12 Nutrient Mix), integrati con FBS all'1%.
- Imaging confocale dal vivo
- Sostituire il terreno di coltura con terreno di coltura fresco contenente FBS all'1% con tetrametilrodamina 20 nM, estere metilico (TMRM) per 45 minuti.
NOTA: Il TMRM è un colorante fluorescente permeabile per le cellule, sequestrato dai mitocondri attivi, e questo periodo di incubazione consente al TMRM di raggiungere un equilibrio ed essere assorbito dai mitocondri polarizzati11. L'imaging deve essere avviato prima che venga stabilito l'equilibrio, poiché l'intensità del segnale TMRM potrebbe aumentare artificialmente durante l'imaging. - Collocare le cellule in un'incubatrice collegata a un microscopio confocale a scansione laser a 37 °C.
- Cattura immagini utilizzando un obiettivo apocromatico a immersione in olio 63x, con una dimensione dell'immagine di 512 x 512 pixel ottenuta con un'apertura stenopeica di 1 unità airy, catturando una serie temporale di 15 fotogrammi da cinque diversi campi visivi in ciascuna piastra cellulare e una pila z di 8 piani z equidistanti. Il file .lsm risultante è uno stack di tempo, posizione e z.
NOTA: Le impostazioni per il guadagno, il contrasto e la luminosità devono essere inizialmente ottimizzate utilizzando la potenza laser minima necessaria per utilizzare l'intera gamma dinamica del rilevatore e mantenute costanti durante lo studio, assicurando che tutte le immagini vengano eseguite nelle stesse condizioni. In questa combinazione hardware-software è possibile registrare fino a nove diverse posizioni e il microscopio passa automaticamente da una posizione all'altra.
- Sostituire il terreno di coltura con terreno di coltura fresco contenente FBS all'1% con tetrametilrodamina 20 nM, estere metilico (TMRM) per 45 minuti.
2. Analisi delle immagini
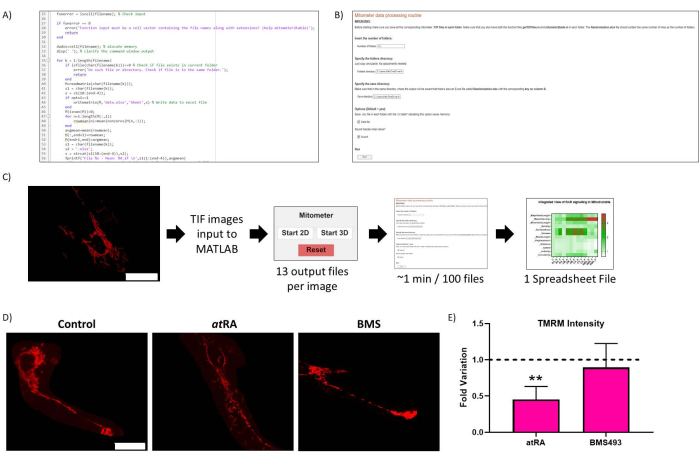
Figura 2: Ottimizzazione di routine. (A) Codice rappresentativo dell'ottimizzazione di routine. (B) Ottimizzazione di routine Live-Script. (C) Flusso di lavoro di ottimizzazione di routine. (D) Validazione dei risultati dell'ottimizzazione di routine: immagine rappresentativa dei mitocondri in cellule non trattate (pannello di sinistra), trattate con AR(10-7 M, 72 h, pannello centrale) e trattate con l'antagonista RAR BMS493 (10-7 M, 72 h, pannello di destra), visualizzate dopo incubazione con TMRM (20 nM, incubazione di 45 min). Barra della scala = 30 μm. (E) TMRM Intensità nei mitocondri del corpo cellulare. Diminuzione significativa con il trattamento con acido all-trans retinoico (all'AR, 10-7 M, 72 h), rispetto al controllo (p=0,0062), non osservata quando trattata con l'antagonista RAR (BMS493, 10-7 M, 72 ore). Cinque cellule sono state quantificate da ciascuna delle tre ripetizioni per condizione. Fare clic qui per visualizzare una versione più grande di questa figura.
- Elaborazione di immagini
- Aprire i file in ImageJ 2.1.0 e separare le pile di posizioni in base al campo visivo: aprire ImageJ e fare clic su Barra dei menu | Immagine | Duplica | Inserire sezioni/numero di fotogramma | Va bene.
NOTA: Per ridurre gli input ripetitivi al software, è stata sviluppata una macro ImageJ per facilitare la duplicazione e il salvataggio delle immagini del campo visivo.
- Aprire i file in ImageJ 2.1.0 e separare le pile di posizioni in base al campo visivo: aprire ImageJ e fare clic su Barra dei menu | Immagine | Duplica | Inserire sezioni/numero di fotogramma | Va bene.
- Protocollo macro
- Disegna una regione di interesse (ROI) intorno alla cella con lo strumento di selezione libera.
- Eseguire la macro aprendo ImageJ e passando a Barra dei menu | Plugin | Macro | Esegui Cancella lo sfondo e salva la macro delle immagini.
NOTA: le immagini possono essere randomizzate in questo processo, memorizzando la chiave della soluzione in base ai requisiti di ottimizzazione. - Determinare la dimensione dei pixel e la profondità del voxel: aprire ImageJ | l'immagine e passare alla barra dei menu | Immagine | Proprietà.
- Protocollo diretto alternativo
NOTA: questa alternativa non utilizza Macro per elaborare le immagini- Trovare la dimensione dei pixel e la profondità del voxel: Apri ImageJ | Apri Immagine e vai a Barra dei menu | Immagine | Proprietà.
- Disegna un ROI intorno alla cella con lo strumento di selezione libera, assicurandoti che comprenda l'intera cella attraverso i 15 fotogrammi.
- Passare alla barra dei menu | Modifica | Sgombro all'esterno.
- Passare alla barra dei menu | Archivio | Salva con nome | Selezionare Tiff.
- Seleziona Cartella di salvataggio e fai clic su Salva.
NOTA: A questo punto, le immagini possono essere cieche/randomizzate manualmente prima di continuare con l'analisi.
- Analisi automatizzata delle immagini
- Preparare le cartelle per i file di analisi.
- Crea tre cartelle principali, intitolate "Tutte le tracce", "Tracce perinucleari" e "Tracce telenucleari".
NOTA: Corrispondono alle principali opzioni di tracciamento automatizzato. - In ogni cartella principale, creare una sottocartella per ogni immagine da elaborare, identificata numericamente da 1 in su.
- Aggiungere una copia dei due file supplementari di ottimizzazione di routine (mitometer2table.m e getTXTfiles.m) a ciascuna cartella di immagini.
NOTA: Questi file aiutano nell'analisi dei dati e nella disposizione finale del formato. Il numero di cartelle deve corrispondere al numero di elementi nel foglio di calcolo randomizzato (.xlsx). Dopo aver creato tutte le sottocartelle numerate con file supplementari per un set di dati, è possibile copiarle e incollarle in batch nei set di dati rimanenti.
- Crea tre cartelle principali, intitolate "Tutte le tracce", "Tracce perinucleari" e "Tracce telenucleari".
- Preparare le cartelle per i file di analisi.
- Utilizza l'applicazione MATLAB Mitometer per analizzare le immagini.
NOTA: Questo protocollo è stato eseguito su un mitometro, installato su MATLAB R2022a. Carica le immagini in batch di 30 per ottimizzare il tempo di esecuzione e il bilanciamento dell'output. La dimensione massima del file MAT è imposta dal file system nativo: per impostazione predefinita, le operazioni di "salvataggio" possono creare un file < 231 byte (~2 GB); Al suo posto è possibile utilizzare MAT-Files versione 7.3, che consente dimensioni variabili massime superiori a 2 GB.- Identificare/modificare la versione predefinita del file MAT: nella scheda Home della sezione Ambiente , fare clic su Preferenze e selezionare MATLAB | Generale | File MAT.
- Usa lo strumento MATLAB Mitometer per analizzare: Apri MATLAB e vai alla barra dei menu | APPLICAZIONI | Mitometro aperto | Selezionare Avvia 3D | Dati di input (Dimensione pixel (μm): 0,1395089/Tempo tra i fotogrammi (s): 2/Numero piani z: 8/Distanza assiale tra i piani z (μm): 0,418809 | Selezionare le immagini da inserire.
- Vai al menu laterale del mitometro, seleziona Immagine, fai clic su Seleziona evidenziato, vai alla barra dei menu del mitometro | Selezione delle tracce | Visualizzazione delle tracce | selezionare All-Tracks, Telenucleare o Perinucleare | Barra dei menu del mitometro | Selezionare Analisi | Scegliere un elemento (ad esempio, Lunghezza) | selezionare Salva in ".txt".
NOTA: È possibile scaricare più di un parametro contemporaneamente, se selezionato contemporaneamente. - Estrarre i file dei risultati nelle cartelle create (2.2.1).
- Ottimizzazione di routine e analisi dei dati
- Preparare/adattare il file "Randomization.xlsx" contenente la chiave per la codifica delle immagini.
- Inserire un elenco di numeri interi consecutivi, da 1 in su, nella colonna A.
NOTA: Si consiglia di conservare una cartella duplicata con le immagini originariamente denominate. - Inserire le variabili analitiche nella colonna B, composta da caratteri alfanumerici.
NOTA: il numero di righe nel documento deve essere coerente con il numero di cartelle presenti nel set di dati principale. Copiare e incollare questo file "Randomization.xlsx" negli altri due set di dati principali.
- Inserire un elenco di numeri interi consecutivi, da 1 in su, nella colonna A.
- Analisi dei dati ottimizzata
- Fare doppio clic su "executable.mlx", inserire il numero di cartelle, specificare la directory delle cartelle (copiare la directory dall'inizio della cartella) | la directory di salvataggio (copiare la directory dalla parte superiore della cartella) | il nome del foglio di calcolo nel file di output e fare clic su Esegui.
- Eseguire l'analisi statistica in base alle esigenze.
NOTA: È possibile selezionare la creazione facoltativa di un file di .xlsx in ciascuna cartella con i dati .txt e l'avviso sonoro alla fine dell'analisi. Live Script genera un singolo foglio di calcolo in formato tabella. In questo output, le colonne rappresentano i parametri analizzati (ad esempio, "Lunghezza asse maggiore"; "Intensità") e le linee rappresentano i campi visivi delle variabili analitiche (ad esempio, "Controllo").
- Preparare/adattare il file "Randomization.xlsx" contenente la chiave per la codifica delle immagini.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per migliorare e accelerare l'analisi dei file di output in .txt formato, è stata codificata un'ottimizzazione di routine che legge i dati coerenti con i file di output di Mitometer .txt, con colonne che rappresentano un frame e linee che rappresentano i mitocondri identificati. L'ottimizzazione di routine produce dati in un unico valore per parametro calcolando la media dei frame per ogni mitocondri identificato e quindi calcolando la media dei risultati di tutti i mitocondri per campo visivo. La routine sviluppata leg...
Access restricted. Please log in or start a trial to view this content.
Discussione
L'imaging di cellule vive produce file di grandi dimensioni che richiedono una seria elaborazione informatica, ma anche gli strumenti più recenti richiedono un ampio input manuale per l'elaborazione. Questa ottimizzazione di routine si concentra sulla semplificazione del processo di analisi dei mitocondri sul mitometro perché questo strumento presenta un ottimo equilibrio tra l'input dell'utente e l'output dei dati. In precedenza è stato esaminato un confronto completo tra diversi strumenti per l'analisi delle immagin...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
L'acquisizione delle immagini è stata eseguita nella struttura LiM di iBiMED, un nodo di PPBI (Piattaforma Portoghese di BioImaging): POCI-01-0145-FEDER-022122. Questo lavoro è stato sostenuto da FCT (EXPL/BTM-SAL/0902/2021), LCF (CI21-00276), una sovvenzione a DT dalla Fundação para a Ciência e Tecnologia del Ministério da Educação e Ciência (2020.02006.CEECIND), una sovvenzione da ATG-The Gabba Alumni Association a VP e dall'Istituto di Biomedicina-iBiMED, Università di Aveiro.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| AM580 | Sigma-Aldrich | A8843 | |
| BDNF | Thermo-Fisher | RP8642 | |
| BMS493 | Tocris Bioscience | 3409 | |
| CD2314 | Tocris Bioscience | 3824 | |
| Ch55 | Tocris Bioscience | 2020 | |
| Foetal Bovine Serum | Thermo-Fisher | 10270106 | |
| GraphPad Prism v4.0 | GraphPad Software, La Jolla | n/a | |
| Ham’s F12 Nutrient Mix | Thermo-Fisher | 21765029 | |
| MATLAB R2022a | MathWorks | n/a | |
| Minimal Essential Medium | Thermo-Fisher | 31095 | |
| Nunc Glass Bottom Dishes | Thermo-Fisher | 150680 | |
| Phosphate Buffer Saline Solution | Thermo-Fisher | 28372 | |
| Retinoic acid | Sigma-Aldrich | R2625 | |
| TMRM | Thermo-Fisher | T668 | |
| Zeiss LSM 510 | Carl Zeiss | n/a | Equiped with live-cell imaging culture chamber and 63x oil immersion objective |
Riferimenti
- Trigo, D., Avelar, C., Fernandes, M., Sa, J., da Cruz, E. S. O. Mitochondria, energy, and metabolism in neuronal health and disease. FEBS Letters. 596 (9), 1095-1110 (2022).
- Zong, W. X., Rabinowitz, J. D., White, E. Mitochondria and cancer. Molecular Cell. 61 (5), 667-676 (2016).
- Clare, D. K., Saibil, H. R. ATP-driven molecular chaperone machines. Biopolymers. 99 (11), 846-859 (2013).
- Tourniaire, F., et al. All-trans retinoic acid induces oxidative phosphorylation and mitochondria biogenesis in adipocytes. Journal of Lipid Research. 56 (6), 1100-1109 (2015).
- Psarra, A. M., Sekeris, C. E. Nuclear receptors and other nuclear transcription factors in mitochondria: regulatory molecules in a new environment. Biochimica et Biophysica Acta. 1783 (1), 1-11 (2008).
- Trigo, D., Goncalves, M. B., Corcoran, J. P. T. The regulation of mitochondrial dynamics in neurite outgrowth by retinoic acid receptor beta signaling. FASEB Journal. 33 (6), 7225-7235 (2019).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Current Protocols in Cell Biology. Chapter 4 (Unit 4), 1-21 (2010).
- Sajic, M., et al. Impulse conduction increases mitochondrial transport in adult mammalian peripheral nerves in vivo. PLoS Biology. 11 (12), e1001754(2013).
- Lefebvre, A., Ma, D., Kessenbrock, K., Lawson, D. A., Digman, M. A. Automated segmentation and tracking of mitochondria in live-cell time-lapse images. Nature Methods. 18 (9), 1091-1102 (2021).
- Chu, C. -H., Tseng, W. -W., Hsu, C. -M., Wei, A. -C. Image analysis of the mitochondrial network morphology with applications in cancer research. Frontiers in Physics. 10, 855775(2022).
- Creed, S., McKenzie, M. Measurement of mitochondrial membrane potential with the fluorescent dye tetramethylrhodamine methyl ester (TMRM). Methods in Molecular Biology. 1928, 69-76 (2019).
- Kovalevich, J., Langford, D. Considerations for the use of SH-SY5Y neuroblastoma cells in neurobiology. Methods in Molecular Biology. 1078, 9-21 (2013).
- Sahin, M., Oncu, G., Yilmaz, M. A., Ozkan, D., Saybasili, H. Transformation of SH-SY5Y cell line into neuron-like cells: Investigation of electrophysiological and biomechanical changes. Neuroscience Letters. 745, 135628(2021).
- Trigo, D., et al. Mitochondria dysfunction and impaired response to oxidative stress promotes proteostasis disruption in aged human cells. Mitochondrion. 69, 1-9 (2022).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon