É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise Automatizada Otimizada da Modulação da Homeostase das Mitocôndrias Neuronais Vivas por Receptores de Ácido Retinóico Isoform-Específicos
Neste Artigo
Resumo
A rede mitocondrial é extremamente complexa, tornando-a muito difícil de analisar. Uma nova ferramenta MATLAB analisa mitocôndrias confocais ao vivo em imagens de timelapse, mas resulta em um grande volume de saída que requer atenção manual individual. Para resolver esse problema, uma otimização de rotina foi desenvolvida, permitindo uma análise rápida dos arquivos.
Resumo
A complexa rede mitocondrial torna muito desafiador segmentar, acompanhar e analisar células vivas. As ferramentas MATLAB permitem a análise de mitocôndrias em arquivos timelapse, simplificando e acelerando consideravelmente o processo de processamento de imagens. No entanto, as ferramentas existentes produzem um grande volume de saída, exigindo atenção manual individual, e as configurações experimentais básicas têm uma saída de milhares de arquivos, cada um exigindo manuseio extenso e demorado.
Para resolver essas questões, uma otimização de rotina foi desenvolvida, tanto em código MATLAB quanto em formulários live-script, permitindo uma análise rápida de arquivos e reduzindo significativamente a leitura de documentos e o processamento de dados. Com uma velocidade de 100 arquivos/min, a otimização permite uma análise geral rápida. A otimização alcança a saída de resultados pela média de dados específicos de quadros para mitocôndrias individuais ao longo de períodos de tempo, analisando dados de maneira definida, consistente com a saída de ferramentas existentes. Imagens confocais ao vivo foram realizadas usando o corante tetrametilrodamina éster, e a otimização de rotina foi validada pelo tratamento de células neuronais com agonistas do receptor do ácido retinóico (RAR), cujos efeitos sobre as mitocôndrias neuronais estão estabelecidos na literatura. Os resultados foram consistentes com a literatura e permitiram uma caracterização mais detalhada do comportamento das redes mitocondriais em resposta à modulação RAR isoform-específica.
Esta nova metodologia permitiu a caracterização rápida e validada da rede mitocondrial de neurônios inteiros, mas também permite a diferenciação entre axônio e mitocôndrias de corpo celular, uma característica essencial para aplicação no campo da neurociência. Além disso, esse protocolo pode ser aplicado a experimentos utilizando tratamentos de ação rápida, permitindo a obtenção de imagens das mesmas células antes e após os tratamentos, transcendendo o campo da neurociência.
Introdução
As mitocôndrias celulares estão no centro de todos os estados fisiológicos, e uma compreensão completa de sua homeostase (mitostase) e comportamento é fundamental para auxiliar na identificação do tratamento farmacológico para uma ampla gama de doenças, incluindo câncer e doença de Alzheimer 1,2.
As mitocôndrias desempenham papéis celulares cruciais na homeostase energética, geração de ATP, tamponamento de cálcio e regulação de EROs, e a mitostase é essencial para manter a homeostase proteica, uma vez que as chaperonas moleculares são dependentes de energia3. Estes requerem uma modulação e adaptação de rede constante e dinâmica para atender eficientemente às necessidades celulares, e o transporte mitocondrial é regulado por diferentes vias de sinalização; trabalhos anteriores descreveram uma dessas vias, a dos receptores do ácido retinóico (RARs)4,5. O ácido retinóico (AR) promove crescimento axonal e neurítico via ativação RAR. Em neurônios corticais primários de camundongos, a ativação do RAR-β estimula o crescimento, a velocidade e a mobilidade mitocondrial no neurito6.
Considerando a adaptabilidade e dinâmica da rede mitocondrial, a possibilidade de avaliar a mitostase em "tempo real" é essencial não apenas para investigar a homeostase energética, mas também para proteostase, saúde celular, proliferação ou sinalização. Um método comumente utilizado para avaliação de mitostases baseia-se na microscopia confocal após o realce mitocondrial usando um corante ou marcador fluorescente, bem como uma configuração de microscopia específica que permite a regulação da temperatura e/ou do CO2 7. Esse tipo de arranjo experimental implica que uma réplica experimental seja realizada de cada vez. Além da repetição experimental de diferentes tratamentos, deve-se considerar que a maioria dos experimentos deve ter suas réplicas técnicas (onde mais de uma posição é imageada por placa), com uma série de planos focais (z-stacks) sendo registrados em uma série de pontos de tempo. Assim, um delineamento experimental com três repetições de um controle e dois tratamentos, com cinco posições de imagem por placa, e 15 pontos de tempo, resultou em 225 pilhas a serem processadas. Classicamente, os vídeos de mitocôndrias vivas eram analisados por meio de quimografias plotadas, que seriam analisadasindividualmente8, em um processo demorado que exigia extensa entrada manual, mesmo quando dependia de ferramentas computacionais.
Recentemente foi descritoum algoritmo 9 que permite segmentação e rastreamento automatizado de mitocôndrias em arquivos time-lapse 2-D e 3-D de células vivas. Outras técnicas de quantificação estão disponíveis e todas apresentam suas limitações10. O mitômetro, um aplicativo automatizado de código aberto, é particularmente adequado para análise de lapso de tempo e dinâmica mitocondrial, exigindo baixa entrada do usuário. Esta aplicação tem uma série de vantagens sobre outras ferramentas existentes baseadas no MATLAB, nomeadamente permitindo o processamento automático de pilhas TIF individuais, utilizando até 13 parâmetros diferentes, particularmente interessantes para as neurociências, uma vez que diferencia entre mitocôndrias peri e telenucleares.
No entanto, para um experimento como o descrito acima, esses 13 parâmetros aplicados a 225 pilhas resultam em 2.925 arquivos de saída individuais. Estes requerem quatro entradas de computador individuais, que somam mais de 10.000 entradas manuais sendo necessárias para baixar todos os arquivos de saída. Para grandes projetos experimentais, isso resulta em uma análise desnecessariamente extremamente demorada de cada arquivo e integração de dados. Aqui apresentamos uma rotina de otimização que permite a análise rápida de arquivos, reduzindo consideravelmente a leitura de documentos e o processamento de dados, analisando os dados de forma definida, consistente com a saída das ferramentas existentes.
Access restricted. Please log in or start a trial to view this content.
Protocolo
OBS: Este protocolo tem duas etapas principais: uma etapa de laboratório úmido, envolvendo cultura de células e microscopia confocal viva para obtenção de imagens de mitocôndrias vivas (Figura 1) e uma etapa in silico para análise das imagens obtidas (Figura 2). Para a análise automatizada dos dados de mitocôndrias com imagens ao vivo em 3D, foi utilizado o mitômetro de aplicação MATLAB, conforme fornecido por Lefebvre et al.9. A otimização de rotina é escrita no MATLAB. O software, as versões atualizadas e o processamento de Macros ImageJ estão disponíveis gratuitamente on-line através do GitHub, no https://github.com/JoseJoaoMV/Routine_Optimization_Mitometer_APP_MATLAB.
1. Microscopia ao vivo
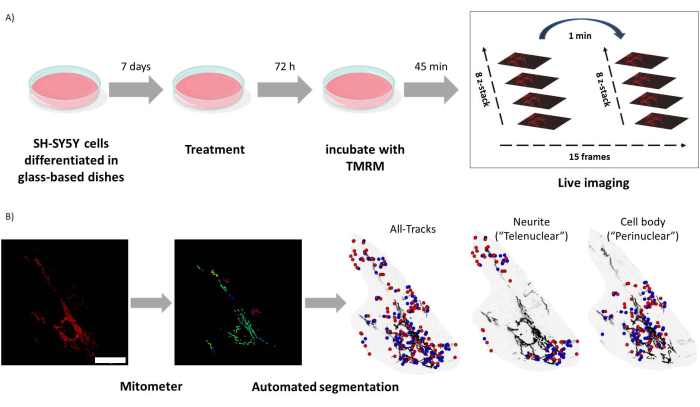
Figura 1: Protocolo experimental. As células SH-SY5Y foram diferenciadas e tratadas com retinoides. (A) TMRM foi usado para imagens vivas de mitocôndrias saudáveis em células tratadas usando um microscópio confocal, capturando um z-stack de lapso de tempo de cinco campos visuais. (B) Aplicação de mitômetro O MATLAB segmenta e analisa automaticamente as imagens das mitocôndrias. Além de analisar, este software discrimina automaticamente as mitocôndrias de acordo com a proximidade nuclear. Os pontos azuis são posições iniciais mitocondriais; pontos vermelhos são posições finais. Barra de escala = 30 μm. Abreviação: TMRM = tetrametilrodamina, éster metílico. Clique aqui para ver uma versão maior desta figura.
- Cultura celular
- Incubar células SH-SY5Y a 37 °C em atmosfera umidificada de 5% de CO2 e 95% de ar, cultivadas em partes iguais de meio MEM (Minimal Essential Medium) e F12 (Ham's F12 Nutrient Mix), suplementado com 10% de soro fetal bovino (FBS).
- Células SH-SY5Y em placas de células com fundo de vidro a uma densidade de 15 × 104 células/mL.
- Diferenciar células com 5 dias de tratamento com 10 μM de ácido all-trans retinóico em meio de cultura contendo FBS a 1%, seguido de 2 dias de tratamento com 10 ƞg/mL de fator neurotrófico derivado do cérebro (BDNF).
- Tratamento celular
- Lavar as células com solução salina tamponada com fosfato estéril (PBS) e tratar por 72 h com agonistas isoformes de RAR 10-7 M, em partes iguais de meio MEM (Minimal Essential Medium) e F12 (Ham's F12 Nutrient Mix), suplementado com FBS a 1%.
LEGENDA: O agonista RARα utilizado foi o AM580; O agonista RARβ utilizado foi o CD2314; Ch55 foi usado como agonista como RARα e β co-agonista; emA AR foi utilizada como controle positivo; BMS493 foi usado como pan-antagonista RAR.
- Lavar as células com solução salina tamponada com fosfato estéril (PBS) e tratar por 72 h com agonistas isoformes de RAR 10-7 M, em partes iguais de meio MEM (Minimal Essential Medium) e F12 (Ham's F12 Nutrient Mix), suplementado com FBS a 1%.
- Imagem confocal ao vivo
- Substitua o meio de cultura por meio de cultura fresco contendo FBS a 1% por tetrametilrodamina 20 nM, éster metílico (TMRM) por 45 min.
NOTA: O TMRM é um corante fluorescente pertençoso por células, sequestrado por mitocôndrias ativas, e esse período de incubação permite que o TMRM atinja um equilíbrio e seja absorvido por mitocôndrias polarizadas11. A aquisição de imagens deve ser iniciada antes que o equilíbrio seja estabelecido, pois a intensidade do sinal TMRM pode aumentar artificialmente durante a aquisição de imagens. - Colocar as células numa incubadora ligada a um microscópio confocal de varredura a laser a 37 °C.
- Capture imagens usando uma objetiva apocromática de imersão em óleo de 63x, com um tamanho de imagem de 512 x 512 pixels obtida com uma abertura pinhole de 1 unidade arejada, capturando uma série temporal de 15 quadros de cinco campos visuais diferentes em cada placa celular, e uma pilha z de 8 planos z equidistantes. O arquivo .lsm resultante é uma pilha de tempo, posição e z.
NOTA: As configurações de ganho, contraste e brilho devem ser inicialmente otimizadas usando a potência mínima do laser necessária para usar toda a faixa dinâmica do detector e mantidas constantes durante todo o estudo, garantindo que todas as imagens sejam realizadas nas mesmas condições. Até nove posições diferentes podem ser registradas nesta combinação hardware-software e os ciclos do microscópio entre as posições de imagem automaticamente.
- Substitua o meio de cultura por meio de cultura fresco contendo FBS a 1% por tetrametilrodamina 20 nM, éster metílico (TMRM) por 45 min.
2. Análise das imagens
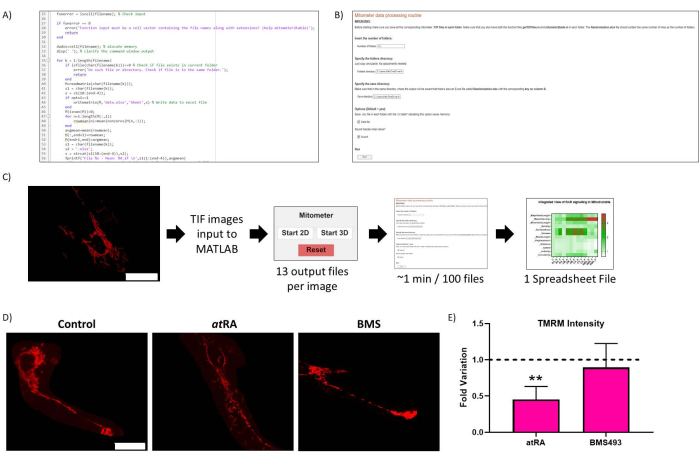
Figura 2: Otimização de rotina. (A) Código representativo da otimização de rotina. (B) Otimização de rotina Live-Script. (C) Fluxo de trabalho de otimização de rotina. (D) Validação dos resultados da otimização de rotina: imagem representativa das mitocôndrias em células não tratadas (painel esquerdo), tratadas com RA (10-7 M, 72 h, painel médio) e tratadas com antagonistaRAR BMS493 (10-7 M, 72 h, painel direito), obtida após incubação com TMRM (20 nM, 45 min de incubação). Barra de escala = 30 μm. (E) TMRM Intensidade em mitocôndrias do corpo celular. Redução significativa com o tratamento com ácido trans-retinóico (AR, 10-7 M, 72 h), em comparação com o controle (p=0,0062), não observada quando tratada com antagonista RAR (BMS493, 10-7 M, 72 horas). Cinco células foram quantificadas de cada uma das três repetições por condição. Clique aqui para ver uma versão maior desta figura.
- Processamento de imagens
- Abra arquivos no ImageJ 2.1.0 e separe pilhas de posição por campo visual: Abra o ImageJ e clique em Barra de menus | Imagem | Duplicar | Fatias de entrada/número de quadro | Está bem.
NOTA: Para diminuir as entradas repetitivas para o software, um ImageJ Macro foi desenvolvido para facilitar a duplicação e salvamento de imagens de campo visual.
- Abra arquivos no ImageJ 2.1.0 e separe pilhas de posição por campo visual: Abra o ImageJ e clique em Barra de menus | Imagem | Duplicar | Fatias de entrada/número de quadro | Está bem.
- Protocolo de macro
- Desenhe uma região de interesse (ROI) ao redor da célula com a ferramenta de seleção gratuita.
- Execute a macro abrindo o ImageJ e navegando até a Barra de Menus | Plugins | Macros | Execute Limpar o plano de fundo e salvar imagens macro.
NOTA: As imagens podem ser randomizadas neste processo, armazenando a chave da solução de acordo com os requisitos de otimização. - Determine o tamanho do pixel e a profundidade do voxel: Abra o ImageJ | a Imagem e navegue até a Barra de Menus | Imagem | Propriedades.
- Protocolo direto alternativo
Observação : essa alternativa não usa macro para processar imagens- Encontre o tamanho do pixel e a profundidade do voxel: Abra o ImageJ | Abra a Imagem e navegue até a Barra de Menus | Imagem | Propriedades.
- Desenhe um ROI ao redor da célula com a ferramenta de seleção gratuita, garantindo que ela abranja toda a célula ao longo dos 15 quadros.
- Navegue até a barra de menus | Editar | Limpe do lado de fora.
- Navegue até a barra de menus | Arquivo | Salvar como | Selecione Tiff.
- Selecione Salvar pasta e clique em Salvar.
NOTA: As imagens podem ser cegas/randomizadas manualmente neste ponto antes de continuar a análise.
- Análise automatizada de imagens
- Prepare as pastas para arquivos de análise.
- Crie três pastas principais, intituladas "Todas as faixas", "Trilhas perinucleares" e "Trilhas telenucleares".
NOTA: Eles correspondem às principais opções de faixa automatizada. - Em cada pasta principal, crie uma subpasta para cada imagem a ser processada, identificada numericamente de 1 para cima.
- Adicione uma cópia dos dois arquivos suplementares de otimização de rotina (mitometer2table.m e getTXTfiles.m) a cada pasta de imagem.
NOTA: Esses arquivos ajudam na análise de dados e na disposição final do formato. O número de pastas deve corresponder ao número de elementos na planilha aleatória (.xlsx). Depois de criar todas as subpastas numeradas com arquivos suplementares para um conjunto de dados, elas podem ser copiadas em lote e coladas nos conjuntos de dados restantes.
- Crie três pastas principais, intituladas "Todas as faixas", "Trilhas perinucleares" e "Trilhas telenucleares".
- Prepare as pastas para arquivos de análise.
- Use o aplicativo MATLAB Mitometer para analisar imagens.
NOTA: Este protocolo foi executado em um mitômetro, instalado no MATLAB R2022a. Carregue imagens em lotes de 30 para otimizar o tempo de execução e o equilíbrio de saída. O tamanho máximo do arquivo MAT é imposto pelo sistema de arquivos nativo: por padrão, as operações de "salvar" podem criar um arquivo < 231 bytes (~2 GB); salvar formato MAT-Files Versão 7.3 pode ser usado em vez disso, pois permite tamanhos variáveis máximos maiores do que 2 GB.- Identificar/alterar a versão padrão do arquivo MAT: Na guia Página Inicial da seção Ambiente, clique em Preferências e selecione MATLAB | Geral | Arquivos MAT.
- Use a ferramenta MATLAB Mitometer para analisar: Abra o MATLAB e navegue até a Barra de menus | APLICATIVOS | Miômetro Aberto | Selecione Iniciar 3D | Dados de entrada (Tamanho do pixel (μm): 0,1395089/Tempo entre quadros (s): 2/Número z-planos: 8/Distância axial entre z-planos (μm): 0,418809 | Selecione as imagens a serem inseridas.
- Vá para o Menu Lateral do Mitômetro, selecione Imagem, clique em Selecionar Realçado, navegue até a Barra de Menus do mitômetro | Selecionar faixas | Rastrear visualizações | selecione All-Tracks, Telenuclear ou Perinuclear | mitômetro Barra de Menus | Selecionar Análise | Escolha um elemento (ou seja, Comprimento) | selecione Salvar em ".txt".
Observação : mais de um parâmetro pode ser baixado de uma só vez, se selecionado concomitantemente. - Extraia arquivos de resultado para as pastas criadas (2.2.1).
- Otimização de rotina e análise de dados
- Prepare/adapte o arquivo "Randomization.xlsx" que contém a chave para codificação de imagem.
- Insira uma lista de inteiros consecutivos, de 1 para cima, na coluna A.
NOTA: É aconselhável manter uma pasta duplicada com imagens originalmente nomeadas. - Coloque as variáveis analíticas na coluna B, composta por caracteres alfanuméricos.
Observação : o número de linhas no documento deve ser consistente com o número de pastas presentes no conjunto de dados principal. Copie e cole esse arquivo "Randomization.xlsx" nos outros dois conjuntos de dados principais.
- Insira uma lista de inteiros consecutivos, de 1 para cima, na coluna A.
- Análise de dados otimizada
- Clique duas vezes em "executable.mlx", insira o número de pastas, especifique o diretório de pastas (copie o diretório da parte superior da pasta) | o diretório de salvamento (copie o diretório da parte superior da pasta) | o nome da planilha no arquivo de saída e clique em Executar.
- Realizar análise estatística conforme necessário.
Observação : criação opcional de um arquivo de .xlsx em cada pasta com os dados .txt e som alerta para o final da análise pode ser selecionado. O Live Script gera um único arquivo de planilha em um formato de tabela. Nessa saída, as colunas representam os parâmetros analisados (por exemplo, "Comprimento do eixo principal"; "Intensidade") e linhas representam campos visuais de variáveis analíticas (por exemplo, "Controle").
- Prepare/adapte o arquivo "Randomization.xlsx" que contém a chave para codificação de imagem.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para aprimorar e acelerar a análise de arquivos de saída em .txt formato, foi codificada uma rotina de otimização que lê dados consistentes com os arquivos de saída do Mitometer .txt, com colunas representando um quadro e linhas representando mitocôndrias identificadas. A otimização de rotina produz dados em um único valor por parâmetro, calculando a média dos quadros para cada mitocôndria identificada e, em seguida, calculando a média dos resultados de todas as mitocôndrias por campo visual. A rotina dese...
Access restricted. Please log in or start a trial to view this content.
Discussão
A imagem de células vivas produz arquivos grandes que exigem processamento de computação sério, mas mesmo as ferramentas mais recentes exigem extensa entrada manual para processar. Esta otimização de rotina é focada em simplificar o processo de análise de mitocôndrias no Mitômetro, pois esta ferramenta apresenta um equilíbrio muito bom entre a entrada do usuário e a saída de dados. Uma comparação abrangente entre diferentes ferramentas para análise de imagens mitocondriais foi previamente revisada
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
A aquisição das imagens foi realizada no equipamento LiM do iBiMED, um nó do PPBI (Plataforma Portuguesa de BioImagem): POCI-01-0145-FEDER-022122. Este trabalho contou com o apoio da FCT (EXPL/BTM-SAL/0902/2021) LCF (CI21-00276), uma bolsa a DT da Fundação para a Ciência e Tecnologia do Ministério da Educação e Ciência (2020.02006.CEECIND), uma bolsa da ATG-The Gabba Alumni Association to VP, e do Instituto de Biomedicina-iBiMED, Universidade de Aveiro.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| AM580 | Sigma-Aldrich | A8843 | |
| BDNF | Thermo-Fisher | RP8642 | |
| BMS493 | Tocris Bioscience | 3409 | |
| CD2314 | Tocris Bioscience | 3824 | |
| Ch55 | Tocris Bioscience | 2020 | |
| Foetal Bovine Serum | Thermo-Fisher | 10270106 | |
| GraphPad Prism v4.0 | GraphPad Software, La Jolla | n/a | |
| Ham’s F12 Nutrient Mix | Thermo-Fisher | 21765029 | |
| MATLAB R2022a | MathWorks | n/a | |
| Minimal Essential Medium | Thermo-Fisher | 31095 | |
| Nunc Glass Bottom Dishes | Thermo-Fisher | 150680 | |
| Phosphate Buffer Saline Solution | Thermo-Fisher | 28372 | |
| Retinoic acid | Sigma-Aldrich | R2625 | |
| TMRM | Thermo-Fisher | T668 | |
| Zeiss LSM 510 | Carl Zeiss | n/a | Equiped with live-cell imaging culture chamber and 63x oil immersion objective |
Referências
- Trigo, D., Avelar, C., Fernandes, M., Sa, J., da Cruz, E. S. O. Mitochondria, energy, and metabolism in neuronal health and disease. FEBS Letters. 596 (9), 1095-1110 (2022).
- Zong, W. X., Rabinowitz, J. D., White, E. Mitochondria and cancer. Molecular Cell. 61 (5), 667-676 (2016).
- Clare, D. K., Saibil, H. R. ATP-driven molecular chaperone machines. Biopolymers. 99 (11), 846-859 (2013).
- Tourniaire, F., et al. All-trans retinoic acid induces oxidative phosphorylation and mitochondria biogenesis in adipocytes. Journal of Lipid Research. 56 (6), 1100-1109 (2015).
- Psarra, A. M., Sekeris, C. E. Nuclear receptors and other nuclear transcription factors in mitochondria: regulatory molecules in a new environment. Biochimica et Biophysica Acta. 1783 (1), 1-11 (2008).
- Trigo, D., Goncalves, M. B., Corcoran, J. P. T. The regulation of mitochondrial dynamics in neurite outgrowth by retinoic acid receptor beta signaling. FASEB Journal. 33 (6), 7225-7235 (2019).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Current Protocols in Cell Biology. Chapter 4 (Unit 4), 1-21 (2010).
- Sajic, M., et al. Impulse conduction increases mitochondrial transport in adult mammalian peripheral nerves in vivo. PLoS Biology. 11 (12), e1001754(2013).
- Lefebvre, A., Ma, D., Kessenbrock, K., Lawson, D. A., Digman, M. A. Automated segmentation and tracking of mitochondria in live-cell time-lapse images. Nature Methods. 18 (9), 1091-1102 (2021).
- Chu, C. -H., Tseng, W. -W., Hsu, C. -M., Wei, A. -C. Image analysis of the mitochondrial network morphology with applications in cancer research. Frontiers in Physics. 10, 855775(2022).
- Creed, S., McKenzie, M. Measurement of mitochondrial membrane potential with the fluorescent dye tetramethylrhodamine methyl ester (TMRM). Methods in Molecular Biology. 1928, 69-76 (2019).
- Kovalevich, J., Langford, D. Considerations for the use of SH-SY5Y neuroblastoma cells in neurobiology. Methods in Molecular Biology. 1078, 9-21 (2013).
- Sahin, M., Oncu, G., Yilmaz, M. A., Ozkan, D., Saybasili, H. Transformation of SH-SY5Y cell line into neuron-like cells: Investigation of electrophysiological and biomechanical changes. Neuroscience Letters. 745, 135628(2021).
- Trigo, D., et al. Mitochondria dysfunction and impaired response to oxidative stress promotes proteostasis disruption in aged human cells. Mitochondrion. 69, 1-9 (2022).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados