Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Clasificación unicelular de células madre mesenquimales inmunofenotipadas de dientes deciduos exfoliados humanos
En este artículo
Resumen
Este protocolo describe el uso de la clasificación celular activada por fluorescencia de células madre mesenquimales humanas utilizando el método de clasificación de una sola célula. Específicamente, el uso de la clasificación de una sola célula puede lograr una pureza del 99% de las células inmunofenotipadas de una población heterogénea cuando se combina con un enfoque basado en citometría de flujo multiparamétrica.
Resumen
Las células madre mesenquimales (MSC) de un organismo poseen una extraordinaria capacidad para diferenciarse en múltiples linajes de células adultas en el cuerpo y son conocidas por sus propiedades inmunomoduladoras y antiinflamatorias. El uso de estas células madre es una bendición para el campo de la biología regenerativa, pero al mismo tiempo, una pesadilla para la medicina regenerativa y la terapéutica debido a las múltiples ambigüedades celulares asociadas con ellas. Estas ambigüedades pueden surgir de la diversidad en la fuente de estas células madre y de sus condiciones de crecimiento in vitro , las cuales reflejan su heterogeneidad funcional.
Esto justifica metodologías para proporcionar poblaciones purificadas y homogéneas de MSC para aplicaciones terapéuticas. Los avances en el campo de la citometría de flujo han permitido la detección de poblaciones unicelulares mediante un enfoque multiparamétrico. Este protocolo describe una forma de identificar y purificar las células madre de los dientes deciduos exfoliados humanos (SHED) a través de la clasificación de células individuales asistida por fluorescencia. La expresión simultánea de marcadores de superficie, a saber, el isotiocianato de fluoresceína CD90 (FITC), la proteína CD73-peridinina-clorofila (PerCP-Cy5.5), la aloficocianina CD105 (APC) y la CD44-V450, identificó los expresores positivos "brillantes" de las MSC mediante citometría de flujo multiparamétrica. Sin embargo, se observó una disminución significativa en los porcentajes de expresores cuádruples de estos marcadores positivos desde el pasaje 7 en adelante hasta los pasajes posteriores.
Las subpoblaciones inmunofenotipadas se clasificaron utilizando el modo de clasificación de una sola célula, donde solo dos marcadores positivos y uno negativo constituyeron los criterios de inclusión. Esta metodología garantizó la viabilidad celular de las poblaciones clasificadas y mantuvo la proliferación celular después de la clasificación. La aplicación posterior para dicha clasificación se puede utilizar para evaluar la diferenciación específica del linaje para las subpoblaciones cerradas. Este enfoque se puede aplicar a otros sistemas de una sola célula para mejorar las condiciones de aislamiento y para adquirir información de marcadores de superficie celular múltiple.
Introducción
Las células madre mesenquimales (MSC) pueden considerarse como una fuente escalable de células adecuadas para terapias basadas en células y pueden considerarse un sistema de referencia en medicina regenerativa. Estas células se pueden aislar de una variedad de fuentes en el cuerpo con diferentes orígenes tisulares1. Dependiendo de su tejido de origen, cada tipo de MSC muestra un comportamiento in vitro ambiguo2. Esto se observa bien en sus propiedades morfológicas y funcionales3. Múltiples estudios han demostrado variación intraclonal en las dimensiones, incluyendo la diferenciación tisular adulta, el estado genómico y la arquitectura metabólica y celular de las MSCs 2,4.
El inmunofenotipado de células ha sido una aplicación común de la citometría de flujo para la identificación de células madre y esto fue utilizado por la Sociedad Internacional de Terapia Celular y Génica (ISCT) en 2006 para prescribir una lista de criterios mínimos para identificar células como MSC. Afirmó que junto con la adherencia plástica y la capacidad de diferenciarse en tres linajes (osteogénico, condrogénico y adipogénico) in vitro, ≥95% de la población celular debe expresar CD105, CD73, CD90, y estas células deben carecer de la expresión (≤2% positiva) de CD34, CD45, CD11b, CD14 y HLA-DR, medida por citometría de flujo5. A pesar de que las MSC se definieron mediante un conjunto de biomarcadores bajo los criterios mínimos de la ISCT, sus propiedades inmunes no pudieron compararse con estos biomarcadores y se necesitaba más allá de estos criterios para facilitar la cuantificación de las comparaciones entre estudios y las variaciones clonales2.
A pesar de las directrices marcadas por el ISCT, una amplia investigación sobre las MSC ha demostrado que existe heterogeneidad en esta población, lo que podría deberse a una multitud de factores, principalmente debido a la naturaleza ubicua de la heterogeneidad que surge entre los donantes de MSC6, las fuentes de tejidos7, las células individuales dentro de una población clonal8 y las condiciones de cultivo 2,9. 10. La caracterización y purificación de estas células primarias a partir de una variedad de fuentes de tejidos para garantizar la calidad y el destino celular son pasos clave en su producción. La necesidad de comprender las variaciones mostradas entre la población requiere un método eficiente para resolverlas en subpoblaciones que puedan dividirse y recogerse por separado11. Los análisis a nivel de una sola célula ayudan a superar los desafíos de la variación célula-célula, reducen el ruido biológico que surge de una población heterogénea y ofrecen la capacidad de investigar y caracterizar células raras12.
En función del propósito y los parámetros elegidos, se pueden emplear varios métodos para clasificar y enriquecer las poblaciones seleccionadas. Las técnicas de clasificación por células pueden comprender tanto la clasificación masiva como los métodos de clasificación por células. Mientras que la clasificación masiva puede enriquecer las poblaciones objetivo a través de la clasificación de células activadas magnéticamente (MACS)13, el fraccionamiento14 y la elutriación 15, la clasificación de células individuales puede enriquecer poblaciones más homogéneas mediante la clasificación de células activadas por fluorescencia (FACS)11. En la Tabla 1 se destaca un análisis comparativo de cada uno de estos métodos con su propio conjunto de ventajas y desventajas.
Tabla 1: Análisis comparativos de diferentes técnicas: MACS, Fraccionamiento, Elutriación y FACS destacando las diferencias en su principio y las ventajas y desventajas de elegir una técnica en particular sobre otra. Abreviaturas: MACS = Clasificación celular activada magnéticamente; FACS = Clasificación celular activada por fluorescencia. Haga clic aquí para descargar esta tabla.
Desde el advenimiento de la técnica, la citometría de flujo de una sola célula ha desempeñado un papel importante en la enumeración16, detección y caracterización de una población celular específica en una muestra heterogénea17. Hewitt et al. en 2006 sentaron las bases de la metodología de clasificación celular automatizada para mejorar el aislamiento de grupos homogéneos de células madre embrionarias humanas diferenciadas (hESCs)18. La clasificación unicelular enriqueció la población de células madre embrionarias transducidas por GFP, facilitando el aislamiento de clones modificados genéticamente, lo que abrió una nueva dimensión en la investigación clínica. Para mejorar la eficiencia de la clasificación, generalmente se han adoptado dos enfoques; O bien se modifican los medios de recolección de las poblaciones clasificadas para mantener la viabilidad y proliferación de las células posclasificadas19 o bien se modifica adecuadamente el algoritmo/software de clasificación celular12.
Con el avance de la tecnología, los citómetros de flujo comerciales y los clasificadores celulares han podido ayudar a abordar los desafíos que se enfrentaron al clasificar asépticamente poblaciones celulares frágiles y raras, especialmente células madre de diferentes orígenes. Uno de los principales desafíos de los biólogos de células madre ha sido el aislamiento clonal de células madre pluripotentes humanas siguiendo los protocolos de transfección requeridos en los estudios de edición génica19. Esto se abordó mediante la clasificación de células individuales en placas de 96 pocillos que se recubrieron con fibroblastos embrionarios de ratón (MEF) junto con suplementos e inhibidores comerciales de ROCK de moléculas pequeñas. Sin embargo, las estrategias de aislamiento celular podrían refinarse en gran medida con el uso de la clasificación por índices, una característica del algoritmo de clasificación que identifica el inmunofenotipo de las células individuales clasificadas12. Esta modalidad refinada en la clasificación de células individuales ayudó no solo a mejorar la eficiencia de clasificación de las células madre, especialmente en lo que respecta a las poblaciones de células madre hematopoyéticas raras, sino que también vinculó de manera eficiente los clones de células individuales a sus ensayos funcionales posteriores20.
Este artículo se centra en la clasificación unicelular de células madre inmunofenotipadas de dientes deciduos exfoliados humanos (SHEDs) para el enriquecimiento de subpoblaciones con el fin de estudiar sus capacidades de diferenciación funcional. Utilizando una combinación de dos marcadores MSC positivos, CD90 y CD73, y un marcador hematopoyético negativo CD45, se inmunofenotiparon las MSC y se identificaron los expresores dim y null. Con base en su inmunofenotipo, las subpoblaciones se identificaron como MSC puras, poblaciones positivas simples y poblaciones negativas dobles. Se clasificaron utilizando el modo de clasificación de una sola célula para obtener subpoblaciones puras y enriquecidas para estudios funcionales posteriores con el fin de identificar si la expresión diferencial de los marcadores era un artefacto de las condiciones de cultivo in vitro o si también tiene algún efecto sobre las propiedades funcionales. Las células que no eran expresoras homogéneas de los "marcadores MSC positivos" se clasificaron para estudiar sus propiedades funcionales.
Protocolo
Aprobación ética y consentimiento para participar: Se recibieron muestras de pulpa dental caducifolia exfoliada humana después de obtener el consentimiento informado y la aprobación ética completa por parte del Departamento Oral y Maxilofacial del Sri Rajiv Gandhi Dental College and Hospital (SRGCDS), Bangalore, de acuerdo con los estándares establecidos por el Comité de Autorización Ética Hospitalaria, SRGCDS. Después de lo cual el aislamiento, el cultivo, el mantenimiento y la aplicación de SHEDs fueron aprobados y en cumplimiento de las directrices recomendadas por el Comité Institucional para la Investigación con Células Madre (IC-SCR) en el Instituto Manipal de Medicina Regenerativa, MAHE - Bangalore. Consulte la Tabla de materiales para obtener detalles sobre todos los materiales y reactivos utilizados en este protocolo.
1. Preparación de reactivos y tampones
- Para el mantenimiento de la cultura
- Preparar medios de cultivo celular MSC (10%) utilizando medio basal para hESCs indiferenciadas, 10% de suero fetal bovino (FBS), 1% de L-glutamina y 1% de penicilina-estreptomicina (Pen-strep) (Tabla 2).
- Preparar medios de cultivo celular MSC (20%) utilizando medio basal para hESCs indiferenciadas, 20% de FBS, 1% de L-glutamina y 1% de Pen-streptococo (Tabla 2).
- Preparar medios neutralizantes utilizando medio basal para hESCs indiferenciadas, L-glutamina al 1% y antibiótico-antimicótico (Anti-Anti) al 1% (Tabla 2).
- Para el análisis y la clasificación por citometría de flujo
- Prepare el tampón de tinción utilizando FBS al 2 % en solución salina tamponada con fosfato (PBS).
- Prepare una solución madre de 4′,6-diamidino-2-fenilindol (DAPI) (1 μg/mL) añadiendo 1 μL en 1 mL de PBS
- Para la diferenciación específica del linaje de las MSC
- Preparar medios de diferenciación para la diferenciación osteogénica, condrogénica y adipogénica de acuerdo con la composición descrita en la Tabla 3. Almacenar las alícuotas preparadas a 4 °C durante todo el experimento.
- Prepare medios sin suero (medio al 2 %) utilizando medio basal para hESC indiferenciadas, FBS al 2 %, L-glutamina al 1 % y Pen-streptococo al 1 % (Tabla 2).
| TIPO DE MEDIO | FINALIDAD DE LOS MEDIOS DE COMUNICACIÓN | COMPOSICIÓN PARA 50 mL | ||||||||
| FBS | Pen-Estreptococo | L-glutamina | MEDIO BASAL PARA C/ indiferenciadas | |||||||
| 10% medios de comunicación | Cultura y mantenimiento de MSC | 5 mL | 500 μL | 500 μL | 44 mL | |||||
| 20% medios de comunicación | Ensayo de UFC-F | 10 mL | 500 μL | 500 μL | 39 mL | |||||
| Medios hambrientos de suero (2%) | Medios para pozos de control en el protocolo de diferenciación | 1 ml | 500 μL | 500 μL | 48 mL | |||||
| Medios neutralizantes | Medios para neutralizar la suspensión celular después de la tripsinización | - | 500 μL | 500 μL | 49 mL | |||||
Tabla 2: Medios de cultivo celular para mantenimiento de cultivos y ensayos. Abreviaturas: MSC = célula madre mesenquimatosa; UFC-F = unidad formadora de colonias-fibroblasto.
| COMPONENTES | MEDIOS OSTEOGÉNICOS | MEDIOS CONDROGÉNICOS | MEDIOS ADIPÓGENOS |
| Medios basales | 90 mL | 90 mL | 90 mL |
| Medios de inducción | 10 mL | 10 mL | 10 mL |
| Volumen total | 100 ml | 100 ml | 100 ml |
Tabla 3: Medios de diferenciación para la diferenciación trilinaje de los SHEDs.
2. Cultivo y mantenimiento de los cobertizos
- Mantenga las células en medios de cultivo de MSC al 10% y realice cambios de medios cada 2 días o según sea necesario.
- Tripsinar las células al 95% de confluencia utilizando tripsina-EDTA al 0,25%.
- Neutralizar las células después de la tripsinización utilizando medios neutralizantes.
- Centrifugar el tubo a 300 × g durante 6 min a temperatura ambiente para obtener un gránulo celular.
- Decantar el sobrenadante y resuspender el gránulo celular en medios de cultivo de MSC al 10%.
- Siembre las células en placas de cultivo celular recién preparadas que contengan un 10% de medios de cultivo de MSC para experimentos posteriores o subcultivos.
NOTA: La densidad óptima de siembra para los SHEDs es de 0,2 × 10 6 celdas en un plato de 100 mm y de 0,8 × 10 6 celdas en un matraz T-75, para obtener 1,5 × 106 celdas y 4 × 106 celdas, respectivamente, con una confluencia del 90-100%.
3. Caracterización de las MSC
- Ensayo de crecimiento celular a corto plazo
- Siembre 4 × 104 células/pocillo por triplicado en placas de 6 pocillos en medios de cultivo de MSC al 10%.
- Incubar las placas durante 7 días a 37 °C y realizar cambios de medio cada 2 días.
- Recolectar las células los días 2, 4 y 8 con un tratamiento de tripsina al 0,25% y lavar con medios de cultivo.
- Centrifugar las células a 300 g durante 6 min a temperatura ambiente y resuspender el gránulo en 1 mL de medio.
- Contar las células con un hemocitómetro y determinar su viabilidad mediante el método de exclusión de azul de tripano.
- Calcule la tasa de proliferación utilizando la ecuación (1):
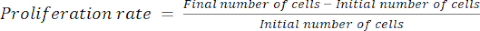 (1)
(1)
- Ensayo de fibroblastos unitarios formadores de colonias (UFC-F)
- Siembre 10.000 células en una placa de 100 mm y cultívelas en medios de cultivo MSC al 20%.
- Incubar las placas durante 14 días a 37 °C y realizar cambios de medio cada 3 días.
- Después de 14 días, enjuague las colonias con PBS, fíjelas con tinte violeta cristalino en metanol y enjuague nuevamente con PBS para eliminar la mancha residual.
- Contar e imaginar las colonias.
NOTA: Cuente las colonias con >50 celdas. La eficiencia de formación de colonias se calcula como el número de unidades de formación de colonias.
- Ensayo de inmunofluorescencia
- Siembre las MSC en platos de 35 mm y déjelas crecer hasta un 80-90% de confluencia.
- Retire el medio y enjuague los platos con PBS una vez.
- Fijar las células con 1 mL de paraformaldehído (PFA) al 4% incubando durante 1 h a temperatura ambiente o durante la noche a 4 °C.
- Después de la fijación, lave los pocillos con PBST durante 3 x 5 minutos en el balancín.
- Añadir Triton X-100 al 0,3% en PBST (solución de Tween 20 al 0,05% en PBS) para permeabilizar las células. Manténgalo en el balancín durante 15 minutos a temperatura ambiente.
- Lave los pocillos con PBST durante 3 x 5 minutos en el balancín.
- Añadir un 3% de albúmina sérica bovina (BSA) para el bloqueo y mantenerla en el balancín durante 1 h a temperatura ambiente.
- Lave los pocillos con PBST durante 3 x 5 minutos en el balancín.
- Añadir 800 μL de dilución 1:500 de anti-vimentina para ratones, mantenerla en el balancín durante 1 h a temperatura ambiente y transferir la placa a 4 °C para la incubación durante la noche.
- Al día siguiente, retire el anticuerpo primario y lave los pocillos con PBST durante 3 x 5 minutos en el balancín.
- Añadir 800 μL de dilución 1:1.000 del anticuerpo secundario adsorbido cruzado IgG (H+L) antiratón de cabra, Alexa Fluor 555, y mantenerlo durante 3 h a temperatura ambiente en el balancín.
- Lave los pocillos con PBST durante 3 x 5 minutos en el balancín.
- Añadir Alexa fluor 488 Pfaloidin Sondas 240 μL en 1.000 μL de PBS e incubar a temperatura ambiente durante 60 min en el balancín.
- Lave los pocillos con PBST durante 3 x 5 minutos en el balancín.
- Agregue 700 μL de montante DAPI y observe las células bajo un microscopio.
- Diferenciación específica del linaje
- Diferenciación siguiendo la cultura 2D
- Tome dos placas de 48 pocillos y etiquételas como linajes osteogénicos y adipogénicos, respectivamente.
- Siembre 15.000 células/pocillo en cuatro pocillos de cada placa y cultívelas en medios de cultivo de MSC al 10%.
- Una vez que la monocapa de células haya alcanzado el 90% de confluencia, etiquete los dos primeros pocillos como "Control" y reemplace el medio existente con medios hambrientos de suero (2% de medio). En los dos últimos pocillos etiquetados como 'Test', agregue medios de diferenciación de cualquiera de los dos linajes, adipogénicos u osteogénicos. Márcalo como Día 0.
- Reemplace suavemente los medios cada tres días para evitar la descamación y tenga cuidado de evitar la contaminación.
- Mantener estas condiciones hasta el vigésimo primer día; luego procese para el experimento de tinción.
- Diferenciación siguiendo la cultura 3D
- Tome dos tubos de 15 ml para realizar la diferenciación condrogénica mediante cultivo de gránulos en 3D.
- Transfiera 1 × 106 celdas a cada tubo y centrifugue a 300 × g durante 6 minutos para formar un gránulo. Etiquete un tubo como "Control" y agréguele un 10% de medios de cultivo MSC; etiquete el otro tubo como 'Prueba' y agregue medios de diferenciación condrogénicos. Coloque los tubos con cuidado en la incubadora con las tapas sin enroscar. Márcalo como Día 0.
- Cambie el medio cada tercer día con cuidado, para no desalojar/desintegrar el gránulo durante los cambios de medio.
- Mantenga estas condiciones hasta el vigésimo primer día y luego, procese las células para futuros experimentos.
- Diferenciación siguiendo la cultura 2D
- Tinción citoquímica específica del linaje
- Para cultivos 2D, utilice las placas de MSC diferenciadas (prueba) e indiferenciadas (control) (del paso 3.4.1.5.) para la tinción y, en primer lugar, retire el medio y lave dos veces con PBS. Fije las células con PFA al 4% durante 30 minutos a temperatura ambiente, retire el sobrenadante y lave una vez con PBS. Realice la tinción para cada linaje de la siguiente manera.
- Para el linaje de los adipocitos, agregue la solución de permeabilización del kit e incube la placa durante 5 minutos a temperatura ambiente. Preparar y añadir 1 mL de solución de trabajo de Oil red O y conservar durante 10 min. Retira la mancha y realiza cinco lavados con agua destilada.
- Para el linaje de los osteocitos, agregue un 5% de nitrato de plata recién preparado (en agua destilada) a cada pocillo y mantenga la placa bajo rayos UV durante 1 h. Retire la solución y agregue 2,5% de tiosulfato de sodio para eliminar la plata sin reaccionar; Guárdalo durante 5 min. Retire la mancha, lave dos veces con agua destilada y observe las células teñidas bajo un microscopio.
- Para cultivos 3D, recoja el gránulo después del final del período de diferenciación del paso 3.4.2.3 y obtenga criosecciones del tejido diferenciado en forma de gránulo. Deje que los portaobjetos se sequen al aire y estén a temperatura ambiente antes de proceder a la tinción.
- Para el linaje de condrocitos, siga las instrucciones del kit de tinción para agregar un volumen suficiente de solución de lavado, retírelo, agregue la solución de fijación e incube durante 30 minutos. Lavar con agua destilada, agregar la solución de tinción e incubar durante 30 min. Lavar tres veces con ácido clorhídrico 0,1 N; Agregue agua destilada para neutralizar la acidez. Observe las células teñidas bajo un microscopio de campo claro.
- Para cultivos 2D, utilice las placas de MSC diferenciadas (prueba) e indiferenciadas (control) (del paso 3.4.1.5.) para la tinción y, en primer lugar, retire el medio y lave dos veces con PBS. Fije las células con PFA al 4% durante 30 minutos a temperatura ambiente, retire el sobrenadante y lave una vez con PBS. Realice la tinción para cada linaje de la siguiente manera.
4. Tinción de la superficie celular para inmunofenotipado
NOTA: Las placas de cultivo celular recomendadas para obtener un número óptimo de células en los pasos 4.2-4.5 son placas de 100 mm o matraces T75.
- Preparación celular para experimentos de citometría de flujo
- Tripsinizar y recoger las células de una placa/matraz confluente y centrifugar a 300 × g durante 6 min para obtener el gránulo de células.
- Resuspender el pellet en 1 mL de medio y determinar el recuento de células viables utilizando un hemocitómetro siguiendo el método de exclusión de azul de tripano.
- Vuelva a centrifugar la suspensión celular después de contar y dé dos lavados más al gránulo con 1 ml de tampón de tinción.
- Deseche el sobrenadante y, finalmente, vuelva a suspender el gránulo en un volumen adecuado del tampón de tinción según el protocolo (consulte los pasos 4.2 a 4.5).
- Elaboración de controles de compensación
- Tome siete tubos FACS y etiquételos como Sin teñir, DAPI, V450, FITC, PE, PerCP-Cy 5.5 y APC.
- Vuelva a suspender el gránulo final en el tampón de tinción (ver paso 4.1.) manteniendo una densidad celular de 0,5 × 106 células por 50 μL por tubo para los tubos sin tinción y DAPI.
- Prepare los tubos de una sola tinción para la compensación como se describe en la Tabla 4.
- Agite suavemente cada tubo después de la preparación e incube en la oscuridad durante 30 minutos.
- Después de la incubación, realizar dos lavados añadiendo 1 ml de tampón de tinción a cada tubo, seguido de un breve vórtice y centrifugación a 200 g durante 10 min a temperatura ambiente.
- Deseche el sobrenadante, vuelva a suspender el gránulo en 500 μL de tampón de tinción y déjelo a un lado hasta la adquisición.
- Para el tubo DAPI, realice un tratamiento de choque térmico incubándolo en un baño de agua a 60 °C durante 5 minutos seguido de 15 minutos en hielo. Agregue 5 μL de DAPI a la suspensión y manténgala en la oscuridad hasta la adquisición de la carrera.
- Preparación de controles de fluorescencia menos uno (FMO)
- Vuelva a suspender el gránulo final en el tampón de tinción (ver paso 4.1) manteniendo una densidad celular de 0,5 × 106 células por 50 μL para cada tubo.
- Tome cinco tubos FACS y etiquételos como isotipo CD44-V450, CD90-FITC, CD45-PE, isotipo CD73-PerCP Cy5.5 e isotipo CD105-APC.
- Prepare la suspensión celular y de anticuerpos de acuerdo con la Tabla 5.
- Agite los tubos suavemente e incube durante 30 minutos a temperatura ambiente en la oscuridad.
- Después de la incubación, administrar dos lavados con 1 ml de tampón de tinción a cada tubo, seguidos de un breve vórtice y centrifugación a 200 g durante 10 min a temperatura ambiente.
- Deseche el sobrenadante, vuelva a suspender el gránulo en 500 μL de tampón de tinción y déjelo a un lado hasta su adquisición.
- Preparación de muestras para análisis
- Vuelva a suspender el gránulo final en el tampón de tinción (ver paso 4.1) manteniendo una densidad celular de 0,5 × 106 células por 50 μL para cada tubo.
- Tome dos tubos FACS y etiquételos como tubos mixtos 1 y 2.
- Prepare la célula y la suspensión de anticuerpos de acuerdo con la Tabla 6.
- Agite los tubos suavemente e incube durante 30 minutos a temperatura ambiente en la oscuridad.
- Después de la incubación, administrar dos lavados con 1 ml de tampón de tinción a cada tubo, seguidos de un breve vórtice y centrifugación a 200 g durante 10 min a temperatura ambiente.
- Deseche el sobrenadante, vuelva a suspender el gránulo en 500 μL de tampón de tinción y deje reposar hasta su adquisición.
- Preparación de muestras para la clasificación de células individuales
- Tome dos tubos FACS, agregue 1 ml de FBS y haga rodar el tubo hasta que se forme una capa uniforme de FBS en el interior de cada tubo. Incube esto durante 1-2 h mientras procesa la muestra para teñirla. Etiquete estos tubos como tubos mixtos 1 y 2.
- Vuelva a suspender el gránulo final en el tampón de tinción (ver paso 4.1) manteniendo una densidad celular de 2-3 × 106 células por 50 μL para cada tubo.
- Preparar la célula y la suspensión de anticuerpos en los tubos mixtos 1 y 2 de acuerdo con la Tabla 7.
- Agite los tubos suavemente e incube durante 30 minutos a temperatura ambiente en la oscuridad.
- Después de la incubación, administrar dos lavados con 1 ml de tampón de tinción a cada tubo, seguidos de un breve vórtice y centrifugación a 200 g durante 10 min a temperatura ambiente.
- Deseche el sobrenadante, vuelva a suspender el gránulo en 500 μL de tampón de tinción y reserve. Agregue 5 μL de DAPI 15 min antes de la clasificación.
NOTA: Tenga en cuenta que para los experimentos de clasificación se recomienda una mayor densidad de células por tubo.
| Tipo de tubo | Cuentas Comp Positivas* | Cuentas Comp negativas* | Células | Anticuerpos añadidos | |
| Tubo FITC | 1 gota | 1 gota | – | Anti-humano CD90-FITC (2 μL) | |
| Tubo V450 | 1 gota | 1 gota | – | Antihumano CD44-V450 (2 μL) | |
| Tubo PerCP-Cy 5.5 | 1 gota | 1 gota | – | Anti-humano CD73-PerCP Cy 5.5 (2 μL) | |
| Tubo de PE | 1 gota | 1 gota | – | CD45-PE antihumano (2 μL) | |
| Tubo APC | 1 gota | 1 gota | – | Anti-humano CD105-APC (2 μL) | |
| Tubo DAPI | – | – | 50 μL | – | |
| Tubo sin teñir | – | – | 50 μL | – | |
| *1 gota = 60 μL de suspensión de cordones | |||||
Tabla 4: Muestras de control de compensación. Abreviaturas: Comp = compensación; DAPI = 4',6-diamidino-2-fenilindol; FITC = isotiocianato de fluoresceína; APC = aloficocianina; PE = ficoeritrina; PerCP = peridinina-clorofila-proteína.
| TIPO DE TUBO | ANTICUERPO CONTRA MARCADOR POSITIVO (2 μL) | ANTICUERPO CONTRA MARCADOR NEGATIVO (2 μL) | ANTICUERPOS DE ISOTIPO AÑADIDOS (2 μL) | VOLUMEN TOTAL DE ANTICUERPOS | VOLUMEN DE SUSPENSIÓN CELULAR AÑADIDO | VOLUMEN DE TAMPÓN DE TINCIÓN AÑADIDO | ||
| Tubo CD90-FITC FMO | - Antihumano CD44-V450 | Anti-humano CD45-PE | Isotipo FITC IgG1 | 10 μL | 50 μL | 40 μL | ||
| - Anti-humano CD73 PerCP Cy 5.5 | ||||||||
| - CD105-APC antihumano | ||||||||
| Tubo CD73-PerCP Cy5.5 FMO | - Antihumano CD44-V450 | Anti-humano CD45-PE | Isotipo PerCP Cy 5.5 IgG1 | 10 μL | 50 μL | 40 μL | ||
| - Anti-humano CD90-FITC | ||||||||
| - CD105-APC antihumano | ||||||||
| Válvula CD44-V450 FMO | - Anti-humano CD90-FITC | Anti-humano CD45-PE | Isotipo V450 IgG1 | 10 μL | 50 μL | 40 μL | ||
| - Anti-humano CD73-PerCP Cy 5.5 | ||||||||
| - CD105-APC antihumano | ||||||||
| Tubo CD105-APC FMO | - Antihumano CD44-V450 | Anti-humano CD45-PE | Isotipo APC IgG1 | 10 μL | 50 μL | 40 μL | ||
| - Anti-humano CD90-FITC | ||||||||
| - Anti-humano CD73-PerCP Cy 5.5 | ||||||||
| Tubo CD45-PE FMO | - Antihumano CD44-V450 | - | Isotipo PE IgG1 | 10 μL | 50 μL | 40 μL | ||
| - Anti-humano CD90-FITC | ||||||||
| - Anti-humano CD73-PerCP Cy 5.5 | ||||||||
| - CD105-APC antihumano | ||||||||
Tabla 5: Muestras de control de FMO. Abreviaturas: FMO = fluorescencia menos uno; FITC = isotiocianato de fluoresceína; APC = aloficocianina; PE = ficoeritrina; PerCP = peridinina-clorofila-proteína.
| TIPO DE TUBO | ANTICUERPO CONTRA MARCADOR POSITIVO (2 μL) | ANTICUERPO CONTRA MARCADOR NEGATIVO (2 μL) | VOLUMEN TOTAL DE ANTICUERPOS | VOLUMEN DE SUSPENSIÓN CELULAR AÑADIDO | VOLUMEN DE TAMPÓN DE TINCIÓN AÑADIDO | |
| Tubo mixto 1 | - Antihumano CD44-V450 | Anti-humano CD45-PE | 10 μL | 50 μL | 40 μL | |
| - Anti-humano CD90-FITC | ||||||
| - Anti-humano CD73-PerCP Cy5.5 | ||||||
| - CD105-APC antihumano | ||||||
| Tubo mixto 2 | - Antihumano CD44-V450 | Anti-humano CD45-PE | 10 μL | 50 μL | 40 μL | |
| - Anti-humano CD90-FITC | ||||||
| - Anti-humano CD73-PerCP Cy5.5 | ||||||
| - CD105-APC antihumano | ||||||
Tabla 6: Tubos de muestra para inmunofenotipado multicolor de SHEDs. Abreviaturas: SHEDs = células madre de dientes deciduos humanos exfoliados; PE = ficoeritrina.
| TIPO DE TUBO | ANTICUERPO CONTRA MARCADOR POSITIVO (3 μL) | ANTICUERPO CONTRA MARCADOR NEGATIVO (3 μL) | VOLUMEN TOTAL DE ANTICUERPOS | VOLUMEN DE SUSPENSIÓN CELULAR AÑADIDO | VOLUMEN DE TAMPÓN ANTIMANCHAS AÑADIDO | |
| Tubo mixto 1 | - Anti-humano CD90-FITC | Anti-humano CD45-PE | 9 μL | 50 μL | 41 μL | |
| - Anti-humano CD73-PerCP Cy5.5 | ||||||
| Tubo mixto 2 | - Anti-humano CD90-FITC | Anti-humano CD45-PE | 9 μL | 50 μL | 41 μL | |
| - Anti-humano CD73-PerCP Cy5.5 | ||||||
Tabla 7: Tubos de reacción de clasificación de una sola célula. Abreviaturas: FITC = isotiocianato de fluoresceína; PE = ficoeritrina; PerCP = peridinina-clorofila-proteína.
5. Clasificación de una sola célula
- Preparación del clasificador de células
- Instale una boquilla de 100 μm en el clasificador.
NOTA: La boquilla adecuada tiene al menos cinco veces el diámetro de la partícula que se va a clasificar. El fluido de la vaina que se utilizará para la clasificación debe decidirse en función del tipo de muestra y la sensibilidad del experimento; Para este experimento, se ha utilizado un fluido de vaina patentado. - Realice el control de calidad (QC) diario del instrumento y configure el clasificador para el experimento. Consulte el manual del instrumento para obtener una guía detallada sobre la configuración del instrumento.
- Instale una boquilla de 100 μm en el clasificador.
- Configuración de la matriz de compensación
- Ajuste la matriz de compensación utilizando tubos de compensación de una sola mancha del paso 4.2.
- En el software propietario, seleccione Experimento en la barra de herramientas y haga clic en Configuración de compensación. Abra Crear controles de compensación.
- Revisa los marcadores y confirma. Se agrega un nuevo espécimen llamado "Compensación" bajo el cual el software agrega automáticamente nuevos tubos denominados controles marcadores.
- Seleccione el tubo Unstained y ejecútelo para registrar 5.000 eventos. Arrastre la puerta a la población de celdas y aplíquela a todos los controles de compensación. Esto es para establecer los voltajes y la puerta negativa para cada parámetro fluorescente.
- Del mismo modo, cargue los tubos de compensación de una sola mancha por separado y registre y guarde los datos. Seleccione la puerta que delimita la población de interés y aplíquela a todos los controles de compensación. Esto es para establecer las puertas positivas para cada parámetro fluorescente.
- Seleccione Experimento en la barra de herramientas y haga clic en Calcular valores de compensación | enlace y guardar.
NOTA: Una vez que se genera la matriz de auto-composición utilizando el software, los parámetros de voltaje de los fluorocromos no se pueden cambiar para ninguno de los canales de los tubos mixtos.
- Adquisición de datos
- Registre 10.000 células en cada tubo de los pasos 4.3. y 4.4 para recoger datos para el análisis del inmunofenotipo de las células.
- Preparación de los dispositivos de recogida
- Dependiendo del propósito de las poblaciones celulares ordenadas, elija entre placas de 6 pocillos, 24 pocillos, 48 pocillos o 96 pocillos.
- Cubra los pocillos con 200-500 μL de FBS y mantenga las placas intactas durante 2 h.
- Después de 2 h, retire el FBS residual y agregue 200-500 μL de medio de cultivo MSC al 10%.
- Clasificación de celdas en modo de clasificación de una sola celda
- Ejecute el tubo mixto 1 (del paso 4.5) y registre 10.000 eventos para establecer las puertas en la población de interés que se va a ordenar, utilizando la estrategia de compuerta adecuada.
- Cargue una placa de recolección y establezca los números de celda objetivo entre 2.500 y 5.000 celdas/pocillo y seleccione la máscara de pureza de clasificación de una sola celda.
- Recoja las poblaciones clasificadas en el dispositivo de recolección y manténgalas en hielo hasta el final del experimento de clasificación.
- Una vez hecho esto, transfiera las placas a la incubadora de CO2 al 5% para mantener los cultivos a 37 °C.
NOTA: Después de la adquisición, los archivos de datos sin procesar se exportaron en formato de archivo .fcs (v.3.0. en adelante). Los informes de ordenación generados después de cada experimento registraron el número de eventos/celdas ordenados por pozo asignado e indicaron el número de conflictos que se abortaron.
Resultados
Los SHEDs se caracterizaron con ensayos de inmunofluorescencia estándar que mostraron la expresión de vimentina (rojo, filamentos intermedios tipo III), filamentos de actina (Alexa fluor 488 Phalloidin Probes) y núcleos teñidos con DAPI (Figura 1A). Para estimar sus capacidades proliferativas y formadoras de colonias, se realizaron ensayos estándar de crecimiento celular a corto plazo. En la Figura 1B se muestra un aumento de 14,3 veces en la tasa de prolif...
Discusión
En el campo de la ingeniería de tejidos y la medicina regenerativa, entre las fuentes postnatales, las MSC derivadas de tejidos orales han atraído un profundo interés debido a sus obligaciones éticas mínimas y a su notable potencial de diferenciación multilinaje21. Las células madre de la pulpa dental (CPD) del tercer molar impactado y las SHEDs han atraído la mayor atención entre las MSC dentales por su potencial terapéutico en enfermedades neurodegenerativas y traumáticas
Divulgaciones
Los autores declaran que no existe ningún conflicto de intereses con respecto a la publicación de este artículo.
Agradecimientos
Agradecemos a la Instalación de Celdas de Flujo del Centro Jawaharlal Nehru para la Investigación Científica Avanzada, Bangalore, India, por el uso de la instalación central de citometría de flujo. El criocorte del cultivo de gránulos de células diferenciadas se realizó en el Laboratorio de Referencia Neuberg Anand, Bangalore, India. Este trabajo fue apoyado por el financiamiento interno de la UC de la Academia de Educación Superior de Manipal (MAHE), India. AG agradece el apoyo de la Beca Dr. T. M. A. Pai de MAHE.
Materiales
| Name | Company | Catalog Number | Comments |
| Alcian Blue Stain | HiMedia | CCK029-1KT | |
| Antibiotic-Antimycotic (100x) | Gibco by ThermoFisher | 15240062 | |
| BD CompBead Plus Anti-Mouse Ig, κ/Negative Control (BSA) Compensation Plus (7.5 µm) Particles Set | BD Biosciences | 560497 | |
| BD FACS Accudrop Beads | BD Biosciences | 345249 | Used to set up the Laser delay when the sort module opens. |
| BD FACS Aria Fusion Flow cytometer | BD Biosciences | --- | |
| BD FACS Diva 9.4 | BD Biosciences | --- | |
| BD FACS Sheath Fluid | BD Biosciences | 342003 | Used as sheath fluid for both analysis and sorting experiments in the BD FACSAria Fusion |
| BD FACSDiva CS&T Research Beads | BD Biosciences | 655050 | Used for Instrument configuration depending on the nozzle size. |
| BD Horizon V450 Mouse Anti-Human CD44 | BD Biosciences | 561292 | |
| BD Horizon V450 Mouse IgG2b, κ Isotype Control | BD Biosciences | 560374 | CD44-V450 isotype |
| BD Pharmingen APC Mouse Anti-Human CD105 | BD Biosciences | 562408 | |
| BD Pharmingen APC Mouse IgG1, κ Isotype Control | BD Biosciences | 555751 | CD105-APC isotype |
| BD Pharmingen DAPI Solution | BD Biosciences | 564907 | DAPI Stock solution of 1 mg/mL |
| BD Pharmingen FITC Mouse Anti-Human CD90 | BD Biosciences | 555595 | |
| BD Pharmingen FITC Mouse IgG1, κ Isotype Control | BD Biosciences | 555748 | CD90-FITC isotype |
| BD Pharmingen PE Mouse Anti-Human CD45 | BD Biosciences | 555483 | |
| BD Pharmingen PE Mouse IgG1, κ Isotype Control | BD Biosciences | 555749 | CD45-PE isotype |
| BD Pharmingen PerCP-Cy 5.5 Mouse Anti-Human CD73 | BD Biosciences | 561260 | |
| BD Pharmingen PerCP-Cy 5.5 Mouse IgG1, κ Isotype Control | BD Biosciences | 550795 | CD73-PerCP-Cy 5.5 isotype |
| BD Pharmingen Purified Mouse Anti-Vimentin | BD Biosciences | 550513 | |
| Bovine serum albumin | Hi-Media | TC548-5G | |
| Crystal violet | Nice chemical pvt ltd | C33809 | |
| Dulbecco's Phosphate Buffered Saline | Sigma-aldrich | D5652-50L | dPBS used for culture work and maintenance. |
| Ethanol | --- | --- | Used for general sterlization. |
| Fetal Bovine Serum | Gibco by ThermoFisher | 10270-106 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 555 | ThermoFisher Scientific | A-21422 | |
| KO-DMEM | Gibco by ThermoFisher | 10829018 | Basal medium for undifferentiated hESCs, used in the preparation of culture media |
| L-Glutamine 200mM (100x) | Gibco by ThermoFisher | 25030-081 | |
| Methanol, for Molecular Biology | Hi-Media | MB113 | |
| Oil red O | HiMedia | CCK013-1KT | |
| Paraformaldehyde | loba chemie | 30525-89-4 | |
| Penicillin Streptomycin (100x) | Gibco by ThermoFisher | 15140- 122 | |
| Phalloidin (ActinGreen 488 ReadyProbes reagent) | Invitrogen | R37110 | |
| Silver Nitrate | HiMedia | MB156-25G | |
| Sodium Thiosulphate pentahydrate | Chemport | 10102-17-7 | |
| Sphero Rainbow Fluorescent Particles, 3.0 - 3.4 µm | BD Biosciences | 556291 | |
| Staining buffer | Prepared in MIRM | ---- | It was prepared using 2% FBS in PBS |
| StemPro Adipogenesis Differentiation Basal Media | Gibco by ThermoFisher | A10410-01 | Basal media for Adipogenic media |
| StemPro Adipogenesis Supplement | Gibco by ThermoFisher | A10065-01 | Induction media for Adipogenic media |
| StemPro Chondrogenesis Supplement | Gibco by ThermoFisher | A10064-01 | Induction media for Chondrogenic media |
| StemPro Osteogenesis Supplement | Gibco by ThermoFisher | A10066-01 | Induction media for Osteoogenic media |
| StemPro Osteogenesis/Chondrogenesis Differentiation Basal Media | Gibco by ThermoFisher | A10069-01 | Basal media for both Ostegenic and Chondrogenic media |
| Triton-X-100 | Hi-Media | MB031 | |
| Trypan Blue | Gibco by life technologies | 15250-061 | |
| Trypsin - EDTA Solution 1x | Hi-media | TCL049 | |
| Tween-20 | MERCK | 9005-64-5 |
Referencias
- Kobolak, J., Dinnyes, A., Memic, A., Khademhosseini, A., Mobasheri, A. Mesenchymal stem cells: Identification, phenotypic characterization, biological properties and potential for regenerative medicine through biomaterial micro-engineering of their niche. Methods. 99, 62-68 (2016).
- Wilson, A., Hodgson-Garms, M., Frith, J. E., Genever, P. Multiplicity of mesenchymal stromal cells: finding the right route to therapy. Frontiers in Immunology. 10, 1112 (2019).
- Li, J., et al. Comparison of the biological characteristics of human mesenchymal stem cells derived from exfoliated deciduous teeth, bone marrow, gingival tissue, and umbilical cord. Molecular Medicine Reports. 18 (6), 4969-4977 (2018).
- McLeod, C. M., Mauck, R. L. On the origin and impact of mesenchymal stem cell heterogeneity: new insights and emerging tools for single cell analysis. European Cells & Materials. 34, 217-231 (2017).
- Dominici, M., et al. Minimal criteria for defining multipotent mesenchymal stromal cells. The International Society for Cellular Therapy position statement. Cytotherapy. 8 (4), 315-317 (2006).
- Wang, J., Liao, L., Wang, S., Tan, J. Cell therapy with autologous mesenchymal stem cells-how the disease process impacts clinical considerations. Cytotherapy. 15 (8), 893-904 (2013).
- Kern, S., Eichler, H., Stoeve, J., Kluter, H., Bieback, K. Comparative analysis of mesenchymal stem cells from bone marrow, umbilical cord blood, or adipose tissue. Stem Cells. 24 (5), 1294-1301 (2006).
- Dunn, C. M., Kameishi, S., Grainger, D. W., Okano, T. Strategies to address mesenchymal stem/stromal cell heterogeneity in immunomodulatory profiles to improve cell-based therapies. Acta Biomaterialia. 133, 114-125 (2021).
- Yang, Y. K., Ogando, C. R., Wang See, C., Chang, T. Y., Barabino, G. A. Changes in phenotype and differentiation potential of human mesenchymal stem cells aging in vitro. Stem Cell Research & Therapy. 9 (1), 131 (2018).
- Costa, L. A., et al. Functional heterogeneity of mesenchymal stem cells from natural niches to culture conditions: implications for further clinical uses. Cellular and Molecular Life Sciences. 78 (2), 447-467 (2021).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Review of Scientific Instruments. 43 (3), 404-409 (1972).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
- Liao, X., Makris, M., Luo, X. M. Fluorescence-activated cell sorting for purification of plasmacytoid dendritic cells from the mouse bone marrow. Journal of Visualized Experiments. (117), (2016).
- Roda, B., et al. A novel stem cell tag-less sorting method. Stem Cell Reviews and Reports. 5 (4), 420-427 (2009).
- Hall, S. R., et al. Identification and isolation of small CD44-negative mesenchymal stem/progenitor cells from human bone marrow using elutriation and polychromatic flow cytometry. Stem Cells Translational Medicine. 2 (8), 567-578 (2013).
- Eggleton, M. J., Sharp, A. A. Platelet counting using the Coulter electronic counter. Journal of Clinical Pathology. 16 (2), 164-167 (1963).
- Porwit-Ksiazek, A., Aman, P., Ksiazek, T., Biberfeld, P. Leu 7+ (HNK-1+) cells. II. Characterization of blood Leu 7+ cells with respect to immunophenotype and cell density. Scandinavian Journal of Immunology. 18 (6), 495-449 (1983).
- Hewitt, Z., et al. Fluorescence-activated single cell sorting of human embryonic stem cells. Cloning and Stem Cells. 8 (3), 225-234 (2006).
- Singh, A. M. An efficient protocol for single-cell cloning human pluripotent stem cells. Frontiers in Cell and Developmental Biology. 7, 11 (2019).
- Wilson, N. K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Zhou, L. L., et al. Oral mesenchymal stem/progenitor cells: the immunomodulatory masters. Stem Cells Inernational. 2020, 1327405 (2020).
- Fawzy El-Sayed, K. M., et al. Adult mesenchymal stem cells explored in the dental field. Advances in Biochemical Engineering/Biotechnology. 130, 89-103 (2013).
- Hardy, W. R., et al. Transcriptional networks in single perivascular cells sorted from human adipose tissue reveal a hierarchy of mesenchymal stem cells. Stem Cells. 35 (5), 1273-1289 (2017).
- . . FACSAria Fusion User's Guide. , (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados