È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Ordinamento a singola cellula di cellule staminali mesenchimali immunofenotipizzate da denti decidui esfoliati umani
In questo articolo
Riepilogo
Questo protocollo descrive l'uso della selezione cellulare attivata dalla fluorescenza di cellule staminali mesenchimali umane utilizzando il metodo di selezione a singola cellula. In particolare, l'uso del sorting a singola cellula può raggiungere una purezza del 99% delle cellule immunofenotipizzate da una popolazione eterogenea se combinato con un approccio basato sulla citometria a flusso multiparametrica.
Abstract
Le cellule staminali mesenchimali (MSC) di un organismo possiedono una straordinaria capacità di differenziarsi in più lignaggi di cellule adulte nel corpo e sono note per le loro proprietà immunomodulatorie e antinfiammatorie. L'uso di queste cellule staminali è una manna per il campo della biologia rigenerativa, ma allo stesso tempo, una rovina per la medicina rigenerativa e le terapie a causa delle molteplici ambiguità cellulari ad esse associate. Queste ambiguità possono derivare dalla diversità nella fonte di queste cellule staminali e dalle loro condizioni di crescita in vitro , che si riflettono entrambe sulla loro eterogeneità funzionale.
Ciò garantisce metodologie per fornire popolazioni purificate e omogenee di MSC per applicazioni terapeutiche. I progressi nel campo della citometria a flusso hanno permesso di rilevare popolazioni di singole cellule utilizzando un approccio multiparametrico. Questo protocollo delinea un modo per identificare e purificare le cellule staminali dai denti decidui esfoliati umani (SHED) attraverso la selezione di singole cellule assistita da fluorescenza. L'espressione simultanea di marcatori di superficie, vale a dire, CD90-isotiocianato di fluoresceina (FITC), CD73-peridinina-clorofilla-proteina (PerCP-Cy5.5), CD105-alloficocianina (APC) e CD44-V450, ha identificato gli espressiori positivi "luminosi" delle MSC utilizzando la citometria a flusso multiparametrica. Tuttavia, è stato osservato un calo significativo nelle percentuali di quadrupli espressiori di questi marcatori positivi dal passaggio 7 in poi ai passaggi successivi.
Le sottopopolazioni immunofenotipizzate sono state ordinate utilizzando la modalità di ordinamento a singola cellula in cui solo due marcatori positivi e uno negativo costituivano i criteri di inclusione. Questa metodologia ha garantito la vitalità cellulare delle popolazioni selezionate e ha mantenuto la proliferazione cellulare dopo lo smistamento. L'applicazione a valle per tale ordinamento può essere utilizzata per valutare la differenziazione specifica del lignaggio per le sottopopolazioni gated. Questo approccio può essere applicato ad altri sistemi a singola cellula per migliorare le condizioni di isolamento e per acquisire informazioni multiple sui marcatori di superficie cellulare.
Introduzione
Le cellule staminali mesenchimali (MSC) possono essere considerate una fonte scalabile di cellule adatte alle terapie cellulari e possono essere considerate un sistema gold standard nella medicina rigenerativa. Queste cellule possono essere isolate da una varietà di fonti nel corpo con diverse origini tissutali1. A seconda del tessuto di origine, ogni tipo di MSC mostra un comportamento in vitro ambiguo2. Ciò è ben osservato nelle loro proprietà morfologiche e funzionali3. Diversi studi hanno mostrato variazioni intraclonali nelle dimensioni, tra cui la differenziazione dei tessuti adulti, lo stato genomico e l'architettura metabolica e cellulare delle MSC 2,4.
L'immunofenotipizzazione delle cellule è stata un'applicazione comune della citometria a flusso per l'identificazione delle cellule staminali ed è stata utilizzata dall'International Society for Cell and Gene Therapy (ISCT) nel 2006 per prescrivere un elenco di criteri minimi per identificare le cellule come MSC. Ha affermato che insieme all'aderenza plastica e alla capacità di differenziarsi in tre linee (osteogeniche, condrogeniche e adipogeniche) in vitro, il ≥95% della popolazione cellulare deve esprimere CD105, CD73, CD90 e queste cellule devono mancare dell'espressione (≤2% positiva) di CD34, CD45, CD11b, CD14 e HLA-DR, come misurato dalla citometria a flusso5. Sebbene le MSC fossero definite da una serie di biomarcatori secondo i criteri minimi dell'ISCT, le loro proprietà immunitarie non potevano essere confrontate con questi biomarcatori e c'era bisogno di ulteriori oltre questi criteri per rendere più facili da quantificare i confronti tra studi e le variazioni clonali2.
Nonostante le linee guida stabilite dall'ISCT, un'ampia ricerca sulle MSC ha dimostrato che esiste un'eterogeneità in questa popolazione, che potrebbe derivare da una moltitudine di fattori, principalmente a causa della natura ubiquitaria dell'eterogeneità che si verifica tra i donatori di MSC6, le fonti di tessuto7, le singole cellule all'interno di una popolazione clonale8 e le condizioni di coltura2,9, Ore 10. La caratterizzazione e la purificazione di queste cellule primarie da una varietà di fonti tissutali per garantire la qualità e il destino cellulare sono fasi chiave della loro produzione. La necessità di comprendere le variazioni mostrate all'interno della popolazione richiede un metodo efficiente per risolverle in sottopopolazioni che possono essere suddivise e raccolte separatamente11. Le analisi a livello di singola cellula aiutano a superare le sfide della variazione cellula-cellula, riducono il rumore biologico derivante da una popolazione eterogenea e offrono la possibilità di studiare e caratterizzare cellule rare12.
In base allo scopo e ai parametri scelti, possono essere impiegati diversi metodi per ordinare e arricchire le popolazioni selezionate. Le tecniche di selezione cellulare possono comprendere sia metodi di selezione alla rinfusa che metodi di selezione a singola cellula. Mentre lo smistamento di massa può arricchire le popolazioni bersaglio attraverso lo smistamento cellulare attivato magneticamente (MACS)13, il frazionamento 14 e l'elutriazione 15, lo smistamento a cellula singola può arricchire popolazioni più omogenee per mezzo del selezionatore cellulare attivato dalla fluorescenza (FACS)11. Un'analisi comparativa di ciascuno di questi metodi, con il proprio insieme di vantaggi e svantaggi, è evidenziata nella Tabella 1.
Tabella 1: Analisi comparative di diverse tecniche: MACS, Frazionamento, Elutriazione e FACS evidenziando le differenze nel loro principio e i vantaggi e gli svantaggi della scelta di una particolare tecnica rispetto ad un'altra. Abbreviazioni: MACS = Magnetic-activated cell sorting; FACS = Selezione cellulare attivata dalla fluorescenza. Clicca qui per scaricare questa tabella.
Dall'avvento della tecnica, la citometria a flusso a singola cellula ha svolto un ruolo importante nell'enumerazione16, nel rilevamento e nella caratterizzazione di una specifica popolazione cellulare in un campione eterogeneo17. Hewitt et al. nel 2006 hanno gettato le basi di una metodologia di selezione cellulare automatizzata per migliorare l'isolamento di pool omogenei di cellule staminali embrionali umane differenziate (hESCs)18. Lo smistamento a singola cellula ha arricchito la popolazione di hESC trasdotte con GFP facilitando l'isolamento di cloni geneticamente modificati, aprendo una nuova dimensione nella ricerca clinica. Per migliorare l'efficienza dello smistamento, sono stati generalmente adottati due approcci; O i terreni di raccolta delle popolazioni selezionate vengono modificati per sostenere la vitalità e la proliferazione delle cellule post-selezionate19 o l'algoritmo/software di selezione delle cellule viene opportunamente modificato12.
Con il progresso della tecnologia, i citometri a flusso commerciali e i selezionatori cellulari sono stati in grado di aiutare ad affrontare le sfide che sono state affrontate durante lo smistamento asettico di popolazioni cellulari fragili e rare, in particolare cellule staminali di origini diverse. Una delle principali sfide dei biologi delle cellule staminali è stata l'isolamento clonale di cellule staminali pluripotenti umane seguendo i protocolli di trasfezione richiesti negli studi di editing genetico19. Questo problema è stato affrontato ordinando singole cellule in piastre a 96 pozzetti rivestite con fibroblasti embrionali di topo (MEF) insieme a integratori e inibitori commerciali di piccole molecole ROCK. Tuttavia, le strategie di isolamento cellulare potrebbero essere in gran parte perfezionate con l'uso dell'index sorting, una caratteristica dell'algoritmo di sorting che identifica l'immunofenotipo delle singole cellule selezionate12. Questa raffinata modalità di selezione di singole cellule ha contribuito non solo a migliorare l'efficienza di selezione per le cellule staminali, in particolare per quanto riguarda le rare popolazioni di cellule staminali ematopoietiche, ma ha anche collegato in modo efficiente i cloni a singola cellula ai loro saggi funzionali a valle20.
Questo articolo si concentra sullo smistamento di singole cellule di cellule staminali immunofenotipizzate da denti decidui esfoliati umani (SHED) per l'arricchimento di sottopopolazioni per studiare le loro capacità di differenziazione funzionale. Utilizzando una combinazione di due marcatori MSC-positivi, CD90 e CD73, e di un marcatore ematopoietico negativo CD45, le MSC sono state immunofenotipizzate e sono stati identificati gli espressiori deboli e nulli. Sulla base del loro immunofenotipo le sottopopolazioni sono state identificate come MSC pure, popolazioni singole positive e doppie negative. Sono stati ordinati utilizzando la modalità di ordinamento a singola cellula per ottenere sottopopolazioni pure e arricchite per ulteriori studi funzionali per identificare se l'espressione differenziale dei marcatori fosse un artefatto delle condizioni di coltura in vitro o se avesse qualche effetto anche sulle proprietà funzionali. Le cellule che non erano espressioni omogenee dei "marcatori MSC positivi" sono state ordinate per studiare le loro proprietà funzionali.
Protocollo
Approvazione etica e consenso alla partecipazione: i campioni di polpa dentale decidua esfoliata umana sono stati ricevuti dopo aver ottenuto il consenso informato e la piena approvazione etica da parte del Dipartimento orale e maxillofacciale dello Sri Rajiv Gandhi Dental College and Hospital (SRGCDS), Bengaluru, in conformità con gli standard stabiliti dal Comitato di autorizzazione etica dell'ospedale, SRGCDS. A seguito di ciò, l'isolamento, la coltura, il mantenimento e l'applicazione di SHED sono stati approvati e in conformità con le linee guida raccomandate dal Comitato Istituzionale per la Ricerca sulle Cellule Staminali (IC-SCR) presso l'Istituto di Medicina Rigenerativa Manipal, MAHE - Bengaluru. Vedere la Tabella dei materiali per i dettagli su tutti i materiali e i reagenti utilizzati in questo protocollo.
1. Preparazione di reagenti e tamponi
- Per la manutenzione delle colture
- Preparare i terreni di coltura cellulare MSC (10%) utilizzando terreno basale per hESC indifferenziate, 10% di siero fetale bovino (FBS), 1% di L-glutammina e 1% di penicillina-streptomicina (Pen-streptococco) (Tabella 2).
- Preparare i terreni di coltura cellulare MSC (20%) utilizzando terreno basale per hESC indifferenziate, 20% FBS, 1% L-glutammina e 1% Pen-strep (Tabella 2).
- Preparare i terreni neutralizzanti utilizzando un terreno basale per hESC indifferenziate, L-glutammina all'1% e antibiotico-antimicotico (Anti-Anti) all'1% (Tabella 2).
- Per l'analisi citofluorimetrica e lo smistamento
- Preparare il tampone di colorazione utilizzando FBS al 2% in soluzione salina tamponata con fosfato (PBS).
- Preparare una soluzione madre di 4′,6-diamidino-2-fenilindolo (DAPI) (1 μg/mL) aggiungendo 1 μL in 1 mL di PBS
- Per la differenziazione specifica del lignaggio delle MSC
- Preparare i terreni di differenziazione per la differenziazione osteogenica, condrogenica e adipogenica secondo la composizione descritta nella Tabella 3. Conservare le aliquote preparate a 4 °C per tutta la durata dell'esperimento.
- Preparare terreni affamati di siero (2% di terreno) utilizzando terreno basale per hESC indifferenziate, 2% FBS, 1% L-glutammina e 1% Pen-strep (Tabella 2).
| TIPO DI SUPPORTO | SCOPO DEI MEDIA | COMPOSIZIONE PER 50 mL | ||||||||
| FBS | Penna-Streptococco | L-Glutammina | TERRENO BASALE PER hESC INDIFFERENZIATE | |||||||
| 10% media | Cultura e manutenzione MSC | 5 mL | 500 μL | 500 μL | 44 ml | |||||
| 20% media | Test CFU-F | 10 ml | 500 μL | 500 μL | 39 ml | |||||
| Terreni affamati di siero (2%) | Terreni per pozzetti di controllo in protocollo di differenziamento | 1 mL | 500 μL | 500 μL | 48 ml | |||||
| Mezzi neutralizzanti | Terreno per neutralizzare la sospensione cellulare dopo tripsinizzazione | - | 500 μL | 500 μL | 49 ml | |||||
Tabella 2: Terreni di coltura cellulare per il mantenimento della coltura e i saggi. Abbreviazioni: MSC = cellula staminale mesenchimale; CFU-F = unità formante colonie-fibroblasto.
| COMPONENTI | TERRENI OSTEOGENICI | TERRENI CONDROGENICI | TERRENI ADIPOGENICI |
| Terreni basali | 90 ml | 90 ml | 90 ml |
| Mezzi di induzione | 10 ml | 10 ml | 10 ml |
| Volume totale | 100 ml | 100 ml | 100 ml |
Tabella 3: Mezzi di differenziazione per la differenziazione trilineage degli SHED.
2. Cultura e manutenzione degli SHED
- Mantenere le cellule in terreni di coltura MSC al 10% ed eseguire cambi di terreno ogni 2 giorni o secondo necessità.
- Tripsinizzare le cellule al 95% di confluenza utilizzando lo 0,25% di tripsina-EDTA.
- Neutralizzare le cellule dopo la tripsinizzazione utilizzando mezzi neutralizzanti.
- Centrifugare la provetta a 300 × g per 6 minuti a temperatura ambiente per ottenere un pellet cellulare.
- Decantare il surnatante e risospendere il pellet cellulare in terreni di coltura MSC al 10%.
- Semina le cellule in piastre di coltura cellulare appena preparate contenenti il 10% di terreni di coltura MSC per ulteriori esperimenti o sub-coltura.
NOTA: La densità di semina ottimale per i capannoni è di 0,2 × 10 6 celle in una capsula da 100 mm e di 0,8 × 10 6 celle in un matraccio T-75, per ottenere rispettivamente 1,5 × 10 6 celle e 4 × 10 6 cellule, al 90-100% di confluenza.
3. Caratterizzazione delle MSC
- Saggio di crescita cellulare a breve termine
- Seme 4 × 104 cellule/pozzetto in triplice copia in piastre da 6 pozzetti in terreni di coltura MSC al 10%.
- Incubare le piastre per 7 giorni a 37 °C ed effettuare cambi di terreno ogni 2 giorni.
- Raccogliere le cellule nei giorni 2, 4 e 8 utilizzando un trattamento con tripsina allo 0,25% e lavare con terreni di coltura.
- Centrifugare le cellule a 300 g per 6 minuti a temperatura ambiente e risospendere il pellet in 1 mL di terreno.
- Contare le cellule utilizzando un emocitometro e determinarne la vitalità con il metodo di esclusione del blu tripano.
- Calcolare il tasso di proliferazione utilizzando l'equazione (1):
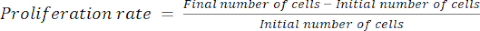 (1)
(1)
- Saggio dell'unità formante coloni-fibroblasti (CFU-F)
- Seminare 10.000 cellule in un piatto da 100 mm e coltivarle in terreni di coltura MSC al 20%.
- Incubare le piastre per 14 giorni a 37 °C ed effettuare cambi di terreno ogni 3 giorni.
- Dopo 14 giorni, sciacquare le colonie con PBS, fissarle con colorante crystal violet in metanolo e risciacquare nuovamente con PBS per rimuovere la macchia residua.
- Conta e immagina le colonie.
NOTA: Contare le colonie con >50 cellule. L'efficienza di formazione delle colonie è calcolata come numeri di unità formanti colonie.
- Test di immunofluorescenza
- Seminare le MSC su piastre da 35 mm e lasciarle crescere fino all'80-90% di confluenza.
- Rimuovere il supporto e sciacquare le stoviglie con PBS una volta.
- Fissare le cellule con 1 mL di paraformaldeide al 4% (PFA) incubandole per 1 ora a temperatura ambiente o per una notte a 4 °C.
- Dopo il fissaggio, lavare i pozzetti con PBST per 3 x 5 minuti sul bilanciere.
- Aggiungere lo 0,3% di Triton X-100 in PBST (soluzione di Tween 20 allo 0,05% in PBS) per permeabilizzare le cellule. Tienilo sul bilanciere per 15 minuti a temperatura ambiente.
- Lavare i pozzetti con PBST per 3 x 5 minuti sul bilanciere.
- Aggiungere il 3% di albumina sierica bovina (BSA) per bloccarlo e tenerlo sul bilanciere per 1 ora a temperatura ambiente.
- Lavare i pozzetti con PBST per 3 x 5 minuti sul bilanciere.
- Aggiungere 800 μL di diluizione 1:500 di antivimentina per topi, tenerlo sul bilanciere per 1 ora a temperatura ambiente e trasferire la piastra a 4 °C per l'incubazione notturna.
- Il giorno successivo, rimuovere l'anticorpo primario e lavare i pozzetti con PBST per 3 x 5 minuti sul bilanciere.
- Aggiungere 800 μL di diluizione 1:1.000 dell'anticorpo secondario adsorbito incrociato IgG (H+L) anti-topo di capra, Alexa Fluor 555, e tenerlo per 3 ore a temperatura ambiente sul bilanciere.
- Lavare i pozzetti con PBST per 3 x 5 minuti sul bilanciere.
- Aggiungere le sonde per falloidina Alexa fluor 488 da 240 μL in 1.000 μL di PBS e incubare a temperatura ambiente per 60 minuti sul bilanciere.
- Lavare i pozzetti con PBST per 3 x 5 minuti sul bilanciere.
- Aggiungere 700 μL di montante DAPI e osservare le cellule al microscopio.
- Differenziazione specifica del lignaggio
- Differenziazione in base alla cultura 2D
- Prendi due piastre da 48 pozzetti ed etichettale rispettivamente come linee osteogeniche e adipogeniche.
- Seminare 15.000 cellule/pozzetto in quattro pozzetti di ciascuna piastra e coltivarle in terreni di coltura MSC al 10%.
- Una volta che il monostrato di cellule ha raggiunto il 90% di confluenza, etichettare i primi due pozzetti come "Controllo" e sostituire il terreno esistente con terreno affamato di siero (2% di terreno). Negli ultimi due pozzetti etichettati come "Test", aggiungere i mezzi di differenziazione di uno dei due lignaggi, adipogenico o osteogenico. Contrassegnalo come Giorno 0.
- Sostituisci delicatamente i terreni ogni tre giorni per evitare la desquamazione e fai attenzione a evitare contaminazioni.
- Mantieni queste condizioni fino al ventunesimo giorno; quindi il processo per l'esperimento di colorazione.
- Differenziazione in base alla cultura 3D
- Prelevare due provette da 15 mL per eseguire la differenziazione condrogenica utilizzando la coltura di pellet 3D.
- Trasferire 1 × 10 6 cellule in ciascuna provetta e centrifugare a 300 × g per6 minuti per formare un pellet. Etichettare una provetta come "Controllo" e aggiungervi il 10% di terreno di coltura MSC; etichettare l'altra provetta come 'Test' e aggiungere il mezzo di differenziazione condrogenica. Posizionare con cura le provette nell'incubatrice con i tappi avvitati senza stringere. Contrassegnalo come Giorno 0.
- Cambiare il supporto ogni tre giorni con attenzione, in modo da non spostare/disintegrare il pellet durante i cambi di supporto.
- Mantieni queste condizioni fino al ventunesimo giorno e poi elabora le cellule per ulteriori esperimenti.
- Differenziazione in base alla cultura 2D
- Colorazione citochimica specifica per il lignaggio
- Per le colture 2D, utilizzare le piastre MSC differenziate (test) e indifferenziate (controllo) (dal punto 3.4.1.5.) per la colorazione e, per prima cosa, rimuovere il terreno e lavare due volte con PBS. Fissare le cellule con PFA al 4% per 30 minuti a temperatura ambiente, rimuovere il surnatante e lavare una volta con PBS. Eseguire la colorazione per ogni lignaggio come segue.
- Per il lignaggio degli adipociti, aggiungere la soluzione di permeabilizzazione dal kit e incubare la piastra per 5 minuti a temperatura ambiente. Preparare e aggiungere 1 mL di soluzione di lavoro Oil red O e conservarlo per 10 min. Rimuovere la macchia e fare cinque lavaggi con acqua distillata.
- Per il lignaggio degli osteociti, aggiungere il 5% di nitrato d'argento appena preparato (in acqua distillata) a ciascun pozzetto e tenere la piastra sotto UV per 1 ora. Rimuovere la soluzione e aggiungere il 2,5% di tiosolfato di sodio per rimuovere l'argento non reagito; Conservare per 5 min. Rimuovere la macchia, lavare due volte con acqua distillata e osservare le cellule macchiate al microscopio.
- Per le colture 3D, raccogliere il pellet dopo la fine del periodo di differenziazione di cui al punto 3.4.2.3 e ottenere criosezioni del tessuto differenziato sotto forma di pellet. Lasciare asciugare i vetrini all'aria e a temperatura ambiente prima di procedere alla colorazione.
- Per il lignaggio dei condrociti, seguire le indicazioni del kit di colorazione per aggiungere un volume sufficiente di soluzione di lavaggio, rimuoverla, aggiungere la soluzione fissante e incubare per 30 minuti. Lavare con acqua distillata, aggiungere la soluzione colorante e incubare per 30 minuti. Lavare tre volte con acido cloridrico 0,1 N; Aggiungere acqua distillata per neutralizzare l'acidità. Osservare le cellule colorate al microscopio in campo chiaro.
- Per le colture 2D, utilizzare le piastre MSC differenziate (test) e indifferenziate (controllo) (dal punto 3.4.1.5.) per la colorazione e, per prima cosa, rimuovere il terreno e lavare due volte con PBS. Fissare le cellule con PFA al 4% per 30 minuti a temperatura ambiente, rimuovere il surnatante e lavare una volta con PBS. Eseguire la colorazione per ogni lignaggio come segue.
4. Colorazione della superficie cellulare per l'immunofenotipizzazione
NOTA: Le piastre di coltura cellulare consigliate per ottenere un numero ottimale di cellule nei passaggi 4.2-4.5 sono piastre da 100 mm o matracci T75.
- Preparazione delle cellule per esperimenti di citofluorimetria
- Tripsinizzare e raccogliere le cellule da un piatto/matraccio confluente e centrifugare a 300 × g per 6 minuti per ottenere il pellet cellulare.
- Risospendere il pellet in 1 mL di terreno e determinare la conta delle cellule vitali utilizzando un emocitometro seguendo il metodo di esclusione del blu di tripano.
- Centrifugare nuovamente la sospensione cellulare dopo il conteggio e somministrare altri due lavaggi al pellet con 1 mL di tampone colorante.
- Scartare il surnatante e infine risospendere il pellet in un volume appropriato del tampone colorante a seconda del protocollo (vedere i passaggi da 4.2 a 4.5).
- Preparazione dei controlli di compensazione
- Prendi sette provette FACS ed etichettale come Unstained, DAPI, V450, FITC, PE, PerCP-Cy 5.5 e APC.
- Risospendere il pellet finale nel tampone di colorazione (vedere il punto 4.1.) mantenendo una densità cellulare di 0,5 × 10,6 cellule per 50 μL per provetta per le provette non colorate e DAPI.
- Preparare le provette monocoloranti per la compensazione come descritto nella Tabella 4.
- Dopo la preparazione, agitare delicatamente ogni provetta e incubare al buio per 30 minuti.
- Dopo l'incubazione, effettuare due lavaggi aggiungendo 1 mL di tampone colorante a ciascuna provetta, seguiti da un breve vortex e centrifugazione a 200 g per 10 minuti a temperatura ambiente.
- Scartare il surnatante, risospendere il pellet in 500 μL di tampone colorante e metterlo da parte fino all'acquisizione.
- Per il tubo DAPI, eseguire un trattamento a shock termico incubandolo a bagnomaria a 60 °C per 5 minuti seguiti da 15 minuti su ghiaccio. Aggiungere 5 μL di DAPI alla sospensione e tenerla al buio fino all'acquisizione della corsa.
- Preparazione dei controlli di fluorescenza meno uno (FMO)
- Risospendere il pellet finale nel tampone colorante (vedere il punto 4.1) mantenendo una densità cellulare di 0,5 × 106 cellule per 50 μL per ciascuna provetta.
- Prendi cinque provette FACS ed etichettale come isotipo CD44-V450, CD90-FITC, CD45-PE, isotipo CD73-PerCP Cy5.5 e isotipo CD105-APC.
- Preparare la sospensione cellulare e anticorpale secondo la Tabella 5.
- Agitare delicatamente le provette e incubarle per 30 minuti a temperatura ambiente al buio.
- Dopo l'incubazione, somministrare due lavaggi con 1 mL di tampone colorante a ciascuna provetta, seguiti da un breve vortex e centrifugazione a 200 g per 10 minuti a temperatura ambiente.
- Scartare il surnatante, risospendere il pellet in 500 μL di tampone colorante e metterlo da parte fino all'acquisizione.
- Preparazione dei campioni per l'analisi
- Risospendere il pellet finale nel tampone di colorazione (vedere il passaggio 4.1) mantenendo una densità cellulare di 0,5 × 106 cellule per 50 μL per ciascuna provetta.
- Prendi due provette FACS ed etichettale come provette miste 1 e 2.
- Preparare la sospensione cellulare e anticorpale secondo la Tabella 6.
- Agitare delicatamente le provette e incubarle per 30 minuti a temperatura ambiente al buio.
- Dopo l'incubazione, somministrare due lavaggi con 1 mL di tampone colorante a ciascuna provetta, seguiti da un breve vortex e centrifugazione a 200 g per 10 minuti a temperatura ambiente.
- Scartare il surnatante, risospendere il pellet in 500 μL di tampone colorante e mettere da parte fino all'acquisizione.
- Preparazione dei campioni per la selezione di singole cellule
- Prendi due provette FACS, aggiungi 1 ml di FBS e arrotola la provetta fino a formare uno strato uniforme di FBS all'interno di ciascuna provetta. Incubare per 1-2 ore durante l'elaborazione del campione per la colorazione. Etichettare queste provette come provette miste 1 e 2.
- Risospendere il pellet finale nel tampone di colorazione (vedere il punto 4.1) mantenendo una densità cellulare di 2-3 × 10,6 cellule per 50 μL per ciascuna provetta.
- Preparare la sospensione cellulare e anticorpale in provette miste 1 e 2 secondo la Tabella 7.
- Agitare delicatamente le provette e incubarle per 30 minuti a temperatura ambiente al buio.
- Dopo l'incubazione, somministrare due lavaggi con 1 mL di tampone colorante a ciascuna provetta, seguiti da un breve vortex e centrifugazione a 200 g per 10 minuti a temperatura ambiente.
- Scartare il surnatante, risospendere il pellet in 500 μL di tampone colorante e mettere da parte. Aggiungere 5 μL di DAPI 15 minuti prima della cernita.
NOTA: Si noti che per gli esperimenti di smistamento si consiglia una maggiore densità cellulare per provetta.
| Tipo di tubo | Perline Comp Positive* | Perline Comp Negative* | Cellule | Anticorpo aggiunto | |
| Tubo FITC | 1 goccia | 1 goccia | – | CD90-FITC anti-umano (2 μL) | |
| Tubo V450 | 1 goccia | 1 goccia | – | CD44-V450 anti-umano (2 μL) | |
| Tubo PerCP-Cy 5.5 | 1 goccia | 1 goccia | – | CD73-PerCP anti-umano Cy 5,5 (2 μL) | |
| Tubo in PE | 1 goccia | 1 goccia | – | CD45-PE anti-umano (2 μL) | |
| Tubo APC | 1 goccia | 1 goccia | – | CD105-APC anti-umano (2 μL) | |
| Tubo DAPI | – | – | 50 μL | – | |
| Tubo non colorato | – | – | 50 μL | – | |
| *1 goccia = 60 μL di sospensione del tallone | |||||
Tabella 4: Campioni di controllo della compensazione. Abbreviazioni: Comp = compensazione; DAPI = 4',6-diamidino-2-fenilindolo; FITC = isotiocianato di fluoresceina; APC = alloficocianina; PE = ficoeritrina; PerCP = proteina peridinina-clorofilla.
| TIPO DI TUBO | ANTICORPO CONTRO IL MARCATORE POSITIVO (2 μL) | ANTICORPO CONTRO IL MARCATORE NEGATIVO (2 μL) | AGGIUNTA DI ANTICORPI ISOTIPICI (2 μL) | VOLUME TOTALE DEGLI ANTICORPI | VOLUME DI SOSPENSIONE CELLULARE AGGIUNTO | VOLUME DI TAMPONE COLORANTE AGGIUNTO | ||
| Tubo FMO CD90-FITC | - Anti-umano CD44-V450 | CD45-PE anti-umano | Isotipo FITC IgG1 | 10 μL | 50 μL | 40 μL | ||
| - CD73 anti-umano PerCP Cy 5.5 | ||||||||
| - CD105-APC anti-umano | ||||||||
| CD73-PerCP Valvola FMO Cy5.5 | - Anti-umano CD44-V450 | CD45-PE anti-umano | Isotipo PerCP Cy 5.5 IgG1 | 10 μL | 50 μL | 40 μL | ||
| - CD90-FITC anti-umano | ||||||||
| - CD105-APC anti-umano | ||||||||
| Tubo FMO CD44-V450 | - CD90-FITC anti-umano | CD45-PE anti-umano | Isotipo V450 IgG1 | 10 μL | 50 μL | 40 μL | ||
| - CD73-PerCP anti-umano Cy 5.5 | ||||||||
| - CD105-APC anti-umano | ||||||||
| Tubo FMO CD105-APC | - Anti-umano CD44-V450 | CD45-PE anti-umano | Isotipo APC IgG1 | 10 μL | 50 μL | 40 μL | ||
| - CD90-FITC anti-umano | ||||||||
| - CD73-PerCP anti-umano Cy 5.5 | ||||||||
| Tubo FMO CD45-PE | - Anti-umano CD44-V450 | - | Isotipo PE IgG1 | 10 μL | 50 μL | 40 μL | ||
| - CD90-FITC anti-umano | ||||||||
| - CD73-PerCP anti-umano Cy 5.5 | ||||||||
| - CD105-APC anti-umano | ||||||||
Tabella 5: Campioni di controllo FMO. Abbreviazioni: FMO = fluorescenza meno uno; FITC = isotiocianato di fluoresceina; APC = alloficocianina; PE = ficoeritrina; PerCP = proteina peridinina-clorofilla.
| TIPO DI TUBO | ANTICORPO CONTRO IL MARCATORE POSITIVO (2 μL) | ANTICORPO CONTRO IL MARCATORE NEGATIVO (2 μL) | VOLUME TOTALE DEGLI ANTICORPI | VOLUME DI SOSPENSIONE CELLULARE AGGIUNTO | VOLUME DI TAMPONE COLORANTE AGGIUNTO | |
| Tubo misto 1 | - Anti-umano CD44-V450 | CD45-PE anti-umano | 10 μL | 50 μL | 40 μL | |
| - CD90-FITC anti-umano | ||||||
| - CD73-PerCP anti-umano Cy5.5 | ||||||
| - CD105-APC anti-umano | ||||||
| Tubo misto 2 | - Anti-umano CD44-V450 | CD45-PE anti-umano | 10 μL | 50 μL | 40 μL | |
| - CD90-FITC anti-umano | ||||||
| - CD73-PerCP anti-umano Cy5.5 | ||||||
| - CD105-APC anti-umano | ||||||
Tabella 6: Provette per campioni per l'immunofenotipizzazione multicolore di SHED. Abbreviazioni: SHEDs = cellule staminali da denti decidui esfoliati umani; PE = ficoeritrina.
| TIPO DI TUBO | ANTICORPO CONTRO MARCATORE POSITIVO (3 μL) | ANTICORPO CONTRO IL MARCATORE NEGATIVO (3 μL) | VOLUME TOTALE DEGLI ANTICORPI | VOLUME DI SOSPENSIONE CELLULARE AGGIUNTO | VOLUME DI TAMPONE COLORATO AGGIUNTO | |
| Tubo misto 1 | - CD90-FITC anti-umano | CD45-PE anti-umano | 9 μL | 50 μL | 41 μL | |
| - CD73-PerCP anti-umano Cy5.5 | ||||||
| Tubo misto 2 | - CD90-FITC anti-umano | CD45-PE anti-umano | 9 μL | 50 μL | 41 μL | |
| - CD73-PerCP anti-umano Cy5.5 | ||||||
Tabella 7: Provette di reazione per cernita a singola cellula. Abbreviazioni: FITC = isotiocianato di fluoresceina; PE = ficoeritrina; PerCP = proteina peridinina-clorofilla.
5. Selezione a cellula singola
- Preparazione del selezionatore cellulare
- Installare un ugello da 100 μm nello smistatore.
NOTA: L'ugello appropriato è almeno cinque volte il diametro della particella da selezionare. Il fluido di guaina da utilizzare per la cernita deve essere deciso in base al tipo di campione e alla sensibilità dell'esperimento; Per questo esperimento è stato utilizzato un fluido di guaina brevettato. - Eseguire il controllo di qualità (QC) giornaliero dello strumento e impostare il selezionatore per l'esperimento. Fare riferimento al manuale dello strumento per una guida dettagliata alla configurazione dello strumento.
- Installare un ugello da 100 μm nello smistatore.
- Impostazione della matrice di retribuzione
- Impostare la matrice di compensazione utilizzando tubi di compensazione a macchia singola dal punto 4.2.
- Nel software proprietario, selezionare Esperimento dalla barra degli strumenti e fare clic su Impostazione compensazione. Apri Crea controlli di compensazione.
- Controllare i marcatori e confermare. Viene aggiunto un nuovo campione denominato "Compensazione" in base al quale i nuovi tubi denominati controlli del marcatore vengono aggiunti automaticamente dal software.
- Seleziona il tubo non colorato ed eseguilo per registrare 5.000 eventi. Trascinare il gate sulla popolazione di celle e applicarlo a tutti i controlli di compensazione. Questo per impostare le tensioni e il gate negativo per ogni parametro fluorescente.
- Allo stesso modo, caricare separatamente i tubi di compensazione delle macchie singole, registrare e salvare i dati. Selezionare il cancello che delimita la popolazione di interesse e applicarlo a tutti i controlli di compensazione. In questo modo è possibile impostare le porte positive per ogni parametro fluorescente.
- Seleziona Esperimento dalla barra degli strumenti e fai clic sul collegamento Calcola valori di compensazione | e salva.
NOTA: Una volta generata la matrice auto-comp utilizzando il software, i parametri di tensione dei fluorocromi non possono essere modificati per nessuno dei canali nei tubi misti.
- Acquisizione dati
- Registrare 10.000 cellule in ogni provetta dal punto 4.3. e 4.4 per raccogliere dati per l'analisi dell'immunofenotipo delle cellule.
- Preparazione dei dispositivi di raccolta
- A seconda dello scopo delle popolazioni cellulari ordinate, scegliere tra piastre a 6, 24 pozzetti, 48 pozzetti o 96 pozzetti.
- Rivestire i pozzetti con 200-500 μL di FBS e mantenere le piastre indisturbate per 2 ore.
- Dopo 2 ore, rimuovere l'FBS residuo e aggiungere 200-500 μL di terreno di coltura MSC al 10%.
- Ordinamento delle celle in modalità di ordinamento a cella singola
- Eseguire il canale misto 1 (dal passaggio 4.5) e registrare 10.000 eventi per impostare i gate sulla popolazione di interesse da ordinare, utilizzando la strategia di gating appropriata.
- Caricare una piastra di raccolta e impostare il numero di cellule target tra 2.500 e 5.000 cellule/pozzetto e selezionare la maschera di purezza per l'ordinamento a cellula singola.
- Raccogli le popolazioni ordinate nel dispositivo di raccolta e tienile sul ghiaccio fino alla fine dell'esperimento di smistamento.
- Una volta terminato, trasferire le piastre nell'incubatore al 5% di CO2 per mantenere le colture a 37 °C.
NOTA: Dopo l'acquisizione, i file di dati grezzi sono stati esportati in formato .fcs (v.3.0. e successive). I report di ordinamento generati dopo ogni esperimento registravano il numero di eventi/celle ordinati per ogni pozzetto assegnato e indicavano il numero di conflitti che erano stati interrotti.
Risultati
Gli SHED sono stati caratterizzati con saggi di immunofluorescenza standard che mostrano l'espressione di vimentina (filamenti intermedi rossi di tipo III), filamenti di actina (Alexa fluor 488 Phalloidin Probes) e nuclei colorati con DAPI (Figura 1A). Per stimare le loro capacità proliferative e di formazione di colonie, sono stati eseguiti saggi standard di crescita cellulare a breve termine. Nella Figura 1B è stato mostrato un aumento di 14,3 volte del tass...
Discussione
Nel campo dell'ingegneria tissutale e della medicina rigenerativa, tra le fonti postnatali, le MSC orali derivate da tessuto hanno suscitato un profondo interesse a causa dei loro obblighi etici minimi e del notevole potenziale di differenziazione multilignaggio21. Le cellule staminali della polpa dentale (DPSC) del terzo molare impattato e le SHED hanno attirato la maggior parte dell'attenzione tra le MSC dentali per il loro potenziale terapeutico nelle malattie neurodegenerative e traumatiche
Divulgazioni
Gli autori dichiarano che non vi è alcun conflitto di interessi per quanto riguarda la pubblicazione di questo articolo.
Riconoscimenti
Ringraziamo la Flow Cell Facility del Jawaharlal Nehru Centre for Advanced Scientific Research, Bengaluru, India, per l'utilizzo della struttura centrale della citometria a flusso. La crio-sezione della coltura di pellet di cellule differenziate è stata eseguita presso il Neuberg Anand Reference Laboratory, Bengaluru, India. Questo lavoro è stato sostenuto dal finanziamento intramurale dell'UC da parte della Manipal Academy of Higher Education (MAHE), in India. AG è grata per il supporto della borsa di studio Dr. T. M. A. Pai di MAHE.
Materiali
| Name | Company | Catalog Number | Comments |
| Alcian Blue Stain | HiMedia | CCK029-1KT | |
| Antibiotic-Antimycotic (100x) | Gibco by ThermoFisher | 15240062 | |
| BD CompBead Plus Anti-Mouse Ig, κ/Negative Control (BSA) Compensation Plus (7.5 µm) Particles Set | BD Biosciences | 560497 | |
| BD FACS Accudrop Beads | BD Biosciences | 345249 | Used to set up the Laser delay when the sort module opens. |
| BD FACS Aria Fusion Flow cytometer | BD Biosciences | --- | |
| BD FACS Diva 9.4 | BD Biosciences | --- | |
| BD FACS Sheath Fluid | BD Biosciences | 342003 | Used as sheath fluid for both analysis and sorting experiments in the BD FACSAria Fusion |
| BD FACSDiva CS&T Research Beads | BD Biosciences | 655050 | Used for Instrument configuration depending on the nozzle size. |
| BD Horizon V450 Mouse Anti-Human CD44 | BD Biosciences | 561292 | |
| BD Horizon V450 Mouse IgG2b, κ Isotype Control | BD Biosciences | 560374 | CD44-V450 isotype |
| BD Pharmingen APC Mouse Anti-Human CD105 | BD Biosciences | 562408 | |
| BD Pharmingen APC Mouse IgG1, κ Isotype Control | BD Biosciences | 555751 | CD105-APC isotype |
| BD Pharmingen DAPI Solution | BD Biosciences | 564907 | DAPI Stock solution of 1 mg/mL |
| BD Pharmingen FITC Mouse Anti-Human CD90 | BD Biosciences | 555595 | |
| BD Pharmingen FITC Mouse IgG1, κ Isotype Control | BD Biosciences | 555748 | CD90-FITC isotype |
| BD Pharmingen PE Mouse Anti-Human CD45 | BD Biosciences | 555483 | |
| BD Pharmingen PE Mouse IgG1, κ Isotype Control | BD Biosciences | 555749 | CD45-PE isotype |
| BD Pharmingen PerCP-Cy 5.5 Mouse Anti-Human CD73 | BD Biosciences | 561260 | |
| BD Pharmingen PerCP-Cy 5.5 Mouse IgG1, κ Isotype Control | BD Biosciences | 550795 | CD73-PerCP-Cy 5.5 isotype |
| BD Pharmingen Purified Mouse Anti-Vimentin | BD Biosciences | 550513 | |
| Bovine serum albumin | Hi-Media | TC548-5G | |
| Crystal violet | Nice chemical pvt ltd | C33809 | |
| Dulbecco's Phosphate Buffered Saline | Sigma-aldrich | D5652-50L | dPBS used for culture work and maintenance. |
| Ethanol | --- | --- | Used for general sterlization. |
| Fetal Bovine Serum | Gibco by ThermoFisher | 10270-106 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 555 | ThermoFisher Scientific | A-21422 | |
| KO-DMEM | Gibco by ThermoFisher | 10829018 | Basal medium for undifferentiated hESCs, used in the preparation of culture media |
| L-Glutamine 200mM (100x) | Gibco by ThermoFisher | 25030-081 | |
| Methanol, for Molecular Biology | Hi-Media | MB113 | |
| Oil red O | HiMedia | CCK013-1KT | |
| Paraformaldehyde | loba chemie | 30525-89-4 | |
| Penicillin Streptomycin (100x) | Gibco by ThermoFisher | 15140- 122 | |
| Phalloidin (ActinGreen 488 ReadyProbes reagent) | Invitrogen | R37110 | |
| Silver Nitrate | HiMedia | MB156-25G | |
| Sodium Thiosulphate pentahydrate | Chemport | 10102-17-7 | |
| Sphero Rainbow Fluorescent Particles, 3.0 - 3.4 µm | BD Biosciences | 556291 | |
| Staining buffer | Prepared in MIRM | ---- | It was prepared using 2% FBS in PBS |
| StemPro Adipogenesis Differentiation Basal Media | Gibco by ThermoFisher | A10410-01 | Basal media for Adipogenic media |
| StemPro Adipogenesis Supplement | Gibco by ThermoFisher | A10065-01 | Induction media for Adipogenic media |
| StemPro Chondrogenesis Supplement | Gibco by ThermoFisher | A10064-01 | Induction media for Chondrogenic media |
| StemPro Osteogenesis Supplement | Gibco by ThermoFisher | A10066-01 | Induction media for Osteoogenic media |
| StemPro Osteogenesis/Chondrogenesis Differentiation Basal Media | Gibco by ThermoFisher | A10069-01 | Basal media for both Ostegenic and Chondrogenic media |
| Triton-X-100 | Hi-Media | MB031 | |
| Trypan Blue | Gibco by life technologies | 15250-061 | |
| Trypsin - EDTA Solution 1x | Hi-media | TCL049 | |
| Tween-20 | MERCK | 9005-64-5 |
Riferimenti
- Kobolak, J., Dinnyes, A., Memic, A., Khademhosseini, A., Mobasheri, A. Mesenchymal stem cells: Identification, phenotypic characterization, biological properties and potential for regenerative medicine through biomaterial micro-engineering of their niche. Methods. 99, 62-68 (2016).
- Wilson, A., Hodgson-Garms, M., Frith, J. E., Genever, P. Multiplicity of mesenchymal stromal cells: finding the right route to therapy. Frontiers in Immunology. 10, 1112 (2019).
- Li, J., et al. Comparison of the biological characteristics of human mesenchymal stem cells derived from exfoliated deciduous teeth, bone marrow, gingival tissue, and umbilical cord. Molecular Medicine Reports. 18 (6), 4969-4977 (2018).
- McLeod, C. M., Mauck, R. L. On the origin and impact of mesenchymal stem cell heterogeneity: new insights and emerging tools for single cell analysis. European Cells & Materials. 34, 217-231 (2017).
- Dominici, M., et al. Minimal criteria for defining multipotent mesenchymal stromal cells. The International Society for Cellular Therapy position statement. Cytotherapy. 8 (4), 315-317 (2006).
- Wang, J., Liao, L., Wang, S., Tan, J. Cell therapy with autologous mesenchymal stem cells-how the disease process impacts clinical considerations. Cytotherapy. 15 (8), 893-904 (2013).
- Kern, S., Eichler, H., Stoeve, J., Kluter, H., Bieback, K. Comparative analysis of mesenchymal stem cells from bone marrow, umbilical cord blood, or adipose tissue. Stem Cells. 24 (5), 1294-1301 (2006).
- Dunn, C. M., Kameishi, S., Grainger, D. W., Okano, T. Strategies to address mesenchymal stem/stromal cell heterogeneity in immunomodulatory profiles to improve cell-based therapies. Acta Biomaterialia. 133, 114-125 (2021).
- Yang, Y. K., Ogando, C. R., Wang See, C., Chang, T. Y., Barabino, G. A. Changes in phenotype and differentiation potential of human mesenchymal stem cells aging in vitro. Stem Cell Research & Therapy. 9 (1), 131 (2018).
- Costa, L. A., et al. Functional heterogeneity of mesenchymal stem cells from natural niches to culture conditions: implications for further clinical uses. Cellular and Molecular Life Sciences. 78 (2), 447-467 (2021).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Review of Scientific Instruments. 43 (3), 404-409 (1972).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
- Liao, X., Makris, M., Luo, X. M. Fluorescence-activated cell sorting for purification of plasmacytoid dendritic cells from the mouse bone marrow. Journal of Visualized Experiments. (117), (2016).
- Roda, B., et al. A novel stem cell tag-less sorting method. Stem Cell Reviews and Reports. 5 (4), 420-427 (2009).
- Hall, S. R., et al. Identification and isolation of small CD44-negative mesenchymal stem/progenitor cells from human bone marrow using elutriation and polychromatic flow cytometry. Stem Cells Translational Medicine. 2 (8), 567-578 (2013).
- Eggleton, M. J., Sharp, A. A. Platelet counting using the Coulter electronic counter. Journal of Clinical Pathology. 16 (2), 164-167 (1963).
- Porwit-Ksiazek, A., Aman, P., Ksiazek, T., Biberfeld, P. Leu 7+ (HNK-1+) cells. II. Characterization of blood Leu 7+ cells with respect to immunophenotype and cell density. Scandinavian Journal of Immunology. 18 (6), 495-449 (1983).
- Hewitt, Z., et al. Fluorescence-activated single cell sorting of human embryonic stem cells. Cloning and Stem Cells. 8 (3), 225-234 (2006).
- Singh, A. M. An efficient protocol for single-cell cloning human pluripotent stem cells. Frontiers in Cell and Developmental Biology. 7, 11 (2019).
- Wilson, N. K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Zhou, L. L., et al. Oral mesenchymal stem/progenitor cells: the immunomodulatory masters. Stem Cells Inernational. 2020, 1327405 (2020).
- Fawzy El-Sayed, K. M., et al. Adult mesenchymal stem cells explored in the dental field. Advances in Biochemical Engineering/Biotechnology. 130, 89-103 (2013).
- Hardy, W. R., et al. Transcriptional networks in single perivascular cells sorted from human adipose tissue reveal a hierarchy of mesenchymal stem cells. Stem Cells. 35 (5), 1273-1289 (2017).
- . . FACSAria Fusion User's Guide. , (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon