Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Einzelzellsortierung von immunphänotypisierten mesenchymalen Stammzellen aus humanen abgeblätterten Milchzähnen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Verwendung der fluoreszenzaktivierten Zellsortierung von humanen mesenchymalen Stammzellen unter Verwendung der Einzelzellsortierungsmethode. Insbesondere kann durch den Einsatz der Einzelzellsortierung eine Reinheit von 99 % der immunphänotypisierten Zellen aus einer heterogenen Population erreicht werden, wenn sie mit einem multiparametrischen Durchflusszytometrie-basierten Ansatz kombiniert wird.
Zusammenfassung
Die mesenchymalen Stammzellen (MSCs) eines Organismus besitzen eine außergewöhnliche Fähigkeit, sich in mehrere Linien adulter Zellen im Körper zu differenzieren und sind für ihre immunmodulatorischen und entzündungshemmenden Eigenschaften bekannt. Die Verwendung dieser Stammzellen ist ein Segen für das Gebiet der regenerativen Biologie, aber gleichzeitig ein Fluch für die regenerative Medizin und Therapeutika, da sie mit zahlreichen zellulären Mehrdeutigkeiten verbunden sind. Diese Mehrdeutigkeiten können sich aus der Vielfalt der Quelle dieser Stammzellen und aus ihren In-vitro-Wachstumsbedingungen ergeben, die beide ihre funktionelle Heterogenität widerspiegeln.
Dies erfordert Methoden, um gereinigte, homogene Populationen von MSCs für therapeutische Anwendungen bereitzustellen. Fortschritte auf dem Gebiet der Durchflusszytometrie haben den Nachweis von Einzelzellpopulationen mit einem multiparametrischen Ansatz ermöglicht. Dieses Protokoll beschreibt einen Weg zur Identifizierung und Reinigung von Stammzellen aus menschlichen exfolierten Milchzähnen (SHEDs) durch fluoreszenzgestützte Einzelzellsortierung. Die gleichzeitige Expression von Oberflächenmarkern, nämlich CD90-Fluoresceinisothiocyanat (FITC), CD73-Peridin-Chlorophyll-Protein (PerCP-Cy5.5), CD105-Allophycocyanin (APC) und CD44-V450, identifizierte die "hellen", positiven Exprimatoren von MSCs mittels multiparametrischer Durchflusszytometrie. Es wurde jedoch ein signifikanter Rückgang des Prozentsatzes der vierfachen Exprimatoren dieser positiven Marker von Passage 7 bis zu den späteren Passagen beobachtet.
Die immunphänotypisierten Subpopulationen wurden im Einzelzell-Sortiermodus sortiert, wobei nur zwei positive und ein negativer Marker die Einschlusskriterien darstellten. Diese Methodik stellte die Zelllebensfähigkeit der sortierten Populationen sicher und hielt die Zellproliferation nach der Sortierung aufrecht. Die nachgeschaltete Anwendung für eine solche Sortierung kann verwendet werden, um die linienspezifische Differenzierung für die abgegrenzten Teilpopulationen zu evaluieren. Dieser Ansatz kann auf andere Einzelzellsysteme angewendet werden, um die Isolationsbedingungen zu verbessern und Informationen über mehrere Zelloberflächenmarker zu erhalten.
Einleitung
Mesenchymale Stammzellen (MSCs) können als skalierbare Zellquelle angesehen werden, die für zellbasierte Therapien geeignet ist, und können als Goldstandardsystem in der regenerativen Medizin angesehen werden. Diese Zellen können aus einer Vielzahl von Quellen im Körper mit unterschiedlicher Gewebeherkunft isoliert werden1. Abhängig von ihrem Ausgangsgewebe zeigt jede Art von MSC ein mehrdeutiges In-vitro-Verhalten 2. Dies zeigt sich gut in ihren morphologischen und funktionellen Eigenschaften3. Mehrere Studien haben intraklonale Variationen in den Dimensionen gezeigt, einschließlich der Differenzierung von adultem Gewebe, des genomischen Zustands sowie der metabolischen und zellulären Architektur von MSCs 2,4.
Die Immunphänotypisierung von Zellen ist eine gängige Anwendung der Durchflusszytometrie zur Identifizierung von Stammzellen und wurde 2006 von der Internationalen Gesellschaft für Zell- und Gentherapie (ISCT) genutzt, um eine Liste von Mindestkriterien zur Identifizierung von Zellen als MSCs vorzuschreiben. Darin heißt es, dass neben der plastischen Adhärenz und der Fähigkeit, sich in drei Linien (osteogen, chondrogen und adipogen) in vitro zu differenzieren, ≥95 % der Zellpopulation CD105, CD73 und CD90 exprimieren müssen, und diesen Zellen muss die Expression (≤2 % positiv) von CD34, CD45, CD11b, CD14 und HLA-DR fehlen, gemessen durch Durchflusszytometrie5. Obwohl die MSCs durch eine Reihe von Biomarkern nach den Minimalkriterien des ISCT definiert wurden, konnten ihre Immuneigenschaften nicht mit diesen Biomarkern verglichen werden, und es bestand ein Bedarf an weiteren über diese Kriterien hinausgehenden Kriterien, um studienübergreifende Vergleiche und klonale Variationen leichter quantifizierbar zu machen2.
Trotz der vom ISCT festgelegten Richtlinien haben umfangreiche Forschungen zu MSCs gezeigt, dass in dieser Population Heterogenität besteht, die auf eine Vielzahl von Faktoren zurückzuführen sein könnte, hauptsächlich aufgrund der Allgegenwart der Heterogenität, die zwischen MSC-Spendern6, Gewebequellen7, einzelnen Zellen innerhalb einer klonalen Population8 und den Kulturbedingungen 2,9 entsteht. 10. Anmelden Die Charakterisierung und Reinigung dieser Primärzellen aus einer Vielzahl von Gewebequellen, um die Qualität und das Zellschicksal sicherzustellen, sind wichtige Schritte bei ihrer Herstellung. Die Notwendigkeit, die angezeigten Variationen innerhalb der Grundgesamtheit zu verstehen, erfordert eine effiziente Methode, um sie in Teilpopulationen aufzulösen, die unterteilt und separat gesammelt werden können11. Analysen auf Einzelzellebene tragen dazu bei, die Herausforderungen der Zell-Zell-Variation zu überwinden, das biologische Rauschen zu reduzieren, das durch eine heterogene Population entsteht, und bieten die Möglichkeit, seltene Zellen zu untersuchen und zu charakterisieren12.
Basierend auf dem Zweck und den gewählten Parametern können verschiedene Methoden angewendet werden, um die ausgewählten Populationen zu sortieren und anzureichern. Zellsortiertechniken können sowohl Massensortier- als auch Einzelzellensortierverfahren umfassen. Während die Massensortierung Zielpopulationen durch magnetisch aktivierte Zellsortierung (MACS)13, Fraktionierung 14 und Elutriation15 anreichern kann, kann die Einzelzellsortierung homogenere Populationen mittels fluoreszenzaktivierter Zellsortierung (FACS)11 anreichern. Eine vergleichende Analyse jeder dieser Methoden mit ihren eigenen Vor- und Nachteilen ist in Tabelle 1 dargestellt.
Tabelle 1: Vergleichende Analysen verschiedener Techniken: MACS, Fraktionierung, Elutriation und FACS, wobei die Unterschiede in ihrem Prinzip und die Vor- und Nachteile der Wahl einer bestimmten Technik gegenüber einer anderen hervorgehoben werden. Abkürzungen: MACS = Magnetisch aktivierte Zellsortierung; FACS = Fluoreszenz-aktivierte Zellsortierung. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Seit dem Aufkommen der Technik spielt die Einzelzell-Durchflusszytometrie eine wichtige Rolle bei der Zählung16, dem Nachweis und der Charakterisierung einer spezifischen Zellpopulation in einer heterogenen Probe17. Hewitt et al. legten 2006 den Grundstein für eine automatisierte Zellsortierungsmethode, um die Isolierung homogener Pools differenzierter humaner embryonaler Stammzellen (hESCs) zu verbessern18. Die Einzelzellsortierung reicherte die Population der GFP-transduzierten hES-Zellen an und erleichterte die Isolierung genetisch veränderter Klone, was eine neue Dimension in der klinischen Forschung eröffnete. Um die Sortiereffizienz zu verbessern, wurden im Allgemeinen zwei Ansätze gewählt; Entweder werden die Sammelmedien der sortierten Populationen modifiziert, um die Lebensfähigkeit und Proliferation der nachsortierten Zellen19 aufrechtzuerhalten, oder der Zellsortierungsalgorithmus/die Zellsortierungssoftware wird entsprechend modifiziert12.
Mit dem technologischen Fortschritt konnten kommerzielle Durchflusszytometer und Zellsortierer dazu beitragen, Herausforderungen zu bewältigen, die bei der aseptischen Sortierung fragiler und seltener Zellpopulationen, insbesondere Stammzellen unterschiedlicher Herkunft, bewältigt wurden. Eine der größten Herausforderungen für Stammzellbiologen war die klonale Isolierung menschlicher pluripotenter Stammzellen nach Transfektionsprotokollen, die in Gen-Editing-Studien erforderlich sind19. Dies wurde durch die Sortierung einzelner Zellen in 96-Well-Platten gelöst, die mit embryonalen Fibroblasten (MEFs) der Maus zusammen mit Nahrungsergänzungsmitteln und kommerziellen niedermolekularen ROCK-Inhibitoren beschichtet wurden. Die Strategien zur Zellisolierung konnten jedoch durch die Verwendung der Indexsortierung, einer Funktion des Sortieralgorithmus, die den Immunphänotyp einzelner sortierter Zellen identifiziert, weitgehend verfeinert werden12. Diese verfeinerte Modalität in der Einzelzellsortierung trug nicht nur dazu bei, die Sortiereffizienz von Stammzellen zu verbessern, insbesondere im Hinblick auf seltene hämatopoetische Stammzellpopulationen, sondern verknüpfte auch Einzelzellklone effizient mit ihren nachgeschalteten funktionellen Assays20.
Diese Arbeit konzentriert sich auf die Einzelzellsortierung von immunphänotypisierten Stammzellen aus menschlichen exfolierten Milchzähnen (SHEDs) zur Anreicherung von Subpopulationen, um ihre funktionellen Differenzierungsfähigkeiten zu untersuchen. Mit einer Kombination aus zwei MSC-positiven Markern, CD90 und CD73, und einem negativen hämatopoetischen Marker CD45 wurden die MSCs immunphänotypisiert und die Dim- und Null-Expressionoren identifiziert. Basierend auf ihrem Immunphänotyp wurden die Subpopulationen als reine MSCs, einfach positive und doppelt negative Populationen identifiziert. Sie wurden im Einzelzell-Sortiermodus sortiert, um reine und angereicherte Subpopulationen für weitere funktionelle Studien zu erhalten, um festzustellen, ob die differentielle Expression von Markern ein Artefakt der In-vitro-Kulturbedingungen ist oder ob sie auch einen Einfluss auf die funktionellen Eigenschaften hat. Zellen, die keine homogenen Exprimatoren der "positiven MSC-Marker" waren, wurden sortiert, um ihre funktionellen Eigenschaften zu untersuchen.
Protokoll
Ethische Genehmigung und Zustimmung zur Teilnahme: Menschliche Proben von gefolterter Zahnpulpa wurden nach Einholung der Einverständniserklärung und der vollständigen ethischen Genehmigung durch die Mund-, Kiefer- und Gesichtsabteilung des Sri Rajiv Gandhi Dental College and Hospital (SRGCDS), Bengaluru, in Übereinstimmung mit den vom Hospital Ethical Clearance Committee, SRGCDS, festgelegten Standards erhalten. Danach wurden Isolierung, Kultur, Aufrechterhaltung und Anwendung von SHEDs genehmigt und in Übereinstimmung mit den vom Institutional Committee for Stem Cell Research (IC-SCR) am Manipal Institute of Regenerative Medicine, MAHE - Bengaluru empfohlenen Richtlinien genehmigt. In der Materialtabelle finden Sie Einzelheiten zu allen Materialien und Reagenzien, die in diesem Protokoll verwendet werden.
1. Herstellung von Reagenzien und Puffern
- Für die Kulturpflege
- Bereiten Sie MSC-Zellkulturmedien (10 %) unter Verwendung von Basalmedium für undifferenzierte hES-Zellen, 10 % fötales Kälberserum (FBS), 1 % L-Glutamin und 1 % Penicillin-Streptomycin (Pen-Streptokokken) vor (Tabelle 2).
- Bereiten Sie MSC-Zellkulturmedien (20 %) unter Verwendung von Basalmedium für undifferenzierte hES-Werte, 20 % FBS, 1 % L-Glutamin und 1 % Pen-Streptokokken vor (Tabelle 2).
- Neutralisierendes Medium mit Basalmedium für undifferenzierte hES-Werte, 1 % L-Glutamin und 1 % Antibiotikum-Antimykotikum (Anti-Anti) herstellen (Tabelle 2).
- Zur durchflusszytometrischen Analyse und Sortierung
- Färbepuffer mit 2 % FBS in phosphatgepufferter Kochsalzlösung (PBS) herstellen.
- Bereiten Sie eine Stammlösung von 4′,6-Diamidino-2-phenylindol (DAPI) (1 μg/ml) vor, indem Sie 1 μl in 1 ml PBS zugeben
- Zur linienspezifischen Differenzierung von MSCs
- Differenzierungsmedien für die osteogene, chondrogene und adipogene Differenzierung gemäß der in Tabelle 3 beschriebenen Zusammensetzung herstellen. Lagern Sie die vorbereiteten Aliquots für die Dauer des Versuchs bei 4 °C.
- Bereiten Sie serumarme Medien (2 % Medien) unter Verwendung von Basalmedium für undifferenzierte hES-Werte, 2 % FBS, 1 % L-Glutamin und 1 % Pen-Streptokokken vor (Tabelle 2).
| ART DES MEDIUMS | ZWECK DER MEDIEN | ZUSAMMENSETZUNG FÜR 50 ml | ||||||||
| FBS | Pen-Streptokokken | L-Glutamin | BASALES MEDIUM FÜR UNDIFFERENZIERTE hES-Zellen | |||||||
| 10% Medien | MSC-Kultur und -Pflege | 5 ml | 500 μl | 500 μl | 44 ml | |||||
| 20% Medien | CFU-F-Assay | 10 ml | 500 μl | 500 μl | 39 ml | |||||
| Serumarme (2 %) Medien | Medien für Kontrollwellen im Differenzierungsprotokoll | 1 ml | 500 μl | 500 μl | 48 ml | |||||
| Neutralisierende Medien | Medien zur Neutralisierung der Zellsuspension nach der Trypsinisierung | - | 500 μl | 500 μl | 49 ml | |||||
Tabelle 2: Zellkulturmedien für die Kulturpflege und Assays. Abkürzungen: MSC = mesenchymale Stammzelle; CFU-F = koloniebildende Einheit-Fibroblasten.
| KOMPONENTEN | OSTEOGENE MEDIEN | CHONDROGENE MEDIEN | ADIPOGENE MEDIEN |
| Basales Medium | 90 ml | 90 ml | 90 ml |
| Induktionsmedien | 10 ml | 10 ml | 10 ml |
| Gesamtvolumen | 100 ml | 100 ml | 100 ml |
Tabelle 3: Differenzierungsmedien für die Trilineage-Differenzierung von SHEDs.
2. Kultur und Pflege von SHEDs
- Halten Sie die Zellen in 10%igen MSC-Nährmedien und führen Sie alle 2 Tage oder nach Bedarf einen Medienwechsel durch.
- Trypsinisierung von Zellen bei 95 % Konfluenz unter Verwendung von 0,25 % Trypsin-EDTA.
- Neutralisieren Sie die Zellen nach der Trypsinisierung mit neutralisierenden Medien.
- Zentrifugieren Sie das Röhrchen bei 300 × g für 6 Minuten bei Raumtemperatur, um ein Zellpellet zu erhalten.
- Dekantieren Sie den Überstand und resuspendieren Sie das Zellpellet in 10%igen MSC-Nährmedien.
- Besiedeln Sie Zellen in frisch zubereitete Zellkulturschalen, die 10 % MSC-Nährmedien enthalten, für weitere Experimente oder Subkulturen.
HINWEIS: Die optimale Aussaatdichte für SHEDs beträgt 0,2 × 10 6 Zellen in einer 100-mm-Schale und 0,8 × 10 6 Zellen in einem T-75-Kolben, um 1,5 × 10 6 Zellen bzw. 4 × 10 6 Zellen bei 90-100 % Konfluenz zu erhalten.
3. Charakterisierung von MSCs
- Kurzzeit-Zellwachstums-Assay
- Säen Sie 4 × 104 Zellen/Well in dreifacher Ausführung in 6-Well-Platten in 10%igen MSC-Nährmedien.
- Inkubieren Sie die Platten 7 Tage lang bei 37 °C und führen Sie alle 2 Tage einen Medienwechsel durch.
- Entnehmen Sie die Zellen an den Tagen 2, 4 und 8 mit 0,25 % Trypsin und waschen Sie sie mit Nährmedien.
- Zentrifugieren Sie die Zellen bei 300 g für 6 Minuten bei Raumtemperatur und resuspendieren Sie das Pellet in 1 ml Medium.
- Zählen Sie die Zellen mit einem Hämozytometer und bestimmen Sie ihre Lebensfähigkeit mit der Trypanblau-Ausschlussmethode.
- Berechnen Sie die Proliferationsrate mit Gleichung (1):
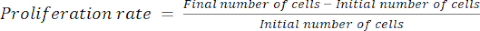 (1)
(1)
- Koloniebildender Unit-Fibroblasten-Assay (CFU-F)
- Säen Sie 10.000 Zellen in einer 100-mm-Schale und kultivieren Sie sie in 20%igen MSC-Nährmedien.
- Inkubieren Sie die Platten 14 Tage lang bei 37 °C und führen Sie alle 3 Tage einen Medienwechsel durch.
- Spülen Sie die Kolonien nach 14 Tagen mit PBS, fixieren Sie sie mit kristallviolettem Farbstoff in Methanol und spülen Sie erneut mit PBS, um den Restfleck zu entfernen.
- Zähle und bilde die Kolonien.
HINWEIS: Zählen Sie Kolonien mit >50 Zellen. Die koloniebildende Effizienz wird als koloniebildende Einheitszahlen berechnet.
- Immunfluoreszenz-Assay
- Säen Sie die MSCs auf 35-mm-Schalen und lassen Sie sie bis zu 80-90% Konfluenz wachsen.
- Entfernen Sie das Medium und spülen Sie das Geschirr einmal mit PBS ab.
- Fixieren Sie die Zellen mit 1 ml 4%igem Paraformaldehyd (PFA), indem Sie entweder 1 Stunde bei Raumtemperatur oder über Nacht bei 4 °C inkubieren.
- Nach der Fixierung die Vertiefungen mit PBST 3 x 5 min auf der Wippe waschen.
- Fügen Sie 0,3 % Triton X-100 in PBST (0,05 % Tween-20-Lösung in PBS) hinzu, um die Zellen zu permeabilisieren. Halten Sie es 15 Minuten lang bei Raumtemperatur auf der Wippe.
- Waschen Sie die Vertiefungen mit PBST für 3 x 5 min auf der Wippe.
- Fügen Sie 3 % Rinderserumalbumin (BSA) zum Blockieren hinzu und lassen Sie es 1 Stunde lang bei Raumtemperatur auf der Wippe.
- Waschen Sie die Vertiefungen mit PBST für 3 x 5 min auf der Wippe.
- Fügen Sie 800 μl einer 1:500-Verdünnung von Maus-Antivimentin hinzu, lassen Sie es 1 Stunde lang bei Raumtemperatur auf der Wippe und überführen Sie die Platte zur Inkubation über Nacht auf 4 °C.
- Entfernen Sie am nächsten Tag den primären Antikörper und waschen Sie die Vertiefungen mit PBST für 3 x 5 min auf der Wippe.
- 800 μl 1:1.000 Verdünnung des kreuzadsorbierten Ziegen-Anti-Maus-IgG (H+L)-Sekundärantikörpers Alexa Fluor 555 zugeben und 3 Stunden bei Raumtemperatur auf der Wippe aufbewahren.
- Waschen Sie die Vertiefungen mit PBST für 3 x 5 min auf der Wippe.
- Alexa fluor 488 Phalloidin-Sonden 240 μl in 1.000 μl PBS geben und bei Raumtemperatur 60 Minuten auf der Wippe inkubieren.
- Waschen Sie die Vertiefungen mit PBST für 3 x 5 min auf der Wippe.
- Fügen Sie 700 μl DAPI-Eindeckmittel hinzu und beobachten Sie die Zellen unter einem Mikroskop.
- Abstammungsspezifische Differenzierung
- Differenzierung nach 2D-Kultur
- Nehmen Sie zwei 48-Well-Platten und kennzeichnen Sie sie als osteogene bzw. adipogene Abstammungslinien.
- Säen Sie 15.000 Zellen/Well in vier Wells jeder Platte und kultivieren Sie sie in 10%igen MSC-Nährmedien.
- Sobald die Monoschicht der Zellen eine Konfluenz von 90 % erreicht hat, kennzeichnen Sie die ersten beiden Vertiefungen als "Kontrolle" und ersetzen Sie die vorhandenen Medien durch serumarme Medien (2 % Medien). In den letzten beiden Vertiefungen, die als "Test" gekennzeichnet sind, fügen Sie Differenzierungsmedien einer der beiden Abstammungslinien hinzu, adipogen oder osteogen. Markieren Sie dies als Tag 0.
- Tauschen Sie das Medium jeden dritten Tag vorsichtig aus, um ein Ablösen zu vermeiden, und achten Sie darauf, eine Kontamination zu vermeiden.
- Halte diese Bedingungen bis zum einundzwanzigsten Tag aufrecht; dann Prozess für das Färbeexperiment.
- Differenzierung nach 3D-Kultur
- Nehmen Sie zwei 15-ml-Röhrchen zur Durchführung einer chondrogenen Differenzierung mit einer 3D-Pelletkultur.
- 1 × 106 Zellen in jedes Röhrchen überführen und bei 300 × g 6 min zentrifugieren, um ein Pellet zu bilden. Beschriften Sie ein Röhrchen als "Kontrolle" und fügen Sie 10 % MSC-Nährmedien hinzu. Kennzeichnen Sie das andere Röhrchen als "Test" und fügen Sie chondrogene Differenzierungsmedien hinzu. Legen Sie die Röhrchen vorsichtig mit locker verschraubten Kappen in den Inkubator. Markieren Sie dies als Tag 0.
- Wechseln Sie das Medium jeden dritten Tag sorgfältig, um das Pellet während des Medienwechsels nicht zu lösen/aufzulösen.
- Halten Sie diese Bedingungen bis zum einundzwanzigsten Tag aufrecht und verarbeiten Sie dann die Zellen für weitere Experimente.
- Differenzierung nach 2D-Kultur
- Abstammungsspezifische zytochemische Färbung
- Verwenden Sie für 2D-Kulturen die differenzierten (Test) und undifferenzierten MSCs (Kontrollplatten) (aus Schritt 3.4.1.5.) für die Färbung, entfernen Sie zunächst das Medium und waschen Sie es zweimal mit PBS. Fixieren Sie die Zellen mit 4% PFA für 30 Minuten bei Raumtemperatur, entfernen Sie den Überstand und waschen Sie sie einmal mit PBS. Führen Sie die Färbung für jede Linie wie folgt durch.
- Für die Adipozyten-Abstammung fügen Sie Permeabilisierungslösung aus dem Kit hinzu und inkubieren Sie die Platte 5 Minuten lang bei Raumtemperatur. Bereiten Sie 1 ml Ölrot-O-Arbeitslösung vor und fügen Sie sie hinzu und bewahren Sie sie 10 Minuten lang auf. Entfernen Sie den Fleck und waschen Sie ihn fünf Mal mit destilliertem Wasser.
- Für die Osteozyten-Abstammung 5% frisch zubereitetes Silbernitrat (in destilliertem Wasser) in jede Vertiefung geben und die Platte 1 Stunde lang unter UV-Strahlung halten. Entfernen Sie die Lösung und fügen Sie 2,5% Natriumthiosulfat hinzu, um das nicht umgesetzte Silber zu entfernen. 5 Minuten einwirken lassen. Entfernen Sie den Fleck, waschen Sie ihn zweimal mit destilliertem Wasser und beobachten Sie die gefärbten Zellen unter dem Mikroskop.
- Bei 3D-Kulturen wird das Pellet nach dem Ende des Differenzierungszeitraums aus Schritt 3.4.2.3 entnommen und Kryoschnitte des differenzierten Gewebes in Form des Pellets erhalten. Lassen Sie die Objektträger an der Luft trocknen und haben sie Raumtemperatur, bevor Sie mit dem Färben fortfahren.
- Befolgen Sie für die Chondrozytenlinie die Anweisungen des Färbekits, um eine ausreichende Menge an Waschlösung hinzuzufügen, entfernen Sie sie, fügen Sie die Fixierlösung hinzu und inkubieren Sie 30 Minuten lang. Mit destilliertem Wasser waschen, die Färbelösung hinzufügen und 30 Minuten inkubieren. Dreimal mit 0,1 N Salzsäure waschen; Füge destilliertes Wasser hinzu, um die Säure zu neutralisieren. Beobachten Sie die gefärbten Zellen unter einem Hellfeldmikroskop.
- Verwenden Sie für 2D-Kulturen die differenzierten (Test) und undifferenzierten MSCs (Kontrollplatten) (aus Schritt 3.4.1.5.) für die Färbung, entfernen Sie zunächst das Medium und waschen Sie es zweimal mit PBS. Fixieren Sie die Zellen mit 4% PFA für 30 Minuten bei Raumtemperatur, entfernen Sie den Überstand und waschen Sie sie einmal mit PBS. Führen Sie die Färbung für jede Linie wie folgt durch.
4. Zelloberflächenfärbung zur Immunphänotypisierung
HINWEIS: Empfohlene Zellkulturplatten, um eine optimale Anzahl von Zellen in den Schritten 4.2-4.5 zu erhalten, sind 100-mm-Schalen oder T75-Kolben.
- Zellvorbereitung für durchflusszytometrische Experimente
- Trypsinisieren und sammeln Sie die Zellen aus einer konfluierenden Schale/einem Kolben und zentrifugieren Sie sie bei 300 × g für 6 Minuten, um das Zellpellet zu erhalten.
- Resuspendieren Sie das Pellet in 1 ml Medium und bestimmen Sie die Anzahl der lebensfähigen Zellen mit einem Hämozytometer nach der Trypanblau-Ausschlussmethode.
- Zentrifugieren Sie die Zellsuspension nach dem Zählen erneut und waschen Sie das Pellet zweimal mit 1 ml Färbepuffer.
- Verwerfen Sie den Überstand und resuspendieren Sie das Pellet schließlich in einem geeigneten Volumen des Färbepuffs je nach Protokoll (siehe Schritte 4.2 bis 4.5).
- Vorbereitung von Vergütungskontrollen
- Nehmen Sie sieben FACS-Röhrchen und kennzeichnen Sie sie als Unstained, DAPI, V450, FITC, PE, PerCP-Cy 5.5 und APC.
- Resuspendieren Sie das fertige Pellet im Färbepuffer (siehe Schritt 4.1) und behalten Sie dabei eine Zelldichte von 0,5 × 106 Zellen pro 50 μl pro Röhrchen für die ungefärbten und DAPI-Röhrchen bei.
- Bereiten Sie die einfach gefärbten Röhrchen für die Kompensation vor, wie in Tabelle 4 beschrieben.
- Jedes Röhrchen nach der Zubereitung vorsichtig vortexen und 30 Minuten im Dunkeln inkubieren.
- Nach der Inkubation werden zwei Wäschen durchgeführt, indem 1 ml Färbepuffer in jedes Röhrchen gegeben werden, gefolgt von einem kurzen Wirbel und einer Zentrifugation bei 200 g für 10 Minuten bei Raumtemperatur.
- Verwerfen Sie den Überstand, resuspendieren Sie das Pellet in 500 μl Färbepuffer und legen Sie es bis zur Aufnahme beiseite.
- Führen Sie für das DAPI-Röhrchen eine Hitzeschockbehandlung durch, indem Sie es 5 Minuten lang in einem 60 °C warmen Wasserbad und anschließend 15 Minuten auf Eis inkubieren. Geben Sie 5 μl DAPI in die Aufhängung und lassen Sie sie bis zur Lauferfassung im Dunkeln.
- Herstellung von Fluoreszenz-Minus-Eins-Kontrollen (FMO)
- Resuspendieren Sie das fertige Pellet in Färbepuffer (siehe Schritt 4.1) und behalten Sie dabei eine Zelldichte von 0,5 × 106 Zellen pro 50 μl für jedes Röhrchen bei.
- Nehmen Sie fünf FACS-Röhrchen und kennzeichnen Sie sie als CD44-V450-Isotyp, CD90-FITC, CD45-PE, CD73-PerCP Cy5.5-Isotyp und CD105-APC-Isotyp.
- Bereiten Sie die Zell- und Antikörpersuspension gemäß Tabelle 5 vor.
- Die Röhrchen vorsichtig vortexen und 30 min bei Raumtemperatur im Dunkeln inkubieren.
- Nach der Inkubation werden zwei Wäschen mit 1 ml Färbepuffer in jedes Röhrchen gegeben, gefolgt von kurzem Wirbeln und Zentrifugieren bei 200 g für 10 Minuten bei Raumtemperatur.
- Verwerfen Sie den Überstand, resuspendieren Sie das Pellet in 500 μl Färbepuffer und legen Sie es bis zur Aufnahme beiseite.
- Vorbereitung der Proben für die Analyse
- Resuspendieren Sie das fertige Pellet im Färbepuffer (siehe Schritt 4.1) und behalten Sie dabei eine Zelldichte von 0,5 × 106 Zellen pro 50 μl für jedes Röhrchen bei.
- Nehmen Sie zwei FACS-Röhrchen und beschriften Sie sie als gemischte Röhrchen 1 und 2.
- Bereiten Sie die Zell- und Antikörpersuspension gemäß Tabelle 6 vor.
- Die Röhrchen vorsichtig vortexen und 30 min bei Raumtemperatur im Dunkeln inkubieren.
- Nach der Inkubation werden zwei Wäschen mit 1 ml Färbepuffer in jedes Röhrchen gegeben, gefolgt von kurzem Wirbeln und Zentrifugieren bei 200 g für 10 Minuten bei Raumtemperatur.
- Verwerfen Sie den Überstand, resuspendieren Sie das Pellet in 500 μl Färbepuffer und legen Sie es bis zur Akquisition beiseite.
- Vorbereitung von Proben für die Einzelzellsortierung
- Nehmen Sie zwei FACS-Röhrchen, fügen Sie 1 ml FBS hinzu und rollen Sie das Röhrchen herum, bis sich auf der Innenseite jedes Röhrchens eine gleichmäßige FBS-Schicht bildet. Inkubieren Sie dies für 1-2 Stunden, während Sie die Probe für die Färbung verarbeiten. Beschriften Sie diese Röhrchen als gemischte Röhrchen 1 und 2.
- Das fertige Pellet wird in den Färbepuffer (siehe Schritt 4.1) resuspendiert, wobei eine Zelldichte von 2-3 × 106 Zellen pro 50 μl für jedes Röhrchen beibehalten wird.
- Bereiten Sie die Zell- und Antikörpersuspension in gemischten Röhrchen 1 und 2 gemäß Tabelle 7 vor.
- Die Röhrchen vorsichtig vortexen und 30 min bei Raumtemperatur im Dunkeln inkubieren.
- Nach der Inkubation werden zwei Wäschen mit 1 ml Färbepuffer in jedes Röhrchen gegeben, gefolgt von kurzem Wirbeln und Zentrifugieren bei 200 g für 10 Minuten bei Raumtemperatur.
- Verwerfen Sie den Überstand, resuspendieren Sie das Pellet in 500 μl Färbepuffer und stellen Sie es beiseite. 5 μl DAPI 15 Minuten vor dem Sortieren hinzufügen.
HINWEIS: Beachten Sie, dass für Sortierexperimente eine höhere Zelldichte pro Röhrchen empfohlen wird.
| Röhren-Typ | Positive Comp Perlen* | Negative Comp Perlen* | Zellen | Antikörper hinzugefügt | |
| FITC-Schlauch | 1 Tropfen | 1 Tropfen | – | Anti-Human-CD90-FITC (2 μl) | |
| V450 Röhre | 1 Tropfen | 1 Tropfen | – | Anti-Human-CD44-V450 (2 μl) | |
| PerCP-Cy 5.5 Röhre | 1 Tropfen | 1 Tropfen | – | Anti-humanes CD73-PerCP Cy 5,5 (2 μl) | |
| PE-Rohr | 1 Tropfen | 1 Tropfen | – | Anti-humanes CD45-PE (2 μl) | |
| APC-Röhre | 1 Tropfen | 1 Tropfen | – | Anti-Human-CD105-APC (2 μl) | |
| DAPI-Röhre | – | – | 50 μl | – | |
| Ungefärbte Röhre | – | – | 50 μl | – | |
| *1 Tropfen = 60 μl Kügelchensuspension | |||||
Tabelle 4: Kompensationskontrollproben. Abkürzungen: Comp = Kompensation; DAPI = 4',6-Diamidino-2-phenylindol; FITC = Fluoresceinisothiocyanat; APC = Allophycocyanin; PE = Phycoerythrin; PerCP = Peridin-Chlorophyll-Protein.
| RÖHRCHEN-TYP | ANTIKÖRPER GEGEN POSITIVEN MARKER (2 μL) | ANTIKÖRPER GEGEN NEGATIVEN MARKER (2 μL) | HINZUGEFÜGTE ISOTYP-ANTIKÖRPER (2 μL) | GESAMTVOLUMEN DER ANTIKÖRPER | VOLUMEN DER ZUGEGEBENEN ZELLSUSPENSION | VOLUMEN DES ZUGEGEBENEN FÄRBEPUFFS | ||
| CD90-FITC FMO-Röhre | - Anti-Human-CD44-V450 | Anti-Human-CD45-PE | FITC IgG1 Isotyp | 10 μl | 50 μl | 40 μl | ||
| - Anti-Human-CD73 PerCP Cy 5.5 | ||||||||
| - Anti-humanes CD105-APC | ||||||||
| CD73-PerCP Cy5.5 FMO-Röhre | - Anti-Human-CD44-V450 | Anti-Human-CD45-PE | PerCP Cy 5.5 IgG1 Isotyp | 10 μl | 50 μl | 40 μl | ||
| - Anti-Human-CD90-FITC | ||||||||
| - Anti-humanes CD105-APC | ||||||||
| CD44-V450 FMO-Röhre | - Anti-Human-CD90-FITC | Anti-Human-CD45-PE | V450 IgG1 Isotyp | 10 μl | 50 μl | 40 μl | ||
| - Anti-Human-CD73-PerCP Cy 5.5 | ||||||||
| - Anti-humanes CD105-APC | ||||||||
| CD105-APC FMO-Röhre | - Anti-Human-CD44-V450 | Anti-Human-CD45-PE | APC IgG1 Isotyp | 10 μl | 50 μl | 40 μl | ||
| - Anti-Human-CD90-FITC | ||||||||
| - Anti-Human-CD73-PerCP Cy 5.5 | ||||||||
| CD45-PE FMO-Röhrchen | - Anti-Human-CD44-V450 | - | PE IgG1 Isotyp | 10 μl | 50 μl | 40 μl | ||
| - Anti-Human-CD90-FITC | ||||||||
| - Anti-Human-CD73-PerCP Cy 5.5 | ||||||||
| - Anti-humanes CD105-APC | ||||||||
Tabelle 5: FMO-Kontrollproben. Abkürzungen: FMO = Fluoreszenz minus eins; FITC = Fluoresceinisothiocyanat; APC = Allophycocyanin; PE = Phycoerythrin; PerCP = Peridin-Chlorophyll-Protein.
| RÖHRCHEN-TYP | ANTIKÖRPER GEGEN POSITIVEN MARKER (2 μL) | ANTIKÖRPER GEGEN NEGATIVEN MARKER (2 μL) | GESAMTVOLUMEN DER ANTIKÖRPER | VOLUMEN DER ZUGEGEBENEN ZELLSUSPENSION | VOLUMEN DES ZUGEGEBENEN FÄRBEPUFFS | |
| Gemischte Röhre 1 | - Anti-Human-CD44-V450 | Anti-Human-CD45-PE | 10 μl | 50 μl | 40 μl | |
| - Anti-Human-CD90-FITC | ||||||
| - Anti-humanes CD73-PerCP Cy5.5 | ||||||
| - Anti-humanes CD105-APC | ||||||
| Gemischte Röhre 2 | - Anti-Human-CD44-V450 | Anti-Human-CD45-PE | 10 μl | 50 μl | 40 μl | |
| - Anti-Human-CD90-FITC | ||||||
| - Anti-humanes CD73-PerCP Cy5.5 | ||||||
| - Anti-humanes CD105-APC | ||||||
Tabelle 6: Probenröhrchen für die mehrfarbige Immunphänotypisierung von SHEDs. Abkürzungen: SHEDs = Stammzellen aus menschlichen abgeblätterten Milchzähnen; PE = Phycoerythrin.
| RÖHRCHEN-TYP | ANTIKÖRPER GEGEN POSITIVEN MARKER (3 μL) | ANTIKÖRPER GEGEN NEGATIVEN MARKER (3 μL) | GESAMTVOLUMEN DER ANTIKÖRPER | VOLUMEN DER ZUGEGEBENEN ZELLSUSPENSION | VOLUMEN DES HINZUGEFÜGTEN FLECKENPUFFERS | |
| Gemischte Röhre 1 | - Anti-Human-CD90-FITC | Anti-Human-CD45-PE | 9 μl | 50 μl | 41 μl | |
| - Anti-humanes CD73-PerCP Cy5.5 | ||||||
| Gemischte Röhre 2 | - Anti-Human-CD90-FITC | Anti-Human-CD45-PE | 9 μl | 50 μl | 41 μl | |
| - Anti-humanes CD73-PerCP Cy5.5 | ||||||
Tabelle 7: Einzelzell-Sortierreaktionsröhrchen. Abkürzungen: FITC = Fluoresceinisothiocyanat; PE = Phycoerythrin; PerCP = Peridin-Chlorophyll-Protein.
5. Sortierung einzelner Zellen
- Vorbereitung des Zellsortierers
- Installieren Sie eine 100-μm-Düse im Sortierer.
HINWEIS: Die geeignete Düse hat mindestens das Fünffache des Durchmessers des zu sortierenden Partikels. Die für die Sortierung zu verwendende Mantelflüssigkeit muss auf der Grundlage des Probentyps und der Empfindlichkeit des Experiments entschieden werden. Für dieses Experiment wurde proprietäre Mantelflüssigkeit verwendet. - Führen Sie die tägliche Qualitätsprüfung (QC) des Instruments durch und richten Sie den Sortierer für das Experiment ein. Im Gerätehandbuch finden Sie eine detaillierte Anleitung zur Geräteeinrichtung.
- Installieren Sie eine 100-μm-Düse im Sortierer.
- Einrichten der Vergütungsmatrix
- Stellen Sie die Kompensationsmatrix mit Hilfe von Einzelflecken-Kompensationsröhrchen aus Schritt 4.2 ein.
- Wählen Sie in der proprietären Software in der Symbolleiste Experiment aus und klicken Sie auf Kompensations-Setup. Öffnen Sie Steuerelemente für die Kompensation erstellen.
- Überprüfen Sie die Markierungen und bestätigen Sie. Es wird eine neue Probe mit dem Namen "Kompensation" hinzugefügt, unter der neue Röhrchen, die als Marker-Steuerelemente bezeichnet werden, automatisch von der Software hinzugefügt werden.
- Wählen Sie die ungefärbte Röhre aus, und führen Sie sie aus, um 5.000 Ereignisse aufzuzeichnen. Ziehen Sie das Gatter auf die Grundgesamtheit der Zellen und wenden Sie es auf alle Kompensationssteuerelemente an. Damit werden die Spannungen und das negative Gate für jeden Fluoreszenzparameter eingestellt.
- Legen Sie auf ähnliche Weise die Röhrchen zur Korrektur einzelner Flecken separat ein und zeichnen Sie die Daten auf und speichern Sie sie. Wählen Sie das Gate aus, das die Grundgesamtheit von Interesse abgrenzt, und wenden Sie es auf alle Kompensationskontrollen an. Damit werden die positiven Gates für jeden Fluoreszenzparameter eingestellt.
- Wählen Sie in der Symbolleiste Experiment aus und klicken Sie auf Kompensationswerte berechnen | verknüpfen und speichern.
HINWEIS: Sobald die Auto-Comp-Matrix mit der Software generiert wurde, können die Spannungsparameter der Fluorochrome für keinen der Kanäle in den gemischten Röhren geändert werden.
- Datenerfassung
- Zeichnen Sie 10.000 Zellen in jedem Röhrchen aus Schritt 4.3 auf. und 4.4 zur Erhebung von Daten zur Analyse des Immunphänotyps der Zellen.
- Vorbereitung der Auffangvorrichtungen
- Je nach Verwendungszweck der sortierten Zellpopulationen wählen Sie zwischen 6-Well-, 24-Well-, 48-Well- oder 96-Well-Platten.
- Beschichten Sie die Vertiefungen mit 200-500 μl FBS und lassen Sie die Platten 2 h lang ungestört.
- Entfernen Sie nach 2 h das restliche FBS und fügen Sie 200-500 μl 10%ige MSC-Nährmedien hinzu.
- Zellensortierung im Einzelzellen-Sortiermodus
- Führen Sie Mixed Tube 1 (ab Schritt 4.5) aus, und zeichnen Sie 10.000 Ereignisse auf, um die Gatter für die zu sortierende Grundgesamtheit mit der entsprechenden Gating-Strategie festzulegen.
- Laden Sie eine Sammelplatte, stellen Sie die Anzahl der Zielzellen zwischen 2.500 und 5.000 Zellen/Well ein und wählen Sie die Reinheitsmaske für die Einzelzellsortierung aus.
- Sammeln Sie die sortierten Populationen in der Sammelvorrichtung und lassen Sie sie bis zum Ende des Sortierexperiments auf Eis.
- Sobald dies erledigt ist, werden die Platten in den 5% CO2 -Inkubator überführt, um die Kulturen bei 37 °C zu halten.
HINWEIS: Nach der Erfassung wurden die Rohdatendateien im .fcs-Dateiformat exportiert (ab Version 3.0). Die Sortierberichte, die nach jedem Experiment erstellt wurden, zeichneten die Anzahl der Ereignisse/Zellen auf, die nach zugewiesenen Vertiefungen sortiert waren, und gaben die Anzahl der Konflikte an, die abgebrochen wurden.
Ergebnisse
Die SHEDs wurden mit Standard-Immunfluoreszenzassays charakterisiert, die die Expression von Vimentin (rot, Typ-III-Intermediärfilamente), Aktinfilamenten (Alexa fluor 488 Phalloidin-Sonden) und mit DAPI gefärbten Zellkernen zeigten (Abbildung 1A). Um ihre proliferativen und koloniebildenden Fähigkeiten abzuschätzen, wurden Standard-Kurzzeit-Zellwachstumsassays durchgeführt. Ein 14,3-facher Anstieg der Proliferationsrate von Tag 2 bis Tag 8 ist in Abbildung 1B
Diskussion
Auf dem Gebiet des Tissue Engineering und der regenerativen Medizin haben unter den postnatalen Quellen aus oralem Gewebe gewonnene MSCs aufgrund ihrer minimalen ethischen Verpflichtungen und ihres bemerkenswerten Multilineage-Differenzierungspotenzials großes Interesse geweckt21. Zahnpulpa-Stammzellen (DPSCs) aus dem betroffenen dritten Molaren und SHEDs haben aufgrund ihres therapeutischen Potenzials bei neurodegenerativen und traumatischen Erkrankungen die größte Aufmerksamkeit unter dentale...
Offenlegungen
Die Autoren erklären, dass es keinen Interessenkonflikt in Bezug auf die Veröffentlichung dieses Papiers gibt.
Danksagungen
Wir danken der Flow Cell Facility am Jawaharlal Nehru Centre for Advanced Scientific Research, Bengaluru, Indien, für die Nutzung der Core Facility für Durchflusszytometrie. Der Kryoschnitt der Pelletkultur differenzierter Zellen wurde am Neuberg Anand Reference Laboratory, Bengaluru, Indien, durchgeführt. Diese Arbeit wurde durch die interne Finanzierung der UC durch die Manipal Academy of Higher Education (MAHE), Indien, unterstützt. AG bedankt sich für die Unterstützung durch das Dr. T. M. A. Pai Stipendium von MAHE.
Materialien
| Name | Company | Catalog Number | Comments |
| Alcian Blue Stain | HiMedia | CCK029-1KT | |
| Antibiotic-Antimycotic (100x) | Gibco by ThermoFisher | 15240062 | |
| BD CompBead Plus Anti-Mouse Ig, κ/Negative Control (BSA) Compensation Plus (7.5 µm) Particles Set | BD Biosciences | 560497 | |
| BD FACS Accudrop Beads | BD Biosciences | 345249 | Used to set up the Laser delay when the sort module opens. |
| BD FACS Aria Fusion Flow cytometer | BD Biosciences | --- | |
| BD FACS Diva 9.4 | BD Biosciences | --- | |
| BD FACS Sheath Fluid | BD Biosciences | 342003 | Used as sheath fluid for both analysis and sorting experiments in the BD FACSAria Fusion |
| BD FACSDiva CS&T Research Beads | BD Biosciences | 655050 | Used for Instrument configuration depending on the nozzle size. |
| BD Horizon V450 Mouse Anti-Human CD44 | BD Biosciences | 561292 | |
| BD Horizon V450 Mouse IgG2b, κ Isotype Control | BD Biosciences | 560374 | CD44-V450 isotype |
| BD Pharmingen APC Mouse Anti-Human CD105 | BD Biosciences | 562408 | |
| BD Pharmingen APC Mouse IgG1, κ Isotype Control | BD Biosciences | 555751 | CD105-APC isotype |
| BD Pharmingen DAPI Solution | BD Biosciences | 564907 | DAPI Stock solution of 1 mg/mL |
| BD Pharmingen FITC Mouse Anti-Human CD90 | BD Biosciences | 555595 | |
| BD Pharmingen FITC Mouse IgG1, κ Isotype Control | BD Biosciences | 555748 | CD90-FITC isotype |
| BD Pharmingen PE Mouse Anti-Human CD45 | BD Biosciences | 555483 | |
| BD Pharmingen PE Mouse IgG1, κ Isotype Control | BD Biosciences | 555749 | CD45-PE isotype |
| BD Pharmingen PerCP-Cy 5.5 Mouse Anti-Human CD73 | BD Biosciences | 561260 | |
| BD Pharmingen PerCP-Cy 5.5 Mouse IgG1, κ Isotype Control | BD Biosciences | 550795 | CD73-PerCP-Cy 5.5 isotype |
| BD Pharmingen Purified Mouse Anti-Vimentin | BD Biosciences | 550513 | |
| Bovine serum albumin | Hi-Media | TC548-5G | |
| Crystal violet | Nice chemical pvt ltd | C33809 | |
| Dulbecco's Phosphate Buffered Saline | Sigma-aldrich | D5652-50L | dPBS used for culture work and maintenance. |
| Ethanol | --- | --- | Used for general sterlization. |
| Fetal Bovine Serum | Gibco by ThermoFisher | 10270-106 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 555 | ThermoFisher Scientific | A-21422 | |
| KO-DMEM | Gibco by ThermoFisher | 10829018 | Basal medium for undifferentiated hESCs, used in the preparation of culture media |
| L-Glutamine 200mM (100x) | Gibco by ThermoFisher | 25030-081 | |
| Methanol, for Molecular Biology | Hi-Media | MB113 | |
| Oil red O | HiMedia | CCK013-1KT | |
| Paraformaldehyde | loba chemie | 30525-89-4 | |
| Penicillin Streptomycin (100x) | Gibco by ThermoFisher | 15140- 122 | |
| Phalloidin (ActinGreen 488 ReadyProbes reagent) | Invitrogen | R37110 | |
| Silver Nitrate | HiMedia | MB156-25G | |
| Sodium Thiosulphate pentahydrate | Chemport | 10102-17-7 | |
| Sphero Rainbow Fluorescent Particles, 3.0 - 3.4 µm | BD Biosciences | 556291 | |
| Staining buffer | Prepared in MIRM | ---- | It was prepared using 2% FBS in PBS |
| StemPro Adipogenesis Differentiation Basal Media | Gibco by ThermoFisher | A10410-01 | Basal media for Adipogenic media |
| StemPro Adipogenesis Supplement | Gibco by ThermoFisher | A10065-01 | Induction media for Adipogenic media |
| StemPro Chondrogenesis Supplement | Gibco by ThermoFisher | A10064-01 | Induction media for Chondrogenic media |
| StemPro Osteogenesis Supplement | Gibco by ThermoFisher | A10066-01 | Induction media for Osteoogenic media |
| StemPro Osteogenesis/Chondrogenesis Differentiation Basal Media | Gibco by ThermoFisher | A10069-01 | Basal media for both Ostegenic and Chondrogenic media |
| Triton-X-100 | Hi-Media | MB031 | |
| Trypan Blue | Gibco by life technologies | 15250-061 | |
| Trypsin - EDTA Solution 1x | Hi-media | TCL049 | |
| Tween-20 | MERCK | 9005-64-5 |
Referenzen
- Kobolak, J., Dinnyes, A., Memic, A., Khademhosseini, A., Mobasheri, A. Mesenchymal stem cells: Identification, phenotypic characterization, biological properties and potential for regenerative medicine through biomaterial micro-engineering of their niche. Methods. 99, 62-68 (2016).
- Wilson, A., Hodgson-Garms, M., Frith, J. E., Genever, P. Multiplicity of mesenchymal stromal cells: finding the right route to therapy. Frontiers in Immunology. 10, 1112 (2019).
- Li, J., et al. Comparison of the biological characteristics of human mesenchymal stem cells derived from exfoliated deciduous teeth, bone marrow, gingival tissue, and umbilical cord. Molecular Medicine Reports. 18 (6), 4969-4977 (2018).
- McLeod, C. M., Mauck, R. L. On the origin and impact of mesenchymal stem cell heterogeneity: new insights and emerging tools for single cell analysis. European Cells & Materials. 34, 217-231 (2017).
- Dominici, M., et al. Minimal criteria for defining multipotent mesenchymal stromal cells. The International Society for Cellular Therapy position statement. Cytotherapy. 8 (4), 315-317 (2006).
- Wang, J., Liao, L., Wang, S., Tan, J. Cell therapy with autologous mesenchymal stem cells-how the disease process impacts clinical considerations. Cytotherapy. 15 (8), 893-904 (2013).
- Kern, S., Eichler, H., Stoeve, J., Kluter, H., Bieback, K. Comparative analysis of mesenchymal stem cells from bone marrow, umbilical cord blood, or adipose tissue. Stem Cells. 24 (5), 1294-1301 (2006).
- Dunn, C. M., Kameishi, S., Grainger, D. W., Okano, T. Strategies to address mesenchymal stem/stromal cell heterogeneity in immunomodulatory profiles to improve cell-based therapies. Acta Biomaterialia. 133, 114-125 (2021).
- Yang, Y. K., Ogando, C. R., Wang See, C., Chang, T. Y., Barabino, G. A. Changes in phenotype and differentiation potential of human mesenchymal stem cells aging in vitro. Stem Cell Research & Therapy. 9 (1), 131 (2018).
- Costa, L. A., et al. Functional heterogeneity of mesenchymal stem cells from natural niches to culture conditions: implications for further clinical uses. Cellular and Molecular Life Sciences. 78 (2), 447-467 (2021).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Review of Scientific Instruments. 43 (3), 404-409 (1972).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
- Liao, X., Makris, M., Luo, X. M. Fluorescence-activated cell sorting for purification of plasmacytoid dendritic cells from the mouse bone marrow. Journal of Visualized Experiments. (117), (2016).
- Roda, B., et al. A novel stem cell tag-less sorting method. Stem Cell Reviews and Reports. 5 (4), 420-427 (2009).
- Hall, S. R., et al. Identification and isolation of small CD44-negative mesenchymal stem/progenitor cells from human bone marrow using elutriation and polychromatic flow cytometry. Stem Cells Translational Medicine. 2 (8), 567-578 (2013).
- Eggleton, M. J., Sharp, A. A. Platelet counting using the Coulter electronic counter. Journal of Clinical Pathology. 16 (2), 164-167 (1963).
- Porwit-Ksiazek, A., Aman, P., Ksiazek, T., Biberfeld, P. Leu 7+ (HNK-1+) cells. II. Characterization of blood Leu 7+ cells with respect to immunophenotype and cell density. Scandinavian Journal of Immunology. 18 (6), 495-449 (1983).
- Hewitt, Z., et al. Fluorescence-activated single cell sorting of human embryonic stem cells. Cloning and Stem Cells. 8 (3), 225-234 (2006).
- Singh, A. M. An efficient protocol for single-cell cloning human pluripotent stem cells. Frontiers in Cell and Developmental Biology. 7, 11 (2019).
- Wilson, N. K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Zhou, L. L., et al. Oral mesenchymal stem/progenitor cells: the immunomodulatory masters. Stem Cells Inernational. 2020, 1327405 (2020).
- Fawzy El-Sayed, K. M., et al. Adult mesenchymal stem cells explored in the dental field. Advances in Biochemical Engineering/Biotechnology. 130, 89-103 (2013).
- Hardy, W. R., et al. Transcriptional networks in single perivascular cells sorted from human adipose tissue reveal a hierarchy of mesenchymal stem cells. Stem Cells. 35 (5), 1273-1289 (2017).
- . . FACSAria Fusion User's Guide. , (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten